A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تعيين بروتوكول لاستخدام الجينات إثراء التحليل لتحديد نموذج الحيوان المناسب للبحوث متعدية
In This Article
Summary
نحن نقدم على بروتوكول موحد لاستخدام الجينات إثراء مجموعة تحليل البيانات ترانسكريبتوميك لتحديد طراز ماوس مثالية للبحوث متعدية الجنسيات.
هذا البروتوكول يمكن استخدامها مع دنا وبيانات تسلسل الحمض النووي الريبي، ويمكن تمديد مرة أخرى للبيانات اوميكس الأخرى إذا كانت البيانات متوفرة.
Abstract
الدراسات التي أجريت مؤخرا أنه بالمقارنة مع مجموعات البيانات ترانسكريبتوميك من الأمراض التي تصيب الإنسان مع مجموعات البيانات من نماذج الماوس باستخدام التقنيات التقليدية الجينات بالجينات مقارنة أدت إلى استنتاجات متناقضة فيما يتعلق بالأهمية نماذج حيوانية متعدية البحث. سببا رئيسيا للتباين بين الجينات المختلفة تعبير تحاليل هو تصفية التعسفي الأثران عن الجينات. وعلاوة على ذلك، يقتصر مقارنة الجينات واحد بين الأنواع المختلفة ومنصات غالباً ما الفرق الفنية، مما يؤدي إلى سوء فهم يخدع/الخلافات بين البيانات من النماذج البشرية والحيوانية. وهكذا، يلزم اتباع نهج موحد لتحليل البيانات بصورة منتظمة. للتغلب على تصفية ذاتية الجينات ومقارنات الجينات بالجينات غير فعالة، أظهرنا مؤخرا أن تحليل إثراء مجموعة الجينات (جسيا) لديه القدرة على تجنب هذه المشاكل. ولذلك، قمنا بتطوير بروتوكول موحد لاستخدام جسيا للتمييز بين النماذج الحيوانية الملائمة وغير الملائمة للبحوث متعدية الجنسيات. هذا البروتوكول غير مناسب للتنبؤ بكيفية تصميم نظم جديدة لنموذج بداهة–، كما أنه يتطلب بيانات اوميكس التجريبية الحالية. ومع ذلك، يصف البروتوكول كيفية تفسير البيانات الموجودة بطريقة موحدة بغية تحديد أنسب نموذج الحيوان، وبالتالي تجنب التجارب على الحيوانات لا لزوم لها ومضللة الدراسات متعدية الجنسيات.
Introduction
نماذج حيوانية تستخدم على نطاق واسع لدراسة الأمراض البشرية، بسبب ما تشابه المفترضة للبشر فيما يتعلق بعلم الوراثة، وعلم التشريح وعلم وظائف الأعضاء. وعلاوة على ذلك، نماذج حيوانية غالباً ما تعمل كحراس للعلاج السريري ويمكن أن يكون لها تأثير كبير على نجاح البحوث متعدية الجنسيات. الاختيار الدقيق للنموذج الحيواني الأمثل يمكن أن تقلل من العدد الدراسات الحيوانية مضللة. في الآونة الأخيرة، أهمية نماذج حيوانية للبحوث متعدية مثير للجدل نوقشت، خاصة وأن تحليل مجموعات البيانات نفسها التي تم الحصول عليها من أمراض التهابات البشرية ونماذج الماوس المتصلة بها أدت إلى نتائج متناقضة 1،2. وكشفت هذه المناقشة مشكلة أساسية أثناء تحليل البيانات اوميكس: هناك حاجة إلى اتباع نهج موحد لتحليل البيانات بصورة منتظمة بغية الحد من اختيار الجينات متحيزة وزيادة متانة المقارنات فيما بين 3.
تقليديا، تحليل البيانات ترانسكريبتوميكس (وغيرها من البيانات اوميكس) يتم على مستوى واحد--جين ويشمل خطوة أولية لتحديد الجينات على أساس معايير صارمة وقف إنتاج المواد الانشطارية (مثلاً، تغيير إضعاف > قيمة p 2.0، < 0.05). ومع ذلك، الإعداد معلمات الأولى وقف إنتاج المواد الانشطارية غالباً ذاتية والتعسفي وغير المبرر بيولوجيا، ويمكن أن يؤدي حتى إلى عكس النتائج1،2. وعلاوة على ذلك، اختيار الجينات الأولى عموما تقييد التحليل إلى عدد قليل جداً حتى-دوونريجولاتيد الجينات وهكذا ليست حساسة بما يكفي ليشمل غالبية الجينات التي تم الإعراب عنها متفاوتاً إلى حد أقل.
مع ظهور عصر الجينوم في وقت مبكر 2000s، والمعرفة المتزايدة للممرات البيولوجية والسياقات، وضعت النهج الإحصائية البديلة التي سمحت للتحايل على القيود المفروضة على تحليل مستوى واحد--جين. مجموعة الجينات إثراء التحليل (جسيا)4، واحد أساليب مقبولة على نطاق واسع لتحليل البيانات ترانسكريبتوميكس، يجعل استخدام بداهة تعريف مجموعات من الجينات (مثلاً، مما يشير إلى مسارات، موقع الدانية على كروموسوم إلخ.). أولاً خرائط جسيا جميع الجينات غير المصفى تم الكشف عنها إلى مجموعات المورثات المقصودة (مثلاً، مسارات)، بصرف النظر عن تغير الفردية في التعبير. ويشمل هذا النهج وبالتالي أيضا تنظيم معتدل الجينات التي لولا ذلك فقدت بتحليل مستوى واحد--جين. بعد ذلك يتم التغيير المضافة في التعبير داخل مجموعات الجينات استخدام إحصاءات مجموع قيد التشغيل.
وعلى الرغم من استخدامه على نطاق واسع في البحوث الطبية، جسيا والنهج المتصلة إثراء مجموعة لا طبيعة الحال تؤخذ في الاعتبار لتحليل البيانات المعقدة اوميكس. هنا، يمكننا وصف بروتوكول لمقارنة البيانات اوميكس من العينات البشرية مع تلك من نماذج الماوس لتحديد النموذج المثالي للدراسات متعدية الجنسيات. علينا أن نظهر مدى انطباق البروتوكول استناداً إلى مجموعة من نماذج الماوس التي يتم استخدامها لمحاكاة اضطرابات التحريضية البشرية. ومع ذلك، أنابيب تحليل هذا لا يقتصر على المقارنات بين الإنسان والفأر وأسئلة تنظيمي لمواصلة البحث.
Protocol
1-تحميل "البرنامج جسيا" و "قاعدة بيانات التواقيع الجزيئية"
- الذهاب إلى موقع معهد واسع جسيا الرسمية (http://software.broadinstitute.org/gsea/index.jsp) وقم بالتسجيل للحصول على حق الوصول إلى برنامج جسيا أداة و "قاعدة بيانات التواقيع الجزيئية" (مسيجدب)-
- تحميل تطبيقات سطح المكتب جافاجسيا أو خيار برامج بديلة (مثلاً، برنامج نصي R)-
ملاحظة: جميع خيارات تنفيذ نفس خوارزمية. البرنامج جسيا متاحة بحرية للأفراد في الأوساط الأكاديمية والصناعة لأغراض البحوث الداخلية- - لمزيد التفاصيل عن البرنامج جسيا، انتقل إلى الوثائق في موقع على شبكة الإنترنت (http://software.broadinstitute.org/cancer/software/gsea/wiki/index.php/Main_Page)، ودليل المستخدم جسيا (http://software.broadinstitute.org/gsea/doc/ GSEAUserGuideFrame.html)-
- تحميل "قاعدة بيانات التواقيع الجزيئية" (مسيجدب) من موقع جسيا للحصول على الوصول إلى مجموعات مجموعة الجينات الفردية-
ملاحظة: مسيجدب مجموعة من مجموعات الجينات المشروح للاستخدام مع برامج جسيا أو لأغراض أخرى. ويمكن تقسيم مجموعات الجينات وفقا لإشارات مسارات ومصطلحات علم الوجود الجينات، زخارف رابطة الدول المستقلة التنظيمية، والتوقيعات التجريبية والآخرين. تتم تسمية الجينات من مسيجدب دائماً على رمز الجين هوغو (منظمة المجين البشري) الرسمية. للمقارنة بين تنظيم المسار بين المعطى اضطراب البشرية والماوس المختلفة نماذج من المستحسن تحميل ' جميع مسارات المتعارف عليه، ورموز الجينات ' الملف (c2.cp.v5.2.symbols.gmt). هذا الملف يضم مجموعات الجينات المشروح، ونظمتها في مسارات إشارات كج 5 ، 6 و 7 ، رياكتومي 8 وبيوكارتا 9-السلسلة ' v5.2 ' يمثل معلومات الإصدار للمجموعة. تأكد من تحميل أحدث إصدار من الملفات. مسيجدب متاحة بحرية للأفراد في الأوساط الأكاديمية والصناعة لأغراض البحوث الداخلية. لا حاجة لتحميل مسيجدب، إذا كان الاتصال بإنترنت يتم توفيرها من خلال التحليل. وفي هذه الحالة مسيجدب يمكن أن يكون اختيار مباشرة داخل واجهة المستخدم جسيا.
رقاقة - "الحمض النووي تحميل" ملفات التعليقات التوضيحية (الصفيف) من موقع جسيا لترجمة المعرفات التحقيق الخاصة بمجموعة عامة رموز الجينات هوغو (مثلاً، Mouse430_2.chip).
ملاحظة: لا حاجة لتحميل شروح رقاقة الحمض النووي، إذا لم يتم توفير اتصال إنترنت أثناء التحليل. وفي هذه الحالة يمكن أن يكون اختيار شروح رقاقة الحمض النووي مباشرة داخل واجهة المستخدم جسيا. يمكن أيضا استخدام البروتوكول مع بيانات تسلسل الحمض النووي الريبي. وفي هذه الحالة، لا حاجة لتنزيل ملفات الشرح. بدلاً من ذلك، استخدم أداة جسيا بريرانكيد لتحليل البيانات التعبير الجيني (راجع الخطوة 4، 12)-
2. تنزيل "البيانات التجريبية في التعبير الجيني" لاضطراب البشرية والنماذج الحيوانية المناسبة
- تحديد الجينات التجريبية التعبير (transcriptomics) دراسات للاضطراب البشرية الاختيار (مثلاً، ملامح التعبير الجيني من الكريات البيضاء المستمدة من المرضى الذين يعانون من اضطراب التعفين، GSE9960)-
- المثل، البحث عن الحيوان عدة نماذج التي من المفترض أن تكون المقارنة مع الدراسات الإنسانية (مثلاً، التشكيلات الجانبية للتعبير الجيني من خلايا الدم المستمدة من الفئران بعد الحقن المكوّرات العنقودية الذهبية (S. aureus)، GSE20524). في هذه الخطوة استخدام المعرفة السابقة للاختيار الأولى للنماذج الحيوانية التي قد تكون مناسبة لمحاكاة الوضع البشري-
- لهذا الغرض الإشارة إلى الأدب وقواعد البيانات مثل الجامع التعبير الجيني (GEO) قاعدة البيانات 10 أو 11 من أراييكسبريس وتنزيل البيانات ترانسكريبتوميكس طبيعية للفائدة. حفظ البيانات كملفات نصية على القرص الثابت المحلي. لقاعدة بيانات توقعات البيئة العالمية، ينصح بتحميل الملفات النصية مصفوفة سلسلة المفصول. كما تحيط علما بمنهاج العمل (نوع الصفيف) المستخدمة لهذه الدراسة، حيث أن هذه المعلومات ضرورية لترجمة المعرفات التحقيق الخاصة بالمصفوفة إلى رموز الجينات هوغو عام.
ملاحظة: تأكد من ذاكرة كافية لتخزين البيانات، كما تضم مجموعات البيانات transcriptomics عادة عدة مئات ميغابايت-
3. بيانات معالجة وتنسيق
- قبل استيراد البيانات التعبير الجيني التجريبية إلى أداة البرنامج جسيا، النظر في بنية البيانات المطلوبة. لكل دراسة يدوياً إنشاء الملفين مختلفة: 1) ملف بيانات تعبير جينات التي تحتوي على قيم القياس للجينات وعينات مختلفة، و 2) ملف النمط الظاهري الذي يحتوي على تسميات عينة لتجميع عينات فردية (مثلاً، إلى مجموعات العلاج).
لمزيد التفاصيل والبيانات هيكل خيارات انتقل إلى صفحة تنسيق البيانات جسيا (http://software.broadinstitute.org/cancer/software/gsea/wiki/index.php/Data_formats)-
ملاحظة: عموما، جميع أشكال البيانات ترانسكريبتوميكس غير متوافق مع البروتوكول، بما في ذلك تجارب ميكرواري الحمض النووي والحمض النووي الريبي seq أو الدراسات seq رقاقة. في حالة استخدام التجارب ميكرواري الحمض النووي، ينبغي أن يتضمن ملف البيانات التعبير الجيني المعرف التحقيق الخاصة بالمصفوفة أو هوغو رموز الجينات لكل الجينات (المسبار معرفات سوف تترجم إلى رموز الجينات هوغو أثناء التحليل، انظر الخطوات 1.5 و 4-10). في حالة استخدام بيانات تسلسل الحمض النووي الريبي أو seq رقاقة، ينبغي أن تستخدم مقاييس المجموعة المحسوبة يدوياً للبيانات التعبير الجيني (مثلاً نسبة متوسط مجموعة) بدلاً من بيانات العينة الفردية. يجب أن ثم يتم تحليل هذه المقاييس مجموعة باستخدام أداة بريرانكيد جسيا (راجع الخطوة 4، 12). البيانات التعبير الجيني يكون تطبيع كالمعتاد قبل استيرادها إلى البرنامج جسيا. نوع التطبيع (مثلاً أو ربعي أو المفتاح مكعب) عموما متروك للباحث-
ملف - البيانات التعبير الجيني: استخدام تنسيق الملف (*.txt) نص محدد بعلامات جدولة لوصف dataset تعبير كما هو مبين في الشكل 1 ألف. انظر أيضا الملف المثال المدعومة GSE20524_expression.txt.
ملاحظة: يتضمن ملف البيانات التعبير الجيني قيم التعبير جميع الجينات القابلة للاكتشاف (أو المسابر)، أيضا للجينات التي ربما لا يمكن التعبير عنها متفاوتاً. الملف ولذلك عادة ما تضم عدة آلاف من الجينات. وهو ينظم كما هو مبين في الشكل 1 ألف. السطر الأول يحتوي على اسم بطاقة العنونة (مثلاً، والجينات الرمز أو التحقيق معرف) متبوعاً بالمعرف لكل عينة في dataset (مثلاً، نموذج 1، نموذج 2 إلخ). ما تبقى الملف يحتوي على قيم التعبير لكل من الجينات وكل عينة في dataset. أداة البرنامج جسيا ينفذ العمليات الحسابية لمجموعة المقاييس (مثلاً، المجموعة يعني نسبة أو إشارة إلى نسبة الضوضاء)، ولذلك من المستحسن تضمين البيانات لكل عينة الفردية. وبدلاً من ذلك، فمن الممكن لاستخدام المقاييس المحسوبة خارجياً مجموعة البيانات التعبير الجيني (انظر الشكل 1B).
ملف - النمط الظاهري: إنشاء ملف منفصل لتحديد وتصنيف المجموعات التي تضم العينات الفردية كما هو مبين في < طبقة قوية= "إكسفيج" > 2 الرقم. استخدام مسافات أو علامات التبويب لفصل الحقول. حفظه بتنسيق ملف CLS (تعريف فئة c + +). انظر أيضا الملف المثال المدعومة GSE20524_pheno_infection.cls.
ملاحظة: يحتوي السطر الأول على العدد الإجمالي للعينات، وكذلك العدد من المجموعات ( الشكل 2). في حين أن عدد العينات التي يجب أن تتوافق مع ملف البيانات التعبير الجيني (انظر 3.2)، عدد المجموعات التي تعتمد على تصميم الدراسة. الحقل الثالث من السطر الأول دائماً ' 1 '.
يحتوي السطر الثاني في ملف CLS على اسم لكل مجموعة. يجب أن يبدأ السطر بعلامة جنيه (#) متبوعة بمسافة ( الشكل 2).
السطر الثالث يحتوي على تسمية فريق لكل عينة. يمكن أن تكون تسمية المجموعة عدد التعسفي أو النص. هو فقط ترتيب التسميات التي تحدد الرابطة لكل عينة للمجموعات: يتم تعيين التسمية الأولى المستخدمة للمجموعة الأولى في السطر الثاني؛ التسمية الثانية فريد تم تعيينه إلى المجموعة الثانية وهلم جرا. التأكد من أن كل عينة من نفس المجموعة نفس التسمية في هذه الخطوة، وأن عدد التسميات هو نفس عدد العينات المحدد في السطر الأول. وأخيراً، قم بحفظ الملف كملف نصي محدد بعلامات جدولة (*.txt) وتغيير ملحق اسم الملف يدوياً إلى (*.cls)- - ملفات "قاعدة بيانات تعيين الجينات" (اختياري): تحديد مجموعات الجينات المخصصة. استخدام تنسيق الملف (الجينات مصفوفة تحويل) بتوقيت جرينتش المفصول لمجموعات الجينات كما هو مبين في الشكل 3. انظر أيضا الملف المثال المدعومة Gene_sets_Inflammation_BIOCARTA_KEGG_REACTOME.gmt.
ملاحظة: تحديد مجموعات الجينات المخصصة يمكن أن تكون مفيدة على سبيل المثال لتقييد الجينات تعيين تحليل الإثراء إلى مسارات ذات الأهمية الخاصة (مثلاً، علم المناعة مما يشير إلى دراسات الانتان)، أو لحيثياته تعريف مجموعات الجينات الخاصة (مثلاً، المنشط و تثبيط الجينات في الدراسات التي يجب أن تكون المقارنة). يتم تنظيم الملف كما هو مبين في الشكل 3. في تنسيق توقيت جرينتش، يمثل كل صف مجموعة جينات ( الشكل 3). يتم وصف كل مجموعة الجينات اسماً ووصفاً والجينات في مجموعة الجينات. العمود الأول يحتوي على أسماء مجموعة الجينات فريدة من نوعها. السطر الثاني قد بشكل اختياري تحتوي على وصف لمجموعة الجينات. الأعمدة التالية تحتوي على أسماء الجينات (رموز الجينات هوغو الرسمية) مجموعة الجينات المقابلة. وأخيراً، قم بحفظ الملف كملف نصي محدد في علامة التبويب (*.txt) ويدويًا تغيير ملحق اسم الملف إلى (*.gmt)-
4. أداء في جسيا
- فتح أداة البرمجيات جسيا (انظر 1-2)-
- انقر ' تحميل بيانات ' الزر الموجود على الجانب الأيسر من الإطار الرئيسي ( الشكل 4 أ). سيتم فتح علامة تبويب جديدة لاستيراد ملفات البيانات المطلوبة ( الشكل 4 باء). تصفح في علامة التبويب الجديدة إلى ملف البيانات (*.txt) التعبير الجيني (انظر 3.2)، ملف النمط الظاهري (*.cls) (انظر 3.3)، واختيارياً، إلى مجموعات الجينات المخصصة (*.gmt) ملف ( الشكل 4B).
- في حالة جسيا يتعذر الاتصال بالإنترنت، وأيضا تحميل مسيجدب الذي تم تنزيله (*.gmt) الملفات (انظر مثلاً، c2.cp.v5.2.symbols.gmt لمسارات، 1.4) والحمض النووي رقاقة الشروح (الصفيف) (*.chip) الملفات (على سبيل المثال، Mouse430_2.chip، انظر 1.5). تظهر البيانات التي تم استيرادها بنجاح في ' تحميل بيانات ' القسم ( الشكل 4).
ملاحظة: كل دراسة التعبير الجيني يجب تحليل مع جسيا على حدة. سيتم إجراء المقارنة بين الدراستين (مثل اضطراب البشرية مقابل طراز الماوس) في الخطوة 5.
- في حالة جسيا يتعذر الاتصال بالإنترنت، وأيضا تحميل مسيجدب الذي تم تنزيله (*.gmt) الملفات (انظر مثلاً، c2.cp.v5.2.symbols.gmt لمسارات، 1.4) والحمض النووي رقاقة الشروح (الصفيف) (*.chip) الملفات (على سبيل المثال، Mouse430_2.chip، انظر 1.5). تظهر البيانات التي تم استيرادها بنجاح في ' تحميل بيانات ' القسم ( الشكل 4).
- انقر ' "تشغيل جسيا" ' الزر الموجود على الجانب الأيسر من الإطار الرئيسي. سيتم فتح علامة تبويب جديدة من أجل تعيين المعلمات للتحليل ( الشكل 4). علامة التبويب ينقسم إلى ثلاثة أجزاء: الحقول المطلوبة والحقول الأساسية و مجالات متقدمة.
- في الحقول المطلوبة، اختر أولاً dataset التعبير تم تحميله في الخطوة 4، 2 ( الشكل 4).
- اختيار الجينات تحدد قاعدة البيانات، من موقع متصل أو من الملف مجموعة الجينات المستوردة يدوياً ( الشكل 4).
- تحرير تسميات النمط الظاهري لتحديد مجموعات العينات التي من المفترض أن تكون مقارنة لبعضها البعض (مثل، العلاج س. المذهبة مقابل مراقبة صحية) ( الشكل 4).
- انهيار dataset إلى رموز الجينات (= true) من أجل ترجمة مسبار معرفات في dataset التعبير الرسمي هوجو الجينات الرموز المستخدمة في قاعدة بيانات مجموعات الجينات. حدد كاذبة، إذا كان التعبير dataset يحتوي بالفعل على رموز الجينات هوغو ( الشكل 4).
- تعيين الإعداد الافتراضي عدد التباديل مبلغ 000 1 ( الشكل 4).
ملاحظة: لأرقام أعلى الوقت الحوسبة سيزداد بصورة كبيرة- - تغيير نوع التقليب إلى ' مجموعة الجينات '، حيث يوصي التقليب النمط الظاهري فقط عندما يكون هناك أكثر من سبع عينات في كل النمط الظاهري ( الشكل 4).
- وأخيراً، حدد النظام الأساسي الرقائق المستخدمة لتوليد البيانات التعبير الجيني، أما من موقع ويب متصلة أو من الحمض النووي المستوردة يدوياً رقاقة (الصفيف) شروح الملف ( الشكل 4).
ملاحظة: هذه الخطوة ضروري فقط، إذا كان يتم استخدام مسبار معرفات في dataset التعبير الذي تم تحميله. - في الحقول الأساسية تحرير على الأقل قسم تحليل الاسم و الحفظ النتائج في هذا المجلد للبحث مرة أخرى عن ملف النتائج ( الشكل 4). وبالإضافة إلى ذلك، يمكن تغيير المزيد من المعلمات الإحصائية. لمزيد التفاصيل على المعلمات والمقطع حقول متقدمة الرجاء الانتقال إلى دليل المستخدم جسيا (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html)-
- (اختياري): في حالة المقاييس المجموعة المحسوبة خارجياً للتعبير الجيني قد البيانات (مثلاً، نسبة متوسط المجموعة) لاستخدامها بدلاً من بيانات العينة الفردية، استخدام أداة جسيا بريرانكيد. ثم يجري التحليل استناداً إلى قائمة بسيطة من الجينات التي تم تعيينها مع المقاييس مجموعة محسوبة مسبقاً التي يتم استخدامها لترتيب الجينات. بعد تحميل الملف التعبير الجيني البديلة الذهاب إلى شريط التنقل الرئيسي وانقر فوق أدوات/جسيبريرانكيد. وبالمثل، سيتم فتح علامة تبويب جديدة لتعيين معلمات للتحليل ( 4E الشكل)-
ملاحظة: باستخدام أداة بريرانكيد جسيا يوصي للدراسات التي ليس لديها بيانات التعبير الفردية الخاصة بعينه الجينات. يمكن أن يكون الحال إذا كانت الإحصاءات الخاصة أو إجراءات التطبيع وأجريت على البيانات المؤدية إلى مجموعة القيم الوسطية بدلاً من بيانات العينة الفردية. من المستحسن استخدام الأداة جسيا بريرانكيد لبيانات تسلسل الحمض النووي الريبي. تطبيع البيانات التعبير تسلسل الحمض النووي الريبي وحساب مجموعة مقاييس للعينات (مثلاً، سجل التغيير إضعاف)، التي يمكن استخدامها لترتيب الجينات وفقا للتعبير عنها- - اضغط ' تشغيل ' زر على الجزء السفلي الأيمن من النافذة.
ملاحظة: التحليل ثم قد يستغرق عدة دقائق اعتماداً على سرعة الكمبيوتر. تتبع التقدم المحرز في التحليل في قسم التقارير جسيا في الجزء السفلي الأيسر من الإطار. بعد الانتهاء من التحليل، والوضع ' نجاح ' يظهر في المقطع تقارير جسيا.
تحليل - انقر على ينجح في قسم التقارير جسيا لفتح نتائج التحليل-
ملاحظة: سيتم فتح قائمة تنقل جديدة في نافذة مستعرض يلخص جميع النتائج وإعدادات المعلمة ( الشكل 5). وتشمل القسمين العلوي من قائمة التنقل تعيين الجينات نتائج الإثراء لمجموعات محددة (مثل تخصيب اليورانيوم في عينات المذهبة تعامل س أو عينات مراقبة صحية). تظهر الأسطر الأولى من كلا القسمين موجزاً لنتائج الإحصاء. وتعتبر مجموعات الجينات التي هي إلى حد كبير أثري بمعدل اكتشاف false (FDR) أقل من 25% أثري في التفسير التالي. ويمكن الاطلاع على المزيد من التفاصيل حول تفسير التحليل في دليل المستخدم جسيا (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html)- - انقر على نتائج تخصيب مفصلة في excel تنسيق تصدير نتائج التحليل إلى جدول بيانات ( الشكل 6A). تصدير excel نتائج تخصيب مفصلة في كل على حدة لتعمل معا ( الشكل 5)، والانضمام إلى نتائج البيانات في ملف جدول بيانات واحد. للمقارنة اللاحقة بين البيانات التعبير الجيني للعديد من الدراسات، الحفاظ على الأقل اسم مجموعة الجينات (العمود A)، نقاط تخصيب طبيعية (متنوعة) (العمود F) وقيمته فرانكلين روزفلت (معدل اكتشاف كاذبة) (العمود H) ( الشكل 6B )-
ملاحظة: ملف جدول البيانات يحتوي على بيانات ضخمة لكل من تحليل الجينات المحددة، بما في ذلك اسم مجموعة الجينات (العمود A)، حجمه (أي عدد الجينات التي تم الكشف عنها في البيانات التعبير الجيني، العمود D)، نس به (مقياس كمي للاتجاه و حد الإثراء، العمود F)، قيمته الاسمية ف (الأعمدة التي لم يتم تصحيحها، ز) وقيمته فرانكلين روزفلت (تصحيح للفرضية متعددة اختبار، العمود H). لمزيد من التفاصيل بشأن التفسير يرجى الرجوع إلى دليل المستخدم جسيا (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html)- - تكرار الجينات تعيين إثراء التحليل (خطوات 4.1 إلى 4.15) للدراسة الثانية (مثلاً، المذهبة س. GSE9960) وكل إجراء المزيد من الدراسات التي من المفترض أن تكون مقارنة لبعضها البعض. وتشمل العديد من الدراسات السريرية البشرية ونماذج الماوس مختلفة قدر الإمكان لتحديد النموذج الأمثل الماوس للسؤال البحثي متعدية.
5. مقارنة "نتائج جسيا"
- لتحديد النموذج الأمثل لمحاكاة مقارنة الحالة البشرية الحيوانية جسيا نتائج جميع الدراسات إلى بعضها البعض. استخدام عشرات الإثراء وقيم فرانكلين روزفلت لتصنيف المسارات (مجموعات الجينات) كما المنشط (الدائرة الوطنية للتوظيف > فرانكلين روزفلت 0, < 25%)، وتحول دون (نس < فرانكلين روزفلت 0, < 25%) أو لا شيء على حد سواء (فرانكلين روزفلت > 25%). لمقارنة كل من هاتين الدراستين، عد للتركيبات الممكنة تسعة من تنظيم مسار كما هو مبين بجدول 3 × 3 للطوارئ ( الشكل 7 أ). تقييم العلاقة بين هاتين الدراستين بحساب القيمة التنبؤية الإيجابية (ppv) والقيمة التنبؤية السلبية (npv)، التي هي بحكم التعريف الجزء من المسارات التي تظهر نفس التنظيم (تنشيط أو تثبيط) في دراستين من
- .
ببف
- حساب وصافي القيمة الحالية وفقا للصيغ التالية (1) و (2):
(1)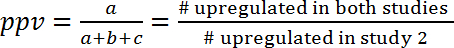
(2)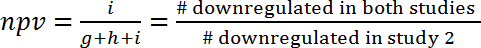
ملاحظة: نظراً للتداخل يمكن أن تكون محض مصادفة، في ppv وصافي القيمة الحالية يجب أن زيادة مقارنة بالقيم المتوقعة بالصدفة. ويسمح هذا النهج تقدير كمية المعلومات التي يمكن الحصول عليها من دراسة واحدة للتنبؤ بالآثار في دراسة أخرى. على سبيل المثال، إذا كانت عمليات التنظيم في هذين النموذجين كانت مستقلة عن بعضها البعض (وإلا تتداخل بالصدفة)، وإذا كانت في النموذج الأول 10% الممرات أوبريجولاتيد، مما ببف للنموذج الثاني سيكون أيضا 10%، وكان هناك إضافية لا الحصول على الوقود النووي المشع أورميشن. على الجانب الآخر، إذا تم ربط كلا النموذجين من آليات التنظيم المشتركة، ثم ppv (وصافي القيمة الحالية) سيكون أكبر بكثير مما كان متوقعا بالصدفة. على سبيل المثال، هو ببف للتنبؤ بالتغيرات تعبير الجينات خلال الانتان البشري (GSE9960) من آثار في المذهبة س مورين حقن نموذج (GSE20524)، 43% (6/(6+8+0)) وصافي القيمة الحالية هو 61% (11/(0+7+11)). وبعبارة أخرى، يتم أيضا تنشيط 43% مسارات المنشط في المذهبة S. موريني حقن طراز (GSE20524) خلال الانتان البشري (GSE9960). وبالمثل، كما تحول دون 61% مسارات تحول دون في المذهبة S. موريني حقن طراز (GSE20524) خلال الانتان البشري (GSE9960) ( الشكل 7). يمكن أيضا تحديد ppv وصافي القيمة الحالية لكوكبة معكوس (وهذا يعني توقع من الدراسة 1 دراسة 2)-
- حساب وصافي القيمة الحالية وفقا للصيغ التالية (1) و (2):
- لحساب التداخل عن طريق الصدفة الرجوع إلى الجدول 3 × 3 للطوارئ ( الشكل 7)، وحساب بفتشانسي ونبفتشانسي وفقا للصيغ التالية (3) و (4):
(3) < img alt = "المعادلة 3" src = "/files/ ftp_upload/55768/55768eq3.jpg "/>
(4)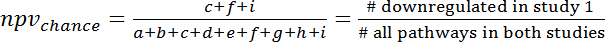
ملاحظة: على سبيل المثال، للتنبؤ بالتغيرات تعبير الجينات خلال الانتان البشري (GSE9960) من آثار في المذهبة س مورين نموذج حقن (GSE20524) بفتشانسي هو 13 في المائة (8/64) ونبفتشانسي و 22% (14/64). - حساب مكسب ببف مقابل فرصة بطرح بفتشانسي من ppv. حساب تبعاً لذلك لصافي القيمة الحالية:
(5)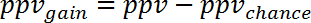
(6)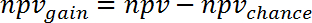
ملاحظة: على سبيل المثال، للتنبؤ بالجينات التعبير يتغير خلال الانتان البشري (GSE9960) من آثار في المذهبة س مورين حقن نموذج (GSE20524) التغير في ppv وهو صافي القيمة الحالية مقابل فرصة + 30% (13%-43%) و 39% (61%-22%)، على التوالي. - حساب مكسب المعلومات التي يمكن الحصول عليها من دراسة 2 فيما يتعلق بالدراسة 1 بالمتوسط بفجاين ونبفجاين:
(7)
- استخدام الجدول للطوارئ المحددة في الخطوة 5، 1 زوج من الدراسات (study1.pathway, study2.pathway) لحساب قيمة p من test. كاي تربيع
تخزين بيانات الجدول للطوارئ في مصفوفة عاشرا إجراء اختبار كاي تربيع، مثلاً، باستخدام R يعمل chisq.test.
ملاحظة: على سبيل المثال، مقارنة الدراسة الانتان البشري المحدد (GSE9960) مع موريني المذهبة س. حقن (GSE20524) يبين نموذج تداخل يعتد به إحصائيا في تنظيم مسار التحريضية:
> chisq.test(X,simulate.p.value=F)$ p.value
3.82e-07
6. تحديد "نموذج الحيوان الأمثل"
- مقارنة جسيا النتائج لكافة تركيبات من الدراسات التي تم تحديدها للتحليل-
ملاحظة: من المستحسن أيضا لمقارنة الدراسات البشرية (مشابهة) إلى بعضها البعض، فضلا عن مختلف الدراسات الحيوانية إلى بعضها البعض. هذه المقارنة يمكن أن توفر نظرة ثاقبة الفرق يبلغ من الدراسات السريرية (أو الاضطرابات) ونماذج حيوانية مختلفة. ومن المتوقع أن تظهر الدراسات السريرية اكتساب تداخل مقبولة وعلى معلومات هامة، لأن خلاف ذلك قد تكون الدراسات السريرية متغايرة جداً لإيجاد نموذج حيوان الذي يمكن أن تحاكي الوضع البشري. وفي هذه الحالة، من المستحسن أن تدرج فقط الدراسات الإنسانية التي مماثلة لبعضها البعض للتعرف على نماذج حيوانية مناسبة. - فرز كافة تركيبات من كسب المعلومات (الخطوة 5، 5). للمقارنة بين العديد من مجموعات البيانات، استخدام مصفوفة وتصور النتائج باستخدام heatmap ملونة أو ما شابه ذلك ( الشكل 8).
- حدد نموذج الحيوان مع كسب أعلى من المعلومات. بغية تقييم أهمية كسب معلومات، كما أن اختبار كاي تربيع (الخطوة 5، 6) يراعي.
ملاحظة: نماذج حيوانية ينبغي إلا يعتبر مناسباً إذا كسب معلومات جوهرية، وإذا كانت قيمة ف لاختبار مربع كاي أدناه مستوى الأهمية. عتبات User-defined عموما يعتمد على عدة عوامل: 1) المعرفة ما قبل الدراسة على إمكانية تحويل النتائج من نموذج الحيوانية إلى البشر (مثل مماثلة فسيولوجيا)، 2) الفائدة المتوقعة بالنسبة للبشر بنجاح مفترضة، 3) العملية انطباق هذه التجربة الحيوانية، و 4) الألم المتوقع، والمعاناة، أو الأضرار التي لحقت بالمعملالحيوانات الخطابة.
النتائج
وأظهرت جسيا سير العمل ولقطات من البيانات النموذجية. ويبين الشكل 1 الجينات التعبير ملف البيانات الذي يحتوي على البيانات ترانسكريبتوميك من الفائدة. لكل دراسة ملف وصفي النمط الظاهري المطلوب الذي يظهر في الشكل 2. يتم تعريف مجموعات الجينات الم...
Discussion
منذ فترة طويلة تم تطبيق نماذج حيوانية للتحقيق في آليات المرض وتطوير استراتيجيات علاجية جديدة. ومع ذلك، بدأت الشكوك بشأن بريديكتيفيتي نماذج حيوانية تنتشر في أعقاب الفشل في التجارب السريرية12. وعلاوة على ذلك، أثيرت مناقشات مثيرة للجدل حول الاستراتيجيات المناسبة لتحليل وتفسير ...
Disclosures
الكتاب يعلن أن لديهم لا تضارب المصالح المالية.
Acknowledgements
تم تمويل هذا العمل من المعهد الاتحادي الألماني "تقييم المخاطر" (BfR).
Materials
| Name | Company | Catalog Number | Comments |
| Excel | Microsoft Corporation |
References
- Seok, J., et al. Genomic responses in mouse models poorly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 110 (9), 3507-3512 (2013).
- Takao, K., Miyakawa, T. Genomic responses in mouse models greatly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 112 (4), 1167-1172 (2015).
- Weidner, C., Steinfath, M., Opitz, E., Oelgeschläger, M., Schönfelder, G. Defining the optimal animal model for translational research using gene set enrichment analysis. EMBO Mol Med. 8 (8), 831-838 (2016).
- Subramanian, A., et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. Proc Natl Acad Sci U S A. 102 (43), 15545-15550 (2005).
- Kanehisa, M., Sato, Y., Kawashima, M., Furumichi, M., Tanabe, M. KEGG as a reference resource for gene and protein annotation. Nucleic Acids Res. 44 (D1), D457-D462 (2016).
- Kanehisa, M., Goto, S. KEGG: kyoto encyclopedia of genes and genomes. Nucleic Acids Res. 28 (1), 27-30 (2000).
- Fabregat, A., et al. The Reactome pathway Knowledgebase. Nucleic Acids Res. 44 (D1), D481-D487 (2016).
- Croft, D., et al. The Reactome pathway knowledgebase. Nucleic Acids Res. 42 (Database issue), D472-D477 (2014).
- Nishimura, D. BioCarta. Biotech Software & Internet Report. 2 (3), 117-120 (2001).
- Edgar, R., Domrachev, M., Lash, A. E. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Res. 30 (1), 207-210 (2002).
- Kolesnikov, N., et al. ArrayExpress update--simplifying data submissions. Nucleic Acids Res. 43 (Database issue), D1113-D1116 (2015).
- Cohen, J., et al. Sepsis: a roadmap for future research. Lancet Infect Dis. 15 (5), 581-614 (2015).
- Spinelli, L., Carpentier, S., Montanana Sanchis, F., Dalod, M., Vu Manh, T. P. BubbleGUM: automatic extraction of phenotype molecular signatures and comprehensive visualization of multiple Gene Set Enrichment Analyses. BMC Genomics. 16 (1), 814 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved