Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un protocole pour l’utilisation de gènes série d’analyses d’enrichissement pour identifier le modèle Animal approprié de recherche translationnelle
Dans cet article
Résumé
Nous fournissons un protocole normalisé pour l’utilisation du gène enrichissement set analyse des données transcriptomiques pour identifier un modèle souris idéal pour la recherche translationnelle.
Ce protocole peut être utilisé avec les puces à ADN et des données de séquençage de l’ARN et encore peut être étendu à d’autres données omics si des données sont disponibles.
Résumé
Récentes études comparant les ensembles de données transcriptomique des maladies humaines avec les ensembles de données de modèles de souris en utilisant des techniques traditionnelles de gènes comparaison a abouti à des conclusions contradictoires au sujet de la pertinence de modèles animaux pour translationnelle recherche. Des principales raisons des écarts entre les analyses d’expression de gène différent sont le filtrage arbitraire de gènes différentiellement exprimés. En outre, la comparaison d’un simple gène entre espèces différentes et souvent les plates-formes est limitée par la technique variance, conduisant à une interprétation erronée de la con/discordance entre les données provenant de modèles humains et animaux. Ainsi, les méthodes normalisées d’analyse des données systématiques sont nécessaires. Pour surmonter le gène subjective de filtrage et les comparaisons de gène à gène inefficaces, nous a récemment démontré que le gène enrichissement set analyse (GSEA) a le potentiel pour éviter ces problèmes. Par conséquent, nous avons développé un protocole normalisé pour l’utilisation de GSEA pour distinguer les modèles animaux appropriés et inappropriés pour la recherche translationnelle. Ce protocole ne convient pas à prédire comment concevoir de nouveaux systèmes de modèle a priori–, puisqu’elle requiert des données expérimentales omics existantes. Toutefois, le protocole décrit comment interpréter les données existantes d’une manière standardisée afin de sélectionner le modèle animal plus adapté, ainsi évitant les expérimentations animales inutiles et trompeuses études translationnelles.
Introduction
Modèles animaux sont largement utilisés pour l’étude des maladies humaines, en raison de leur ressemblance supposée à l’homme sur le plan génétique, l’anatomie et la physiologie. En outre, des modèles animaux souvent servent portiers aux thérapies cliniques et peuvent avoir un impact énorme sur le succès de la recherche translationnelle. Une sélection rigoureuse du modèle animal optimale peut réduire le nombre d’études animales trompeuses. Récemment, la pertinence de modèles animaux pour la recherche translationnelle a été controversée, notamment parce que l’analysant les ensembles de données même obtenus à partir des maladies inflammatoires humaines et modèles murins connexes ont conduit à des conclusions contradictoires 1,2. Cette discussion a révélé un problème fondamental au cours de l’analyse des données omique : des approches normalisées pour l’analyse de données systématiques sont nécessaires afin de réduire la sélection de gènes partiale et d’augmenter la robustesse des comparaisons interspécifiques 3.
Traditionnellement, l’analyse des données transcriptomique (et autres données omics) se fait au niveau monogéniques et comprend une première étape de sélection génétique basée sur les paramètres de seuil strictes (par exemple, les changements de pli > 2.0, la valeur p < 0,05). Cependant, le réglage des paramètres de seuil initial souvent est subjectif, arbitraire et pas biologiquement justifiée et peut même conduire à des conclusions opposées1,2. En outre, sélection de gènes initial généralement restreint l’analyse aux quelques très en amont et réprimés gènes et n’est donc pas suffisamment sensible pour inclure la plupart des gènes qui sont exprimés dans une moindre mesure.
Avec la montée de l’ère de la génomique dans les années 2000 et la connaissance croissante des voies biologiques et des contextes, des approches statistiques ont été développés qui permettait de contourner les limites des analyses de niveau monogéniques. Gène set enrichissement analyse (GSEA)4, qui est l’une des méthodes largement acceptées pour l’analyse des données transcriptomique, fait appel à des groupes définis a priori des gènes (par exemple, signalisation, localisation proximale sur un chromosome etc..). GSEA mappe tout d’abord tous les gènes non filtrées détectés pour les ensembles de gènes prévue (p. ex., voies), quel que soit leur changement individuel dans l’expression. Ainsi, cette approche comprend aussi modérément réglementé de gènes qui seraient autrement perdus avec analyses de niveau monogéniques. Le changement d’additif dans l’expression à l’intérieur des ensembles de gènes s’effectue par la suite en utilisant les statistiques de somme en cours d’exécution.
Malgré sa large utilisation dans la recherche médicale, GSEA et approches connexes enrichissement set ne sont pas évidemment pris en compte pour l’analyse des données complexes omics. Nous décrivons ici un protocole pour comparer les omiques données provenant d’échantillons humains avec ceux des modèles de souris afin d’identifier le modèle idéal pour les études translationnelles. Nous démontrons l’applicabilité du protocole basé sur une collection de modèles de souris utilisées pour imiter les troubles inflammatoires humaines. Toutefois, ce pipeline d’analyse ne se limite pas à l’homme-souris comparaisons et recherche modifiable en plus d’autres questions.
Protocole
1. téléchargement du logiciel GSEA et la base de données de Signatures moléculaires
- aller sur le site officiel de l’Institut large GSEA (http://software.broadinstitute.org/gsea/index.jsp) et s’inscrire pour avoir accès au logiciel GSEA outil et la base de données de Signatures moléculaires (MSigDB).
- Télécharger l’application de bureau javaGSEA ou une option de logiciel alternatif (par exemple, le script de R).
Remarque : Toutes les options appliquer exactement le même algorithme. Le logiciel GSEA est librement disponible aux individus dans les universités et l’industrie à des fins de recherches internes. - Pour plus amples détails sur le logiciel GSEA aller au site Web de la documentation (http://software.broadinstitute.org/cancer/software/gsea/wiki/index.php/Main_Page) et le guide de l’utilisateur GSEA (http://software.broadinstitute.org/gsea/doc/ GSEAUserGuideFrame.html).
- Télécharger la base de données de Signatures moléculaires (MSigDB) sur le site GSEA pour accéder aux collections ensemble de gènes individuels.
Remarque : Le MSigDB est une collection d’ensembles de gènes annotés pour une utilisation avec le logiciel GSEA ou d’autres fins. Ensembles de gènes peuvent être classés par la signalisation des voies, termes d’ontologie génique, motifs cis-régulation, signatures expérimentales et d’autres. Gènes de la MSigDB sont toujours nommés par leur symbole officiel du gène HUGO (Human Genome Organisation). Pour la comparaison du règlement de la voie entre une maladie humaine donnée et l’autre souris modèles il est recommandé de télécharger le ' toutes les voies canoniques, symboles du gène ' file (c2.cp.v5.2.symbols.gmt). Ce fichier comprend des ensembles de gènes qui ont été annotées et organisés dans les voies de signalisation par KEGG 5 , 6, Reactome 7 , 8 et BioCarta 9. la chaîne ' v5.2 ' représente les informations de version de la collection. Assurez-vous de télécharger la dernière version des fichiers. Le MSigDB est disponible gratuitement aux personnes dans les milieux universitaires et industriels à des fins de recherche interne. Il n’est pas nécessaire de télécharger le MSigDB, si la connexion internet est fournie lors de l’analyse. Dans ce cas, le MSigDB peut directement être choisi au sein de l’interface utilisateur GSEA. - Télécharger DNA chip fichiers annotations (tableau) sur le site GSEA à traduire les identificateurs de sonde tableau spécifique à HUGO gène symboles généraux (p. ex., Mouse430_2.chip).
Remarque : Il n’est pas nécessaire de télécharger les annotations de puce ADN, si la connexion internet est fournie lors de l’analyse. Dans ce cas les annotations de puce ADN peuvent directement être choisies au sein de l’interface utilisateur GSEA. Le protocole peut également servir avec les données de séquençage de l’ARN. Dans ce cas, il n’est pas nécessaire de télécharger les fichiers d’annotation. Au lieu de cela, utilisez l’outil preranked GSEA pour analyser les données d’expression de gène (Voir l’étape 4.12).
2. Télécharger des données expérimentales d’Expression génique pour la maladie humaine et des modèles appropriés d’Animal
- identifier des études d’expression (transcriptomique) gène expérimentale pour la maladie humaine de choix (p. ex., profils d’expression génique de leucocytes provenant de patients atteints de troubles septique, GSE9960).
- Même, recherche d’animaux plusieurs modèles qui sont censés être comparées avec les études sur les humains (p. ex., profils d’expression génique des cellules sanguines provenant de souris après injection de staphylocoque doré (Staphylococcus aureus), GSE20524). À cette étape, utiliser les connaissances préalables pour la présélection des modèles animaux qui pourraient convenir pour imiter la situation humaine.
- Pour cela se référer à la littérature et les bases de données telles que le Gene Expression Omnibus (GEO) 10 ou 11 de la ArrayExpress des bases de données et téléchargement les données transcriptomique normalisée d’intérêt. Enregistrer les données sous forme de fichiers texte sur le disque dur local. Pour la base de données GEO, le téléchargement de fichiers de texte délimité par des tabulations série matrix est recommandé. Également prendre note de la plateforme (type array) utilisé pour cette étude, puisque cette information est nécessaire pour traduire les identificateurs de sonde tableau spécifique aux symboles généraux de gène HUGO.
Remarque : S’assurer suffisamment de mémoire pour stocker les données, comme des ensembles de données transcriptomique comprennent généralement plusieurs centaines de Mo.
3. Données de manutention et de mise en forme
- avant d’importer les données d’expression génique expérimental dans l’outil logiciel GSEA, examiner la structure de données requises. Pour chaque étude manuellement créer deux fichiers différents : 1) un fichier de données d’expression de gène contenant les valeurs de mesure pour des exemples et des gènes différents et 2) un phénotype de fichier contenant les étiquettes de l’échantillon pour grouper des échantillons individuels (par exemple, à des groupes de traitement).
Pour plus amples détails et structure de données options aller à la page de format de données GSEA (http://software.broadinstitute.org/cancer/software/gsea/wiki/index.php/Data_formats).
Remarque : En règle générale, toutes les formes de données transcriptomique sont compatibles avec le protocole, y compris les expériences de microréseau d’ADN, RNA-seq ou études de ChIP-seq. Dans le cas à l’aide d’expériences de microréseau d’ADN, le fichier de données d’expression de gène devrait contenir identificateur de sonde tableau spécifique ou symboles de gène de HUGO pour chaque gène (sonde identificateurs se traduira pour les symboles de gène HUGO pendant l’analyse, voir les étapes 1.5 et 4.10). En cas d’utilisation de données RNA-seq ou ChIP-seq, mesures de groupe calculé manuellement des données d’expression de gène (par exemple, le ratio moyen groupe) doivent être utilisés au lieu de données échantillon individuel. Ces mesures de groupe doivent ensuite être analysées avec l’outil preranked GSEA (voir étape 4.12). Données d’expression doivent être normalisées comme d’habitude avant d’importer dans le logiciel GSEA. Le type de normalisation (p. ex., quartile ou spline cubique) est généralement laissé au chercheur.
Fichier - données d’expression génique : utilisez le format texte délimité par des tabulations (*.txt) pour décrire un ensemble de données d’expression, comme illustré à la Figure 1 a. Voir aussi le fichier d’exemple de prise en charge GSE20524_expression.txt.
Remarque : Le fichier de données d’expression de gène contient des valeurs d’expression pour tous les gènes détectable (ou sondes), également pour les gènes qui ne pourraient pas être exprimés. Le fichier comprend donc généralement plusieurs milliers de gènes. Il est organisé comme l’illustre la Figure 1 a. La première ligne contient le nom d’étiquette (par exemple, le gène symbole ou sonde ID) suivi par un identificateur pour chaque échantillon dans le groupe de données (par exemple, exemple 1, exemple 2 etc.). Le reste du fichier contient les valeurs de l’expression pour chacun des gènes et pour chaque échantillon dans le dataset. L’outil logiciel GSEA effectue des calculs pour les mesures de groupe (par exemple, ratio moyen de groupe ou rapport signal-à-bruit), il est donc recommandé d’inclure les données pour chaque échantillon individuel. Alternativement, il est possible d’utiliser des mesures de groupe externe calculée pour les données d’expression de gène (voir Figure 1 b).
Fichier - phénotype : créer un fichier distinct de définition et l’étiquetage des groupes qui comprennent des échantillons individuels telle que décrite par < forte classe= « xfig » > Figure 2. Utilisez des espaces ou des tabulations pour séparer les champs. Enregistrez-le dans un format de fichier CLS (définition de la classe C++). Voir aussi le fichier d’exemple de prise en charge GSE20524_pheno_infection.cls.
Remarque : La première ligne contient le nombre total d’échantillons et plus le nombre de groupes ( Figure 2). Alors que le nombre d’échantillons doit correspondre pour le fichier de données d’expression de gène (voir 3.2), le nombre de groupes dépend de la conception de l’étude. Le troisième champ de la première ligne est toujours ' 1 '.
La deuxième ligne dans un fichier CLS contient le nom de chaque groupe. La ligne doit commencer par un signe dièse (#) suivi d’un espace ( Figure 2).
La troisième ligne contient une étiquette de groupe pour chaque échantillon. L’étiquette de groupe peut être un nombre arbitraire ou du texte. C’est seulement l’ordre des étiquettes qui détermine l’association de chaque échantillon pour les groupes : le premier label utilisé est attribué au premier groupe sur la deuxième ligne ; la seconde étiquette unique est attribuée au second groupe, et ainsi de suite. S’assurer que chaque échantillon du même groupe le libellé est identique à cette étape, et que le nombre d’étiquettes est le même que le nombre d’échantillons spécifiés dans la première ligne. Enfin, enregistrez le fichier en tant que fichier texte délimité par des tabulations (*.txt) et modifier manuellement l’extension de nom de fichier à (*.cls). - (optionnel) fichiers de base de données de la valeur de Gene : définir des ensembles de gènes personnalisé. Utilisez le format de fichier délimité par des tabulations GMT (Gene matrice transposée) pour les jeux de gène, tel que représenté dans la Figure 3. Voir aussi le fichier d’exemple de prise en charge Gene_sets_Inflammation_BIOCARTA_KEGG_REACTOME.gmt.
NOTE : Définir des jeux de gène personnalisé peut être utile par exemple pour limiter la gène série d’analyses de l’enrichissement de voies d’intérêt spécial (p. ex., immunologie de signalisation pour les études de septicémie) ou pour définir des jeux de gène propre en reprenant (p. ex., activé et inhibent les gènes dans les études qui doivent être comparées). Le fichier est organisé tel que représenté dans la Figure 3. Au format GMT, chaque ligne représente un ensemble de gènes ( Figure 3). Chaque ensemble de gènes est décrite par un nom et une description des gènes dans l’ensemble de gènes. La première colonne contient les noms uniques de gène. La deuxième ligne peut éventuellement contenir une description de l’ensemble de gènes. Les colonnes suivantes contiennent les noms de gène (symboles de gène de HUGO officiels) de l’ensemble de gènes correspondants. Enfin, enregistrez le fichier sous l’onglet fichier de texte délimité (*.txt) et modifier manuellement l’extension de nom de fichier à (* .gmt).
4. Effectuant la GSEA
- Ouvrez l’outil de logiciel GSEA (voir 1.2).
- Cliquez sur le ' charger des données ' bouton sur le côté gauche de la fenêtre principale ( Figure 4 a). Un nouvel onglet s’ouvre pour l’importation des fichiers de données requis ( Figure 4 b). Naviguer dans le nouvel onglet fichier de données (*.txt) expression génique (voir 3.2), le fichier de phénotype (*.cls) (voir 3.3) et, éventuellement, pour les ensembles de gènes personnalisé (* .gmt) fichier ( Figure 4 b).
- Dans l’affaire GSEA impossible de se connecter à internet, également charger le MSigDB téléchargé (* .gmt) fichiers (par exemple, c2.cp.v5.2.symbols.gmt pour les voies, voir 1.4) et l’ADN puce annotations (array) (* .chip) fichiers (par exemple, Mouse430_2.chip, Voir 1.5). Les données importées avec succès apparaissent dans le ' charger des données ' section ( Figure 4).
Remarque : Chaque étude d’expression de gène doit être analysée individuellement avec GSEA. La comparaison entre les deux études (p. ex. une maladie humaine vs modèle murin) sera effectuée à l’étape 5.
- Dans l’affaire GSEA impossible de se connecter à internet, également charger le MSigDB téléchargé (* .gmt) fichiers (par exemple, c2.cp.v5.2.symbols.gmt pour les voies, voir 1.4) et l’ADN puce annotations (array) (* .chip) fichiers (par exemple, Mouse430_2.chip, Voir 1.5). Les données importées avec succès apparaissent dans le ' charger des données ' section ( Figure 4).
- Cliquez sur le ' exécuter GSEA ' bouton sur le côté gauche de la fenêtre principale. Un nouvel onglet s’ouvre afin de définir les paramètres de l’analyse ( Figure 4). L’onglet est divisé en trois parties : les champs obligatoires, les champs de base et avancée de champs.
- Dans les champs requis, choisissez d’abord l' expression dataset chargé à l’étape 4.2 ( Figure 4).
- Choisir le gène affecte la base de données, soit de site Web connecté le fichier manuellement importées de gène d’ensemble ( Figure 4).
- Modifier les étiquettes de phénotype pour sélectionner les groupes d’échantillons qui sont censés être comparés les uns aux autres (p. ex., traitement vs contrôle sain de S. aureus) ( Figure 4).
- Effondrement dataset aux symboles de gène (= true) afin de traduire les identificateurs de sonde dans le dataset d’expression pour les symboles officiels de gène HUGO utilisés dans la base de données des ensembles de gènes. Sélectionnez false, si l’expression dataset contient déjà des symboles de gène de HUGO ( Figure 4).
- La valeur par défaut le nombre de permutations pour 1000 ( Figure 4).
Remarque : Pour un plus grand nombre le temps de calcul augmente considérablement. - Modifier le type de permutation pour ' ensemble de gènes ', étant donné que la permutation de phénotype n’est recommandée que lorsqu’il y a plus de sept échantillons dans chaque phénotype ( Figure 4).
- Enfin, sélectionnez la plate-forme de la puce utilisée pour générer les données d’expression de gène, de site Web connecté ou de l’ADN manuellement importée chip fichier d’annotations (array) ( Figure 4).
Remarque : Cette étape n’est nécessaire, si la sonde les identificateurs sont utilisés dans le dataset téléchargé expression. - Dans les champs de base de modifier au moins la section nom de l’analyse et l’enregistrement des résultats dans ce dossier pour retrouver le fichier de résultats ( Figure 4). En outre, outre les paramètres statistiques peuvent être modifiés. Pour plus détails sur les paramètres et la section champs avancés veuillez consulter le guide de l’utilisateur GSEA (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html).
- (Facultatif) : dans l’affaire mesures de groupe extérieurement calculé pour l’expression des gènes données (par exemple, la proportion moyenne groupe) doivent être utilisés au lieu des exemples de données, utilisez l’outil preranked de GSEA. L’analyse sera effectuée ensuite basé sur une simple liste de gènes assignés avec mesures de groupe pré-calculées qui sont utilisés pour classer les gènes. Après avoir chargé le fichier d’expression de gène alternatives vont à la barre de navigation principale et cliquez sur Outils/GseaPreranked. De même, un nouvel onglet s’ouvrira pour définir les paramètres pour l’analyse ( Figure 4F).
Remarque : En utilisant l’outil preranked GSEA est recommandée pour les études qui n’ont pas de données d’expression spécifiques à échantillon individuel. Cela pourrait être le cas si les statistiques spéciales ou des procédures de normalisation ont été effectuées sur les données conduisant à des valeurs moyennes de groupe au lieu de données échantillon individuel. À l’aide de l’outil preranked GSEA est recommandé pour les données de séquençage de l’ARN. Normaliser les données d’expression de séquençage RNA et calculer les mesures de groupe pour les échantillons (par exemple, journal des changements de pli), qui peuvent être utilisées pour classer les gènes selon leur expression. - Cliquez sur le ' Run ' bouton en bas à droite de la fenêtre.
NOTE : L’analyse peut prendre plusieurs minutes selon la vitesse de calcul. Suivez les progrès de l’analyse dans la section rapports GSEA sur le bas gauche de la fenêtre. Après avoir terminé l’analyse, le statut ' succès ' apparaît dans la section rapports GSEA. - Cliquer sur l’aboutie analyse dans la section rapports GSEA pour ouvrir les résultats d’analyse.
Remarque : Un nouveau menu de navigation s’ouvre dans une fenêtre de navigateur qui récapitule tous les résultats et les réglages des paramètres ( Figure 5). La partie supérieure comprennent deux sections du menu navigation gène déterminer les résultats de l’enrichissement pour les groupes définis (par exemple, l’enrichissement dans les échantillons de S. aureus traités ou échantillons témoins sains). Les premières lignes de ces deux sections montrent un résumé des résultats statistiques. Des ensembles de gènes qui sont significativement enrichis à un taux de faux-découverte (FDR) inférieur à 25 % sont considérées comme enrichie dans l’interprétation suivante. On trouvera des précisions sur l’interprétation de l’analyse dans le guide de l’utilisateur GSEA (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html). - Cliquez sur les résultats détaillés de l’enrichissement en format excellent pour exporter les résultats d’analyse dans une feuille de calcul ( Figure 6 a). Exporter les résultats détaillés de l’enrichissement dans excellent séparément pour les deux phénotypes ( Figure 5) et joindre les données de résultats dans un fichier de feuille de calcul. Pour une comparaison ultérieure entre les données d’expression de gène de plusieurs études, conserver au moins le nom de l’ensemble de gènes (colonne A), son enrichissement normalisée score (NES) (colonne F) et sa valeur FDR (taux de fausse découverte) (colonne H) ( Figure 6 b ).
Remarque : Le fichier de feuille de calcul contient des données énormes pour chacun de l’analysa gène la valeur, y compris le nom de l’ensemble de gènes (colonne A), sa taille (c'est-à-dire le nombre de gènes détectés dans les données d’expression de gène, colonne D), son NDA (une mesure quantitative de la direction et la mesure de l’enrichissement, la colonne F), sa valeur nominale p (sans correction, colonne G) et sa valeur FDR (corrigées pour l’hypothèse de multiples essais, colonne H). Pour plus amples détails sur l’interprétation veuillez vous référer au guide d’utilisation GSEA (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html). - Répéter que le gène mis enrichissement analyse (4.1 à 4,15) pour la seconde étude (p. ex., S. aureus GSE9960) et pour toutes les autres études qui sont censés être comparés les uns aux autres. Inclure autant des études cliniques et des modèles de souris différents que possible pour identifier le modèle de souris optimale pour la question de la recherche translationnelle.
5. En comparant les résultats GSEA
- pour identifier le modèle animal optimal pour imiter la situation humaine de comparer les résultats GSEA d’études tous les uns aux autres. Utiliser les notes de l’enrichissement et les valeurs FDR pour classer les voies (ensembles de gènes) comme activé (NES > FDR 0, < 25 %), inhibé (NES < FDR 0, < 25 %) ou aucun des deux (FDR > 25 %). Pour chaque comparaison des deux études, compter le nombre de réalisations des neuf combinaisons possibles de règlement de la voie indiquée par un tableau de contingence de 3 x 3 ( Figure 7 a).
- Évaluer la corrélation entre les deux études de calcul de la valeur prédictive positive (VPP) et la valeur prédictive négative (VPN), qui est par définition la partie des voies qui montrent le même règlement (activé ou inhibé) dans deux études .
- Calculer ppv et Van selon les formules suivantes (1) et (2) :
(1)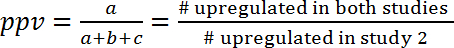
(2)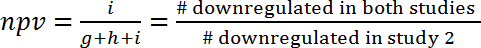
Remarque : Étant donné le chevauchement pourrait être purement fortuite, le ppv et la van ont à encore comparer avec les valeurs attendues par hasard. Cette approche permet l’estimation de la quantité d’informations qui peuvent être tirées d’une étude pour prédire les effets dans une autre étude. Par exemple, si les processus de règlement en deux modèles étaient indépendants l’un de l’autre (et seulement se chevauchent pas par hasard), et si dans le premier modèle, 10 % des voies étaient surexprimés, que le ppv pour le second modèle serait également 10 % et il n’y avait pas de plus gain de inf ormation. De l’autre côté, si les deux modèles étaient liées par des mécanismes de régulation commune, puis le ppv (et npv) serait sensiblement plus forte que prévu par hasard. Par exemple, pour la prévision des modifications de l’expression génique au cours de l’infection humaine (GSE9960) des effets dans un modèle murin de l’injection du aureus S. (GSE20524), la VPP est de 43 % (6/(6+8+0)) et la van est de 61 % (11/(0+7+11)). En d’autres termes, 43 % des voies activées dans le modèle murin d’injection pour les aureus S. (GSE20524) sont également activées au cours de l’infection humaine (GSE9960). De même, 61 % des voies inhibées dans le modèle murin d’injection pour les aureus S. (GSE20524) sont inhibés aussi au cours de l’infection humaine (GSE9960) ( Figure 7 b). PPV et Van peuvent aussi être établies pour la constellation inverse (autrement dit, prédisant d’étude 1 pour étude 2).
- Calculer ppv et Van selon les formules suivantes (1) et (2) :
- Pour calculer le chevauchement par hasard, consultez le tableau de contingence de 3 x 3 ( Figure 7) et calculer ppvchance et npvchance selon les formules suivantes (3) et (4) :
(3) < img alt = « Équation 3 » src = « /files/ ftp_upload/55768/55768eq3.jpg » / >
(4)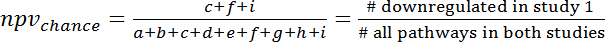
Remarque : par exemple, pour la prévision des modifications d’expression génique lors septicémie humaine (GSE9960) des effets chez un murin S. aureus modèle injection (GSE20524) le ppvchance est de 13 % (8/64) et le npvchance est et 22 % (14/64). - Calculer le gain de la sharka vs chance en soustrayant ppvchance de ppv. Calculer en conséquence pour la van :
(5)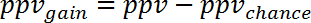
(6)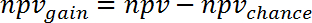
Remarque : par exemple, pour la prédiction du gène expression est modifiée au cours de l’infection humaine (GSE9960) des effets dans un modèle murin de l’injection du aureus S. (GSE20524) le changement de ppv et Van vs chance est de + 30 % (43 % - 13 %) et + 39 % (61 % - 22 %), respectivement. - Calculer le gain d’information qui peut être obtenue d’étude 2 au sujet de l’étude 1 en faisant la moyenne ppvgain et npvgain :
(7)
- utiliser la table de contingence définie à l’étape 5.1 d’une paire d’études (study1.pathway, study2.pathway), à calculer la valeur de p par un test de khi-deux.
Stocker les données de la table de contingence dans une matrice de X. effectuer le test de khi-deux, par exemple, par l’utilisation de la R de la fonction chisq.test.
NOTE : par exemple, comparant l’étude sélectionnée septicémie humaine (GSE9960) avec un murin S. aureus injection modèle (GSE20524) montre un chevauchement significatif dans le règlement de la voie inflammatoires :
> chisq.test(X,simulate.p.value=F)$ p.value
3.82e-07
6. Identifier le modèle Optimal d’Animal
- comparer le GSEA résultats pour toutes les combinaisons des études qui ont été retenues pour l’analyse.
Remarque : Il est également recommandé de comparer les études humaines (similaires) à un autre ainsi que différentes études chez l’animal à l’autre. Cette comparaison peut donner un aperçu de la variation intraspécifique des études cliniques (ou troubles) et les différents modèles animaux. Il est prévu que les études cliniques devraient montrer un chevauchement acceptable et une information significative gain, parce que sinon, les études cliniques peuvent être trop hétérogènes pour trouver un modèle animal qui peut imiter la situation humaine. Dans ce cas, il est recommandé d’inclure seulement les études humaines qui sont semblables les uns aux autres pour l’identification de modèles animaux appropriés. - Trier toutes les combinaisons par le gain d’information (étape 5.5). Pour la comparaison de plusieurs séries de données, utilisez une matrice et visualiser les résultats au moyen d’un heatmap colorée ou similaires ( Figure 8).
- Sélectionner le modèle animal dont le gain est plus élevé d’informations. Afin d’évaluer l’importance du gain de l’information, compte également le test de khi-deux (point 5.6).
Remarque : Modèles animaux devraient seulement être considérés comme approprié si le gain d’information est important et si la valeur p du test khi-deux est inférieure au seuil de signification. User-Defined seuils généralement dépendent de plusieurs facteurs : 1) la connaissance de l’étude préliminaire sur la transférabilité des résultats du modèle animal à l’humain (p. ex. la physiologie similaire), 2) le bénéfice escompté pour les humains par un succès présumé, 3) la pratique applicabilité de l’expérimentation animale et 4) l’attendu douleur, souffrance ou préjudice infligé à la labanimaux de l’oratoire.
Résultats
GSEA workflow et des captures d’écran de données exemplaires sont démontrés. La figure 1 montre le fichier de données d’expression de gène qui contient les données transcriptomiques d’intérêt. Pour chaque étude, un fichier descriptif de phénotype est nécessaire qui est illustré à la Figure 2. Jeux de gènes annotés (p. ex., voies) sont définis dans le fichier de base de données set de gène (
Discussion
Des modèles animaux ont longtemps été appliquées pour l’étude des mécanismes des maladies et le développement de nouvelles stratégies thérapeutiques. Cependant, le scepticisme au sujet de la valeur prédictive des modèles animaux commencé à se répandre après les échecs des essais cliniques,12. En outre, des discussions controversées sur les stratégies appropriées pour analyser et interpréter les omiques grandes données issues des essais précliniques ont été soulevées par ...
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont aucun intérêt financier concurrentes.
Remerciements
Ce travail a été financé par l’Institut fédéral allemand pour l’évaluation des risques (BfR).
matériels
| Name | Company | Catalog Number | Comments |
| Excel | Microsoft Corporation |
Références
- Seok, J., et al. Genomic responses in mouse models poorly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 110 (9), 3507-3512 (2013).
- Takao, K., Miyakawa, T. Genomic responses in mouse models greatly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 112 (4), 1167-1172 (2015).
- Weidner, C., Steinfath, M., Opitz, E., Oelgeschläger, M., Schönfelder, G. Defining the optimal animal model for translational research using gene set enrichment analysis. EMBO Mol Med. 8 (8), 831-838 (2016).
- Subramanian, A., et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. Proc Natl Acad Sci U S A. 102 (43), 15545-15550 (2005).
- Kanehisa, M., Sato, Y., Kawashima, M., Furumichi, M., Tanabe, M. KEGG as a reference resource for gene and protein annotation. Nucleic Acids Res. 44 (D1), D457-D462 (2016).
- Kanehisa, M., Goto, S. KEGG: kyoto encyclopedia of genes and genomes. Nucleic Acids Res. 28 (1), 27-30 (2000).
- Fabregat, A., et al. The Reactome pathway Knowledgebase. Nucleic Acids Res. 44 (D1), D481-D487 (2016).
- Croft, D., et al. The Reactome pathway knowledgebase. Nucleic Acids Res. 42 (Database issue), D472-D477 (2014).
- Nishimura, D. BioCarta. Biotech Software & Internet Report. 2 (3), 117-120 (2001).
- Edgar, R., Domrachev, M., Lash, A. E. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Res. 30 (1), 207-210 (2002).
- Kolesnikov, N., et al. ArrayExpress update--simplifying data submissions. Nucleic Acids Res. 43 (Database issue), D1113-D1116 (2015).
- Cohen, J., et al. Sepsis: a roadmap for future research. Lancet Infect Dis. 15 (5), 581-614 (2015).
- Spinelli, L., Carpentier, S., Montanana Sanchis, F., Dalod, M., Vu Manh, T. P. BubbleGUM: automatic extraction of phenotype molecular signatures and comprehensive visualization of multiple Gene Set Enrichment Analyses. BMC Genomics. 16 (1), 814 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon