Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un protocolo para el uso de gen Set enriquecimiento análisis para identificar el modelo Animal adecuado para la investigación traslacional
En este artículo
Resumen
Proporcionamos un protocolo estandarizado para el uso del gen enriquecimiento conjunto análisis transcriptómico datos para identificar un modelo de ratón ideal para la investigación traslacional.
Este protocolo se puede utilizar con microarrays de ADN y datos de la secuencia de RNA y puede ampliarse aún más a otros datos ómicos si se dispone de datos.
Resumen
Estudios recientes que compararon datos transcriptómicos de enfermedades humanas con conjuntos de datos de modelos de ratón mediante técnicas de comparación de gen a gen tradicional dio lugar a conclusiones contradictorias con respecto a la pertinencia de modelos animales para traslacional investigación. Una de las principales razones para las diferencias entre análisis de expresión de genes diferentes es la arbitraria filtrado de genes diferencialmente expresados. Además, la comparación de solo genes entre diferentes especies y plataformas a menudo está limitada por variación técnica, llevando a interpretaciones erróneas de la con/discordancia entre los datos de modelos animales y humanos. Por lo tanto, se necesitan métodos estandarizados de análisis sistemático de datos. Para superar el gen subjetivo filtrado y comparaciones ineficaces de gen a gen, hemos demostrado recientemente que el análisis del enriquecimiento conjunto gene (GSEA) tiene el potencial para evitar estos problemas. Por lo tanto, hemos desarrollado un protocolo estandardizado para el uso de GSEA distinguir entre modelos animales apropiados e inapropiados para la investigación traslacional. Este protocolo no es adecuado para predecir cómo diseñar nuevos sistemas de modelo a priori–, ya que requiere de datos experimental de ómicas. Sin embargo, el protocolo describe cómo interpretar los datos existentes de manera estandarizada con el fin de seleccionar el modelo animal más conveniente, evitando innecesarios experimentos con animales y engañosa estudios traslacionales.
Introducción
Se utilizan modelos animales para estudiar enfermedades humanas, debido a su supuesta semejanza a los seres humanos en términos de genética, anatomía y fisiología. Por otra parte, los modelos animales a menudo sirven como guardianes a los tratamientos clínicos y pueden tener un impacto enorme en el éxito de la investigación traslacional. Una cuidadosa selección del modelo animal óptima puede reducir el número de estudios animales engañosos. Recientemente, la pertinencia de modelos animales para investigación traslacional ha sido discutida polémico, particularmente porque analizando el mismo conjuntos de datos obtenidos de enfermedades inflamatorias humanas y modelos relacionados con ratón condujo a conclusiones contradictorias 1,2. Esta discusión revela un problema fundamental en análisis de datos ómicos: métodos estandarizados para el análisis de datos sistemáticos son necesarios para reducir la selección del gene parcial y aumentar la robustez de las comparaciones entre las especies 3.
Tradicionalmente, el análisis de los datos de la transcriptómica (y otras ómicas) se realiza a nivel de gen único e incluye un primer paso de selección genética basado en parámetros estrictos de corte (por ejemplo, cambio de doblez > 2.0, valor de p < 0.05). Sin embargo, el ajuste de parámetros de corte inicial a menudo es subjetiva, arbitraria y no biológicamente justificada y puede llevar a conclusiones opuestas1,2. Además, selección génica inicial generalmente restringe el análisis a unos cuantos muy arriba y o genes y por lo tanto no es sensible a la mayoría de los genes que se expresan diferencialmente en menor medida.
Con el surgimiento de la era de la genómica en el 2000s temprano y el conocimiento creciente de caminos biológicos y contextos, enfoques estadísticos alternativos fueron desarrollados permite eludir las limitaciones de análisis nivel de gen único. Gen set enriquecimiento análisis (GSEA)4, que es uno de los métodos ampliamente aceptados para el análisis de los datos de la transcriptómica, hace uso de los grupos definido a priori de genes (por ejemplo, señalización de vías, de localización proximal en un cromosoma etcetera.). GSEA primero mapas de todos los genes detectados sin filtrar a los conjuntos gen prevista (p.ej., caminos), independientemente de su cambio individual de expresión. Así, este enfoque incluye también moderadamente regulados genes que de otra manera se perderían con análisis nivel del solo-gene. El cambio aditivo de expresión dentro de conjuntos de genes se realiza posteriormente utilizando corriente suma estadística.
A pesar de su amplio uso en la investigación médica, GSEA y enriquecimiento conjunto relacionados con enfoques no evidentemente toman en cuenta para el análisis de datos complejos ómicas. Aquí, describimos un protocolo para comparar datos de omics de muestras humanas con las de modelos de ratón con el fin de identificar el modelo ideal para los estudios traslacionales. Se demuestra la aplicabilidad del protocolo basado en una colección de modelos de ratón que se utilizan para la mímica humanos trastornos inflamatorios. Sin embargo, este gasoducto de análisis no se limita a humanos-ratón comparaciones y preguntas de investigación modificable a otros.
Protocolo
1. descargar el Software de GSEA y la base de datos de firmas moleculares
- ir a la página oficial del Instituto amplio GSEA (http://software.broadinstitute.org/gsea/index.jsp) y registrarte para tener acceso al software de GSEA herramienta y la base de datos de firmas moleculares (MSigDB).
- Descargar la aplicación de escritorio de javaGSEA o una opción de software alternativo (por ejemplo, escritura de R).
Nota: Todas las opciones de aplicación exactamente el mismo algoritmo. El software GSEA es libremente disponible para individuos en academia e industria con fines de investigación internos. - Para más detalles sobre el software GSEA a la Web de documentación (http://software.broadinstitute.org/cancer/software/gsea/wiki/index.php/Main_Page) y la guía del usuario GSEA (http://software.broadinstitute.org/gsea/doc/ GSEAUserGuideFrame.html).
- descargar la base de datos de firmas moleculares (MSigDB) del sitio web de GSEA acceder a colecciones set de genes individuales.
Nota: El MSigDB es una colección de conjuntos de genes anotados para su uso con el software GSEA u otros propósitos. Gen sets pueden dividirse según la señalización de vías, términos de ontología del gen, motivos cis-reguladoras, firmas experimentales y otros. Genes de lo MSigDB siempre son nombrados por su símbolo oficial del gene de HUGO (organización humana del genoma). Para la comparación de la regulación de la vía entre un determinado desorden humano y ratón diferente modelos lo es recomendable que descargues el ' todos los caminos canónicos, símbolos del gene ' archivo (c2.cp.v5.2.symbols.gmt). Este archivo compone de conjuntos de genes que fueron anotadas y organizadas en vías de señalización por KEGG 5 , 6, Reactome 7 , 8 y BioCarta 9. la cadena ' v5.2 ' representa la información de versión de la colección. Asegúrese de descargar la última versión de los archivos. El MSigDB está disponible para individuos en academia e industria con fines de investigación interna. No es necesario descargar el MSigDB, si la conexión a internet se proporciona durante el análisis. En este caso el MSigDB puede ser elegido directamente en la interfaz de usuario GSEA. - DNA descargar chip archivos de anotaciones (array) del sitio web de GSEA traducir identificadores específicos de matriz sonda general símbolos de los gene de HUGO (p. ej., Mouse430_2.chip).
Nota: No es necesario descargar las anotaciones de chip de ADN, si la conexión a internet se proporciona durante el análisis. En este caso las anotaciones de chip de ADN pueden ser elegidas directamente en la interfaz de usuario GSEA. El protocolo también puede utilizarse con datos de la secuencia de RNA. En este caso, no es necesario descargar los archivos de anotación. En su lugar, utilice la herramienta preranked de GSEA para analizar los datos de expresión génica (ver paso 4.12).
2. Descargar datos de expresión genética Experimental para el desorden humano y modelos animales apropiados
- identificar estudios de expresión (transcriptómica) de genética experimental para la enfermedad humana de elección (por ejemplo, perfiles de expresión génica de leucocitos derivan de pacientes con trastorno séptico, GSE9960).
- De la misma, búsqueda de la animal varios modelos que deben ser comparados con los estudios en seres humanos (por ejemplo, perfiles de expresión génica de las células sanguíneas derivadas de ratones después de la inyección de Staphylococcus aureus (S. aureus), GSE20524). En este paso usar el conocimiento previo para la preselección de modelos animales que podrían ser adecuados para mímico la situación humana.
- Para este propósito se refieren a la literatura y bases de datos como el Omnibus de expresión del Gene (GEO) base de datos 10 o ArrayExpress 11 y descargar los datos de la transcriptómica normalizado de interés. Guardar los datos como archivos de texto en el disco duro local. La base de datos de GEO, se recomienda la descarga de archivos de texto delimitado por tabuladores de la serie matriz. También tomar nota de la plataforma (tipo matriz) utilizado para este estudio, ya que esta información es necesaria para la traducción de los identificadores de matriz específico sonda general símbolos del gene de HUGO.
Nota: Asegúrese de suficiente memoria para el almacenamiento de datos, como conjuntos de datos de transcriptómica generalmente comprenden varios cientos MB.
3. Datos de manejo y formato
- antes de importar datos de expresión genética experimental en la herramienta de software GSEA, considerar la estructura de datos requerida. Para cada estudio manualmente crear dos archivos diferentes: 1) un archivo de datos de expresión génica que contiene valores de medición de varios genes y muestras y 2) un archivo de fenotipo que contiene las etiquetas de muestra para muestras individuales (por ejemplo, a grupos de tratamiento) del grupo.
Para más detalles y datos estructura opciones ir a la página de formato de datos GSEA (http://software.broadinstitute.org/cancer/software/gsea/wiki/index.php/Data_formats).
Nota: Generalmente, todas las formas de transcriptómica datos son compatibles con el protocolo, incluyendo experimentos de microarrays de DNA, RNA-seq o estudios de ChIP-seq. En caso de utilizar experimentos de microarrays de ADN, el archivo de datos de expresión génica debe contener identificador de sondeo de matriz específico o símbolos del gene de HUGO para cada gen (sonda identificadores serán traducidos a símbolos del gene de HUGO durante el análisis, ver pasos 1.5 y 4.10). En caso de utilizar datos de RNA-seq o ChIP-seq, grupo calculado manualmente métrica de datos de expresión genética (por ejemplo, cociente malo de grupo) debe utilizarse en lugar de datos individuales. Estas métricas grupo entonces deben ser analizadas con la herramienta preranked de GSEA (ver paso 4.12). Datos de expresión genética deben ser normalizada como de costumbre antes de importar en el software GSEA. El tipo de normalización (por ejemplo, cuartil o spline cúbico) generalmente se deja al investigador.
Archivo de - datos de expresión genética: Utilice el formato texto delimitado por tabulaciones (*.txt) para describir un conjunto de datos de expresión como se muestra en la figura 1A. Vea también el archivo de ejemplo compatibles GSE20524_expression.txt.
Nota: El archivo de datos de expresión génica contiene valores de expresión de todos los genes detectables (o sondas), también para los genes que no pueden ser expresados diferencialmente. Por lo tanto el archivo normalmente consta de muchos miles de genes. Se organiza como se muestra en la figura 1A. La primera línea contiene el nombre de etiqueta (por ejemplo, el gen símbolo o identificador de la sonda) seguido por el identificador para cada muestra del conjunto de datos (por ejemplo, muestra 1, muestra 2 etc.). El resto del archivo contiene los valores de expresión para cada uno de los genes y para cada muestra del conjunto de datos. La herramienta de software GSEA realiza cálculos de indicadores de grupo (por ejemplo, el cociente malo de grupo o relación señal a ruido), por lo tanto, se recomienda incluir datos de cada muestra individual. Alternativamente, es posible utilizar métricas grupo exterior calculado para los datos de expresión genética (ver figura 1B).
Archivo - fenotipo: crear un archivo independiente para definir y etiquetar grupos que conforman las muestras individuales como se describe en < clase fuerte= "xfig" > Figura 2. Utilice espacios o tabulaciones para separar los campos. Guardarlo en un formato de archivo CLS (definición de la clase de C++). Vea también el archivo de ejemplo compatibles GSE20524_pheno_infection.cls.
Nota: La primera línea contiene el número total de muestras y aún más el número de grupos ( figura 2). Mientras que el número de muestras debe corresponderse con el archivo de datos de expresión génica (véase 3.2), el número de grupos depende el diseño del estudio. El tercer campo de la primera línea es siempre ' 1 '.
La segunda línea en un archivo CLS contiene el nombre de cada grupo. La línea debe comenzar con un signo de libra (#) seguido de un espacio ( figura 2).
La tercera línea contiene una etiqueta de grupo para cada muestra. La etiqueta de grupo puede ser un número arbitrario o texto. Es sólo el orden de las etiquetas que determina la Asociación de cada muestra a los grupos: la primera etiqueta que se utiliza es asignada al primer grupo en la segunda línea; la segunda etiqueta única es asignada al grupo de segundo y así sucesivamente. Asegúrese de que cada muestra del mismo grupo tiene la misma etiqueta en este paso, y que el número de etiquetas es el mismo que el número de muestras especificado en la primera línea. Por último, guarde el archivo como archivo de texto delimitado por tabulaciones (*.txt) y cambiar manualmente la extensión de nombre de archivo a (*.cls). - (opcionales) archivos de base de datos conjunto de Gene: definir conjuntos gen personalizados. Utilice el formato de archivo delimitado por tabuladores de GMT (gen matriz transpuesta) de conjuntos de genes como se muestra en la figura 3. Ver también el archivo de ejemplo compatibles Gene_sets_Inflammation_BIOCARTA_KEGG_REACTOME.gmt.
Nota: Definir sistemas gene personalizado puede ser útil por ejemplo restringir el gen establece análisis de enriquecimiento a las vías de especial interés (p. ej., Inmunología para estudios de sepsis), o novo de definir conjuntos de genes propios (por ejemplo, activados y inhibir genes en estudios que tienen que ser comparados). El archivo se organiza como se muestra en la figura 3. En el formato GMT, cada fila representa un conjunto de genes ( figura 3). Cada conjunto de genes es descrito por un nombre, una descripción y los genes en el conjunto de genes. La primera columna contiene nombres determinados de gen único. La segunda línea puede contener opcionalmente una descripción del conjunto de genes. Las columnas siguientes contienen los nombres de gene (oficial HUGO gene los símbolos) del correspondiente conjunto de genes. Por último, guarde el archivo como archivo de texto delimitado ficha (*.txt) y cambiar manualmente la extensión de nombre de archivo (* .gmt).
4. Realizar la GSEA
- Abra la herramienta de software GSEA (véase 1.2).
- , Haga clic en el ' datos de la carga ' botón en el lado izquierdo de la ventana principal ( Figura 4A). Se abrirá una nueva pestaña para la importación de los archivos de datos necesarios ( Figura 4B). Buscar en la pestaña para el archivo de datos (*.txt) de expresión de genes (ver 3.2), el archivo de fenotipo (*.cls) (véase 3.3) y, opcionalmente, a los conjuntos gen personalizado (* .gmt) archivo ( Figura 4B).
- En el caso de GSEA no se puede conectar a internet, también carga el MSigDB descargado (* .gmt) archivos (p. ej., c2.cp.v5.2.symbols.gmt de caminos, ver 1.4) y la DNA chip anotaciones (matriz) (* .chip) archivos (p. ej., Mouse430_2.chip, Ver 1.5). Datos importados con éxito aparecen en la ' datos de la carga ' sección ( figura 4).
Nota: Cada estudio de expresión génica se debe analizar con GSEA individualmente. La comparación entre dos estudios (e.g. desorden humano vs modelo de ratón) se realizará en el paso 5.
- En el caso de GSEA no se puede conectar a internet, también carga el MSigDB descargado (* .gmt) archivos (p. ej., c2.cp.v5.2.symbols.gmt de caminos, ver 1.4) y la DNA chip anotaciones (matriz) (* .chip) archivos (p. ej., Mouse430_2.chip, Ver 1.5). Datos importados con éxito aparecen en la ' datos de la carga ' sección ( figura 4).
- Haga clic en el ' GSEA ejecutar ' botón en el lado izquierdo de la ventana principal. Se abrirá una nueva pestaña para definir los parámetros para el análisis ( figura 4). La ficha se divide en tres partes: campos obligatorios, campos básicos y campos avanzados.
- En los campos requeridos, primero elige la expresión dataset cargado en el paso 4.2 ( figura 4).
- Elegir el gen establece la base de datos, en el sitio conectado o desde el archivo de conjunto de gene importado manualmente ( figura 4).
- Editar las etiquetas de fenotipo para seleccionar los grupos de muestras que deben compararse entre sí (p. ej., S. aureus tratamiento vs control sano) ( figura 4).
- Conjunto de colapso a los símbolos del gene (= verdadero) para los identificadores de la punta de prueba del conjunto de datos de expresión oficial HUGO gene símbolos utilizados en la base de datos de conjuntos de genes. Seleccione false, si el conjunto de datos de expresión ya contiene símbolos del gene de HUGO ( figura 4).
- Ajustar el número de permutaciones para defecto en 1.000 ( figura 4).
Nota: Para un número mayor el tiempo de computación aumentará considerablemente. - Cambiar el tipo de permutación para ' gen set ', puesto que la permutación de fenotipo sólo se recomienda cuando hay más de siete muestras en cada fenotipo ( figura 4).
- Por último, seleccione la plataforma chip utilizada para generar los datos de expresión genética, desde el sitio Web conectado o del ADN manualmente importado viruta archivo de anotaciones (matriz) ( figura 4).
Nota: Este paso sólo es necesario, si se utilizan identificadores de sonda en el dataset cargado de expresión. - En los campos básicos de edición al menos la sección nombre de análisis y resultados de la operación de guardar en esta carpeta para encontrar de nuevo el archivo de resultados ( figura 4). Además, otros parámetros estadísticos se pueden cambiar. Para más detalles sobre los parámetros y la sección campos avanzados por favor ir a la guía de usuario GSEA (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html).
- (Opcional): en el caso métricas de grupo externamente calculados para la expresión del gene datos (p. ej., proporción media grupo) tienen que ser utilizados en lugar de datos individuales, utilice la herramienta preranked de GSEA. El análisis entonces se realizará en base a una simple lista de genes asignados con métricas de grupo previamente calculadas que se utilizan para clasificar los genes. Después de cargar el archivo de expresión de genes alternativos a la barra de navegación principal y haga clic en Herramientas/GseaPreranked. Del mismo modo, se abrirá una nueva pestaña para configurar los parámetros para el análisis ( figura 4E).
Nota: Usando la herramienta preranked de GSEA está recomendada para estudios que no tienen datos de expresión genética específica de muestra individual. Podría ser el caso si las estadísticas especiales o procedimientos de normalización fueron realizados en los datos a valores promedio de grupo en lugar de datos individuales. Se recomienda el uso de la herramienta preranked de GSEA para datos de secuencias de RNA. Normalizar los datos de expresión de la secuencia de RNA y calcular métricas de grupo para las muestras (p. ej., registro del doble cambio), que pueden utilizarse para clasificar los genes según su expresión. - Haga clic en el ' funcionamiento ' botón en la parte inferior derecha de la ventana.
Nota: El análisis entonces puede tomar hasta varios minutos dependiendo de la velocidad de computación. Seguir el progreso del análisis en la sección de informes GSEA en la parte inferior izquierda de la ventana. Después de terminar el análisis, el estado ' éxito ' aparece en la sección de informes GSEA.
Análisis de - clic en el tuvo éxito en la sección de informes GSEA para abrir los resultados del análisis.
Nota: Se abrirá un nuevo menú de navegación en una ventana del explorador que resume todos los resultados y ajustes de los parámetros ( figura 5). El superior comprenden dos secciones del menú de navegación gen set resultados de enriquecimiento para los grupos definidos (por ejemplo, enriquecimiento en S. aureus tratadas las muestras o muestras sanas de control). Las primeras líneas de ambas secciones muestran un resumen de los resultados de la estadística. Conjuntos de genes que se enriquecen significativamente a una tasa de falso-descubrimiento (FDR) por debajo del 25% se consideran enriquecido en la siguiente interpretación. Más detalles sobre la interpretación de los análisis pueden encontrarse en la guía del usuario GSEA (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html). - Click sobre el resultado de enriquecimiento detallada en formato de excel para exportar los resultados del análisis a una hoja de cálculo ( figura 6A). Exportar los resultados enriquecimiento detallados en excel por separado para ambos fenotipos ( figura 5) y a los datos de resultados en un archivo de hoja de cálculo. Para la posterior comparación entre datos de expresión genética de varios estudios, mantener por lo menos el nombre del conjunto de genes (columna A), su enriquecimiento normalizado puntuación (NES) (columna F) y su valor FDR (tarifa falsa del descubrimiento) (columna H) ( Figura 6B ).
Nota: El archivo de hoja de cálculo contiene gran cantidad de datos para cada uno de los analizados conjunto de genes, incluyendo el nombre del conjunto de genes (columna A), su tamaño (es decir, el número de genes detectados en los datos de expresión génica, columna D), su NES (una medida cuantitativa de la dirección y grado de enriquecimiento, columna F), su valor nominal p (sin corregir, columna G) y su valor FDR (corregido por múltiples pruebas de hipótesis, columna H). Para más detalles sobre la interpretación, consulte la guía de usuario GSEA (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html). - Repita que el gen establece análisis de enriquecimiento (pasos 4.1 a 4.15) para el segundo estudio (p. ej., S. aureus GSE9960) y para todos los estudios adicionales que deben compararse entre sí. Incluyen todos los estudios clínicos en humanos y modelos de ratón diferentes como sea posible para identificar el modelo de ratón óptimo para la pregunta de investigación traslacional.
5. Comparación de los resultados de GSEA
- para identificar el modelo animal óptima para mímico el situación humana de comparar los resultados GSEA de todos los estudios entre sí. Utilizar las puntuaciones de enriquecimiento y los valores FDR para clasificar los caminos (Gen sets) como activa (NES > FDR 0, < 25%), inhibición (NES < FDR 0, < 25%) o ninguno de ambos (FDR > 25%). Para cada comparación de dos estudios, contar el número de realizaciones de las nueve posibles combinaciones de la regulación de la vía como se indica en una tabla de contingencia de 3 x 3 ( Figura 7A).
- Evaluar la correlación entre los dos estudios por el cálculo del valor predictivo positivo (VPP) y el valor predictivo negativo (VPN), que es por definición la parte de las vías que el mismo Reglamento (activados o inhibidos) en dos estudios de .
- Calcular VPP y VPN según las siguientes fórmulas (1) y (2):
(1)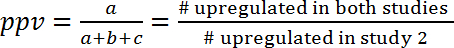
(2)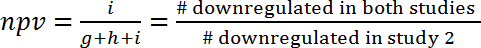
Nota: Puesto que la superposición podría ser pura coincidencia, el VPP y el VPN deben ser más en comparación con los valores de lo esperados por azar. Este enfoque permite la estimación de la cantidad de información que puede obtenerse de un estudio para predecir los efectos en otro estudio. Por ejemplo, si los procesos de regulación en dos modelos eran independientes uno del otro (sólo se superponen por casualidad), y si en el primer modelo de 10% de las vías de alza, que el ppv para el segundo modelo sería 10% y había no más ganar de inf ormación. En el otro lado, si ambos modelos estaban relacionados por mecanismos de regulación común, entonces el ppv (y van) sería significativamente mayores de lo esperado por azar. Por ejemplo, para la predicción de los cambios de expresión génica durante la sepsis humana (GSE9960) de efectos en un modelo murino de inyección aureus de S. (GSE20524), el VPP es de 43% (6/(6+8+0)) y el VPN es del 61% (11/(0+7+11)). En otras palabras, el 43% de las vías activadas en el modelo murino S. aureus inyección (GSE20524) también se activan durante la sepsis humana (GSE9960). Asimismo, 61% de las vías de inhibición en el modelo murino S. aureus inyección (GSE20524) también se inhiben durante la sepsis humana (GSE9960) ( figura 7B). también se pueden determinar VPP y VPN para la inversa de la constelación (que significa predicción de estudio 1 estudio 2).
- Calcular VPP y VPN según las siguientes fórmulas (1) y (2):
- Para calcular la superposición por casualidad consulte la tabla de contingencia de 3 x 3 ( figura 7) y calcular ppvchance y npvchance según las siguientes fórmulas (3) y (4):
(3) < img alt = "Ecuación 3" src = "/files/ ftp_upload/55768/55768eq3.jpg"/ >
(4)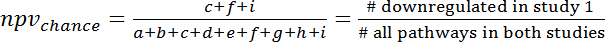
Nota: por ejemplo, para la predicción de los cambios de expresión génica durante la sepsis humana (GSE9960) de efectos en un ratón S. aureus modelo de la inyección (GSE20524) el ppvchance es del 13% (8/64) y el npvchance y el 22% (14/64). - Calcular la ganancia del ppv vs oportunidad restando ppvchance de ppv. Calcular en consecuencia para el VPN:
(5)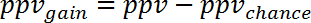
(6)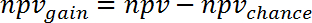
Nota: por ejemplo, para la predicción del gene expresión cambia durante sepsis humana (GSE9960) de efectos en un modelo murino de inyección aureus de S. (GSE20524) el cambio en ppv y npv vs oportunidad es + 30% (43% - 13%) y 39% (61% - 22%), respectivamente. - Calcular la ganancia de información que puede obtenerse del estudio 2 estudio 1 con un promedio de ppvgain y npvgain:
(7)
- Utilice la tabla de contingencia definida en el paso 5.1 de un par de estudios (study1.pathway, study2.pathway) para calcular el valor de p por un test Chi-cuadrado
Almacenar los datos de la tabla de contingencia en una matriz X. Realice la prueba de Chi-cuadrada, p. ej., por el uso de la R la función chisq.test.
Nota: por ejemplo, comparar el estudio de las sepsis humana (GSE9960) con un ratón S. aureus inyección modelo (GSE20524) muestra una superposición estadísticamente significativa en la regulación de la vía inflamatoria:
> $ chisq.test(X,simulate.p.value=F) p.Value
3.82e-07
6. Identificación del modelo Animal óptima
- compara la GSEA resultados para todas las combinaciones de los estudios que fueron seleccionados para el análisis.
Nota: Se recomienda también para comparar los estudios en humanos (similar) unos a otros así como los estudios animales diferentes entre sí. Esta comparación puede proporcionar la penetración en la variación interespecífica de los estudios clínicos (o trastornos) y los diferentes modelos animales. Se espera que los estudios clínicos deben mostrar que un compromiso aceptable y una información importante ganan, porque de otro modo, los estudios clínicos podrían ser demasiado heterogéneos para encontrar un modelo animal que puede imitar la situación humana. En este caso, se recomienda incluir sólo los estudios en humanos que son similares entre sí para la identificación de modelos animales adecuados. - Ordenar todas las combinaciones por la ganancia de información (paso 5.5). Para la comparación de muchos conjuntos de datos, utilice una matriz y visualizar los resultados por el uso de un heatmap color o similar ( figura 8).
- Seleccione el modelo animal con la mayor ganancia de información. Con el fin de evaluar la importancia de la ganancia de información, también considerar la prueba de ji cuadrado (paso 5.6).
Nota: Los modelos animales sólo pueden considerarse como adecuado si la ganancia de información es sustancial y el valor de p de la prueba chi-cuadrado es por debajo del nivel de significación. Umbrales User-Defined general dependerá de varios factores: 1) el conocimiento previo estudio de la transferibilidad de los resultados del modelo animal a los seres humanos (e.g. similar fisiología), 2) el beneficio esperado para los seres humanos por un presunto éxito, 3) la práctica aplicabilidad de ese experimento animal y 4) el esperado dolor, el sufrimiento o el daño infligido en el laboratorioanimales de oratorio.
Resultados
El flujo de trabajo GSEA y capturas de pantalla de datos de ejemplares se demuestran. La figura 1 muestra el archivo de datos de expresión de genes que contiene los datos transcriptómicos de interés. Cada estudio un archivo descriptivo del fenotipo se requieren se muestra en la figura 2. Conjuntos de genes anotado (p.ej., caminos) se definen en el archivo de base de datos conjunto de gene (figura ...
Discusión
Durante mucho tiempo se han aplicado modelos animales para la investigación de los mecanismos de la enfermedad y el desarrollo de nuevas estrategias terapéuticas. Sin embargo, escepticismo con respecto a la predecibilidad de los modelos animales empieza a difundirse después de fracasos de ensayos12. Además, polémicas discusiones sobre estrategias apropiadas para el análisis e interpretación de datos ómicos grande de ensayos preclínicos fueron levantadas por opuestas conclusiones extraída...
Divulgaciones
Los autores declaran que no tienen intereses financieros que compiten.
Agradecimientos
Este trabajo fue financiado por el Instituto Federal alemán para la evaluación del riesgo (BfR).
Materiales
| Name | Company | Catalog Number | Comments |
| Excel | Microsoft Corporation |
Referencias
- Seok, J., et al. Genomic responses in mouse models poorly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 110 (9), 3507-3512 (2013).
- Takao, K., Miyakawa, T. Genomic responses in mouse models greatly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 112 (4), 1167-1172 (2015).
- Weidner, C., Steinfath, M., Opitz, E., Oelgeschläger, M., Schönfelder, G. Defining the optimal animal model for translational research using gene set enrichment analysis. EMBO Mol Med. 8 (8), 831-838 (2016).
- Subramanian, A., et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. Proc Natl Acad Sci U S A. 102 (43), 15545-15550 (2005).
- Kanehisa, M., Sato, Y., Kawashima, M., Furumichi, M., Tanabe, M. KEGG as a reference resource for gene and protein annotation. Nucleic Acids Res. 44 (D1), D457-D462 (2016).
- Kanehisa, M., Goto, S. KEGG: kyoto encyclopedia of genes and genomes. Nucleic Acids Res. 28 (1), 27-30 (2000).
- Fabregat, A., et al. The Reactome pathway Knowledgebase. Nucleic Acids Res. 44 (D1), D481-D487 (2016).
- Croft, D., et al. The Reactome pathway knowledgebase. Nucleic Acids Res. 42 (Database issue), D472-D477 (2014).
- Nishimura, D. BioCarta. Biotech Software & Internet Report. 2 (3), 117-120 (2001).
- Edgar, R., Domrachev, M., Lash, A. E. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Res. 30 (1), 207-210 (2002).
- Kolesnikov, N., et al. ArrayExpress update--simplifying data submissions. Nucleic Acids Res. 43 (Database issue), D1113-D1116 (2015).
- Cohen, J., et al. Sepsis: a roadmap for future research. Lancet Infect Dis. 15 (5), 581-614 (2015).
- Spinelli, L., Carpentier, S., Montanana Sanchis, F., Dalod, M., Vu Manh, T. P. BubbleGUM: automatic extraction of phenotype molecular signatures and comprehensive visualization of multiple Gene Set Enrichment Analyses. BMC Genomics. 16 (1), 814 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados