Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein Protokoll für die Verwendung von Gen Set Anreicherung Analyse zur Ermittlung von geeigneten Tiermodell für die translationale Forschung
In diesem Artikel
Zusammenfassung
Wir bieten ein standardisiertes Protokoll für die Nutzung der Genanalyse Set Anreicherung von transkriptomischen Daten, um eine ideale Maus-Modell für die translationale Forschung zu identifizieren.
Dieses Protokoll kann mit DNA-Microarray und RNA Sequenzierungsdaten verwendet werden und kann weiter mit anderen Daten der Omics verlängert werden, wenn Daten vorhanden sind.
Zusammenfassung
Neuere Studien, die transkriptomischen-Datasets von menschlichen Krankheiten mit Datensätzen von Mausmodellen mit traditionellen gen-gen-Vergleich Techniken verglichen führten zu widersprüchlichen Schlussfolgerungen bezüglich der Relevanz von Tiermodellen für Translationale Forschung. Ein wesentlicher Grund für die Unterschiede zwischen den verschiedenen Genexpressionsanalysen ist die willkürliche Filterung differentiell exprimierten Gene. Außerdem ist der Vergleich einzelner Gene zwischen verschiedenen Arten und Plattformen oft durch technische Varianz, was zu Fehlinterpretationen der Con/Discordance zwischen Daten aus menschlichen und tierischen Modelle beschränkt. Daher sind standardisierte Ansätze zur systematischen Analyse erforderlich. Um subjektive gen filtern und unwirksam gen-gen-Vergleiche zu überwinden, wir kürzlich gezeigt, dass Set Bereicherung Genanalyse (GSEA) das Potenzial hat, diese Probleme zu vermeiden. Daher entwickelten wir ein standardisiertes Protokoll für die Verwendung von GSEA zu passenden und unpassenden Tiermodelle für die translationale Forschung unterscheiden. Dieses Protokoll eignet sich nicht, wie neue Modellsysteme–von vornherein, design vorherzusagen, da es bestehende experimentelle Omics Daten erfordert. Jedoch beschreibt das Protokoll die vorhandene Daten in einer standardisierten Weise zu interpretieren, um die am besten geeigneten Tiermodell, damit unnötige Tierversuche zu vermeiden und irreführende translationale Studien zu wählen.
Einleitung
Tiermodelle sind am meisten benutzt, um menschliche Krankheiten wegen ihrer vermeintlichen Ähnlichkeit zu den Menschen in Bezug auf die Genetik, Anatomie und Physiologie zu studieren. Darüber hinaus Tiermodellen oft dienen als Pförtner, Therapien und haben einen großen Einfluss auf den Erfolg der translationalen Forschung. Sorgfältige Auswahl der optimalen Tiermodell reduzieren die Zahl der Tierversuche irreführend. Vor kurzem hat die Relevanz von Tiermodellen für die translationale Forschung kontrovers diskutiert, vor allem, weil die Analyse der gleichen Datasets gewonnenen menschlichen entzündliche Erkrankungen und damit verbundene Mausmodellen zu widersprüchlichen Schlussfolgerungen geführt, 1,2. Diese Diskussion zeigte ein grundlegendes Problem während der Omics Datenanalyse: standardisierte Ansätze zur systematischen Datenanalyse sind erforderlich, um einseitige Genselektion zu senken und die Robustheit der Interspezies Vergleiche 3erhöhen.
Traditionell, die Analyse der Transkriptom (und andere Omics-Daten) erfolgt auf der Single-gen-Ebene und enthält einen ersten Schritt der Genselektion basierend auf strengen Cut-off-Parametern (z.B., Falte Änderung > 2.0, p-Wert < 0,05). Allerdings die Einstellung der ersten Cut-off Parameter oft ist subjektiv, willkürlich und nicht biologisch begründete, und sogar zu anderen Schlussfolgerungen1,2führen kann. Darüber hinaus erste Genselektion in der Regel schränkt die Analyse auf ein paar sehr up- und herunterreguliert Gene und ist somit nicht empfindlich genug, um die Mehrzahl der Gene enthalten, die differentiell in geringerem Maße zum Ausdruck gebracht wurden.
Mit dem Aufstieg der Genomik-Ära in den frühen 2000er Jahren und die zunehmende Kenntnis der biologischer Signalwege und Kontexte wurden alternative statistische Ansätze entwickelt, die erlaubt, die Grenzen der einzelnen Level Genanalysen zu umgehen. Gen gesetzt Bereicherung Analyse (GSEA)4, das ist eine der weithin akzeptierten Methoden für die Analyse von Transkriptom Daten, nutzt a Priori definierte Gruppen von Genen (z. B.Signalwege, proximalen Ort auf einem Chromosom etc..). GSEA Karten zuerst alle erkannten ungefilterte Gene auf die beabsichtigte Gen-Sets (z.B. Wege), unabhängig von ihren individuellen Ausdruck. Dieser Ansatz beinhaltet somit auch mäßig regulierten Gene, die sonst mit Single-Level-Genanalysen verloren wären. Die additive Veränderung Ausdruck in Gen-Sets erfolgt anschließend über laufende Summe Statistiken.
Trotz seiner breite Verwendung in der medizinischen Forschung sind GSEA und verwandte Set Bereicherung Ansätze nicht selbstverständlich für die Analyse von komplexen Omics Daten berücksichtigt. Hier beschreiben wir ein Protokoll für den Vergleich von Omics Daten aus humanen Proben mit denen von Mausmodellen um das ideale Modell für Translationale Studien zu identifizieren. Wir zeigen die Anwendbarkeit des Protokolls basiert auf einer Sammlung von Maus-Modellen, die für die Nachahmung menschlicher entzündlicher Erkrankungen verwendet werden. Allerdings diese Pipeline Analyse beschränkt sich nicht auf Mensch-Maus-Vergleiche und ist amendable zur weiteren Forschungsfragen.
Protokoll
1. GSEA-Software und die molekulare Signaturen-Datenbank herunterladen
- auf der offiziellen Website der GSEA breite Institute (http://software.broadinstitute.org/gsea/index.jsp) gehen und registrieren, um Zugriff auf die GSEA Software erhalten Werkzeug und die molekulare Signaturen-Datenbank (MSigDB).
- Herunterladen, die JavaGSEA desktop-Applikation oder eine alternative Softwareoption (z. B. R-Skript).
Hinweis: Alle Optionen setzen genau den gleichen Algorithmus. Die GSEA Software ist frei verfügbar für Personen aus Wissenschaft und Industrie für interne Recherchezwecke. - Für weitere Details über die GSEA-Software zur Dokumentation Website (http://software.broadinstitute.org/cancer/software/gsea/wiki/index.php/Main_Page) und das GSEA Benutzerhandbuch (http://software.broadinstitute.org/gsea/doc/ gehen GSEAUserGuideFrame.html).
- die molekulare Signaturen-Datenbank (MSigDB) von der GSEA Website Zugriff auf einzelne gen Set Sammlungen herunterladen.
Hinweis: Die MSigDB ist eine Sammlung von kommentierten Gen-Sets für den Einsatz mit der GSEA-Software oder andere Zwecke. Gen-Sets lassen sich einteilen nach Signalisierung Wege, gen Ontologie Begriffe, Cis-regulatorische Motive, experimentelle Signaturen und andere. Gene aus der MSigDB sind immer von ihren offiziellen HUGO (Human Genome Organisation) gen Symbol benannt. Für der Vergleich von Weg-Verordnung zwischen einem bestimmten menschlichen Störung und andere Maus-Modelle empfiehlt sich zum download der ' alle kanonischen Wege, gen Symbole ' Datei (c2.cp.v5.2.symbols.gmt). Diese Datei besteht aus Gen-Sets, die wurden kommentiert und in Signalwege KEGG 5 , 6, Reactome 7 , 8 und BioCarta geordnet 9. die Zeichenfolge ' V5. 2 ' die Versionsinformationen der Sammlung darstellt. Achten Sie darauf, die neueste Version der Dateien herunterladen. Die MSigDB ist frei verfügbar für Personen aus Wissenschaft und Industrie für interne Recherchezwecke. Es ist nicht notwendig, um die MSigDB herunterzuladen, wenn Internet-Verbindung während der Analyse zur Verfügung gestellt wird. In diesem Fall kann die MSigDB direkt in der Benutzeroberfläche GSEA gewählt werden. - Download DNA-chip (Array) Anmerkungen Dateien von der Website GSEA, Array-spezifische Sonde-IDs allgemeine HUGO gen-Symbole (z.B. Mouse430_2.chip) zu übersetzen.
Hinweis: Es ist nicht notwendig, laden Sie die DNA-Chip-Anmerkungen, wenn Internet-Verbindung während der Analyse zur Verfügung gestellt wird. In diesem Fall können die DNA-Chip-Anmerkungen direkt innerhalb der Benutzeroberfläche GSEA gewählt werden. Das Protokoll kann auch mit RNA Sequenzierungsdaten verwendet werden. In diesem Fall ist es nicht erforderlich, Annotation Dateien herunterzuladen. Verwenden Sie stattdessen die GSEA-preranked-Tool für die Analyse der Genexpressionsdaten (siehe Schritt 4.12).
2. Experimentelle Genexpressionsdaten für menschliche Störung und geeigneter Tiermodelle herunterladen
- identifizieren experimentell (Transkriptom) Genexpressionsstudien für die menschliche Störung der Wahl (z.B., gen Expressionsprofile von Leukozyten abgeleitet von Patienten mit septischen Erkrankung, GSE9960).
- Desgleichen, Suche nach mehreren Tier-Modelle, die mit den menschlichen Studien (z. B. gen Expressionsprofile von Blut Zellen aus Mäusen nach Injektion von Staphylococcus Aureus (S. Aureus), GSE20524) verglichen werden sollen. In diesem Schritt verwenden die Vorkenntnisse für die Vorauswahl von Tiermodellen, die für die Nachahmung der menschlichen Situation geeignet sein könnte.
- Für diesen Zweck beziehen sich auf Literatur und Datenbanken wie Gene Expression Omnibus (GEO) 10 oder ArrayExpress 11-Datenbank und laden Sie die normalisierten Transkriptom-Daten von Interesse. Speichern Sie die Daten als Text-Dateien auf der lokalen Festplatte. Für die GEO-Datenbank empfiehlt sich der Download von Tabulatorzeichen Serie Matrix Textdateien. Beachten Sie auch die Plattform (Array-Typ) für diese Studie verwendet, da diese Informationen für die Array-spezifische Sonde Bezeichner zu allgemeinen HUGO gen Symbole übersetzen benötigt wird.
Hinweis: Darauf achten genügend Speicher zum Speichern von Daten, da Transkriptom Datensätze in der Regel mehrere hundert MB umfassen.
3. Daten Handling und Formatierung
- vor dem Importieren der experimentellen Genexpressionsdaten in das Software-Tool GSEA, betrachten Sie die erforderlichen Daten-Struktur. Für jede Studie zwei verschiedene Dateien manuell erstellen: (1) ein Gen Ausdruck Datendatei enthält Messwerte für verschiedene Gene und Proben, und (2) ein Phänotyp mit Beispiel-Etiketten um einzelne Proben (z. B. Behandlung Gruppen) zu gruppieren.
Weitere Details und Daten Struktur Optionen finden Sie unter der GSEA Datenseite Format (http://software.broadinstitute.org/cancer/software/gsea/wiki/index.php/Data_formats).
Hinweis: Im Allgemeinen sind alle Formen von Transkriptom Daten kompatibel mit dem Protokoll, einschließlich DNA-Microarray Experimente, RNA-Seq oder ChIP-Seq-Studien. Bei der Verwendung von DNA-Microarray Experimente, sollte die gen-Ausdruck-Datendatei enthalten, Array-spezifische Sonde Bezeichner oder HUGO gen Symbole für jedes Gen (Sonde, die Kennungen zu HUGO gen Symbole während der Analyse, siehe Schritte 1,5 und 4.10 übersetzt werden). Bei der Verwendung von RNA-Seq oder ChIP-Seq-Daten, sollte anstelle von einzelnen Beispieldaten manuell berechneten Gruppe Metriken für die Genexpressionsdaten (z. B. Gruppe mittlere Verhältnis) verwendet werden. Diese Gruppe Kennzahlen sollten dann mit dem GSEA preranked Tool analysiert werden (siehe Schritt 4.12). Genexpressionsdaten haben wie üblich normalisiert werden, bevor Sie in die GSEA-Software importieren. Die Art der Normalisierung (z.B., Quartil oder kubische Spline) ist in der Regel dem Forscher links. - Genexpressionsdaten Datei: verwenden Sie das Tabulatorzeichen Text-Dateiformat (*.txt) für einen Ausdruck Dataset zu beschreiben, wie in Figur 1A dargestellt. Siehe auch die unterstützten Beispieldatei GSE20524_expression.txt.
Hinweis: Die gen-Ausdruck-Datendatei enthält Ausdruckswerte für alle nachweisbaren Gene (oder Sonden), auch für Gene, die nicht differenziell ausgedrückt werden könnte. Die Datei umfasst daher in der Regel viele Tausende von Genen. Es ist organisiert wie in Figur 1A dargestellt. Die erste Zeile enthält den Label-Namen (z.B., gen symbol oder Sonde ID) gefolgt von der Bezeichner für jede Probe in das Dataset (z. B. Probe 1, Probe 2 etc.). Der Rest der Datei enthält Ausdruckswerte für jedes der Gene und für jede Probe in das Dataset. Die GSEA-Software-Tool führt Berechnungen für Gruppe Kennzahlen (z.B., Gruppe mittlere Verhältnis oder Signal-Rausch-Verhältnis), daher es wird empfohlen, die Daten für jede einzelne Probe enthalten. Alternativ ist es möglich, extern berechneten Gruppe Metriken für Genexpressionsdaten verwenden (siehe Abbildung 1 b). - Phänotyp Datei: Erstellen Sie eine separate Datei für Festlegung und Kennzeichnung von Gruppen, die Einzelproben umfassen, wie in dargestellt < starke Klasse= "Xfig" > Abbildung 2. Verwenden Sie Leerzeichen oder Tabulatoren zum Trennen der Felder. Speichern Sie es in einem Dateiformat CLS (C++-Klassendefinition). Siehe auch die unterstützten Beispieldatei GSE20524_pheno_infection.cls.
Hinweis: Die erste Zeile enthält die Gesamtzahl der Proben und weiter die Anzahl der Gruppen ( Abbildung 2). Während die Anzahl der Samples der gen-Ausdruck-Datendatei entsprechen sollte (vgl. 3.2), die Anzahl der Gruppen hängt das Studiendesign. Das dritte Feld der ersten Zeile ist immer ' 1 '.
Die zweite Zeile in einer CLS-Datei enthält den Namen für jede Gruppe. Die Zeile sollte beginnen mit einem Nummernzeichen (#) gefolgt von einem Leerzeichen ( Abbildung 2).
Die dritte Zeile enthält eine Gruppenbezeichnung für jede Probe. Die Gruppenbezeichnung kann eine beliebige Zahl oder Text. Es ist nur die Reihenfolge der Etiketten, die die Vereinigung von jeder Probe den Gruppen bestimmt: das erste Etikett verwendet, erhält die erste Gruppe in der zweiten Zeile; die zweite eindeutige Bezeichnung erhält die zweite Gruppe und so weiter. Stellen Sie sicher, dass jede Probe derselben Gruppe bei diesem Schritt die gleiche Bezeichnung hat, und dass die Anzahl der Etiketten ist identisch mit der Anzahl der Stichproben, die in der ersten Zeile angegeben. Abschließend speichern Sie die Datei als tabulatorgetrennte Textdatei (*.txt) und manuell ändern Sie die Dateinamenerweiterung in (*.cls). - (optional) Gen Set Datenbankdateien: benutzerdefinierte Gen-Sets definieren. Verwenden Sie das Tabulatorzeichen GMT (Gene Matrix umgesetzt) Dateiformat für Gen-Sets wie in Abbildung 3 dargestellt. Siehe auch die unterstützten Beispieldatei Gene_sets_Inflammation_BIOCARTA_KEGG_REACTOME.gmt.
Hinweis: Benutzerdefinierte Gen-Sets definieren kann nützlich sein, zum Beispiel, das Gen zu beschränken soll die Anreicherung Analyse Wege von besonderem Interesse (z. B. Immunologie Signalisierung für Sepsis-Studien) oder de Novo eigenen Gen-Sets definieren (z.B., aktiviert und gehemmt, Gene in Studien, die verglichen werden). Die Datei wird organisiert, wie in Abbildung 3 dargestellt. Im GMT-Format stellt jede Zeile eine gen-Set ( Abbildung 3). Jedes Gen-Set wird durch einen Namen, eine Beschreibung und die Gene im gen Satz beschrieben. Die erste Spalte enthält einzigartige gen Namen festlegen. Die zweite Zeile kann optional eine Beschreibung des gen-Sets enthalten. Die folgenden Spalten enthalten die gen-Namen (offizielle HUGO gen Symbole) der das entsprechende Gen-Set. Abschließend speichern Sie die Datei als Registerkarte durch Trennzeichen getrennte Textdatei (*.txt) und manuell ändern Sie die Dateinamenerweiterung (* .gmt).
4. Durchführung der GSEA
- GSEA-Software-Tool zu öffnen (siehe 1.2).
- Klicken Sie auf die ' Daten laden ' Schaltfläche auf der linken Seite des Hauptfensters ( Abb. 4A). Eine neue Registerkarte öffnet sich für den Import der erforderlichen Daten-Dateien ( Abbildung 4 b). Stöbern Sie in der neuen Registerkarte mit der gen Ausdruck Datendatei (*.txt) gespeichert (siehe 3.2), der Phänotyp (*.cls) Datei (siehe 3.3) und gegebenenfalls auf die benutzerdefinierte Gen-Sets (* .gmt) Datei ( Abbildung 4 b).
- Für den Fall, dass GSEA mit dem Internet verbinden kann nicht laden auch die heruntergeladene MSigDB (* .gmt) Dateien (z.B. c2.cp.v5.2.symbols.gmt für Wege, siehe 1.4) und die DNA-chip (Array) Anmerkungen (* .chip) Dateien (z. B. Mouse430_2.chip, (siehe 1.5). Erfolgreich importierte Daten erscheinen in der ' Daten laden ' Abschnitt ( Abbildung 4).
Hinweis: Jede gen-Ausdruck-Studie muss mit GSEA einzeln analysiert werden. Der Vergleich zwischen beiden Studien (z.B. menschliche Störung vs. Mausmodell) erfolgt bei Schritt 5.
- Für den Fall, dass GSEA mit dem Internet verbinden kann nicht laden auch die heruntergeladene MSigDB (* .gmt) Dateien (z.B. c2.cp.v5.2.symbols.gmt für Wege, siehe 1.4) und die DNA-chip (Array) Anmerkungen (* .chip) Dateien (z. B. Mouse430_2.chip, (siehe 1.5). Erfolgreich importierte Daten erscheinen in der ' Daten laden ' Abschnitt ( Abbildung 4).
- Klicken Sie auf die ' laufen GSEA ' Schaltfläche auf der linken Seite des Hauptfensters. Eine neue Registerkarte wird öffnen, um die Parameter für die Analyse ( Abbildung 4). Die Registerkarte ist in drei Teile gegliedert: erweiterte Felder Pflichtfelder, Basisfelder und.
- In die erforderlichen Felder, wählen Sie zuerst den Ausdruck Dataset geladen in Schritt 4.2 ( Abbildung 4).
- Wählen Sie die Gen Datenbank setzt, der angeschlossenen Website oder aus der Set-Datei manuell importierte gen ( Abbildung 4).
- Bearbeiten der Phänotyp Etiketten um Gruppen von Proben auszuwählen, die (z. B. S. Aureus vs. gesunde Therapiekontrolle) miteinander verglichen werden sollen ( Abbildung 4).
- Zusammenbruch Dataset zu gen-Symbole (= true) um die Sonde Bezeichner im Ausdruck-Dataset zu offiziellen HUGO gen Symbolen in der Gen-Sets-Datenbank zu übersetzen. Wählen Sie falsch, wenn der Ausdruck Dataset bereits HUGO gen Symbole ( Abbildung 4 enthält).
- Legen Sie die Anzahl der Permutationen, Standardeinstellung bei 1.000 ( Abbildung 4).
Hinweis: Für höhere Zahlen die Rechenzeit wird ansteigen. - Ändern der Permutation Typ, ' gen Satz ', da Phänotyp Permutation nur empfohlen ist, wenn es mehr als sieben Proben in jeder Phänotyp ( Abbildung 4 gibt).
- Wählen Sie schließlich die Chip-Plattform für die Generierung der Genexpressionsdaten verwendet, entweder von der angeschlossenen Website oder von der manuell importierte DNA chip (Array) Anmerkungen Datei ( Abbildung 4).
Hinweis: Dieser Schritt ist nur erforderlich, wenn die Sonde Bezeichner im hochgeladenen Ausdruck Dataset verwendet werden. - In den grundlegenden Bereichen bearbeiten mindestens Abschnitt Analysenamen und Speichern ergibt sich in diesem Ordner um die Ergebnisdatei ( Abbildung 4) wieder zu finden. Darüber hinaus können weitere statistische Parameter geändert werden. Für weitere Details zu den Parametern und der Abschnitt "erweiterte Felder" Sie bitte zur GSEA-Anleitung (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html gehen).
- (Optional): In den Fall extern berechneten Gruppe Metriken für die Genexpression Daten (z. B. Gruppe mittlere Verhältnis) anstelle von einzelnen Beispieldaten verwendet werden müssen, verwenden Sie das GSEA preranked Werkzeug. Die Analyse erfolgt dann anhand einer einfachen Liste von Genen mit vorberechneten Gruppe Metriken, die verwendet werden, um die Gene Rang zugewiesen. Nach dem Laden der alternativen gen-Ausdruck-Datei in der Hauptnavigation und klicken Sie auf Tools/GseaPreranked. In ähnlicher Weise wird eine neue Registerkarte öffnen, für die Einstellung der Parameter für die Analyse ( Abb. 4E).
Hinweis: Mit dem GSEA preranked Werkzeug für Studien empfiehlt sich, die nicht einzelnen Probe-spezifische Genexpressionsdaten verfügen. Das könnte der Fall sein, wenn spezielle Statistiken oder Normalisierung Verfahren auf die Daten zu Gruppe Mittelwerte anstelle von einzelnen Beispieldaten durchgeführt wurden. Mit dem GSEA preranked Werkzeug empfiehlt sich für RNA Sequenzierungsdaten. Die RNA Sequenzierung Ausdruck Daten normalisieren und berechnen Gruppe Metriken für die Proben (z. B. Protokoll der Falte Änderung), das verwendet werden können, um die Gene nach ihren Ausdruck Rang. - Klicken Sie auf die ' Run ' Schaltfläche am unteren rechten Rand des Fensters.
Hinweis: Die Analyse kann dann abhängig von der Rechengeschwindigkeit einige Minuten dauern. Verfolgen Sie den Fortschritt der Analyse im Abschnitt GSEA "Berichte" auf der linken unteren Rand des Fensters. Nach Abschluss der Analysis den Status ' Erfolg ' erscheint im Abschnitt Berichte GSEA. - Klicken Sie auf die erfolgreiche Analyse im Abschnitt GSEA Berichte, die Analyseergebnisse zu öffnen.
Hinweis: Ein neue Navigations-Menü öffnet sich in einem Browserfenster, das fasst alle Ergebnisse und Parameter-Einstellungen ( Abbildung 5). Die oberen zwei Abschnitte des Navigationsmenüs umfassen gen stellen Bereicherung Ergebnisse für die definierten Gruppen (z. B. Anreicherung in S. Aureus behandelt Proben oder gesunde Kontrollproben). Die ersten Zeilen der beiden Abschnitte zeigen die statistischen Ergebnisse im Überblick. Gen-Sets, die mit einer False Discovery Rate (FDR) unter 25 % wesentlich bereichert sind gelten als in der folgenden Interpretation bereichert. Weitere Einzelheiten über die Auslegung der Analyse finden Sie im Benutzerhandbuch GSEA (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html). - Klicken Sie auf die detaillierte Bereicherung Ergebnisse im excel-Format, die Analyse-Ergebnisse in einer Tabelle ( Abb. 6A) exportieren. Exportieren Sie die detaillierte Bereicherung Ergebnisse in excel, getrennt für beide Phänotypen ( Abbildung 5) und beitreten Sie die Ergebnisdaten in einer Tabellenkalkulationsdatei. Für nachfolgende Vergleich zwischen Genexpressionsdaten aus mehreren Studien erhalten mindestens den Namen des gen-Sets (Spalte A), seine normalisierte Anreicherung score (NES) (Spalte F) und dessen FDR (false Discovery Rate) Wert (Spalte H) ( Abbildung 6 b ).
Hinweis: Die Kalkulationstabellendatei enthält riesige Daten für jede der untersuchten gen festgelegt, einschließlich des eingestellten gen (Spalte A), seine Größe (d. h. die Anzahl der Gene entdeckt in der Genexpressionsdaten, Spalte D), seine NES (ein quantitatives Maß für die Richtung und Umfang der Bereicherung, Spalte F), seinen nominalen p-Wert (unkorrigiert, Spalte G) und Nutzwert FDR (für mehrere Hypothesentests, Spalte H korrigiert). Für weitere Einzelheiten über die Auslegung der GSEA Benutzerhandbuch (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html) siehe. - Wiederholen, dass das Gen Anreicherung Analyse (Stufen 4.1 bis 4.15) festgelegt, für die zweite Studie (z. B. S. Aureus GSE9960) und für alle weiteren Studien, die miteinander verglichen werden sollen. Geben Sie möglichst viele klinische Studien am Menschen und verschiedenen Mausmodellen wie möglich, die optimale Maus-Modell für die translationale Forschungsfrage zu identifizieren.
5. Vergleicht man die Ergebnisse der GSEA
- , optimale Tiermodell für die Nachahmung der menschlichen Situation vergleichen die GSEA Ergebnisse aller Studien miteinander zu identifizieren. Verwenden der Bereicherung Partituren und die FDR-Werte, um die Wege (Gen-Sets) zu klassifizieren, da aktiviert (NES > 0, FDR < 25 %), gehemmt (NES < 0, FDR < 25 %) oder keines von beiden (FDR > 25 %). Für jeden Vergleich von zwei Studien, die Anzahl der Realisierungen von neun möglichen Kombinationen von Weg-Verordnung durch eine 3 x 3 Kontingenztafel ( Abb. 7A).
- Bewerten die Korrelation zwischen zwei Studien durch Berechnung der positive Vorhersagewert (Ppv) und die negativen prädiktiven Wert (Npv), die definitionsgemäß die Wege, die den gleichen Verordnung gehört (aktiviert oder gehemmt) in zwei Studien zeigen .
- Berechnen Ppv und Kapitalwert nach folgenden Formeln (1) und (2):
(1)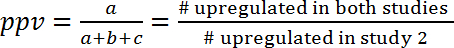
(2)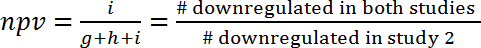
Hinweis: Da die Überlappung rein zufällig sein kann, haben die Ppv und dem Kapitalwert weiter die Werte, denen sich durch Zufall verglichen werden soll. Dieser Ansatz erlaubt die Schätzung der Höhe der Informationen, die aus einer Studie für die Vorhersage der Effekte in einer anderen Studie gewonnen werden können. Zum Beispiel wenn Regulationsprozesse in zwei Modelle waren unabhängig voneinander (und nur durch Zufall überlappen) und 10 % der Wege im ersten Modell hochreguliert, wurden als die Ppv, das zweite Modell auch 10 wäre % und es keine zusätzlichen gab gewinnen der inf ormationen verwendet. Auf der anderen Seite wenn beide Modelle durch gemeinsame Regulationsmechanismen verbunden waren wäre dann die Ppv (und Npv) deutlich größer, als durch Zufall zu erwarten. Die Ppv ist beispielsweise für die Vorhersage von Veränderungen der Genexpression in menschlichen Sepsis (GSE9960) von Effekten in einem murinen S. Aureus Injektion Modell (GSE20524), 43 % (6/(6+8+0)) und dem Kapitalwert beträgt 61 % (11/(0+7+11)). Das heißt, sind 43 % der aktivierten Wege in der S. Aureus Injektion Mausmodell (GSE20524) auch bei menschlichen Sepsis (GSE9960) aktiviert. In ähnlicher Weise 61 % der gehemmten Wege in der S. Aureus Injektion Mausmodell (GSE20524) sind auch gehemmt, während menschliche Sepsis (GSE9960) ( Abb. 7 b). PPV und Npv können auch für die umgekehrte Konstellation (das heißt die Vorhersage aus Studie 1, 2 zu studieren) ermittelt werden.
- Berechnen Ppv und Kapitalwert nach folgenden Formeln (1) und (2):
- Um die Überlappung zu berechnen durch Zufall beziehen sich auf die 3 x 3 Kontingenztabelle ( Abbildung 7) und berechnen Sie Ppvchance und Npvchance nach den folgenden Formeln (3) und (4):
(3) < Img Alt = "Gleichung 3" Src = "/files/ ftp_upload/55768/55768eq3.jpg"/ >
(4)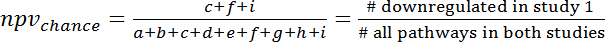
Hinweis: Z. B. für die Vorhersage von Veränderungen der Genexpression in menschlichen Sepsis (GSE9960) aus Effekten in einem murinen S. Aureus Injektion-Modell (GSE20524) der Ppvchance ist 13 % (8/64) und die Npvchance ist und 22 % (14/64). - Die Gewinn der Ppv vs. Chance durch Subtrahieren Ppvchance von Ppv berechnen. Entsprechend berechnen für die Npv:
(5)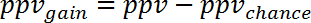
(6)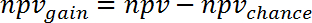
Hinweis: Z. B. für die Vorhersage des Gens Ausdruck ändert während menschliche Sepsis (GSE9960) von Effekten in einem murinen S. Aureus Injektion Modell (GSE20524) die Veränderung der Ppv und Npv vs. Chance ist + 30 % (43 % - 13 %) und + 39 % (61 % - 22 %), beziehungsweise. - Berechnung der Zugewinn an Information, die sich aus Studie 2 Studie 1 kann durch Mittelung, Ppvgain und Npvgain:
(7)
- verwenden die Kontingenztabelle im Schritt 5.1 ein paar Studien (study1.pathway, study2.pathway) zur Berechnung des p-Wertes durch einen Chi-Quadrat-Test definiert
Speichern Sie die Daten von der Kontingenztabelle in einer Matrix X. führen Sie die Chi-Quadrat-Test, z.B., durch Verwendung der R-Funktion chisq.test.
Hinweis: Z. B. Vergleich der ausgewählten menschlichen Sepsis-Studie (GSE9960) mit einem murinen S. Aureus Injektion Modell (GSE20524) zeigt eine statistisch signifikante Überschneidung im entzündlichen Weg Verordnung:
> chisq.test(X,simulate.p.value=F)$ p.Value
3.82e-07
6. Ermittlung der optimalen Tier-Modell
- vergleichen die GSEA ergibt sich für alle Kombinationen von den Studien, die für die Analyse ausgewählt wurden.
Hinweis: Es wird auch empfohlen, der (ähnlich wie) Studien am Menschen zueinander sowie die verschiedenen tierexperimentellen Studien miteinander zu vergleichen. Dieser Vergleich kann geben Einblick in die Wiederkäuer Varianz der klinischen Studien (oder Störungen) und die verschiedenen Tiermodellen. Es wird erwartet, dass die klinische Studien zeigen sollte, dass eine akzeptable Überlappung und eine aussagekräftige Informationen gewinnen, weil sonst die klinischen Studien möglicherweise zu heterogen, einem Tiermodell zu finden, die die menschliche Situation nachahmen kann. In diesem Fall es wird empfohlen, nur Studien am Menschen enthalten, die für die Identifizierung von geeigneten Tiermodellen einander ähnlich sind. - Sortieren alle Kombinationen durch den Zugewinn an Information (Schritt 5.5). Für den Vergleich von vielen Datensätzen, verwenden Sie eine Matrix und visualisieren Sie die Ergebnisse durch eine farbige Heatmap oder dergleichen ( Abbildung 8).
- Wählen Sie aus das Tiermodell mit der höchsten Zugewinn an Information. Bei der Beurteilung der Bedeutung der Zugewinn an Information berücksichtigen auch die Chi-Quadrat-Test (Schritt 5,6).
Hinweis: Tiermodelle sollte nur betrachtet werden je nach Bedarf wenn der Zugewinn an Information beträchtlich ist und der p-Wert des Chi-Quadrat-Tests unter dem Signifikanzniveau ist. User-Defined Schwellenwerte werden im Allgemeinen von mehreren Faktoren abhängig: (1) die Vorstudie wissen über die Übertragbarkeit der Ergebnisse von Tiermodell für den Menschen (z.B. ähnliche Physiologie), (2) der zu erwartende Nutzen für den Menschen von einem mutmaßlichen Erfolg (3) die praktische Anwendbarkeit dieser Tierversuch und 4) die erwarteten Schmerzen, leiden oder Schäden zugefügt, LaborOratorium Tiere.
Ergebnisse
Die GSEA Workflow und Screenshots von beispielhaften Daten werden vorgeführt. Abbildung 1 zeigt die gen-Ausdruck-Datendatei, die die transkriptomischen relevanten Daten enthält. Für jede Studie eine beschreibende Phänotyp-Datei erforderlich ist, die in Abbildung 2dargestellt. Kommentierte Gen-Sets (z.B. Wege) sind in der gen-Set Datenbank-Datei (Abbildung 3) definiert.
Diskussion
Tiermodelle wurden lange für die Erforschung von Krankheitsmechanismen und die Entwicklung neuer therapeutischer Strategien angewendet. Skepsis gegenüber der Prädiktivität von Tiermodellen begann jedoch nach Versagen der klinischen Studien12zu verbreiten. Darüber hinaus wurden Kontroverse Diskussionen über geeignete Strategien zur Analyse und Interpretation der großen Omics Daten aus präklinischen Studien von entgegengesetzten Schlussfolgerungen aus den gleichen Daten nach Anwendung von un...
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessenkonflikte.
Danksagungen
Diese Arbeit wurde durch das deutsche Bundesinstitut für Risikobewertung (BfR) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Excel | Microsoft Corporation |
Referenzen
- Seok, J., et al. Genomic responses in mouse models poorly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 110 (9), 3507-3512 (2013).
- Takao, K., Miyakawa, T. Genomic responses in mouse models greatly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 112 (4), 1167-1172 (2015).
- Weidner, C., Steinfath, M., Opitz, E., Oelgeschläger, M., Schönfelder, G. Defining the optimal animal model for translational research using gene set enrichment analysis. EMBO Mol Med. 8 (8), 831-838 (2016).
- Subramanian, A., et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. Proc Natl Acad Sci U S A. 102 (43), 15545-15550 (2005).
- Kanehisa, M., Sato, Y., Kawashima, M., Furumichi, M., Tanabe, M. KEGG as a reference resource for gene and protein annotation. Nucleic Acids Res. 44 (D1), D457-D462 (2016).
- Kanehisa, M., Goto, S. KEGG: kyoto encyclopedia of genes and genomes. Nucleic Acids Res. 28 (1), 27-30 (2000).
- Fabregat, A., et al. The Reactome pathway Knowledgebase. Nucleic Acids Res. 44 (D1), D481-D487 (2016).
- Croft, D., et al. The Reactome pathway knowledgebase. Nucleic Acids Res. 42 (Database issue), D472-D477 (2014).
- Nishimura, D. BioCarta. Biotech Software & Internet Report. 2 (3), 117-120 (2001).
- Edgar, R., Domrachev, M., Lash, A. E. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Res. 30 (1), 207-210 (2002).
- Kolesnikov, N., et al. ArrayExpress update--simplifying data submissions. Nucleic Acids Res. 43 (Database issue), D1113-D1116 (2015).
- Cohen, J., et al. Sepsis: a roadmap for future research. Lancet Infect Dis. 15 (5), 581-614 (2015).
- Spinelli, L., Carpentier, S., Montanana Sanchis, F., Dalod, M., Vu Manh, T. P. BubbleGUM: automatic extraction of phenotype molecular signatures and comprehensive visualization of multiple Gene Set Enrichment Analyses. BMC Genomics. 16 (1), 814 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten