JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
유전자를 사용 하 여 프로토콜 변환 연구에 대 한 적절 한 동물 모델을 식별 하기 위해 농축 분석 설정
요약
우리는 유전자 세트 농축 분석 transcriptomic 데이터를 변환 연구를 위한 이상적인 마우스 모델의 사용에 대 한 표준화 된 프로토콜을 제공 합니다.
이 프로토콜 DNA microarray 및 RNA 시퀀싱 데이터와 함께 사용할 수 있습니다 그리고 더 확장 될 수 있습니다 다른 omics 데이터에 데이터를 사용할 수 있는 경우.
초록
Transcriptomic 전통적인 유전자-유전자 비교 기술을 사용 하 여 마우스 모델에서 데이터 집합으로 인간의 질병의 데이터 집합을 비교 하는 최근 연구 결과 대 한 동물 모델의 관련성에 관한 모순 된 결론에 변환 연구입니다. 다른 유전자 표정 분석 사이의 불일치에 대 한 주요 이유는 차동 표현한 유전자의 임의 필터링입니다. 또한, 다른 종과 자주 플랫폼 사이의 단일 유전자의 비교는 인간과 동물 모델에서 데이터 간의 콘/부조화의 오해로 이어지는 기술 차이 의해 제한 됩니다. 따라서, 체계적인 데이터 분석에 대 한 표준화 된 접근법이 필요 합니다. 극복 하기 위해 주관적인 유전자 필터링 및 효과 없는 유전자-유전자 비교, 우리는 최근 유전자 세트 농축 분석 (GSEA) 이러한 문제를 방지 하려면 가능성이 있다 시연. 따라서, 우리는 GSEA 변환 연구에 대 한 적절 하 고 부적 절 한 동물 모델을 구분 하기 위해 사용에 대 한 표준화 된 프로토콜을 개발 했다. 이 프로토콜은 기존 실험 omics 데이터 필요-선험적 새로운 모델 시스템을 설계 하는 방법을 예측할 적당 하다. 그러나, 프로토콜 표준화 된 방식으로 가장 적합 한 동물 모델, 따라서 불필요 한 동물 실험을 피하 하 고 오해의 소지가 변환 연구를 선택 하기 위해 기존 데이터를 해석 하는 방법을 설명 합니다.
서문
동물 모델 널리 유전학, 해부학 및 생리학 인 간에 게 그들의 가정된 유사 때문에 인간의 질병을 연구 하는 데 사용 됩니다. 또한, 동물 모델 자주 임상 치료에 문지기로 봉사 하 고 변환 연구의 성공에 큰 영향을 미칠 수 있습니다. 최적의 동물 모델의 신중한 선택은 잘못 된 동물 연구의 수를 줄일 수 있습니다. 최근, 변환 연구를 위한 동물 모델의 관련성 논쟁 논의 되었습니다, 특히 때문에 모순 된 결론 주도 인간의 염증 성 질환과 관련 된 마우스 모델에서 얻은 동일한 데이터 집합을 분석 1,2. 이 토론 omics 데이터를 분석 하는 동안 근본적인 문제를 계시 했다: 체계적인 데이터 분석에 대 한 표준화 된 접근 interspecies 비교 3의 안정성을 향상 하 고 한쪽으로 치우친된 유전자 선택을 줄이기 위해 필요 하다.
전통적으로, transcriptomics 데이터 (및 기타 omics 데이터)의 분석 단일 유전자 수준에서 수행 되 고 엄격한 차단 매개 변수 기반 유전자 선택의 초기 단계를 포함 한다 (예를들면, 배 변경 > 2.0, p 값 < 0.05). 그러나, 초기 절단 매개 변수 설정을 종종 주관적, 임의적이 고 생물학 정당화 이며 반대 결론1,2으로 이어질 수 있다. 또한, 초기 유전자 선택은 일반적으로 몇 가지 매우 업 및 downregulated 유전자 분석을 제한 하 고 충분히 차동에 표현 된 유전자의 대다수를 포함 하는 구분 하지 않습니다 따라서.
2000 년대 초반에 게놈의 상승 및 생물 학적 경로 및 컨텍스트 증가 지식, 다른 통계 접근은 단일 유전자 수준 분석의 한계를 우회할 수 개발 되었다. 농축 분석 (GSEA)4, transcriptomics 데이터의 분석을 위해 광범위 하 게 허용된 방법 중 하나를 설정 하는 유전자, 유전자 (예를 들어, 경로, 염색체 등 인접 위치 신호.)의 한 선험적 정의 된 그룹을 사용. GSEA 처음 식에 그들의 개인적인 변화에 관계 없이 (예를 들어, 경로), 대상된 유전자 세트를 모든 검색된 필터링 되지 않은 유전자를 매핑합니다. 이 방법은 따라서 또한 알맞게 조절된 유전자를 그렇지 않으면 단일 유전자 수준 분석을 포함 한다. 유전자 집합 내의 식에 첨가제 변화 이후에 실행 합계 통계를 사용 하 여 수행 됩니다.
의료 연구에 있는 그것의 광범위 한 사용에 불구 하 고 GSEA 및 관련된 설정된 농축 접근은 하지 self-evidently 고려 복잡 한 omics 데이터의 분석에 대 한. 여기, 우리가 마우스 모델에서 인간의 샘플에서 omics 데이터 변환 연구에 대 한 이상적인 모델을 식별 비교 하기 위한 프로토콜을 설명 합니다. 우리 인간의 염증 성 질환을 흉내 낸에 사용 되는 마우스 모델의 컬렉션을 기반 프로토콜의 적용을 보여 줍니다. 그러나,이 분석 파이프라인 인간 마우스 비교를 제한 하지 않습니다 이며 더 amendable 연구 질문.
프로토콜
1. GSEA 소프트웨어 및 분자 서명 데이터베이스 다운로드
- 공식 GSEA 광범위 한 연구소 웹사이트 (http://software.broadinstitute.org/gsea/index.jsp)에 고 GSEA 소프트웨어에 대 한 액세스를 얻을 등록 도구 및 분자 서명 데이터베이스 (MSigDB).
- 다운로드 javaGSEA 데스크톱 응용 프로그램 또는 다른 소프트웨어 옵션 (예를 들어, R 스크립트).
참고: 모든 옵션은 정확히 같은 알고리즘을 구현합니다. GSEA 소프트웨어는 학계 및 업계 내부 연구 목적을 위해 개인에 게 자유롭게 제공. - GSEA 소프트웨어에 대 한 내용은 설명서 웹 사이트 (http://software.broadinstitute.org/cancer/software/gsea/wiki/index.php/Main_Page) 및 GSEA 사용자 가이드 (http://software.broadinstitute.org/gsea/doc/가 더 GSEAUserGuideFrame.html).
- 개별 유전자 설정된 컬렉션에 접근할 GSEA 웹사이트에서 분자 서명 데이터베이스 (MSigDB)를 다운로드.
참고: MSigDB는 GSEA 소프트웨어 또는 다른 목적으로 사용 하기 위해 주석된 유전자 집합의 모음입니다. 유전자 집합 신호 경로, 유전자 존재론 기간, cis 규제 모티프, 실험적인 서명 및 다른 사람에 따라 나눌 수 있습니다. 유전자는 MSigDB에서 항상 그들의 공식적인 휴고 (인간 게놈 조직) 유전자 기호로 이름이 지정 됩니다. 그것은 모델 주어진된 인간의 무질서와 다른 마우스 사이 통로 규제의 비교에 대 한 것이 좋습니다 다운로드는 ' 모든 정식 경로, 유전자 기호 ' 파일 (c2.cp.v5.2.symbols.gmt). 이 파일은 주석 되었고 KEGG 5 , 6, Reactome 7 , 8, BioCarta 신호 경로에 주최 유전자 세트 9. 문자열 ' v5.2 ' 컬렉션의 버전 정보를 나타냅니다. 파일의 최신 버전을 다운로드 해야 합니다. MSigDB는 학계 및 업계 내부 연구 목적을 위해 개인에 게 자유롭게 제공 됩니다. 그것은 분석 하는 동안 인터넷 연결이 제공 되는 경우 MSigDB, 다운로드 필요 하지 않습니다. 이 경우에 MSigDB 직접 GSEA 사용자 인터페이스 내에서 선택할 수. - 다운로드 DNA 칩 배열 전용 프로브 식별자 일반 휴고 유전자 기호 (예: Mouse430_2.chip)를 번역 하는 GSEA 웹사이트에서 (배열) 주석 파일.
참고: 그것은 필요 하지 DNA 칩 주석 다운로드 인터넷 연결 분석 하는 동안 제공 됩니다. 이 경우에 DNA 칩 주석 GSEA 사용자 인터페이스 내에서 직접 선택할 수 있습니다. 프로토콜은 또한 RNA 시퀀싱 데이터와 함께 사용할 수 있습니다. 이 경우에, 주석 파일을 다운로드 필요 하지 않습니다. 대신, GSEA preranked 도구를 사용 하 여 (단계 4.12 참조) 유전자 표현 데이터를 분석 하기 위한.
2. 인간의 무질서와 적절 한 동물 모델에 대 한 실험적인 유전자 표현 데이터를 다운로드
- (예를들면, 유전자 표현 프로필의 선택의 인간의 장애에 대 한 확인 실험 진 식 (transcriptomics) 연구 백혈구 정화 조 장애, GSE9960 가진 환자에서 파생).
- 마찬가지로, 여러 동물에 대 한 검색 모델 인간의 연구 (예를 들면, 황색 포도상구균 (S. 구 균), GSE20524의 주입 후 마우스에서 파생 하는 혈액 세포의 유전자 식 프로필)와 비교 하는. 이 단계에서 인간의 상황을 흉내 낸 적합 있을 동물 모델의 미리 선택에 대 한 사전 지식을 사용.
- 가이 문학을 참조 하 고 데이터베이스 유전자 식 옴니 버스 (지 오) 등 10 또는 ArrayExpress 11 데이터베이스 정규화 된 transcriptomics 데이터를 다운로드에 대 한. 로컬 하드 디스크에 텍스트 파일로 데이터를 저장. GEO 데이터베이스 탭 구분 시리즈 매트릭스 텍스트 파일의 다운로드 것이 좋습니다. 또한 주의 플랫폼 (배열 형식)의 일반 휴고 유전자 기호를 배열 전용 프로브 식별자 변환 하기 위해이 정보가 필요 때문에 그 연구에 대 한 사용.
주: transcriptomics 데이터 세트는 일반적으로 몇 백 MB를 구성 데이터 저장을 위한 충분 한 메모리를 확인 하십시오.
3. 데이터 처리 및 서식
- GSEA 소프트웨어 도구에 실험적인 유전자 표현 데이터를 가져오기 전에 필요한 데이터 구조를 고려 하십시오. 각 연구에 대 한 두 개의 다른 파일 수동으로 만들: 1) 유전자 표현 데이터 파일 포함 하는 다양 한 유전자 및 샘플, 측정 값 및 샘플 라벨 개별 샘플 (예를 들어, 치료 그룹) 그룹을 포함 2) 형 파일.
추가 세부 사항 및 데이터 구조에 대 한 옵션 페이지로 이동 GSEA 데이터 형식 (http://software.broadinstitute.org/cancer/software/gsea/wiki/index.php/Data_formats).
참고: 일반적으로, 모든 형태의 transcriptomics 데이터 프로토콜을 포함 하 여 DNA microarray 실험, RNA-seq 또는 칩 seq 연구와 호환 됩니다. DNA microarray 실험을 사용 하는 경우 유전자 표현 데이터 파일 배열 전용 프로브 식별자 또는 각 유전자 (프로브 식별자 휴고 유전자 기호 분석, 1.5, 4.10 참조 단계 동안 번역 될 것입니다)에 대 한 휴고 유전자 기호를 포함 해야 합니다. RNA-seq 또는 칩 seq 데이터를 사용 하는 경우 수동으로 계산된 그룹 메트릭 유전자 표현 데이터 (예: 그룹 평균 비율)에 대 한 개별 샘플 데이터 대신 사용 되어야 한다. 이러한 그룹 메트릭 GSEA preranked 도구와 분석 다음 합니다 (단계 4.12 참조). 유전자 표현 데이터 GSEA 소프트웨어로 가져오기 전에 평소 처럼 정규화 해야 합니다. 정규화 (예를 들어, 사분 위 수 또는 입방 스플라인)의 종류는 일반적으로 연구원에 남아. - 유전자 표현 데이터 파일: 탭으로 구분 된 텍스트 파일 (*.txt) 형식 사용 하 여 그림 1A에서 같이 식 데이터 집합을 설명 하기 위한. 지원 되는 예제 파일을 참고 GSE20524_expression.txt.
참고: 유전자 표현 데이터 파일 들어 있습니다 모든 감지 유전자 (또는 프로브) 식 값 또한 유전자 차동 표현 하지. 파일 따라서 일반적으로 유전자의 많은 수천을 함유 한다. 그것은 그림 1A에서 같이 구성 됩니다. 첫 번째 행 레이블 이름을 포함 (예를 들어, 유전자 기호 또는 프로브 ID) 뒤에 각 샘플 데이터 집합 (예를 들어, 샘플 1, 샘플 2 등)에 대 한 식별자. 파일의 나머지 각 유전자에 대 한 및 각 샘플 데이터 집합에서 식 값을 포함합니다. 그룹 메트릭 (예를 들어, 그룹 평균 비율 또는 신호 대 잡음 비율)에 대 한 계산을 수행 하는 GSEA 소프트웨어 도구, 따라서 각 개별 샘플 데이터를 포함 하려면 것이 좋습니다. 또는, 그것은 유전자 표현 데이터 ( 그림 1B) 외부 계산된 그룹 메트릭 사용 가능. - 형 파일: 정의 및 표시 그룹을 구성 하는 개별 샘플 에서처럼 대 한 별도 파일을 만들고 < 강한 클래스"xfig" = > 그림 2. 공백이 나 탭을 사용 하 여 필드를 구분 하. CLS (c + + 클래스 정의) 파일 형식으로 저장 합니다. 지원 되는 예제 파일을 참고 GSE20524_pheno_infection.cls.
참고: 첫 번째 줄은 샘플의 총 수와 그룹 ( 그림 2)의 수를 더 포함 되어 있습니다. 샘플 수 유전자 표현 데이터 파일에 해당 하는 동안 (3.2 참조) 그룹의 연구 설계에 따라 다릅니다. 첫 번째 줄의 세 번째 필드는 항상 ' 1 '.
CLS 파일의 두 번째 줄에는 각 그룹에 대 한 이름을 포함합니다. 선 공간 ( 그림 2) 뒤에 파운드 기호 (#)로 시작 한다.
세 번째 줄에는 각 샘플에 대 한 그룹 레이블을 포함 되어 있습니다. 임의의 숫자 또는 텍스트 그룹 레이블을 수 있습니다. 그것은 그룹에 각 샘플의 협회 결정 하는 레이블 주문만: 사용 하는 첫 번째 레이블; 두 번째 줄에 첫 번째 그룹에 할당 두 번째 고유 라벨 두 번째 그룹에 할당 됩니다. 같은 그룹의 각 샘플은 동일한 레이블을 사용 하 여이 단계에서 확인 레이블 수는 첫 번째 줄에 지정 된 샘플의 수와 동일 합니다. 마지막으로, 파일을 탭 구분 텍스트 파일 (*.txt)로 저장 하 고 (*.cls)에 파일 이름 확장명을 수동으로 변경. - (옵션) 유전자 데이터베이스 설정 파일: 사용자 지정 유전자 세트를 정의. 그림 3 에서처럼 유전자 집합에 대 한 탭으로 구분 된 GMT (유전자 매트릭스 Transposed) 파일 형식을 사용 합니다. 또한 지원 되는 예제 파일을 참조 하십시오 Gene_sets_Inflammation_BIOCARTA_KEGG_REACTOME.gmt.
참고: 사용자 지정 유전자 세트를 정의 유용한 예를 들어 유전자를 제한 설정할 수 농축 분석 특별 한 관심 (예를 들면, 패 혈 증 연구에 대 한 신호 면역학), 또는 자신의 유전자 세트를 정의 하는 데 노 보에 대 한 경로를 (예를 들어, 활성화 및 비교할 수 있는 연구에 유전자 억제). 파일은 그림 3에서 같이 구성 됩니다. 그리니치 표준시 형식에서 각 행 유전자 집합을 ( 그림 3)을 나타냅니다. 각 유전자 세트는 이름, 설명 및 유전자 세트에 유전자에 의해 설명 되어 있습니다. 첫 번째 열에는 독특한 유전자 세트 이름을 포함 되어 있습니다. 두 번째 줄은 선택적으로 유전자 세트의 설명을 포함할 수 있습니다. 다음 열에는 해당 유전자 세트의 유전자 이름 (공식 휴고 유전자 기호) 포함 되어 있습니다. 마지막으로, 파일을 탭 구분 된 텍스트 파일 (*.txt)로 저장 하 고 파일 이름 확장명을 수동으로 변경 (*.gmt).
4. 수행 하는 GSEA
- GSEA 소프트웨어 도구를 열고 (1.2 참조).
- 클릭에 ' 데이터 로드 ' 주 창 ( 그림 4A)의 왼쪽에 버튼. 새로운 탭 ( 그림 4B) 필요한 데이터 파일을 가져오기 위해 열 것 이다. 유전자 표현 데이터 (*.txt) 파일에 새 탭에서 탐색 (3.2 참조), 형 (*.cls) 파일 (3.3 참조) 하 고, 필요에 따라 사용자 지정 유전자 세트를 (*.gmt) 파일 ( 그림 4B).
- GSEA는 인터넷에 연결할 수 없는 경우에 또한 로드 한다 다운로드 MSigDB (*.gmt) 파일 (예를 들어, c2.cp.v5.2.symbols.gmt 통로, 1.4 참조) 및 DNA 칩 (배열) 주석 (*.chip) 파일 (예: Mouse430_2.chip, 1.5 참조). 성공적으로 가져온된 데이터에 표시는 ' 데이터 로드 ' 섹션 ( 그림 4C).
참고: 각 진 식 연구 되어야 분석 GSEA와 개별적으로. 두 개의 연구 (예: 마우스 모델 대 인간의 장애) 사이 비교 5 단계에서 수행 됩니다.
- GSEA는 인터넷에 연결할 수 없는 경우에 또한 로드 한다 다운로드 MSigDB (*.gmt) 파일 (예를 들어, c2.cp.v5.2.symbols.gmt 통로, 1.4 참조) 및 DNA 칩 (배열) 주석 (*.chip) 파일 (예: Mouse430_2.chip, 1.5 참조). 성공적으로 가져온된 데이터에 표시는 ' 데이터 로드 ' 섹션 ( 그림 4C).
- 클릭에 ' 실행 GSEA ' 메인 윈도우의 왼쪽에 버튼. 새로운 탭 ( 그림 4D) 분석에 대 한 매개 변수를 설정 하려면 열 것 이다. 탭은 세 부분으로 세분화 됩니다: 필수 필드, 기본 필드 및 고급 필드.
- 필수 필드에 처음 단계 4.2 ( 그림 4D)에서 로드 식 데이터 집합 선택.
- 수동으로 가져온된 유전자 설정된 파일 ( 그림 4D) 또는 연결 된 웹 사이트에서 데이터베이스를 설정 하는 유전자는, 선택.
- 서로 (예를 들면, S. 구 균 치료와 건강 한 컨트롤)를 비교 하는 샘플 그룹 선택 형 레이블을 편집 ( 그림 4D).
- 유전자 기호 데이터 집합 붕괴 (= true) 유전자 집합 데이터베이스에 사용 된 공식 휴고 유전자 기호를 식 데이터 집합에서 프로브 식별자를 번역 하기 위하여. 식 데이터 집합 휴고 유전자 기호 ( 그림 4D)에 이미 포함 되어 있는 경우 false 선택.
- 1000 ( 그림 4D) 수를 순열의 기본 설정은 설정.
참고: 높은 숫자에 대 한 컴퓨팅 시간 늘어난다 상당히. - 순열 형식 변경 ' 유전자 세트 ', 모든 표현 형 ( 그림 4D)에 이상 7 샘플 때 표현 형 순열 좋습니다만.
- 마지막으로, 유전자 표현 데이터를 생성 하는 데 사용 하는 칩 플랫폼 선택, 연결 된 웹 사이트에서 또는 수동으로 가져온된 DNA 칩 (배열) 주석 파일 ( 그림 4D).
참고:이 단계는 필요한 프로브 식별자 업로드 된 식 데이터 집합에 사용 되는 경우만. - 기본 필드에서 적어도 결과 파일 ( 그림 4D)을 다시 찾으려고 분석 이름 및 이 폴더에는 저장 결과 섹션 편집. 또한, 추가 통계 매개 변수 바뀔 수 있다. 추가 매개 변수 및 고급 필드 섹션에 세부 정보로 이동 하십시오 GSEA 사용자 가이드 (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html)에 대 한.
- (선택 사항): GSEA preranked 도구를 사용 하는 경우 외부 계산된 그룹 메트릭 데이터 (예를 들어, 그룹 평균 비율) 개별 샘플 데이터를 대신 사용 해야 하는 유전자 발현에 대 한,에서. 분석은 유전자 유전자를 평가 하는 데 사용 되는 미리 계산 된 그룹 메트릭 할당의 간단한 목록에 따라 실시 다음 됩니다. 대체 진 식 파일을 로드 한 후 주 탐색 모음을 이동 하 고 도구/GseaPreranked 클릭 합니다. 마찬가지로, 새 탭 ( 그림 4E) 분석에 대 한 매개 변수 설정에 대 한 열립니다.
참고: GSEA preranked 도구를 사용 하 여 개별 샘플 특정 유전자 표현 데이터가 없는 연구에 대 한 것이 좋습니다. 특별 한 통계 또는 정규화 절차 그룹 개별 샘플 데이터 대신 평균값을 선도 하는 데이터에 수행 하는 경우 있는 경우 수 있습니다. GSEA preranked 도구를 사용 하 여 RNA 시퀀싱 데이터에 대 한 것이 좋습니다. RNA 시퀀싱 식 데이터를 정상화 하 고 그들의 표현에 따르면 유전자 순위를 사용할 수 있는 샘플 (예를 들어, 로그 배 변화), 그룹 메트릭 계산. - 클릭 합니다 ' 실행 ' 윈도우의 오른쪽 하단에 버튼.
참고: 분석 다음 걸릴 수 있습니다 컴퓨팅 속도 따라 몇 분 정도. 윈도우의 왼쪽된 하단에 GSEA 보고서 섹션에서 분석의 진행을 따라 합니다. 상태 분석을 마친 후 ' 성공 ' GSEA 보고서 섹션에 나타납니다.
분석 결과 여 GSEA 보고서 섹션에는 성공에 - 클릭 분석.
참고: 새 탐색 메뉴 모든 결과 및 매개 변수 설정 ( 그림 5)를 요약 하는 브라우저 창에서 열립니다. 탐색 메뉴의 위쪽 두 개의 섹션 구성 유전자는 정의 된 그룹 (예: 미 균 처리 샘플 또는 건강 한 컨트롤 샘플 농축)에 대 한 농축 결과 설정합니다. 두 섹션의 첫 번째 라인 통계 결과의 요약을 보여줍니다. False-검색 속도 (루즈벨트) 25%에 풍성 하 게 크게 하는 유전자 세트는 다음과 같은 해석에 농축으로 간주 됩니다. 분석의 해석에 대 한 자세한 내용은 GSEA 사용자 가이드 (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html)에서 찾을 수 있습니다. - 클릭에는 에 상세한 농축 결과 형식 excel 스프레드시트 ( 그림 6A) 분석 결과 내보내려면. 에 상세한 농축 결과 excel 두 고기 ( 그림 5)에 대해 별도로 내보내고 한 스프레드시트 파일에서 결과 데이터를 가입. 이후의 여러 연구의 유전자 표현 데이터 비교 유지 적어도 유전자 집합 (A 열)의 이름에 대 한 그것의 표준화 된 농축 점수 (NES) (열 F) 그리고 루즈벨트 (틀린 발견 비율) 값 (열 H) ( 그림 6B ).
참고: 스프레드시트 파일 들어 거 대 한 데이터는 분석의 각 유전자 설정, 유전자 세트 (A 열), 그것의 크기 (즉, 수 열 D 유전자 표현 데이터에 감지 하는 유전자), 이름을 포함 하 여 그것의 NES (방향의 정량적 측정 및 농축, 열 F의 범위), 그것의 명목상 p 값 (교정된, 열 G)와 (여러 가설 테스트, 열 H에 대 한 수정)는 루즈벨트 값. 더 해석에 대 한 내용은를 참조 하십시오 GSEA 사용자 가이드 (http://software.broadinstitute.org/gsea/doc/GSEAUserGuideFrame.html). - (예를 들면, S. 구 균 GSE9960) 두 번째 연구 결과 서로 비교 하는 모든 추가 연구에 대 한 유전자 설정 농축 분석 (4.1 4.15 단계)를 반복 합니다. 많은 인간의 임상 연구와 다른 마우스 모델 변환 연구 질문에 대 한 최적의 마우스 모델을 식별 하는 가능한 포함.
5. GSEA 결과 비교
- GSEA 결과 서로 게 모든 연구의 인간의 상황 비교를 흉내 낸 위한 최적의 동물 모델을 식별 하. 농축 점수 및 루즈벨트 값을 사용 하 여 활성화로 경로 (유전자 세트)를 분류 (NES > 0, 루즈벨트 < 25%), 금 (NES < 0, 루즈벨트 < 25%) 또는 둘 다의 (루즈벨트 > 25%). 3 x 3 우발 테이블 ( 그림 7A)에 표시 된 통로의 9 개의 가능한 조합의 현실화의 수를 계산 하는 두 연구의 각 비교.
- 긍정적인 예측 값 (ppv)의 계산에 의해 두 개의 연구와 부정적인 예측 값 (npv), 정의의 일부인 두 연구에서 같은 규제 (활성화 또는 억제)를 보여 주는 경로 사이의 상관 관계 평가 .
- 계산 ppv 및 다음 수식 (1) 및 (2)에 따라 npv:
(1)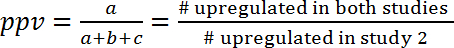
(2)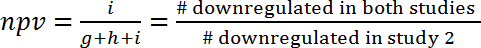
참고: 오버랩, 순전히 우연의 일치 될 수 있는 때문에 ppv와는 npv 우연히 예상 값에 비해 추가 될 수 있다. 이 접근의 또 다른 연구에서는 효과 예측에 대 한 한 연구에서 얻을 수 있는 정보의 양 추정 수 있습니다. 예를 들어, 두 가지 모델로 규정 프로세스 서로 독립 (이었고 우연히 겹치는), 그리고 만약 첫 번째 모델에는 통로의 10 %upregulated, 보다 두 번째 모델에 ppv는 또한 10% 더 추가 했다 inf의 이득 ormation입니다. 다른 측면에서 만약 두 모델 공통 규제 메커니즘에 의해 연결 되었다 다음 ppv (및 npv) 것 우연히 예상 보다 상당히 큰. 예를 들어 murine 미 균 주입 모델 (GSE20524) 효과에서 인간의 패 혈 증 (GSE9960) 동안 유전자 식 변화의 예측에는 ppv 43% 이다 (6/(6+8+0))와는 npv는 61% (11/(0+7+11)). 즉, murine 미 균 주입 모델 (GSE20524)에서 활성화 통로의 43%는 또한 인간의 패 혈 증 (GSE9960) 동안 활성화 됩니다. 마찬가지로, murine 미 균 주입 모델 (GSE20524)에 저해 통로의 61%는 또한 인간의 패 혈 증 (GSE9960) 동안 저해 ( 그림 7B). ppv와 npv (연구 2 연구 1에서에서 예측을 즉) 역 별자리에 대 한 확인할 수 있습니다.
- 계산 ppv 및 다음 수식 (1) 및 (2)에 따라 npv:
- 오버랩을 계산 하 우연히 3 x 3 우발 테이블 ( 그림 7)을 참조 하 고 ppvchance 및 npvchance 다음 수식 (3) 및 (4) 계산:
(3) < img alt = "식 3" src = "/files/ ftp_upload/55768/55768eq3.jpg"/ >
(4)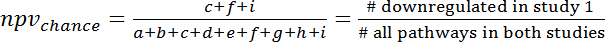
참고: 예: murine S. 구 균에 효과에서 인간의 패 혈 증 (GSE9960) 동안 유전자 식 변화의 예측 사출 모델 (GSE20524)에 ppvchance은 13% (8/64)는 npvchance 이며 22% (14/64). - 는 Ppv에서 ppvchance를 빼서 기회 대 ppv의 이득을 계산 합니다. 따라 계산는 npv에 대 한:
(5)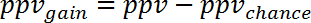
(6)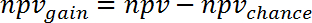
참고: 예를 들어 유전자의 예측에 대 한 식 변경 인간의 패 혈 증 (GSE9960) 동안 murine 미 균 주입 모델 (GSE20524) 효과에서 ppv에서 변화 하 고 기회 대 npv는 + 30% (43%-13%)와 + 39% (61%-22%), 각각. - Ppvgain 및 npvgain를 평균 하 여 연구 1에 관한 연구 2에서에서 얻을 수 있는 정보 이득 계산:
(7)
- 우발 테이블 (study1.pathway, study2.pathway) 카이 제곱 테스트에 의해 p 값을 계산 하는 연구의 한 쌍의 5.1 단계에서 정의 된 사용
X. 수행 카이 제곱 테스트, 예를 들어 행렬에 대비한 테이블의 데이터를 저장, R의 사용 하 여 chisq.test 기능.
참고: 예를 들어 비교 murine S. 구 균으로 선택한 인간의 패 혈 증 연구 (GSE9960) 주입 모델 (GSE20524)에서는 통계적 중복 염증 통로 규제:
> chisq.test(X,simulate.p.value=F)$ p.value
3.82e-07
6. 최적의 동물 모델 식별
- 비교는 GSEA 분석을 위해 선택한 연구의 모든 조합에 대 한 결과.
참고: 그것은 또한 다른 (유사한) 인간의 연구 뿐만 아니라 서로 다른 동물 연구를 비교 권장. 이 비교 임상 연구 (또는 장애)의 공략 분산에 대 한 통찰력을 제공할 수 있습니다 및 다른 동물 모델. 임상 연구 표시 해야 허용 중복 및 중요 한 정보를 얻을 있기 때문에 그렇지 않으면 임상 연구는 인간의 상황을 흉내낼 수 있는 동물 모델을 찾기 위해 너무 이질적인 있을 전망 이다. 이 경우에, 적절 한 동물 모델의 식별을 위해 서로 게 유사한 인간 연구를 포함 하도록 것이 좋습니다. - 정보 (단계 5.5)의 이득으로 모든 조합을 정렬합니다. 여러 데이터 집합의 비교에 대 한 행렬을 사용 하 고 컬러 heatmap 또는 같은 사용 하 여 결과 시각화 ( 그림 8).
- 정보의 높은 이득 갖는 동물 모델을 선택합니다. 정보의 증가의 중요성을 평가, 또한 고려 카이 제곱 테스트 (5.6 단계).
참고: 동물 모델만 간주 되어야 합니다 적절 한 정보 이득 상당한 고 카이 제곱 테스트의 p 값 의미 수준 이하로 경우. User-defined 임계값 일반적으로 여러 가지 요인에 따라 달라 집니다: 1) 사전 연구에 대 한 지식 2) 추정된 성공, 3) 실천에 의해 인 간에 대 한 예상된 혜택 (예: 비슷한 생리학), 인 간에 게 동물 모델에서 결과의 양도 동물 실험, 및 4의 적용) 예상된 고통, 고통, 또는 실험실에 해성사 동물.
결과
GSEA 워크플로 및 모범적인 데이터의 screenshots는 설명 했다. 그림 1 는 transcriptomic 데이터를 포함 하는 유전자 표현 데이터 파일을 보여 줍니다. 모든 연구 설명 형 파일은 필요를 그림 2에 표시 됩니다. 주석이 달린된 유전자 집합 (예를 들어, 경로) 유전자 데이터베이스 설정된 파일 (그림 3)에서 정의 ...
토론
동물 모델을가지고 오랫동안 질병 메커니즘의 조사와 새로운 치료 전략의 개발에 대 한 적용 되었습니다. 그러나, 회의론 동물 모델의 predictivity에 관한 임상 시험12의 실패 다음 확산 시작 했다. 또한, 적절 한 전략을 분석 하 고 전 임상 시험에서 큰 omics 데이터 해석에 대 한 논란이 토론 다른 데이터 분석 전략1 적용 한 후 같은 데이터에서 도출 하는 반대 결론에 의...
공개
저자 들은 아무 경쟁 금융 관심사 선언 합니다.
감사의 말
이 작품은 위험 평가 (BfR)에 대 한 독일 연방 연구소에 의해 융자 되었다.
자료
| Name | Company | Catalog Number | Comments |
| Excel | Microsoft Corporation |
참고문헌
- Seok, J., et al. Genomic responses in mouse models poorly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 110 (9), 3507-3512 (2013).
- Takao, K., Miyakawa, T. Genomic responses in mouse models greatly mimic human inflammatory diseases. Proc Natl Acad Sci U S A. 112 (4), 1167-1172 (2015).
- Weidner, C., Steinfath, M., Opitz, E., Oelgeschläger, M., Schönfelder, G. Defining the optimal animal model for translational research using gene set enrichment analysis. EMBO Mol Med. 8 (8), 831-838 (2016).
- Subramanian, A., et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. Proc Natl Acad Sci U S A. 102 (43), 15545-15550 (2005).
- Kanehisa, M., Sato, Y., Kawashima, M., Furumichi, M., Tanabe, M. KEGG as a reference resource for gene and protein annotation. Nucleic Acids Res. 44 (D1), D457-D462 (2016).
- Kanehisa, M., Goto, S. KEGG: kyoto encyclopedia of genes and genomes. Nucleic Acids Res. 28 (1), 27-30 (2000).
- Fabregat, A., et al. The Reactome pathway Knowledgebase. Nucleic Acids Res. 44 (D1), D481-D487 (2016).
- Croft, D., et al. The Reactome pathway knowledgebase. Nucleic Acids Res. 42 (Database issue), D472-D477 (2014).
- Nishimura, D. BioCarta. Biotech Software & Internet Report. 2 (3), 117-120 (2001).
- Edgar, R., Domrachev, M., Lash, A. E. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Res. 30 (1), 207-210 (2002).
- Kolesnikov, N., et al. ArrayExpress update--simplifying data submissions. Nucleic Acids Res. 43 (Database issue), D1113-D1116 (2015).
- Cohen, J., et al. Sepsis: a roadmap for future research. Lancet Infect Dis. 15 (5), 581-614 (2015).
- Spinelli, L., Carpentier, S., Montanana Sanchis, F., Dalod, M., Vu Manh, T. P. BubbleGUM: automatic extraction of phenotype molecular signatures and comprehensive visualization of multiple Gene Set Enrichment Analyses. BMC Genomics. 16 (1), 814 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유