A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
هندسة الجينوم كريسبر-Cas9-تعتمد لتوليد نماذج مراسل جوركات للإصابة بفيروس نقص المناعة البشرية-1 مع مواقع مختارة من التكامل بروفيرال
In This Article
Summary
نقدم جينوم سير عمل الهندسي لتوليد نماذج جديدة في المختبر للإصابة بفيروس نقص المناعة البشرية-1 أن الخص التكامل بروفيرال في مواقع مختارة الجينوم. استهداف الصحفيين المستمدة من فيروس نقص المناعة البشرية يسهل التلاعب جينوم كريسبر-Cas9-بوساطة، والمعينة للموقع. يتم توفير بروتوكولات مفصلة لتوليد استنساخ الخلايا المفردة والفحص والتحقق الاستهداف الصحيح.
Abstract
فيروس نقص المناعة البشرية (فيروس الإيدز) يدمج الحمض النووي بروفيرال غير عشوائي في جينوم الخلية المضيفة في مواقع المتكررة، والنقاط الساخنة الجينوم. هنا نقدم بروتوكول مفصل لتوليد نماذج في المختبر رواية للإصابة بفيروس نقص المناعة البشرية مع مواقع التكامل الجينوم الذي تم اختياره باستخدام الجينوم كريسبر-Cas9-تعتمد تكنولوجيا الهندسة. مع هذا الأسلوب، تسلسل مراسل الاختيار يمكن إدماج محور الجينوم المستهدف، والذي تم اختياره، تعكس مواقع التكامل ذات الصلة سريرياً.
ويرد في البروتوكول، تصميم مراسل المستمدة من فيروس نقص المناعة البشرية واختيار الهدف تسلسل الموقع وجرنة. ناقل استهداف مع الأسلحة التماثل التي شيدت وترانسفيكتيد في خلايا تي جوركات. التسلسل مراسل يستهدفها إلى موقع جينومي المحدد جزئ مثلى سهلت من كسر مزدوج-حبلا بوساطة Cas9 في الموقع المستهدف. استنساخ الخلايا المفردة التي تم إنشاؤها وفرزهم لاستهداف الأحداث بالتدفق الخلوي وبكر. ثم يتم توسيع استنساخ المحدد، واستهداف الصحيح يمكن التحقق منه ببكر وتسلسلها والنشاف جنوبي. ويتم تحليل الأحداث المحتملة خارج الهدف من هندسة الجينوم كريسبر-Cas9-بوساطة.
باستخدام هذا البروتوكول، ونظم الثقافة الخلية رواية يمكن أن تتولد أن الإصابة بفيروس نقص المناعة البشرية النموذجي في المواقع ذات الصلة سريرياً التكامل. ورغم أن توليد استنساخ الخلايا المفردة، والتحقق من تكامل التسلسل الصحيح مراسل مضيعة للوقت، خطوط الاستنساخ الناتجة أدوات قوية لتحليل وظيفيا اختيار موقع التكامل بروفيرال.
Introduction
إدماج الحمض النووي بروفيرال في جينوم مضيف عند الإصابة خطوة حاسمة في دورة حياة فيروس نقص المناعة البشرية (HIV). وبعد الاندماج، استمرت فيروس نقص المناعة البشرية بوضع الكمون في خلايا CD4 + "تي" خلية فرعية المعمرة مثل خلايا CD4 + T الذاكرة. إدماج فيروس نقص المناعة البشرية، على ما يبدو، غير عشوائية1،2. تم الكشف عن عدد من النقاط الساخنة الجينوم مع الحمض النووي بروفيرال المتكاملة متكرر في عدة دراسات عن طريق تسلسل مواقع التكامل في الأفراد المصابين حادة ومزمنة2،،من34 ،،من56،،من78. من المثير للاهتمام، في بعض من هذه المواقع التكامل، تم الكشف عن المكان نفسه في جزء كبير من الخلايا المصابة، مما يؤدي إلى فكرة أن التكامل في المواقع المتكررة قد تؤثر تأثيراً إيجابيا على التوسع في الاستنساخ1.
المضي قدما في فهمنا لأهمية مواقع التكامل المتكررة، يجب استكشاف خيار التكامل بروفيرال الموقع. ومع ذلك، العديد من الجوانب التقنية تعيق دراسة إدماج فيروس نقص المناعة البشرية الموقع الاختيار والعواقب. تستخدم على نطاق واسع نماذج ثقافة الخلية لزمن انتقال فيروس نقص المناعة البشرية مثل خطوط الخلايا جلات لا تعكس التكامل المتكررة ذات الصلة سريرياً مواقع9. دراسات على الخلايا الأولية المستمدة من المريض، من ناحية، تمكين وصف للتكامل الموقع المشهد بالتسلسل ولكن لا تسمح للتحليلات الفنية. على حد علمنا، يتوفر لا نموذج تجريبي كافية لتحليل المواقع المختارة ذات الصلة سريرياً الاندماج وظيفيا.
نقدم هنا سير عمل مفصلة لإنشاء نماذج جديدة بعدوى فيروس نقص المناعة البشرية باستخدام تكنولوجيا الجينوم كريسبر-Cas9-تعتمد الهندسة. يمكن استخدام سير العمل الموضحة هنا لإنشاء خطوط الخلايا المشتقة من الخلايا T مراسل أن نموذج الإصابة بفيروس نقص المناعة البشرية، تحمل مراسل بروفيرال جينوميكالي متكاملة في موقع تكامل الذي تم اختياره. وهكذا يقضون كأدوات جديدة لاستكشاف كيف يمكن أن تؤثر موقع التكامل بروفيرال بيولوجيا فيروس نقص المناعة البشرية، وكيف بروفيروس يستجيب لاستراتيجيات علاجية مختلفة (مثل، إيندوسيبيليتي بعكس وكلاء الكمون). يستخدم أسلوب لدينا مزايا هندسة الجينوم كريسبر-Cas9-تعتمد، في أي اندماج المراسل يسر تسلسل من جزئ مثلى فاصل مزدوج-حبلا المستحثة نوكلاس Cas9 في الموقع المستهدف. ويتم اختيار المواقع المستهدفة للاندماج وفقا لقربها من مواقع وصف التكامل المتكررة من الدراسات المتعلقة بالأفراد المصابين بفيروس نقص المناعة البشرية، ووجود زخارف بام مناسبة للهندسة Cas9 بوساطة الجينوم.
في نتائج مثالية، وركزنا على موضع الجينات BACH2، أي رموز للجهة النسخي BTB والتماثل باستخدام الحاسب الآلي 2. في الأفراد المصابين بأمراض مزمنة على العلاج المضاد للفيروسات الرجعية، BACH2 واحد من المكاني عرض تخصيب المتكاملة فيروس نقص المناعة البشرية-1 تسلسل3،6،7،،من810. وقد اخترنا مراسل المستمدة من فيروس نقص المناعة البشرية الحد أدنى تتكون مشتقة من فيروس نقص المناعة البشرية-1 طويلة طرفية تكرار (لاتينية) وتسلسل ترميز تدتوماتو وهرمون النمو البقري (محكمة العدل الاتحادية) بوليادينيليشن إشارة (السلطة الفلسطينية)، التي كنا قد استهدفت موقعين من مواقع محددة في إنترون BACH2 5. البروتوكول المقدم هو الأمثل للخلايا جوركات والبشرية CD4 + خط الخلية المستمدة من الخلايا T تعليق، ولكن خلية أخرى يمكن أن تستخدم خطوط وبروتوكول تكييفها تبعاً لذلك. نقدم سير عمل مفصلة لاختيار الموقع المستهدف، بناء ناقلات الأمراض المستهدفة مع الأسلحة التماثل، كريسبر-Cas9-بوساطة استهداف المراسل في موقع جينومي المختار، وتوليد واختيار الخطوط الاستنساخ، وشاملة التحقق خطوط الخلية مراسل المستهدف، والذي تم إنشاؤه حديثا.
Protocol
1-استراتيجية استهداف للجينوم الهندسية وتستهدف تصميم المتجهات (التلفزيون)
ملاحظة: الخطوة الأولى لهندسة الجينوم ينطوي على التحديد وجيل من الأدوات اللازمة لاستهداف كريسبر-Cas9-بوساطة. اختيار موضع موقع التكامل الجينوم، اختيار نوع الخلية لاستهداف، وتصميم لمراسل المستمدة من فيروس نقص المناعة البشرية للتكامل ينبغي أن تسبق هذه الخطوة. ويصف هذا البروتوكول استهداف مراسل الحد الأدنى لفيروس نقص المناعة البشرية--LTR_tdTomato_BGH--السلطة الفلسطينية إلى خلايا الهدف جوركات. مخطط تدفق سير العمل كريسبر-Cas9-تعتمد استهداف جيل والفحص والتحقق من خطوط الاستنساخ هو مبين في الشكل 1. يستخدم استراتيجية تستهدف وصف Cas9 س. المقيّحة (SpCas9) لإنشاء فواصل دسدنا جرنا الموجهة في موقع تكامل محدد. ثم يستهدف المراسل في موضع الجينوم الذي تم اختياره من خلال اقترانها مثلى بتوفير غير خطيا استهداف ناقل (التلفزيون) الذي يحتوي على تسلسل مراسل يحف به ما يسمى 5 'و 3' التماثل الأسلحة (ها)11.
-
اختيار المكان المستهدف، جرنا، واستهداف ناقلات التصميم
- اختر موقعا ثيجينوميك استهداف استناداً إلى مسألة العلمية الفردية. استخدام القوائم المنشورة من مواقع التكامل المتكررة لفيروس نقص المناعة البشرية وجدت في المرضى في مختلف الدراسات2،3،4،،من56،،من78 المبدأ التوجيهي. في السيليكون استخراج تسلسل الجينوم من محور الجينوم المطلوب أن تكون مستهدفة (تسلسل الجين كاملة) أو على الأقل 5 كيلو بايت لتسلسل الجينوم باستخدام مستعرض الجينوم التسخن (http://genome.edu.ucsc.edu).
- اختر دليل الكشف (جرناس) من 20 nt لاستهداف موضع الجينوم المختارة باستخدام ويبتول هش ه (http://www.e-crisp.org).
- حدد "GRCh38 الإنسان العاقل" كالكائن الحي. Bp الإدخال 2,000 تسلسل الجينوم يغطي موضع الجينوم المطلوب استخراج في الخطوة 1.1.1.
- بدء بحث جرنة باستخدام إعدادات التطبيق المتوسطة (أي أم أي 5 ' قاعدة قبالة أهداف تحمل عدم التطابق، وجزر إينترونس/البد مستبعدة). مع تصاميم جرنة ممكن ستظهر قائمة، مرتبة من أعلى إلى أدنى درجات الدقة والكفاءة.
- حدد جرنة التي يفضل أن يظهر درجة عالية من الدقة والكفاءة وهو أقرب ما يكون إلى موضع الجينوم المرجوة استهداف.
ملاحظة: قد حلاً وسطا بين القرب من موضع الجينوم المرجوة وتصميم جرنة محددة وتتسم بالكفاءة التي يمكن العثور عليها.
- انفجار التسلسل الذي تم اختياره جرنة ضد الجينوم مرجع باستخدام المستعرض الانفجار نكبي (https://blast.ncbi.nlm.nih.gov) للتأكد من تفرد موقع الربط جرنة.
- حدد "الإنسان" الجينوم. إدخال تسلسل جرنة كسلسلة استعلام. حدد "تسلسل مماثلة جداً" (ميجابلاست) كالبرنامج. تأكد من أن تسلسل جرنة فريدة من نوعها. إذا لم يكن كذلك، اختار من جرنة مختلفة من خطوة 1.1.2.3 والانفجار مرة أخرى.
- حالما يتم اختيار تسلسل جرنة، حدد في السيليكون 1,000 bp المنبع والمصب من تسلسل جرنة من تسلسل الجينوم استخراج في الخطوة 1.1.1 5 '3' ها تبعاً لذلك.
ملاحظة: جرناس ينبغي أن تكون مثلى لموضع الموقع المختار التكامل الجينوم وجوار فكرة المتاخمة بروتوسباسير (بام؛ ومثل، نج ل SpCas9) (الشكل 2 أ). التلفزيون يحتوي على تسلسل مراسل هو 5 'و 3' يحيط بها قد. وقد تغطي 1000 شركة بريتيش بتروليوم المنبع والمصب من تسلسل جرنة11. لا ينبغي أن يتضمنها التسلسل الكامل جرنة ها. تداخل ليصل إلى 5 nt هو مقبول (الشكل 2a).
-
جيل جرنة واستهداف ناقلات
ملاحظة: لمخططات ناقل، انظر الشكل 2b.- لإيجاد وسيلة للتعبير عن SpCas9 وجرنة، استخدم في pX330-U6-Chimeric_BB-cBh-hSpCas9 كالعمود الفقري الذي SpCas9 والحمض النووي الريبي (سجرنا) دليل واحد يمكن في نفس الوقت التعبير عن. لاستنساخ تسلسل جرنة في العمود الفقري، استخدام مواقع التقييد بسي12.
- لإنشاء جهاز التلفزيون، اختر بلازميد عالية-نسخة كالعمود الفقري (على سبيل المثال pMK، أو cDNA3.1).
- تجميع أولاً، المراسل (في هذا البروتوكول: LTR_tdTomato_BGH--السلطة الفلسطينية) في بناء العمود الفقري "الجمعية جيبسون" كلونج13 استخدام إنشاء جمعية تجارية استنساخ كيت، وإدخال 5 'و 3' المرافقة تقييد مواقع (مثل، 5 'باشي و 3' سماي) لتقييد اللاحقة استنساخ الهضم قد.
- تضخيم bp 1000 من الشظايا هكتار تم اختيارها في الخطوة 1.1.4 من الحمض النووي (جدنا) من نوع الخلية لاستهداف (في هذا البروتوكول: الخلايا جوركات) باستخدام بوليميراز الدنا مع تصحيح التجارب المطبعية النشاط (انظر الجدولين 1 و 2 للمكونات PCR و ركوب الدراجات الشروط). ثم يعرض مراسل المرافقة مواقع التقييد على طرفي 5 'و 3' لكل هكتار (باتشي في 5 'هكتار في كلا طرفي، سماي على 3' هكتار على طرفي).
- وقد استنساخ تسلسلياً في العمود الفقري بناء الفعل الذي يحتوي على المراسل (تم إنشاؤه في الخطوة 1.2.2.1) بتقييد إنزيم الاستنساخ14،15. أولاً، استنساخ في 5 'هكتار باستخدام مواقع التقييد باشي، ثم استنساخ في ها 3' استخدام مواقع تقييد سماي.
ملاحظة: إذا احتوى العمود الفقري التلفزيون على مراسل فلورسنت إضافية، يمكن تقييم التكامل العمود الفقري غير المرغوب فيها من خلال التدفق الخلوي (انظر الخطوات 3.2.2 و 3.2.3). إذا احتوى العمود الفقري التلفزيون على لا مراسل الفلورسنت، يجب تقييم التكامل العمود الفقري باستخدام PCR (راجع الخطوة 3.2.8).
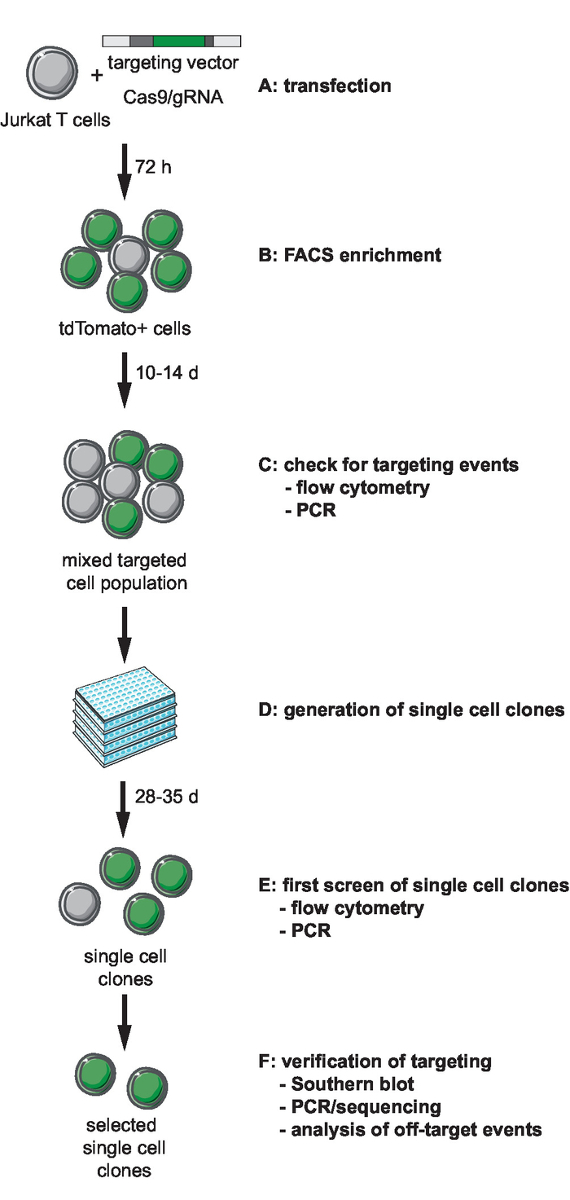
رقم 1: خطوط سير العمل كريسبر-Cas9-بوساطة تستهدف توليد واختيار المراسل الاستنساخ مع تكامل محدد الموقع. (أ) إنشاء ناقلات الأمراض المستهدفة وترانسدوسي الخلايا T جوركات مع ناقلات الأمراض المستهدفة و Cas9/جرنة التعبير بلازميد. (ب) إثراء تعداء بوست ح 72 transfected الخلايا بنظام مراقبة الأصول الميدانية. (ج) ترك الخلايا تنمو لمدة 10 إلى 14 يوما، وتأكيد وقوع استهداف الأحداث بواسطة PCR والتدفق الخلوي. (د) إنشاء استنساخ خلية واحدة بالحد من الحيوانات المستنسخة والسماح لتمييع تنمو لمدة 3 أسابيع. (ﻫ) شاشة المستنسخين لاستهداف الصحيح ببكر والتدفق الخلوي في شكل 96-جيدا. قم بتوسيع النسخ المحددة. (و) التحقق من الاستهداف في استنساخ المحدد بوصمة عار جنوب، بكر وتسلسلها، وتحليل الأحداث خارج الهدف من النشاط اندونوكليسي Cas9 الصحيح. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
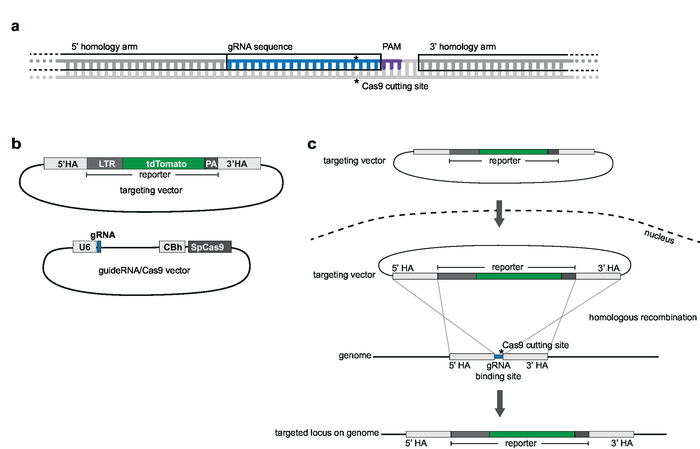
رقم 2: استهداف تصميم الاستراتيجية وناقلات. () جرنة واختيار الأسلحة التماثل. 20 nt جرنة مثلى إلى الموقع المستهدف الجينوم الذي تم اختياره، ويقع المتاخمة للتماثل PAM. الأسلحة مكملة إلى 1,000 bp أعلى-والمتلقين للمعلومات من جرنة ولا ينبغي أن تشمل التسلسل جرنة. (ب) الخطط لاستهداف ناقلات الأمراض ومكافحة ناقلات جرنة/Cas9. ناقل استهداف يتكون من تسلسل المراسل الذي تم اختياره هو 5 'و 3' الذي كان واقفاً إلى جانب الأسلحة التماثل. ناقل جرنة/Cas9 يستند إلى العمود الفقري pX330-U6-Chimeric_BB-cBh-hSpCas9. (ج) التخطيطي لاستهداف جزئ مثلى. ناقلات الأمراض المستهدفة وناقلات جويديرنا/Cas9 هي ترانسفيكتيد إلى خلايا جوركات. Cas9 يتوسط فاصل حبلا مزدوجة في موقع جينومي الهدف (المؤشرة *) وتسهل جزئ مثلى والتكامل لتسلسل مراسل في المكان المستهدف الجينوم. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
2-كريسبر-Cas9-على أساس استهداف الخلايا جوركات
-
تعداء الخلايا جوركات
- 24 ساعة قبل تعداء، لوحة 1.25 × 106 ر جوركات الخلايا 2.5 مل من 1640 RPMI تستكمل مع مصل العجل الجنين 10% (v/v) (FCS) و 4 مم الجلوتامين ل [يشار إلى "ربمي w.o. المضادات الحيوية (AB)"] كل من صفيحة ثقافة الخلية 6-جيدا جيدا. لتجربة استهداف واحد، وإعداد لوحة 6-جيدا كاملة واحدة (أي، 6 آبار كل منها 2.5 مل تعليق خلية).
- اليوم التالي، شارك ترانسفيكت الخلايا مع التلفزيون و pX330-U6-Chimeric_BB-cBh-hSpCas9/جرنة استخدام محدد كاشف تعداء الخلايا جوركات دائرية.
- إضافة 2 ميكروغرام للتلفزيون التعميم و 2 ميكروغرام من pX330-U6-Chimeric_BB-cBh-hSpCas9/جرنة الواحدة وكذلك إلى 250 ميليلتر من تجاري ربمي المتوسطة مع تركيز المصل الحد الأمثل تعداء (ربمي مع تخفيض 50% في مصل الدم) في أنبوب رد فعل ومزيج جيد.
- إضافة 12 ميليلتر من تعداء الكاشف ببطء للحمض النووي والمتوسطة دون لمس جدار الأنبوبة ودوامه. واسمحوا المخلوط احتضانها لمدة 15 دقيقة، وإضافة دروبويسي إلى بئر واحدة من الخلايا. احتضان الخلايا في 37 درجة مئوية و 5% CO2.
ملاحظة: ويمكن الارتقاء بإعداد رد فعل تعداء. مطلوب أي تغيير المتوسطة بعد تعداء.
-
تخصيب transfected الخلايا خلية تنشيط fluorescence الفرز (نظام مراقبة الأصول الميدانية)
- ح 72 تعداء بعد، تجمع transfected الخلايا، يعول عليها، والتحضير لتخصيب اليورانيوم بنظام مراقبة الأصول الميدانية. جمع الخلايا في أنبوب 50 مل مخروطية، أجهزة الطرد المركزي في 300 × ز ودرجة حرارة الغرفة (RT) لمدة 4 دقائق، يغسل الخلايا ذات مرة في برنامج تلفزيوني، والطرد المركزي مرة أخرى، تعليق بيليه بمبلغ مناسب لنظام مراقبة الأصول الميدانية المخزن المؤقت (برنامج تلفزيوني تكملها FCS 1% + 1 مم يدتا) في 1 × 10 7 خلايا/مل، ونقل أخيرا إلى أنبوب نظام مراقبة الأصول الميدانية.
- تخضع الخلايا لنظام مراقبة الأصول الميدانية وفرز تلك التي تعبر عن الفلورسنت مراسل التلفزيون (مثل، تدتوماتو في هذا البروتوكول). جمع الخلايا في 1640 RPMI تستكمل مع السفح 10%، 4 مم ل الجلوتامين، و 50 البنسلين يو/مليلتر وستربتوميسين (يشار إليها على أنها "ربمي ث/AB").
- بعد الفرز من نظام مراقبة الأصول الميدانية، تغسل الخلايا مرة واحدة عن طريق إضافة 20 مل ربمي ث/AB لفرز الخلايا والطرد المركزي في 300 x غ لمدة 4 دقائق في "الرايت ريسوسبيند" بيليه الخلية في مبلغ مناسب من ربمي ث/AB ولوحة الخلايا في بئر واحدة من صفيحة ثقافة الخلية مع حجم المناسبة وفقا لخلية رقم الوظيفة-نظام مراقبة الأصول الميدانية.
ملاحظة: ثقافة فرز الخلايا الموجودة في وحدة تخزين صغيرة (مثل، 24-أيضا)، كما لوحظت مستويات كبيرة من موت الخلايا في الأسبوع الأول بعد استهداف (تصل إلى 80 – 90%). - قم بتوسيع سكان الخلية المستهدفة المختلطة حتى بكثافة 1 × 106 خلايا/مل في قارورة ثقافة الخليوي cm ² 75. سوف تستغرق هذه العملية حوالي 10 – 14 يوما ونظام مراقبة الأصول الميدانية-وظيفة الفرز.
-
تأكيدا لاستهداف الأحداث بالتدفق الخلوي في الخلية المستهدفة المختلطة السكان
- بعد 10-14 يوما توسع (عندما وصلت الخلايا كثافة 1 × 106 خلايا/مل في قارورة ثقافة الخليوي2 75 سم)، استهدفت لوحة بئرين مع 1 × 106 خلايا المختلط سكان الخلية في 1 مل ربمي ث/AB في ثقافة خلية 12-جيدا لوحة.
- حمل "لتر فيروس نقص المناعة البشرية" للمراسل (يرد وصف جيل تلفزيون في خطوة 1.2.2.1.) في أحد الآبار بإضافة 50 نانوغرام/مل phorbol 12-ميريستاتي 13-خلات (سلطة النقد الفلسطينية) و 1 ميكرومتر إيونوميسين (المشار إليها كسلطة النقد الفلسطينية-الحيز). استخدام الخلايا في البئر الثانية كعنصر التحكم غير المستحث. الثقافة في المستحث والخلايا المستحثة بغير أجل ح 24.
- تأخذ 0.5 مل تعليق خلية من الخلايا المستحثة بغير والمستحث (كل) وغسلها مرة في برنامج تلفزيوني، وتعليق كل واحد في 200 ميليلتر من المخزن المؤقت لنظام مراقبة الأصول الميدانية.
- تحليل الخلايا 100,000 بالتدفق الخلوي. بوابة واحدة قابلة للحياة-الخلايا استناداً إلى حجم في المبعثر إلى الأمام وسيديوارد، وتحليل التعبير الجيني مراسل الفلورسنت.
ملاحظة: في هذه الخطوة (10 – 14 يوما ونظام مراقبة الأصول الميدانية-وظيفة الفرز)، ينبغي أن يكون التعبير عابر لمراسل الفلورسنت من تعداء لم تعد قابلة للاكتشاف. التعبير الفلورسنت عند هذه النقطة الزمنية تشير إلى التكامل الجينوم تسلسل مراسل.
-
الكشف عن استهداف الأحداث بواسطة PCR على الحمض النووي للخلية المستهدفة المختلطة السكان
ملاحظة: للكشف عن الأحداث تستهدف عبر PCR، تصميم اثنين التمهيدي أزواج محددة لملتقى التكامل (عدد صحيح) 5 'و int. تقاطع 3'. ل 5 'تقاطع int. بكر، ينبغي ربط التمهيدي إلى الأمام المنبع 5' هكتار وعكس التمهيدي في لتر المراسل (كبسولة تفجير P1 و P2 في الشكل 3 ألف). زوج التمهيدي لملتقى int. 3 'ينبغي أن تمتد بكر من السلطة الفلسطينية للمراسل إلى 100-200 bp المصب من 3' هكتار (كبسولة تفجير P3 و P4 في الشكل 3 ألف). كبسولة تفجير P1 و P4 سيعمل أيضا لتضخيم اليل غير المستهدفة في المختلطة السكان المستهدفين. لتخطيطي، انظر الشكل 3 ألف.- إعداد جدنا من 2 مل تعليق الخلية للخلية المستهدفة المختلطة السكان من الخطوة 2.2.4. استخدام أدوات استخراج جدنا وفقا للبروتوكول الخاص بالشركة المصنعة. قم بإعداد جدنا الخلايا غير المستهدفة كعنصر تحكم.
- أداء مفرق صحيح تقارير إتمام المشروعات (كبسولة تفجير P1/P2 و P3/P4 5 '3' مفرق صحيح، على التوالي) واستخدام غير المستهدفة اليل بكر (كبسولة تفجير P1/P4) عالية دقة بوليميريز الحمض النووي (انظر الجداول 3 و 4 للمكونات بكر وظروف ركوب) . تحليل 5 ميليلتر من منتجات PCR في هلام [اغروس]/تاي 1.5%.
ملاحظة: إذا كان السكان المستهدفين مختلط يحتوي على الخلايا التي خضعت لهندسة الجينوم، ينبغي مراعاة منتج PCR محددة التي لا يمكن كشفها في جدنا خلايا غير المستهدفة (مراقبة سلبية). اليل PCR غير المستهدفة، ينبغي للمرء أن يلاحظ منتج من نفس الحجم لكلا المستهدفة وغير المستهدفة الخلايا (مراقبة إيجابية للجينوم الإشعال P1 و P4). إذا لوحظت لا عصابات، النظر في تغيير ظروف ركوب PCR بزيادة عدد الدورات أو تغيير المخزن المؤقت بكر (على سبيل المثال عن طريق إضافة [دمس] أو مبالغ متزايدة من Mg2 +)، أو بواسطة تغيير بوليميراز.
3-توليد خطوط الاستنساخ والفحص لاستهداف الصحيح
ملاحظة: بعد التأكد الأحداث التي تستهدف السكان مختلطة الخلية المستهدفة حسب التدفق الخلوي وبكر (أقسام 2.2-2.4)، تولد استنساخ الخلايا المفردة (المدة: 28 إلى 35 يوما) وشاشة للتكامل الصحيح لتسلسل مراسل.
- جيل من الحيوانات المستنسخة من خلية واحدة من خلال تخفيف الطلاء
- إعداد مكيفة جوركات المتوسطة مقدما: تقلع ربمي ث/AB المتوسطة من الخلايا T جوركات صحية، غير المعالجة نمت إلى 1 × 106 خلايا/مل، أجهزة الطرد المركزي لمدة 5 دقائق في 300 س ز، وتصفية المادة طافية باستخدام وحدة تصفية حقنه 0.22 ميكرومتر.
ملاحظة: تبقى متوسطة مكيفة في 4 درجات مئوية للتخزين القصير الأجل أو عند-20 درجة مئوية لتخزين أطول من أسبوع واحد. إعداد 20 إلى 30 مل متوسطة مكيفة قبل تخفيف الطلاء. - عد الخلايا المستهدفة من الخطوة 2.2.4 بعد 10-14 يوما من التوسع وتمييع لهم في ربمي ث/AB بتركيز 1 × 105 خلايا/مل. تأخذ 100 ميليلتر من 1 × 105 خلايا/مل الحل وتمييع مع 9.9 مل متوسطة تحقيق تركيز 1,000 خلايا/مل. أخذ 1 مل من 1,000 خلايا/مل الحل وتمييع مع 9 مل متوسطة تحقيق تركيز 100 خلايا/مل.
- لوحات لوحة 96-جيدا خلية 1 تحتوي على كل بئر والخلايا 2 كل بئر. 1 الخلية الواحدة وكذلك، أخذ 1 مل من 100 خلايا/مل الحل وتخلط برفق مع 5 مل متوسطة مكيفة و 4 مل من متوسطة جديدة في خزان كاشف عقيمة.
- 2 الخلايا الواحدة كذلك، أخذ 2 مل من 100 خلايا/مل الحل وتخلط بلطف مع 5 مل متوسطة مكيفة و 3 مل من متوسطة جديدة. لوحة 96-بئر مستديرة-أسفل لوحات مع 100 ميليلتر لتمييع كل منهما الخلية الواحدة وكذلك استخدام بيبيت الأقنية.
ملاحظة: 5 إلى 10 لوحات 96-البئر الواحدة تستهدف بناء تكفي للحصول على الحيوانات المستنسخة للفحص. - المكدس لوحات 96-جيدا وتغطية كل مكدس مع لوحة 6-كذلك يتضمن 3 مل من برنامج تلفزيوني في كل بئر، واحتضان لوحات عند 37 درجة مئوية في حاضنة ثقافة خلية هوميديفيد مع 5% CO2 لمدة 3 أسابيع.
ملاحظة: لا تقم بتغيير مستنبت الخلية خلال هذا الوقت. لا تقم بفتح الحاضنة أكثر من مرة أو مرتين في أسبوع. ولوحظت نتائج أفضل في الحاضنات مع خزان المياه المفتوحة. - بعد 3 أسابيع حضانة، بصريا تأكيد وجود نمت المستعمرات استخدام مجهر الضوء (4 X التكبير) ومارك الآبار مع المستعمرات نمت حتى تكون مرئية كنقاط على الجزء السفلي من الآبار.
- إعداد واحد 96-بئر مستديرة-أسفل لوحة مع 100 ميليلتر من ربمي ث آب كل بئر. ريسوسبيند بلطف خلايا ملحوظ جيدا من قبل بيبيتينج. نقل 100 ميليلتر من تعليق خلية في بئر واحدة من لوحة 96-بئر جديدة فعلا تحتوي على 100 ميليلتر من ربمي ث/AB، ثم تخلط بلطف بيبيتينج. نقل 100 ميليلتر من هذا تعليق خلية في فارغ ثانية 96-بئر مستديرة-أسفل لوحة لتكرار اللوحة.
- تواصل مع جميع الآبار ملحوظ مع نمت المستعمرات. ملء جميع الآبار فارغة مع 200 ميليلتر من ربمي ث المتوسطة AB. احتضان اللوحات في 37 درجة مئوية و 5% CO2-
ملاحظة: واحدة من هذه اللوحات ستكون بمثابة للتوسع في استنساخ خلية واحدة ("لوحة الأسهم")، والأخرى "لوحة مكررة" للفحص.
- إعداد مكيفة جوركات المتوسطة مقدما: تقلع ربمي ث/AB المتوسطة من الخلايا T جوركات صحية، غير المعالجة نمت إلى 1 × 106 خلايا/مل، أجهزة الطرد المركزي لمدة 5 دقائق في 300 س ز، وتصفية المادة طافية باستخدام وحدة تصفية حقنه 0.22 ميكرومتر.
- الفرز لاستنساخ الخلايا المفردة بالتدفق الخلوي وبكر
ملاحظة: بينما يتم توسيع استنساخ خلية واحدة، استخدم لوحة مكررة من الخطوة 3.1.8 لاستنساخ الخلايا المفردة الشاشة لوجود تسلسل المراسل ببكر (الخطوات 3.2.4–3.2.12) والتعبير عن مراسل الفلورسنت بالتدفق الخلوي (الخطوات 3.2.2–3.2.3) (الشكل 3 ج).- تسمح لوحة مكررة احتضانها ح 24 إلى 48 وتكرار اللوحة مرة أخرى. للقيام بذلك، إضافة 100 ميليلتر من ربمي ث/AB لكل بئر وتخلط بلطف بيبيتينج، ونقل 100 ميليلتر لصفيحة 96-بئر مستديرة القاع جديدة استخدام بيبيت الأقنية. استخدم لوحة واحدة للفحص الخلوي التدفق، والأخرى للفرز على أساس PCR.
- لفحص التدفق الخلوي، تحفز الخلايا مع سلطة النقد الفلسطينية-الحيز. إعداد mastermix من 0.1 ميليلتر من إيونوموسين (1 مم من الأوراق المالية)، ميليلتر 0.1 من سلطة النقد الفلسطينية (50 ميكروغرام/ميليلتر الأسهم)، و 4.8 ميليلتر من ربمي ث/AB في عدد من الآبار، ثم قم بإضافة 5 ميليلتر من mastermix كل بئر.
ملاحظة: التعريفي ضروري لتحديد نجاح استنساخ، حيث لتر قد يكون ترانسكريبتيونالي صامت وذلك لا يعبر عنه المراسل الفلورسنت. - السماح للخلايا احتضانها ح 24 وإعداد الخلايا للتدفق الخلوي كما هو موضح في الخطوة 2.3.3. بوابة أي واحدة قابلة للحياة-الخلايا استناداً إلى حجم في المبعثر إلى الأمام وسيديوارد وتحليل التعبير الجيني مراسل الفلورسنت بالتدفق الخلوي (على سبيل المثال النتائج، انظر الشكل 3 ج). إذا كان العمود الفقري التلفزيون يحتوي على مراسل فلورسنت ثانية مع المروج (مثل، التجارة والنقل)، الشاشة أي استنساخ للعمود الفقري مراسل التعبير أيضا (راجع الخطوة 1.2.2 والملاحظة التالية لشرح).
ملاحظة: العمود الفقري مراسل التعبير يشير إلى التكامل غير المرغوب فيها من تسلسل العمود الفقري. - وبمجرد استنساخ في اللوحة الثانية مكررة قد نمت بما فيه الكفاية (عادة 24 إلى 48 ح بعد الازدواجية في لوحة 96-جيدا)، إعداد ليساتيس الخلية التي تحتوي على جدنا لفحص PCR. اللوحة أجهزة الطرد المركزي لمدة 10 دقيقة في 300 x ز في "الرايت بدقة" الإقلاع المادة طافية دون إزعاج بيليه الخلية.
ملاحظة: يمكن أن يؤديها جميع الخطوات لإعداد ليساتيس وردود الفعل PCR مع الممصات متعددة القنوات. - تغسل الخلايا مع 100 ميليلتر من برنامج تلفزيوني لطيف بيبيتينج ولوحة لمدة 5 دقائق في 300 x ز في الرايت سينتريفوجينج تقلع في برنامج تلفزيوني وإضافة 200 ميليلتر من المخزن المؤقت لتحلل [200 ملم كلوريد الصوديوم، 100 ملم تريس-HCl pH 8-8.5، 5 مم يدتا، الحزب الديمقراطي الصربي 0.1%؛ ثم إضافة 250 – 1,000 ميكروغرام/مل من البروتيناز ك (المجففة بالتبريد مسحوق، وزنها في طازجة)] كل بئر. المزيج بلطف بيبيتينج، ونقل التعليق لوح بكر جديدة.
- ختم اللوحة مع الفيلم البارافين واحتضانها ح 1 في 55 درجة مئوية في ثيرموسيكلير. الطرد المركزي بأقصى سرعة لمدة 10 دقائق (3,000 س ز) تدور أسفل الخلية الحطام، ونقل المادة طافية على صفيحة بكر جديدة.
ملاحظة: ليساتيس خلية في لوحات يمكن تخزينها في هذه المرحلة في 4 درجات مئوية حتى استخدامها مرة أخرى. - إعداد لوحة بكر 96-جيدا مع ميليلتر 110 درهم2س وإضافة 10 ميليلتر من الخلية ليستي (01:12 إضعاف). قد يكون ليساتيس خلية لزج ويصعب بيبيت. استخدم على الأقل 20 ميليلتر بيبيت نصائح.
- إلغاء تنشيط بروتيناز ك بالحضانة لمدة 10 دقيقة على 99 درجة مئوية في ثيرموسيكلير. وفي وقت لاحق استخدام ليساتيس خلية المعطل والمخفف لفحص PCR.
- تصميم كبسولة تفجير لفحص PCR (P5 و P6) استناداً إلى تسلسل المراسل الذي تم اختياره لتضخيم bp 500-800 تسلسل مراسل. لمراقبة إيجابية PCR، استخدم bp الإشعال P7 و P8 تضخيم 630 من محور الجينوم البرية من نوع، غير المستهدفة (الجينNUP188 ) (الشكل 3 جيم و الجدول 5). تصميم زوج التمهيدي ثالث أن يستفيض bp 500-600 من العمود الفقري التلفزيون كعنصر تحكم لتكامل غير محدد من التلفزيون متواليات العمود الفقري (العمود الفقري PCR).
- للفحص والمراقبة والعمود الفقري بكر، استخدم تجارية PCR mastermix (انظر الجداول 6 و 7 للمكونات بكر وظروف ركوب الدراجات). استخدام 2 ميليلتر من المخفف والمعطل ليستي أعد الخطوة 3.2.8 كقالب وتشغيل PCR لدورات 38 إلى 40 من [بكر] تضخيم في شكل 96-جيدا.
- تحليل 5 ميليلتر من منتجات PCR في هلام [اغروس]/تاي 1.5%.
ملاحظة: لمراقبة بكر، عصابة محددة من 630 ينبغي التقيد بي بي لكل عينة، يؤكد أن نوعية الخلية ليساتيس كافية لبكر. عصابة محددة في فحص PCR (500-800 شركة بريتيش بتروليوم اعتماداً على التصميم التمهيدي) يشير إلى التكامل في التسلسل مراسل. فرقة خاصة للعمود الفقري بكر (500-600 شركة بريتيش بتروليوم، اعتماداً على التصميم التمهيدي) يشير إلى التكامل غير المرغوب فيها من تسلسل العمود الفقري التلفزيون (على سبيل المثال النتائج، انظر الشكل 3 ج). - الجمع بين النتائج من التدفق الخلوي (الخطوة 3.2.3) والفحص على أساس بكر (الخطوة 3.2.12). قم بتحديد النسخ التي تبين أحجام الصحيح لمنتجات PCR في فحص PCR ومراقبة إيجابية PCR والتعبير عن مراسل الفلورسنت بعد التعريفي مع سلطة النقد الفلسطينية-الحيز في التدفق الخلوي. استبعاد الحيوانات المستنسخة التي تظهر أي منتج PCR في العمود الفقري PCR أو التعبير عن تلفزيون ترميز العمود الفقري الفلورسنت البروتين، مشيراً إلى تكامل غير محدد من التلفزيون العمود الفقري تسلسل.
- تدريجيا توسيع نطاق استنساخ المحدد من لوحة الأسهم 96-جيدا إلى صيغ جيدا أكبر (48/24/12/6-جيد) حتى تحقيق شكل قارورة ثقافة الخليوي T75 عن طريق إضافة جديدة متوسطة كل 2 إلى 3 أيام. المحافظة على كثافة خلية بين 1 × 105 و 1 × 106 خلايا/مل.
- تأكد من أن إعداد مخزون الخلية من الحيوانات المستنسخة خلال التوسع: عد الخلايا والطرد المركزي في 300 غرام x لمدة 5 دقائق في الرايت وتجاهل المادة طافية وتعليق الخلايا برفق في السفح + 10% [دمس] في 5 × 106 خلايا/مل. قاسمة في قنينات المبردة وحاوية استخدام cryo-تجميد تجميد الخلايا إلى 80 درجة مئوية إلى 1 درجة مئوية/دقيقة. للتخزين طويل الأجل، وتحويلها إلى سائل ن2.
ملاحظة: فإنه من المستحسن الاحتفاظ قارورة ثقافة الخليوي T75 (أي، نحو 1 × 107 خلايا) أثناء التوسع في إعداد جدنا للتحقق من استهداف جنوب النشاف (انظر الفرع 3، 4).
- تحقق مواقع التكامل ببكر/التسلسل في استنساخ المحدد
ملاحظة: تقاطعات int. 5 'و 3' من استنساخ المحدد هي بكر تضخيم والمقدمة إلى سانغر التسلسل للتحقق من تصحيح الاستهداف على مستوى تسلسل الحمض النووي.- إعداد جدنا استنساخ المحدد وجوركات البرية من نوع الخلايا باستخدام مجموعة أدوات استخراج تجاري جدنا.
- باستخدام أزواج التمهيدي ملزم 5 'نهاية المراسل والمنبع من 5' هكتار ل 5 'int. مفترق الطرق (كبسولة تفجير P1 و P2) وفي نهاية 3' المراسل والمتلقين للمعلومات من 3 'هكتار ل 3' int. مفترق الطرق (كبسولة تفجير P3 و P4) كما هو موضح في الخطوة 2، 4. استخدام كبسولة تفجير P1 و P4 لتضخيم في موقع التكامل المستهدفة في اليل دون مساواة مراسل (الشكل 4 أ).
- إعداد ردود فعل PCR مع 100-200 ng جدنا كقالب وأداء بكر استخدام بوليميريز مع تصحيح التجارب المطبعية النشاط (انظر الجدولين 1 و 2 للمكونات بكر وظروف ركوب الدراجات).
ملاحظة: إذا لوحظت لا عصابات، النظر في تغيير الظروف ركوب PCR بزيادة عدد الدورات أو تغيير المخزن المؤقت بكر (على سبيل المثال، عن طريق إضافة [دمس] أو مبالغ متزايدة من Mg2 +)، أو عن طريق تغيير بوليميراز. - تحليل 5 ميليلتر من منتجات PCR في هلام [اغروس]/تاي 1.5%. إذا لوحظت أحجام الفرقة الصحيحة، تنقية المنتج بكر المتبقية باستخدام مجموعة أدوات تجارية وإخضاعها سانغر التسلسل. التحقق من تسلسل int. مفرق 5 '، 3' تقاطع int. والموقع المستهدف من اليل دون مراسل مكمل بمواءمتها مع تسلسل المتوقعة.
ملاحظة: سوف تظهر اليل مثلى حيث أدمج المراسل لا يحتمل Cas9 بوساطة التغييرات في الموقع المستهدف، مثل النوكليوتيدات عمليات الإدراج أو الحذف (الشكل 4 أ). - للحيوانات المستنسخة التي تظهر int. الصحيح تسلسل مفرق بعد المحاذاة، إجراء PCR تضخيم المراسل المستهدفة كلياً وإخضاعها سانغر التسلسل للتحقق من التسلسل الصحيح لمساواة.
- تحليل لطخة الجنوبية للتحقق من الاستهداف في استنساخ المحدد
ملاحظة: تحليل لطخة الجنوبية من استنساخ المحدد المطلوب للتحقق من استهداف الصحيح واستبعاد اقترانها بوساطة Cas9 الأحداث التي قد تكون حدثت في موقع التكامل المستهدفة.- وضع استراتيجية الهضم جدنا المناسبة والتحقيق في التصميم قبل البدء بالتجربة.
- حدد إنزيم التقييد لتقييد جدنا الذي يولد الأجزاء المناسبة من طول 2 إلى 10 كيلو بايت في الموقع المستهدف. الإنزيمات قيود معينة، مثل آسيا والمحيط الهادئ718، أممرحبا، Bglالأول والثاني، بغل الإيكولوجيةRV، ثالثا هند، ضابط صفالأول، الباسيفيكيالأول والثاني، من بفو Scaالأول، ستوالأول، و درجة حرارة سطح البحر أنا قد استخدمت بنجاح لهضم جدنا عالية الوزن الجزيئي.
- تصميم المجسات الجنوبي مختلفة اثنين: التحقيق الخاصة بمراسل، ومسبار الجينوم. التحقيق الخاصة بمراسل هيبريديزيس على تسلسل داخل المراسل (أي التحقيق الخاصة تدتوماتو، ). التحقيق المجينية هيبريديزيس إلى منطقة الجينوم قريبة من (ولكن ليست متداخلة) هكتار واحد.
- اختر المسبار الجينوم حتى أن الشظايا الناتجة عن الهضم جدنا سوف يتم الكشف عنها بواسطة ملزمة التحقيق الجينوم تختلف في الطول (أكثر من 2 كيلو بايت) من الآليلات المستهدفة وغير المستهدفة (الشكل 4 باء). طول مسبار 400 إلى 1000 شركة بريتيش بتروليوم ويوصي.
- تصميم بكر كبسولة تفجير لتضخيم المسابير المطلوب اثنين. تضخيم التحقيق الخاصة بمراسل من التلفزيون قالب باستخدام دقة عالية بوليميريز الحمض النووي (انظر الجداول 3 و 4 للمكونات بكر وظروف ركوب الدراجات).
- تضخيم المجس الجينوم من البرية من نوع جوركات جدنا على استعداد مع مجموعة استخراج تجاري جدنا استخدام بوليميراز الدنا مع تصحيح التجارب المطبعية النشاط (انظر الجدولين 1 و 2 للمكونات بكر وظروف ركوب الدراجات). تنقية منتجات PCR على هلام [اغروس]/تاي واستخراج الشظايا باستخدام مجموعة أدوات استخراج جل تجاري وفقا لإرشادات الشركة المصنعة.
- استخراج جدنا عالية الوزن الجزيئي من 1 × 107 الخلايا البرية من نوع جوركات الخلايا واستنساخ المحدد من الخطوة 3.2.14.
- بيليه الخلايا باستخدام الطرد المركزي في 300 x ز لمدة 5 دقائق في الرايت وغسله مرة واحدة مع برنامج تلفزيوني، وتعليق بيليه في 4 مل من المخزن المؤقت لتحلل [200 ملم كلوريد الصوديوم، 100 ملم تريس-HCl pH 8، 5 ملم يدتا، الحزب الديمقراطي الصربي 0.1%؛ ثم إضافة 250 – 1,000 ميكروغرام/مل بروتيناز ك (مسحوق المجففة بالتبريد ، في وزن طازجة)]. احتضان o/n في 55 درجة مئوية، تهتز في 350 دورة في الدقيقة في ثيرموميكسير سطح المنضدة.
- إضافة 4 مل الكحول، ومزيج من انعكاس 10 إلى 20 مرة. جدنا ينبغي أن تصبح مرئية ترسبات بيضاء. بكرة جدنا سرع على طرف ماصة زجاجية الغرامة، وتغسل الناشئة في ميليلتر 750 70% EtOH، واسمحوا جافة في الرايت (5 إلى 10 دقيقة).
- إلقاء متسرعا في أنبوب رد فعل 1.5 مل تحتوي على 500 ميليلتر من المخزن المؤقت تي س 1 (الرقم الهيدروجيني 10 ملم تريس-HCl 8.0، يدتا 1 مم) وترك حل س/ن في 4 درجات مئوية، تهز 350 لفة في الدقيقة. وينبغي أن يتم أي بيبيتينج جدنا من هذه المرحلة على مستوى تحمل نصائح لتجنب إمالة.
ملاحظة: إعداد جدنا عالية الوزن الجزيئي ضروري لتحليل لطخة جنوب، ومجموعات الإعداد المتاحة تجارياً جدنا ليست مناسبة.
- خلاصة (مرتين) 15 ميكروغرام لجدنا لاستنساخ المحدد والبرية من نوع جوركات الخلايا مع إنزيم التقييد المحدد (راجع الخطوة 3.4.1.1) في رد فعل على 60 ميليلتر مع ميليلتر 6 من إنزيم (20 وحدات/ميليلتر): أولاً، إضافة الحمض النووي، المخزن المؤقت الهضم، و ddH2س، احتضان o/n في 37 درجة مئوية ، ثم إضافة الإنزيم واحتضان o/n عند درجة حرارة محددة إنزيم الهضم. مطلوب 15 ميكروغرام لجدنا هضمها كل مسبار الجنوبي.
- استخدام ميليلتر 7 خلاصة القيد 60 ميليلتر للتفريد جل التحليلية في جل 1% [اغروس]/تاي. تشويه يشير إلى الهضم كاملة وذات نوعية جيدة من الحمض النووي لتحليل لطخة الجنوبي.
- يعجل في ملخص القيود المتبقية عن طريق إضافة 01:10 م 3 خلات الصوديوم ووحدات التخزين 2 100% EtOH، ثم احتضانها ح 1-80 درجة مئوية والطرد المركزي لمدة 30 دقيقة في س 15,600 ز في 4 درجات مئوية.
- تجاهل المادة طافية وتغسل بيليه مع 70% EtOH. الطرد المركزي لمدة 15 دقيقة في س 15,600 ز في 4 درجات مئوية، وتجاهل المادة طافية، اسمحوا بيليه الجاف لفترة وجيزة في RT، وتذوب في 20 ميليلتر ddH2o.
- تشغيل 1% [اغروس]/تاي النشاف هلام، تحميل ماي 20 من جدنا هضمها كل حارة. تشغيل الهلام ح 2 في 60 الخامس، 400 mA.
ملاحظة: نسبة مئوية من [اغروس] هلام وتشغيل الوقت/الجهد يمكن تعديلها وفقا لحجم الشظايا المتوقعة للكشف عن وصمة عار جنوب المحسوبة في الخطوة 3.4.1.1. الخطوات التالية لتحليل لطخة جنوب موصوفة بالتفصيل في بروتوكول تكميلي (الخطوات من 1 إلى 18). وتشمل هذه الخطوات من: غسل الهلام blotting النشاف على غشاء النايلون، جيل مسبار المشعة، مسبار التهجين، والتنمية من الفيلم أوتوراديوجراف. مقارنة نمط النطاقات التي تم الحصول عليها بعد التنمية أوتوراديوجراف في الخطوة 18 (البروتوكول الإضافي) مع النمط المتوقع وفقا لاستراتيجية الجنوبي (على سبيل المثال النتائج، انظر الشكل 4 باء).
- وضع استراتيجية الهضم جدنا المناسبة والتحقيق في التصميم قبل البدء بالتجربة.
- تحليل الأحداث خارج الهدف
ملاحظة: منذ هندسة الجينوم كريسبر-Cas9-بوساطة يمكن أن تولد آثار خارج الهدف، بكر تضخيم العشرة أعلى مرتبة في السيليكون-تنبأت مواقع خارج الهدف في تحديد الحيوانات المستنسخة وإخضاعها سانغر التسلسل.- استخدام ككتوب16 (http://crispr.cos.uni-heidelberg.de) لإنشاء قائمة من عشرة أعلى مرتبة في السيليكون وتوقع تسلسل خارج الهدف.
- إدخال تسلسل جرنة بما في ذلك بام، كما تستخدم لاستهداف كسلسلة استعلام. حدد "نج" كأم و "الجينوم البشري" كالمرجع للتنبؤ خارج الهدف.
- تعيين عدم التطابق التام القصوى "4" والهدف الموقع طول على طول جرنة دون أم. ملف الإخراج ستوفر قائمة مرتبة من الجينوم خارج الهدف مواقع لكل منها جرنة.
- في السيليكون استخراج تسلسل الجينوم 500 bp المنبع والمصب لكل من يضرب عشرة خارج الهدف أعلى مرتبة باستخدام "مستعرض الجينوم التسخن" (http://genome.edu.ucsc.edu) وموقف خارج الهدف ضرب من قائمة النتائج ككتوب.
- لكل قبالة المواقع المستهدفة تحليلها، يسهب التصميم تمهيدي بكر زوج أن جزء من 600 إلى 700 شركة بريتيش بتروليوم في طول بما في ذلك الموقع خارج الهدف المتوقع.
- استخراج جدنا من استنساخ المحدد وجوركات البرية من نوع الخلايا باستخدام مجموعة أدوات استخراج تجاري جدنا. لكل موقع خارج الهدف، إجراء PCR استخدام بوليميراز الدنا مع تصحيح التجارب المطبعية النشاط (انظر الجدولين 1 و 2 للمكونات بكر وظروف ركوب) في البرية من نوع وجدنا المستمدة من استنساخ كل منهما.
- تحليل 5 ميليلتر من منتجات PCR في هلام [اغروس]/تاي 1.5%. إذا لوحظت أحجام الفرقة الصحيحة، تنقية المنتج بكر المتبقية باستخدام مجموعة أدوات تنقية بكر تجاري وإخضاعها سانغر التسلسل. قارن تسلسل المواقع خارج الهدف في الخلايا جوركات والمستنسخين المستهدفة.
- استخدام ككتوب16 (http://crispr.cos.uni-heidelberg.de) لإنشاء قائمة من عشرة أعلى مرتبة في السيليكون وتوقع تسلسل خارج الهدف.
النتائج
في هذه التجربة التمثيلية اخترناه لاستهداف مراسل مشتقة من فيروس نقص المناعة البشرية-1 الحد أدنى تتكون لتر والترميز تدتوماتو التسلسل، والتسلسل بوليا-إشارة إلى اثنين المكاني في إنترون 5 من الجينات BACH217. وقد تم اختيار المكاني لاستهداف وفقا لقربها من المواقع الم?...
Discussion
وهنا يصف لنا وضع بروتوكول لإنشاء نماذج مراسل جوركات مشتقة من فيروس نقص المناعة البشرية-1 مع المواقع المختارة التكامل بروفيرال تطبيق هندسة الجينوم كريسبر-Cas9-تعتمد.
نقاط عدة من البروتوكول تتطلب عناية فائقة أثناء مرحلة التخطيط. أولاً، موضع استهداف ينبغي أن تختار بعناية، كما ب...
Disclosures
الكتاب ليس لها علاقة بالكشف عن.
Acknowledgements
ونشكر ويسيلوه Britta وهابيل بيتينا للمساعدة التقنية. كما نشكر أرني Düsedau وجانا هنسن (التدفق الخلوي منصة التكنولوجيا، معهد بيت هاينريش) لتقديم الدعم التقني.
Materials
| Name | Company | Catalog Number | Comments |
| pX330-U6-Chimeric_BB-cBh-hSpCas9 | Addgene | 42230 | vector for expression of SpCas9 and gRNA |
| pMK | GeneArt | mammalian expression vector for cloning | |
| cDNA3.1 | Invitrogen | V79020 | mammalian expression vector for cloning |
| BbsI | New England Biolabs | R0539S | restriction enzyme |
| NEBuilder Hifi DNA Assembly Cloning Kit | New England Biolabs | E5520S | Assembly cloning kit used for target vector generation |
| TaqPlus Precision PCR System | Agilent Technologies | 600210 | DNA polymerase with proofreading activity used for amplification of homology arms (step 1.2.2.2), verification of integration site and reporter sequence (step 3.3.3 and 3.3.5), generation of genomic probe for Southern blot (step 3.4.1.5) and analysis of off-target events (step 3.5.4) |
| 96-well tissue culture plate (round-bottom) | TPP | 92097 | tissue culture plates for dilution plating |
| Phusion High-Fidelity DNA polymerase | New England Biolabs | M0530 L | DNA polymerase used for detection of targeting events (step 2.4.2) and generation ofreporter-specific probe for Southern blot (step 3.4.1.4) |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D9170 | dimethyl sulfoxide as PCR additive |
| Magnesium Chloride (MgCl2) Solution | New England Biolabs | B9021S | MgCl2 solution as PCR additive |
| Deoxynucleotide (dNTP) Solution Mix | New England Biolabs | N0447S | dNTP mixture with 10 mM of each nt for PCR reactions |
| 5PRIME HotMasterMix | 5PRIME | 2200400 | ready-to-use PCR mix used for screening PCR (step 3.2.11) |
| QIAamp DNA blood mini kit | Qiagen | 51106 | DNA isolation and purification kit |
| QIAquick PCR Purification Kit | Qiagen | 28106 | PCR Purification Kit |
| RPMI 1640 without glutamine | Lonza | BE12-167F | cell culture medium |
| Fetal Bovine Serum South Africa Charge | PAN Biotech | P123002 | cell culture medium supplement |
| L-glutamine | Biochrom | K 0282 | cell culture medium supplement |
| Penicillin/Streptomycin 10.000 U/mL/ 10.000 µg/mL | Biochrom | A 2212 | cell culture medium supplement |
| Gibco Opti-MEM Reduced Serum Media | Thermo Fisher Scientific | 31985062 | cell culture medium with reduced serum concentration optimized for transfection |
| TransIT-Jurkat | Mirus Bio | MIR2125 | transfection reagent |
| phorbol 12-myristate 13-acetate | Sigma-Aldrich | P8139-1MG | cell culture reagent |
| Ionomycin | Sigma-Aldrich | I0634-1MG | cell culture reagent |
| Syringe-driven filter unit, PES membrane, 0,22 µm | Millex | SLGP033RB | filter unit for sterile filtration |
| Heracell 150i incubator | Thermo Fisher Scientific | 51026280 | tissue culture incubator |
| Amershan Hybond-N+ | GE Healthcare | RPN1520B | positively charged nylon membrane for DNA and RNA blotting |
| Stratalinker 1800 | Stratagene | 400072 | UV crosslinker |
| High Prime | Roche | 11585592001 | kit for labeling of DNA with radioactive dCTP using random oligonucleotides as primers |
| illustra ProbeQuant G-50 Micro Columns | GE Healthcare | 28-9034-08 | chromatography spin-columns for purification of labeled DNA |
References
- Hughes, S. H., Coffin, J. M. What Integration Sites Tell Us about HIV Persistence. Cell Host and Microbe. 19 (5), 588-598 (2016).
- Marini, B., Kertesz-Farkas, A., et al. Nuclear architecture dictates HIV-1 integration site selection. Nature. 521 (7551), 227-231 (2015).
- Cesana, D., Santoni de Sio, F. R., et al. HIV-1-mediated insertional activation of STAT5B and BACH2 trigger viral reservoir in T regulatory cells. Nature Communications. 8 (1), 498 (2017).
- Cohn, L. B., Silva, I. T., et al. HIV-1 Integration Landscape during Latent and Active Infection. Cell. 160 (3), 420-432 (2015).
- Han, Y., Lassen, K., et al. Resting CD4+ T cells from human immunodeficiency virus type 1 (HIV-1)-infected individuals carry integrated HIV-1 genomes within actively transcribed host genes. Journal of Virology. 78 (12), 6122-6133 (2004).
- Ikeda, T., Shibata, J., Yoshimura, K., Koito, A., Matsushita, S. Recurrent HIV-1 integration at the BACH2 locus in resting CD4+ T cell populations during effective highly active antiretroviral therapy. The Journal of Infectious Diseases. 195 (5), 716-725 (2007).
- Wagner, T. A., Mclaughlin, S., et al. Proliferation of cells with HIV integrated into cancer genes contributes to persistent infection. Science. 345 (6196), 570-573 (2014).
- Maldarelli, F., Wu, X., et al. Specific HIV integration sites are linked to clonal expansion and persistence of infected cells. Science. 345 (6193), 179-183 (2014).
- Jordan, A., Bisgrove, D., Verdin, E. HIV reproducibly establishes a latent infection after acute infection of T cells in vitro. The EMBO Journal. 22 (8), 1868-1877 (2003).
- Mack, K. D., Jin, X., et al. HIV insertions within and proximal to host cell genes are a common finding in tissues containing high levels of HIV DNA and macrophage-associated p24 antigen expression. Journal of Acquired Immune Deficiency Syndromes. 33 (3), 308-320 (2003).
- Byrne, S. M., Ortiz, L., Mali, P., Aach, J., Church, G. M. Multi-kilobase homozygous targeted gene replacement in human induced pluripotent stem cells. Nucleic Acids Research. 43 (3), 1-12 (2014).
- CRISPR Genome Engineering Toolbox: Target Sequence Cloning Protocol. Addgene website Available from: https://www.addgene.org/static/cms/filer_public/e6/5a/e65a9ef8-c8ac-4f88-98da-3b7d7960394c/zhang-lab-general-cloning-protocol.pdf (2013)
- Gibson Assembly Protocol. Addgene website Available from: https://www.addgene.org/protocols/gibson-assembly/ (2009)
- Addgene Plasmid Cloning by PCR. Addgene website Available from: https://www.addgene.org/protocols/pcr-cloning/ (2014)
- Addgene Plasmid Cloning by Restriction Enzyme Digest (aka Subcloning). Addgene website Available from: https://www.addgene.org/protocols/subcloning/ (2013)
- Stemmer, M., Thumberger, T., Del Sol Keyer, M., Wittbrodt, J., Mateo, J. L. CCTop: An intuitive, flexible and reliable CRISPR-Cas9 target prediction tool. Public Library of Science (PLoS) ONE. 10 (4), (2015).
- Lange, U. C., Bialek, J. K., Walther, T., Hauber, J. Pinpointing recurrent proviral integration sites in new models for latent HIV-1 infection. Virus Research. 249, (2018).
- Bialek, J. K., Dunay, G. A., et al. Targeted HIV-1 Latency Reversal Using CRISPR-Cas9-Derived Transcriptional Activator Systems. PloS ONE. 11 (6), e0158294 (2016).
- Lee, C. M., Davis, T. H., Bao, G. Examination of CRISPR-Cas9 design tools and the effect of target site accessibility on Cas9 activity. Experimental Physiology. 103 (4), 456-460 (2018).
- Jensen, K. T., Fløe, L., et al. Chromatin accessibility and guide sequence secondary structure affect CRISPR-Cas9 gene editing efficiency. FEBS Letters. 591 (13), 1892-1901 (2017).
- Simonetti, F. R., Sobolewski, M. D., et al. Clonally expanded CD4 + T cells can produce infectious HIV-1 in vivo. Proceedings of the National Academy of Sciences. 113 (7), 1883-1888 (2016).
- Chen, H. C., Martinez, J. P., Zorita, E., Meyerhans, A., Filion, G. J. Position effects influence HIV latency reversal. Nature Structural and Molecular Biology. 24 (1), 47-54 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved