Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Génie de génome CRISPR dotés de Cas9 pour produire des modèles de Jurkat Reporter pour l’Infection à VIH-1 avec les Sites d’intégration Proviral sélectionnés
Dans cet article
Résumé
Nous présentons un flux de travail de génie du génome pour la génération de nouveaux modèles in vitro pour l’infection à VIH-1 qui récapitulent l’intégration provirale dans certains sites génomiques. Ciblage des journalistes dérivé du VIH est facilitée par la manipulation du génome CRISPR Cas9-médiation, in situ. Des protocoles détaillés pour la génération de cellule unique clone, dépistage et vérification de ciblage correcte sont fournis.
Résumé
Virus de l’immunodéficience humaine (VIH) intègre son ADN proviral non aléatoirement dans le génome de cellule hôte sites récurrents et génomiques hotspots. Nous présentons ici un protocole détaillé pour la génération de nouveaux modèles in vitro d’infection à VIH avec des sites choisis intégration génomique à l’aide des technologies d’ingénierie génomique CRISPR dotés de Cas9. Avec cette méthode, une séquence de journaliste de choix peut être intégrée dans un locus génomique ciblé et choisi, reflétant des sites d’intégration cliniquement pertinente.
Dans le protocole, la conception d’un journaliste dérivé du VIH et en choisissant une séquence cible de site et gRNA sont décrites. Un vecteur de ciblage avec les bras de l’homologie est construit et transfecté dans des cellules T Jurkat. La séquence de journaliste s’adresse au site génomique sélectionné par recombinaison homologue, facilitée par une médiation Cas9 bicaténaires pause sur le site cible. Clones de cellules individuelles sont générées et projetés pour le ciblage des événements par cytométrie en flux et par PCR. Clones sélectionnés sont ensuite développées, et un ciblage correct est vérifiée par PCR, séquençage et de Southern. Événements hors-cible du génie de génome CRISPR Cas9-médiation sont analysés.
En utilisant ce protocole, les systèmes de cultures cellulaires roman cette infection à VIH modèle à sites d’intégration cliniquement pertinente peut être générée. Bien que la génération de clones de cellules individuelles et la vérification de l’intégration de séquence correcte journaliste prend du temps, les lignées clonales qui en résultent sont des outils puissants pour analyser fonctionnellement choix site intégration proviral.
Introduction
Intégration de l’ADN proviral dans le génome de l’hôte à l’infection est une étape essentielle dans le cycle de vie du virus de l’immunodéficience humaine (VIH). Après intégration, VIH persiste en établissant la latence en sous-ensembles de cellules T CD4 + vivaces telles que les cellules T CD4 + mémoire. Intégration du VIH semble être non aléatoire1,2. On a décelé un certain nombre de hotspots génomiques avec l’ADN proviral intégré de manière récurrente dans plusieurs études à travers le séquençage des sites d’intégration aux personnes infectées de manière aiguë et chronique2,3,4 ,5,6,7,8. Fait intéressant, à certains de ces sites d’intégration, un même locus a été détecté dans une grande partie des cellules infectées, conduisant à l’idée que l’intégration aux sites récurrents pourrait affecter positivement expansion clonale1.
Pour faire progresser notre compréhension de l’importance des sites d’intégration récurrente, choix du site intégration proviral doit être explorée. Cependant, plusieurs aspects techniques entravent étudie l’intégration du VIH du site choix et les conséquences. Largement utilisé des modèles de culture de cellules pour la latence du VIH comme les lignées cellulaires JLat ne reflètent pas cliniquement pertinente intégration récurrente sites9. Études sur des cellules primaires dérivés de patient, d’une part, permettent la description du paysage du site intégration par séquençage mais ne permettent pas d’analyses fonctionnelles. À notre connaissance, aucun modèle expérimental adéquat n’est disponible pour analyser fonctionnellement certains sites d’intégration cliniquement pertinente.
Nous présentons ici un flux de travail détaillé pour générer de nouveaux modèles pour l’infection par le VIH en utilisant des technologies d’ingénierie génomique CRISPR dotés de Cas9. Le flux de travail décrit ci-après peut être utilisé pour générer des lignées de cellules de culture cellulaire T journaliste qui modélisent l’infection par le VIH, transportant un journaliste proviral génomiquement intégré à un site d’intégration choisie. Ils servent ainsi que de nouveaux outils pour explorer comment le site intégration proviral peut avoir un impact biologie le VIH et comment le provirus répond aux différentes stratégies de traitement (p. ex., inductibilité par inversion des agents de latence). Notre méthode utilise les avantages du génie de génome CRISPR dotés de Cas9, dans laquelle intégration du reporter séquence par recombinaison homologue est facilitée par une pause de bicaténaires induite par la nucléase Cas9 sur le site cible. Les sites cibles pour l’intégration sont choisis selon la proximité avec les sites décrits intégration récurrente d’études sur les personnes infectées par le VIH et la présence de motifs de PAM appropriés pour l’ingénierie du génome induite par le Cas9.
Dans nos résultats exemplaires, nous avons mis l’accent sur le locus du gène BACH2, qui code pour le régulateur transcriptionnel BTB et CNC homologie 2. Chez les personnes chroniquement infectées par le VIH sous traitement antirétroviral, BACH2 est un des locus montrant l’enrichissement du integrated HIV-1 séquences3,6,7,8,10. Nous avons choisi un journaliste dérivé du VIH minimal consistant en dérivés du VIH-1 long terminal repeat (LTR), tdTomato séquence codante et signal de polyadénylation de l’hormone de croissance bovine (BGH) (PA), dont nous avons ciblé deux sites spécifiques dans l’intron BACH2 5. Le protocole présenté est optimisé pour Jurkat cellules, une lignée de cellules humaine CD4 + suspension dérivé de lymphocytes T, mais autre cellule lignées peuvent être utilisées et le protocole adapté en conséquence. Nous présentons un flux de travail détaillé pour la sélection du site cible, construction du vecteur de la cible avec les bras de l’homologie, CRISPR Cas9-médiation ciblant le reporter dans le site choisi de génomique, la génération et la sélection des lignées clonales et complètes vérification des lignées cellulaires de journaliste nouvellement généré et ciblées.
Protocole
1. cibler stratégie pour génome d’ingénierie et le ciblage de dessin vectoriel (tv)
Remarque : La première étape de l’ingénierie du génome consiste sélection et génération des outils nécessaires pour cibler les CRISPR Cas9-induite. Sélection d’un locus du site intégration génomique, choix du type de cellule afin de cibler et conception d’un reporter dérivé du VIH pour intégration doivent précéder cette étape. Ce protocole décrit le ciblage d’un journaliste minime du VIH-LTR_tdTomato_BGH-PA dans des cellules Jurkat cible. Un diagramme du flux de travail basés sur CRISPR-Cas9 ciblage, génération, dépistage et vérification des lignées clonales est représenté dans la Figure 1. La stratégie de ciblage décrite utilise le Cas9 S. pyogenes (SpCas9) pour générer des sauts de dsDNA gRNA-réalisé sur un site d’intégration sélectionnés. Le journaliste est alors ciblé dans le locus génomique choisi par recombinaison homologue en fournissant un non-linéarisé ciblage vecteur (tv) qui contient la séquence de journaliste flanquée de ce que l'on appelle 5' et 3' homologie armes (HA)11.
-
Choix du locus ciblées, gRNA et ciblage vector design
- Choisissez thegenomic locus ciblé repose sur la question scientifique individuelle. Utilisation publié des listes de sites récurrents de l’intégration du VIH trouvés chez les patients dans différentes études2,3,4,5,6,7,8 comme un ligne directrice. In silico , extraire la séquence génomique du locus génomique désiré être ciblée (séquence du gène complet) ou au moins 5 Ko de la séquence génomique UCSC genome navigateur (http:// genome.edu.ucsc.edu).
- Choisir guide RNAs (gRNAs) de 20 nt pour le ciblage du locus génomique choisi à l’aide du webtool E-CRISP (http://www.e-crisp.org).
- Sélectionnez « Homo sapiens GRCh38 » comme l’organisme. Entrée 2 000 bp de la séquence génomique couvrant le locus génomique désiré extrait à l’étape 1.1.1.
- Lancer une recherche de gRNA à l’aide de paramètres d’application moyenne (tout PAM, tout 5' base, off-cibles tolèrent incompatibilités, et îles d’introns/CPG sont exclus). Une liste avec des conceptions possibles gRNA apparaîtra, au classement du plus élevé au bas scores pour la spécificité et l’efficacité.
- Sélectionnez un gRNA qui affiche un score élevé pour la spécificité et l’efficacité de préférence et est aussi proche que possible du locus génomiques désiré d’être la cible.
Remarque : Un compromis entre la proximité du locus génomiques désiré et de la conception de gRNA spécifique et efficace doit être trouvée.
- Souffle la séquence choisie gRNA contre le génome de référence en utilisant le navigateur de souffle NCBI (https://blast.ncbi.nlm.nih.gov) pour vérifier l’unicité de l’accepteur gRNA.
- Sélectionnez « humaine » comme le génome. Entrez la séquence gRNA comme la séquence requête. Sélectionnez « séquences très similaires » (megablast) que le programme. S’assurer que la séquence gRNA est unique. Sinon, a choisi un gRNA différent à l’étape 1.1.2.3 et souffle à nouveau.
- Une fois gRNA séquence choisie, sélectionnez silico 1 000 bp en amont et en aval de gRNA séquence de la séquence génomique extrait à l’étape 1.1.1 5' et 3' HA en conséquence.
Remarque : Le gRNAs doit être homologue au locus du site choisi intégration génomique et adjacent à un motif adjacent de protospacer (PAM, par exemple, NGG pour SpCas9) (Figure 2 a). Le téléviseur contient la séquence de journaliste qui est 5' et 3' flanqué par des AP. A couverture 1000 PB en amont et en aval de la séquence de gRNA11. La séquence complète gRNA ne devrait pas figurer dans l’AP. Un chevauchement de 5 nt est acceptable (Figure 2 a).
-
Génération de gRNA et ciblage des vecteurs
Remarque : Pour les régimes de vecteur, voir Figure 2 b.- Pour générer un vecteur d’expression de SpCas9 et gRNA, utilisez le pX330-U6-Chimeric_BB-cBh-hSpCas9 comme l’épine dorsale d'où les SpCas9 et le seul guide RNA (sgRNA) peuvent être en même temps exprimés. Pour cloner la séquence gRNA dans la colonne vertébrale, utiliser la BbsI restriction sites12.
- Pour générer la tv, choisissez un plasmide haute-copie comme épine dorsale (pMK, ou cDNA3.1).
- Assemblez tout d’abord, le journaliste (dans le présent protocole : LTR_tdTomato_BGH-PA) dans la colonne vertébrale de construction par assemblage de Gibson clong13 à l’aide d’un ensemble commercial clonage kit et introduire les 5' et 3' accompagnement des sites de restriction (par exemple, 5' PacI et 3' SmaI) pour le clonage de digestion de restriction ultérieure des AP.
- Amplifier 1000 PB des fragments HA choisie à l’étape 1.1.4 de l’ADN génomique (ADNg) du type cellulaire ciblé (dans le présent protocole : cellules Jurkat) en utilisant une ADN polymérase avec activité de relecture (voir tableaux 1 et 2 pour les ingrédients de la PCR et conditions du vélo). Puis, va présenter le journaliste flanquant les sites de restriction sur les extrémités 5' et 3' de chaque HA (PacI sur 5' HA sur les deux extrémités, SmaI sur 3' HA sur les deux extrémités).
- Séquentiellement clone a en construction ossature contenant déjà le journaliste (généré à l’étape 1.2.2.1) par l’enzyme de restriction clonage14,15. Tout d’abord, clone en 5' HA à l’aide de sites de restriction de PacI, puis cloner en 3' HA à l’aide de sites de restriction SmaI.
Remarque : Si colonne vertébrale tv contient un journaliste fluorescent supplémentaire, intégration dorsale non désirés peut être évaluée par cytométrie en flux (voir étapes 3.2.2 et 3.2.3). Si colonne vertébrale tv ne contient aucun journaliste fluorescent, intégration de la colonne vertébrale doit être évaluée à l’aide de la PCR (voir étape 3.2.8).
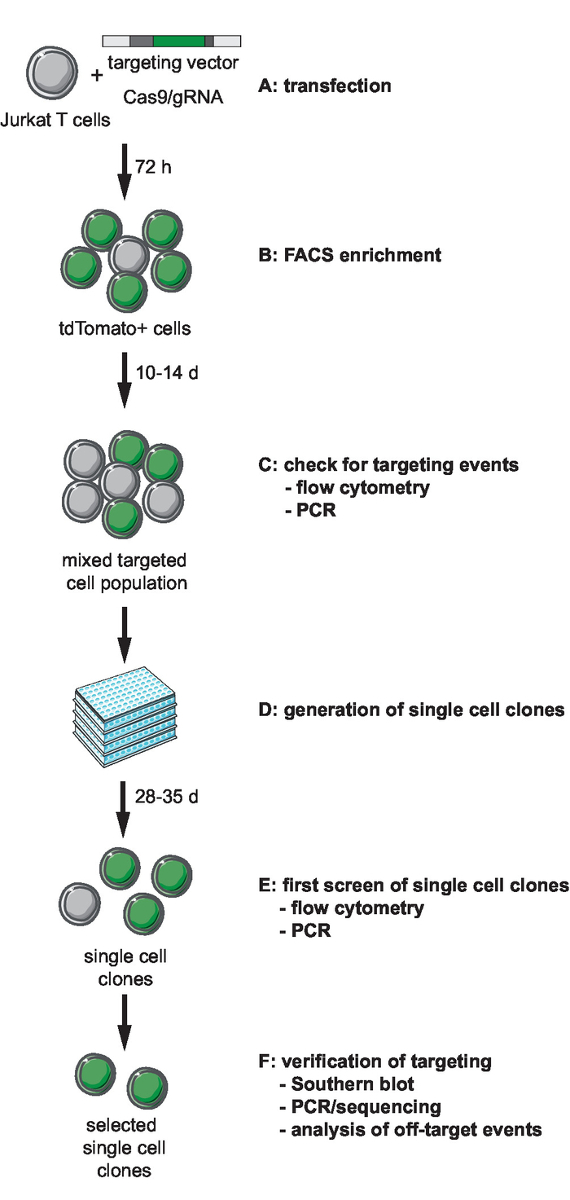
Figure 1 : "Workflow" pour CRISPR Cas9-médiation cibler, génération et sélection clonale journaliste de lignes avec site d’intégration défini. (A) mettre sur pied le vecteur de la cible et transduce cellules Jurkat T avec le vecteur de la cible et le plasmide expression Cas9/gRNA. Enrich (B) la transfection de post de 72 h de cellules transfectées par FACS. (C) laissez les cellules croissent pendant 10 à 14 jours et confirment la présence du ciblage des événements par PCR et cytométrie en flux. (D) générer de clones unicellulaires en limitant les clones de dilution et de laisser se développer pendant 3 semaines. (E) les clones pour viser correct par PCR de l’écran et cytométrie en flux au format 96 puits. Développez des clones sélectionnés. (F) vérifier le ciblage des clones sélectionnés par Southern blot, PCR et séquençage et analyse des événements hors-cible de l’activité endonucléasique Cas9 correct. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
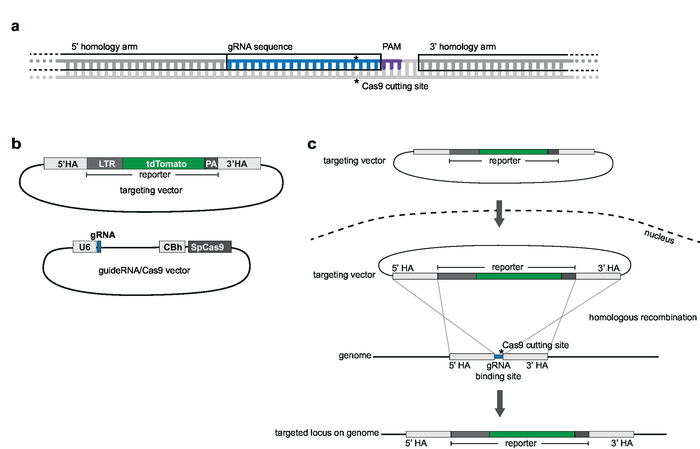
Figure 2 : ciblage design stratégie et vecteur. (un) gRNA et le choix des armes d’homologie. 20 nt gRNA est homologue au site choisi cible génomique et situé à côté d’une homologie PAM. bras sont complémentaires aux 1 000 bp haut - et en aval de la gRNA et ne doivent pas inclure la séquence gRNA. (b) schémas de ciblage vecteur et le vecteur de gRNA/Cas9. Le ciblage vecteur est constitué de la séquence choisie journaliste qui est 5' et 3' flanqué par les bras de l’homologie. Le vecteur gRNA/Cas9 repose sur la colonne vertébrale pX330-U6-Chimeric_BB-cBh-hSpCas9. (c) Représentation schématique du ciblage par recombinaison homologue. Vecteur de cible et guideRNA/Cas9 vector sont transfectés dans des cellules Jurkat. Cas9 intervient dans un saut double brin au site cible génomique (indiquées par *) et facilite la recombinaison homologue et l’intégration des séquences journaliste dans le locus génomiques cible. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
2. CRISPR Cas9-ciblage basé sur des cellules Jurkat
-
Transfection des cellules Jurkat
- 24 h avant la transfection, plaque 1,25 x 106 T Jurkat cellules dans 2,5 mL du milieu RPMI 1640 complétée avec 10 % (v/v) de sérum de veau foetal (FCS) et L-glutamine [dénommé « Antibiotiques de w.o. RPMI (AB) »] / puits d’une plaque de culture de cellules de 6 puits de 4 mM. Pour une expérience unique de ciblage, préparer un plat de 6 puits complet (p. ex., 6 puits chacune avec 2,5 mL de suspension cellulaire).
- Le lendemain, co transfecter les cellules avec tv et pX330-U6-Chimeric_BB-cBh-hSpCas9/gRNA utilisant un spécifique de réactif de transfection de cellules Jurkat circulaires.
- Ajouter 2 µg de tv circulaire et 2 µg de pX330-U6-Chimeric_BB-cBh-hSpCas9/gRNA / puits de 250 µL de milieu RPMI commercial avec concentration sérique réduite optimisée pour la transfection (RPMI avec réduction de 50 % dans le sérum) dans un tube à essais et bien mélanger.
- Ajouter 12 µL de réactif de transfection lentement à l’ADN/médium sans toucher les parois du tube et agiter. Laissez le mélange incuber pendant 15 min et ajouter goutte à goutte dans un puits de cellules. Incuber les cellules à 37 ° C et 5 % de CO2.
Remarque : La préparation de la réaction de transfection peut évoluer vers le haut. Aucun changement médiatique n’est nécessaire après la transfection.
-
Enrichissement des cellules transfectées par cellule activée par fluorescence triant (FACS)
- piscine de 72 h après la transfection, les cellules transfectées, les compter et préparer pour l’enrichissement de FACS. Recueillir les cellules dans un tube conique de 50 mL, centrifuger à 300 x g et à température ambiante (RT) pendant 4 min, laver les cellules une fois dans du PBS, centrifuger à nouveau, suspendre le culot dans une quantité appropriée de mémoire tampon de FACS (PBS additionné de 1 % FCS + EDTA 1 mM) à 1 x 10 7 cellules/mL et enfin transférer dans un tube de FACS.
- Soumettre les cellules à FACS et trier ceux qui expriment le fluorescent reporter de la télévision (par exemple, tdTomato dans le présent protocole). Recueillir les cellules en milieu RPMI 1640 additionné de 10 % FCS, 4 mM de L-glutamine et 50 U/mL de pénicilline et la streptomycine (dénommé « RPMI w / AB »).
- Après FACS Trier, laver les cellules une fois en ajoutant 20 mL de milieu RPMI w / AB aux cellules triées et centrifuger à 300 g pendant 4 min à RT. Resuspendre le culot dans une quantité appropriée de RPMI w / AB et blanc sur plaque de cellules dans un puits d’une plaque de culture cellulaire avec le volume approprié selon la cellule numéro post-FACS.
Remarque : La culture trier les cellules dans un petit volume (p. ex., 24 puits), comme des niveaux considérables de la mort cellulaire ont été observés dans la première semaine après ciblage (jusqu'à 80-90 %). - Développez la population ciblée les cellules mixtes jusqu'à une densité de 1 x 106 cellules/mL dans un flacon de culture cellulaire 75 cm². Cette opération peut prendre autour de 10 à 14 jours post-FACS Trier.
-
Confirmation du ciblage des événements par cytométrie en flux dans la population ciblée les cellules mixtes
- Après 10 à 14 jours d’expansion (lorsque les cellules ont atteint une densité de 1 x 106 cellules/mL dans un flacon de culture cellulaire 75 cm2 ), deux puits plaque avec 1 x 106 cellules de la mixte ciblées population cellulaire dans 1 mL de milieu RPMI w / AB dans une culture cellulaire de 12 puits plaque.
- Induire la LTR du VIH du reporter (génération de tv est décrit à l’étape 1.2.2.1.) dans un puits en ajoutant 50 ng/mL phorbol 12-myristate 13-acétate (PMA) et 1 µM ionomycine (dénommée AGP-Iono). Utiliser les cellules dans le deuxième puits que le contrôle non induite. Culture de l’induit et les cellules non induite pendant 24 h.
- Prendre de 0,5 mL de suspension cellulaire des cellules non induite et induits (chacun), les laver une fois dans du PBS et suspendre chacun dans 200 µL de tampon de FACS.
- Analyser les 100 000 cellules par cytométrie en flux. Porte les single-cellules viables selon la taille en nuages de points avant et latéralement et analyser l’expression du gène rapporteur fluorescent.
Remarque : à cette étape (10 – 14 jours post-FACS Trier), expression transitoire du reporter fluorescent par transfection ne devrait plus être détectable. Fluorescente expression à ce moment indique intégration génomique des séquence de journaliste.
-
Détection du ciblage des événements par PCR sur l’ADN génomique de la population ciblée les cellules mixtes
Remarque : Pour détecter les événements ciblage par PCR, conception deux paires d’amorces spécifiques pour la jonction 5' intégration (int) et int. jonction de 3'. Pour la jonction 5' int. PCR, l’apprêt avant se coince en amont de la 5' HA et l’apprêt inverse dans le LTR du reporter (amorces P1 et P2 dans la Figure 3 a). La paire d’amorces pour la jonction 3' int. PCR devrait s’étendent de l’autorité palestinienne du reporter à 100 à 200 PB en aval de la 3' HA (amorces P3 et P4 à la Figure 3 a). Amorces P1 et P4 servira aussi pour l’amplification de l’allèle non ciblée dans la population cible mixte. Pour un schéma, voir la Figure 3 a.- Préparer l’ADNg de 2 mL de suspension cellulaire de la population ciblée les cellules mixtes de l’étape 2.2.4. Utiliser un kit d’extraction ADNg selon le protocole du fabricant. Préparer ensuite la génomique des cellules indifférenciés comme un contrôle.
- Effectuer la jonction int. PCRs (amorces P1/P2 et P3/P4 pour 5' et 3' int. junction, respectivement) et indifférenciés allèle PCR (amorces P1/P4) en utilisant une ADN polymérase haute fidélité (voir tableaux 3 et 4 pour les ingrédients PCR et conditions de cyclisme) . Analyser 5 µL de produits PCR sur un gel d’agarose/TAE de 1,5 %.
Remarque : Si la population ciblée mixte contient des cellules qui ont subi le génie du génome, on devrait observer un produit PCR spécifique qui n’est pas détectable en génomique des cellules indifférenciés (témoin négatif). Pour l’allèle indifférenciés PCR, on devrait observer un produit de la même taille pour les deux les cellules ciblées et non ciblées (contrôle positif pour les amorces de P1 et P4 génomiques). Si aucune bande n’est observées, pensez modifiant les conditions de cyclisme de PCR en augmentant le nombre de cycles ou en modifiant la mémoire tampon d’ACP (par exemple par ajout de DMSO ou quantités accrues de Mg2 +), ou en changeant la polymérase.
3. génération des lignées clonales et de dépistage pour le ciblage Correct
Remarque : Après la confirmation des événements ciblant la population ciblée les cellules mixtes par cytométrie en flux et par PCR (sections 2.2 à 2.4), générer des clones de cellules individuelles (durée : 28 à 35 jours) et l’écran pour la bonne intégration de la séquence de journaliste.
- Génération de clones de cellules individuelles par étalement de dilutions
- Préparer Jurkat conditionné le milieu à l’avance : décoller RPMI w / milieu de AB des cellules T Jurkat sains et non traités passé à 1 x 106 cellules/mL, centrifuger pendant 5 min à 300 x get filtrer le surnageant à l’aide d’une unité de filtre de seringue de 0,22 µm.
Remarque : Garder le milieu conditionné à 4 ° C pour le stockage à court terme ou à-20 ° C pour le stockage de plus de 1 semaine. Préparer 20 à 30 mL de milieu conditionné avant étalement de dilutions. - Compter les cellules ciblées de l’étape 2.2.4 après 10 à 14 jours d’expansion et Diluez-les dans RPMI w / AB à une concentration de 1 x 105 cellules/mL. Prélever 100 µL de solution de 1 x 105 cellules/mL et diluer avec 9,9 mL de milieu pour atteindre une concentration de 1 000 cellules/mL. Prélever 1 mL de 1 000 cellules/mL de solution et diluer avec 9 mL de milieu pour atteindre une concentration de 100 cellules/mL.
- Plaque 96 puits plaques contenant 1 cellule par puits et 2 cellules par puits. Pour 1 cellule par puits, prendre 1 mL de solution à 100 cellules/mL et mélanger doucement avec 5 mL de milieu conditionné et 4 mL de milieu frais dans un réservoir stérile.
- 2 cellules / puits, prélever 2 mL de solution 100 cellules/mL et mélanger doucement avec 5 mL de milieu conditionné et 3 mL de milieu frais. Plaques de fond rond plaque 96 puits avec 100µl de la dilution cellulaires respectives par bien à l’aide d’une pipette multicanaux.
Remarque : 5 à 10 plaques de 96 puits par ciblage construct suffisent à obtenir des clones pour le dépistage. - Empiler les plaques de 96 puits, couvrir chaque cheminée avec une plaque de 6 puits contenant 3 mL de PBS dans chaque puits et incuber les plaques à 37 ° C dans un incubateur de culture cellulaire humidifié avec 5 % de CO2 pendant 3 semaines.
Remarque : Ne changent pas le milieu de culture cellulaire pendant cette période. Ne pas ouvrir l’incubateur à plus d’une fois ou deux fois par semaine. Les meilleurs résultats sont observés en couveuse avec réservoir d’eau ouvert. - Après 3 semaines d’incubation, confirmer visuellement la présence de colonies cultivées au microscope photonique (grossissement de 4 X) et marquez les puits avec des colonies cultivées afin qu’ils soient visibles comme des points sur le fond des puits.
- Préparer une plaque de 96 puits fond rond avec 100 µL de milieu RPMI w / AB / puits. Resuspendre doucement les cellules d’un marqué par pipetage. Transférer 100 µL de suspension cellulaire dans un puits de la nouvelle plaque de 96 puits contenant déjà 100 µL de milieu RPMI w / AB, puis mélanger doucement en pipettant également. Transférer 100 µL de cette suspension cellulaire dans un vide deuxième plaque 96 puits fond rond pour dupliquer la plaque.
- Continuer avec tous les puits marqués en colonies cultivées. Remplissez tous les puits vides avec 200 µL de RPMI w / milieu de AB. Incuber les plaques à 37° C et 5 % CO2.
Remarque : Une de ces plaques serviront pour l’expansion des clones unicellulaires (« planche de stock ») et l’autre une plaque « en double » pour le dépistage.
- Préparer Jurkat conditionné le milieu à l’avance : décoller RPMI w / milieu de AB des cellules T Jurkat sains et non traités passé à 1 x 106 cellules/mL, centrifuger pendant 5 min à 300 x get filtrer le surnageant à l’aide d’une unité de filtre de seringue de 0,22 µm.
- Dépistage des unicellulaires clones par cytométrie en flux et par PCR
Remarque : Tandis que les clones de cellules individuelles sont en expansion, utilisez la plaque double étape 3.1.8 aux clones de cellule unique écran pour présence de séquence journaliste par PCR (mesures 3.2.4–3.2.12) et l’expression de journaliste fluorescent par cytométrie en flux (pas 3.2.2–3.2.3) (Figure 3C).- Laissez la plaque double incuber pendant 24 à 48 h et dupliquer la plaque encore une fois. Pour ce faire, ajouter 100 µL de milieu RPMI w / AB dans chaque puits, mélanger doucement de pipetage et transférer 100 µL dans une nouvelle plaque de 96 puits fond rond à l’aide d’une pipette multicanaux. Utiliser une plaque pour le dépistage de cytométrie de flux et l’autre pour dépistage basé sur la PCR.
- Pour le dépistage de cytométrie en flux, stimuler les cellules avec AGP-Iono. Préparer un mastermix 0.1 µl d’Ionomocin (1 mM stock), 0.1 µL de PMA (stock de 50 µg/µL), 4,8 µL de RPMI w / AB par nombre de puits, puis ajouter 5 µL de mastermix / puits.
Remarque : L’induction est nécessaire pour identifier avec succès des clones, où le LTR est peut-être transcriptionally silencieux et c’est pourquoi le Rapporteur fluorescent n’est pas exprimé. - Laisser les cellules incuber pendant 24 h et préparer des cellules pour la cytométrie en flux comme indiqué au point 2.3.3. Porte tout seul-cellules viables selon la taille en nuages de points avant et latéralement et analyser l’expression du gène rapporteur fluorescent par cytométrie en flux (par exemple résultats, voir Figure 3C). Si l’épine dorsale tv contient un deuxième journaliste fluorescent avec promoteur (p. ex., GFP), écran des clones pour l’expression du Rapporteur épine dorsale également (voir étape 1.2.2 et la note suivante pour l’explication).
Remarque : Colonne vertébrale journaliste expression indique indésirable intégration de séquences de la colonne vertébrale. - Une fois que les clones dans la deuxième plaque en double sont devenus suffisamment (généralement 24 à 48 h après la duplication de la plaque de 96 puits), préparer des lysats de cellules contenant ADNg pour le dépistage de la PCR. Centrifuger la plaque pendant 10 min à 300 x g à RT. soigneusement enlever le surnageant sans déranger le culot cellulaire.
Remarque : Toutes les étapes pour la préparation des lysats et réactions de PCR peuvent être effectuées avec les pipettes multicanaux. - Laver les cellules avec 100 µL de PBS de doux de pipetage et de centrifugation de la plaque pendant 5 min à 300 x g à température ambiante. Décoller le PBS et ajouter 200 µL de tampon de lyse [200 mM NaCl, 100 mM Tris-HCl pH 8-8,5, 5 mM EDTA, SDS 0,1 % ; puis ajouter 250 – 1 000 µg/mL de protéinase K (lyophilisé en poudre, pesée dans fraîchement)] / puits. Mélanger doucement en pipettant également et transférer la suspension d’une nouvelle plaque PCR.
- Sceller la plaque avec le film de paraffine et incuber pendant 1 heure à 55 ° C dans un thermocycleur. Centrifuger à vitesse maximale pendant 10 min (3 000 x g) pour filer vers le bas de débris cellulaires et transférer le liquide surnageant d’une nouvelle plaque PCR.
Remarque : Des lysats de cellules en plaques peuvent être stockées à ce stade à 4 ° C jusqu'à l’utilisation ultérieure. - Préparer une plaque à 96 puits PCR avec 110 µL de dH2O et ajouter 10 µL de lysat cellulaire (01:12 dilution). Lysats cellulaires peuvent être visqueux et difficiles à pipette. Utilisez au moins embouts 20 µL.
- Inactiver protéinase K par incubation pendant 10 min à 99 ° C dans un thermocycleur. Par la suite utiliser les lysats de cellules inactivé et diluée pour le dépistage de la PCR.
- Concevoir des amorces pour la PCR (P5 et P6) basé sur la séquence choisie journaliste pour amplifier 500 – 800 bp de la séquence de journaliste de dépistage. Pour contrôle positif PCR, utiliser des amorces P7 et P8 qui amplifient 630 bp d’un locus génomique sauvage et non ciblées (gèneNUP188 ) (Figure 3C et le tableau 5). Concevoir une troisième paire d’amorces qui amplifie bp 500 – 600 de l’épine dorsale de tv comme un contrôle pour l’intégration non spécifique de séquences de tv colonne vertébrale (épine dorsale PCR).
- Pour le dépistage, le contrôle et colonne vertébrale PCR, utiliser un commercial mastermix PCR (voir tableaux 6 et 7 pour des conditions cyclisme et ingrédients PCR). Utiliser 2 µL de dilué et inactivé lysat préparée à l’étape 3.2.8 comme gabarit et PCR pour 38 à 40 cycles d’amplification PCR au format 96 puits.
- Analyser 5 µL de produits PCR sur un gel d’agarose/TAE de 1,5 %.
Remarque : Pour contrôle PCR, un groupe spécifique de 630 bp doit être observé pour chaque échantillon, confirmant que la qualité des lysats cellulaires est adéquate pour l’ACP. Un groupe spécifique de dépistage PCR (500 – 800 bp selon la conception d’amorce) indique l’intégration de la séquence de journaliste. Un groupe spécifique pour épine dorsale PCR (500 – 600 bp, selon la conception d’amorce) indique indésirable intégration de séquences d’épine dorsale tv (par exemple résultats, voir Figure 3C). - Combiner les résultats de la cytométrie en flux (étape 3.2.3) et dépistage basé sur la PCR (étape 3.2.12). Sélectionnez les clones qui montrent les tailles correctes des produits PCR PCR et contrôle positif PCR et l’expression du journaliste fluorescent après induction avec AGP-Iono en cytométrie de dépistage. Exclure les clones qui montrent tout produit de la PCR dans la colonne vertébrale PCR ou l’expression de tv colonne vertébrale fluorescent protéine codée, indiquant l’intégration non spécifique de séquence d’épine dorsale de tv.
- Étendre progressivement des clones sélectionnés de la plaque 96 puits stock aux formats bien plus grandes (48/24/12/6 puits) jusqu'à obtenir un format de flacon de culture T75 cellule en ajoutant un milieu frais tous les 2 à 3 jours. Maintenir une densité cellulaire entre 1 x 10,5 et 1 x 106 cellules/mL.
- N’oubliez pas de préparer des stocks de cellules de clones au cours de l’expansion : compter les cellules non vides, centrifuger à 300 x g pendant 5 min à ta, éliminer le surnageant et suspendre les cellules doucement dans FCS + 10 % DMSO à 5 x 106 cellules/mL. Aliquote dans flacons cryogéniques et utiliser un gel cryo conteneur pour geler les cellules à 80 ° C à 1 ° C/min. Pour le stockage à long terme, transférez-les sur liquide N2.
Remarque : Il est conseillé de conserver un flacon de culture cellulaire T75 (p. ex., environ 1 x 107 cellules) au cours de l’expansion en vue de l’ADNg pour vérification du ciblage par transfert de Southern (voir section 3.4).
- Vérification des sites d’intégration par PCR/séquençage de clones sélectionnés
Remarque : 5' et 3' int. jonctions des clones sélectionnés sont PCR amplifiés et soumis à Sanger séquençage pour vérifier corriger ciblage au niveau de séquences de l’ADN.- Préparer l’ADNg des clones sélectionnés et Jurkat sauvage de cellules à l’aide d’un kit d’extraction commerciale ADNg.
- Utiliser des paires d’amorces liant l’extrémité 5' du reporter et en amont du 5' HA pour 5' jonction int. (amorces P1 et P2) et l’extrémité 3' du reporter et en aval de 3' HA pour 3' jonction int. (amorces P3 et P4) comme indiqué au point 2.4. Utiliser les amorces P1 et P4 pour amplifier le site intégration ciblée sur l’allèle sans journaliste intégrante (Figure 4 a).
- Préparer des réactions de PCR avec 100 – 200 ng d’ADNg comme modèle et effectuez des PCR à l’aide une polymérase avec activité de relecture (voir tableaux 1 et 2 pour les ingrédients PCR et conditions cyclisme).
Remarque : Si aucune bande n’est observés, envisager de modifier les conditions de cyclisme PCR en augmentant le nombre de cycles ou en modifiant la mémoire tampon d’ACP (par exemple, en ajoutant de DMSO ou quantités accrues de Mg2 +), ou en changeant la polymérase. - Analyser 5 µL de produits PCR sur un gel d’agarose/TAE de 1,5 %. Si on observe des tailles correctes, purifier le produit restant de la PCR à l’aide d’une trousse commerciale et protégez-la Sanger séquençage. Vérifier les séquences de 5' jonction int., 3' int. junction et site ciblé de l’allèle sans journaliste intégrante en les alignant avec séquences attendues.
Remarque : L’allèle homologue où le journaliste n’a pas intégré montrera probablement Cas9 changements sur le site cible, tels que les nucléotides insertions ou les suppressions (Figure 4 a). - Pour les clones qui montrent correct int. séquences de jonction après mise en conformité, effectuer une PCR amplifiant le reporter toute ciblé et protégez-la Sanger séquençage pour vérifier la bonne séquence de l’intégrante.
- Analyse Southern pour vérification du ciblage dans les clones sélectionnés
Remarque : Analyse Southern de clones sélectionnés est requise pour vérifier un ciblage correct et exclure des événements de recombinaison médiée par Cas9 pouvant avoir eu lieu sur le site d’intégration ciblées.- Élaborer une stratégie pour une digestion appropriée ADNg et sondent la conception avant de commencer l’expérience.
- Sélectionnez une enzyme de restriction restriction ADNg qui génère des fragments appropriés de longueur de 2 à 10 Ko au site ciblé. Certaines enzymes de restriction, comme Asp718, BamHI, Bglje, BglII, EcoRV, HindIII, NcoI, PstI, PvuII, ScaI, StuI, et SST J’ai été utilisé avec succès pour digérer ADNg de haut poids moléculaire.
- Conception des deux sondes Sud différents : une sonde spécifique journaliste et sonde génomique. La sonde spécifique journaliste hybride une séquence dans le reporter (p. ex. sonde de tdTomato spécifique de, ). La sonde génomique hybride une région génomique près de (mais ne pas chevaucher) un HA.
- Choisir la sonde génomique afin que le fragment généré par digestion ADNg qui sera détectée par liaison sonde génomique se distingue par la longueur (plus de 2 Ko) à partir des allèles ciblés et non ciblés (Figure 4 b). Une longueur de sonde de 400 à 1 000 bp est recommandé.
- Concevoir des amorces PCR pour amplifier les deux sondes requis. Amplifier la sonde journaliste spécifiques du modèle de tv à l’aide d’une haute fidélité ADN polymérase (voir tableaux 3 et 4 pour les ingrédients PCR et conditions cyclisme).
- Amplifier la sonde génomique du type sauvage Jurkat ADNg préparé avec un kit d’extraction ADNg commercial utilisant une ADN polymérase avec activité de relecture (voir tableaux 1 et 2 pour les ingrédients PCR et conditions cyclisme). Purifier les produits PCR sur gel d’agarose/TAE et extraire les fragments à l’aide d’un kit d’extraction gel commercial selon les instructions du fabricant.
- Extrait 1 x 107 cellules des cellules Jurkat sauvage et des clones sélectionnés à l’étape 3.2.14 de haut poids moléculaire ADNg.
- Les cellules de granule par centrifugation à 300 x g pendant 5 min à ta, lavez-le une fois avec du PBS et suspendre le culot dans 4 mL de tampon de lyse [200 mM NaCL, 100 mM Tris-HCl pH 8, 5 mM EDTA, SDS 0,1 % ; puis ajoutez protéinase de 250 à 1 000 µg/mL K (poudre lyophilisée pèsent fraîchement)]. Incuber les o/n à 55 ° C, secouant à 350 tr/min dans une table thermomixer.
- Ajouter 4 mL d’isopropanol et mélanger par retournement de 10 à 20 fois. L’ADNg devienne visible dans le précipité blanc. La bobine de l’ADNg précipité sur la fine pointe de la pipette de verre, lavez en émergeant dans 750 µL de 70 % EtOH et laissez sécher à RT (5 à 10 min).
- Jeter le précipité dans un tube de réaction de 1,5 mL contenant 500 µL de tampon de TE des x 1 (10 mM Tris-HCl pH 8,0, EDTA 1 mM) et laisser pour dissoudre o/n à 4 ° C, secouant à 350 tr/min. Toute pipetage d’ADNg dès cette étape devrait être fait avec l’échelle-alésage conseils pour éviter la tonte.
Remarque : Préparation de haut poids moléculaire ADNg est essentielle pour l’analyse par transfert Southern, et kits de préparation ADNg disponibles dans le commerce ne conviennent pas.
- Digest (deux fois) 15 µg de génomique des clones sélectionnés et des cellules Jurkat sauvage avec certaines enzymes de restriction (voir étape 3.4.1.1) dans une réaction de 60 µL avec 6 µL d’enzyme (20 unités/µL) : tout d’abord, ajoutez l’ADN, tampon de digestion et FD2O, incuber o/n à 37 ° C , puis ajouter des enzymes et incuber o/n à la température de digestion enzymatique spécifique. Il faut 15 ug d’ADNg digérée par sonde Sud.
- Utilisez 7 µL de la Sommaire de restriction 60 µL d’électrophorèse analytique sur un gel d’agarose/TAE 1 %. Un frottis indique la digestion complète et bonne qualité d’ADN pour l’analyse par transfert Southern.
- Précipiter le Sommaire de restriction restant en ajoutant 01:10 l’acétate de sodium 3M et 2 volumes 100 % EtOH, puis incuber pendant 1 heure à-80 ° C et centrifuger pendant 30 min à 15 600 g à 4 ° C.
- Jeter le surnageant et laver le culot avec 70 % EtOH. Centrifuger à x 15 600 g à 4 ° C pendant 15 minutes, éliminer le surnageant, le culot sécher brièvement à la RT et dissoudre dans 20 µL de ddH2O.
- Exécutez le 1 % d’agarose/TAE buvard gel, 20 uL d’ADNg digérée par voie de chargement. Exécutez le gel pendant 2 h à 60 V, 400 mA.
Remarque : Le pourcentage d’agarose gel et fonctionnement temps/tension peut être réglé selon la taille des fragments attendus pour la détection de Southern blot calculée à l’étape 3.4.1.1. Les étapes suivantes de l’analyse par transfert Southern sont décrites en détail dans un protocole additionnel (étapes 1 à 18). Ces mesures comprennent : lavage du gel Blot, éponger sur une membrane de nylon, génération de sonde radioactive, hybridation de la sonde et le développement du film autoradiogramme. Comparez le modèle des bandes obtenu après développement autoradiogramme à l’étape 18 (protocole additionnel) avec le modèle prévu selon la stratégie du Sud (par exemple résultats, voir Figure 4 b).
- Élaborer une stratégie pour une digestion appropriée ADNg et sondent la conception avant de commencer l’expérience.
- Analyse des événements hors-cible
Remarque : Étant donné que le génie de génome CRISPR Cas9-médiation peut générer des effets hors cible, PCR-amplifier les dix mieux classés en silico-sites hors cible sélectionné les clones et les soumettent à Sanger a prédit le séquençage.- CCTop16 (http://crispr.cos.uni-heidelberg.de) permet de générer une liste des dix plus hauts classés en silico prédit séquences hors cible.
- Entrez la séquence gRNA, y compris le PAM comme employée pour viser comme la séquence requête. Sélectionnez « NGG » comme PAM et « Génome humain » comme la référence pour la prédiction de hors-cible.
- La valeur maximale totale inadéquation à « 4 » et la cible du site longueur à la longueur de la gRNA sans PAM. Le fichier de sortie fournira une liste de sites génomiques de hors-cible pour gRNA respectif.
- In silico extrait de la séquence génomique 500 bp en amont et en aval de chacun des dix mieux classés hors cible hits utilisant UCSC Genome Browser (http://genome.edu.ucsc.edu) et la position de la cible touchée depuis la liste de résultats de CCTop.
- Pour chaque arrêt les sites cibles à analyser, conception une amorce PCR paire qui amplifie un fragment de 600 à 700 bp dont le site hors cible prévu.
- Extraire ADNg des clones sélectionnés et des cellules de type sauvage Jurkat à l’aide d’un kit d’extraction commerciale ADNg. Pour tous les sites hors cible, effectuez une PCR en utilisant une ADN polymérase avec activité de relecture (voir tableaux 1 et 2 pour des conditions cyclisme et ingrédients PCR) sur le type sauvage et l’ADNg clone dérivés respectif.
- Analyser 5 µL de produits PCR sur un gel d’agarose/TAE de 1,5 %. Si on observe des tailles correctes, purifier le produit restant de la PCR à l’aide d’une trousse commerciale de purification des PCR et protégez-la Sanger séquençage. Comparer les séquences des sites hors-cible dans les cellules Jurkat et les clones ciblées.
- CCTop16 (http://crispr.cos.uni-heidelberg.de) permet de générer une liste des dix plus hauts classés en silico prédit séquences hors cible.
Résultats
Dans cette expérience de représentant, nous avons choisi de cibler un journaliste dérivés du VIH-1 minime consistant en une LTR, séquence codant pour tdTomato et la séquence polyA-signal à deux loci dans l’intron 5 du gène BACH217. Les locus pour le ciblage ont été choisis selon la proximité avec les sites publiés intégration récurrente trouvés dans différentes études sur des cellules de T primaires de patients infectés par le VIH
Discussion
Nous décrivons ici un protocole visant à générer des modèles de journaliste Jurkat dérivés du VIH-1 avec des sites d’intégration proviral choisi appliquant le génie du génome CRISPR dotés de Cas9.
Plusieurs points du protocole nécessitent une attention particulière durant la phase de planification. Tout d’abord, le locus d’être la cible doit être choisi avec soin, car certains loci pourrait être plus facile à la cible que d’autres (par exemple, selon l’état...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Britta Weseloh et Bettina Abel pour l’assistance technique. Nous remercions également Arne Düsedau et Jana Hennesen (plate-forme de technologie dans la cytométrie en flux, Heinrich Pette Institut) pour le support technique.
matériels
| Name | Company | Catalog Number | Comments |
| pX330-U6-Chimeric_BB-cBh-hSpCas9 | Addgene | 42230 | vector for expression of SpCas9 and gRNA |
| pMK | GeneArt | mammalian expression vector for cloning | |
| cDNA3.1 | Invitrogen | V79020 | mammalian expression vector for cloning |
| BbsI | New England Biolabs | R0539S | restriction enzyme |
| NEBuilder Hifi DNA Assembly Cloning Kit | New England Biolabs | E5520S | Assembly cloning kit used for target vector generation |
| TaqPlus Precision PCR System | Agilent Technologies | 600210 | DNA polymerase with proofreading activity used for amplification of homology arms (step 1.2.2.2), verification of integration site and reporter sequence (step 3.3.3 and 3.3.5), generation of genomic probe for Southern blot (step 3.4.1.5) and analysis of off-target events (step 3.5.4) |
| 96-well tissue culture plate (round-bottom) | TPP | 92097 | tissue culture plates for dilution plating |
| Phusion High-Fidelity DNA polymerase | New England Biolabs | M0530 L | DNA polymerase used for detection of targeting events (step 2.4.2) and generation ofreporter-specific probe for Southern blot (step 3.4.1.4) |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D9170 | dimethyl sulfoxide as PCR additive |
| Magnesium Chloride (MgCl2) Solution | New England Biolabs | B9021S | MgCl2 solution as PCR additive |
| Deoxynucleotide (dNTP) Solution Mix | New England Biolabs | N0447S | dNTP mixture with 10 mM of each nt for PCR reactions |
| 5PRIME HotMasterMix | 5PRIME | 2200400 | ready-to-use PCR mix used for screening PCR (step 3.2.11) |
| QIAamp DNA blood mini kit | Qiagen | 51106 | DNA isolation and purification kit |
| QIAquick PCR Purification Kit | Qiagen | 28106 | PCR Purification Kit |
| RPMI 1640 without glutamine | Lonza | BE12-167F | cell culture medium |
| Fetal Bovine Serum South Africa Charge | PAN Biotech | P123002 | cell culture medium supplement |
| L-glutamine | Biochrom | K 0282 | cell culture medium supplement |
| Penicillin/Streptomycin 10.000 U/mL/ 10.000 µg/mL | Biochrom | A 2212 | cell culture medium supplement |
| Gibco Opti-MEM Reduced Serum Media | Thermo Fisher Scientific | 31985062 | cell culture medium with reduced serum concentration optimized for transfection |
| TransIT-Jurkat | Mirus Bio | MIR2125 | transfection reagent |
| phorbol 12-myristate 13-acetate | Sigma-Aldrich | P8139-1MG | cell culture reagent |
| Ionomycin | Sigma-Aldrich | I0634-1MG | cell culture reagent |
| Syringe-driven filter unit, PES membrane, 0,22 µm | Millex | SLGP033RB | filter unit for sterile filtration |
| Heracell 150i incubator | Thermo Fisher Scientific | 51026280 | tissue culture incubator |
| Amershan Hybond-N+ | GE Healthcare | RPN1520B | positively charged nylon membrane for DNA and RNA blotting |
| Stratalinker 1800 | Stratagene | 400072 | UV crosslinker |
| High Prime | Roche | 11585592001 | kit for labeling of DNA with radioactive dCTP using random oligonucleotides as primers |
| illustra ProbeQuant G-50 Micro Columns | GE Healthcare | 28-9034-08 | chromatography spin-columns for purification of labeled DNA |
Références
- Hughes, S. H., Coffin, J. M. What Integration Sites Tell Us about HIV Persistence. Cell Host and Microbe. 19 (5), 588-598 (2016).
- Marini, B., Kertesz-Farkas, A., et al. Nuclear architecture dictates HIV-1 integration site selection. Nature. 521 (7551), 227-231 (2015).
- Cesana, D., Santoni de Sio, F. R., et al. HIV-1-mediated insertional activation of STAT5B and BACH2 trigger viral reservoir in T regulatory cells. Nature Communications. 8 (1), 498 (2017).
- Cohn, L. B., Silva, I. T., et al. HIV-1 Integration Landscape during Latent and Active Infection. Cell. 160 (3), 420-432 (2015).
- Han, Y., Lassen, K., et al. Resting CD4+ T cells from human immunodeficiency virus type 1 (HIV-1)-infected individuals carry integrated HIV-1 genomes within actively transcribed host genes. Journal of Virology. 78 (12), 6122-6133 (2004).
- Ikeda, T., Shibata, J., Yoshimura, K., Koito, A., Matsushita, S. Recurrent HIV-1 integration at the BACH2 locus in resting CD4+ T cell populations during effective highly active antiretroviral therapy. The Journal of Infectious Diseases. 195 (5), 716-725 (2007).
- Wagner, T. A., Mclaughlin, S., et al. Proliferation of cells with HIV integrated into cancer genes contributes to persistent infection. Science. 345 (6196), 570-573 (2014).
- Maldarelli, F., Wu, X., et al. Specific HIV integration sites are linked to clonal expansion and persistence of infected cells. Science. 345 (6193), 179-183 (2014).
- Jordan, A., Bisgrove, D., Verdin, E. HIV reproducibly establishes a latent infection after acute infection of T cells in vitro. The EMBO Journal. 22 (8), 1868-1877 (2003).
- Mack, K. D., Jin, X., et al. HIV insertions within and proximal to host cell genes are a common finding in tissues containing high levels of HIV DNA and macrophage-associated p24 antigen expression. Journal of Acquired Immune Deficiency Syndromes. 33 (3), 308-320 (2003).
- Byrne, S. M., Ortiz, L., Mali, P., Aach, J., Church, G. M. Multi-kilobase homozygous targeted gene replacement in human induced pluripotent stem cells. Nucleic Acids Research. 43 (3), 1-12 (2014).
- CRISPR Genome Engineering Toolbox: Target Sequence Cloning Protocol. Addgene website Available from: https://www.addgene.org/static/cms/filer_public/e6/5a/e65a9ef8-c8ac-4f88-98da-3b7d7960394c/zhang-lab-general-cloning-protocol.pdf (2013)
- Gibson Assembly Protocol. Addgene website Available from: https://www.addgene.org/protocols/gibson-assembly/ (2009)
- Addgene Plasmid Cloning by PCR. Addgene website Available from: https://www.addgene.org/protocols/pcr-cloning/ (2014)
- Addgene Plasmid Cloning by Restriction Enzyme Digest (aka Subcloning). Addgene website Available from: https://www.addgene.org/protocols/subcloning/ (2013)
- Stemmer, M., Thumberger, T., Del Sol Keyer, M., Wittbrodt, J., Mateo, J. L. CCTop: An intuitive, flexible and reliable CRISPR-Cas9 target prediction tool. Public Library of Science (PLoS) ONE. 10 (4), (2015).
- Lange, U. C., Bialek, J. K., Walther, T., Hauber, J. Pinpointing recurrent proviral integration sites in new models for latent HIV-1 infection. Virus Research. 249, (2018).
- Bialek, J. K., Dunay, G. A., et al. Targeted HIV-1 Latency Reversal Using CRISPR-Cas9-Derived Transcriptional Activator Systems. PloS ONE. 11 (6), e0158294 (2016).
- Lee, C. M., Davis, T. H., Bao, G. Examination of CRISPR-Cas9 design tools and the effect of target site accessibility on Cas9 activity. Experimental Physiology. 103 (4), 456-460 (2018).
- Jensen, K. T., Fløe, L., et al. Chromatin accessibility and guide sequence secondary structure affect CRISPR-Cas9 gene editing efficiency. FEBS Letters. 591 (13), 1892-1901 (2017).
- Simonetti, F. R., Sobolewski, M. D., et al. Clonally expanded CD4 + T cells can produce infectious HIV-1 in vivo. Proceedings of the National Academy of Sciences. 113 (7), 1883-1888 (2016).
- Chen, H. C., Martinez, J. P., Zorita, E., Meyerhans, A., Filion, G. J. Position effects influence HIV latency reversal. Nature Structural and Molecular Biology. 24 (1), 47-54 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon