Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
HIV-1 enfeksiyonu seçili Proviral tümleştirme siteleri ile Jurkat muhabir modelleri oluşturmak için CRISPR-Cas9-esaslı genom mühendislik
Bu Makalede
Özet
Biz yeni tüp bebek modelleri üretimi için seçili genomik sitelerdeki proviral tümleştirme özetlemek HIV-1 enfeksiyonu için genom mühendislik iş akışı mevcut. HIV kaynaklı gazetecilere hedefleme CRISPR-Cas9-aracılı, siteye özgü genom manipülasyon tarafından yönetilir. Detaylı iletişim kuralları tek hücreli klon üretimi, tarama ve doğru hedefleme doğrulama için sağlanan.
Özet
İnsan immün yetmezlik virüsü (HIV) proviral DNA'sının ana hücre genomu tekrarlayan siteleri ve genomik etkin noktaları olmayan rasgele tümleştirir. Burada ayrıntılı bir protokol roman tüp bebek modelleri üretimi için HIV enfeksiyonu için CRISPR-Cas9-esaslı genom mühendislik teknolojisi kullanarak seçilen genomik tümleştirme siteleri ile mevcut. Bu yöntemle, seçim bir muhabir dizi bir hedefli, seçilen genomik locus klinik entegrasyon siteleri yansıtan entegre edilebilir.
İletişim kuralında, HIV kaynaklı bir muhabir tasarımı ve bir hedef site ve gRNA dizi seçme açıklanmıştır. Hedefleme vektör Homoloji silah ile inşa edilmiştir ve Jurkat T hücre içine transfected. Muhabir sıra Homolog rekombinasyon bir Cas9-aracılı iki iplikçikli ara hedef sitesindeki kolaylaştırılmış tarafından seçilen genomik siteye hedeflenmektedir. Tek hücre klonlar oluşturulan ve olaylar hedefleme için ekranlı akış sitometresi ve PCR tarafından. Seçili klonlar sonra genişletilir ve doğru hedefleyen PCR, sıralama ve Southern Blot tarafından doğrulanır. CRISPR-Cas9-aracılı genom mühendislik potansiyel hedef kapalı olayları analiz edilir.
Bu iletişim kuralı, roman hücre kültür sistemleri kullanarak bu modeli HIV enfeksiyonu klinik entegrasyon sitelerde oluşturulabilir. Tek hücre klonlar oluşturma ve doğrulama doğru muhabir sıra entegrasyon zaman alıcı olsa da, elde edilen klonal satırları işlevsel olarak proviral tümleştirme site seçim analiz etmek için güçlü araçlardır.
Giriş
Ana bilgisayar genom enfeksiyon üzerine proviral DNA entegrasyonu insan immün yetmezlik virüsü (HIV) yaşam döngüsünün kritik bir adımdır. Tümleştirme, HIV uzun ömürlü CD4 + T hücre alt kümeleri bellek CD4 + T hücreleri gibi gecikme süresi kurarak devam ederse. HIV tümleştirme rasgele1,2gibi görünüyor. Akut ve kronik olarak enfekte bireylerin2,3,4 entegrasyon sitelerin nasıl yapılacağını aracılığıyla çeşitli çalışmalarda yerleştirilmiştir entegre proviral DNA ile genomik etkin noktaları bir dizi algılandı ,5,6,7,8. İlginçtir, entegrasyon sitelerin bazıları enfekte hücreleri, tekrarlayan sitelerin entegrasyon klonal genişleme1olumlu etkileyebilir fikir önde gelen büyük bir kısmını aynı locus algılandı.
Tekrarlayan tümleştirme siteleri önemi bizim anlayışı ilerletmek için proviral tümleştirme site seçim keşfedilmeyi gerekir. Ancak, birkaç teknik yönleri eğitim HIV tümleştirme engel site seçimi ve sonuçları. Geniş JLat hücre hatları klinik tekrarlayan tümleştirme siteleri9yansıtmıyor gibi hücre kültür modelleri HIV gecikme süresi için kullanılır. Çalışmalar birincil hasta elde edilen hücrelerde, bir yandan, entegrasyon sitesi peyzaj açıklaması sıralama tarafından etkinleştirir, ancak fonksiyonel analiz için izin vermez. Bilgimizi, yeterli bir deneysel model işlevsel olarak seçilen klinik entegrasyon siteleri analiz etmek kullanılabilir.
Burada CRISPR-Cas9-esaslı genom mühendislik teknolojisi kullanarak HIV enfeksiyonu için roman modelleri oluşturmak için ayrıntılı bir iş akışı mevcut. Burada açıklanan iş akışı HIV enfeksiyonu, genomically entegre proviral muhabir seçilen tümleştirme sitesinde taşıyan model T hücre kaynaklı muhabiri hücre satırlarını oluşturmak için kullanılabilir. Böylece proviral tümleştirme sitesi HIV biyoloji nasıl etkileyebilir ve provirus farklı tedavi stratejileri (örneğin, inducibility gecikme süresi ters kayıt ajanları tarafından) nasıl yanıt vereceğini keşfetmek için yeni araçları olarak hizmet vermekteyiz. Bizim Yöntem CRISPR-Cas9-esaslı genom mühendislik, muhabir hangi Wuppertal'de sırası Homolog rekombinasyon tarafından bir Cas9 nükleaz kaynaklı iki iplikçikli ara hedef sitesindeki kolaylaştırdı avantajları kullanır. Hedef siteleri tümleştirme için HIV enfekte bireylerin ve Cas9-aracılı genom Mühendisliği için uygun PAM motifleri varlığı üzerine çalışmalar açıklanan tekrarlayan tümleştirme sitelere yakınlık göre seçilir.
Örnek sonuçlarımızda BACH2 gen odağı, BTB ve CNC Homoloji transkripsiyon regülatör 2 için hangi kodları üzerinde odaklanmıştır. Kronik olarak HIV enfekte bireylerin antiretroviral tedavi, BACH2 zenginleştirme entegre HIV-1 dizileri3,6,7,8,10gösterilen loci biridir. Uzun terminal tekrar (LTR) HIV-1-türetilmiş, tdTomato kodlama dizisi ve sığır büyüme hormonu (BGH) Poliadenilasyon sinyal (PA), biz BACH2 intron 5 iki belirli sitelere hedef içeren en az bir HIV kaynaklı muhabir seçtik. Sunulan Protokolü Jurkat hücreleri, bir insan CD4 + T hücre kaynaklı süspansiyon hücre kültürünü, ama diğer hücre hatları kullanılabilir ve buna göre uyarlanmış protokolü için optimize edilmiştir. Biz hedef site, inşaat hedef vektörünün Homoloji silah, CRISPR-Cas9-aracılı seçilen genomik, üretimi ve site seçimi klonal satırlarının muhabir hedefleme ve kapsamlı ile seçim için ayrıntılı bir iş akışı mevcut doğrulama Yeni oluşturulan, hedeflenen muhabiri hücre satırı.
Protokol
1. Mühendislik ve vektör (tv) tasarımı hedefleyen genom için strateji hedefleme
Not: İlk adım genom mühendislik seçimi ve CRISPR-Cas9-aracılı hedefleme için gerekli araçları nesil içerir. Genomik tümleştirme sitesi locus yelpazesi, hedefleme için hücre tipi seçimi ve tasarım bir HIV kaynaklı muhabiri tümleştirme için bu adımı öncesinde. Bu iletişim kuralı bir HIV-LTR_tdTomato_BGH-PA en az muhabir Jurkat hedef hücrelere hedefleme açıklar. CRISPR-Cas9-esaslı hedefleme, üretimi, tarama ve klonal satırları doğrulanması için iş akışının bir akış şeması Şekil 1' de tasvir edilir. S. pyogenes Cas9 açıklanan hedefleme stratejisi kullanır (SpCas9) gRNA Yönetmen: dsDNA sonları seçili tümleştirme sitesinde oluşturmak için. Muhabir sonra sözde 5' ve 3' Homoloji silah (HA)11tarafından çevrili muhabir sıra içeren bir sigara Doğrusallaştırılmış hedefleme vektör (tv) sağlayarak Homolog rekombinasyon yoluyla seçilmiş genomik locus içine hedeflenmektedir.
-
Çeşitli hedeflenen odağı, gRNA ve vektör tasarımı hedefleme
- Thegenomic odağı olmak hedef için bireysel bilimsel soru üzerine dayalı seçin. Tekrarlayan tümleştirme site HIV yayımlanmış listesi bulunan hastalarda farklı kullanım olarak2,3,4,5,6,7,8 çalışmalar bir kılavuz. Silico ayıklamak hedeflenen (dizisi tam gen) veya en az 5 kb genomik sırası olmak istenen genomik locus genomik dizisi UCSC genom tarayıcı (http:// genome.edu.ucsc.edu) kullanarak.
- 20 (gRNAs) RNA'ların Kılavuzu seçin E-net webtool (http://www.e-crisp.org) kullanarak seçilen genomik odağı hedefleme için nt.
- "Homo sapiens GRCh38" organizma olarak seçin. 1.1.1. adımda elde istenen genomik locus kapsayan genomik sırası giriş 2.000 bp.
- (Herhangi bir PAM, herhangi bir 5' temel, hedefleri kapalı uyuşmazlıkları tahammül ve intron/CPG Adaları hariç) orta uygulama ayarları kullanarak bir gRNA arama başlatın. Mümkün gRNA tasarımları ile liste görünür sıralama yüksekten özgüllük ve verimliliği için bir puan daha düşük.
- Tercihen özgüllük ve verimliliği yüksek bir puan gösterir ve var olmak hedef için istenen genomik odağı için mümkün olduğu kadar yakın olan bir gRNA seçin.
Not: Bulunmak istenilen genomik locus yakınlığı ve belirli ve verimli gRNA tasarımını arasında bir uzlaşma vardır.
- Patlama seçilen gRNA dizi benzersizliğini gRNA bağlama sitesini kontrol etmek için NCBI patlama tarayıcı (https://blast.ncbi.nlm.nih.gov) kullanarak başvuru genom karşı.
- "İnsan genom" seçin. Sorgu sırası gRNA sýrasýný girdi. "Son derece benzer dizileri" seçin (megablast) programı olarak. GRNA sıra benzersiz olduğundan emin olun. Aksi takdirde, farklı bir gRNA tekrar adım 1.1.2.3 ve patlama açmadı.
- GRNA dizisi seçilir sonra silico 1000 bp akış yukarı ve aşağı akım gRNA sırası 1.1.1. adımda 5' ve 3' HA buna göre çıkarılan genomik serisinden seçeneğini belirleyin.
Not: GRNAs seçilen genomik tümleştirme sitesi locus homolog olmalı ve bitişiğindedir ve protospacer bitişik motifi (PAM; örneğin, NGG SpCas9 için) (Şekil 2a). Tv 5' ve 3' HAs tarafından çevrili olan muhabir sırasını içerir. Kapak 1000 bp akış yukarı ve aşağı akım gRNA sıra11vardır. Tam gRNA sıra içinde HA dahil edilmemesini. 5'e kadar bir çakışma nt olan kabul edilebilir (Şekil 2a).
-
Nesil gRNA ve vektörel çizimler hedefleme
Not: İçin vektör düzenleri, Şekil 2bbakın.- SpCas9 ve gRNA ifadesi için bir vektör üretmek için pX330-U6-Chimeric_BB-cBh-hSpCas9 hangi SpCas9 ve tek Kılavuzu RNA (sgRNA) aynı anda ifade edilebilir bel kemiği olarak kullanın. GRNA sıra omurga içine clone BbsI kısıtlama siteleri12kullanın.
- Tv üretmek için bir yüksek-kopya plazmid bel kemiği olarak (, pMK veya cDNA3.1 gibi) seçin.
- İlk olarak, muhabir montajı (Bu protokol: LTR_tdTomato_BGH-PA) yapı omurga Gibson derleme clong ile içine klonlama ticari bir derleme kullanarak13 kiti ve 5' ve 3' kısıtlama siteleri kanat tanıtmak (örneğin, 5' PacI ve 3' SmaI) sonraki kısıtlama sindirim HAs klonlama için.
- 1000 bp, adım 1.1.4 genomik DNA (gDNA) seçilen HA parçacıkları var olmak hedef için hücre türü yükseltmek (Bu protokol: Jurkat hücreleri) etkinlik düzelti ile DNA polimeraz kullanarak (bkz: Tablo 1 ve 2 için PCR malzemeler ve «««Bisiklet) koşulları. O zaman, kısıtlama siteleri her HA (PacI üzerinde 5' HA her iki ucundaki SmaI üzerinde 3' HA her iki ucundaki) 5' ve 3' ucunda kanat muhabir tanıtmak.
- Sırayla klon zaten Restriksiyon enzimi tarafından14,15klonlama (1.2.2.1 adımda oluşturulan) muhabir içeren yapı omurga içine vardır. İlk olarak, 5' HA klon PacI kısıtlama sites'ı kullanarak, sonra 3' HA klon SmaI kısıtlama sites'ı kullanarak.
Not: Tv omurga ek bir floresan muhabir içeriyorsa, istenmeyen omurga tümleştirme akış sitometresi tarafından tespit edilebilir (3.2.2 ve 3.2.3 adımlara bakın). TV omurga floresan hiçbir gazeteci içeriyorsa, omurga tümleştirme, PCR kullanarak değerlendirilmesi gerekir (bkz. Adım 3.2.8).
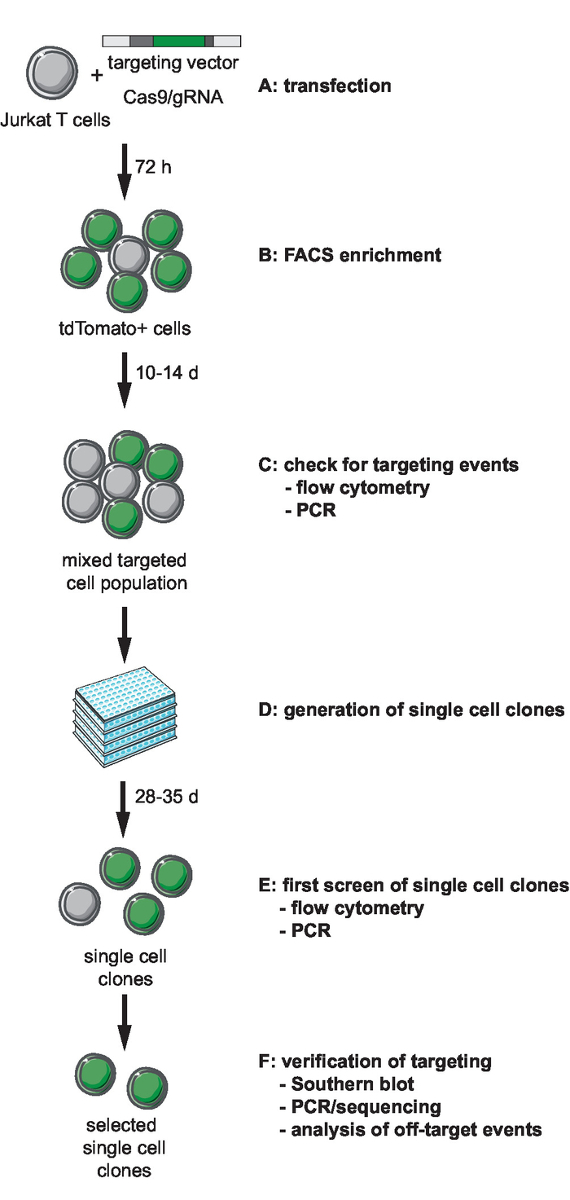
Şekil 1: CRISPR-Cas9-aracılı hedefleme, üretimi ve klonal muhabir yelpazesi için iş akışı hatları ile tanımlanmış tümleştirme site. (A)hedef vektör oluşturmak ve Jurkat T hücreleri hedef vektör ve Cas9/gRNA ifade plazmid transduce. (B) zenginleştirin transfected hücreleri 72 h mesaj transfection FACS tarafından. Hücreleri 10-14 gün boyunca büyümeye ve oluşumu olayları PCR tarafından hedefleme onaylamak ve akış sitometresi (C) Let. 3 hafta boyunca tek hücre klonlar seyreltme ve izin klonlar sınırlandırarak büyümeye (D) oluştur. (E) doğru PCR tarafından hedefleme için klonlar ekran ve akış sitometresi 96-şey biçiminde. Seçili klonlar genişletin. (F) Doğrula Southern blot, PCR ve sıralama ve Cas9 endonükleaz faaliyet hedef dışı olayların analizi tarafından seçilen klonlar hedefleme doğru. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
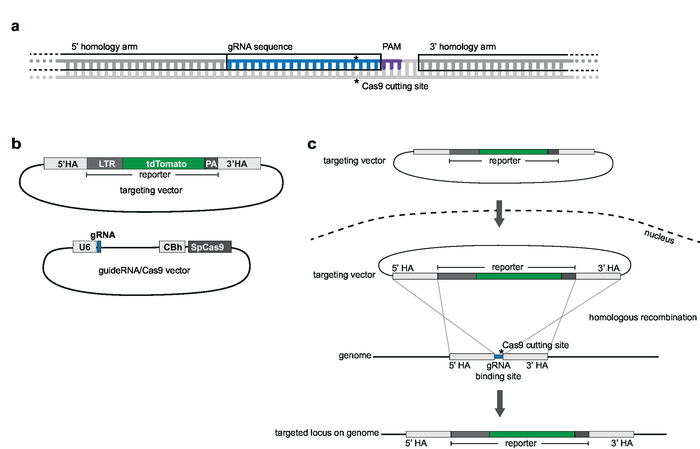
Şekil 2: strateji ve vektör tasarım hedefleme. (bir) gRNA ve Homoloji silah seçimi. 20 nt gRNA seçilen genomik hedef siteye homolog olması ve silah için 1000 bp up - tamamlayıcı ve gRNA aşağı ve gRNA sıra içermemesi gerekir bir mısınız Homoloji bitişik. (b) şemaları vektör ve gRNA/Cas9 vektör hedefleme. Hedefleme vektör 5' ve 3' Homoloji kolu tarafından çevrili olan seçilmiş muhabir sırasını oluşur. GRNA/Cas9 vektör pX330-U6-Chimeric_BB-cBh-hSpCas9 omurga üzerinde temel alır. (c) tarafından Homolog rekombinasyon hedefleme şematik. Hedef vektör ve guideRNA/Cas9 vektör Jurkat hücrelere transfected. Cas9 aracılık eder genomik hedef site Çift Kişilik strand tatilinde (belirttiği *) ve Homolog rekombinasyon ve genomik hedef locus muhabir dizisi entegrasyonunu kolaylaştırır. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
2. CRISPR Cas9-tabanlı Jurkat hücrelerinin hedefleme
-
Jurkat hücre transfection
- RPMI % 10 (v/v) fetal buzağı serum (FCS) ile ve 4 mM L-glutamin ["RPMI w.o. antibiyotik (AB)" 6-şey hücre kültür plaka kuyu sevk] 1640 2.5 ml 24 saat önce transfection, Plaka 1,25 x 106 Jurkat T hücreleri. Tek bir hedefleme deneme için bir tam 6-şey plaka hazırlamak (yani, 6 kuyuları her hücre süspansiyon 2.5 mL).
- Ertesi gün, dairesel tv ve pX330-U6-Chimeric_BB-cBh-hSpCas9/Jurkat hücreler için belirli bir transfection reaktif kullanarak gRNA hücrelerle birlikte transfect.
- 2 µg dairesel TV ve pX330-U6-Chimeric_BB-cBh-hSpCas9/gRNA şey başına 2 µg ticari RPMI orta 250 µL için indirimli serum konsantrasyonu transfection (RPMI serum % 50 azalma) için en iyi duruma getirilmiş bir tepki tüp ve karıştırmak iyi olarak ekleyin.
- Transfection reaktif 12 µL yavaş ekleyin DNA/orta olarak tüp duvarına dokunmadan ve girdap. 15 dakikadır kuluçkaya ve dropwise hücrelerinin bir şey eklemek karışım izin. 37 ° C ve % 5 CO2hücreleri kuluçkaya.
Not: Tepki transfection hazırlanması ölçeklendirilebilir. Hiçbir orta değiştirmek transfection sonra gereklidir.
-
Zenginleştirme floresans aktif hücre (FACS) sıralama transfected hücre
- 72 h sonrası transfection, havuz transfected hücreleri, onları saymak ve zenginleştirme FACS tarafından hazırlamak. 50 mL konik Tüp, santrifüj vasıl 300 x g ve oda sıcaklığında (RT) içinde hücreleri toplamak için 4 dk, PBS hücrelerde bir kez yıkamak, tekrar santrifüj kapasitesi, 1 x 10 Pelet FACS arabellek (PBS takıma % 1 FCS + 1 mM EDTA ile) uygun bir miktarda askıya 7 hücre/mL ve nihayet bir FACS tüp içine aktarın.
- Hücreleri FACS için konu ve bu da tv (örneğin, tdTomato bu protokolü) Floresan muhabir hızlı sıralayın. RPMI % 10 FCS, 4 mM L-glutamin ve 50 U/mL penisilin ve streptomisin ("RPMI w / AB" anılacaktır) ile takıma 1640 hücrelerde toplamak.
- Sıralama FACS sonra bir kez 20 mL RPMI w / AB sıralanmış hücrelere ekleyerek hücreleri yıkayın ve 300 x g 4 dk RT. Resuspend az için de AB w / RPMI uygun bir miktarda hücre Pelet santrifüj kapasitesi ve hücre kültür plaka ile bir şey hücrelerde plakası uygun birim hücre sayı yazı-FACS göre.
Not: Kültür küçük hacimli (örneğin, 24-de), hücrelerde hücre ölümü önemli düzeyde (ilâ 80-%90) sonrası hedefleme ilk haftasında gözlemlediği gibi sıralayın. - 1 x 106 hücre/mL 75 cm² hücre kültür şişesi karma hedeflenen hücre nüfus yoğunluğu kadar genişletin. Bu yazı-FACS 10-14 gün sıralama çevresinde alacaktır.
-
Akış Sitometresi karma hedeflenen hücre nüfus tarafından olayları hedefleme onay
- (1 x 106 hücre/mL 75 cm2 hücre kültür şişesi yoğunluğu hücre ulaştınız zaman) genişleme 10-14 gün sonra plaka iki wells ile karışık 1 x 106 hücreleri hücre 1 ml RPMI w / AB yılında nüfusu 12-şey hücre kültürü hedef plaka.
- HIV LTR bir muhabir neden (tv nesil 1.2.2.1 adımda anlatılan.) bir 50 ng/mL phorbol 12-myristate 13-asetat (PMA) ekleyerek Wells ve 1 µM (PMA-Iono anılacaktır) Ionomycin. Hücreleri ikinci kuyuda sigara kaynaklı denetimi olarak kullanın. Hem bağlı hem de sigara kaynaklı hücreler 24 h için kültür.
- Hücre süspansiyon sigara kaynaklı ve indüklenen hücre (her biri) 0.5 mL al, onları bir kez PBS içinde yıkayın ve her biri 200 µL FACS arabelleği askıya alma.
- 100.000 hücreleri akış sitometresi tarafından analiz. Tek-ileriye ve yana dağılım boyutu temel alan hücrelerin kapısı ve floresan muhabir gen ekspresyonu çözümleyebilirsiniz.
Not: (10-14 gün sonrası FACS sıralama) Bu adımda, floresan muhabir transfection tarafından geçici ifadesi artık tespit olmalıdır. Bu zamanda noktada floresan ifade muhabir sırası genomik Tümleştirme gösterir.
-
Karma hedeflenen hücre nüfusunun genomik DNA PCR olaylardan hedefleme algılama
Not: PCR, üzerinden hedefleme olayları tasarım iki astar çiftleri 5' entegrasyon (int) Kavşağı ve 3' int kavşağı için belirli algılamak için. 5' int kavşağı için PCR, ileri astar bağlama 5' HA ve gazeteci (astar P1 ve P2 Şekil 3a') LTR ters astar ters yönde. Astar çifti 3' int kavşağı için 100-200 kan basıncı aşağı in 3' HA (astar P3 ve P4 Şekil 3a') için muhabir PA PCR yayılacağı. Astar P1 ve P4 de karışık hedeflenen popülasyonda sigara hedefli alleli amplifikasyon için görev yapacak. Bir şema, Şekil 3abakın.- GDNA hücre süspansiyon adım 2.2.4 karma hedeflenen hücre nüfusunun 2 mL den hazırlayın. Bir gDNA ekstraksiyon kiti üreticinin protokolüne göre kullanın. O zaman sigara hedefli hücrelerinin gDNA bir denetim olarak hazırlayın.
- İnt kavşak PCR gerçekleştirmek (astar P1/P2 ve P3/P4 5' ve 3' int Kavşağı, sırasıyla) ve sigara hedefli alleli PCR (astar P1/P4) kullanarak bir yüksek-sadakat DNA polimeraz (bkz: Tablo 3 ve 4 PCR malzemeler ve bisiklet koşullarını) . 5 µL PCR ürünlerinin % 1.5 özel/TAE jel üzerinde analiz.
Not: Karışık hedeflenen nüfus genom mühendislik geçmiştir hücreler içeriyorsa, belirli bir PCR ürünü, Sigara hedef hücreleri (negatif kontrol) gDNA algılanamaz değil dikkat edilmelidir. Sigara hedefli alleli için PCR, tek bir ürün her iki hedeflenen ve sigara hedef hücreler için (olumlu denetim genomik P1 ve P4 astar için) aynı büyüklükte uymak gerekir. Hiçbir grup gözlenir, devir sayısını artırarak veya PCR arabellek (örneğin aracılığıyla DMSO veya artan miktarda Mg2 +eklenmesi) değiştirme veya polimeraz değiştirerek PCR Bisiklet koşullarını değiştirme göz önünde bulundurun.
3. doğru hedefleme için klonal hatları ve nesil tarama
Not: Sonra onay akış sitometresi ve PCR (bölümler 2.2-2.4), karma hedeflenen hücre nüfus hedefleme olayların tek hücre klonlar oluşturmak (süresi: 28-35 gün) ve muhabir sırasının doğru tümleştirme için ekran.
- Tek hücre klonlar seyreltme kaplama aracılığıyla nesil
- Jurkat şartına orta önceden hazırlamak: RPMI w / 1 x 106 hücre/ml, santrifüj, 300 x g, 5 min için yetiştirilen sağlıklı, işlenmemiş Jurkat T hücrelerinin AB ortamından çıkar ve 0,22 µm şırınga filtre birimini kullanarak süpernatant filtre.
Not: Kısa süreli depolama için 4 ° C'de veya depolama 1 haftadan uzun-20 ° C'de şartına orta tutmak. 20-30 mL şartına orta seyreltme kaplama önce hazırlayın. - Adım 2.2.4 10-14 gün sonra genişleme hedeflenen hücreleri saymak ve onları RPMI w / AB içinde bir konsantrasyon 1 x 105 hücre/mL seyreltik. 1 x 105 hücre/mL solüsyon 100 µL al ve 1000 hücre/mL konsantrasyonu elde etmek için orta ile 9.9 mL seyreltik. 1000 hücre/mL solüsyon 1 mL al ve 100 hücre/mL konsantrasyonu elde etmek için orta ile 9 mL seyreltik.
- Plaka 96-şey içeren 1 hücre başına iyi ve iyi başına 2 hücre tabaklar. 1 hücre iyi ücret için 1 mL 100 hücre/mL solüsyon alıp yavaşça 5 mL şartına orta ve 4 mL steril reaktif Reservoir taze orta ile karıştırın.
- 2 hücreleri iyi ücret için 100 hücre/mL solüsyon 2 mL alıp yavaşça şartına orta 5 mL ve taze orta 3 mL ile karıştırın. İyi bir çok kanallı damlalıklı kullanarak başına ilgili hücre seyreltme 100 µL ile plaka 96-şey yuvarlak-alt plakalar.
Not: 5 yapı hedefleme başına 10 96-şey plakaları için klonlar filtreleme için elde etmek yeterlidir. - 96-şey plakaları yığını, her yığında PBS her iyi 3 mL içeren bir 6-şey plaka ile kapak ve plakaları bir oksijen hücre kültür kuluçka %5 CO2 37 ° C'de 3 hafta kuluçkaya.
Not: Hücre kültür orta bu sırada değiştirmeyin. Kuluçka makinesi fazla bir veya iki kez bir hafta açık değil. En iyi sonuçları İnkübatörler açık su deposu ile gözlenir. - 3 hafta sonra kuluçka, görsel olarak yetiştirilen kolonileri ışık mikroskobu (4 X büyütme) kullanarak varlığını doğrulamak ve kuyu dibinde puan olarak görünmüyorsa bu yüzden wells yetişkin koloniler ile işaretleyin.
- RPMI w / AB 100 µL iyi ücret ile bir 96-şey yuvarlak-alt plaka hazırlayın. Yavaşça bir işaretli hücrelerinin resuspend de pipetting tarafından. Hücre süspansiyon, 100 µL zaten RPMI w / AB 100 µL içeren yeni 96-şey plaka bir kuyuya, daha sonra pipetting tarafından karışımı yavaşça. Bu hücre süspansiyon, 100 µL ikinci bir boş plaka çoğaltmak için 96-şey yuvarlak-alt plaka aktarın.
- Tüm işaretli wells yetişkin koloniler ile devam edin. Boş kuyu RPMI 200 µL w / AB orta ile doldurun. 37 ° C ve % 5 tabak kuluçkaya CO2.
Not: Bu plakaları biri hizmet edecek ("hisse senedi plaka") tek hücreli klonların genişleme ve diğer için tarama için bir "yinelenen tabak" olarak.
- Jurkat şartına orta önceden hazırlamak: RPMI w / 1 x 106 hücre/ml, santrifüj, 300 x g, 5 min için yetiştirilen sağlıklı, işlenmemiş Jurkat T hücrelerinin AB ortamından çıkar ve 0,22 µm şırınga filtre birimini kullanarak süpernatant filtre.
- Tek hücre klonlar akış sitometresi ve PCR testi ile taranması
Not: Tek hücre klonlar genişletiyoruz, ekran tek hücre klonlar yinelenen plakasına adım 3.1.8 muhabir sırası PCR (adımları 3.2.4–3.2.12) tarafından varlığı ve floresan muhabir akış sitometresi (adımları 3.2.2–3.2.3) tarafından ifade için kullanın (Şekil 3 c).- 24-48 h için kuluçkaya ve plaka tekrar yinelenen yinelenen tabağı ver. Bunu yapmak için her şey için RPMI AB w / 100 µL eklemek, karışımı yavaşça pipetting tarafından ve bir çok kanallı damlalıklı kullanarak yeni bir 96-şey yuvarlak-alt plaka 100 µL aktarmak. Akış Sitometresi tarama diğeri PCR tabanlı tarama bir levha kullanın.
- Akış Sitometresi tarama için PMA-Iono içeren hücreleri uyarmak. Ionomocin (1 mM hisse senedi), PMA 0.1 µL 0.1 µL mastermix hazırlamak (50 µg/µL hisse senedi) ve RPMI AB sayısına göre wells, w / 4,8 µL mastermix iyi başına 5 µL ekleyin.
Not: İndüksiyon nerede soldan sağa transcriptionally sessiz olabilir ve bu nedenle floresan muhabir değil ifade edilir klonlar, başarılı bir şekilde tanımlamak gereklidir. - 24 saat kuluçkaya ve hücreleri 2.3.3. adımda açıklandığı gibi akış sitometresi hazırlamak hücreleri izin. Herhangi bir tek-ileriye ve yana dağılım boyutu temel alan hücrelerin kapısı ve floresan muhabir gen ekspresyonu akış sitometresi tarafından analiz (örneğin sonuçları, bakınız Şekil 3 c). TV omurga organizatörü ikinci floresan gazeteci içeriyorsa (örneğin, GFP), aynı zamanda herhangi bir klon omurga muhabir ifade için ekran (adım 1.2.2 ve açıklama için aşağıdaki nota bakın).
Not: Omurga muhabir ifade omurga sıralarının istenmeyen Tümleştirme gösterir. - İkinci çift plaka klonlar yeterince büyüdü bir kez (genellikle 24-48 h çoğaltma 96-şey plaka), gDNA PCR tarama için içeren hücre lysates hazırlayın. Santrifüj plaka RT. dikkatle de 300 x g , 10 min için süpernatant al hücre Pelet bozmadan.
Not: Lysates ve PCR reaksiyonları hazırlanması için tüm adımları ile çok kanallı pipetler gerçekleştirilebilir. - Yumuşak pipetting ve RT. de 300 x g , 5 min için plaka centrifuging tarafından PBS 100 µL hücrelerle yıkama PBS almak ve 200 µL lizis arabelleği ekleyin [200 mM NaCl, 100 mM Tris-HCl pH 8-8.5, sonra 250-1000 µg/mL İndinavir K ekleyin 5 mM EDTA, % 0,1 SDS; (toz, tartmak taze liyofilize)] iyi başına. Pipetting tarafından karışımı yavaşça ve süspansiyon yeni bir PCR tabağa aktarın.
- Parafin film ile plaka mühür ve bir thermocycler 55 ° C'de 1 h için kuluçkaya. 10 dk (dönmeye 3000 x g) için maksimum hızda hücre artıkları santrifüj kapasitesi ve süpernatant yeni bir PCR tabağa aktarın.
Not: Hücre lysates tabak içinde saklanabilir, bu aşamada 4 ° c kadar kullanmaya devam etmenize. - DH2O 110 µL 96-şey PCR plakalı hazırlamak ve hücrenin lysate 10 µL ekleyin (1:12 seyreltme). Hücre lysates viskoz ve damlalıklı zor olabilir. En az 20 µL damlalıklı ipuçları.
- Bir thermocycler 99 ° C'de 10 dakika için kuluçka tarafından İndinavir K devre dışı bırakabilirsiniz. Daha sonra Inaktif ve seyreltilmiş hücre lysates PCR tarama için kullanın.
- 500 – 800 bp muhabir sırası yükseltmek için seçilen muhabir sıralamasına dayanan PCR (P5 ve P6) süzmek için astar tasarım. PCR olumlu denetim için bir vahşi-tipi, Sigara hedefli genomik odağı (NUP188 gen) 630 yükseltmek astar P7 ve P8 bp kullanmak (Şekil 3 c ve Tablo 5). Tv omurga sıralarının (omurga PCR) belirsiz tümleştirme için bir denetim olarak 500-600 bp tv omurga yükselterek üçüncü bir astar çifti Tasarla.
- Tarama, denetim ve omurga PCR için ticari bir PCR mastermix kullanın (bkz. Tablo 6 ve 7 için PCR malzemeler ve bisiklet koşullarını). Adım 3.2.8 şablon olarak hazırlanan ve PCR PCR güçlendirme 96-iyi biçimde 38-40 döngüleri çalıştırmak 2 µL seyreltilmiş ve Inaktif lysate kullanın.
- 5 µL PCR ürünlerinin % 1.5 özel/TAE jel üzerinde analiz.
Not: Denetim PCR, 630 belirli bir grup için bp hücre lysates kalitesini PCR için yeterli olduğunu teyit her örnek için gözlenen. PCR (astar tasarım bağlı olarak 500-800 bp) eleme içinde belirli bir grup muhabir sıra bütünleştirilmesi gösterir. Omurga PCR (500-600 bp, astar tasarım bağlı olarak) için belirli bir grup (örneğin sonuçları, bakınız Şekil 3 c) tv omurga sıralarının istenmeyen Tümleştirme gösterir. - Akış Sitometresi (adım 3.2.3) ve PCR tabanlı tarama (adım 3.2.12) sonuçlarını birleştirmek. PCR ve pozitif kontrol PCR ve floresan muhabir ifadeden sonra akış sitometresi içinde PMA-Iono ile indüksiyon eleme PCR ürünlerinin doğru boyutlarını göster klonlar seçin. Herhangi bir omurga PCR PCR ürünü veya ifade belirsiz entegrasyon-in tv omurga sıra gösteren tv omurga kodlanmış floresan protein, göstermek klonlar hariç.
- Yavaş yavaş daha büyük iyi formatları (48/24/12/6-de) seçili klonlar 96-şey hisse senedi plaka T75 hücre kültür şişesi kartvizitlere kadar elde her 2-3 gün taze orta ekleyerek genişletin. 1 x 105 ve 1 x 106 hücre/mL arasında bir hücre yoğunluğu korumak.
- Hücre stokları klonların genişleme sırasında hazırlamak emin olun: hücreleri saymak, santrifüj kapasitesi 300 x g RT, 5 min için de, süpernatant atmak ve hücrelerde yavaşça FCS + % 10 DMSO 5 x 106 hücre/mL, askıya alma. Kriyojenik şişeleri ve 1 ° C/dk 80 ° C'de hücrelere dondurmak için kullanım cryo-dondurma konteyner aliquot. Uzun süreli depolama için onları sıvı N2' ye naklet.
Not: GDNA hazırlık (bakınız Bölüm 3.4) Southern Blot tarafından hedef doğrulama için genişleme sırasında bir T75 hücre kültür şişesi (yani, 1 x 107 hücreleri) korumak için tavsiye edilir.
- PCR/sıralama tarafından entegrasyon Siteler'de seçili klonlar doğrulanması
Not: 5' ve 3' int kavşak seçili klonların vardır gönderilmiş ve güçlendirilmiş PCR Sanger için doğrulamak için sıralama düzeltmek DNA dizisi düzeyinde hedefleme.- GDNA seçili klonlar ve Jurkat vahşi-türü hücre bir ticari gDNA ayıklama kit kullanarak hazırlayın.
- Astar çiftleri 5' uç bir muhabir ve bir 5' HA 5' int Kavşağı (astar P1 ve P2) için ters yönde ve 3' ucu muhabir ve aşağı 3' HA 3' int Kavşağı (astar P3 ve P4) için 2.4 adımda anlatıldığı gibi bağlama kullanın. Astar P1 ve P4 alleli muhabir integrant (Şekil 4a) olmadan hedeflenen entegrasyon ücreti yükseltmek için kullanın.
- PCR reaksiyonları 100 – 200 ng ile gDNA bir şablon olarak hazırlamak ve PCR etkinlik düzelti ile bir polimeraz kullanarak gerçekleştirmek ( Tablo 1 ve 2 PCR malzemeler ve bisiklet koşullarını için bakınız).
Not: Yok bantları görülmektedir döngüleri artırıldığında veya (örneğin, DMSO veya artan miktarda Mg2 +ekleyerek), PCR arabellek değiştirme PCR Bisiklet koşullarını değiştirme göz önünde bulundurun ve polimeraz değiştirerek. - 5 µL PCR ürünlerinin % 1.5 özel/TAE jel üzerinde analiz. Eğer doğru şerit boyutları gözlenir, ticari bir seti kullanarak kalan PCR ürünü arındırmak ve Sanger için konu sıralaması. 5' int Kavşağı, 3' int kavşak ve hedef sitenin muhabir integrant olmadan alleli dizileri ile beklenen dizileri yerleştirerek, ikisi doğrulayın.
Not: Nerede muhabir entegre homolog alleli büyük olasılıkla Cas9-aracılı değişiklikleri hedef sitede nükleotit eklemelerin veya silmelerin (Şekil 4a) gibi gösterir. - Doğru int. bağlantı dizileri sonra hizalama göstermek klonlar için bütün hedeflenen muhabir yükseltecek bir PCR gerçekleştirmek ve Sanger için konu sıralama integrant doğru sırasını doğrulayın.
- Southern blot Analizi doğrulama seçili klonlar hedefleme için
Not: Seçili klonlar Southern blot Analizi doğru hedefleme doğrulamak ve hedeflenen tümleştirme sitesinde oluşmuş olabilir Cas9-aracılı rekombinasyon olaylar dışlamak için gereklidir.- Uygun gDNA sindirim için bir strateji geliştirmek ve tasarım deneme başlamadan önce yoklama.
- Restriksiyon enzimi 2 ile 10 kb uzunluğunda hedeflenen sitesinde uygun parçaları oluşturur gDNA kısıtlama için seçin. Bazı enzimleri, Asp718, BamHI, Bglgibi ben, BglII, EkoRV, HindIII, Astsubayben Pstben, PvuII, ani kalp durmasıben Stu, ve SST Yüksek molekül ağırlıklı gDNA sindirerek için başarılı bir şekilde kullanılmıştır.
- İki farklı Güney problar tasarlayın: bir gazeteci özel soruşturma ve genomik sonda. Muhabir özgü sonda bir diziye muhabir (yani, tdTomato özgü sonda) içinde hybridizes. Genomik sonda genomik bölgesine yakın (değil örtüşen ama) hybridizes bir HA.
- Genomik sonda bağlama tarafından tespit gDNA sindirim tarafından oluşturulan parçası uzunluğu (fazla 2 kb) hedefli ve sigara hedefli gen (Şekil 4b) üzerinden farklı genomik sonda seçin. Bir sonda uzunluğu 400 ila 1000 bp önerilir.
- PCR astar iki gerekli problar yükseltmek için tasarım. Muhabir özgü sonda bir yüksek-sadakat DNA polimeraz kullanarak tv şablondan yükseltmek (bkz: Tablo 3 ve 4 PCR malzemeler ve bisiklet koşullarını).
- Genomik sonda gelen DNA polimeraz etkinlik düzelti ile kullanarak bir ticari gDNA ekstraksiyon kiti ile hazırlanan vahşi tipi Jurkat gDNA yükseltmek ( Tablo 1 ve 2 PCR malzemeler ve bisiklet koşullarını için bakınız). Bir özel/TAE jel PCR ürünlerde arındırmak ve üreticinin yönergelerine göre ticari jel ekstraksiyon kiti kullanarak parçaları ayıklayın.
- Yüksek molekül ağırlıklı gDNA vahşi tipli Jurkat hücreleri ve adım 3.2.14 seçili klonlar 1 x 107 hücreleri ayıklamak.
- Hücreleri tarafından Santrifüjü 300 x g RT, 5 min için de cips, bir kez PBS ile yıkayın ve Pelet 4 mL lizis arabellek askıya alma [200 mM NaCL, 100 mM Tris-HCl pH 8, 5 mM EDTA, % 0,1 SDS; sonra 250-1000 µg/mL İndinavir K (liyofilize toz ekleyin taze tartmak içinde)]. O/n 55 ° c, Masa üstü thermomixer 350 rpm'de sallayarak kuluçkaya.
- İsopropanol 4 mL ve inversiyon tarafından 10-20 kez karıştırın. GDNA beyaz çökelti görünür hale gelmelidir. Üzerine ince bir cam pipet ucu zarlarını gDNA biriktirmek, %70 alkol ve izin 750 µL RT (5-10 dk) Kuru ortaya çıkan tarafından yıka.
- Çökelti 500 µL 1 x TE arabelleği (10 mM Tris-HCl pH 8.0, 1 mM EDTA) içeren bir 1,5 mL tepki tüp içine dökmek ve o/n 350 rpm'de sallayarak 4 ° C'de çözülmeye bırakın. Herhangi bir gDNA bu sahneden pipetting wide-geçişli ipuçları ile kesme önlemek için yapılmalıdır.
Not: Yüksek molekül ağırlıklı gDNA hazırlıktır Southern blot analizi için temel ve piyasada bulunan gDNA hazırlık setleri uygun değildir.
- GDNA seçili klonlar ve vahşi-tipi Jurkat hücre seçili Restriksiyon enzimi ile Özet (iki kez) 15 µg (bkz. Adım 3.4.1.1) enzim (20 adet/µL) 6 µL ile bir 60 µL tepki: Birincisi, DNA, eklemek sindirim tampon ve GKD2O, o/n 37 ° C'de kuluçkaya , daha sonra enzim ekleyin ve o/n, enzim özgü sindirim sıcaklığında kuluçkaya. sindirilmiş gDNA 15 ug Güney sonda gereklidir.
- 60 µL kısıtlama Özet 7 µL % 1'özel / TAE jel üzerinde analitik Jel Elektroforez için kullanın. Bir smear tam sindirim ve DNA kaliteli Southern blot analizi için gösterir.
- 1:10 ekleyerek kalan kısıtlama Özet çökelti 3 M sodyum asetat ve 2 Cilt %100 alkol,-80 ° C ve santrifüj 15,600 x g 4 ° C'de 30 min için 1 h için kuluçkaya
- Süpernatant atmak ve Pelet % 70 ile yıkayın alkol. 15,600 x g 4 ° c de 15 dakika santrifüj kapasitesi, süpernatant atmak, kısaca RT kuru ve GKD2O. 20 µL içinde erimesi Pelet izin
- Sindirilmiş gDNA lane başına 20 uL yükleniyor % 1'özel / TAE kurutma jel, çalıştırın. 2s için jel 60 V, 400 çalışan anne.
Not: Özel jel ve koşma zaman/gerilim yüzdesi 3.4.1.1 adımda hesaplanan Southern blot algılama için beklenen parça büyüklüğüne göre ayarlanabilir. Southern blot Analizi aşağıdaki adımlardan ayrıntılı ek Protokolü (adım 1-18) olarak açıklanmıştır. Aşağıdaki adımları oluşmaktadır: naylon membran, radyoaktif sonda üretimi, sonda hibridizasyon ve autoradiograph film gelişimi kurutma blotting jel yıkama. Adım 18 (Ek Protokolü) ile beklenen kalıba uygun Güney strateji geliştirmede autoradiograph sonra elde edilen bantlama deseni karşılaştırın (örneğin sonuçları, bakınız Şekil 4b).
- Uygun gDNA sindirim için bir strateji geliştirmek ve tasarım deneme başlamadan önce yoklama.
- Hedef dışı olayların analizi
Not: CRISPR-Cas9-aracılı genom mühendislik hedef kapalı etkileri, sıralaması en yüksek içinde silisPCR-yükseltmek on oluşturabilirsiniz beri-hedef Siteler'de seçili klonlar ve onları Sanger için tabi tahmin sıralama.- Sıralaması en yüksek on bir listesini oluşturmak için CCTop16 (http://crispr.cos.uni-heidelberg.de) kullanın silico öngörülen hedef kapalı dizileri.
- Sorgu sırası yönlendirilmesi gibi PAM de dahil olmak üzere gRNA sıra giriş. PAM ve "İnsan genomu" olarak "NGG" için hedef tahmin referans olarak seçin.
- Ayarla maksimal toplam uyuşmazlıkları "4" ve hedef site PAM olmadan gRNA uzunluğu uzunluğu. Çıktı dosyası için ilgili gRNA sırada genomik hedef kapalı sitelerin listesini sağlayacaktır.
- Silico genomik sıra 500 bp akış yukarı ve aşağı akım her birinin UCSC genom tarayıcı (http://genome.edu.ucsc.edu) ve off-CCTop sonuç listesinden vurmak hedef konumunu kullanarak 10 sıralaması en yüksek hedef kapalı sayısı ayıklayın.
- Her analiz için hedef siteleri off, 600-700 parçası PCR kısaca çift tasarım yükselterek bp öngörülen hedef kapalı site de dahil olmak üzere uzunluğu.
- GDNA seçili klonlar ve Jurkat vahşi tipi hücreler bir ticari gDNA ayıklama kit kullanarak ayıklayın. Her hedef kapalı site için bir PCR DNA polimeraz etkinlik düzelti ile kullanarak gerçekleştirmek (bkz. Tablo 1 ve 2 için PCR malzemeler ve bisiklet koşullarını) vahşi-türü ve ilgili klon elde edilen gDNA.
- 5 µL PCR ürünlerinin % 1.5 özel/TAE jel üzerinde analiz. Eğer doğru şerit boyutları gözlenir, ticari bir PCR arıtma kit kullanarak kalan PCR ürünü arındırmak ve Sanger için konu sıralaması. Hedef kapalı siteleri Jurkat hücrelerde ve hedeflenen klonlar dizileri karşılaştırın.
- Sıralaması en yüksek on bir listesini oluşturmak için CCTop16 (http://crispr.cos.uni-heidelberg.de) kullanın silico öngörülen hedef kapalı dizileri.
Sonuçlar
Temsilcisi bu deneyde LTR, tdTomato kodlama dizisi ve iki loci intron 5 BACH2 gen17dizisine polyA-sinyal oluşan en az bir HIV-1-türetilmiş muhabir hedeflemek seçtiniz. Hedefleme için loci yayımlanmış tekrarlayan tümleştirme yerler HIV infekte hastaların2,4,5,6birincil T hücreleri üzerinde farklı çalışmalarda bulundu ...
Tartışmalar
Burada, CRISPR-Cas9-esaslı genom mühendislik uygulama seçilen proviral tümleştirme sitelerle HIV-1-türetilmiş Jurkat muhabir modelleri oluşturmak için bir protokol açıklayın.
İletişim kuralı birkaç puan planlama aşamasında dikkat gerektirir. Gibi bazı loci diğerlerinden daha hedef için daha kolay olabilir ilk olarak, var olmak hedef için odağı dikkatli seçilmelidir (örneğin, bölge ve hedef kromatin durumuna bağlı olarak kendisi sıra). Tekrarlayan diziler...
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Britta Weseloh ve Bettina Abel teknik yardım için teşekkür ederiz. Biz de Arne Düsedau ve Jana Hennesen (akış sitometresi teknoloji platformu, Heinrich Pette Institut) Teknik destek için teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| pX330-U6-Chimeric_BB-cBh-hSpCas9 | Addgene | 42230 | vector for expression of SpCas9 and gRNA |
| pMK | GeneArt | mammalian expression vector for cloning | |
| cDNA3.1 | Invitrogen | V79020 | mammalian expression vector for cloning |
| BbsI | New England Biolabs | R0539S | restriction enzyme |
| NEBuilder Hifi DNA Assembly Cloning Kit | New England Biolabs | E5520S | Assembly cloning kit used for target vector generation |
| TaqPlus Precision PCR System | Agilent Technologies | 600210 | DNA polymerase with proofreading activity used for amplification of homology arms (step 1.2.2.2), verification of integration site and reporter sequence (step 3.3.3 and 3.3.5), generation of genomic probe for Southern blot (step 3.4.1.5) and analysis of off-target events (step 3.5.4) |
| 96-well tissue culture plate (round-bottom) | TPP | 92097 | tissue culture plates for dilution plating |
| Phusion High-Fidelity DNA polymerase | New England Biolabs | M0530 L | DNA polymerase used for detection of targeting events (step 2.4.2) and generation ofreporter-specific probe for Southern blot (step 3.4.1.4) |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D9170 | dimethyl sulfoxide as PCR additive |
| Magnesium Chloride (MgCl2) Solution | New England Biolabs | B9021S | MgCl2 solution as PCR additive |
| Deoxynucleotide (dNTP) Solution Mix | New England Biolabs | N0447S | dNTP mixture with 10 mM of each nt for PCR reactions |
| 5PRIME HotMasterMix | 5PRIME | 2200400 | ready-to-use PCR mix used for screening PCR (step 3.2.11) |
| QIAamp DNA blood mini kit | Qiagen | 51106 | DNA isolation and purification kit |
| QIAquick PCR Purification Kit | Qiagen | 28106 | PCR Purification Kit |
| RPMI 1640 without glutamine | Lonza | BE12-167F | cell culture medium |
| Fetal Bovine Serum South Africa Charge | PAN Biotech | P123002 | cell culture medium supplement |
| L-glutamine | Biochrom | K 0282 | cell culture medium supplement |
| Penicillin/Streptomycin 10.000 U/mL/ 10.000 µg/mL | Biochrom | A 2212 | cell culture medium supplement |
| Gibco Opti-MEM Reduced Serum Media | Thermo Fisher Scientific | 31985062 | cell culture medium with reduced serum concentration optimized for transfection |
| TransIT-Jurkat | Mirus Bio | MIR2125 | transfection reagent |
| phorbol 12-myristate 13-acetate | Sigma-Aldrich | P8139-1MG | cell culture reagent |
| Ionomycin | Sigma-Aldrich | I0634-1MG | cell culture reagent |
| Syringe-driven filter unit, PES membrane, 0,22 µm | Millex | SLGP033RB | filter unit for sterile filtration |
| Heracell 150i incubator | Thermo Fisher Scientific | 51026280 | tissue culture incubator |
| Amershan Hybond-N+ | GE Healthcare | RPN1520B | positively charged nylon membrane for DNA and RNA blotting |
| Stratalinker 1800 | Stratagene | 400072 | UV crosslinker |
| High Prime | Roche | 11585592001 | kit for labeling of DNA with radioactive dCTP using random oligonucleotides as primers |
| illustra ProbeQuant G-50 Micro Columns | GE Healthcare | 28-9034-08 | chromatography spin-columns for purification of labeled DNA |
Referanslar
- Hughes, S. H., Coffin, J. M. What Integration Sites Tell Us about HIV Persistence. Cell Host and Microbe. 19 (5), 588-598 (2016).
- Marini, B., Kertesz-Farkas, A., et al. Nuclear architecture dictates HIV-1 integration site selection. Nature. 521 (7551), 227-231 (2015).
- Cesana, D., Santoni de Sio, F. R., et al. HIV-1-mediated insertional activation of STAT5B and BACH2 trigger viral reservoir in T regulatory cells. Nature Communications. 8 (1), 498 (2017).
- Cohn, L. B., Silva, I. T., et al. HIV-1 Integration Landscape during Latent and Active Infection. Cell. 160 (3), 420-432 (2015).
- Han, Y., Lassen, K., et al. Resting CD4+ T cells from human immunodeficiency virus type 1 (HIV-1)-infected individuals carry integrated HIV-1 genomes within actively transcribed host genes. Journal of Virology. 78 (12), 6122-6133 (2004).
- Ikeda, T., Shibata, J., Yoshimura, K., Koito, A., Matsushita, S. Recurrent HIV-1 integration at the BACH2 locus in resting CD4+ T cell populations during effective highly active antiretroviral therapy. The Journal of Infectious Diseases. 195 (5), 716-725 (2007).
- Wagner, T. A., Mclaughlin, S., et al. Proliferation of cells with HIV integrated into cancer genes contributes to persistent infection. Science. 345 (6196), 570-573 (2014).
- Maldarelli, F., Wu, X., et al. Specific HIV integration sites are linked to clonal expansion and persistence of infected cells. Science. 345 (6193), 179-183 (2014).
- Jordan, A., Bisgrove, D., Verdin, E. HIV reproducibly establishes a latent infection after acute infection of T cells in vitro. The EMBO Journal. 22 (8), 1868-1877 (2003).
- Mack, K. D., Jin, X., et al. HIV insertions within and proximal to host cell genes are a common finding in tissues containing high levels of HIV DNA and macrophage-associated p24 antigen expression. Journal of Acquired Immune Deficiency Syndromes. 33 (3), 308-320 (2003).
- Byrne, S. M., Ortiz, L., Mali, P., Aach, J., Church, G. M. Multi-kilobase homozygous targeted gene replacement in human induced pluripotent stem cells. Nucleic Acids Research. 43 (3), 1-12 (2014).
- CRISPR Genome Engineering Toolbox: Target Sequence Cloning Protocol. Addgene website Available from: https://www.addgene.org/static/cms/filer_public/e6/5a/e65a9ef8-c8ac-4f88-98da-3b7d7960394c/zhang-lab-general-cloning-protocol.pdf (2013)
- Gibson Assembly Protocol. Addgene website Available from: https://www.addgene.org/protocols/gibson-assembly/ (2009)
- Addgene Plasmid Cloning by PCR. Addgene website Available from: https://www.addgene.org/protocols/pcr-cloning/ (2014)
- Addgene Plasmid Cloning by Restriction Enzyme Digest (aka Subcloning). Addgene website Available from: https://www.addgene.org/protocols/subcloning/ (2013)
- Stemmer, M., Thumberger, T., Del Sol Keyer, M., Wittbrodt, J., Mateo, J. L. CCTop: An intuitive, flexible and reliable CRISPR-Cas9 target prediction tool. Public Library of Science (PLoS) ONE. 10 (4), (2015).
- Lange, U. C., Bialek, J. K., Walther, T., Hauber, J. Pinpointing recurrent proviral integration sites in new models for latent HIV-1 infection. Virus Research. 249, (2018).
- Bialek, J. K., Dunay, G. A., et al. Targeted HIV-1 Latency Reversal Using CRISPR-Cas9-Derived Transcriptional Activator Systems. PloS ONE. 11 (6), e0158294 (2016).
- Lee, C. M., Davis, T. H., Bao, G. Examination of CRISPR-Cas9 design tools and the effect of target site accessibility on Cas9 activity. Experimental Physiology. 103 (4), 456-460 (2018).
- Jensen, K. T., Fløe, L., et al. Chromatin accessibility and guide sequence secondary structure affect CRISPR-Cas9 gene editing efficiency. FEBS Letters. 591 (13), 1892-1901 (2017).
- Simonetti, F. R., Sobolewski, M. D., et al. Clonally expanded CD4 + T cells can produce infectious HIV-1 in vivo. Proceedings of the National Academy of Sciences. 113 (7), 1883-1888 (2016).
- Chen, H. C., Martinez, J. P., Zorita, E., Meyerhans, A., Filion, G. J. Position effects influence HIV latency reversal. Nature Structural and Molecular Biology. 24 (1), 47-54 (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır