A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تطبيق التصوير الخلوي المباشر والتصوير المقطعي بالتبريد الإلكترون لحل الميزات الزمنية للالفيلقية الهوائية Dot/Icm نظام إفراز
In This Article
Summary
تصوير الخلايا البكتيرية هو نهج بيولوجيا الأنظمة الناشئة الذي يركز على تحديد العمليات الثابتة والديناميكية التي تملي وظيفة الآلات الجزيئية الكبيرة. هنا ، يتم استخدام التكامل بين التصوير الكمي للخلايا الحية والتصوير المقطعي بالإلكترونات الباردة لدراسة Legionella الرئوية من نوع IV بنية نظام الإفراز ووظائفه.
Abstract
نظام إفراز Dot/Icm من الفيلق الرئوي Legionella هو نظام إفراز من النوع الرابع (T4SS) جهاز نانوي معقد يُجتار في القطب البكتيري ويتوسط في تسليم ركائز البروتين والحمض النووي إلى الخلايا المستهدفة ، وهي عملية تتطلب بشكل عام الاتصال المباشر من خلية إلى خلية. لقد حللنا مؤخرًا بنية جهاز Dot /Icm بواسطة التصوير المقطعي بالإلكترونات (cryo-ET) وأظهرنا أنه يشكل قناة ممتدة للغلاف الخلوي تتصل بمجمع سيتوبلازمي. تطبيق نهجين متكاملين التي تحافظ على الهيكل الأصلي للعينة، المجهر الفلوري في الخلايا الحية والتبريد-ET، يسمح في الموقع تصور البروتينات واستيعاب قياس الخلايا وتوقيت إنتاج كل مكون آلة بالنسبة إلى غيرها من الوحدات الفرعية نقطة / Icm. للتحقيق في متطلبات تحديد المواقع القطبية وتوصيف الميزات الديناميكية المرتبطة بالتكوين الحيوي لآلة T4SS ، قمنا بدمج جين ترميز بروتين الفلورسنت الأخضر فوق المجلد إلى جينات Dot / Icm ATPase في مواقعها الأصلية على الكروموسوم. تدمج الطريقة التالية المجهر المجهري للفلورية الكمية للخلايا الحية والتبريد-ET لتحديد التوطين القطبي وديناميكيات وبنية هذه البروتينات في الخلايا البكتيرية السليمة. تطبيق هذه النهج لدراسة الفيلق ية الهوائية T4SS مفيد لتوصيف وظيفة نظام نقطة / Icm ويمكن تكييفها لدراسة مجموعة واسعة من مسببات الأمراض البكتيرية التي تستخدم T4SS أو أنواع أخرى من مجمعات إفراز البكتيريا.
Introduction
الفيلقية الرئوية (L. الرئوية),العامل المسببات لمرض الفيلق, يسكن خزانات المياه العذبة, حيث تنتشر البكتيريا عن طريق إصابة وتكرار داخل البروتوزوا المائية الحرة السباحة. L. الرئوي يسبب تفشي الأمراض في البشر عندما استنشاق البكتيريا الهباء الجوي من مصادر مياه الشرب يحدث. في الخلايا المصابة، تخريب مسارات المضيف يسمح L. الرئوي لتأخير النضج الانديسيمن اللافوال الذي يقيم فيه وتعزيز التكوين الحيوي للحجرة الخلوية التي تدعم النسخ البكتيري. والدافع وراء هذه العملية من قبل نوع البكتيريا المتخصصة IVB إفراز النظام (T4BSS) المعروفة باسم نقطة / Icm ذخيرتها من أكثر من 300 "المؤثرات" البروتينات التي يتم نقلها إلى السيتوسول المضيف أثناء العدوى لتسهيل التلاعب وظائف الخلوية1،2،3،4،5. المسوخ التي تفتقر إلى جهاز نقطة وظيفية / Icm تفشل في تقديم المؤثرات في السيتوسول المضيف ، معيبة للتكرار داخل الخلايا ، وهي في نماذج الحيوانات من المرض6،7.
وقد وضعت العديد من الأنواع البكتيرية آلات متعددة المكونات معقدة للغاية وديناميكية المطلوبة لعمليات العدوى. T4BSS أخرى مثل نظام نقطة / Icm ضرورية أيضا لتكرار داخل الخلايا من مسببات الأمراض البكتيرية مثل Coxiella burnetii وريكيتسيلا غريللي. على الرغم من أن T4BSS ترتبط تطورًا بأنظمة IVA من النوع النموذجي ، والتي تتوسط في نقل الحمض النووي ويمكن أن توفر ذخيرة محدودة من البروتينات المؤثرات ، فإن نظام Dot / Icm يحتوي على ضعف العديد من مكونات الجهاز ويسلم مجموعة واسعة من المؤثرات. ويفترض أن هذا التوسع في عدد من المكونات قد مكنت جهاز نقطة / Icm لاستيعاب ودمج المؤثرات الجديدة بسهولة8،9.
لقد استخدمنا مؤخرًا التصوير المقطعي بالإلكترونات (cryo-ET) لحل بنية جهاز Dot/Icm في الموقع وأظهرنا أنه يشكل قناة ممتدة للغلاف الخلوي تتصل بمجمع سيتوبلازمي. وكشف مزيد من التحليل أن CYTOsolic ATPase DotB المنتسبين مع نظام نقطة / Icm في القطب L. خلية الرئوية من خلال التفاعلات مع ATPase DotO الخلوي. لقد اكتشفنا أن DotB يعرض حركة دورية في معظم الخلايا البكتيرية ، مما يشير إلى أن هذا ATPase موجود في مجموعة دورية ديناميكية ولكنه يرتبط أيضًا بمجمعات Dot / Icm القطبية. بالإضافة إلى ذلك ، تشكل DotO تجميعًا حشسيًا من أجهزة الديمون DotO المرتبطة بمجمع الغشاء الداخلي ، وينضم سداسي DotB إلى قاعدة هذا المجمع السيتوبلازمي. تجميع مجمع الطاقة DotB-DotO يخلق قناة السيتوبلازمية التي توجه نقل الركائز من خلال T4SS(الشكل 1)10.
على الرغم من هذه التطورات الأخيرة ، لا يعرف سوى القليل عن كيفية وظائف نظام Dot / Icm وكيف يتجمع كل بروتين لتشكيل جهاز نشط8. الكشف عن الدوائر التنظيمية للنقطة / Icm T4SS أمر أساسي لفهم الآليات الجزيئية للتفاعلات المضيفة الممرض. لذلك ، نناقش كيفية استخدام المجهر الخلية الحية والتبريد ET للكشف عن وتوصيف الأساسية L. الرئوية دوت / Icm مكونات النظام التي يتم وضع علامة مع سوبر المجلد GFP (sfGFP). باستخدام المجهر الفلوري الكمي ، سيتم تعريف التعريب القطبي لـ DotB في خلفية من النوع البري أو عند حذف نظام النوع الرابع. سيتم استخدام المجهر الفاصل الزمني لقياس الاختلافات في التعريب والديناميكيات بين ATPases Cytosolic Dot/Icm.
التطبيق المشترك لنهجين متكاملين مثل التصوير الحي والتبريد ET يوفر ميزة بالمقارنة مع غيرها من الأنظمة المختبرية. يتم تنفيذ كلتا الطريقتين في خلايا سليمة والحفاظ على البيئة الطبيعية للT4BSS، وبالتالي تقليل اضطراب الهيكل الأصلي أثناء إعداد العينة. لأن الإفراط في التعبير عن البروتينات قد يضعف قياس التروية من جهاز إفراز، يتم إرجاع الاندماجsfGFP عن طريق تبادل أليليإلى كروموسوم الفيلقية بحيث يتم ترميز كل الانصهار في نسخة واحدة ويحرك التعبير من قبل المروج الذاتية. تصور الانصهار الكروموسومات المشفرة تمكن من القياس الكمي للمستوى الدقيق للبروتين الذي يتم التعبير عنه في نقطة زمنية محددة. Cryo-ET أيضا العديد من المزايا لتحديد هيكل أنظمة إفراز. الميزة الأبرز هي أن عينات Cryo-ET تتكون من خلايا مجمدة سليمة تحافظ على المجمعات الأصلية في سياق بنية الخلايا البكتيرية. وبالتالي، قد يكون من الأفضل للكبوو-ET نهج تنقية البيوكيميائية، التي تستخرج المجمعات الأغشية وقد تجرد البروتينات الطرفية من الجهاز الأساسي أو تعديل الهيكل العام. بالإضافة إلى ذلك ، وضع علامات على بروتين مهم مع بروتين ضخم مثل sfGFP يضيف كتلة يمكن اكتشافها بواسطة Cryo-ET ويمكن أن تساعد في رسم خرائط المجمعات الفرعية المختلفة لجهاز Dot / Icm على الهيكل الذي تم الحصول عليه بواسطة cryo-ET.
هذا النهج هو أداة قوية للكشف عن المعلومات الهيكلية حول المجمعات المتعددة الجزيئية التي تتجمع في غشاء الخلية البكتيرية. سيساعد تفسير الهياكل التي تم توضيحها باستخدام هذه التقنيات الحقل على فهم كيفية عمل مكونات T4BSS ، ولماذا هناك حاجة إلى العديد من المكونات للوظيفة ، وكيف تتفاعل المكونات داخل المجمع الأكبر ، وما هي وظائف هذه التجميعات الفرعية تنفيذ.
Protocol
ملاحظة: يجب إجراء جميع الإجراءات التي تنطوي على نمو وتلاعب وتصوير L. الرئوي ة في مختبر مستوى السلامة البيولوجية 2 وفقا للمبادئ التوجيهية المحلية.
1. إدراج sfGFP في L. كروموسوم الرئوي باستخدام تبادل أليليتش واستراتيجية الاختيار المزدوج(الشكل 2، الشكل 3)
- استنساخ في ناقلات استبدال الجينات pSR47S11 التسلسل التالي: 1000 bp المنبع من موقع الفائدة، ثم تسلسل sfGFP، ثم 1000 bp المصب من موقع الفائدة(الشكل 2). يجب وضع تسلسل sfGFP في الإطار إلى N-terminus أو C-terminus ينتهي مع الرابط الذي يحتوي على أربعة إلى ثمانية الأحماض الأمينية. تحويل ناقلات الناتجة إلى E. coli DH5αаpir. في وقت لاحق، سلسلة L. الرئوي (المتلقي) لمستعمرات واحدة على استخراج الفحم الخميرة (CYE) أغار12 التي تحتوي على 100 ميكروغرام / مل ستريبتومسين وتنمو لمدة 5 أيام في 37 درجة مئوية(الشكل 3).
- الشرائط L. الرئوية على CYE-agar-streptomycin وتنمو لمدة 2 أيام في 37 درجة مئوية (التصحيح الثقيلة)10. المتتالية E. coli DH5α تحولت مع pRK600 المساعد plasmid (مساعد)13 على LB أغار تحتوي على 25 ميكروغرام / مل الكلورامفينيكول. Streak E. coli DH5αаpir (المانحة) على LB أجار تحتوي على 50 ميكروغرام / مل kanamycin.
- قم بالتزاوج الثلاثي الوالدين: احتضان مستعمرة المساعد، مستعمرة المتبرع، والمتلقي عن طريق تراكب بقع من السلالات الثلاث على لوحة أغار CYE دون اختيار واحتضان لمدة 4-8 ساعة عند 37 درجة مئوية. كما الضوابط السلبية احتضان المساعد + المتلقي مزيج سلالة والمانح + المتلقي سلالة مزيج لنفس الفترات من الزمن.
- إعادة تعليق ردود فعل التزاوج في 500 ميكرولتر من ddH2O. لوحة 20 ميكرولتر و 50 ميكرولتر من ردود الفعل على CYE أغار التي تحتوي على 100 ميكروغرام / مل ستريبتومسين و 10 ميكروغرام / مل kanamycin وتنمو لمدة 5 أيام عند 37 درجة مئوية. المتتالية أربعة من المستنسخات الناتجة على CYE أجار التي تحتوي على 100 ميكروغرام / مل ستريبتومسين وتنمو لمدة 5 أيام عند 37 درجة مئوية.
- المتتالية 16 استنساخ على أجار CYE تحتوي على 5٪ السكروز و 100 ميكروغرام / مل ستربتوموسين وتنمو لمدة 5 أيام في 37 درجة مئوية. ثم، سلسلة 32 من هذه المستنسخات على CYE أغار تحتوي على 100 ميكروغرام/مل ستريبتومسين وعلى CYE agar التي تحتوي على 100 ميكروغرام/مل ستريبتومسين و 10 ميكروغرام/مل كاناموسين وتنمو لمدة 5 أيام عند 37 درجة مئوية.
2. عزل المستنسخين التي دمجت sfGFP في كروموسوم L. الرئوي
- المستنسخات المتتالية التي كانت حساسة لkanamycin على لوحات CYE-agar-streptomycin وتأكيد إدخال sfGFP في الكروموسوم مع PCR مستعمرة. استخدام التمهيديات التي هي مكملة للجين sfGFP والمنطقة الكروموسومية ذات الأهمية لتضخيم تقاطع الإدراج.
- مزيج 0.5 ميكرولتر من كل من مُحلول الكبيات 10 ميكرومتر ومستعمرة واحدة إلى حجم نهائي قدره 12.5 ميكرولتر وتشوه لمدة 10 دقيقة عند 95 درجة مئوية. تبرد على الجليد لمدة 10 دقيقة، إضافة 12.5 ميكرولتر من محلول المزيج الرئيسي PCR 2x، وإجراء تحليل PCR.
- تنمو بقع ثقيلة من المستعمرات المعزولة على لوحات CYE-agar-streptomycin لمدة يومين في 37 درجة مئوية. فحص مستويات التعبير والاستقرار من الانصهار sfGFP عن طريق النفخ المناعي مع الأجسام المضادة GFP.
3. تصوير الخلية الحية من L. الرئوي مع الفلورسنت الموسومة نقطة / Icm مكونات
- إعداد منصات أغاروز
- جعل حوالي 30 مل من محلول أغروس منخفض الذوبان بنسبة 1٪ في الماء. الميكروويف في قارورة زجاجية لحوالي 90 ث، يحوم في بعض الأحيان، حتى يذوب الأغروس تماما.
- ضع شريحتين زجاجيتين 22 × 22 × 0.15 مم3 على حافة شريحة زجاجية 25 × 75 × 1.1 مم3 ، واحدة فوق الأخرى. كومة اثنين أكثر 22 × 22 × 0.15 ملم3 شرائح الزجاج على الحافة الأخرى.
- ماشيت حوالي 1 مل من agarose المنصهر في الشريحة الوسطي بين اثنين من الشرائح الزجاجية العليا، ثم وضع آخر 25 × 75 × 1.1 ملم3 الشريحة على رأس agarose المنصهر. في محاولة لتجنب تشكيل فقاعات الهواء. تبريد الشرائح في 4 درجة مئوية لمدة 15 دقيقة.
- باستخدام مشرط أو شفرة حلاقة، وقطع بلطف وسادة إلى مربعات صغيرة، ~ 5 × 5 ملم2. قم بإصلاح إطار لاصق على الوجهين 17 × 28 × 0.25 مم3 على شريحة زجاجية مقاس 25 × 75 × 1.1 مم3 ووضع عدة منصات على الشريحة.
- الحصول على صورة
ملاحظة: يتم وصف الخطوات التالية للمجهر الذي هو تحت سيطرة SlideBook 6.0 ومجهزة مضيئة الحالة الصلبة، وكاميرا أحادية اللون CCD، وعدسة موضوعية 100x (1.4 الفتحة العددية). إذا لزم الأمر، استخدم أجهزة فحص مجهرية بديلة مع تكوينات الأجهزة والبرامج المناسبة التي يمكن تخصيصها وفقًا لإعدادات البروتوكول.- حل رقعة ثقيلة من L. الرئوي في 1 مل من ddH2O، دوامة وpipet 2-3 μL من تخفيف على منصات. ضع قسيمة تغطية 50 × 24 × 0.15 مم3 بلطف فوق الإطار اللاصق.
- في نافذة الالتقاط ضبط ND إلى 180. ضبط binning إلى 2 ×2 واستخدام قناة 488 نانومتر لفضح العينة بين 500-1000 ms. التحقق من خصوصية إشارة الفلورية عن طريق التصوير L. الرئوي غير الموسومة مع نفس المعلمات(الشكل 4).
4. القياس الكمي للتوطين القطبي وديناميات مكونات نقطة / Icm
ملاحظة: تم تصميم الخطوات التالية للصور مع 0.129 ميكرومتر لكل بكسل التي تم الحصول عليها مع 2 × 2 binning.
- قياس كمية القطبية لبروتينات الانصهار sfGFP(الشكل 5)
- ضبط تباين الصورة بحيث تكون البكتيريا مرئية بوضوح. استخدم أداة المنطقة لوضعمستطيل 2 0.25 × 1.3 ميكرومتر يبدأ من القطب ويمتد إلى السيتوبلازم. يجب أن يبقى المستطيل على وجه التحديد داخل الحدود البكتيرية.
- ضع علامة على ما لا يقل عن 200 بكتيريا واستخدم زر Region to Mask لإنشاء أقنعة للمناطق المثيرة للاهتمام. ضمن إحصائيات القناع ونطاق القناع،اختر الكائن. ثم، تحت الميزات والكثافة،علامة متوسط كثافة والتباين.
- تصدير البيانات وحساب درجات القطبية من كل بكتيريا كنسبة بين الفرق إلى متوسط كثافة.
- للتطبيقات عالية الإنتاجية استخدام هدف المرحلة وإعداد مكثف المناسبة للحصول على الصور مع المرحلة وقنوات 488 نانومتر. تأكد من اختيار مجالات الرؤية حيث يتم فصل البكتيريا تماما.
- ضبط تباين قناة المرحلة للصورة إلى مستوى تكون فيه البكتيريا مرئية بوضوح. افتح صورة القناة المزدوجة، وأطلق نافذة إنشاء قناع الشريحة وغيّر القناة إلى مرحلة.
- ضبط عتبة مناسبة وإزالة الكائنات الصغيرة مع الزر تعريف الكائنات. ضمن تحسين قناع اختيار إزالة حواف الكائنات وأقنعة منفصلة في وقت لاحق من البكتيريا التي هي متجاورة مع بعضها البعض.
- احسب درجات الاستقطاب للإشارة في القناة 488 لكل خلية كما تم وصفها سابقًا في الخطوات 4.1.2-4.1.3.
- القياس الكمي لديناميكيات بروتينات الاندماج sfGFP(الشكل 6)
ملاحظة: اتبع الإرشادات الواردة في القسم 3 لإعداد عينة للحصول على صورة. يتم تعريف الديناميكيات على أنها تغييرات في الكثافة بمرور الوقت ويتم تصميم الخطوات التالية لفترات التصوير القصيرة (أي عدة دقائق). إضافة إلى لوحة المكملات الغذائية المناسبة إذا كانت مطلوبة فترات التصوير أطول.- في إطار التقاط الصور، ضع علامة Timelapse،أدخل وقت الفواصل الزمنية في مربع الفاصل الزمني، وأدخل 2 في مربع # النقاط الزمنية. الحصول على صورتين متتالية من L. الرئوي التعبير عن البروتين الفلوري من الفائدة.
- ضبط تباين الصورة حتى تكون الخلايا مرئية بوضوح. اتبع الشكل 6A والأوصاف أدناه لوضع ثلاثة أقنعة مختلفة. استخدم أداة المنطقة لوضع مربع 0.25 × 0.25 ميكرومتر في منتصف 400 خلية على الأقل.
- استخدم زر المنطقة إلى قناع لإنشاء قناع (قناع 1) من مربعات الاهتمام. إنشاء قناع فارغ جديد (قناع 2) واستخدام أداة بكسل أو أداة المضلع لوضع علامة على منطقة الخلية بأكملها من ما لا يقل عن 25 خلية عشوائية، والتي سيتم استخدامها لحساب تبييض الفلورية. إنشاء قناع فارغ جديد (قناع 3) واستخدام أداة فرشاة كبيرة لوضع علامة على المناطق بين الخلايا، والتي سيتم استخدامها للطرح الخلفية.
- ضمن إحصاءات القناع ونطاق القناع، اختر الكائن للقناع 1 والقناع 2. ثم، تحت الميزات والكثافة،واختيار متوسط كثافة وتصدير البيانات من أقنعة اثنين. للقناع 3، تصدير متوسط كثافة القناع بأكمله.
- حساب التغيير في كثافة الفلورية لكل كائن في قناع 1 باستخدام الصيغ التالية:

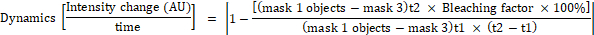
حيث القناع 1 هو 0.25 ميكرومتر × 0.25 ميكرومتر مربع وضعت في مركز الخلية ، قناع 2 يغطي الخلية بأكملها ، قناع 3 هو الخلفية بين الخلايا ، t1 هو متوسط كثافة في نقطة المرة الأولى ، وt2 هو متوسط كثافة في نقطة الوقت الثاني.
5. الكشف عن كثافة كتلة sfGFP مع Cryo-ET
- إعداد العينات وجمع البيانات وإعادة الإعمار
- تنمو رقعة ثقيلة من L. الرئوي التعبير عن بروتين نقطة / Icm الموسومة بالفلور على لوحات CYE-agar-streptomycin لمدة 48 ساعة في 37 درجة مئوية. إعادة تعليق الخلايا في ddH2O إلى OD600 ~ 0.7. أضف 5 ميكرولتر من جزيئات الذهب الغروانية (متتبع BSA، 10 نانومتر) إلى 20 ميكرولتر من تعليق الخلية.
- ماصة 5 μL من خليط الخلية على الطازجة توهج تفريغها شبكة الكربون حريف (R 2/1 على شبكة Cu 200) والسماح للوقوف لمدة 1 دقيقة. Blot مع ورقة تصفية وتجميد في إيثان السائل باستخدام جهاز الضخ الجاذبية يحركها كما هو موضح سابقا14,15.
- صورة العينات المجمدة رطب مع المجهر الإلكتروني انتقال 300 كيلوفولت مجهزة بندقية الانبعاثات الميدانية، ومرشح الطاقة، لوحة المرحلة فولتا، وجهاز الكشف المباشر. اجمع سلسلة الإمالة أحادية المحور عند 26,000 x و42,000 x التكبيرات، مما يؤدي إلى أحجام بكسل على مستوى العينة من 5.4 Å/pixel أو 3.4 Å/pixel، على التوالي.
- استخدم الحزمة الضخمة SerialEM لجمع أكوام الصور عند إزالة التركيز ~ 0 ميكرومتر ، مع مجموعة من زوايا الميل بين -60 درجة و +60 درجة مع زيادة خطوة 3 درجة وجرعة تراكمية تبلغ ~ 60 e-/Å2،16. محاذاة صور الفيلم الجرعة مجزأة في كل كومة باستخدام MotionCor217. تجميع المكدسات المصححة بالانجراف باستخدام TOMOAUTO14.
- محاذاة المكدسات المصححة للانجراف بواسطة المحاذاة المعتمدة على علامة IMOD18. إعادة بناء التوموجرامباستخدام طريقة SIRT19 للتجزئات وتحليل الصورة المباشر وطريقة WBP20.
- تحليل الغلوزموغرام لعينات الاندماج sfGFP
- استخدام حزمة التوموغرافية I3 (0.9.9.3) لتحليل subtomogram14،21،22.
ملاحظة: تتم متابعة المحاذاة بشكل متكرر، حيث يتكون كل تكرار من ثلاثة أجزاء يتم فيها إنشاء المراجع وأقنعة التصنيف، ويتم محاذاة المخططات الفرعية وتصنيفها، ويتم محاذاة متوسطات الفئات مع بعضها البعض. - استخدم 4 × 4 × 4 مخططات فرعية للمحاذاة الأولية. دمج الجسيمات التي تنتمي إلى متوسطات الفئة وتظهر كثافة الإلكترون المقابلة لsfGFP. بعد فرز الجسيمات مع الانصهار sfGFP، استخدم 2 × 2 × 2 بينيد subtomograms لمحاذاة مركزة من منطقة ذات أهمية (مثل مجمع ATPase CYTOPLASMIC Dot/Icm) للحصول على بنية عالية الدقة.
- استخدام حزمة التوموغرافية I3 (0.9.9.3) لتحليل subtomogram14،21،22.
النتائج
تم استخدام إعادة التركيب المتجانس مع التحديد المزدوج في خطوتين لبناء الإدراج المحدد لـ sfGFP. في الخطوة الأولى، تم تنفيذ التزاوج ثلاثي الوالدين، حيث تم تعبئة bRK600 بلازميد الزوجية (بلازميد IncP) من سلالة مساعد الإشريكية القولونية E. MT616 إلى سلالة الإشريكية القولونية المانحة مع ناقل الا...
Discussion
إن توضيح وظائف أنظمة الإفراز البكتيري هو المفتاح لفهم كامل للتفاعلات بين المضيف والعامل الممرض. أنظمة الإفراز هي آلات معقدة يمكنها حقن البروتينات المؤثرات في الخلايا المضيفة ، وفي بعض الحالات تعزز إنشاء مكانة دون خلوية تدعم النسخ البكتيري. الأسلوب أعلاه يوفر أدوات جديدة هامة لدراسة نظام...
Disclosures
وليس لدى صاحبي البلاغ ما يكشفان عنه.
Acknowledgements
تم دعم العاصمة وC.R.R. من قبل المعاهد القومية للصحة (R37AI041699 وR21AI130671). تم دعم D.P. و B.H. و J.L. من قبل المعاهد الوطنية للصحة (R01AI087946 وR01GM107629).
Materials
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold particles | Aurion | 25486 | |
| 100x Plan Apo objective (1.4 NA) | Nikon | ||
| ACES | Sigma-Aldrich | A9758 | |
| Activated charcoal | Sigma-Aldrich | C5510 | |
| Agaroze GPG/LMP, low melt | American bioanalytical | AB00981 | |
| Bacto dehydrated agar | BD | 214010 | |
| CoolSNAP EZ 20 MHz digital monochrome camera | Photometrics | ||
| Gene Frame, 1.7x2.8 cm, 125 µL | Fisher Scientific | AB-0578 | |
| Holey Carbon grid R 2/1 Cu 200 mesh | Quantifoil | Q225-CR1 | |
| Iron(III) nitrate nonahydrate | Sigma-Aldrich | 216828 | |
| K2 Summit camera for cryo-EM | GATAN | ||
| L-Cysteine | Sigma-Aldrich | C7352 | |
| Microscope cover slides 22x22 mm | Fisher Scientific | 12-542B | |
| Microscope cover slides 24x50 mm | Fisher Scientific | 12-545K | |
| Microscope slides 25x75x1 mm | Globe Scientific | 1380 | |
| SlideBook 6.0 | Intelligent Imaging Innovations | ||
| Spectra X light engine | Lumencor | ||
| Taq 2X Master Mix | New England BioLabs | M0270 | |
| Titan Krios | Thermo Fisher Scientific | ||
| Yeast Extract | BD | 212750 |
References
- Franco, I. S., Shuman, H. A., Charpentier, X. The perplexing functions and surprising origins of Legionella pneumophila type IV secretion effectors. Cellular Microbiology. 11, 1435-1443 (2009).
- Burstein, D., et al. Genome-scale identification of Legionella pneumophila effectors using a machine learning approach. PLOS Pathogens. 5, 1000508 (2009).
- Ninio, S., Roy, C. R. Effector proteins translocated by Legionella pneumophila: strength in numbers. Trends in Microbiology. 15, 372-380 (2007).
- Vogel, J. P., Andrews, H. L., Wong, S. K., Isberg, R. R. Conjugative transfer by the virulence system of Legionella pneumophila. Science. 279, 873-876 (1998).
- Isberg, R. R., O'Connor, T. J., Heidtman, M. The Legionella pneumophila replication vacuole: making a cosy niche inside host cells. Nature Reviews Microbiology. 7, 13-24 (2009).
- Roy, C. R., Berger, K. H., Isberg, R. R. Legionella pneumophila DotA protein is required for early phagosome trafficking decisions that occur within min of bacterial uptake. Molecular Microbiology. 28, 663-674 (1998).
- Archer, K. A., Roy, C. R. MyD88-Dependent Responses Involving Toll-Like Receptor 2 Are Important for Protection and Clearance of Legionella pneumophila in a Mouse Model of Legionnaires' Disease. Infection and Immunity. 74, 3325-3333 (2006).
- Nagai, H., Kubori, T. Type IVB Secretion Systems of Legionella and Other Gram-Negative Bacteria. Frontiers in Microbiology. 2, 136 (2011).
- Kubori, T., Nagai, H. The Type IVB secretion system: an enigmatic chimera. Current Opinion in Microbiology. 29, 22-29 (2016).
- Chetrit, D., Hu, B., Christie, P. J., Roy, C. R., Liu, J. A unique cytoplasmic ATPase complex defines the Legionella pneumophila type IV secretion channel. Nature Microbiology. 3, 678-686 (2018).
- Merriam, J. J., Mathur, R., Maxfield-Boumil, R., Isberg, R. R. Analysis of the Legionella pneumophila fliI gene: intracellular growth of a defined mutant defective for flagellum biosynthesis. Infection and Immunity. 65, 2497-2501 (1997).
- Feeley, J. C., et al. Charcoal-yeast extract agar: primary isolation medium for Legionella pneumophila. Journal of Clinical Microbiology. 10, 437-441 (1979).
- Andrews, H. L., Vogel, J. P., Isberg, R. R. Identification of linked Legionella pneumophila genes essential for intracellular growth and evasion of the endocytic pathway. Infection and Immunity. 66, 950-958 (1998).
- Morado, D. R., Hu, B., Liu, J. Using Tomoauto: A Protocol for High-throughput Automated Cryo-electron Tomography. Journal of Visualized Experiments. (107), e53608 (2016).
- Hu, B., Lara-Tejero, M., Kong, Q., Galan, J. E., Liu, J. In Situ Molecular Architecture of the Salmonella Type III Secretion Machine. Cell. 168, 1065-1074 (2017).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152, 36-51 (2005).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14, 331-332 (2017).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Gilbert, P. Iterative methods for the three-dimensional reconstruction of an object from projections. Journal of Theoretical Biology. 36, 105-117 (1972).
- Radermacher, M. Weighted Back-projection Methods. Electron Tomography. , 245-273 (2007).
- Winkler, H., et al. Tomographic subvolume alignment and subvolume classification applied to myosin V and SIV envelope spikes. Journal of Structural Biology. 165, 64-77 (2009).
- Winkler, H., Taylor, K. A. Accurate marker-free alignment with simultaneous geometry determination and reconstruction of tilt series in electron tomography. Ultramicroscopy. 106, 240-254 (2006).
- Prevost, M. S., Waksman, G. X-ray crystal structures of the type IVb secretion system DotB ATPases. Protein Science. 27, 1464-1475 (2018).
- Miklos, G. L., Rubin, G. M. The role of the genome project in determining gene function: insights from model organisms. Cell. 86, 521-529 (1996).
- Reyrat, J. M., Pelicic, V., Gicquel, B., Rappuoli, R. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis. Infection and Immunity. 66, 4011-4017 (1998).
- Yu, J. Single-Molecule Studies in Live Cells. Annual Review of Physical Chemistry. 67, 565-585 (2016).
- Stewart, P. L. Cryo-electron microscopy and cryo-electron tomography of nanoparticles. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 9, (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved