É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Aplicando imagens de células vivas e tomografia crio-elétron para resolver características espaciales do Sistema de Secreção Legionella pneumophila Dot/ICM
Neste Artigo
Resumo
A imagem de células bacterianas é uma abordagem de biologia de sistemas emergentes focada na definição de processos estáticos e dinâmicos que ditam a função de grandes máquinas macromoleculares. Aqui, a integração da imagem quantitativa de células vivas e tomografia crio-elétron é usada para estudar a arquitetura e funções do sistema de secreção legionella tipo IV.
Resumo
O sistema de secreção Dot/ICM da pneumophila Legionella é uma nanomáquina complexa do sistema de secreção tipo IV (T4SS) que localiza no pólo bacteriano e media a entrega de substratos de proteína e DNA às células-alvo, um processo que geralmente requer contato direto entre células. Recentemente, resolvemos a estrutura do aparelho Dot/Icm por tomografia crio-elétron (crio-ET) e mostramos que ele forma um canal de abrangência de envelopes de células que se conecta a um complexo citoplasmático. A aplicação de duas abordagens complementares que preservam a estrutura nativa do espécime, a microscopia fluorescente em células vivas e crio-ET, permite a visualização in situ de proteínas e assimilação da estequiometria e tempo de produção de cada componente da máquina em relação a outras subunidades do Ponto/Icm. Para investigar os requisitos para o posicionamento polar e para caracterizar características dinâmicas associadas à biogênese da máquina T4SS, fundimos uma proteína fluorescente verde de codificação de genes de superpasta para genes Dot/Icm ATPase em suas posições nativas no cromossomo. O método a seguir integra a microscopia quantitativa de fluorescência de células vivas e crio-ET para quantificar a localização polar, a dinâmica e a estrutura dessas proteínas em células bacterianas intactas. A aplicação dessas abordagens para o estudo da Legionella pneumophila T4SS é útil para caracterizar a função do sistema Dot/Icm e pode ser adaptada para estudar uma grande variedade de patógenos bacterianos que utilizam o T4SS ou outros tipos de complexos de secreção bacteriana.
Introdução
Legionella pneumophila (L. pneumophila), o agente etiológico da doença dos Legionários, habita reservatórios de água doce, onde as bactérias se propagam infectando e replicando dentro de protozoários aquáticos de natação livre. L. pneumophila causa surtos de doenças em humanos quando ocorre inalação de bactérias aerossolizadas de fontes de água potável. Nas células infectadas, a subversão das vias hospedeiras permite que a L. pneumophila atrase a maturação endocítica do vacúolo em que reside e promova a biogênese de um compartimento celular que suporta a replicação bacteriana. Este processo é conduzido por um sistema especializado de secreção de tipo bacteriano IVB (T4BSS) conhecido como Ponto/Icm e seu repertório de mais de 300 proteínas "eficazes" que são translocadas no citosol hospedeiro durante a infecção para facilitar a manipulação das funções celulares1,2,3,4,5. Mutantes sem um aparelho funcional Dot/Icm não conseguem entregar os efeitos no citosol hospedeiro, são defeituosos para replicação intracelular e são aviudores em modelos animais da doença6,7.
Muitas espécies bacterianas desenvolveram máquinas multicomponentes extremamente complexas e dinâmicas que são necessárias para processos de infecção. Outros T4BSS como o sistema Dot/Icm também são essenciais para a replicação intracelular de patógenos bacterianos como Coxiella burnetii e Rickettsiella grylli. Embora o T4BSS esteja evolutivamente relacionado aos sistemas iva do tipo prototípico, que mediam a transferência de DNA e podem fornecer um repertório limitado de proteínas eficazes, o sistema Dot/Icm tem quase o dobro de componentes da máquina e fornece uma grande variedade de efeitos. Presumivelmente, essa expansão no número de componentes permitiu que o aparelho Dot/Icm acomodasse e integrasse facilmente novos efeitos8,9.
Recentemente usamos a tomografia crio-elétron (crio-ET) para resolver a estrutura do aparelho Dot/Icm in situ e mostramos que ele forma um canal de abrangência de envelopes de células que se conecta a um complexo citoplasmático. Análises posteriores revelaram que o citosolicatat ATPase DotB associa-se ao sistema Dot/Icm no pólo celular L. pneumophila através de interações com o citosolicatATPase DotO. Descobrimos que o DotB apresenta um movimento citosólico na maioria das células bacterianas, indicando que este ATPase está presente em uma população citossólica dinâmica, mas também se associa aos complexos polares do Dot/Icm. Além disso, DotO forma uma montagem hexamérica de dimers DotO associados com o complexo de membrana interna, e um Hexamer DotB se junta à base deste complexo citoplasmático. A montagem do complexo energético DotB-DotO cria um canal citoplasmático que direciona a translocação de substratos através do T4SS (Figura 1)10.
Apesar desses avanços recentes, pouco se sabe sobre como funciona o sistema Ponto/Icm e como cada proteína se reúne para formar um aparelho ativo8. Descobrir os circuitos regulatórios do Ponto/Icm T4SS é fundamental para compreender os mecanismos moleculares das interações hospedeira-patógena. Portanto, discutimos como usar a microscopia de células vivas e crio-ET para detectar e caracterizar componentes essenciais do sistema L. pneumophila Dot/Icm que são marcados com a super pasta GFP (sfGFP). Utilizando microscopia quantitativa de fluorescência, a localização polar do DotB será definida em um tipo de fundo selvagem ou quando o sistema tipo IV for excluído. A microscopia de lapso de tempo será usada para quantificar diferenças na localização e dinâmica entre os ATPas citosolic do TT/ICM.
A aplicação combinada de duas abordagens complementares, como imagem ao vivo e crio-ET, proporciona uma vantagem em comparação com outros sistemas in vitro. Ambos os métodos são realizados em células intactas e preservam o ambiente natural do T4BSS, minimizando assim a interrupção da estrutura nativa durante a preparação da amostra. Como a superexpressão das proteínas pode prejudicar a estequiometria do aparelho de secreção, as fusões sfGFP são devolvidas através da troca alélica ao cromossomo Legionella para que cada fusão seja codificada em cópia única e a expressão seja conduzida pelo promotor endógeno. A visualização de fusões codificadas cromossomicamente permite quantificação do nível exato de proteína sendo expressa em um ponto de tempo definido. O Cryo-ET também tem muitas vantagens para determinar a estrutura dos sistemas de secreção. A vantagem mais notável é que as amostras crio-ET são compostas de células intactas congeladas que preservam complexos nativos no contexto da arquitetura celular bacteriana. Consequentemente, o crio-ET pode ser preferível às abordagens de purificação bioquímica, que extraem complexos de membrana e podem retirar proteínas periféricas do aparelho central ou modificar a estrutura geral. Além disso, a marcação de uma proteína de interesse com uma proteína volumosa como o sfGFP adiciona uma massa que é detectável pelo crio-ET e pode auxiliar no mapeamento dos diferentes subcomplexos do aparelho Dot/Icm na estrutura obtida pelo crio-ET.
Esta abordagem é uma poderosa ferramenta para descobrir informações estruturais sobre complexos multimoleculares que se reúnem na membrana celular bacteriana. A interpretação das estruturas elucidadas usando essas técnicas ajudará o campo a entender como funcionam os componentes T4BSS, por que tantos componentes são necessários para a função, como os componentes interagem dentro do complexo maior e quais funções esses subconjuntos funcionam.
Protocolo
NOTA: Todos os procedimentos envolvendo o crescimento, manipulação e imagem de L. pneumophila devem ser realizados em um laboratório de segurança biológica nível 2 em conformidade com as diretrizes locais.
1. Inserção de sfGFP no Cromossomo L. pneumophila utilizando a Estratégia de Troca Adélia e Dupla Seleção(Figura 2, Figura 3)
- Clone no vetor de substituição genética pSR47S11 a seguinte seqüência: a 1.000 bp upstream do local de interesse, depois a seqüência sfGFP, em seguida, a 1.000 bp downstream do site de interesse(Figura 2). A seqüência sfGFP deve ser colocada no quadro para as extremidades N-terminus ou C-terminus com um linker que contém quatro a oito aminoácidos. Transforme o vetor resultante em E. coli DH5αλpir. Posteriormente, estria L. pneumophila (o receptor) para colônias únicas no extrato de levedura de carvão (CYE) ágar12 contendo 100 μg/mL de estreptomicina e crescer durante 5 dias a 37 °C(Figura 3).
- Raia L. pneumofa em CYE-ágar-estreptomicina e crescer por 2 dias a 37 °C (patch pesado)10. Raia E. coli DH5α transformada com pRK600 assistente plasmid (ajudante)13 no ágar LB contendo 25 μg/mL de clorofenicol. Streak o E. coli DH5αλpir (doador) em ágar LB contendo 50 μg/mL de kanamicina.
- Realizar o acasalamento triparental: incubar uma colônia do ajudante, uma colônia do doador e o receptor sobrepondo manchas das três cepas em uma placa de ágar CYE sem seleção e incubando por 4-8 h a 37 °C. À medida que os controles negativos incubam uma mistura de tensão helper+recipient e uma mistura de cepa doador+receptora pelos mesmos períodos de tempo.
- Resuspender as reações de acasalamento em 500 μL de ddH2O. Placa 20 μL e 50 μL das reações no ágar CYE contendo 100 μg/mL de estreptomicina e 10 μg/mL de kanamicina e crescer por 5 dias a 37 °C. Streak quatro dos clones resultantes no ágar CYE contendo 100 μg/mL de estreptomicina e crescem por 5 dias a 37 °C.
- Estrias de 16 clones no ágar CYE contendo 5% de sacarose e 100 μg/mL de estreptomicina e cresçam por 5 dias a 37 °C. Em seguida, estria 32 desses clones no ágar CYE contendo 100 μg/mL de estreptomicina e em ágar CYE contendo 100 μg/mL de estreptomicina e 10 μg/mL de kanamicina e crescer por 5 dias a 37 °C.
2. Isolamento de Clones que Integraram sfGFP no Cromossomo L. pneumophila
- Clones de raia que eram sensíveis à kanamicina em placas CYE-ágar-estreptomicina e confirmam a inserção de sfGFP no cromossomo com pcr colônia. Use primers complementares ao gene sfGFP e à região de interesse cromossômica para amplificar a junção de inserção.
- Misture 0,5 μL de cada uma das soluções de primers de 10 μM e uma colônia a um volume final de 12,5 μL e desnaturação por 10 min a 95 °C. Esfrie no gelo por 10 min, adicione 12,5 μL de solução master mix 2x PCR e realize uma análise PCR.
- Cultivar manchas pesadas das colônias isoladas em placas CYE-ágar-estreptomicina por 2 dias a 37 °C. Examine os níveis de expressão e a estabilidade das fusões sfGFP por imunoblotting com um anticorpo anti-GFP.
3. Imagem celular viva de L. pneumofa com componentes de ponto/icm marcados fluorescentemente
- Preparação de almofadas de agarose
- Faça cerca de 30 mL de uma solução de agarose de 1% de baixo derretimento na água. Microondas em um frasco de vidro por cerca de 90 s, girando ocasionalmente, até que a agarose seja completamente dissolvida.
- Coloque duas lâminas de vidro 22 x 22 x 22 x 0,15 mm3 na borda de um escorregador de vidro de 25 x 75 x 1,1 mm3, um em cima do outro. Empilhe mais dois slides de vidro 22 x 22 x 22 x 0,15 mm3 na outra borda.
- Pipeta cerca de 1 mL da agarose derretida no deslizamento central entre as duas lâminas de vidro superiores, em seguida, coloque outro deslizamento de 25 x 75 x 1,1 mm3 em cima da agarose derretida. Tente evitar a formação de bolhas de ar. Esfrie os slides a 4 °C por 15 min.
- Usando um bisturi ou lâmina de barbear, corte suavemente a almofada em quadrados pequenos, ~5 x 5 mm2. Fixar um adesivo de dupla lateral 17 x 28 x 0,25 mm3 quadro em um slide de vidro de 25 x 75 x 1,1 mm3 e coloque várias almofadas no slide.
- Aquisição de imagens
NOTA: As seguintes etapas são descritas para um microscópio que está o controle do SlideBook 6.0 e equipado com iluminadores de estado sólido, câmera monocromática CCD e uma lente objetiva de 100x (abertura numérica de 1,4). Se necessário, use dispositivos alternativos de microscopia com configurações adequadas de hardware e software que podem ser personalizadas de acordo com as configurações do protocolo.- Dissolva um pedaço pesado de L. pneumophila em 1 mL de ddH2O, vórtice e pipeta 2-3 μL da diluição nas almofadas. Coloque um deslizamento de cobertura de 50 x 24 x 0,15 mm3 suavemente sobre a estrutura do adesivo.
- Na janela de captura ajuste o ND para 180. Ajuste o binning para 2×2 e use o canal de 488 nm para expor a amostra entre 500-1.000 ms. Valide a especificidade do sinal de fluorescência por imagem não marcada L. pneumófilia com os mesmos parâmetros(Figura 4).
4. Quantificação da Localização Polar e Dinâmica dos Componentes Dot/Icm
NOTA: As seguintes etapas são projetadas para imagens com 0,129 μm por pixel que foram adquiridas com 2 x 2 binning.
- Quantificação da polaridade para proteínas de fusão sfGFP (Figura 5)
- Ajuste o contraste da imagem para que as bactérias sejam claramente visíveis. Use a ferramenta de região para colocar um retângulo de 0,25 x 1,3 μm2 começando no pólo e estendendo-se até o citoplasma. O retângulo deve permanecer precisamente dentro das fronteiras bacterianas.
- Marque pelo menos 200 bactérias e use o botão Região para Máscara para criar máscaras para as regiões de interesse. Em Estatísticas de máscara e escopo da máscara,escolha objeto. Em seguida, em Características e Intensidade, marque Intensidade média e variância.
- Exportar os dados e calcular os escores de polaridade de cada bactéria como a razão entre a variância à intensidade média.
- Para aplicações de alto throughput use um objetivo de fase e uma configuração de condensador apropriado para adquirir imagens com a fase e canais de 488 nm. Certifique-se de escolher campos de visão onde as bactérias estão totalmente separadas.
- Ajuste o contraste do canal de fase da imagem a um nível onde as bactérias são claramente visíveis. Abra a imagem de canal duplo, inicie a janela Criar máscara de segmento e mude o canal para fase.
- Ajuste um limiar apropriado e remova pequenos objetos com o botão Definir objetos. Em Refine Mask, escolha Remover bordas objetos e, posteriormente, máscaras separadas de bactérias adjacentes umas às outras.
- Calcule os escores de polaridade do sinal no canal 488 para cada célula, como foi descrito anteriormente nas etapas 4.1.2-4.1.3.
- Quantificação da dinâmica para proteínas de fusão sfGFP (Figura 6)
NOTA: Siga as instruções da seção 3 para preparar uma amostra para aquisição de imagens. A dinâmica é definida como mudanças de intensidade ao longo do tempo e as seguintes etapas são projetadas para curtos períodos de imagem (ou seja, vários minutos). Adicione à almofada os suplementos apropriados se forem desejados períodos de imagem mais longos.- Na janela Captura de imagem, marque Timelapse, digite o tempo dos intervalos na caixa de intervalo e digite 2 na caixa # de Pontos de Tempo. Adquira duas imagens sucessivas de L. pneumophila expressando a proteína fluorescente de interesse.
- Ajuste o contraste da imagem até que as células fiquem claramente visíveis. Siga a Figura 6A e as descrições abaixo para colocar três máscaras diferentes. Use a ferramenta de região para colocar um quadrado de 0,25 x 0,25 μm no meio de pelo menos 400 células.
- Use o botão Região para Máscara para criar uma máscara (máscara 1) dos quadrados de interesse. Crie uma nova máscara vazia (máscara 2) e use a ferramenta pixel ou a ferramenta polígono para marcar toda a área celular de pelo menos 25 células aleatórias, que serão usadas para calcular o branqueamento da fluorescência. Crie uma nova máscara vazia (máscara 3) e use a grande ferramenta de pincel para marcar áreas entre as células, que serão usadas para subtração de fundo.
- Em Mask Statistics e Mask Scope, escolha Objeto para máscara 1 e máscara 2. Em seguida, em Características e Intensidade,escolha Intensidade Média e exporte os dados das duas máscaras. Para a máscara 3, exporte a intensidade média de toda a máscara.
- Calcule a mudança na intensidade da fluorescência para cada objeto na máscara 1 usando as seguintes fórmulas:

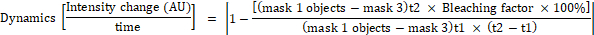
onde a máscara 1 é um 0,25 μm × 0,25 μm quadrado colocado no centro celular, máscara 2 cobre toda a célula, máscara 3 é o fundo entre as células, t1 é a intensidade média no primeiro ponto de tempo, e t2 é a intensidade média no segundo ponto de tempo.
5. Detecção da densidade de massa sfGFP com Crio-ET
- Preparação de amostras, coleta de dados e reconstrução
- Cultivar um pedaço pesado de L. pneumophila expressando uma proteína Dot/Icm fluorescentemente marcada em placas CYE-ágar-estreptomicina por 48 horas a 37 °C. Resuspender as células em ddH2O para OD600 ~0.7. Adicione 5 μL de partículas de ouro coloidais (BSA Tracer, 10 nm) a 20 μL da suspensão celular.
- Pipeta 5 μL da mistura celular na grade de carbono furada recém-descarregada (R 2/1 na malha 200) e deixe ficar por 1 min. Mancha com papel filtro e congelar em etano líquido usando um aparelho de êmbolo acionado pela gravidade como descrito anteriormente14,15.
- Imagem das amostras hidratadas congeladas com um microscópio eletrônico de transmissão de 300 kV equipado com uma arma de emissão de campo, um filtro de energia, placa de fase Volta e um dispositivo de detecção direta. Coletar séries de inclinação de um eixo único em ampliações de 26.000x e 42.000x, que resultam em tamanhos de pixels no nível da amostra de 5,4 Å/pixel ou 3,4 Å/pixel, respectivamente.
- Use o pacote tomográfico SerialEM para coletar pilhas de imagem a ~0 μm dedefocus, com uma gama de ângulos de inclinação entre -60° e +60° com incremento de 3° de passo e dose acumulada de ~60 e-/Å2,16. Alinhar imagens de filme fracionadas em cada pilha usando MotionCor217. Montar pilhas corrigidas à deriva usando TOMOAUTO14.
- Alinhar pilhas corrigidas à deriva pelo alinhamento dependente do marcador IMOD18. Reconstruir tomogramas com o método SIRT19 para segmentações e análise direta de imagem e o método WBP20.
- Análise de subtomograma de amostras de fusões sfGFP
- Use o pacote tomográfico I3 (0.9.9.3) para análise de subtomograma14,21,22.
NOTA: O alinhamento prossegue iterativamente, com cada iteração composta por três partes nas quais são geradas referências e máscaras de classificação, subtomogramas alinhados e classificados, e as médias de classe alinhadas entre si. - Use 4 x 4 x 4 subtomogramas binados para um alinhamento inicial. Mesclar partículas que pertencem às médias de classe e mostram densidade de elétrons correspondente ao sfGFP. Depois de classificar partículas com fusões sfGFP, use subtomogramas binados 2 x 2 x 2 para um alinhamento focalizado de uma região de interesse (como o complexo ATPase citoplasmático Dot/Icm) para obter uma estrutura de alta resolução.
- Use o pacote tomográfico I3 (0.9.9.3) para análise de subtomograma14,21,22.
Resultados
A recombinação homólogo com dupla seleção em duas etapas foi utilizada para construir a inserção definida do sfGFP. Na primeira etapa, foi realizado o acasalamento triparental, onde o plasmídeo conjugado pRK600 (um plasmid IncP) da cepa auxiliar E. coli MT616 foi mobilizado para o doador E. coli cepa com o vetor suicida pSR47S contendo o gene sfGFP ladeado pelas duas regiões homólogas, a origem da transferência oriT e o bacillus subseleção genética Em seguida, o sistema d...
Discussão
Elucidar as funções dos sistemas de secreção bacteriana é a chave para uma compreensão completa das interações hospedeira-patógena. Os sistemas de secreção são máquinas complexas que podem injetar proteínas de efeitos em células hospedeiras, e em alguns casos promovem o estabelecimento de um nicho subcelular que suporta a replicação bacteriana. O método acima fornece novas ferramentas importantes para estudar o sistema de secreção Dot/ICM do patógeno respiratório Legionella pneumophila, dan...
Divulgações
Os autores não têm nada para revelar.
Agradecimentos
D.C. e C.R.R. foram apoiados pelo NIH (R37AI041699 e R21AI130671). D.P., B.H., e J.L foram apoiados pelos Institutos Nacionais de Saúde (R01AI087946 e R01GM107629).
Materiais
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold particles | Aurion | 25486 | |
| 100x Plan Apo objective (1.4 NA) | Nikon | ||
| ACES | Sigma-Aldrich | A9758 | |
| Activated charcoal | Sigma-Aldrich | C5510 | |
| Agaroze GPG/LMP, low melt | American bioanalytical | AB00981 | |
| Bacto dehydrated agar | BD | 214010 | |
| CoolSNAP EZ 20 MHz digital monochrome camera | Photometrics | ||
| Gene Frame, 1.7x2.8 cm, 125 µL | Fisher Scientific | AB-0578 | |
| Holey Carbon grid R 2/1 Cu 200 mesh | Quantifoil | Q225-CR1 | |
| Iron(III) nitrate nonahydrate | Sigma-Aldrich | 216828 | |
| K2 Summit camera for cryo-EM | GATAN | ||
| L-Cysteine | Sigma-Aldrich | C7352 | |
| Microscope cover slides 22x22 mm | Fisher Scientific | 12-542B | |
| Microscope cover slides 24x50 mm | Fisher Scientific | 12-545K | |
| Microscope slides 25x75x1 mm | Globe Scientific | 1380 | |
| SlideBook 6.0 | Intelligent Imaging Innovations | ||
| Spectra X light engine | Lumencor | ||
| Taq 2X Master Mix | New England BioLabs | M0270 | |
| Titan Krios | Thermo Fisher Scientific | ||
| Yeast Extract | BD | 212750 |
Referências
- Franco, I. S., Shuman, H. A., Charpentier, X. The perplexing functions and surprising origins of Legionella pneumophila type IV secretion effectors. Cellular Microbiology. 11, 1435-1443 (2009).
- Burstein, D., et al. Genome-scale identification of Legionella pneumophila effectors using a machine learning approach. PLOS Pathogens. 5, 1000508 (2009).
- Ninio, S., Roy, C. R. Effector proteins translocated by Legionella pneumophila: strength in numbers. Trends in Microbiology. 15, 372-380 (2007).
- Vogel, J. P., Andrews, H. L., Wong, S. K., Isberg, R. R. Conjugative transfer by the virulence system of Legionella pneumophila. Science. 279, 873-876 (1998).
- Isberg, R. R., O'Connor, T. J., Heidtman, M. The Legionella pneumophila replication vacuole: making a cosy niche inside host cells. Nature Reviews Microbiology. 7, 13-24 (2009).
- Roy, C. R., Berger, K. H., Isberg, R. R. Legionella pneumophila DotA protein is required for early phagosome trafficking decisions that occur within min of bacterial uptake. Molecular Microbiology. 28, 663-674 (1998).
- Archer, K. A., Roy, C. R. MyD88-Dependent Responses Involving Toll-Like Receptor 2 Are Important for Protection and Clearance of Legionella pneumophila in a Mouse Model of Legionnaires' Disease. Infection and Immunity. 74, 3325-3333 (2006).
- Nagai, H., Kubori, T. Type IVB Secretion Systems of Legionella and Other Gram-Negative Bacteria. Frontiers in Microbiology. 2, 136 (2011).
- Kubori, T., Nagai, H. The Type IVB secretion system: an enigmatic chimera. Current Opinion in Microbiology. 29, 22-29 (2016).
- Chetrit, D., Hu, B., Christie, P. J., Roy, C. R., Liu, J. A unique cytoplasmic ATPase complex defines the Legionella pneumophila type IV secretion channel. Nature Microbiology. 3, 678-686 (2018).
- Merriam, J. J., Mathur, R., Maxfield-Boumil, R., Isberg, R. R. Analysis of the Legionella pneumophila fliI gene: intracellular growth of a defined mutant defective for flagellum biosynthesis. Infection and Immunity. 65, 2497-2501 (1997).
- Feeley, J. C., et al. Charcoal-yeast extract agar: primary isolation medium for Legionella pneumophila. Journal of Clinical Microbiology. 10, 437-441 (1979).
- Andrews, H. L., Vogel, J. P., Isberg, R. R. Identification of linked Legionella pneumophila genes essential for intracellular growth and evasion of the endocytic pathway. Infection and Immunity. 66, 950-958 (1998).
- Morado, D. R., Hu, B., Liu, J. Using Tomoauto: A Protocol for High-throughput Automated Cryo-electron Tomography. Journal of Visualized Experiments. (107), e53608 (2016).
- Hu, B., Lara-Tejero, M., Kong, Q., Galan, J. E., Liu, J. In Situ Molecular Architecture of the Salmonella Type III Secretion Machine. Cell. 168, 1065-1074 (2017).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152, 36-51 (2005).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14, 331-332 (2017).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Gilbert, P. Iterative methods for the three-dimensional reconstruction of an object from projections. Journal of Theoretical Biology. 36, 105-117 (1972).
- Radermacher, M. Weighted Back-projection Methods. Electron Tomography. , 245-273 (2007).
- Winkler, H., et al. Tomographic subvolume alignment and subvolume classification applied to myosin V and SIV envelope spikes. Journal of Structural Biology. 165, 64-77 (2009).
- Winkler, H., Taylor, K. A. Accurate marker-free alignment with simultaneous geometry determination and reconstruction of tilt series in electron tomography. Ultramicroscopy. 106, 240-254 (2006).
- Prevost, M. S., Waksman, G. X-ray crystal structures of the type IVb secretion system DotB ATPases. Protein Science. 27, 1464-1475 (2018).
- Miklos, G. L., Rubin, G. M. The role of the genome project in determining gene function: insights from model organisms. Cell. 86, 521-529 (1996).
- Reyrat, J. M., Pelicic, V., Gicquel, B., Rappuoli, R. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis. Infection and Immunity. 66, 4011-4017 (1998).
- Yu, J. Single-Molecule Studies in Live Cells. Annual Review of Physical Chemistry. 67, 565-585 (2016).
- Stewart, P. L. Cryo-electron microscopy and cryo-electron tomography of nanoparticles. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 9, (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados