È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Applicazione di Live Cell Imaging e tomografia Cryo-Electron a risolvere le caratteristiche spatiotemporali del sistema di secrezione Legionella pneumophila Dot/Icm
In questo articolo
Riepilogo
L'imaging delle cellule batteriche è un approccio biologico dei sistemi emergenti incentrato sulla definizione di processi statici e dinamici che dettano la funzione di grandi macchine macromolecolari. Qui, l'integrazione dell'imaging quantitativo delle cellule vive e della tomografia crioelettronica viene utilizzata per studiare l'architettura e le funzioni del sistema di secrezione Legionella pneumophila tipo IV.
Abstract
Il sistema di secrezione Dot/Icm della Legionella pneumophila è una nanomacchina complessa di secrezione IV (T4SS) che localizza al polo batterico e media la somministrazione di substrati proteici e del DNA alle cellule bersaglio, un processo che generalmente richiede un contatto diretto tra cellule. Recentemente abbiamo risolto la struttura dell'apparato Punto/Icm mediante tomografia crioelettronica (cryo-ET) e dimostrato che forma un canale di involucro cellulare che si collega a un complesso citoplasmatico. L'applicazione di due approcci complementari che preservano la struttura nativa dell'esemplare, la microscopia fluorescente nelle cellule viventi e il crio-ET, consente la visualizzazione in situ delle proteine e l'assimilazione della stoichiometria e la tempistica della produzione di ciascuna componente macchina rispetto ad altre sottounità punto/icm. Per studiare i requisiti per il posizionamento polare e caratterizzare le caratteristiche dinamiche associate alla biogenesi della macchina T4SS, abbiamo fuso una proteina fluorescente verde di codifica genica ai geni AtPase di Dot/Icm nelle loro posizioni native sul cromosoma. Il seguente metodo integra la microscopia quantitativa a fluorescenza delle cellule viventi e il crio-ET per quantificare la localizzazione, la dinamica e la struttura polari di queste proteine nelle cellule batteriche intatte. L'applicazione di questi approcci per lo studio della Legionella pneumophila T4SS è utile per caratterizzare la funzione del sistema Dot/Icm e può essere adattata per studiare un'ampia varietà di patogeni batterici che utilizzano il T4SS o altri tipi di complessidi di secrezione batterica.
Introduzione
La legionella pneumophila (L. pneumophila), l'agente eziologico della malattia dei Legionari, abita serbatoi d'acqua dolce, dove i batteri si propagano infettando e replicandoalli protozoi acquatici di nuoto libero. L. pneumophila provoca focolai di malattia nell'uomo quando si verifica l'inalazione di batteri aerosolizzati da fonti di acqua potabile. Nelle cellule infette, la sovversione delle vie ospiti consente a L. pneumophila di ritardare la maturazione endocitica del vacuolo in cui risiede e di promuovere la biogenesi di un compartimento cellulare che supporta la replicazione batterica. Questo processo è guidato da un sistema di secrezione iVB di tipo batterico specializzato (T4BSS) noto come Punto/Icm e dal suo repertorio di oltre 300 proteine "effettore" che vengono traslocate nel citosol ospite durante l'infezione per facilitare la manipolazione delle funzioni cellulari1,2,3,4. I mutanti privi di un apparato funzionale Dot/Icm non riescono a fornire gli effettisori nel citosol ospite, sono difettosi per la replicazione intracellulare e sono avirulenti nei modelli animali della malattia6,7.
Molte specie batteriche hanno sviluppato macchine multicomponente estremamente complesse e dinamiche che sono necessarie per i processi di infezione. Altri T4BSS come il sistema Dot/Icm sono essenziali anche per la replicazione intracellulare di patogeni batterici come Coxiella burnetii e Rickettsiella grylli. Sebbene i T4BSS siano evolutivamente correlati a sistemi prototipici di IVA di tipo, che mediano il trasferimento del DNA e possono fornire un repertorio limitato di proteine efvigoreri, il sistema Dot/Icm ha quasi il doppio dei componenti della macchina e offre un'ampia varietà di eseguzioni. Presumibilmente, questa espansione del numero di componenti ha permesso all'apparato Punto/Icm di ospitare e integrare facilmente nuovi effaranti8,9.
Recentemente abbiamo usato la tomografia crioelettronica (crio-ET) per risolvere la struttura dell'apparato Punto/Icm in situ e abbiamo dimostrato che forma un canale di involucro cellulare che si collega a un complesso citoplastico. Ulteriori analisi hanno rivelato che il citosolico ATPase DotB si associa al sistema Dot/Icm al polo cellulare L. pneumophila attraverso interazioni con il citosolico ATPase DotO. Abbiamo scoperto che DotB mostra un movimento citosolico nella maggior parte delle cellule batteriche, indicando che questo ATPase è presente in una popolazione citosolica dinamica, ma si associa anche ai complessi polari Dot/Icm. Inoltre, DotO forma un assemblaggio hexameric di dimeri DotO associati al complesso della membrana interna, e un esamemerDot si unisce alla base di questo complesso citoplasmatico. L'assemblaggio del complesso energetico DotB-DotO crea un canale citoplasmico che dirige la traslocazione dei substrati attraverso il T4SS (Figura 1)10.
Nonostante questi recenti progressi, si sa poco su come funziona il sistema Dot/Icm e su come ogni proteina si assembla per formare un apparato attivo8. Scoprire i circuiti regolatori del Dot/Icm T4SS è fondamentale per comprendere i meccanismi molecolari delle interazioni host-patogen. Pertanto, discutiamo come utilizzare la microscopia a cellule vive e il crio-ET per rilevare e caratterizzare i componenti essenziali del sistema L. pneumophila Dot/Icm che sono contrassegnati con GFP super-folder (sfGFP). Utilizzando la microscopia quantitativa a fluorescenza, la localizzazione polare di DotB sarà definita in uno sfondo di tipo selvaggio o quando il sistema di tipo IV viene eliminato. La microscopia time-lapse sarà utilizzata per quantificare le differenze nella localizzazione e nella dinamica tra l'ATPases citosolico Dot/Icm.
L'applicazione combinata di due approcci complementari come l'imaging dal vivo e il crio-ET offre un vantaggio rispetto ad altri sistemi in vitro. Entrambi i metodi vengono eseguiti in celle intatte e preservano l'ambiente naturale del T4BSS, riducendo così al minimo l'interruzione della struttura nativa durante la preparazione del campione. Poiché la sovraespressione delle proteine può compromettere la stoichiometria dell'apparato di secrezione, le fusioni sfGFP vengono restituite attraverso lo scambio allelico al cromosoma Legionella in modo che ogni fusione sia codificata in una singola copia e l'espressione sia guidata dal promotore endogeno. La visualizzazione di fusioni cromosomiche con codifica consente la quantificazione dell'esatto livello di proteina espresso in un momento definito. Cryo-ET ha anche molti vantaggi per determinare la struttura dei sistemi di secrezione. Il vantaggio più notevole è che i campioni di crio-ET sono costituiti da cellule intatte congelate che conservano complessi nativi nel contesto dell'architettura delle cellule batteriche. Di conseguenza, il crio-ET può essere preferibile agli approcci di purificazione biochimica, che estraggono complessi di membrana e possono privare le proteine periferiche dall'apparato centrale o modificare la struttura complessiva. Inoltre, etichettare una proteina di interesse con una proteina ingombrante come sfGFP aggiunge una massa rilevabile dal crio-ET e può aiutare a mappare i diversi sottocomplessi dell'apparato Punto/Icm sulla struttura ottenuta da cryo-ET.
Questo approccio è un potente strumento per scoprire informazioni strutturali sui complessi multimolecolari che si assemblano nella membrana cellulare batterica. L'interpretazione delle strutture chiarite utilizzando queste tecniche aiuterà il campo a capire come funzionano i componenti T4BSS, perché così tanti componenti sono necessari per la funzione, come i componenti interagiscono all'interno del complesso maggiore e quali funzioni questi i sottoassiemi vengono eseguiti.
Protocollo
NOTA: Tutte le procedure che comportano la crescita, la manipolazione e l'imaging di L. pneumophila devono essere eseguite in un laboratorio di livello di sicurezza biologica 2 in conformità con le linee guida locali.
1. Inserimento di sfGFP in L. pneumophila Cromosoma utilizzando Lo scambio allelico e doppia strategia di selezione (Figura 2, Figura 3)
- Clone nel vettore di sostituzione genica pSR47S11 la seguente sequenza: l'a monte di 1.000 bp del sito di interesse, quindi la sequenza sfGFP, quindi i 1.000 bp a valle del sito di interesse (Figura 2). La sequenza sfGFP deve essere inserita nel frame del capon o del capoC termina con un linker che contiene da quattro a otto aminoacidi. Trasformare il vettore risultante in E. coli DH5- pir. Più tardi, striscia L. pneumophila (il destinatario) per singole colonie su estratto di carbone-lievito (CYE) agar12 contenente 100 g/mL streptomicina e crescere per 5 giorni a 37 gradi centigradi(Figura 3).
- Streak L. pneumophila su CYE-agar-streptomycin e crescere per 2 giorni a 37 gradi centigradi (patch pesante)10. Streak E. coli DH5 è stata trasformata con pRK600 helper plasmid (helper)13 sull'agar LB contenente 25 g/mL di clorammidenico. Streak l'E. coli DH5-pir (donatore) sull'agar LB contenente 50 g/mL di kanamicine.
- Eseguire l'accoppiamento triparentale: incubare una colonia dell'aiutante, una colonia del donatore e il destinatario sovrapponendo le patch dei tre ceppi su una piastra di agar CYE senza selezione e incubare per 4-8 h a 37 gradi centigradi. Come controlli negativi incubare un aiutante- miscela di deformazione ricevente e un mix di deformazione donatore-ricevente per gli stessi periodi di tempo.
- Risospendere le reazioni di accoppiamento in 500 gradi l di ddH2O. Piastra 20 e 50 l delle reazioni sull'agar CYE contenente 100 streptomici e 10g/mL di kanamycin e crescere per 5 giorni a 37 . Streak quattro dei cloni risultanti sull'agar CYE contenenti 100 g/mL di streptomicina e crescono per 5 giorni a 37 gradi centigradi.
- Streak 16 cloni su agar CYE contenente 5% di saccarosio e 100 g/mL di streptomicina e crescono per 5 giorni a 37 gradi centigradi. Poi, striscia 32 di questi cloni su agar CYE contenente 100 g/mL streptomicina e su agar CYE contenente 100 streptomici e 10 g /mL di kanamycin e crescere per 5 giorni a 37 .
2. Isolamento dei cloni integrati sfGFP nel cromosoma L. pneumophila
- Cloni Streak sensibili alla kanamicicina su piastre CYE-agar-streptomicine e confermano l'inserimento di sfGFP nel cromosoma con colonia PCR. Utilizzare primer complementari al gene sfGFP e alla regione cromosomica di interesse per amplificare la giunzione di inserimento.
- Mescolare 0,5 l l di ciascuna delle soluzioni da 10 m e una colonia ad un volume finale di 12,5 gradi centigradi e denaturare per 10 min a 95 gradi centigradi. Raffreddare sul ghiaccio per 10 min, aggiungere 12,5 litri di 2x soluzione di miscela master PCR ed eseguire un'analisi PCR.
- Coltiva pesanti chiazze delle colonie isolate su piastre CYE-agar-streptomicina per 2 giorni a 37 gradi centigradi. Esaminare i livelli di espressione e la stabilità delle fusioni sfGFP mediante immunoblotting con un anticorpo anti-GFP.
3. Live Cell Imaging di L. pneumophila con fluorescently tagged Dot/Icm Componenti
- Preparazione di pastiglie di agarose
- Fare circa 30 mL di una soluzione di agarose a bassa fusione 1% in acqua. Microonde in una fiaschetta di vetro per circa 90 s, vorticoso di tanto in tanto, fino a quando l'agarose è completamente sciolto.
- Posizionare due vetrini divetro 22 x 22 x 0,15 mm sul bordo di uno scivolo divetro 3 25 x 75 x 1,1 mm, uno sopra l'altro. Impilare altri due vetrini invetro 22 x 22 x 0,15 mm sull'altro bordo.
- Pipette circa 1 mL di agarose fuso nella diapositiva centrale tra i due vetrini superiori, quindi posizionare un altro 25 x 75 x 1,1 mm3 scivolo sulla parte superiore dell'agarose fuso. Cercate di evitare la formazione di bolle d'aria. Raffreddare i vetrini a 4 gradi centigradi per 15 min.
- Utilizzando un bisturi o lama di rasoio, tagliare delicatamente il pad in piccoli quadrati, 5 x 5 mm2. Fissare un adesivo a doppio lato 17 x 28 x 0,25 mm3 fotogramma su un 25 x 75 x 1,1 mm3 vetrini di vetro e posizionare diversi pad sul vetrino.
- Acquisizione di immagini
NOTA: i seguenti passaggi sono descritti per un microscopio che è sotto il controllo di SlideBook 6.0 e dotato di illuminatori a stato solido, fotocamera monocromatica CCD e un obiettivo 100x lente (1.4 apertura numerica). Se necessario, utilizzare dispositivi di microscopia alternativi con configurazioni hardware e software appropriate che possono essere personalizzate in base alle impostazioni del protocollo.- Sciogliere un pezzo pesante di L. pneumophila in 1 mL di ddH2O, vortice e pipet 2–3 della diluizione sulle pastiglie. Posizionare un coperchio 50 x 24 x 0,15 mm3 delicatamente sul telaio adesivo.
- Nella finestra di cattura regolare l'ND a 180. Regolare il binning su 2/2 e utilizzare il canale 488 nm per esporre il campione tra 500-1.000 ms. Convalidare la specificità del segnale di fluorescenza mediante l'imaging senza tag L. pneumophila con gli stessi parametri (Figura 4).
4. Quantificazione della localizzazione polare e della dinamica dei componenti Dot/Icm
NOTA: i passaggi seguenti sono progettati per le immagini con 0,129 m per pixel che sono state acquisite con la binning 2 x 2.
- Quantificazione della polarità per le proteine di fusione sfGFP (Figura 5)
- Regolare il contrasto dell'immagine in modo che i batteri siano chiaramente visibili. Utilizzare lo strumento regione per posizionare un rettangolo di 0,25 x 1,3m2 partendo dal palo e estendendosi nel citoplasma. Il rettangolo deve rimanere preciso all'interno dei confini batterici.
- Contrassegnate almeno 200 batteri e utilizzate il pulsante Regione a maschera (Region to Mask) per creare maschere per le regioni di interesse. In Statistiche maschera e Ambito mascherascegliere Oggetto. Quindi, in Funzioni e intensità, contrassegnare Intensità media e Varianza.
- Esportare i dati e calcolare i punteggi di polarità di ogni batterio come rapporto tra la varianza e l'intensità media.
- Per le applicazioni ad alta velocità effettiva, le applicazioni con velocità elevata utilizzano un obiettivo di fase e un'impostazione del condensatore appropriata per acquisire immagini con la fase e canali di 488 nm. Assicurati di scegliere campi visivi in cui i batteri sono completamente separati.
- Regolare il contrasto del canale di fase dell'immagine a un livello in cui i batteri sono chiaramente visibili. Aprire l'immagine del doppio canale, aprire la finestra Crea maschera segmento e modificare il canale in fase.
- Regolare una soglia appropriata e rimuovere piccoli oggetti con il pulsante Definisci oggetti. In Affina maschera scegliere Rimuovi oggetti bordi e successivamente maschere separate di batteri adiacenti l'uno all'altro.
- Calcolare i punteggi di polarità del segnale nel canale 488 per ogni cella come descritto in precedenza nei passaggi 4.1.2–4.1.3.
- Quantificazione delle dinamiche per le proteine di fusione sfGFP (Figura 6)
NOTA: seguire le istruzioni nella sezione 3 per preparare un esempio per l'acquisizione di immagini. Le dinamiche sono definite come cambiamenti di intensità nel tempo e i seguenti passaggi sono progettati per brevi periodi di imaging (ad esempio, diversi minuti). Aggiungere al pad gli integratori appropriati se si desiderano periodi di imaging più lunghi.- Nella finestra Acquisizione immagine, selezionare Timelapse, immettere il tempo degli intervalli nella casella intervallo e immettere 2 nella casella Numero di punti temporali. Acquisire due immagini successive di L. pneumophila che esprime la proteina fluorescente di interesse.
- Regolare il contrasto dell'immagine fino a quando le celle non sono chiaramente visibili. Seguire la figura 6A e le descrizioni riportate di seguito per posizionare tre maschere diverse. Utilizzare lo strumento regione per posizionare un quadrato di 0,25 x 0,25 m al centro di almeno 400 celle.
- Usate il pulsante Regione a maschera per creare una maschera (maschera 1) dei quadrati di interesse. Creare una nuova maschera vuota (maschera 2) e utilizzare lo strumento pixel o lo strumento poligono per contrassegnare l'intera area della cella di almeno 25 celle casuali, che verrà utilizzato per calcolare lo sbiancamento a fluorescenza. Creare una nuova maschera vuota (maschera 3) e utilizzare lo strumento pennello di grandi dimensioni per contrassegnare le aree tra le celle, che verranno utilizzate per la sottrazione di sfondo.
- In Statistiche maschera e Ambito maschera, scegliere Oggetto per maschera1 e maschera 2. Quindi, in Funzioni e intensità, scegliere Intensità media ed esportare i dati delle due maschere. Per la maschera 3, esportare l'intensità media dell'intera maschera.
- Calcolare la variazione dell'intensità di fluorescenza per ogni oggetto nella maschera 1 utilizzando le seguenti formule:

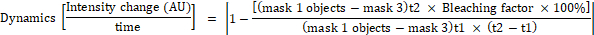
dove la maschera 1 è un quadrato di 0,25 m e 0,25 m posto al centro della cella, la maschera 2 copre l'intera cella, la maschera 3 è lo sfondo tra le celle, t1 è l'intensità media nel primo punto temporale e t2 è l'intensità media nel secondo punto temporale.
5. Rilevamento della densità di massa sfGFP con Cryo-ET
- Preparazione, raccolta e ricostruzione dei campioni
- Coltiva un pezzo pesante di L. pneumophila esprimendo una proteina Dot/Icm fluorescente su piastre CYE-agar-streptomicina per 48 ore a 37 gradi centigradi. Risospendere le celle in ddH2O a600 0,7. Aggiungere 5 -L di particelle d'oro colloidali (BSA Tracer, 10 nm) a 20 -L della sospensione cellulare.
- Pipette 5 - L l della miscela cellulare sulla griglia di carbonio holey appena scaricato dal bagliore (R 2/1 su maglia Cu 200) e lasciare riposare per 1 min. Blot con carta da filtro e congelare in etano liquido utilizzando un apparato di stantuffo a gravità come descritto in precedenza14,15.
- Immagina i campioni congelati con un microscopio elettronico a trasmissione da 300 kV dotato di una pistola ad emissione da campo, un filtro energetico, una piastra di fase Volta e un dispositivo di rilevamento diretto. Raccogliere serie di inclinazione ad asse singolo a 26.000x e 42.000x ingrandimento, che si traducono in dimensioni dei pixel al livello del campione di 5,4 pixel o 3,4 x/pixel, rispettivamente.
- Utilizzare il pacchetto tomografico SerialEM per raccogliere pile di immagini a 0 m di defocus, con una gamma di angoli di inclinazione compresi tra -60 e 60 gradi con incremento di 3 gradini e una dose cumulativa di 60 e-/ 2,16. Allineare le immagini filmate frazionate di dose in ogni pila utilizzando MotionCor217. Assemblare pile corrette dalla deriva utilizzando TOMOAUTO14.
- Allineare le pile corrette dalla deriva mediante l'allineamento dipendente dal marcatore IMOD18. Ricostruire i tomogrammi con il metodo SIRT19 per le segmentazioni e l'analisi diretta delle immagini e il metodo WBP20.
- Analisi del subtomogramma dei campioni di fusioni sfGFP
- Utilizzare il pacchetto tomografico I3 (0.9.9.3) per l'analisi del sottotomogramma14,21,22.
NOTA: l'allineamento procede in modo iterativo, con ogni iterazione costituita da tre parti in cui vengono generati riferimenti e maschere di classificazione, i subtogrammi allineati e classificati e le medie di classe sono allineate tra loro. - Utilizzare 4 x 4 x 4 sottotomogrammi collocati per un tracciato iniziale. Unire le particelle che appartengono alle medie di classe e mostrano la densità degli elettroni corrispondente a sfGFP. Dopo aver smistato particelle con fusioni sfGFP, utilizzare 2 x 2 x 2 sottotomogrammi collocati per un allineamento mirato di una regione di interesse (come il complesso ATPase citoplasmico Dot/Icm) per ottenere una struttura ad alta risoluzione.
- Utilizzare il pacchetto tomografico I3 (0.9.9.3) per l'analisi del sottotomogramma14,21,22.
Risultati
La ricombinazione omologa con doppia selezione in due fasi è stata utilizzata per costruire l'inserimento definito di sfGFP. Nella prima fase, L'accoppiamento triparentale è stato eseguito, dove il plasmide coniugale pRK600 (un plasmide IncP) del ceppo di aiuto E. coli MT616 è stato mobilitato al ceppo E. coli del donatore con il vettore suicida pSR47S contenente il gene sfGFP affiancato dalle due regioni omologhe, l'origine dell'oriT di trasferimento e il gene bacillus subtilselection
Discussione
Chiarire le funzioni dei sistemi di secrezione batterica è fondamentale per una comprensione completa delle interazioni ospite-patogeno. I sistemi di secrezione sono macchine complesse che possono iniettare proteine efricchenti nelle cellule ospiti e in alcuni casi promuovono la creazione di una nicchia subcellulare che supporta la replicazione batterica. Il metodo di cui sopra fornisce importanti nuovi strumenti per studiare il sistema di secrezione punto/Icm del patogeno batterico respiratorio Legionella pneumophi...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
D.C. e C.R.R. sono stati supportati dai NIH (R37AI041699 e R21AI130671). D.P., B.H.e J.L sono stati supportati dai National Institutes of Health (R01AI087946 e R01GM107629).
Materiali
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold particles | Aurion | 25486 | |
| 100x Plan Apo objective (1.4 NA) | Nikon | ||
| ACES | Sigma-Aldrich | A9758 | |
| Activated charcoal | Sigma-Aldrich | C5510 | |
| Agaroze GPG/LMP, low melt | American bioanalytical | AB00981 | |
| Bacto dehydrated agar | BD | 214010 | |
| CoolSNAP EZ 20 MHz digital monochrome camera | Photometrics | ||
| Gene Frame, 1.7x2.8 cm, 125 µL | Fisher Scientific | AB-0578 | |
| Holey Carbon grid R 2/1 Cu 200 mesh | Quantifoil | Q225-CR1 | |
| Iron(III) nitrate nonahydrate | Sigma-Aldrich | 216828 | |
| K2 Summit camera for cryo-EM | GATAN | ||
| L-Cysteine | Sigma-Aldrich | C7352 | |
| Microscope cover slides 22x22 mm | Fisher Scientific | 12-542B | |
| Microscope cover slides 24x50 mm | Fisher Scientific | 12-545K | |
| Microscope slides 25x75x1 mm | Globe Scientific | 1380 | |
| SlideBook 6.0 | Intelligent Imaging Innovations | ||
| Spectra X light engine | Lumencor | ||
| Taq 2X Master Mix | New England BioLabs | M0270 | |
| Titan Krios | Thermo Fisher Scientific | ||
| Yeast Extract | BD | 212750 |
Riferimenti
- Franco, I. S., Shuman, H. A., Charpentier, X. The perplexing functions and surprising origins of Legionella pneumophila type IV secretion effectors. Cellular Microbiology. 11, 1435-1443 (2009).
- Burstein, D., et al. Genome-scale identification of Legionella pneumophila effectors using a machine learning approach. PLOS Pathogens. 5, 1000508 (2009).
- Ninio, S., Roy, C. R. Effector proteins translocated by Legionella pneumophila: strength in numbers. Trends in Microbiology. 15, 372-380 (2007).
- Vogel, J. P., Andrews, H. L., Wong, S. K., Isberg, R. R. Conjugative transfer by the virulence system of Legionella pneumophila. Science. 279, 873-876 (1998).
- Isberg, R. R., O'Connor, T. J., Heidtman, M. The Legionella pneumophila replication vacuole: making a cosy niche inside host cells. Nature Reviews Microbiology. 7, 13-24 (2009).
- Roy, C. R., Berger, K. H., Isberg, R. R. Legionella pneumophila DotA protein is required for early phagosome trafficking decisions that occur within min of bacterial uptake. Molecular Microbiology. 28, 663-674 (1998).
- Archer, K. A., Roy, C. R. MyD88-Dependent Responses Involving Toll-Like Receptor 2 Are Important for Protection and Clearance of Legionella pneumophila in a Mouse Model of Legionnaires' Disease. Infection and Immunity. 74, 3325-3333 (2006).
- Nagai, H., Kubori, T. Type IVB Secretion Systems of Legionella and Other Gram-Negative Bacteria. Frontiers in Microbiology. 2, 136 (2011).
- Kubori, T., Nagai, H. The Type IVB secretion system: an enigmatic chimera. Current Opinion in Microbiology. 29, 22-29 (2016).
- Chetrit, D., Hu, B., Christie, P. J., Roy, C. R., Liu, J. A unique cytoplasmic ATPase complex defines the Legionella pneumophila type IV secretion channel. Nature Microbiology. 3, 678-686 (2018).
- Merriam, J. J., Mathur, R., Maxfield-Boumil, R., Isberg, R. R. Analysis of the Legionella pneumophila fliI gene: intracellular growth of a defined mutant defective for flagellum biosynthesis. Infection and Immunity. 65, 2497-2501 (1997).
- Feeley, J. C., et al. Charcoal-yeast extract agar: primary isolation medium for Legionella pneumophila. Journal of Clinical Microbiology. 10, 437-441 (1979).
- Andrews, H. L., Vogel, J. P., Isberg, R. R. Identification of linked Legionella pneumophila genes essential for intracellular growth and evasion of the endocytic pathway. Infection and Immunity. 66, 950-958 (1998).
- Morado, D. R., Hu, B., Liu, J. Using Tomoauto: A Protocol for High-throughput Automated Cryo-electron Tomography. Journal of Visualized Experiments. (107), e53608 (2016).
- Hu, B., Lara-Tejero, M., Kong, Q., Galan, J. E., Liu, J. In Situ Molecular Architecture of the Salmonella Type III Secretion Machine. Cell. 168, 1065-1074 (2017).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152, 36-51 (2005).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14, 331-332 (2017).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Gilbert, P. Iterative methods for the three-dimensional reconstruction of an object from projections. Journal of Theoretical Biology. 36, 105-117 (1972).
- Radermacher, M. Weighted Back-projection Methods. Electron Tomography. , 245-273 (2007).
- Winkler, H., et al. Tomographic subvolume alignment and subvolume classification applied to myosin V and SIV envelope spikes. Journal of Structural Biology. 165, 64-77 (2009).
- Winkler, H., Taylor, K. A. Accurate marker-free alignment with simultaneous geometry determination and reconstruction of tilt series in electron tomography. Ultramicroscopy. 106, 240-254 (2006).
- Prevost, M. S., Waksman, G. X-ray crystal structures of the type IVb secretion system DotB ATPases. Protein Science. 27, 1464-1475 (2018).
- Miklos, G. L., Rubin, G. M. The role of the genome project in determining gene function: insights from model organisms. Cell. 86, 521-529 (1996).
- Reyrat, J. M., Pelicic, V., Gicquel, B., Rappuoli, R. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis. Infection and Immunity. 66, 4011-4017 (1998).
- Yu, J. Single-Molecule Studies in Live Cells. Annual Review of Physical Chemistry. 67, 565-585 (2016).
- Stewart, P. L. Cryo-electron microscopy and cryo-electron tomography of nanoparticles. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 9, (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon