Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Legionella pnömophila Dot/Icm Salgı Sisteminin Spatiotemporal Özelliklerini Çözmek İçin Canlı Hücre Görüntüleme ve Kriyo-Elektron Tomografisi Uygulama
Bu Makalede
Özet
Bakteri hücrelerinin görüntülenmesi, büyük makromoleküler makinelerin işlevini belirleyen statik ve dinamik süreçleri tanımlamaya odaklanmış yeni ortaya çıkan bir sistem biyolojisi yaklaşımıdır. Burada Lejyonella pnömophila tip IV salgı sistemi mimarisi ve fonksiyonlarını incelemek için kantitatif canlı hücre görüntüleme ve kriyo-elektron tomografisi entegrasyonu kullanılmaktadır.
Özet
Legionella pnömofilinin Nokta/Icm salgı sistemi, bakteri direğinde lokalize olan ve protein ve DNA substratlarının hedef hücrelere teslimine aracılık eden karmaşık bir tip IV salgı sistemidir (T4SS) nanomachine, genellikle doğrudan hücreden hücreye temas gerektiren bir süreçtir. Son zamanlarda kriyo-elektron tomografisi (kriyo-ET) ile Dot/Icm cihazının yapısını çözdük ve sitoplazmik komplekse bağlanan bir hücre zarfı yayılan kanal olduğunu gösterdik. Canlı hücrelerde ve kriyo-ET'de floresan mikroskopi, numunenin doğal yapısını koruyan iki tamamlayıcı yaklaşım uygulayarak, proteinlerin yerinde görüntülenmesine ve her makine bileşeninin diğer Dot/Icm alt birimlerine göre stokiyometrisinin asimilasyonunu ve zamanlamasını sağlar. Polar konumlandırma nın gereklerini araştırmak ve T4SS makine biyogenezi ile ilişkili dinamik özellikleri karakterize etmek için, kromozom üzerindeki doğal konumlarında Dot/Icm ATPaz genlerine süper klasör yeşil floresan proteinini kodlayan bir gen geliştirdik. Aşağıdaki yöntem, polar lokalizasyonu, dinamiği ve bu proteinlerin yapısını sağlam bakteri hücrelerinde ölçmek için canlı hücrelerin nicel floresan mikroskobu ve kriyo-ET'yi entegre eder. Legionella pnömofili T4SS'yi incelemek için bu yaklaşımların uygulanması Dot/Icm sisteminin işlevini karakterize etmek için yararlıdır ve T4SS'i veya diğer bakteriyel salgı komplekslerini kullanan çok çeşitli bakteriyel patojenleri incelemek üzere uyarlanabilir.
Giriş
Legionella pnömofili (L. pnömofili),Lejyoner hastalığının etiyolojik maddesi, bakterinin suda serbest yüzme protozoası içinde enfekte ve çoğalarak yayıldığı tatlı su rezervuarlarında yaşar. L. pnömofili içme suyu kaynaklarından aerosolize bakterilerin teneffüs meydana geldiğinde insanlarda hastalık salgınları neden olur. Enfekte hücrelerde, konak yollarının bozulması L. pnömofili içinde bulunduğu vakuol endositik olgunlaşma geciktirmek ve bakteriyel replikasyon destekleyen bir hücresel kompartman biyogenezi teşvik sağlar. Bu süreç, nokta/Icm olarak bilinen özel bir bakteri tipi IVB salgı sistemi (T4BSS) ve hücresel fonksiyonların manipülasyonunu kolaylaştırmak için enfeksiyon sırasında konak sitosola aktarılan 300'den fazla "efektör" proteininrepertörü tarafından yönlendirilir1,2,3, 4,4,5. Fonksiyonel bir Nokta / Icm aparatyoksun Mutantlar konak sitosol içine efektörler imal etmek için başarısız, hücre içi çoğaltma için kusurlu, ve hastalığın hayvan modellerinde avirulent vardır6,7.
Birçok bakteri türü enfeksiyon süreçleri için gerekli olan son derece karmaşık ve dinamik çok bileşenli makineler geliştirmiştir. Dot / Icm sistemi gibi diğer T4BSS de Coxiella burnetii ve Rickettsiella grylligibi bakteriyel patojenlerin hücre içi replikasyonu için gereklidir. T4BSS evrimsel olarak DNA transferine aracılık eden ve efektör proteinlerin sınırlı bir repertoiresini sunabilen prototip tip IVA sistemleriyle ilişkili olmasına rağmen, Dot/Icm sistemi neredeyse iki kat daha fazla makine bileşenine sahiptir ve çok çeşitli efektörler sunar. Muhtemelen, bileşen sayısındaki bu genişleme Dot/Icm aparatının yeni efektörleri kolayca barındırmasını ve entegre etmesini sağlamıştır8,9.
Son zamanlarda dot/Icm cihazının yapısını yerinde çözmek için kriyo-elektron tomografisi (kriyo-ET) kullandık ve sitoplazmik komplekse bağlanan bir hücre zarfı yayılan kanal olduğunu gösterdik. Daha ileri analizler, sitosolik ATPAz DotB'nin Sitosolik ATPAz DotO ile etkileşimyoluyla L. pnömofili hücre kutbundaki Dot/Icm sistemiyle ilişkilendirdiğini ortaya koymuştur. DotB'nin çoğu bakteri hücrelerinde sitosolik bir hareket gösterdiğini keşfettik, bu ATPaz'ın dinamik bir sitosolik popülasyonda bulunduğunu ve aynı zamanda kutup nokta/Icm kompleksleriyle de ilişkilendirdiğini gösteriyor. Buna ek olarak, DotO iç membran kompleksi ile ilişkili DotO dimers bir hexameric montaj oluşturur ve bir DotB hexamer bu sitoplazmik kompleksin tabanına katılır. DotB-DotO enerji kompleksinin montajı, substratların translokasyonlarını T4SS (Şekil 1)10üzerinden yönlendiren bir sitoplazmik kanal oluşturur.
Bu son gelişmelere rağmen, Nokta/Icm sisteminin nasıl işlediği ve her proteinin aktif bircihazoluşturmak için nasıl bir araya gelerek 8. Dot/Icm T4SS'nin düzenleyici devrelerinin ortaya çıkarılması, konak-patojen etkileşimlerinin moleküler mekanizmalarını anlamak için çok önemlidir. Bu nedenle, süper klasör GFP (sfGFP) ile etiketlenmiş temel L. pnömofili Dot/Icm sistem bileşenlerini tespit etmek ve karakterize etmek için canlı hücre mikroskobu ve kriyo-ET'nin nasıl kullanılacağını tartışıyoruz. Kantitatif floresan mikroskopisi kullanılarak, DotB'nin polar lokalizasyonu vahşi tip bir arka planda veya tip IV sistemi silindiğinde tanımlanır. Nokta/Icm sitosolik ATPaslar arasındaki lokalizasyon ve dinamik farklılıkları ölçmek için hızlandırılmış mikroskopi kullanılacaktır.
Canlı görüntüleme ve kriyo-ET gibi iki tamamlayıcı yaklaşımın birleştirilmiş uygulaması, diğer in vitro sistemlere göre avantaj sağlamaktadır. Her iki yöntem de bozulmamış hücrelerde gerçekleştirilir ve T4BSS'nin doğal ortamıkorunur, böylece numune hazırlama sırasında yerel yapının bozulması en aza indirilir. Proteinlerin aşırı ekspresyonu salgı cihazının stoiyometrisini bozabileceğinden, sfGFP füzyonları allelik değişim yoluyla Lejyonella kromozomuna döndürülür, böylece her füzyon tek bir kopyada kodlanır ve ifade endojen organizatör tarafından yönlendirilir. Kromozomla kodlanmış füzyonların görselleştirilmesi, tanımlanan bir zaman noktasında ifade edilen proteinin tam seviyesinin sayısallaştırılmasını sağlar. Cryo-ET de salgı sistemlerinin yapısını belirlemek için birçok avantajı vardır. En önemli avantajı kriyo-ET örnekleri bakteri hücre mimarisi bağlamında yerli kompleksleri korumak dondurulmuş bozulmamış hücrelerden oluşur olmasıdır. Sonuç olarak, kriyo-ET membran komplekslerini ayıklayan ve çekirdek aygıtından periferik proteinleri sıyırabilecek veya genel yapıyı değiştirebilen biyokimyasal arıtma yaklaşımlarına tercih edilebilir. Buna ek olarak, sfGFP gibi hantal bir protein ile ilgi bir protein etiketleme kriyo-ET tarafından tespit edilebilir bir kitle ekler ve kriyo-ET tarafından elde edilen yapı üzerine Dot / Icm aparatlarının farklı alt kompleksleri haritalama ile yardımcı olabilir.
Bu yaklaşım, bakteri hücre zarında biraraya gelen çok moleküler kompleksler hakkında yapısal bilgileri ortaya çıkarmak için güçlü bir araçtır. Bu teknikler kullanılarak açıklığa kavuşturulacak yapıların yorumlanması, alanın T4BSS bileşenlerinin nasıl çalıştığını, işlev için neden bu kadar çok bileşenin gerekli olduğunu, bileşenlerin büyük kompleks içinde nasıl etkileşime gireceğini ve bu işlevleri anlamalarına yardımcı olacaktır. alt montajlar gerçekleştirir.
Protokol
NOT: L. pnömofilinin büyümesi, manipülasyonu ve görüntülemesini içeren tüm prosedürler, yerel kurallara uygun olarak biyolojik güvenlik düzeyi 2 laboratuvarında yapılmalıdır.
1. Allelik Değişim ve Çift Seçim Stratejisi Kullanarak L. pnömofili Kromozomuna sfGFP takılması (Şekil 2, Şekil 3)
- Gen replasman vektörpSR47S11 içine Klon aşağıdaki sıra: ilgi sitenin 1.000 bp upstream, sonra sfGFP dizisi, sonra ilgi sitenin 1.000 bp downstream (Şekil 2). SfGFP dizisi N-terminus veya C-terminus çerçeve içinde yerleştirilmelidir dört ila sekiz amino asit içeren bir bağlayıcı ile biter. Ortaya çıkan vektörü E. coli DH5αλpir'e dönüştürün. Daha sonra, 100 μg/mL streptomisin içeren kömür-maya ekstresi (CYE) agar12 tek koloniler için seri L. pnömofili (alıcı) ve 37 °C 'de 5 gün boyunca büyür(Şekil 3).
- CYE-agar-streptomisin üzerinde Streak L. pnömofili ve 37 °C (ağır yama)102 gün boyunca büyümek . Streak E. coli DH5α 25 μg/mL kloramfenikol içeren LB agar üzerinde pRK600 yardımcı plazmid (helper)13 ile dönüştürülmüştür. 50 g/mL kanamisin içeren LB agar üzerinde E. coli DH5αλpir (donör) çizgi.
- Üçlü ailede miyonlama yapın: yardımcının kolonisini, donörün kolonisini ve alıcıyı cye agar plakasındaki üç suşun yamaları seçim yapmadan ve 37 °C'de 4-8 saat kuluçkaya yatarak kuluçkaya yatırın. Negatif kontroller bir yardımcı+alıcı gerinim karışımını ve aynı süreler için donör+alıcı gerinim karışımını kuluçkaya yatırır.
- 100 μg/mL streptomisin ve 10 μg/mL kanamisin içeren CYE agar üzerindeki reaksiyonların 500 μL ddH2O. Plaka 20 μL ve 50 μL'lik halindeki miyritleme reaksiyonlarını yeniden askıya alın ve 37 °C'de 5 gün boyunca büyüyün. 100 μg/mL streptomisin içeren CYE agar üzerinde ortaya çıkan klonların dört çizgi ve 37 °C'de 5 gün boyunca büyür.
- % 5 sakaroz ve 100 μg/mL streptomisin içeren CYE agar üzerinde 16 klonlar ve 37 °C'de 5 gün büyür. Daha sonra, 100 μg/mL streptomisin içeren CYE agar ve 100 μg/mL streptomisin ve 10 μg/mL kanamisin içeren CYE agar üzerinde bu klonların 32 çizgi 37 °C'de 5 gün büyür.
2. SfGFP'yi L. pnömofili Kromozomuna Entegre Eden Klonların İzolasyon
- CYE-agar-streptomisin plakalarında kanamisine duyarlı olan seri klonlar ve koloni PCR ile kromozomiçine sfGFP yerleştirimi doğrular. Ekleme kavşağını güçlendirmek için sfGFP geninin tamamlayıcısı olan astarları ve ilgi nin kromozom alacağı bölgeyi kullanın.
- 10°M astar çözeltilerinin her birinden 0,5 μL'yi ve bir koloniyi 12,5 μL'lik son bir hacme ve 10 dakika boyunca 95 °C'de denatüre yedeği karıştırın. 10 dakika boyunca buzüzerinde soğutun, 12,5 μL 2x PCR ana karışım çözeltisi ekleyin ve PCR analizi yapın.
- CYE-agar-streptomisin plakaları üzerinde izole kolonilerin ağır yamalar büyümek 2 gün boyunca 37 °C. SfGFP füzyonlarının ekspresyon düzeylerini ve stabilitesini bir anti-GFP antikor ile immünoblot yaparak inceleyin.
3. Floresan Etiketli Nokta/Icm Bileşenleri ile L. pnömofili Canlı Hücre Görüntüleme
- Agarose pedlerin hazırlanması
- Suda %1 oranında az erimiş agarose çözeltisinin yaklaşık 30 mL'sini yapın. Mikrodalga yaklaşık 90 s için bir cam şişesi, zaman zaman dönen, agarose tamamen çözülmüş kadar.
- Biri üst üste 25 x 75 x 1,1 mm 3 cam kaydırak, 25 x 75 x 1,1 mm3 cam kaydırak kenarına iki adet 22 x 22 x 0,15 mm3 cam kaydırağı yerleştirin. Diğer kenarına iki 22 x 22 x 0,15 mm3 cam kaydırağı daha yığ.
- İki üst cam slayt arasında orta slayt içine erimiş agarose yaklaşık 1 mL pipet, sonra erimiş agarose üstüne başka bir 25 x 75 x 1.1 mm3 slayt yerleştirin. Hava kabarcıkları oluşumunu önlemek için deneyin. 4 °C'de 15 dk kaydırın.
- Neşter veya jilet kullanarak, pedi yavaşça küçük kareler halinde kesin, ~5 x 5 mm2. Çift taraflı yapışkan 17 x 28 x 0,25 mm3 çerçeveyi 25 x 75 x 1,1 mm3 cam kaydırağa sabitleyip slayta birkaç ped yerleştirin.
- Görüntü edinimi
NOT: SlideBook 6.0'ın kontrolü altında bulunan ve katı hal aydınlatıcıları, CCD tek renkli kamera ve 100 x objektif lens (1.4 sayısal diyafram açıklığı) ile donatılmış bir mikroskop için aşağıdaki adımlar açıklanmıştır. Gerekirse, protokol ayarlarına göre özelleştirilebilen uygun donanım ve yazılım yapılandırmalarına sahip alternatif mikroskopi aygıtları kullanın.- DDH2O, girdap ve pipet 2-3 μL seyreltme yastıkları üzerine 1 mL L. pnömofili ağır bir yama eritin. 50 x 24 x 0,15 mm3 kapak kaymasını yapışkan çerçevenin üzerine hafifçe yerleştirin.
- Yakalama penceresinde ND'yi 180 olarak ayarlayın. Binning'i 2×2'ye ayarlayın ve 488 nm kanalını kullanarak numuneyi 500-1.000 ms arasında teşletmek için kullanın.
4. Kutupsal Lokalizasyon ve Nokta/Icm Bileşenlerinin Dinamiğinin Ölçülmesi
NOT: Aşağıdaki adımlar piksel başına 0,129 μm olan ve 2 x 2 binning ile elde edilen görüntüler için tasarlanmıştır.
- sfGFP füzyon proteinleri için polaritenin nicelleştirilmesi (Şekil 5)
- Görüntü kontrastını, bakterilerin açıkça görülebilmesi için ayarlayın. Kutuptan başlayıp sitoplazmaya doğru uzanan 0,25 x 1,3 μm2 dikdörtgen yerleştirmek için bölge aracını kullanın. Dikdörtgen tam olarak bakteri sınırları içinde kalmalıdır.
- En az 200 bakteri işaretleyin ve ilgi çeken bölgelere maske oluşturmak için Bölge'yi Maskelemek düğmesini kullanın. Maske İstatistikleri ve Maske Kapsamıaltında, Nesne'yiseçin. Sonra, Özellikler ve Yoğunlukaltında , mark Ortalama Yoğunluk ve Varyans.
- Verileri dışa aktarın ve her bakterinin polarite puanlarını ortalama yoğunluk arasındaki oran olarak hesaplayın.
- Yüksek işlem uygulamaları için faz ve 488 nm kanallı görüntüler elde etmek için faz hedefi ve uygun bir kondansatör kurulumu kullanın. Bakterilerin tamamen ayrıldığı görüş alanlarını seçtiğinizden emin olun.
- Görüntünün faz kanalı kontrastını bakterilerin açıkça görülebileceği bir seviyeye ayarlayın. Çift kanallı görüntüyü açın, Segment Maskesi Oluştur penceresini başlatın ve kanalı faza çevirin.
- Nesneleri Tanımla düğmesiyle uygun bir eşiği ayarlayın ve küçük nesneleri kaldırın. Maskeyi Hassaslaştır'ın altında Kenarları Kaldır Nesneleri'ni ve daha sonra birbirine bitişik olan bakterileri ayırın maskelerini seçin.
- Daha önce 4.1.2-4.1.3 adımlarında açıklandığı gibi, her hücre için 488 kanaldaki sinyalin polarite puanlarını hesaplayın.
- sfGFP füzyon proteinleri için dinamiğin ölçülmesi (Şekil 6)
NOT: Görüntü edinimi için bir örnek hazırlamak için bölüm 3'teki yönergeleri izleyin. Dinamikler zaman içinde yoğunluk değişiklikleri olarak tanımlanır ve aşağıdaki adımlar kısa görüntüleme dönemleri için tasarlanmıştır (yani, birkaç dakika). Daha uzun görüntüleme süreleri isteniyorsa uygun takviyeleri ped ekleyin.- Görüntü Yakalama penceresinde, Timelapseişaretleyin, aralık kutusuna aralıkların saatini girin ve Zaman Noktaları kutusuna # 2 girin. L. pnömofilinin floresan proteinini ifade eden art arda iki görüntüsünü elde edin.
- Hücreler açıkça görünene kadar görüntü kontrastını ayarlayın. Üç farklı maske yerleştirmek için Şekil 6A'yı ve aşağıdaki açıklamaları takip edin. En az 400 hücrenin ortasına 0,25 x 0,25 m kare yerleştirmek için bölge aracını kullanın.
- İlgi çekici karelerin maskesi (maske 1) oluşturmak için Maskeiçin Bölge düğmesini kullanın. Yeni bir boş maske (maske 2) oluşturun ve floresan ağartma hesaplamak için kullanılacak en az 25 rasgele hücrelerin tüm hücre alanını işaretlemek için piksel aracını veya çokgen aracını kullanın. Yeni bir boş maske (maske 3) oluşturun ve arka plan çıkarma için kullanılacak hücreler arasındaki alanları işaretlemek için büyük fırça aracını kullanın.
- Maske İstatistikleri ve Maske Kapsamıaltında, maske 1 ve maske 2 için Nesne'yi seçin. Daha sonra, Özellikler ve Yoğunlukaltında, Yoğunluk Ortalama'yı seçin ve iki maskenin verilerini dışa aktarın. Maske 3 için, tüm maskenin ortalama yoğunluğunu dışa aktarın.
- Aşağıdaki formülleri kullanarak maske 1'deki her nesne için floresan yoğunluğundaki değişimi hesaplayın:

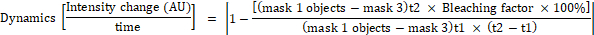
maske 1 hücre merkezine yerleştirilen 0,25 μm × 0,25 μm kare, maske 2 tüm hücre kapsar, maske 3 hücreler arasında arka plan, t1 ilk kez noktada ortalama yoğunluğu ve t2 ikinci zaman noktasında ortalama yoğunluğudur.
5. Cryo-ET ile sfGFP Kütle Yoğunluğunun Saptanması
- Örnek hazırlama, veri toplama ve yeniden yapılandırma
- 37 °C'de 48 saat boyunca CYE-agar-streptomisin plakalarında floresan etiketli Nokta/Icm proteinini ifade eden l. pnömofili nin ağır bir yamasını büyütün. DDH2O'daki hücreleri OD600 ~0.7'ye yeniden askıya alın. Hücre süspansiyonunun 20 μL'sine 5 μL kolloidal altın parçacıkları (BSA Tracer, 10 nm) ekleyin.
- Pipet 5 μL taze parlaklık-deşarj delikli karbon ızgara üzerine hücre karışımı (Cu 200 mesh Üzerinde R 2/1) ve filtre kağıdı ile 1 dakika leke için stand sağlarve daha önce açıklandığı gibi bir yerçekimi tahrikli piston aparatı kullanarak sıvı etandondur.
- Bir alan emisyon tabancası, bir enerji filtresi, Volta faz plakası ve doğrudan algılama cihazı ile donatılmış 300 kV iletim elektron mikroskobu ile dondurulmuş sulu numuneler görüntü. Tek eksenli eğim serilerini 26.000 x ve 42.000 x büyütmelerde toplayın ve bu da piksel boyutlarının sırasıyla 5,4 Å/piksel veya 3,4 Å/piksel seviyesinde sonuçlanmasına neden olabilir.
- -60° ile +60° arasında bir dizi eğim açısına sahip ve ~60 e-/ş2,16'lıkbirikmiş dozile görüntü yığınlarını ~0 μm defokusta toplamak için tomografik paket SerialEM'i kullanın. MotionCor217kullanarak her yığında doz fraksiyonlu film görüntüleri hizalamak. TOMOAUTO14kullanarak sürüklenme düzeltilmiş yığınları bir araya getirin.
- Drift düzeltilmiş yığınları IMOD işaretçiye bağımlı hizalama18ile hizala. Segmentasyonlar ve doğrudan görüntü analizi için SIRT yöntemi19 ve WBP yöntemi20ile tomogramları yeniden yapıla.
- sfGFP füzyon örneklerinin subtomogram analizi
- Subtomogram analizi14,21,22için tomografik paket I3 (0.9.9.3) kullanın.
NOT: Hizalama yinelemeli olarak ilerler, her yineleme, referansların ve sınıflandırma maskelerinin oluşturulduğu, subtomogramların hizalandığı ve sınıflandırıldığı ve sınıf ortalamalarının birbirine hizalandığı üç bölümden oluşur. - İlk hizalama için 4 x 4 x 4 binnli subtomogram kullanın. Sınıf ortalamalarına ait parçacıkları birleştirin ve sfGFP'ye karşılık gelen elektron yoğunluğunu gösterin. SfGFP füzyonları ile parçacıkları sıraladıktan sonra, yüksek çözünürlüklü bir yapı elde etmek için ilgi bir bölgenin (Nokta/Icm sitoplazmik ATPase kompleksi gibi) odaklanmış bir hizalaması için 2 x 2 x 2 binnli subtomogram kullanın.
- Subtomogram analizi14,21,22için tomografik paket I3 (0.9.9.3) kullanın.
Sonuçlar
SfGFP'nin tanımlanan eklemesini oluşturmak için iki adımda çift seçimli homolog rekombinasyon kullanılmıştır. İlk adımda, triparental çiftleşmesi yapıldı, burada PRK600 konjugatif plazmid (bir IncP plazmid) E. coli yardımcı suşu MT616 gelen donör E. coli suşu intihar vektörü pSR47S iki homolog bölgeler, transfer oriT ve Bacillills subselectionsubselection subt istifi ile çevrili içeren sfGFP geni ile seferber edildi . Daha sonra pSR47S türevinin pRK600 destek...
Tartışmalar
Bakteriyel salgı sistemlerinin işlevlerinin aydınlatılması, konak-patojen etkileşimlerinin tam olarak anlaşılmasının anahtarıdır. Salgı sistemleri, etki edici proteinleri konak hücrelerine enjekte edebilen karmaşık makinelerdir ve bazı durumlarda bakteri çoğaltMaişlemini destekleyen bir hücre altı nişin kurulmasını teşvik eder. Yukarıdaki yöntem, solunum bakteriyel patojen lejyonella pnömofili Nin Dot/Icm salgı sistemini incelemek için önemli yeni araçlar sağlayarak patojeniliğ...
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
D.C. ve C.R.R. NIH (R37AI041699 ve R21AI130671) tarafından desteklendi. D.P., B.H., ve J.L Ulusal Sağlık Enstitüleri (R01AI087946 ve R01GM107629) tarafından desteklendi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold particles | Aurion | 25486 | |
| 100x Plan Apo objective (1.4 NA) | Nikon | ||
| ACES | Sigma-Aldrich | A9758 | |
| Activated charcoal | Sigma-Aldrich | C5510 | |
| Agaroze GPG/LMP, low melt | American bioanalytical | AB00981 | |
| Bacto dehydrated agar | BD | 214010 | |
| CoolSNAP EZ 20 MHz digital monochrome camera | Photometrics | ||
| Gene Frame, 1.7x2.8 cm, 125 µL | Fisher Scientific | AB-0578 | |
| Holey Carbon grid R 2/1 Cu 200 mesh | Quantifoil | Q225-CR1 | |
| Iron(III) nitrate nonahydrate | Sigma-Aldrich | 216828 | |
| K2 Summit camera for cryo-EM | GATAN | ||
| L-Cysteine | Sigma-Aldrich | C7352 | |
| Microscope cover slides 22x22 mm | Fisher Scientific | 12-542B | |
| Microscope cover slides 24x50 mm | Fisher Scientific | 12-545K | |
| Microscope slides 25x75x1 mm | Globe Scientific | 1380 | |
| SlideBook 6.0 | Intelligent Imaging Innovations | ||
| Spectra X light engine | Lumencor | ||
| Taq 2X Master Mix | New England BioLabs | M0270 | |
| Titan Krios | Thermo Fisher Scientific | ||
| Yeast Extract | BD | 212750 |
Referanslar
- Franco, I. S., Shuman, H. A., Charpentier, X. The perplexing functions and surprising origins of Legionella pneumophila type IV secretion effectors. Cellular Microbiology. 11, 1435-1443 (2009).
- Burstein, D., et al. Genome-scale identification of Legionella pneumophila effectors using a machine learning approach. PLOS Pathogens. 5, 1000508 (2009).
- Ninio, S., Roy, C. R. Effector proteins translocated by Legionella pneumophila: strength in numbers. Trends in Microbiology. 15, 372-380 (2007).
- Vogel, J. P., Andrews, H. L., Wong, S. K., Isberg, R. R. Conjugative transfer by the virulence system of Legionella pneumophila. Science. 279, 873-876 (1998).
- Isberg, R. R., O'Connor, T. J., Heidtman, M. The Legionella pneumophila replication vacuole: making a cosy niche inside host cells. Nature Reviews Microbiology. 7, 13-24 (2009).
- Roy, C. R., Berger, K. H., Isberg, R. R. Legionella pneumophila DotA protein is required for early phagosome trafficking decisions that occur within min of bacterial uptake. Molecular Microbiology. 28, 663-674 (1998).
- Archer, K. A., Roy, C. R. MyD88-Dependent Responses Involving Toll-Like Receptor 2 Are Important for Protection and Clearance of Legionella pneumophila in a Mouse Model of Legionnaires' Disease. Infection and Immunity. 74, 3325-3333 (2006).
- Nagai, H., Kubori, T. Type IVB Secretion Systems of Legionella and Other Gram-Negative Bacteria. Frontiers in Microbiology. 2, 136 (2011).
- Kubori, T., Nagai, H. The Type IVB secretion system: an enigmatic chimera. Current Opinion in Microbiology. 29, 22-29 (2016).
- Chetrit, D., Hu, B., Christie, P. J., Roy, C. R., Liu, J. A unique cytoplasmic ATPase complex defines the Legionella pneumophila type IV secretion channel. Nature Microbiology. 3, 678-686 (2018).
- Merriam, J. J., Mathur, R., Maxfield-Boumil, R., Isberg, R. R. Analysis of the Legionella pneumophila fliI gene: intracellular growth of a defined mutant defective for flagellum biosynthesis. Infection and Immunity. 65, 2497-2501 (1997).
- Feeley, J. C., et al. Charcoal-yeast extract agar: primary isolation medium for Legionella pneumophila. Journal of Clinical Microbiology. 10, 437-441 (1979).
- Andrews, H. L., Vogel, J. P., Isberg, R. R. Identification of linked Legionella pneumophila genes essential for intracellular growth and evasion of the endocytic pathway. Infection and Immunity. 66, 950-958 (1998).
- Morado, D. R., Hu, B., Liu, J. Using Tomoauto: A Protocol for High-throughput Automated Cryo-electron Tomography. Journal of Visualized Experiments. (107), e53608 (2016).
- Hu, B., Lara-Tejero, M., Kong, Q., Galan, J. E., Liu, J. In Situ Molecular Architecture of the Salmonella Type III Secretion Machine. Cell. 168, 1065-1074 (2017).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152, 36-51 (2005).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14, 331-332 (2017).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Gilbert, P. Iterative methods for the three-dimensional reconstruction of an object from projections. Journal of Theoretical Biology. 36, 105-117 (1972).
- Radermacher, M. Weighted Back-projection Methods. Electron Tomography. , 245-273 (2007).
- Winkler, H., et al. Tomographic subvolume alignment and subvolume classification applied to myosin V and SIV envelope spikes. Journal of Structural Biology. 165, 64-77 (2009).
- Winkler, H., Taylor, K. A. Accurate marker-free alignment with simultaneous geometry determination and reconstruction of tilt series in electron tomography. Ultramicroscopy. 106, 240-254 (2006).
- Prevost, M. S., Waksman, G. X-ray crystal structures of the type IVb secretion system DotB ATPases. Protein Science. 27, 1464-1475 (2018).
- Miklos, G. L., Rubin, G. M. The role of the genome project in determining gene function: insights from model organisms. Cell. 86, 521-529 (1996).
- Reyrat, J. M., Pelicic, V., Gicquel, B., Rappuoli, R. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis. Infection and Immunity. 66, 4011-4017 (1998).
- Yu, J. Single-Molecule Studies in Live Cells. Annual Review of Physical Chemistry. 67, 565-585 (2016).
- Stewart, P. L. Cryo-electron microscopy and cryo-electron tomography of nanoparticles. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 9, (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır