A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
החלת הדמיית תאים בשידור חי וטומוגרפיה ממוחשבת כדי לפתור את התכונות הספאטימיות של הלגיונרים האלה מערכת הפרשות
In This Article
Summary
הדמיה של תאים חיידקיים היא גישה ביולוגיה מערכות מתפתחים התמקדו הגדרת תהליכים סטטיים ודינאמיים המכתיבים את הפונקציה של מכונות macromolecular גדולות. כאן, שילוב של הדמיה תא כמותי לחיות וטומוגרפיה מסוג ההקפאה משמשת לחקר הלגיונרים האלה הפרשת מערכת הפרשה ופונקציות.
Abstract
נקודה/Icm הפרשת מערכת של הלגיונרים משאבה האלה היא סוג מורכב IV הפרשה מערכת (T4SS) nanomachine כי לאתר את הקוטב החיידקי ו סכסוכי את המסירה של חלבונים ו-DNA מצעים כדי למקד את התאים, תהליך בדרך כלל דורש קשר ישיר תא אל התא. לאחרונה פתרנו את המבנה של מנגנון נקודה/Icm על ידי טומוגרפיה ההקפאה-אלקטרון (ההקפאה) והראו כי הוא יוצר מעטפת תא מפורש ערוץ המתחבר למתחם cytoplasmic. החלת שתי גישות משלימות לשימור המבנה היליד של הדגימה, מיקרוסקופ פלורסנט בתאי החיים וההקפאה, מאפשר ויזואליזציה באתרו של חלבונים והטמעה של הסטואיצ'ימטריה והעיתוי של ייצור של כל רכיב מחשב ביחס ליחידות אחרות של נקודה/Icm. כדי לחקור את הדרישות עבור מיצוב קוטבי ולאפיין תכונות דינמיות המשויכות T4SS מחשב biogenesis, יש לנו התמזגו הקידוד הגן הירוק של התיקייה הפלואורסצנט חלבון פלורסנט נקודה/Icm ATPase במיקומים מקוריים שלהם על הכרומוזום. השיטה הבאה משלבת מיקרוסקופ קרינה פלואורסצנטית כמותי של תאים חיים והקפאה-ET כדי לכמת לוקליזציה קוטבית, דינמיקה, ומבנה של חלבונים אלה בתאים חיידקיים שלמים. החלת גישות אלה עבור לימוד הלגיונרים האלה משאבה T4SS היא שימושית לאפיון הפונקציה של מערכת נקודה/Icm והוא יכול להיות מותאם ללמוד מגוון רחב של פתוגנים חיידקיים המשתמשים T4SS או סוגים אחרים של מכלולי הפרשה חיידקיים.
Introduction
הלגיונרים האלה משאבה (L. משאבה), הסוכן הפרוטולוגי של מחלת הלגיונרים, שוכן מאגרי מים מתוקים, שם החיידקים מופצים על ידי הדבקה ושכפול בתוך מימיים שחיה בחינם פרוטותים. ל. משאבה הגורמת להתפרצויות של מחלות בבני אדם כאשר האינהלציה של חיידקים aerosolized ממקורות מים ראויים לשתייה מתרחשת. בתאים נגועים, החתרנות של מסלולים מארחים מאפשר L. משאבה המרחפת כדי לעכב התבגרות endocytic של החללים שבהם הוא שוכן לקדם biogenesis של תא התא התומך שכפול חיידקי. תהליך זה מונע על ידי סוג חיידקי מיוחדות ivb הפרשה (T4BSS) המכונה נקודה/Icm והרפרטואר שלה של מעל 300 "אפקטור" חלבונים כי הם translocated לתוך מארח ציטוסול במהלך הזיהום כדי להקל על מניפולציה של פונקציות סלולריות1,2,3,4,5. מוטציות חסרות נקודה תפקודית/Icm מנגנון להיכשל לספק את העריקים לתוך המחשב המארח ציטוסול, פגומים עבור שכפול תאיים, והם avirulent במודלים של בעלי חיים של מחלה6,7.
זנים חיידקיים רבים פיתחו מכונות רב רכיבי מורכבות ודינמיות מאוד הנדרשות לתהליכי זיהום. אחרים T4BSS כמו מערכת נקודה/Icm חיוניים גם עבור שכפול תאיים של פתוגנים חיידקיים כגון Coxiella בורנתיבי ו ריקטסיאלה grylli. למרות T4BSS הקשורים באבולוצאוסיים מערכות מסוג מערכת ה-DNA, אשר מתווכים העברת דנ א והוא יכול לספק רפרטואר מוגבל של חלבונים אפקטור, מערכת נקודה/Icm יש כמעט פי שניים מרכיבי מחשב ומספק מגוון רחב של מכונות. ככל הנראה, התרחבות זו במספר הרכיבים אפשרה למנגנון הנקודה/Icm להכיל ולשלב בקלות מחדש את האפשרות8,9.
השתמשנו לאחרונה טומוגרפיה אלקטרון-אלקטרונים (ההקפאה) כדי לפתור את המבנה של מנגנון נקודה/Icm באתרו והראה כי זה יוצר תא מעטפה מפורש ערוץ המתחבר למתחם cytoplasmic. ניתוח נוסף עולה כי ציטוסולוג ATPase DotB מקורביו עם מערכת נקודה/Icm בקוטב התא L. משאבה מנועי באמצעות אינטראקציות עם מיקוד. גילינו כי DotB מציג את התנועה ציטוסולג בתאי חיידקי ביותר, המציין כי ATPase זה קיים באוכלוסייה cytosolic דינאמי אך גם מקורביו עם נקודה קוטבית/מתחמי Icm. בנוסף, הקבוצה מהווה הרכבה של הקסאמרס הקשורה למכלול הממברנה הפנימי, והיא מצטרפת לבסיס של מתחם cytoplasmic זה. ההרכבה של מורכבות DotB-לעשות אנרגיה יוצר ערוץ cytoplasmic המנחה את הטרנסלוקציה של מצעים דרך T4SS (איור 1)10.
למרות ההתקדמות האחרונה, מעט ידוע על איך מערכת נקודה/Icm פונקציות ואיך כל חלבון מרכיב ליצור מנגנון פעיל8. חשיפת המעגלים הרגולטורים של הנקודה/Icm T4SS היא בסיסית כדי להבין את המנגנונים המולקולריים של אינטראקציות מארח הפתוגן. לכן, אנו דנים כיצד להשתמש במיקרוסקופיה תא לחיות והקפאה-ET כדי לזהות ולאפיין את הרכיבים החיוניים L. משאבה האנטי/Icm המערכת מתויגים עם סופר תיקיית gfp (sfgfp). באמצעות מיקרוסקופ פלואורסצנטית כמותי, לוקליזציה קוטבי של DotB יוגדר ברקע סוג פראי או כאשר מערכת הרביעי סוג מחיקת. מיקרוסקופ הזמן לשגות ישמש לכמת הבדלים בלוקליזציה ודינמיקה בין הנקודה/Icm ציטוסולטים ATPases.
היישום המשולב של שתי גישות משלימות כגון הדמיה חיה ו-ההקפאה מספק יתרון לעומת אחרים במערכות מבחנה. שתי השיטות מבוצעות בתאים שלמים ושומרות על הסביבה הטבעית של T4BSS, ובכך מזעור השיבוש של המבנה היליד במהלך הכנת המדגם. כי יתר הביטוי של חלבונים עלול לפגוע הסטואיצ'ימטריה של מכשיר הפרשת, sfGFP fusions מוחזרים באמצעות allelic exchange לתוך כרומוזום הלגיונרים , כך שכל היתוך מקודד בעותק אחד והביטוי מונע על ידי מקדם אנדוגני. הדמיה של fusions מקודד כרומוסומלית מאפשרת לכמת את רמת החלבון המדויקת המתבטאת בנקודת זמן מוגדרת. ל-"הקפאה" יש גם יתרונות רבים לקביעת מבנה מערכות הפרשה. היתרון הבולט ביותר הוא שדגימות ההקפאה מורכבות מתאי שלמים קפואים הישמרו על מתחמי מקומיים בהקשר של ארכיטקטורת תאים חיידקיים. כתוצאה מכך, ההקפאה עשויה להיות עדיפה על גישות הטיהור הביוכימי, אשר לחלץ מתחמי ממברנה עשוי להתפשט חלבונים היקפיים ממנגנון הליבה או לשנות את המבנה הכללי. בנוסף, תיוג חלבון של עניין עם חלבון מגושם כגון sfGFP מוסיף מסה שניתן לגילוי על-ידי ההקפאה והוא יכול לסייע עם מיפוי subcomplexes שונים של המנגנון נקודה/Icm על המבנה המתקבל על ידי המקפיא-ET.
גישה זו היא כלי רב עוצמה לגילוי מידע מבני על מתחמים רב מולקולריים להרכיב קרום התא חיידקי. הפרשנות של מבנים הובהר באמצעות טכניקות אלה תסייע לשדה להבין כיצד T4BSS רכיבי הפונקציה, מדוע רכיבים רבים כל כך נדרשים לפונקציה, איך הרכיבים אינטראקציה בתוך המתחם הגדול, ומה פונקציות אלה בצע הרכבות משנה.
Protocol
הערה: יש לבצע את כל ההליכים הכרוכים בצמיחה, מניפולציה והדמיה של L. משאבה ברמת בטיחות ביולוגית 2 בהתאם להנחיות המקומיות.
1. החדרת התכנה לתוך כרומוזום L. משאבה באמצעות Allelic Exchange ואסטרטגיית בחירה כפולה (איור 2, איור 3)
- שיבוט לתוך החלפת גנים וקטור pSR47S11 את הרצף הבא: 1,000 bp במעלה הזרם של העניין, ואז את הרצף sfGFP, ואז 1,000 bp במורד האתר של העניין (איור 2). יש למקם את הרצף sfGFP במסגרת לסוף N-טרמינוס או C-טרמינוס עם מקשר המכיל ארבע עד שמונה חומצות אמינו. הפוך את הווקטור המתקבל לתוך E. coli DH5αλpir. מאוחר יותר, פס ל. משאבה (המטופל) עבור מושבות יחיד על תמצית שמרים פחם (cye) אגר12 המכיל 100 μg/mL סטרפטומיצין ולגדול 5 ימים ב 37 ° צ' (איור 3).
- פס ל. משאבה ב-cye-אגר-סטרפטומיצין וגדלים במשך יומיים ב 37 ° צ' (טלאי כבד)10. רצף E. coli DH5α השתנה עם pRK600 עוזר פלמיד (עוזר)13 על ליברות אגר המכיל 25 μg/mL כלוראמנול. רצף את E. coli DH5αλpir (תורם) על LB אגר המכיל 50 μg/mL kanamycin.
- לבצע הזדווגות הורים: המאכטה מושבה של העוזר, מושבה של התורם, ואת הנמען על ידי הנחת טלאים של שלושה זנים על צלחת באגר CYE ללא בחירה ו הדגירה עבור 4 – 8 h ב 37 ° c. כאשר שולט שלילי משלב המסייע + מערבב זנים של הנמען והתורם + זן הנמען ערבוב עבור אותם פרקי זמן.
- להשעות את תגובות ההזדווגות ב 500 μL של ddH2O. צלחת 20 μl ו 50 μl של התגובות על cye אגר המכיל 100 μg/ml סטרפטומיצין ו 10 Μg/ml kanamycin ולגדול 5 ימים ב 37 ° c. פס ארבע של המשובטים שהתקבל על CYE אגר המכיל 100 μg/mL סטרפטומיצין ולגדול 5 ימים ב 37 ° c.
- פס 16 שיבוטים על CYE אגר המכיל 5% סוכרוז ו-100 μg/mL סטרפטומיצין וגדלים במשך 5 ימים ב 37 ° c. אז, פס 32 של שיבוטים אלה על CYE אגר המכיל 100 μg/mL סטרפטומיצין ועל CYE אגר המכיל 100 μg/mL סטרפטומיצין ו 10 μg/mL kanamycin ולצמוח 5 ימים ב 37 ° c.
2. בידוד של המשובטים כי משולב הספגfp לתוך כרומוזום L. משאבה
- פס שיבוטים שהיו רגישים לקאנאמיצין על CYE-אגר-סטרפטומיצין צלחות ולאשר את החדרת sfGFP לתוך הכרומוזום עם ה-PCR המושבה. השתמש בצבעי היסוד המשלימים את הגן sfGFP ולאזור הכרומוזום של הריבית כדי להגביר את צומת הכניסה.
- מערבבים 0.5 μL של כל אחד 10 הפתרונות התחל באמצעות μM ומושבה אחת לכרך הסופי של 12.5 μL ו-1 הספרות עבור 10 דקות ב 95 ° c. קריר על קרח 10 דקות, להוסיף 12.5 μL של פתרון מאסטר 2x PCR, ולבצע ניתוח PCR.
- הגדל כתמים כבדים של מושבות מבודדות על CYE-אגר-סטרפטומיצין צלחות עבור 2 ימים ב 37 ° c. בדוק את רמות הביטוי והיציבות של הfusions-sfGFP באמצעות נוגדן אנטי-GFP.
3. הדמיה של תא חי של L. משאבה עם שתייגת פלואורוסקופים נקודה/Icm רכיבים
- הכנת רפידות צמח
- הפוך כ-30 מ ל של 1% להמיס מצומצם של פתרון במים. מיקרוגל בבקבוקון זכוכית עבור כ 90 s, מתערבל מדי פעם, עד הצמח הוא התפרקה לחלוטין.
- מקום 2 22 x 22 x 0.15 mm3 שקופיות זכוכית על קצה של 25 x 75 x 1.1 mm3 זכוכית שקופית, אחד על גבי השני. מחסנית 2 יותר 22 x 22 x 0.15 מ"מ3 שקופיות זכוכית על הקצה השני.
- פיפטה בערך 1 מ ל של agarose מותכת לתוך השקופית המרכז בין שתי שקופיות הזכוכית העליונה, ואז מניחים עוד 25 x 75 x 1.1 מ"מ3 השקופית על גבי agarose מותכת. נסה להימנע היווצרות בועות אוויר. לצנן את השקופיות ב 4 ° c עבור 15 דקות.
- באמצעות אזמל או סכין גילוח, בעדינות לחתוך את הלוח לריבועים קטנים, ~ 5 x 5 מ"מ2. לתקן דבק דו צדדי 17 x 28 x 0.25 מ"מ3 מסגרת על 25 x 75 x 1.1 mm3 שקופית זכוכית ומניחים מספר רפידות בשקופית.
- רכישת תמונה
הערה: השלבים הבאים מתוארים עבור מיקרוסקופ כי הוא תחת השליטה של SlideBook 6.0 ומצויד המוארים המדינה מוצק, CCD מצלמה חד-צבעית, ועדשת היעד 100x (1.4 מפתח נומרי). במידת הצורך, השתמש בהתקני מיקרוסקופ חלופיים עם תצורות חומרה ותוכנה מתאימות שניתן להתאים אישית בהתאם להגדרות הפרוטוקול.- התמוססות טלאי כבד של L. משאבה בתוך 1 מ ל של ddh2O, מערבולת ו pipet 2 – 3 μl של הדילול על הרפידות. מקום 50 x 24 x 0.15 מ"מ3 שמיכות בעדינות מעל מסגרת דבק.
- בחלון הלכידה התאימו את ה-ND ל-180. להתאים את binning ל 2 × 2 ולהשתמש בערוץ 488 ננומטר לחשוף את המדגם בין 500-1000 ms. לאמת את הספציפיות של האות הפלואורסצנטית על ידי הדמיה לא מתויגים L. משאבה עם אותם הפרמטרים (איור 4).
4. קוונפיקציה של לוקליזציה קוטבית דינמיקה של רכיבי נקודה/Icm
הערה: השלבים הבאים מיועדים לתמונות עם 0.129 יקרומטר לכל פיקסל שנרכשו עם 2 x 2 binning.
- קוונפיקציה של קוטביות עבור התקני היתוך (איור 5)
- התאימו את הניגודיות של התמונה כך שהחיידקים יהיו גלויים בבירור. השתמש בכלי אזור למקום 0.25 x 1.3 יקרומטר2 מלבן החל בקוטב והארכת לתוך הציטופלסמה. המלבן חייב להישאר בדיוק בתוך הגבולות החיידקיים.
- סמן לפחות 200 חיידקים והשתמש באזור ללחצן מסיכה כדי ליצור מסיכות לאזורי העניין. תחת מסיכת סטטיסטיקה וטווח מסיכות, בחר באפשרות אובייקט. לאחר מכן, תחת תכונות ועוצמה, סמן ממוצע עוצמה וסטיה.
- יצא את הנתונים וחשב את תוצאות הקוטביות של כל חיידק כיחס בין השונות לעוצמה הממוצע.
- עבור יישומים בעלי תפוקה גבוהה, יש להשתמש במטרת שלב ובכיוונון העבה המתאים לרכישת תמונות עם הפאזה וערוצים 488 ננומטר. הקפד לבחור שדות תצוגה היכן החיידקים מופרדים לחלוטין.
- כוונן את הניגודיות של ערוץ הפאזה של התמונה לרמה שבה החיידקים גלויים בבירור. פתחו את תמונת הערוץ הכפול, הפעל את החלון ' צור מסיכת מקטע ' ושנה את הערוץ לשלב.
- התאימו את הסף המתאים והסירו עצמים קטנים בעזרת הלחצן ' הגדר עצמים '. תחת ' מקד מסיכה ' בחרו ' הסר אובייקטי קצוות ' ומסכות נפרדות של חיידקים הסמוכים זה לזה.
- חשב את ציוני הקוטביות של האות בערוץ 488 עבור כל תא כפי שתואר בעבר בשלבים 4.1.2 – 4.1.3.
- הכמת של דינמיקה עבור התקני היתוך חלבונים (איור 6)
הערה: בצע את ההוראות בסעיף 3 כדי להכין מדגם עבור רכישת תמונה. דינמיקות מוגדרות כשינויים באינטנסיביות לאורך זמן והשלבים הבאים מיועדים לתקופות דימות קצרות (כלומר, מספר דקות). הוסף למשטח את תוספי התזונה המתאימים אם יש צורך בתקופות דימות ארוכות יותר.- בחלון לכידת תמונה , סמן אתהזמן הקצוב בתיבה מרווח והזן 2 ב-תיבת נקודות הזמן . להשיג שתי תמונות רצופות של L. משאבה ה, ביטוי חלבון פלורסנט של הריבית.
- כוונן את הניגודיות של התמונה עד שהתאים גלויים בבירור. בצע את איור 6A ואת התיאורים הבאים כדי למקם שלושמסיכות שונות. השתמש בכלי האזור כדי למקם 0.25 x 0.25 יקרומטר כיכר באמצע לפחות 400 תאים.
- השתמש בלחצן אזור למסיכה כדי ליצור מסיכה (מסיכה 1) של ריבועי הריבית. צרו מסכה ריקה חדשה (מסכה 2) והשתמשו בכלי הפיקסל או בכלי המצולע כדי לסמן את כל שטח התא של לפחות 25 תאים אקראיים, אשר ישמשו לחישוב הלבנת זריחה. צרו מסיכה ריקה חדשה (מסיכה 3) והשתמשו בכלי המברשת הגדול כדי לסמן אזורים בין התאים, שישמשו לחיסור רקע.
- תחת מסיכת סטטיסטיקה וטווח מסיכות, בחר אובייקט עבור מסיכה 1 ומסיכה 2. לאחר מכן, תחת תכונות ועוצמה, בחר עוצמה ממוצע ולייצא את הנתונים של שתי המסיכות. למסיכה 3, יצא את העוצמה הממוצע של המסיכה כולה.
- חישוב השינוי בעוצמת הקרינה הפלואורסצנטית עבור כל אובייקט במסיכה 1 באמצעות הנוסחאות הבאות:

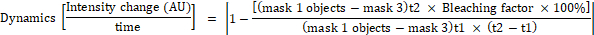
כאשר מסכה 1 הוא 0.25 יקרומטר × 0.25 יקרומטר ריבוע ממוקם במרכז התא, מסכה 2 מכסה את התא כולו, מסכה 3 הוא הרקע בין תאים, t1 הוא העוצמה הממוצע בנקודת הזמן הראשונה, ו t2 הוא העוצמה הממוצע בנקודת הזמן השנייה.
5. זיהוי דחיסות המסה של sfGFP עם ההקפאה
- הכנה לדוגמה, איסוף נתונים ושחזור
- לגדל טלאי כבד של L. משאבה האמההילה ביטוי פלואור מתויג נקודה/Icm חלבון על cye-אגר-סטרפטומיצין צלחות עבור 48 שעות ב 37 ° c. השהה מחדש את התאים ב-ddH2O כדי OD600 ~ 0.7. הוסף 5 μL של חלקיקי זהב (BSA מעקב, 10 ננומטר) עד 20 μL של השעיית התא.
- פיפטה 5 μl של התערובת של התא אל רשת הזוהר הטרייה משוחררת holey פחמן (R 2/1 על Cu 200 רשת שינוי) ולתת לעמוד 1 דקות למחוק עם נייר סינון להקפיא את אתאן נוזלי באמצעות מנגנון כבידה מונחה בבוכנה כמתואר בעבר14,15.
- התמונה של היצורים קפואים עם מיקרוסקופ הילוכים 300 kV מצויד באקדח פליטת שדה, מסנן אנרגיה, צלחת שלב וולטה, ומכשיר זיהוי ישיר. לאסוף סדרת הטיה חד הציר ב 26, 000x ו-42, 000x המאגרים, אשר התוצאה בגדלים פיקסל ברמת הדגימה של 5.4 Å/פיקסל או 3.4 Å/פיקסל, בהתאמה.
- השתמש בחבילת טומוגרפית כדי לאסוף ערימות תמונה ב ~ 0 יקרומטר defocus, עם מגוון של זוויות הטיה בין-60 ° + 60 ° עם התוספת צעד 3 ° ומינון המצטבר של ~ 60 e-/Å2,16. יישר את תמונות הסרט משבר המינון בכל מחסנית באמצעות MotionCor217. להרכיב ערימות להיסחף מתוקן באמצעות TOMOAUTO14.
- יישר ערימות שתוקנו על-ידי הסחף על-ידי יישור התלוי בסמן IMOD18. לשחזר tomograms עם שיטה SIRT19 עבור segmentations וניתוח תמונה ישירה ואת שיטת wbp20.
- Subtomogram ניתוח של הספגfp fusions דגימות
- השתמש בחבילת הטוקסוגרפיות (0.9.9.3) לניתוח subtomogram14,21,22.
הערה: היישור מתקדם בצורה המיטבית, כאשר כל איטראציה מורכבת משלושה חלקים בהם נוצרות הפניות ומסיכות סיווג, subtomograms מיושרות ומסווגים, וממוצעים של מחלקה מיושרים זה לזה. - השתמש ב-4 x 4 x 4 שכבות subtomograms עבור יישור ראשוני. מיזוג חלקיקים השייכים לממוצעים מחלקה ומציגים צפיפות אלקטרונים המתאימה ל-sfGFP. לאחר מיון חלקיקים עם sfGFP fusions, להשתמש 2 x 2 x 2 בנימין subtomograms עבור יישור ממוקד של אזור של עניין (כמו נקודה/Icm cytoplasmic ATPase מורכב) כדי להשיג מבנה ברזולוציה גבוהה.
- השתמש בחבילת הטוקסוגרפיות (0.9.9.3) לניתוח subtomogram14,21,22.
תוצאות
שילוב מחדש של הומוולוגי עם מבחר כפול בשני שלבים שימש לבניית ההכנסה המוגדרת של sfGFP. , בצעד הראשון ההזדווגות triparental ורית בוצעה, שם pRK600 מצונת החומר הנפץ של החיידק הקולי של e. COLI MT616 היה מגוייס החיידק e. coli מאמץ עם וקטור ההתאבדות pSR47S המכיל את הגן sfgfp מוקף על ידי שני אזורים הומולוגיים, מקור ?...
Discussion
להבהיר את הפונקציות של מערכות הפרשה חיידקית הוא המפתח להבנה מלאה של אינטראקציות מארח הפתוגן. מערכות הפרשה הן מכונות מורכבות שיכולות להזריק חלבונים לתוך תאי מחשב מארחים, ובמקרים מסוימים מקדמים את הקמתה של נישה תת-תאית התומכת בשכפול חיידקי. השיטה הנ ל מספקת כלים חדשים חשובים ללימוד נקודה/Icm...
Disclosures
. למחברים אין מה לגלות
Acknowledgements
הבירה ו-C.R.R. תמכו ב-NIH (R37AI041699 וR21AI130671). D.P., B.H. ו-J. L תמכו במוסדות הבריאות הלאומיים (R01AI087946 וR01GM107629).
Materials
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold particles | Aurion | 25486 | |
| 100x Plan Apo objective (1.4 NA) | Nikon | ||
| ACES | Sigma-Aldrich | A9758 | |
| Activated charcoal | Sigma-Aldrich | C5510 | |
| Agaroze GPG/LMP, low melt | American bioanalytical | AB00981 | |
| Bacto dehydrated agar | BD | 214010 | |
| CoolSNAP EZ 20 MHz digital monochrome camera | Photometrics | ||
| Gene Frame, 1.7x2.8 cm, 125 µL | Fisher Scientific | AB-0578 | |
| Holey Carbon grid R 2/1 Cu 200 mesh | Quantifoil | Q225-CR1 | |
| Iron(III) nitrate nonahydrate | Sigma-Aldrich | 216828 | |
| K2 Summit camera for cryo-EM | GATAN | ||
| L-Cysteine | Sigma-Aldrich | C7352 | |
| Microscope cover slides 22x22 mm | Fisher Scientific | 12-542B | |
| Microscope cover slides 24x50 mm | Fisher Scientific | 12-545K | |
| Microscope slides 25x75x1 mm | Globe Scientific | 1380 | |
| SlideBook 6.0 | Intelligent Imaging Innovations | ||
| Spectra X light engine | Lumencor | ||
| Taq 2X Master Mix | New England BioLabs | M0270 | |
| Titan Krios | Thermo Fisher Scientific | ||
| Yeast Extract | BD | 212750 |
References
- Franco, I. S., Shuman, H. A., Charpentier, X. The perplexing functions and surprising origins of Legionella pneumophila type IV secretion effectors. Cellular Microbiology. 11, 1435-1443 (2009).
- Burstein, D., et al. Genome-scale identification of Legionella pneumophila effectors using a machine learning approach. PLOS Pathogens. 5, 1000508 (2009).
- Ninio, S., Roy, C. R. Effector proteins translocated by Legionella pneumophila: strength in numbers. Trends in Microbiology. 15, 372-380 (2007).
- Vogel, J. P., Andrews, H. L., Wong, S. K., Isberg, R. R. Conjugative transfer by the virulence system of Legionella pneumophila. Science. 279, 873-876 (1998).
- Isberg, R. R., O'Connor, T. J., Heidtman, M. The Legionella pneumophila replication vacuole: making a cosy niche inside host cells. Nature Reviews Microbiology. 7, 13-24 (2009).
- Roy, C. R., Berger, K. H., Isberg, R. R. Legionella pneumophila DotA protein is required for early phagosome trafficking decisions that occur within min of bacterial uptake. Molecular Microbiology. 28, 663-674 (1998).
- Archer, K. A., Roy, C. R. MyD88-Dependent Responses Involving Toll-Like Receptor 2 Are Important for Protection and Clearance of Legionella pneumophila in a Mouse Model of Legionnaires' Disease. Infection and Immunity. 74, 3325-3333 (2006).
- Nagai, H., Kubori, T. Type IVB Secretion Systems of Legionella and Other Gram-Negative Bacteria. Frontiers in Microbiology. 2, 136 (2011).
- Kubori, T., Nagai, H. The Type IVB secretion system: an enigmatic chimera. Current Opinion in Microbiology. 29, 22-29 (2016).
- Chetrit, D., Hu, B., Christie, P. J., Roy, C. R., Liu, J. A unique cytoplasmic ATPase complex defines the Legionella pneumophila type IV secretion channel. Nature Microbiology. 3, 678-686 (2018).
- Merriam, J. J., Mathur, R., Maxfield-Boumil, R., Isberg, R. R. Analysis of the Legionella pneumophila fliI gene: intracellular growth of a defined mutant defective for flagellum biosynthesis. Infection and Immunity. 65, 2497-2501 (1997).
- Feeley, J. C., et al. Charcoal-yeast extract agar: primary isolation medium for Legionella pneumophila. Journal of Clinical Microbiology. 10, 437-441 (1979).
- Andrews, H. L., Vogel, J. P., Isberg, R. R. Identification of linked Legionella pneumophila genes essential for intracellular growth and evasion of the endocytic pathway. Infection and Immunity. 66, 950-958 (1998).
- Morado, D. R., Hu, B., Liu, J. Using Tomoauto: A Protocol for High-throughput Automated Cryo-electron Tomography. Journal of Visualized Experiments. (107), e53608 (2016).
- Hu, B., Lara-Tejero, M., Kong, Q., Galan, J. E., Liu, J. In Situ Molecular Architecture of the Salmonella Type III Secretion Machine. Cell. 168, 1065-1074 (2017).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152, 36-51 (2005).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14, 331-332 (2017).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Gilbert, P. Iterative methods for the three-dimensional reconstruction of an object from projections. Journal of Theoretical Biology. 36, 105-117 (1972).
- Radermacher, M. Weighted Back-projection Methods. Electron Tomography. , 245-273 (2007).
- Winkler, H., et al. Tomographic subvolume alignment and subvolume classification applied to myosin V and SIV envelope spikes. Journal of Structural Biology. 165, 64-77 (2009).
- Winkler, H., Taylor, K. A. Accurate marker-free alignment with simultaneous geometry determination and reconstruction of tilt series in electron tomography. Ultramicroscopy. 106, 240-254 (2006).
- Prevost, M. S., Waksman, G. X-ray crystal structures of the type IVb secretion system DotB ATPases. Protein Science. 27, 1464-1475 (2018).
- Miklos, G. L., Rubin, G. M. The role of the genome project in determining gene function: insights from model organisms. Cell. 86, 521-529 (1996).
- Reyrat, J. M., Pelicic, V., Gicquel, B., Rappuoli, R. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis. Infection and Immunity. 66, 4011-4017 (1998).
- Yu, J. Single-Molecule Studies in Live Cells. Annual Review of Physical Chemistry. 67, 565-585 (2016).
- Stewart, P. L. Cryo-electron microscopy and cryo-electron tomography of nanoparticles. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 9, (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved