Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Применение Live Cell Imaging и крио-электронной томографии для устранения пространственно-временной особенностей системы Legionella pneumophila Dot/Icm Secretion
В этой статье
Резюме
Изображение бактериальных клеток является новым подходом биологии систем, ориентированных на определение статических и динамических процессов, которые диктуют функцию крупных макромолекулярных машин. Здесь для изучения архитектуры и функций системы секреции системы секреции legionella pneumophila IV типа используется интеграция количественной визуализации живых клеток и криоэлектронной томографии.
Аннотация
Система секреции Dot/Icm пневмофилы Legionella является сложной системой секреции IV типа (T4SS), которая локализует на бактериальном полюсе и опосредует доставку белков и субстратов ДНК в клетки-мишени, процесс, обычно требующий прямого контакта между клетками и клетками. Недавно мы решили структуру аппарата Dot/Icm с помощью криоэлектронной томографии (крио-ET) и показали, что он образует клеточный конвертографический канал, который соединяется с цитоплазмическим комплексом. Применение двух взаимодополняющих подходов, сохраняющих родную структуру образца, флуоресцентную микроскопию в живых клетках и крио-ET, позволяет на месте визуализировать белки и усвоение стоихиометрии и сроки производства каждого компонента машины по сравнению с другими подразделениями Dot/Icm. Для изучения требований к полярному позиционированию и характеристики динамических особенностей, связанных с биогенезом машины T4SS, мы сплавили ген, кодируя суперфолдер зеленый флуоресцентный белок, к генам Dot/Icm ATPase на их родных позициях на хромосоме. Следующий метод интегрирует количественную флуоресценцию микроскопии живых клеток и крио-ET для количественной оценки полярной локализации, динамики и структуры этих белков в нетронутых бактериальных клетках. Применение этих подходов для изучения Legionella пневмофилы T4SS полезно для характеристики функции Dot / Icm системы и могут быть адаптированы для изучения широкого спектра бактериальных патогенов, которые используют T4SS или других типов бактериальных комплексов секреции.
Введение
Легионелла пневмофила (L. pneumophila), этиологическое вещество болезни легионеров, обитает пресноводных резервуаров, где бактерии размножаются путем заражения и репликации в водной свободного плавания простейшие. L. пневмофила вызывает вспышки болезней у людей при вдыхании аэрозольных бактерий из источников питьевой воды. В инфицированных клетках, подрыв путей хозяина позволяет L. пневмофила отложить эндоцитарное созревание вакуол, в котором он проживает и содействовать биогенеза клеточного отсека, который поддерживает бактериальную репликацию. Этот процесс обусловлен специализированной бактериальной системой секреции IVB (T4BSS), известной как Dot/Icm, и его репертуар из более чем 300 "эффекторов" белков, которые трансвируются в цитозол хозяина во время инфекции для облегчения манипуляции клеточными функциями1,2,3,4,5. Мутанты, не имеющие функционального аппарата Dot/Icm, не могут доставить эффекторов в цитозол хозяина, неполноценны для внутриклеточной репликации и являются avirulent в животных моделях болезни6,7.
Многие бактериальные виды разработали чрезвычайно сложные и динамические многокомпонентные машины, которые необходимы для инфекционных процессов. Другие T4BSS, как Dot / Icm системы также имеют важное значение для внутриклеточной репликации бактериальных патогенов, таких как Coxiella burnetii и Rickettsiella grylli. Хотя T4BSS эволюционно связаны с прототипами типа IVA систем, которые посредничают передачи ДНК и может доставить ограниченный репертуар эффекторных белков, Dot / Icm система имеет почти в два раза больше компонентов машины и обеспечивает широкий спектр эффекторов. Предположительно, это расширение числа компонентов позволило Dot / Icm аппарат для размещения и интеграции новых эффекторов легко8,9.
Недавно мы использовали криоэлектронную томографию (крио-ET) для решения структуры дот-icm аппарата на месте и показали, что он образует клеточный конвертографический канал, который соединяется с цитоплазмическим комплексом. Дальнейший анализ показал, что цитосолико ATPase DotB ассоциируется с системой Dot/Icm на клеточном полюсе L. pneumophila через взаимодействие с цитосолическим ATPase DotO. Мы обнаружили, что DotB отображает цитозоликовое движение в большинстве бактериальных клеток, указывая, что это ATPase присутствует в динамической цитосолильной популяции, но также ассоциируется с полярными комплексами Dot/Icm. Кроме того, DotO образует гексамерную сборку димеров DotO, связанных с внутренним мембранным комплексом, и гексамер DotB присоединяется к основанию этого цитоплазмического комплекса. Сборка энергетического комплекса DotB-DotO создает цитоплазмический канал, который направляет транслокацию субстратов через T4SS(рисунок 1)10.
Несмотря на эти последние достижения, мало что известно о том, как работает система Dot/Icm и как каждый белок собирается, чтобы сформировать активный аппарат8. Раскрытие регулятивной схемы Dot/Icm T4SS имеет основополагающее значение для понимания молекулярных механизмов взаимодействия хоста и патогена. Поэтому мы обсуждаем, как использовать живую клеточную микроскопию и крио-ET для обнаружения и характеристики основных компонентов системы L. pneumophila Dot/Icm, которые помечены супер-папками GFP (sfGFP). Используя количественную флуоресценционную микроскопию, полярная локализация DotB будет определяться в фоновом режиме дикого типа или при удалении системы типа IV. Микроскопия замедленного времени будет использоваться для количественной оценки различий в локализации и динамике между цитосоликами AtPases Dot/Icm.
Комбинированное применение двух взаимодополняющих подходов, таких как живая визуализация и крио-ET, обеспечивает преимущество по сравнению с другими системами in vitro. Оба метода выполняются в нетронутых клетках и сохраняют естественную среду T4BSS, тем самым минимизируя нарушение родной структуры во время подготовки образца. Поскольку переэкспрессия белков может ухудшить стоихиометрию секреционного аппарата, слияние sfGFP возвращается через аллельский обмен на хромосому Legionella, так что каждый синтез закодирован в единственном экземпляре и выражение определяется эндогенным промоутером. Визуализация хромосомно закодированных слияний позволяет количественно определить точный уровень белка, выраженного в определенный момент времени. Cryo-ET также имеет много преимуществ для определения структуры систем секреции. Наиболее заметным преимуществом является то, что образцы крио-ET состоят из замороженных нетронутых клеток, которые сохраняют родные комплексы в контексте архитектуры бактериальных клеток. Следовательно, крио-ET может быть предпочтительнее биохимических подходов к очистке, которые извлекают мембранные комплексы и могут вывести периферийные белки из основного аппарата или изменять общую структуру. Кроме того, пометка белка интерес с громоздким белком, таким как sfGFP добавляет массу, которая обнаруживается крио-ET и может помочь с картированием различных подкомплексов dot/ Icm аппарата на структуру, полученную крио-ET.
Этот подход является мощным инструментом для раскрытия структурной информации о многомолекулярных комплексах, которые собираются в мембране бактериальных клеток. Интерпретация структур, разъясняемых с помощью этих методов, поможет полевому полю понять, как функционируют компоненты T4BSS, почему так много компонентов требуется для работы, как компоненты взаимодействуют в рамках большего комплекса и какие функции эти функции подсборки выполняют.
протокол
ПРИМЕЧАНИЕ: Все процедуры, связанные с ростом, манипуляцией и визуализацией Л. пневмофилы, должны выполняться в лаборатории уровня биологической безопасности 2 в соответствии с местными руководящими принципами.
1. Вставка sfGFP в L. pneumophila Хромосомы использованием аллельной exchange и двойной стратегии отбора(Рисунок 2, Рисунок 3)
- Клон в вектор замены генов pSR47S11 следующая последовательность: 1000 bp вверх по течению сайта интерес, то sfGFP последовательности, то 1000 bp вниз по течению от сайта интерес (Рисунок 2). Последовательность sfGFP должна быть помещена в кадр к N-терминусу или C-конецным концам с связующим звеном, который содержит от четырех до восьми аминокислот. Преобразуйте полученный вектор в E. coli DH5'pir. Позже, полоса L. пневмофила (получатель) для отдельных колоний на древесно-дрожжевой экстракт (CYE) агар12, содержащий 100 мкг /мл стрептомицина и расти в течение 5 дней при 37 КС (Рисунок 3).
- Полоса Л. пневмофилы на CYE-агар-стрептомицин и расти в течение 2 дней при 37 кС (тяжелый патч)10. Полоса E. coli DH5' преобразована с pRK600 помощник плазмид (помощник)13 на АгарLB, содержащий 25 мкг /мЛ хлорамфеникол. Полоса E. coli DH5 'pir (донор) на LB агар, содержащий 50 мкг/мл канамицин.
- Выполните трехродительское спаривание: инкубировать колонию помощника, колонию донора, а реципиента путем наложения пятен трех штаммов на агарную пластину CYE без выбора и инкубации на 4-8 ч при 37 градусах Цельсия. В качестве отрицательного контроля инкубировать помощника реципиента штамма смеси и донора и реципиента штамм амекс и в течение тех же периодов времени.
- Повторяйте реакции спаривания в 500 мкг2O. Плита 20 Л и 50 л реакций на агаре CYE, содержащем 100 мкг/мл стрептомицина и 10 мкг/мл канамицина, и расти в течение 5 дней при 37 градусах Цельсия. Полоса четыре из полученных клонов на агаре CYE, содержащем 100 мкг/мл стрептомицина, и расти в течение 5 дней при 37 градусах Цельсия.
- Полоса 16 клонов на агаре CYE, содержащая 5% сахарозы и 100 мкг/мл стрептомицина и расти в течение 5 дней при 37 градусах Цельсия. Затем, полоса 32 из этих клонов на CYE агар, содержащий 100 мкг /мл стрептомицина и на CYE агар, содержащий 100 мкг/мл стрептомицин и 10 мкг /мл канамицин и расти в течение 5 дней при 37 градусов по Цельсию.
2. Изоляция клонов, которые интегрированы sfGFP в Хромосоме L. pneumophila
- Полоса клонов, которые были чувствительны к канамицин на CYE-агар-стрептомицин пластин и подтвердить вставку sfGFP в хромосому с колонией PCR. Используйте грунтовки, которые дополняют ген sfGFP и хромосомную область, интересуемую для усиления соединения вставки.
- Смешайте 0,5 л каждого из 10 растворов праймеров и одну колонию до окончательного объема в 12,5 л и денатурацию в течение 10 мин при 95 градусах По Цельсия. Охладить на льду в течение 10 минут, добавить 12,5 л 2x PcR мастер-микс раствор, и выполнить анализ ПЦР.
- Выращивайте тяжелые участки изолированных колоний на пластинах CYE-agar-streptomycin в течение 2 дней при 37 градусах Цельсия. Изучите уровень экспрессии и стабильность слияний sfGFP путем иммуноблоттинга с анти-GFP антитела.
3. Live Cell Imaging L. pneumophila с флуоресцентно tagged Dot / Icm компонентов
- Приготовление агарозных прокладок
- Сделайте около 30 мл раствора агарозы в воде на 1%. Микроволновка в стеклянной колбе около 90 с, закрученного время от времени, пока агароза полностью растворяется.
- Поместите два 22 х 22 х 0,15 мм3 стеклянные слайды на краю 25 х 75 х 1,1 мм3 стеклянные слайд, один на вершине другого. Стек еще два 22 х 22 х 0,15 мм3 стеклянные слайды на другой край.
- Пипетка около 1 мл расплавленной агарозы в центр слайдмежду двумя верхними стеклянными горками, а затем поместите еще 25 х 75 х 1,1 мм3 слайд на вершине расплавленной агарозы. Старайтесь избегать образования пузырьков воздуха. Охладите горки при 4 градусах по Цельсию в течение 15 минут.
- Используя скальпель или лезвие бритвы, аккуратно разрежьте площадку на мелкие квадраты, 5 х 5 мм2. Исправьте двусторонний клей 17 x 28 x 0.25 мм3 кадра на 25 x 75 x 1,1 мм3 стеклянных слайда и поместите несколько колодок на слайд.
- Приобретение изображения
ПРИМЕЧАНИЕ: Следующие шаги описаны для микроскопа, который находится под контролем SlideBook 6.0 и оснащен твердотельными иллюминаторами, монохромной камерой CCD и 100-x объективом (1,4 численного отверстия). При необходимости используйте альтернативные микроскопии с соответствующими аппаратными и программными конфигурациями, которые могут быть настроены в соответствии с настройками протокола.- Растворите тяжелый участок L. pneumophila в 1 мл ddH2O, вихрь и труба 2-3 л разбавления на колодки. Поместите 50 х 24 х 0,15 мм3 крышка мягко над клей кадра.
- В окне захвата отрегулируйте ND до 180. Отрегулируйте связующие до 2х2 и используйте канал 488 нм, чтобы выставить образец между 500-1000 мс. Проверить специфику сигнала флуоресценции путем визуализации немаркированной Пневмофилы С теми же параметрами(рисунок 4).
4. Количественная оценка полярной локализации и динамики компонентов Dot/Icm
ПРИМЕЧАНИЕ: Следующие шаги предназначены для изображений с 0,129 мкм на пиксель, которые были приобретены с 2 х 2 binning.
- Количественная оценка полярности для белков синтеза sfGFP(Рисунок 5)
- Отрегулируйте контраст изображения таким образом, чтобы бактерии были хорошо видны. Используйте региональный инструмент, чтобы поместить прямоугольник 0,25 х 1,3 мкм2, начиная с полюса и расширяясь в цитоплазму. Прямоугольник должен оставаться именно в пределах бактериальных границ.
- Отметьте не менее 200 бактерий и используйте кнопку «Регион для маски» для создания масок для интересных регионов. Под маска Статистика и маска сфера,выбрать объект. Затем, под особенности и интенсивность, знак Средняя интенсивность и дисперсия.
- Экспортировать данные и вычислять баллы полярности каждой бактерии как соотношение между дисперсией и средней интенсивностью.
- Для высокопроизводительных приложений используйте фазовую цель и соответствующую установку конденсатора для получения изображений с фазой и 488 нм каналами. Убедитесь в том, чтобы выбрать поля зрения, где бактерии полностью разделены.
- Отрегулируйте контраст фазового канала изображения до уровня, на котором хорошо видны бактерии. Откройте изображение двойного канала, запустите окно Create Segment Mask и измените канал на фазу.
- Отрегулируйте соответствующий порог и удалите небольшие объекты с помощью кнопки «Определить объекты». Под refine Mask выберите Удалить края объектов, а затем отдельные маски бактерий, которые примыкают друг к другу.
- Рассчитайте баллы полярности сигнала в канале 488 для каждой ячейки, как это было ранее описано в шагах 4.1.2-4.1.3.
- Количественная оценка динамики для белков синтеза sfGFP(рисунок 6)
ПРИМЕЧАНИЕ: Следуйте инструкциям в разделе 3, чтобы подготовить образец для приобретения изображения. Динамика определяется как изменения интенсивности с течением времени и следующие шаги предназначены для коротких периодов изображения (т.е. несколько минут). Добавьте к площадке соответствующие добавки, если требуется более длительные периоды визуализации.- В окне захвата изображения отметьте Timelapse,введите время интервалов в интервале коробки и введите 2 в поле q of Time Points. Приобретите два последовательных изображения L. pneumophila, выражающих флуоресцентный белок интереса.
- Отрегулируйте контраст изображения до тех пор, пока ячейки не будут хорошо видны. Следуйте рисунку 6А и описаниям ниже, чтобы разместить три различных масок. Используйте региональный инструмент, чтобы поместить квадрат 0,25 х 0,25 мкм в середине по крайней мере 400 ячеек.
- Используйте кнопку «Маска региона» для создания маски (маска 1) интересуемых квадратов. Создайте новую пустую маску (маску 2) и используйте инструмент пикселя или инструмент полигона для обозначения всей области ячейки, состоящей не менее 25 случайных ячеек, которые будут использоваться для расчета отбеливания флуоресценции. Создайте новую пустую маску (маска 3) и используйте большой инструмент кисти для обозначения областей между ячейками, которые будут использоваться для фонового вычитания.
- Под mask Statistics и Mask Scopeвыберите объект для маски 1 и маски 2. Затем, в соответствии с функциями и интенсивностью,выберите средней интенсивности и экспортировать данные двух масок. Для маски 3, экспортировать средняя интенсивность всей маски.
- Рассчитайте изменение интенсивности флуоресценции для каждого объекта в маске 1 с помощью следующих формул:

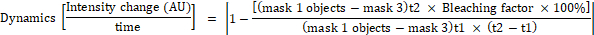
где маска 1 представляет собой 0,25 мкм и 0,25 мкм квадрат, размещенный в центре ячейки, маска 2 охватывает всю ячейку, маска 3 является фоном между клетками, t1 является средней интенсивностью в первой точке времени, и t2 - это средняя интенсивность во второй точке времени.
5. Обнаружение плотности массы sfGFP с помощью Cryo-ET
- Подготовка образцов, сбор и реконструкция
- Выращивайте тяжелый пласт L. pneumophila, выражающий флуоресцентно помеченный белок Dot/Icm на пластинах CYE-agar-streptomycin в течение 48 часов при 37 градусах Цельсия. Приостановите работу ячеек в ddH2O до OD600 0,7. Добавьте 5 кЛ частиц коллоидного золота (BSA Tracer, 10 нм) к 20 злитро-л клеточной подвески.
- Пипетка 5 qL клеточной смеси на свежесветосвеченный дырявый углеродной сетки (R 2/1 на Cu 200 сетки) и пусть стоять в течение 1 мин. Пятно с фильтровальной бумагой и заморозить в жидком этана с использованием гравитационного привода плунжем аппарата, как описано ранее14,15.
- Изображение замороженных гидратированных образцов с электронным микроскопом передачи 300 кВ, оснащенным полевой эмиссионной пушкой, энергетическим фильтром, фазовой пластиной Вольта и устройством прямого обнаружения. Соберите одноосную серию наклонов на уровне 26 000х и 42 000х, что приводит к размеру пикселей на уровне 5,4 евро/пикселей или 3,4 евро/пикселя, соответственно.
- Используйте томографический пакет SerialEM для сбора стеков изображений на 0 мкм дефокусировки, с диапазоном наклона углы между -60 "и "60" с шагом 3 "шаг шагом и накопительной дозы 60 е-/2,16. Выравнивание дозы фракционированных изображений фильма в каждом стеке с помощью MotionCor217. Соберите дрифтерные стеки с помощью TOMOAUTO14.
- Выравнивание стеков, исправленных дрейфом,по выравниванию 18-опове-маркеров IMOD. Реконструкция томограмм с помощью метода SIRT19 для сегментации и прямого анализа изображений и метода WBP20.
- Субтомограмма на анализ образцов слияний sfGFP
- Используйте томографический пакет I3 (0.9.9.3) для субтомограммы анализа14,21,22.
ПРИМЕЧАНИЕ: Выравнивание продолжается итеративно, с каждой итерацией, состоящей из трех частей, в которых ссылки и классификационные маски генерируются, субтомограммы выровнены и классифицированы, а средние классы выровнены друг с другом. - Используйте 4 х 4 х 4 binned субтомограммы для первоначального выравнивания. Слияние частиц, принадлежащих к средним классам, и показывает плотность электронов, соответствующую sfGFP. После сортировки частиц с синтезами sfGFP, используйте 2 х 2 х 2 binned субтомограммы для целенаправленного выравнивания региона, интересующего (например, Dot / Icm цитоплазматический комплекс ATPase) для получения структуры с высоким разрешением.
- Используйте томографический пакет I3 (0.9.9.3) для субтомограммы анализа14,21,22.
Результаты
Для построения определенной вставки sfGFP использовалась гомологовая рекомбинация с двойным отбором в два этапа. На первом этапе было проведено трехродительское спаривание, где pRK600 конъюгативная плазмида (инкп плазмида) из штамма E. coli helper MT616 была мобилизована донору штамма ...
Обсуждение
Выяснение функций бактериальных систем секреции является ключом к полному пониманию взаимодействия хозяина-патогена. Системы секретии являются сложными машинами, которые могут вводить эффекторы белков в клетки-хозяина, а в некоторых случаях способствовать созданию субклеточной ниш...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
D.C. и C.R.R. были поддержаны NIH (R37AI041699 и R21AI130671). D.P., B.H. и J.L. были поддержаны Национальными институтами здоровья (R01AI087946 и R01GM107629).
Материалы
| Name | Company | Catalog Number | Comments |
| 10 nm colloidal gold particles | Aurion | 25486 | |
| 100x Plan Apo objective (1.4 NA) | Nikon | ||
| ACES | Sigma-Aldrich | A9758 | |
| Activated charcoal | Sigma-Aldrich | C5510 | |
| Agaroze GPG/LMP, low melt | American bioanalytical | AB00981 | |
| Bacto dehydrated agar | BD | 214010 | |
| CoolSNAP EZ 20 MHz digital monochrome camera | Photometrics | ||
| Gene Frame, 1.7x2.8 cm, 125 µL | Fisher Scientific | AB-0578 | |
| Holey Carbon grid R 2/1 Cu 200 mesh | Quantifoil | Q225-CR1 | |
| Iron(III) nitrate nonahydrate | Sigma-Aldrich | 216828 | |
| K2 Summit camera for cryo-EM | GATAN | ||
| L-Cysteine | Sigma-Aldrich | C7352 | |
| Microscope cover slides 22x22 mm | Fisher Scientific | 12-542B | |
| Microscope cover slides 24x50 mm | Fisher Scientific | 12-545K | |
| Microscope slides 25x75x1 mm | Globe Scientific | 1380 | |
| SlideBook 6.0 | Intelligent Imaging Innovations | ||
| Spectra X light engine | Lumencor | ||
| Taq 2X Master Mix | New England BioLabs | M0270 | |
| Titan Krios | Thermo Fisher Scientific | ||
| Yeast Extract | BD | 212750 |
Ссылки
- Franco, I. S., Shuman, H. A., Charpentier, X. The perplexing functions and surprising origins of Legionella pneumophila type IV secretion effectors. Cellular Microbiology. 11, 1435-1443 (2009).
- Burstein, D., et al. Genome-scale identification of Legionella pneumophila effectors using a machine learning approach. PLOS Pathogens. 5, 1000508 (2009).
- Ninio, S., Roy, C. R. Effector proteins translocated by Legionella pneumophila: strength in numbers. Trends in Microbiology. 15, 372-380 (2007).
- Vogel, J. P., Andrews, H. L., Wong, S. K., Isberg, R. R. Conjugative transfer by the virulence system of Legionella pneumophila. Science. 279, 873-876 (1998).
- Isberg, R. R., O'Connor, T. J., Heidtman, M. The Legionella pneumophila replication vacuole: making a cosy niche inside host cells. Nature Reviews Microbiology. 7, 13-24 (2009).
- Roy, C. R., Berger, K. H., Isberg, R. R. Legionella pneumophila DotA protein is required for early phagosome trafficking decisions that occur within min of bacterial uptake. Molecular Microbiology. 28, 663-674 (1998).
- Archer, K. A., Roy, C. R. MyD88-Dependent Responses Involving Toll-Like Receptor 2 Are Important for Protection and Clearance of Legionella pneumophila in a Mouse Model of Legionnaires' Disease. Infection and Immunity. 74, 3325-3333 (2006).
- Nagai, H., Kubori, T. Type IVB Secretion Systems of Legionella and Other Gram-Negative Bacteria. Frontiers in Microbiology. 2, 136 (2011).
- Kubori, T., Nagai, H. The Type IVB secretion system: an enigmatic chimera. Current Opinion in Microbiology. 29, 22-29 (2016).
- Chetrit, D., Hu, B., Christie, P. J., Roy, C. R., Liu, J. A unique cytoplasmic ATPase complex defines the Legionella pneumophila type IV secretion channel. Nature Microbiology. 3, 678-686 (2018).
- Merriam, J. J., Mathur, R., Maxfield-Boumil, R., Isberg, R. R. Analysis of the Legionella pneumophila fliI gene: intracellular growth of a defined mutant defective for flagellum biosynthesis. Infection and Immunity. 65, 2497-2501 (1997).
- Feeley, J. C., et al. Charcoal-yeast extract agar: primary isolation medium for Legionella pneumophila. Journal of Clinical Microbiology. 10, 437-441 (1979).
- Andrews, H. L., Vogel, J. P., Isberg, R. R. Identification of linked Legionella pneumophila genes essential for intracellular growth and evasion of the endocytic pathway. Infection and Immunity. 66, 950-958 (1998).
- Morado, D. R., Hu, B., Liu, J. Using Tomoauto: A Protocol for High-throughput Automated Cryo-electron Tomography. Journal of Visualized Experiments. (107), e53608 (2016).
- Hu, B., Lara-Tejero, M., Kong, Q., Galan, J. E., Liu, J. In Situ Molecular Architecture of the Salmonella Type III Secretion Machine. Cell. 168, 1065-1074 (2017).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152, 36-51 (2005).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14, 331-332 (2017).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Gilbert, P. Iterative methods for the three-dimensional reconstruction of an object from projections. Journal of Theoretical Biology. 36, 105-117 (1972).
- Radermacher, M. Weighted Back-projection Methods. Electron Tomography. , 245-273 (2007).
- Winkler, H., et al. Tomographic subvolume alignment and subvolume classification applied to myosin V and SIV envelope spikes. Journal of Structural Biology. 165, 64-77 (2009).
- Winkler, H., Taylor, K. A. Accurate marker-free alignment with simultaneous geometry determination and reconstruction of tilt series in electron tomography. Ultramicroscopy. 106, 240-254 (2006).
- Prevost, M. S., Waksman, G. X-ray crystal structures of the type IVb secretion system DotB ATPases. Protein Science. 27, 1464-1475 (2018).
- Miklos, G. L., Rubin, G. M. The role of the genome project in determining gene function: insights from model organisms. Cell. 86, 521-529 (1996).
- Reyrat, J. M., Pelicic, V., Gicquel, B., Rappuoli, R. Counterselectable markers: untapped tools for bacterial genetics and pathogenesis. Infection and Immunity. 66, 4011-4017 (1998).
- Yu, J. Single-Molecule Studies in Live Cells. Annual Review of Physical Chemistry. 67, 565-585 (2016).
- Stewart, P. L. Cryo-electron microscopy and cryo-electron tomography of nanoparticles. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 9, (2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены