Method Article
تحليل الجينوم على نطاق واسع من ميثيل الحمض النووي في سرطان الجهاز الهضمي
In This Article
Summary
هنا، نحن وصف إجراء لتحليل الجينوم على نطاق واسع من ميثيل الحمض النووي في سرطانات الجهاز الهضمي. هذا الإجراء هو ذات الصلة بالدراسات التي تحقق في العلاقات بين أنماط الميثيل من الجينات والعوامل التي تسهم في التسرطن في سرطان الجهاز الهضمي.
Abstract
إنّ وميض الحمض النووي هو تغيير غير مُضَمّي مهمّ ذو معنى بيولوجيّاً، وهو محور متكرر لأبحاث السرطان. إنّ ميثيل الحمض النووي على نطاق الجينوم هو إجراء مفيد لتوفير تحليل دقيق لحالة المثيل في أمراض الجهاز الهضمي (GI) الخبيثة. وبالنظر إلى الاستخدامات المتعددة المحتملة للمعالجة في تحليل ميثيل الحمض النووي، فإن الأطباء الممارسين وغيرهم من الأطباء الجدد على دراسات الميثيلة بالحمض النووي يحتاجون إلى أن يكونوا قادرين على فهم كيفية إجراء هذه التحليلات على نطاق الجينوم خطوة بخطوة. والهدف من هذا البروتوكول هو تقديم وصف مفصل لكيفية استخدام هذه الطريقة لتحديد العلامات البيولوجية في الأورام الخبيثة لـ GI. والأهم من ذلك، أننا نصف ثلاث خطوات حاسمة ضرورية للحصول على نتائج دقيقة أثناء التحليل على نطاق الجينوم. بوضوح وإيجاز مكتوبة، هذه الأساليب الثلاث غالبا ما تفتقر إلى أن لا تكون ملحوظة لتلك الجديدة على الدراسات اللاجينية. استخدمنا 48 عينة من الورم الخبيث لـ GI (سرطان المعدة) لتسليط الضوء عمليًا على كيفية إجراء تحليل الميثيلية الحمض النووي على نطاق الجينوم للأورام الخبيثة لجي.
Introduction
يشير علم الوراثة اللاجينية إلى التغيرات التي يمكن تَنَتَرُّكها في وظيفة الجينات دون تغيير تسلسل الحمض النووي1. وقد تكون هذه التغيرات ناجمة عن وميض الحمض النووي، حيث يمكن لمجموعات الميثيل على قاعدة الحمض النووي أن تغير التعبير الجيني من خلال التغيرات في تعبئة الكروماتين. قد يحدث تطور السرطان والتقدم إذا كان هذا التأثير يؤدي إلى تغيير التعبير عن الجينات القامع الورم2. الشيخوخة والتهاب مزمن على حد سواء أسباب السرطان والأسباب الرئيسية للتغيرات في ميثييشن الحمض النووي في البشر3,4,5. وبالتالي، يسمح هذا باستخدام ميثيل الحمض النووي كعلامة بيولوجية في تشخيص السرطان، وكهدف للعلاج والوقاية. للكشف المبكر والتكهن بالسرطان، يتم قياس الميثيل الحمض النووي في عينات الورم والدم والبول والبراز6، في حين يتم الآن استخدام العوامل الديموثية لعلاج سرطان الدم مثل متلازمةالنخاعي 7.
يمكن استخدام تحليل الميثيل الحمض النووي على نطاق الجينوم باستخدام منصة صفيف للتقييم المعقد لميثيل الحمض النووي في موضع CPG فردي في الجينوم البشري لدراسة حالة المثيل لأكثر من 450،000 موقع CpG في الحمض النووي الجينومي8،9، مما يسمح باستكشاف الخلايا اللاجينية السرطانية (انظر جدول المواد). لقد غيرت تكنولوجيات تسلسل الجينوم الكامل (WGBS) نهجنا في مجال اللاجينية10،11. ومع ذلك ، هناك بعض العيوب للتكنولوجيات من حيث التكلفة الكبيرة ووقت المعالجة لتحليل اللاجينية لعدد كبير من العينات10،11. ولذلك، فإن منصة الصفيف أكثر جدوى للتقييم المعقد لتكين الحمض النووي في الجينوم البشري. وقد تحسن توافر النهج لتحليلات الميثيل على نطاق الجينوم في السنوات القليلة الماضية ويسمح لنا بتوسيع معرفتنا حول كيفية إسهام ميثيل الحمض النووي في تطور السرطانوتطوره 12. تقدم لنا التقدم الأخير في نهج منصة microarray الأساس المنطقي لتحليل الميثيل على نطاق الجينوم لتحديد توقيع غير جيني جديد في سرطانات الجهاز الهضمي13. والهدف من هذا البروتوكول هو تقديم وصف مفصل لكيفية استخدام هذه الطريقة لتحديد العلامات البيولوجية في الأورام الخبيثة لـ GI.
Protocol
وكانت جميع الإجراءات المتبعة متفقة مع المعايير الأخلاقية للجنة أخلاقيات البحوث البشرية التابعة للمؤسسات. وقد وافق على الدراسة مجلس الاستعراض المؤسسي في مستشفى شيزوكا بجامعة جونتندو، وتم التنازل عن الموافقة الخطية المستنيرة بسبب التصميم الاستعادي.
1. غسل الشرائح
- إعداد 10 ميكرومتر من الأجزاء غير المُضمَّنة للبارافين الثابتة وغير المُطَرَّكة (FFPE).
- ضع الشرائح في حامل شريحة زجاجية: استخدم شرائح مقطعية أكبر من 3 إلى 5 ما لم يكن النسيج في الحد الأدنى، وهناك حاجة إلى المزيد من الشرائح.
- املأ حامل الشريحة بالزيلين، وتأكد من أن جميع الأنسجة على الشريحة مغمورة. السماح للجلوس لمدة 15 دقيقة.
- بعد 15 دقيقة، صب من الزيلين مع الشرائح التي عقدت مع تلميح ماصة بحيث لا تسقط الشرائح.
- صب في أكثر من الزيلين إلى نفس المستوى كما كان من قبل. السماح للجلوس لمدة 15 دقيقة أخرى.
- بعد 15 دقيقة، صب من الزيلين مرة أخرى.
- ملء حامل الشريحة مع الإيثانول 100٪ (EtOH)، وتأكد من أن جميع الأنسجة على الشريحة مغمورة بالكامل. السماح للجلوس لمدة 3 دقائق.
- صب قبالة EtOH في حين عقد بعناية الشرائح. إعادة التعبئة إلى نفس المستوى مع EtOH أكثر. السماح للجلوس لمدة 2 دقيقة.
- صب قبالة EtOH مرة أخرى وإزالة الشريحة. وضعها بعناية مواجهة على منشفة ورقية نظيفة لتجف. السماح للجلوس لمدة 10 دقيقة.
2. كشط الشرائح
- إعداد الهضم / تحلل العازلة مع 650 مل من diethylpyrocarbonate (DEPC) المياه المعالجة، 100 مل من حمض الإيثيلين اديامينتيتراتيكتيك (EDTA)، 50 مل من هيدروكلوريد تريس (تريس-HCL) 2 M PH 8.8، و 200 مل من 10٪ كبريتات دوديسيل الصوديوم (SDS) (انظر جدول المواد).
- ملء 1.5 مل أحادية الاستخدام البولي بروبلين أنبوب مع 80 ميكرولتر من الهضم / تحلل العازلة (انظر جدول المواد).
- وضع طرف ماصة نظيفة في المخزن المؤقت.
- تحديد أنسجة السرطان في منطقة الورم الأكثر ملاءمة ل macrodissection وفقا لH & E المناسبة مقطع الملون.
ملاحظة: يجب تحديد منطقة الورم ل macrodissection من قبل اثنين من علماء الأمراض المؤهلين ويفضل. - Macrodissect الأنسجة السرطانية على أساس علامة H & E مقطع الملون.
- اتخاذ شفرة الحلاقة نظيفة وكشط بلطف الأنسجة السرطانية قبالة الشريحة، في محاولة للحفاظ عليه في قطعة واحدة بحيث يكون من الأسهل للعمل مع.
- خذ طرف ماصة من أنبوب البولي بروبلين أحادي الاستخدام يحتوي على المخزن المؤقت واستخدمه لنقل الأنسجة التي تم التخلص منها إلى قارورة العازلة (انظر جدول المواد).
ملاحظة: يجب أن تجذب التلميح الرطب الأنسجة ، مما يجعل النقل أسهل.
- كرر الخطوة 2.5 مع باقي الشرائح.
- بعد كل الأنسجة في أنبوب البولي بروبلين واحد الاستخدام، واستخدام طرف للتأكد من الأنسجة مغمورة تماما وغير عالق في جدار الأنبوب.
- إضافة 20 ميكرولتر من بروتياز سيرين ذات الصلة subtilisin إلى القارورة ونفض الغبار بلطف لخلط (انظر جدول المواد).
- ضع القارورة في كتلة حرارة 55 درجة مئوية لمدة 4 ساعات على الأقل أو بين عشية وضحاها. تأكد من دوامة قليلا بعد 2 ساعة.
3- علاج بيسولفيت
- استخدام 45 ميكرولتر من محلول الأنسجة المهضمة كعينة.
- قم بإجراء علاج ثنائي الفقار باستخدام الكواشف في مجموعة تحويل ثنائي الفريت وفقًا لتعليمات الشركة المصنعة (انظر جدول المواد).
- إضافة 5 ميكرولتر من عازلة التخفيف إلى عينة الحمض النووي واحتضان في 37 درجة مئوية لمدة 15 دقيقة (انظر جدول المواد).
- في حين أن العينات هي احتضان، وإعداد كاشف تحويل ثنائي الفلتر بإضافة 750 ميكرولتر من dH2O و 210 ميكرولتر من عازلة التخفيف إلى أنبوب واحد من كاشف التحويل CT (انظر جدول المواد). اخلطي الأنابيب عن طريق الدوامة لمدة 10 دقائق.
- بعد 15 دقيقة حضانة، إضافة 100 μL من المعدة CT تحويل كاشف لكل عينة ومزيج عن طريق عكس.
- احتضان العينات في الظلام في 50 درجة مئوية لمدة 12 إلى 16 ساعة.
- بعد الحضانة، وإزالة العينات ومكان على الجليد لمدة 10 دقيقة.
- إضافة 400 ميكرولتر من العازلة ملزمة وخلط كل عينة عن طريق الأنابيب صعودا وهبوطا (انظر جدول المواد). قم بتحميل كل عينة في عمود دوران ووضع العمود في أنبوب جمع 2 مل (انظر جدول المواد).
- أجهزة الطرد المركزي كل عينة في كامل سرعة (10000 س ز)لمدة 1 دقيقة وتجاهل تدفق من خلال.
- إضافة 200 μL من مخزن الغسيل إلى كل عمود وتدور بأقصى سرعة لمدة 1 دقيقة، وتجاهل تدفق من خلال (انظر جدول المواد).
- إضافة 200 ميكرولتر من عازلة إزالة الكبريت لكل عمود والسماح للعمود للوقوف في درجة حرارة الغرفة لمدة 15 دقيقة (انظر جدول المواد). بعد الحضانة، تدور الأعمدة في سرعة كاملة لمدة 1 دقيقة وتجاهل تدفق من خلال.
- إضافة 200 μL من مخزن الغسيل إلى كل عمود وتدور بأقصى سرعة لمدة 1 دقيقة (انظر جدول المواد).
- كرر هذه الخطوة مرة أخرى.
- إضافة 46 ميكرولتر من dH2O إلى كل عمود ووضع كل عمود في جديد معقم 1.5 مل أنبوب البولي بروبلين واحد الاستخدام (انظر جدول المواد). تدور كل أنبوب لمدة 2 دقيقة ل elute الحمض النووي.
- إزالة كل عمود تدور من أنبوب البولي بروبلين ذات الاستخدام الواحد وتجاهل (انظر جدول المواد). الحمض النووي جاهز الآن للتحليل
4- منصة صفيف لتقييم مركّز الحمض النووي في موضع Cpg في الجينوم البشري
- تقييم نوعية الحمض النووي (فحص الجودة: QC) باستخدام اختبار مراقبة الجودة FFPE على أداة تضخيم PCR والكشف في الوقت الحقيقي ، مع تحليل البيانات اللاحقة التي أجريت وفقا لتعليمات المصنعين (انظر جدول المواد).
ملاحظة: يتم معالجة نماذج مع قيم ∆Cq أقل من 5.0 الموصى بها14. - تحليل العينات باستخدام منصة صفيف لتقييم معقد لتكيثيل الحمض النووي لتقييم حالة المثيل من مواقع 450000 CpG في الجينوم (انظر جدول المواد). تتوفر ورقة معلومات المنتج وورقة البيانات وملفات المنتجات الخاصة بالنظام الأساسي للصفيف15.
ملاحظة: يتم إجراء الفحص وفقا لتعليمات الشركة المصنعة للمواد FFPE14. - حساب ميثيل موضع عالي ومنخفض في أداة تحليل بيانات لمنصة صفيف كقيمة β، مع نطاق من 0 إلى 1 على التوالي (انظر جدول المواد). استخدم البرامج المتوفرة تجارياً (انظر جدول المواد)لحساب قيمة β.
- استيراد البيانات التي تم إنشاؤها على منصة صفيف للتقييم المعقد للبرمجيات الميثيل DNA في بيئة البرمجيات R (R v.2.15.1) ومعالجتها باستخدام أداة لتحليل صفائف الميثيل16،17.
ملاحظة: في خريطة الحرارة، التي يتم إنشاؤها باستخدام أداة تحليل البيانات، يتم ترتيب الأعمدة بواسطة التجميع غير الخاضعة للرقابة، في حين أن ترتيب الصفوف يستند إلى الأهمية المتناقصة للإحصائية t لـ المثيلة التفاضلية من أعلى إلى أسفل. - تقسيم خريطة الحرارة إلى مجموعات الميثيل عالية ومنخفضة باستخدام أول مفاضل في التجمع غير الخاضعة للرقابة.
ملاحظة: للتحقق من صحة مع PCR الكمي الخاص بالثلائد (qMSP)، اختر الجينات استناداً إلى قيمة أكبر β بالنسبة إلى جزر Cpg في منطقة المروج، ونظراً لمدى ملاءمتها لتصميم التمهيدي والمسبار لـ qMSP.
5- تحليل PCR الكمي الخاص بالتكين (qMSP)
- استخدام الحمض النووي المعدل من مادة البيسلفيت من الخطوة 3-3 كقالب ل PCR في الوقت الحقيقي القائم على الفلور في qMSP لتقييم مثيلة المنطقة المروجة في كل تحليل جيني.
- تنفيذ qMSP باستخدام أداة PCR في الوقت الحقيقي 96-well (انظر جدول المواد).
- تحقق من حالة الميثيل المروج للجين المستهدف على الحمض النووي المعدلة بسلفوليت باستخدام 200 nM إلى الأمام التمهيدي ، 200 nM التمهيدي العكسي ، و 80 مسابير nM. إعداد مزيج الرئيسي مع 16.6 mM (NH4)2SO4، 67 mM تريس درجة PH 8.8 ، 10 mM β-mercaptoethanol, 10 nM فلوريسين, 0.166 mM من كل ثلاثي الفوسفات ديوكسي نوكليوتيد, و 0.04 U/μl من بوليميراز الحمض النووي (انظر جدول المواد). حجم التفاعل النهائي في كل بئر لجميع المقايسات هو 25 ميكرولتر.
- أداء ركوب الدراجات من qMSP على النحو التالي: 95 درجة مئوية لمدة 5 دقائق، تليها 55 دورة من 95 درجة مئوية لمدة 15 ثانية، 60 درجة مئوية لمدة 1 دقيقة، و 72 درجة مئوية لمدة دقيقة.
ملاحظة: ينبغي اختيار الجين المستهدف بناء على معايير وجود قيم بيتا أكبر، وأن تكون ذات صلة بجزر Cpg في منطقة المروج، وأن تكون مناسبة لتصميم التمهيدي والمسبار لـ qMSP.
- استخدام الجينوم الإنسان تعامل مع ميثيلز CpG (M.SssI) باعتبارها عنصر تحكم ميثيل إيجابي (انظر جدول المواد).
ملاحظة: يتم تعريف القياس الكمي النهائي للميثيل على أنه قيمة المثيل النسبية (RMV)، ويتم حسابه كـ 2-ΔCt لكل تكرار للكشف عن المثيلات مقارنةً مع متوسط Ct β-Actin (ACTB)18. يتم عرض تسلسل التمهيدي والمسبار في الجدول 1. ويستخدم Ct من 100 للتكرار غير المكتشفة، والذي يعطي قيمة 2- ΔCt قريبة من الصفر. وتستخدم الصيغة التالية: يعني 2- ΔCt (RMV) = (2-Δ Ct_replicate_1 + 2- Δ Ct_replicate_2 + 2-Δ Δ Ct_replicate_3)/318.
النتائج
وفيما يلي خصائص 48 مريضاً مصابين بسرطان المعدة في مجموعة التدريب(الجدول 2):كان متوسط عمر المرضى 74 سنة (52-89 سنة)، وضمت الفوج 38 ذكراً (79.2 في المائة)، و10 إناث (20.8 في المائة). وكان هناك 35 مريضاً (72.9٪) مع سرطان المعدة الأولية وفي 13 مريضا (27.1٪ ) مع بقايا سرطان المعدة (سرطان المعدة الأولية: أول حدوث من خبيث غير النقيلي في المعدة; سرطان المعدة المتبقية: السرطان في بقايا المعدة التي وضعت أكثر من 5 سنوات بعد استئصال المعدة القعد, بغض النظر عن السبب في استئصال الأصلي19). وكان هناك 23 مريضاً (47.9 في المائة) مع الانبثاث العقدة الليمفاوية و 25 مريضا (52.1٪) دون. أولاً، تم تحميل جميع العينات الـ 48 (الفوج التدريبي) لتحديد القيم المتطرفة(الشكل 1A). أعطت عينتان قمم أكبر من انحرافين معياريين تم إزاحتها عن الآخرين ، وتمت إزالة هذه العينات (الشكل 1B). ولذلك، تم تجميع 46 عينة بواسطة فرط ميثيل المروج للحمض النووي. تم تقسيم خريطة الحرارة الناتجة إلى مجموعتين على أساس المثيل عالية ومنخفضة (الشكل 2). تسمح هذه الخريطة الحرارية بالتصور من أعلى 50 مسابير داخل 1500 BP من موقع بدء النسخ (TSS) في تحليل الميثيل التفاضلي. تباينت مجموعات الميثيلات العالية والمنخفضة في العوامل السريرية المتعلقة بالنمط الظاهري الخبيث العدواني. وهذا هو نوع من السرطان (سرطان المعدة الأولية: PGC) (ع = 0.01، نسبة الاحتمالات = 9.09 (1.67-50.00)) ووجود ورم خبيث العقدة الليمفاوية (إيجابية) (p = 0.03، نسبة الاحتمالات = 6.82 (1.16-40.08)) ظهرت كعوامل تنبؤية مستقلة كبيرة عندما استخدمت العوامل المرضية كتحاليل متعددة المتغيرات(الجدول 3). وأخيراً، حددنا جين EPB41L320،21 (التمهيدي وتسلسل التحقيق المبين في الجدول 1) لتكون مرتبطة بقوة بتدوين مجموعة التدريب في مجموعات الميثيل عالية ومنخفضة في تحليل microarray. وباستخدام البرنامج الكمي للعلوم ومبيدات الوب، تم التحقق من نتائج تحليل المجهر لـ EPB41L3 في مجموعة الاختبار (126 عينة). وتظهر خصائص المرضى في الفوج اختبار في الجدول 4. وكانت مركبات RMVs من EPB41L3 في أنسجة PGC أعلى بكثير من تلك الموجودة في بقايا سرطان المعدة (RGC) في تحليل أحادي المتغيرات (p = 0.01) (الشكل 3A). وبالمثل، كانت RMVs في العينات مع الانبثاث العقدة الليمفاوية أعلى بكثير من تلك التي لا نقائل العقدة الليمفاوية (ص = 0.03) (الشكل 3B). وبهذه الطريقة، يمكن أن يساعدنا تحليل الحمض النووي على نطاق الجينوم على تحديد جينات محددة لتوصيف حالة سريرية معينة في المرضى الذين يعانون من الأورام الخبيثة في الجهاز الهضمي.
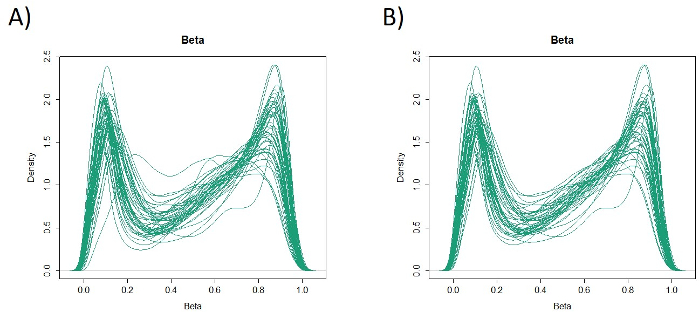
الشكل 1: قيم بيتا في 48 عينة (مجموعة التدريب). تم تحميل جميع العينات الـ 48 (مجموعة التدريب) وفحص القيم المتطرفة (A). وكان اثنين من العينات قمم التي كانت القيم المتطرفة، وتمت إزالة هذه (B). الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
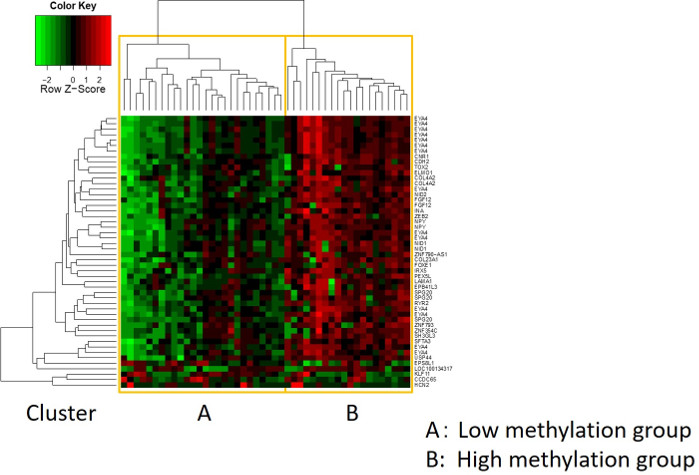
الشكل 2: خريطة الحرارة الناتجة. أما العينات الـ 46 المتبقية فقد تم تجميعها بواسطة فرط مُروِّج الحمض النووي. تم تقسيم خريطة الحرارة إلى مجموعات الميثيل عالية ومنخفضة. تسمح هذه الخريطة الحرارية بالتصور من أعلى 50 مسابير داخل 1500 BP من موقع بدء النسخ (TSS) في تحليل الميثيل التفاضلي. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
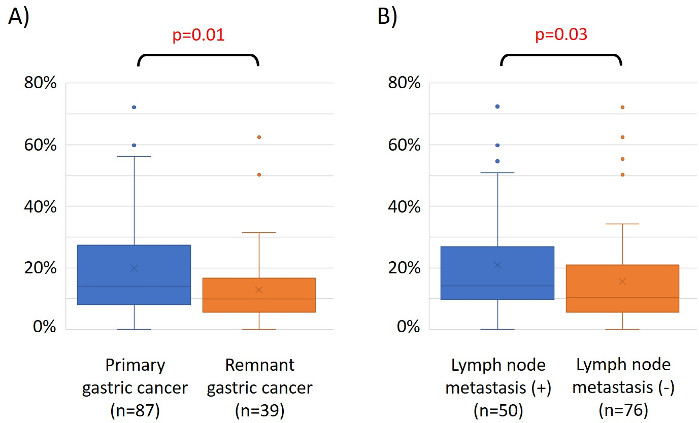
الشكل 3: قيم المثيلات النسبية (RMVs) لـ EPB41L3 في سرطان المعدة الأولية (PGC) مقابل سرطان المعدة المتبقية (RGC)، وفي الحالات مع أو بدون ورم خبيث العقدة الليمفاوية. تم التحقق من نتائج تحليل الراَسَق الصغرى لـ EPB41L3 في مجموعة الاختبار (126 عينة) باستخدام qMSP. (أ)في التحليل أحادي المتغير، كانت مركبات RMVs من EPB41L3 في أنسجة PGC أعلى بكثير من تلك الموجودة في RGC (p = 0.01). (B) وبالمثل، كانت الـ RMVs في العينات ذات الانبثاث العقدة الليمفاوية أعلى بكثير مما كانت عليه في تلك التي لا توجد بها نقائل العقدة الليمفاوية (p = 0.03). الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
| الجينات | إلى الأمام 5'- 3' | عكس 5' - 3 ' | التحقيق |
| B-ACTIN | TAG GGA GTA TAT AGG TTG GGG AAG TT | AAC ACA CAA TAA CAA ACA CAA ATT CAC | \56-FAM\ TGT GGG GTG \ZEN\ GTG هفوة GTT GTT هغه GTT \3IABkFQ\ \ |
| EPB41L3 | GGG ATA GTG GGG TTG ACG C | ATA AAA ATC CCG ACG AAC GA | AAA TTC GAA AAA CCG CGC GAC دول مجلس التعاون الخليجي GAA ACC A |
الجدول 1: تسلسل التمهيدي والمسبار.
| العوامل السريرية | المتغيرات | |
| العمر | 74 (52 - 89) * | |
| الجنسين | ذكر / أنثى | 38 (79.2%) / 10 (20.8%) |
| نوع | PGC / RGC | 35 (72.9%) / 13 (27.1%) |
| ورم خبيث العقدة الليمفاوية | (+) / (-) | 23 (47.9%) / 25 (52.1%) |
| PGC: سرطان المعدة الأولية، RGC: بقايا سرطان المعدة | ||
| * متوسط (الحد الأقصى الأدنى) | ||
الجدول 2: الخصائص في 48 مريضاً بسرطان المعدة في الفوج التدريبي.
| قيمة ف | نسبة الاحتمالات | فاصل ثقة بنسبة 95٪ | |
| النوع (PGC) | 0.01 | 9.09 | 1.67 – 50.00 |
| ورم خبيث العقدة الليمفاوية (+) | 0.03 | 6.82 | 1.16 – 40.08 |
| PGC: سرطان المعدة الأولية | |||
الجدول 3: العوامل التنبؤية لمجموعة الميثيلات العالية (الفئة باء).
| العوامل السريرية | المتغيرات | |
| العمر | 71 (33 - 86) * | |
| الجنسين | ذكر / أنثى | 96 (76.2%) / 30 (23.8%) |
| نوع | PGC / RGC | 87 (69.0%) / 39 (31.0%) |
| ورم خبيث العقدة الليمفاوية | (+) / (-) | 50 (39.7%) / 76 (60.3%) |
| PGC: سرطان المعدة الأولية، RGC: بقايا سرطان المعدة | ||
| * متوسط (الحد الأقصى الأدنى) | ||
الجدول 4: الخصائص في 126 مريضاً بسرطان المعدة في الفوج الاختباري.
Discussion
هناك ثلاث خطوات حاسمة في الحصول على نتائج دقيقة من تحليل الحمض النووي على نطاق الجينوم الميثيل. الأول هو macrodissection من منطقة الورم من قبل اثنين من علماء الأمراض المؤهلين ويفضل استنادا إلى التمثيل H & E المقاطع الملون. يمكن أن يسبب الاضاءة غير الدقيقة التلوث مع الأنسجة غير السرطانية المجاورة، مما يولد نتائج غير موثوقة. وبالتالي، مطلوب الماكروديسينكشن دقيق. والثاني هو تقييم جودة الحمض النووي (فحص الجودة: QC). العينات التي تفشل في QC (∆Cq > 5.0) قد تعطي بيانات ذات جودة رديئة. ولذلك، يجب إزالة العينات مع ∆CQ > 5.0 واستخدامها الآخرين. الخطوة الثالثة هي حساب القيمة β ، والتي يتم تحديدها بواسطة أداة تحليل البيانات لبرنامج منصة الصفيف كإشارة الميثيلية / المجموع (ميثيل + غير الملته) إشارة17. تتراوح القيمة β من 0-1 (أو 0٪-100٪)، وهو أمر بسيط لتفسير بيولوجيا17. المشكلة الرئيسية مع هذه القيمة هي خصائصها الإحصائية السيئة ، حيث أن عدم الاتساق العالي يعني أن التباين عبر العينات في أقصى درجات المدى المُمَسَلّل (β = 0 أو β = 1) ينخفض بدرجة عالية17. وبالإضافة إلى ذلك، وبسبب سوء نوعية العينات، قد لا تظهر القيم β قمم ثنائية الطور قابلة للاستنساخ22، وينبغي استبعاد العينات التي لا تخلو من هذه القمم من الدراسة الإضافية. وبالإضافة إلى ذلك، ينبغي اختيار الجين المستهدف على أساس معايير وجود قيم بيتا أكبر، وأن تكون ذات صلة بجزر Cpg في المنطقة المروجة، وأن تكون مناسبة لتصميم التمهيدي والمسبار لـ qMSP.
يتم تقييم ميثيل الحمض النووي في موضع Cpg في الجينوم البشري باستخدام التكنولوجيا القائمة على microarray مع عدد محدد من المسابير لمسح loci الجينومية محددة. وهو الأسلوب الأكثر استخداما في دراسات الرابطة على نطاق epigenome (EWAS) بسبب تكلفتها المنخفضة، وكمية صغيرة من الحمض النووي المطلوبة، وأقصر بشكل ملحوظ وقت معالجة العينة، والذي يسمح معالجة عالية الإنتاجية للعديد من العينات السريرية23. ومع ذلك، فإن منصة صفيف للتقييم المعقد لتكيثيل الحمض النووي في موضع Cpg الفردية محدودة بعدد وخصوصية المسابير لـ loci المتغيرة وراثياً، مما يمنع استكشاف بعض المناطق الجينومية. وينظر عموما WGBS على أنها طريقة معيار الذهب نظرا لطيف أوسع من التغطية الجينومية10،11. ومع ذلك ، فإن هذه الطريقة لديها تكلفة كبيرة وطويلة نسبيا وقت المعالجة لتحليل عدد كبير من العينات10،11. وبالتالي، فإنه ليس ممكنا دائما. وبالمقارنة، فإن منصة الصفيف للتقييم المعقد لميثيل الحمض النووي في موضع من نوع Cpg في الجينوم البشري معقولة للاستخدام من حيث التكلفة والتغطية الجينية. في الآونة الأخيرة، حصلت على أحدث رقائق الخرز ترقية جاهزة لاستخدام24. يمكن أن تساعدنا هذه المقايسات في تحليل مواقع Cpg المضاءة تقريبًا ، والتي يمكن أن تحقق دراسة رابطة مثالية على نطاق الجينوم (GWAS) لفئات كبيرة من السكان.
وباختصار، فإن تحليل الحمض النووي على نطاق الجينوم مع نظام مجموعة لتقييم معقد لتكبيل الحمض النووي في موضع CPG الفردية في الجينوم البشري يمكن أن توفر معلومات هامة عن المؤشرات الحيوية اللاجينية في سرطان الجهاز الهضمي. مقارنة مع WGBS، هذه الطريقة فعالة من حيث التكلفة ويقلل من وقت معالجة العينة. ولذلك، فإن هذه الطريقة للكشف عن ميثيل الحمض النووي في موضع CPG من المرجح أن تستخدم على نطاق واسع في البحوث العلامات البيولوجية اللاجينية.
Disclosures
ليس لدى أصحاب البلاغ ما يكشفون عنه.
Acknowledgements
ونحن ممتنون بشكل خاص لجميع أعضاء قسم الجراحة، مركز سيدني كيميل الشامل للسرطان في كلية الطب بجامعة جونز هوبكنز لإجراء مناقشات مفيدة ودعم تقني. كما نشكر كريستين رودجرز على التوجيه التقني السخي بشأن إجراءات علاج ثنائي السلفيت وQMSP.
Materials
| Name | Company | Catalog Number | Comments |
| (NH4)2SO4 | Sigma-Aldrich | 14148 | Step 5.2. |
| 10% Sodium dodecyl sulfate (SDS) | Quality Biological | 351-032-721 EA | Step 2.1. |
| 100 % Ethanol | Sigma-Aldrich | 24194 | Step 1.7. |
| ABI StepOnePlus Real-Time PCR System | Applied BioSystems | 4376600 | Step 5.2. 96-well Real-Time PCR instrument |
| CT conversion reagent | Zymo Research | D5001-1 | Step 3.2.3. |
| Deoxynucleotide triphosphate (dNTP) | Invitrogen | 10297-018 | Step 5.2. |
| DEPC-treated water | Quality Biological | 351-068-131 | Step 2.1. |
| Ethylenediaminetetraacetic acid (EDTA) | Corning | 46-034-CL | Step 2.1. |
| EZ DNA Methylation Kit | Zymo Research | D5002 | Step 3.2. |
| Fluorescein | Bio-Rad | #1708780 | Step 5.2. |
| GenomeStudio | omicX | OMICS_00854 | Step 4.3. Data analysis tool for an array platform as a β-value, with a range from 0 to 1 |
| Human genomic DNA | New England Bio Labs | N4002S | Step 5.3. |
| Infinium HD FFPE QC Kit | Illumina | WG-321-1001 | Step 4.1. FFPE QC assay on a real-time PCR amplification |
| Infinium HumanMethylation450 assay | Illumina | WG-314 | Step 4.2. Array platform for complex evaluation of DNA methylation to assess the methylation status of >450,000 CpG sites in the genome |
| LightCycler 480 | Roche | 5015278001 | Step 4.1. |
| M-Binding Buffer | Zymo Research | D5002-3 | Step 3.2.6. |
| M-Desulphonation Buffer | Zymo Research | D5002-5 | Step 3.2.9. |
| M-Dilution Buffer | Zymo Research | D5002-2 | Step 3.2.1. |
| Minfi package | Bioconductor | N/A | Step 4.4. |
| M-Wash Buffer | Zymo Research | D5002-4 | Step 3.2.10. |
| Platinum Taq polymerase | ThermoFisher Scientific | 10966-034 | Step 5.2. |
| Proteinase K | New England Biolabs | P8107S | Step 2.8. |
| Single-use polypropylene (Eppendorf) tube | Eppendorf | 24533495 | Step 2.5.2. |
| Tris hydrochloride (Tris-HCL) 2 M pH 8.8 | Quality Biological | 351-092-101 | Step 2.1. |
| Xylene | Sigma-Aldrich | 214736 | Step 1.3. |
| Zymo Spin 1 Column | Zymo Research | C1003 | Step 3.2.6. |
| β-Mercaptoethanol | Sigma-Aldrich | M3148 | Step 5.2. |
References
- Qiu, J. Epigenetics: unfinished symphony. Nature. 441 (7090), 143-145 (2006).
- Okugawa, Y., Grady, W. M., Goel, A. Epigenetic alterations in colorectal cancer: emerging biomarkers. Gastroenterology. 149 (5), 1204-1225 (2015).
- Ahuja, N., Li, Q., Mohan, A. L., Baylin, S. B., Issa, J. P. Aging and DNA methylation in colorectal mucosa and cancer. Cancer Research. 58 (23), 5489-5494 (1998).
- Hsieh, C. J., et al. Hypermethylation of the p16INK4a promoter in colectomy specimens of patients with long-standing and extensive ulcerative colitis. Cancer Research. 58, 3942-3945 (1998).
- Ushijima, T., Okochi-Takada, E. Aberrant methylations in cancer cells: where do they come from. Cancer Science. 96 (4), 206-211 (2005).
- Coppedè, F. Epigenetic biomarkers of colorectal cancer: Focus on DNA methylation. Cancer Letters. 342 (2), 238-247 (2014).
- Vasilatou, D., Papageorgiou, S. G., Dimitriadis, G., Pappa, V. Epigenetic alterations and microRNAs: new players in the pathogenesis of myelodysplastic syndromes. Epigenetics. 8 (6), 561-570 (2013).
- Ma, X., Wang, Y. W., Zhang, M. Q., Gazdar, A. F. DNA methylation data analysis and its application to cancer research. Epigenomics. 5 (3), 301-316 (2013).
- Morris, T. J., Beck, S. Analysis pipelines and packages for Infinium HumanMethylation450 BeadChip (450k) data. Methods. 72, 3-8 (2015).
- Rauluseviciute, I., Drabløs, F., Rye, M. B. DNA methylation data by sequencing: experimental approaches and recommendations for tools and pipelines for data analysis. Clinical Epigenetics. 11 (1), 193 (2019).
- Cazaly, E. Making sense of the epigenome using data integration approaches. Frontiers in Pharmacology. 10, 126 (2019).
- Leal, A., Sidransky, D., Brait, M. Tissue and cell-free DNA-based epigenomic approaches for cancer detection. Clinical Chemistry. 66 (1), 105-116 (2020).
- Song, W., Ren, J., Wang, W. J., Wang, C. T., Fu, T. Genome-wide methylation and expression profiling identify a novel epigenetic signature in gastrointestinal pan-adenocarcinomas. Epigenomics. 12 (11), (2020).
- Wong, E. M., et al. Tools for translational epigenetic studies involving formalin-fixed paraffin-embedded human tissue: applying the Infinium HumanMethyation450 Beadchip assay to large population-based studies. BMC Research Notes. 8, 543 (2015).
- . Illumina Available from: https://jp.support.illumina.com/downloads/infinium_humanmethylation450_product_files.html (2020)
- Fackler, M. J., et al. Genome-wide methylation analysis identifies genes specific to breast cancer hormone receptor status and risk of recurrence. Cancer Research. 71 (19), 6195-6207 (2011).
- Dedeurwaerder, S., et al. A comprehensive overview of Infinium HumanMethylation450 data processing. Briefings in Bioinformatics. 15 (6), 929-941 (2014).
- Hulbert, A., et al. Early detection of lung cancer using DNA promoter hypermethylation in plasma and sputum. Clinical Cancer Research: An Official Journal of the American Association for Cancer Research. 23 (8), 1998-2005 (2017).
- Shimada, H., Fukagawa, T., Haga, Y., Oba, K. Does remnant gastric cancer really differ from primary gastric cancer? A systematic review of the literature by the Task Force of Japanese Gastric Cancer Association. Gastric Cancer: Official Journal of the International Gastric Cancer Association and the Japanese Gastric Cancer Association. 19 (2), 339-349 (2016).
- Eijsink, J. J., et al. Detection of cervical neoplasia by DNA methylation analysis in cervico-vaginal lavages, a feasibility study. Gynecologic Oncology. 120 (2), 280-283 (2011).
- Sugimoto, K., et al. DNA methylation genome-wide analysis in remnant and primary gastric cancers. Gastric Cancer: Official Journal of the International Gastric Cancer Association and the Japanese Gastric Cancer Association. 22 (6), 1109-1120 (2019).
- Wang, Z., Wu, X., Wang, Y. A framework for analyzing DNA methylation data from Illumina Infinium HumanMethylation450 BeadChip. BMC Bioinformatics. 19, 115 (2018).
- Teh, A. L., et al. Comparison of methyl-capture sequencing vs. Infinium 450K methylation array for methylome analysis in clinical samples. Epigenetics. 11 (1), 36-48 (2016).
- Zhou, W., Laird, P. W., Shen, H. Comprehensive characterization, annotation and innovative use of Infinium DNA methylation BeadChip probes. Nucleic Acids Research. 45 (4), 22 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved