Method Article
Gastrointestinal Kanserde DNA Metilasyonunun Genom Çapında Analizi
Bu Makalede
Özet
Burada, gastrointestinal kanserlerde DNA metilasyonunun genom çapında analizi için bir prosedür tanımladık. Prosedür, gastrointestinal kanserlerde kanserogeneze katkıda bulunan genlerin metilasyon desenleri ile faktörler arasındaki ilişkileri araştıran çalışmalarla ilgilidir.
Özet
DNA metilasyon biyolojik olarak anlamlı ve kanser araştırmalarının sık sık odak noktası olan önemli bir epigenetik değişimdir. Genom çapında DNA metilasyon gastrointestinal (GI) malignitelerin metilasyon durumunun doğru bir analizini sağlamak için yararlı bir önlemdir. DNA metilasyon analizinin birden fazla potansiyel çevirisel kullanımı göz önüne alındığında, pratik klinisyenler ve DNA metilasyon çalışmalarında yeni diğerleri bu genom çapında analizlerin nasıl yapıldığını adım adım anlayabilmek gerekir. Bu protokolün amacı, gi malignitelerinde biyomarker tanımlamaiçin bu yöntemin nasıl kullanıldığına ayrıntılı bir açıklama sağlamaktır. Daha da önemlisi, genom çapında analiz sırasında doğru sonuçlar elde etmek için gereken üç kritik adımı tanımlıyoruz. Açıkça ve özlü yazılmış, bu üç yöntem genellikle eksik ve epigenetik çalışmalara yeni olanlar için fark edilmez. Genom çapında DNA metilasyon analizinin GI maligniteleri için nasıl yapılabilir olduğunu vurgulamak için 48 GI malignitesi (gastrik kanser) üyesi kullandık.
Giriş
Epigenetik, DNA1dizisini değiştirmeden gen fonksiyonlarındaki kalıtsal değişiklikleri ifade eder. Bu tür değişiklikler DNA metilasyon nedeniyle olabilir, hangi bir DNA tabanında metil grupları kromatin ambalaj değişiklikler yoluyla gen ekspresyonunu değiştirebilir. Bu etki tümör baskılayıcı genlerin değişmiş ekspresyonuna neden olursa kanser gelişimi ve ilerlemesi oluşabilir2. Yaşlanma ve kronik inflamasyon hem kanser nedenleri ve insanlarda DNA metilasyon değişiklikleri için ana nedenleri3,4,5. Sonuç olarak, bu kanser tanısında bir biyomarker olarak DNA metilasyon kullanımına olanak sağlar, ve tedavi ve önleme için bir hedef olarak. Erken teşhis ve kanser prognozu için, DNA metilasyon tümör ölçülmektedir, kan, idrar, ve dışkı örnekleri6, demetiye ajanlar şimdi miyelodisplastik sendrom gibi lösemi tedavisinde kullanılmaktadıriken 7.
Genom çapında DNA metilasyon analizi insan genomunda bireysel bir CpG locus DNA metilasyon karmaşık değerlendirilmesi için bir dizi platformu kullanarak genomik DNA'da fazla 450.000 CpG sitelerinin metilasyon durumunu incelemek için kullanılabilir8,9, hangi kanser epigenetik keşif izin verir (Malzemeler Tablosubakınız). Tüm genom bisülfit (WGBS) teknolojileri epigenetik10,11alanında yaklaşımlarımızı değiştirdi. Ancak, çok sayıda örnek10,11epigenetik analizi için önemli bir maliyet ve işlem süresi açısından teknolojileriçin bazı dezavantajları vardır. Bu nedenle, dizi platformu insan genomundaki DNA metilasyonunun karmaşık değerlendirilmesi için daha uygulanabilir. Genom çapında metilasyon analizleri için yaklaşımların kullanılabilirliği son birkaç yıl içinde iyileşmiştir ve DNA metilasyonkanser gelişimi ve ilerlemesine nasıl katkıda bulunduğu bilgimizi genişletmek için bize olanak sağlar12. Mikrodizi-platform yaklaşımlarında son gelişmeler bize gastrointestinal kanserlerde yeni bir epigenetik imza belirlemek için genom çapında metilasyon analizi için mantığı sağlamak13. Bu protokolün amacı, bu yöntemin Gİs malignitelerinde biyomarker tanımlamada nasıl kullanıldığına ayrıntılı bir açıklama sağlamaktır.
Protokol
İzlenen tüm prosedürler kurumların insan araştırma etik komitesinin etik standartlarına uygun du. Çalışma Juntendo Üniversitesi Shizuoka Hastanesi Kurumsal İnceleme Kurulu tarafından onaylandı ve yazılı bilgilendirilmiş onay retrospektif tasarım nedeniyle feragat edildi.
1. Slaytların yıkanması
- 10 μm lekesiz formalin-sabit parafin gömülü (FFPE) kesitler hazırlayın.
- Slaytları cam bir slayt tutucuya yerleştirin: doku minimum olmadığı ve daha fazla slayt alamadığı sürece yaklaşık 3-5 büyük kesitslaytkullanın.
- Slayt tutucuyu ksilen ile doldurun ve slayttaki tüm dokunun batırDığından emin olun. 15 dakika boyunca oturun.
- 15 dk sonra, slaytlar düşmemesi için bir pipet ucu ile düzenlenen slaytlar ile ksilenin dökün.
- Daha önce olduğu gibi aynı seviyeye daha fazla ksilene dökün. Başka bir 15 dakika için oturup bekleyin.
- 15 dk sonra, tekrar ksilenin dökün.
- Slayt tutucuyu %100 etanol (EtOH) ile doldurun ve slayttaki tüm dokunun tamamen batırılmış olduğundan emin olun. 3 dakika boyunca oturun.
- Slaytları dikkatlice tutarken EtOH'u dökün. Daha fazla EtOH ile aynı seviyeye yeniden doldurun. 2 dakika boyunca oturun.
- EtOH'u tekrar dökün ve kaydırağı çıkarın. Dikkatlice kuruması için temiz bir kağıt havlu üzerinde yüz yerleştirin. 10 dakika boyunca oturun.
2. Slaytları kazıma
- 650 mL diethylpyrocarbonate (DEPC) ile işlenmiş su, 100 mL Etilendiamintetraasetik asit (EDTA), 50 ml Tris hidroklorür (Tris-HCL) 2 M pH 8.8 ve 200 mL%10 sodyum dodecyl sülfat (SDS) ile sindirim/lizis tamponu hazırlayın (Bkz. Malzemeler Tablosu).
- 1,5 mL tek kullanımlık polipropilen tüpü 80 μL sindirim/lysis tamponuyla doldurun (bkz.
- Arabelleğe temiz bir pipet ucu koyun.
- Uygun H&E lekeli kesitine göre makrodiskesiçin en uygun tümör bölgesikanser dokularını belirleyin.
NOT: Makrodiszit için tümör alanı tercihen iki nitelikli patolog tarafından tespit edilmelidir. - Belirgin H&E lekeli kesitine göre makrodissect kanser dokusu.
- Temiz bir jilet alın ve yavaşça slayt kapalı kanser dokusu kazıyın, böylece çalışmak daha kolay bir parça da tutmaya çalışıyorum.
- Pipet ucunu tampon içeren tek kullanımlık polipropilen tüpten alın ve hurdaya çıkarılan dokuyu tampon şişeye aktarmak için kullanın (bkz. Malzemeler Tablosu).
NOT: Islönme ucu dokuyu çekmelidir, bu da transferi kolaylaştırır.
- Slaytların geri kalanı ile adım 2.5 tekrarlayın.
- Tüm doku tek kullanımlık polipropilen tüp sonra, doku tamamen batık olduğundan emin olmak için ucu kullanın ve tüp duvarına sıkışmış değildir.
- Şişeye subtilisin ile ilgili serin proteazın 20 μL'sini ekleyin ve karıştırmak için hafifçe hareket ettirin (Bkz. Malzeme Tablosu).
- Şişeyi en az 4 saat veya bir gece boyunca 55 °C'lik bir ısı bloğuna yerleştirin. 2 saat sonra hafifçe girdap emin olun.
3. Bisülfit tedavisi
- Örnek olarak sindirilmiş doku çözeltisinin 45 μL'sini kullanın.
- Üreticinin talimatlarına uygun bir bisülfit dönüşüm kitinde reaktifler kullanarak bisülfit tedavisini gerçekleştirin (bkz. Malzemeler Tablosu).
- DNA örneğine 5 μL seyreltme tamponu ekleyin ve 37 °C'de 15 dakika kuluçkaya yatırın (bkz. Malzemeler Tablosu).
- Numuneler kuluçkaya yatarken, BT dönüşüm reaktifinin bir tüpüne 750 μL dH2O ve 210 μL seyreltme tamponu ekleyerek bisülfit dönüşüm reaktifini hazırlayın (bkz. 10 dakika girdap tarafından tüpleri karıştırın.
- 15 dk kuluçkadan sonra, her numuneye 100 μL'lik ct dönüşüm reaktifini ekleyin ve ters çevrilerek karıştırın.
- Numuneleri karanlıkta 50 °C'de 12 ila 16 saat kuluçkaya yatırın.
- Kuluçkadan sonra, örnekleri çıkarın ve 10 dakika boyunca buz üzerine yerleştirin.
- 400 μL bağlayıcı tampon ekleyin ve her numuneyi yukarı ve aşağı borular oluşturarak karıştırın (bkz. Malzeme Tablosu). Her numuneyi bir spin sütununa yükleyin ve sütunu 2 mL toplama tüpüne yerleştirin (bkz. Malzeme Tablosu).
- Her numuneyi 1 dk boyunca tam hızda (10.000 x g)santrifüj edin ve akışı atın.
- Her sütuna 200 μL yıkama tamponu ekleyin ve akıştan geçenleri atarak 1 dakika boyunca tam hızda döndürün (Bkz. Malzeme Tablosu).
- Her kolona 200 μL desülfürasyon tamponu ekleyin ve sütunun 15 dakika oda sıcaklığında durmasını bekleyin (Bkz. Malzeme Tablosu). Kuluçkadan sonra, 1 dakika boyunca tam hızda sütunları döndürün ve akışı atın.
- Her sütuna 200 μL yıkama tamponu ekleyin ve 1 dakika boyunca tam hızda döndürün (Bkz. Malzeme Tablosu).
- Bu adımı bir kez daha tekrarlayın.
- Her sütuna 46 μL dH2O ekleyin ve her sütunu yeni bir steril 1,5 mL tek kullanımlık polipropilen tüpe yerleştirin (bkz. Malzeme Tablosu). DNA'yı eforlamak için her tüpü 2 dakika döndürün.
- Her spin sütununun tek kullanımlık polipropilen tüpünü çıkarın ve atın (bkz. Malzemeler Tablosu). DNA analiz için hazır.
4. İnsan genomundaki CpG locusundaki DNA metilasyonunun değerlendirilmesi için dizi platformu
- Üreticilerin talimatlarına göre yapılan sonraki veri analizi ile gerçek zamanlı PCR amplifikasyon ve algılama aracında FFPE QC tahlillerini kullanarak DNA'nın kalitesini (kalite kontrolü: QC) değerlendirin (Bkz. Malzeme Tablosu).
NOT: ∆Cq değerleri önerilen 5.0'dan daha az olan numuneler daha da işlenir14. - Dna metilasyonunun karmaşık değerlendirilmesi için örnekleri genomdaki >450.000 CpG bölgesinin metilasyon durumunu değerlendirmek için bir dizi platform ile analiz edin (bkz. Dizi platformuna ait ürün bilgi sayfası, veri sayfası ve ürün dosyaları mevcuttur15.
NOT: FfPE malzeme14için üreticinin talimatlarına göre titreştirilir. - Bir dizi β platformu için bir veri analiz aracında sırasıyla 0 ile 1 arasında değişen bir aralıkla yüksek ve düşük locus metilasyonunu hesaplayın (bkz. Malzemeler Tablosu). β değerini hesaplamak için ticari olarak kullanılabilen yazılımları (bkz. Malzeme Tablosu'nabakın).
- Dna metilasyon platformunun R yazılım ortamına karmaşık değerlendirilmesi için dizi platformunda oluşturulan verileri alma (R v.2.15.1) ve metilasyon dizilerini analiz etmek için bir araç kullanarak işleme16,17.
NOT: Bir veri analiz aracı kullanılarak oluşturulan ısı haritasında, sütunlar denetimsiz kümeleme ile sıralanırken, satır sırası diferansiyel metilasyon için t istatistiğinin yukarıdan aşağıya doğru azalan önemine dayanır. - Isı haritasını, denetimsiz kümelemedeki ilk ayırıcıyı kullanarak yüksek ve düşük metilasyon gruplarına bölün.
NOT: Kantitatif metilasyona özgü PCR (qMSP) ile doğrulama için, organizatör bölgesindeki CpG adalarına göre daha büyük bir β değerine ve qMSP için astar ve prob tasarımına uygunlukları nedeniyle genleri seçin.
5. Kantitatif Metilasyona Özgü PCR (qMSP)
- Her gen analizinde organizatör bölgesinin metilasyonunu değerlendirmek için qMSP'de floresan bazlı gerçek zamanlı PCR için bir şablon olarak adım 3.3'teki bisülfit modifiye DNA'yı kullanın.
- 96-iyi gerçek zamanlı PCR cihazı kullanarak qMSP gerçekleştirin (Bkz. Malzeme Tablosu).
- 200 nM ileri astar, 200 nM ters astar ve 80 nM prob kullanarak bisülfit modifiye DNA'daki hedef genin promotör metilasyon durumunu kontrol edin. Ana karışımı 16,6 mM (NH4)2SO4, 67 mM Tris pH 8,8, 10 mM β-mercaptoetanol, 10 nM floresan, 0,166 mM her deoksinükleotid trifosfat ve 0,04 U/μl DNAas polimeraz ile hazırlayın (Bkz. Malzemeler Tablosu). Tüm tahliller için her kuyudaki son reaksiyon hacmi 25 μL'dir.
- QMSP'nin bisikletini 5 dakika boyunca 95 °C, ardından 15 s,60 °C ve 1 dk için 72 °C'lik 55 devir gerçekleştirin.
NOT: Hedef gen, daha büyük beta değerlerine sahip olma, organizatör bölgesindeki CpG adaları ile ilgili olması ve qMSP için astar ve prob tasarımına uygun olması kriterlerine göre seçilmelidir.
- Pozitif metilasyon kontrolü olarak CpG Methylase (M.SssI) ile tedavi edilen Insan genomik kullanın (Bkz. Malzeme Tablosu).
NOT: Metilasyonun nihai nicelemi bağıl metilasyon değeri (RMV) olarak tanımlanır ve her metilasyon tespiti için 2-ΔΔCt olarak hesaplanır β-Actin (ACTB)18için ortalama Ct ile karşılaştırıldığında. Astar ve sonda dizileri Tablo 1'degösterilmiştir. Tespit edilemeyen çoğaltmalar için 100'lük bir Ct kullanılır ve bu da sıfıra yakın 2-ΔΔCt değeri verir. Aşağıdaki formül kullanılır: ortalama 2–ΔΔCt (RMV) = (2–ΔΔ Ct_replicate_1 + 2–ΔΔ Ct_replicate_2 + 2–ΔΔ Ct_replicate_3)/318.
Sonuçlar
Eğitim kohortundaki 48 mide kanseri hastasının özellikleri şunlardır (Tablo 2): Hastaların ortanca yaşı 74 (52-89 yıl) idi ve kohortta 38 erkek (%79.2) ve 10 kadın (%20.8) yer aldı. 35 hasta (%72.9) idi primer mide kanseri ve 13 hastada (%27.1) kalıntı mide kanseri (primer mide kanseri: midede metastatik olmayan bir malignitenin ilk ortaya çıkması; kalıntı mide kanseri: distal gastrektomiden 5 yıldan fazla bir süre sonra gelişen kalıntı mide kanseri, orijinal rezeksiyon nedeni ne olursa olsun19). 23 hasta (%47.9) idi lenf nodaz metastazı ve 25 hasta (%52.1) Olma -dan. İlk olarak, tüm 48 örnek (eğitim kohort) aykırı ların belirlenmesi için yüklendi(Şekil 1A). İki örnek, diğerlerinden iki standart sapmadan daha büyük zirveler verdi ve bu örnekler kaldırıldı(Şekil 1B). Bu nedenle, 46 örnek DNA organizatörü hipermetilasyon tarafından kümelenmiştir. Ortaya çıkan ısı haritası yüksek ve düşük metilasyona göre iki gruba ayrılmıştır(Şekil 2). Bu ısı haritası, diferansiyel metilasyon analizinde transkripsiyonel başlangıç alanının (TSS) 1.500 bp içinde en iyi 50 probun görüntülenmesine olanak sağlar. Yüksek ve düşük metilasyon grupları agresif malign fenotip ile ilişkili klinikopatolojik faktörlerde farklılık göstermektedir. Yani, kanser türü (primer mide kanseri: PGC) (p = 0.01, oran oranı = 9.09 (1.67-50.00)) ve lenf nodu metastazı varlığı (pozitif) (p = 0.03, oran oranı = 6.82 (1.16-40.08)) klinikopatolojik faktörler çok değişkenli analizde kovariat olarak kullanıldığında anlamlı bağımsız tahmin faktörleri olarak ortaya çıkmıştır(Tablo 3). Son olarak, EPB41L3 geni20,21 (Tablo 1'degösterilen astar ve prob dizileri) eğitim kohortunu mikrodizi analizinde yüksek ve düşük metilasyon gruplarına kodlamakla güçlü bir şekilde ilişkilendirilmiştir. QMSP kullanılarak test kohortundaki (126 örnek) EPB41L3 mikrodizi analizinin sonuçları doğrulandı. Test kohortundaki hastaların özellikleri Tablo 4'tegösterilmiştir. PGC dokularında EPB41L3 rmvs anlamlı olarak tek değişkenli analizde kalıntı mide kanseri (RGC) daha yüksekti (p = 0.01)(Şekil 3A). Benzer şekilde, lenf nodu metastazı olan örneklerdeki RMV'ler lenf nodu metastazı olmayanlara göre anlamlı olarak yüksekti (p = 0.03)(Şekil 3B). Bu şekilde, DNA metilasyon genom u çapında analiz bize Gi maligniteli hastalarda belirli klinik durumu karakterize belirli genleri belirlemek için yardımcı olabilir.
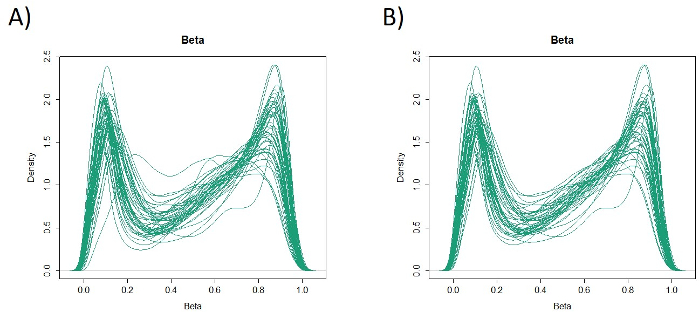
Şekil 1: 48 örnekte beta değerleri (eğitim kohort). 48 numunenin tümü (eğitim kohort) yüklendi ve aykırılar incelendi (A). İki örnek, aykırı olan zirveleri vardı ve bu kaldırıldı(B). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
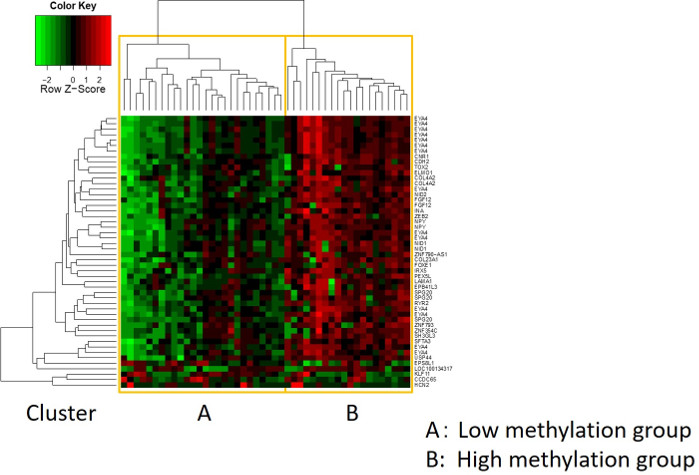
Şekil 2: Ortaya çıkan ısı haritası. Geri kalan 46 örnek DNA organizatörü hipermetilasyon tarafından kümelenmiştir. Isı haritası yüksek ve düşük metilasyon gruplarına ayrılmıştır. Bu ısı haritası, diferansiyel metilasyon analizinde transkripsiyonel başlangıç alanının (TSS) 1.500 bp içinde en iyi 50 probun görüntülenmesine olanak sağlar. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
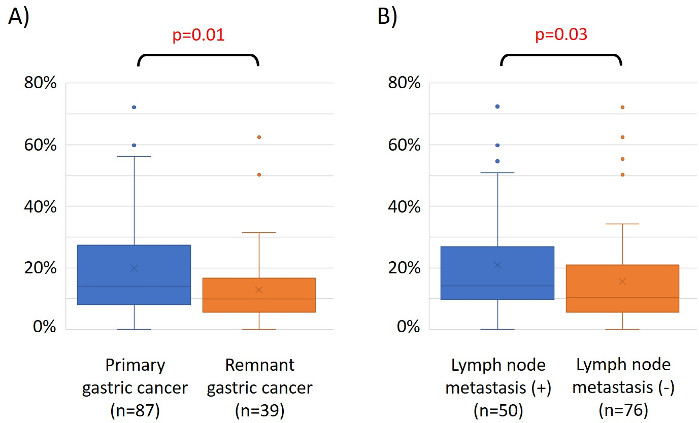
Şekil 3: Primer mide kanserinde EPB41L3 için bağıl metilasyon değerleri (RMV) ve kalıntı mide kanseri (RGC) ve lenf nodülü metastazı olan ve olmayan olgularda. Test kohortundaki EPB41L3 mikrodizi analizinin sonuçları (126 örnek) qMSP kullanılarak doğrulandı. (A) Tek değişkenli analizde, PGC dokularında EPB41L3 rmv'leri RGC'dekilerden anlamlı olarak yüksekti (p = 0.01). (B) Benzer şekilde, lenf nodu metastazı olan örneklerde rcv'ler lenf nodu metastazı olmayanlara göre anlamlı olarak yüksekti (p = 0.03). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Gen | İleri 5' - 3' | Ters 5' - 3' | Sonda |
| B-ACTIN | TAG GGA GTA TAT AGG TTG GGG AAG TT | AAC ACA CAA TAA CAA ACA CAA ATT CAC | \56-FAM\ TGT GGG GTG \ZEN\ GTG ATG GAG GAG GTT TAG \3IABkFQ\ |
| EPB41L3 | GGG ATA GTG GGG TTG ACG C | ATA AAA ATC CCG ACG AAC GA | AAA TTC GAA AAA CCG CGC GAC GCC GAA ACC A |
Tablo 1: Astar ve sonda dizileri.
| Klinikopatolojik faktörler | Değişken | |
| Yaş | 74 (52 - 89) * | |
| Cinsiyet | Erkek / Kadın | 38 (79.2%) / 10 (20.8%) |
| Türü | PGC / RGC | 35 (72.9%) / 13 (27.1%) |
| Lenf nodazı metastazı | (+) / (-) | 23 (47.9%) / 25 (52.1%) |
| PGC: Primer mide kanseri, RGC: Kalıntı mide kanseri | ||
| * Ortanca (minimum-maksimum) | ||
Tablo 2: Eğitim kohortundaki mide kanseri olan 48 hastanın özellikleri.
| P değeri | Oran oranı | %95 Güven aralığı | |
| Türü (PGC) | 0.01 | 9.09 | 1.67 – 50.00 |
| Lenf nodazı metastazı (+) | 0.03 | 6.82 | 1.16 – 40.08 |
| PGC: Primer mide kanseri | |||
Tablo 3: Yüksek metilasyon grubu için tahminfaktörleri (B Kümesi).
| Klinikopatolojik faktörler | Değişken | |
| Yaş | 71 (33 - 86) * | |
| Cinsiyet | Erkek / Kadın | 96 (76.2%) / 30 (23.8%) |
| Türü | PGC / RGC | 87 (69.0%) / 39 (31.0%) |
| Lenf nodazı metastazı | (+) / (-) | 50 (39.7%) / 76 (60.3%) |
| PGC: Primer mide kanseri, RGC: Kalıntı mide kanseri | ||
| * Ortanca (minimum-maksimum) | ||
Tablo 4: Test kohortundaki mide kanseri olan 126 hastanın özellikleri.
Tartışmalar
DNA metilasyon genom u analizinden doğru sonuçlar elde etmek için üç kritik adım vardır. Bunlardan ilki, temsili H&E lekeli kesitlere dayalı tercihen iki nitelikli patolog tarafından tümör bölgesinin makrodisyonudur. Yanlış makrodisksiyon komşu kanser olmayan dokularda kontaminasyona neden olabilir, bu da güvenilmez sonuçlar doğurur; bu nedenle dikkatli makrodisyon gereklidir. İkincisi DNA kalitesinin değerlendirilmesidir (kalite kontrolü: QC). QC (∆Cq > 5.0) başarısız örnekler düşük kaliteli veri verebilir. Bu nedenle, ∆Cq > 5.0 olan örnekler çıkarılmalı ve diğerleri kullanılmalıdır. Üçüncü adım, metilasyonlu sinyal / toplam (metillenmiş + metillenmemiş) sinyal17olarak dizi platformu yazılımı için bir veri analiz aracı tarafından belirlenen β değeri, hesaplanmasıdır. β değeri 0-1 (veya %0-%100) arasında değişmektedir, bu da biyolojik olarak17olarak yorumlanması kolaydır. Yüksek heteroscedasticity metilasyon aralığı aşırı (β = 0 veya β = 1) de numuneler arasında varyans son derece17azalır anlamına gelir beri bu değer ile ana sorun, kötü istatistiksel özellikleri vardır. Buna ek olarak, örnekleme kalitesinin düşük olması nedeniyle, β değerleri tekrarlanabilir bifatik zirvelerigöstermeyebilir 22, ve bu zirveleri olmayan örnekler daha fazla çalışma dışında tutulmalıdır. Buna ek olarak, hedef gen, daha büyük beta değerlerine sahip olma, organizatör bölgesindeki CpG adaları ile ilgili olması ve qMSP için astar ve prob tasarımına uygun olması kriterlerine göre seçilmelidir.
İnsan genomundaki cpg locusundaki DNA metilasyonunun değerlendirilmesi, belirli genomik lokusu incelemek için mikrodizi tabanlı teknoloji ile sabit sayıda prob ile gerçekleştirilir. Düşük maliyeti, az miktarda DNA'sı gerekli olması ve birçok klinik numunenin yüksek işlem lemesine olanak sağlayan numune işlem süresinin az olması nedeniyle epigenom çapında ilişki çalışmalarında (EWAS) en yaygın kullanılan yöntemdir23. Ancak, tek bir CpG locus DNA metilasyon karmaşık değerlendirilmesi için bir dizi platformu epigenetik olarak değiştirilmiş loci için probların sayısı ve özgüllüğü ile sınırlıdır, hangi bazı genomik bölgelerin keşfini önler. WGBS genellikle genomik kapsama geniş spektrumu nedeniyle altın standart yöntem olarak görülüyor10,11. Ancak, bu yöntem önemli bir maliyet ve numuneler10,11çok sayıda analizi için nispeten uzun bir işlem süresi vardır. Böylece, her zaman mümkün değildir. Buna karşılık, insan genomundaki tek bir CpG locusunda DNA metilasyonunun karmaşık bir şekilde değerlendirilmesi için kullanılan dizi platformu, maliyet ve genomik kapsama açısından kullanım için makuldür. Son zamanlarda, en son yükseltilmiş boncuk yongaları24kullanıma hazır aldık. Bu tahliller, büyük örnek popülasyonları için ideal genom çapında ilişkilendirme çalışması (GWAS) elde edebilen neredeyse iki katına ölçülen CpG bölgelerini analiz etmemize yardımcı olabilir.
Özetle, insan genomundaki tek bir CpG lokusta DNA metilasyonunun karmaşık değerlendirilmesi için dizi platformu ile DNA metilasyon genom u ğr'ın analizi gastrointestinal kanserde epigenetik biyobelirteçler hakkında önemli bilgiler sağlayabilir. WGBS ile karşılaştırıldığında, bu yöntem uygun maliyetlidir ve numune işleme süresini azaltır. Bu nedenle, bir CpG lokus DNA metilasyon tespiti için bu yöntem yaygın epigenetik biyomarker araştırmada kullanılması muhtemeldir.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Biz özellikle Cerrahi Bölümü, Sidney Kimmel Kapsamlı Kanser Merkezi Johns Hopkins Üniversitesi Tıp Fakültesi yararlı tartışmalar ve teknik destek için tüm üyelerine müteşekkiriz. Ayrıca bisülfit tedavisi ve qMSP prosedürleri cömert teknik rehberlik için Kristen Rodgers teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| (NH4)2SO4 | Sigma-Aldrich | 14148 | Step 5.2. |
| 10% Sodium dodecyl sulfate (SDS) | Quality Biological | 351-032-721 EA | Step 2.1. |
| 100 % Ethanol | Sigma-Aldrich | 24194 | Step 1.7. |
| ABI StepOnePlus Real-Time PCR System | Applied BioSystems | 4376600 | Step 5.2. 96-well Real-Time PCR instrument |
| CT conversion reagent | Zymo Research | D5001-1 | Step 3.2.3. |
| Deoxynucleotide triphosphate (dNTP) | Invitrogen | 10297-018 | Step 5.2. |
| DEPC-treated water | Quality Biological | 351-068-131 | Step 2.1. |
| Ethylenediaminetetraacetic acid (EDTA) | Corning | 46-034-CL | Step 2.1. |
| EZ DNA Methylation Kit | Zymo Research | D5002 | Step 3.2. |
| Fluorescein | Bio-Rad | #1708780 | Step 5.2. |
| GenomeStudio | omicX | OMICS_00854 | Step 4.3. Data analysis tool for an array platform as a β-value, with a range from 0 to 1 |
| Human genomic DNA | New England Bio Labs | N4002S | Step 5.3. |
| Infinium HD FFPE QC Kit | Illumina | WG-321-1001 | Step 4.1. FFPE QC assay on a real-time PCR amplification |
| Infinium HumanMethylation450 assay | Illumina | WG-314 | Step 4.2. Array platform for complex evaluation of DNA methylation to assess the methylation status of >450,000 CpG sites in the genome |
| LightCycler 480 | Roche | 5015278001 | Step 4.1. |
| M-Binding Buffer | Zymo Research | D5002-3 | Step 3.2.6. |
| M-Desulphonation Buffer | Zymo Research | D5002-5 | Step 3.2.9. |
| M-Dilution Buffer | Zymo Research | D5002-2 | Step 3.2.1. |
| Minfi package | Bioconductor | N/A | Step 4.4. |
| M-Wash Buffer | Zymo Research | D5002-4 | Step 3.2.10. |
| Platinum Taq polymerase | ThermoFisher Scientific | 10966-034 | Step 5.2. |
| Proteinase K | New England Biolabs | P8107S | Step 2.8. |
| Single-use polypropylene (Eppendorf) tube | Eppendorf | 24533495 | Step 2.5.2. |
| Tris hydrochloride (Tris-HCL) 2 M pH 8.8 | Quality Biological | 351-092-101 | Step 2.1. |
| Xylene | Sigma-Aldrich | 214736 | Step 1.3. |
| Zymo Spin 1 Column | Zymo Research | C1003 | Step 3.2.6. |
| β-Mercaptoethanol | Sigma-Aldrich | M3148 | Step 5.2. |
Referanslar
- Qiu, J. Epigenetics: unfinished symphony. Nature. 441 (7090), 143-145 (2006).
- Okugawa, Y., Grady, W. M., Goel, A. Epigenetic alterations in colorectal cancer: emerging biomarkers. Gastroenterology. 149 (5), 1204-1225 (2015).
- Ahuja, N., Li, Q., Mohan, A. L., Baylin, S. B., Issa, J. P. Aging and DNA methylation in colorectal mucosa and cancer. Cancer Research. 58 (23), 5489-5494 (1998).
- Hsieh, C. J., et al. Hypermethylation of the p16INK4a promoter in colectomy specimens of patients with long-standing and extensive ulcerative colitis. Cancer Research. 58, 3942-3945 (1998).
- Ushijima, T., Okochi-Takada, E. Aberrant methylations in cancer cells: where do they come from. Cancer Science. 96 (4), 206-211 (2005).
- Coppedè, F. Epigenetic biomarkers of colorectal cancer: Focus on DNA methylation. Cancer Letters. 342 (2), 238-247 (2014).
- Vasilatou, D., Papageorgiou, S. G., Dimitriadis, G., Pappa, V. Epigenetic alterations and microRNAs: new players in the pathogenesis of myelodysplastic syndromes. Epigenetics. 8 (6), 561-570 (2013).
- Ma, X., Wang, Y. W., Zhang, M. Q., Gazdar, A. F. DNA methylation data analysis and its application to cancer research. Epigenomics. 5 (3), 301-316 (2013).
- Morris, T. J., Beck, S. Analysis pipelines and packages for Infinium HumanMethylation450 BeadChip (450k) data. Methods. 72, 3-8 (2015).
- Rauluseviciute, I., Drabløs, F., Rye, M. B. DNA methylation data by sequencing: experimental approaches and recommendations for tools and pipelines for data analysis. Clinical Epigenetics. 11 (1), 193 (2019).
- Cazaly, E. Making sense of the epigenome using data integration approaches. Frontiers in Pharmacology. 10, 126 (2019).
- Leal, A., Sidransky, D., Brait, M. Tissue and cell-free DNA-based epigenomic approaches for cancer detection. Clinical Chemistry. 66 (1), 105-116 (2020).
- Song, W., Ren, J., Wang, W. J., Wang, C. T., Fu, T. Genome-wide methylation and expression profiling identify a novel epigenetic signature in gastrointestinal pan-adenocarcinomas. Epigenomics. 12 (11), (2020).
- Wong, E. M., et al. Tools for translational epigenetic studies involving formalin-fixed paraffin-embedded human tissue: applying the Infinium HumanMethyation450 Beadchip assay to large population-based studies. BMC Research Notes. 8, 543 (2015).
- . Illumina Available from: https://jp.support.illumina.com/downloads/infinium_humanmethylation450_product_files.html (2020)
- Fackler, M. J., et al. Genome-wide methylation analysis identifies genes specific to breast cancer hormone receptor status and risk of recurrence. Cancer Research. 71 (19), 6195-6207 (2011).
- Dedeurwaerder, S., et al. A comprehensive overview of Infinium HumanMethylation450 data processing. Briefings in Bioinformatics. 15 (6), 929-941 (2014).
- Hulbert, A., et al. Early detection of lung cancer using DNA promoter hypermethylation in plasma and sputum. Clinical Cancer Research: An Official Journal of the American Association for Cancer Research. 23 (8), 1998-2005 (2017).
- Shimada, H., Fukagawa, T., Haga, Y., Oba, K. Does remnant gastric cancer really differ from primary gastric cancer? A systematic review of the literature by the Task Force of Japanese Gastric Cancer Association. Gastric Cancer: Official Journal of the International Gastric Cancer Association and the Japanese Gastric Cancer Association. 19 (2), 339-349 (2016).
- Eijsink, J. J., et al. Detection of cervical neoplasia by DNA methylation analysis in cervico-vaginal lavages, a feasibility study. Gynecologic Oncology. 120 (2), 280-283 (2011).
- Sugimoto, K., et al. DNA methylation genome-wide analysis in remnant and primary gastric cancers. Gastric Cancer: Official Journal of the International Gastric Cancer Association and the Japanese Gastric Cancer Association. 22 (6), 1109-1120 (2019).
- Wang, Z., Wu, X., Wang, Y. A framework for analyzing DNA methylation data from Illumina Infinium HumanMethylation450 BeadChip. BMC Bioinformatics. 19, 115 (2018).
- Teh, A. L., et al. Comparison of methyl-capture sequencing vs. Infinium 450K methylation array for methylome analysis in clinical samples. Epigenetics. 11 (1), 36-48 (2016).
- Zhou, W., Laird, P. W., Shen, H. Comprehensive characterization, annotation and innovative use of Infinium DNA methylation BeadChip probes. Nucleic Acids Research. 45 (4), 22 (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır