Method Article
消化器癌におけるDNAメチル化のゲノムワイド解析
要約
ここでは、消化管癌におけるDNAメチル化のゲノム全体解析の手順について説明する。この手順は、遺伝子のメチル化パターンと胃腸癌の発がんに寄与する因子との関係を調査する研究と関連するものである。
要約
DNAメチル化は、生物学的に意味のある重要なエピジェネティックな変化であり、がん研究の頻繁な焦点です。ゲノムワイドDNAメチル化は、消化器(GI)悪性腫瘍のメチル化状態を正確に分析するのに有用な手段です。DNAメチル化解析の複数の潜在的な翻訳用途を考えると、DNAメチル化研究に新しい臨床医などが、これらのゲノム全体の分析がどのように行われるかを段階的に理解できる必要があります。このプロトコルの目的は、この方法が GI 悪性腫瘍におけるバイオマーカー識別にどのように使用されるかについて詳しく説明することです。重要なことは、ゲノム全体の解析において正確な結果を得るために必要な3つの重要なステップを説明することです。明らかに簡潔に書かれているこれらの3つの方法は、しばしば欠けており、エピジェネティック研究に新しいものには目立たない。GI悪性腫瘍(胃癌)の48サンプルを用いて、GI悪性腫瘍に対してゲノム全体のDNAメチル化解析を実際に行う方法を強調しました。
概要
エピジェネティクスは、DNA1の配列を変化させることなく、遺伝子機能の遺伝性変化を指す。このような変化は、DNAのメチル化に起因する可能性があり、DNAベース上のメチル基はクロマチンパッキングの変化を通じて遺伝子発現を変化させる可能性があります。この効果が腫瘍抑制遺伝子2の発現変化をもたらすと、癌の発症と進行が起こり得る。老化と慢性炎症は、癌の原因とヒト3、4、5におけるDNAメチル化の変化の主な理由である。これにより、がん診断におけるバイオマーカーとしてのDNAメチル化の利用、治療・予防のターゲットとしての利用が可能になります。早期発見と癌予後のために、DNAメチル化は腫瘍、血液、尿および便サンプル6で測定され、脱メチル化剤は骨髄異形成症候群7などの白血病の治療に使用されている。
ヒトゲノム中の個々のCpG遺伝子座におけるDNAメチル化の複雑な評価にアレイプラットフォームを用いたゲノムワイドDNAメチル化解析を利用して、がんエピジェネティクスの探索を可能にするゲノムDNA8,9における450,000以上のCpG部位のメチル化状態を調べることができます(材料表を参照)。全ゲノムバイサルファイトシーケンシング(WGBS)技術は、エピジェネティクス10,11の分野における我々のアプローチを変えた。しかしながら、多数のサンプル10,11のエピジェネティック解析に相当なコストと処理時間の点で、技術にはいくつかの欠点がある。したがって、アレイプラットフォームは、ヒトゲノムにおけるDNAメチル化の複雑な評価に対してより実現可能である。ゲノム全体のメチル化分析のアプローチの利用可能性はここ数年で改善され、DNAメチル化が癌の発症と進行にどのように寄与するかについて知識を広げることができる12.マイクロアレイプラットフォームアプローチの最近の進歩は、消化器癌における新しいエピジェネティックシグネチャを同定するためのゲノムワイドメチル化分析の根拠を提供する13。このプロトコルの目的は、この方法がGI悪性腫瘍におけるバイオマーカー同定にどのように使用されるかについて詳細に説明することです。
プロトコル
すべての手続きは、機関の人間研究倫理委員会の倫理基準に従っていました。この研究は、順天堂大学静岡病院の機関審査委員会によって承認され、遡及的な設計のために書面によるインフォームド・コンセントが放棄された。
1. スライドを洗う
- 10 μmの未染色ホルマリン固定パラフィン埋め込み(FFPE)セクションを用意します。
- スライドをガラススライドホルダに配置する:ティッシュが最小で、より多くのスライドが必要でない限り、約3-5最大の断面スライドを使用してください。
- スライドホルダーにキシレンを充填し、スライド上のすべての組織が水没していることを確認します。15分間座ります。
- 15分後、スライドが抜けないようにピペットチップでスライドを保持したキシレンを注ぎます。
- 以前と同じレベルに多くのキシレンを注ぎます。さらに15分間座ります。
- 15分後、再びキシレンを注ぎます。
- スライドホルダーに100%エタノール(EtOH)を充填し、スライド上のすべての組織が完全に水没していることを確認します。3分間座ります。
- 慎重にスライドを保持しながら、EtOHを注ぎます。より多くのEtOHで同じレベルにリフィルします。2分間座ります。
- もう一度EtOHを注ぎ、スライドを取り外します。清潔なペーパータオルの上に慎重に顔を上げて乾かします。10分間座ります。
2. スライドを削る
- 650 mLのジエチルピーロカーボネート(DEPC)処理水、100 mLのエチレンジアミンテトラ酢酸(EDTA)、50mlのトリス塩酸塩(トリスHCL)2 MpH 8.8、および200 mLのドデシルスルフェートナトリウム(SD)を用いて消化/リシスバッファーを調製する( 材料)。
- 1.5 mLの単回使用ポリプロピレンチューブに80 μLの消化/リシスバッファーを充填します( 材料表を参照)。
- きれいなピペットチップをバッファーに入れます。
- 適切なH&E染色部に従ってマクロ解剖に最も適した腫瘍領域の癌組織を同定する。
注:マクロ解剖のための腫瘍領域は、好ましくは2人の修飾された病理学者によって同定されるべきである。 - マークされたH&E染色部に基づいて癌組織をマクロ解剖する。
- きれいなカミソリの刃を取り、滑らかにスライドから癌組織を削り取り、それを一枚に保ち、作業しやすいようにします。
- バッファーを含む使い捨てのポリプロピレンチューブからピペットチップを取り出し、それを使用して廃棄された組織をバッファバイアルに移します( 材料表を参照)。
注:濡れた先端は、転送が容易になる組織を引き付ける必要があります。
- 残りのスライドでステップ 2.5 を繰り返します。
- すべての組織が使い捨てのポリプロピレンチューブに入った後、ティッシュが完全に水没し、管の壁に付着していないことを確認するために先端を使用してください。
- バイアルに20μLのサチリシン関連セリンプロテアーゼを加え、軽くフリックして混ぜます( 材料表を参照)。
- バイアルを55°Cのヒートブロックに4時間または一晩置きます。2時間後に少し渦を作るようにしてください。
3. バイサルファイト治療
- 消化した組織溶液の45 μLをサンプルとして使用します。
- 製造者の指示に従って、バイサルファイト変換キットで試薬を使用してバイサルファイト処理を行います( 材料表を参照)。
- 5 μL の希釈バッファーを DNA サンプルに加え、37 °C で 15 分間インキュベートします ( 材料表を参照)。
- サンプルがインキュベートしている間に、750 μLのdH2Oと210 μLの希釈バッファーをCT変換試薬の1つのチューブに加えて、バイサルファイト変換試薬を調製します( 材料表を参照)。10分間ボルテックスでチューブを混ぜます。
- 15分インキュベーション後、調製したCT変換試薬を100μLずつ各サンプルに加え、反転によって混合します。
- 50°Cの暗闇の中で12〜16時間、サンプルをインキュベートする。
- インキュベーション後、サンプルを取り出し、氷の上に10分間置きます。
- 400 μL の結合バッファーを追加し、各サンプルを上下にピペット処理して混合します ( 資料表を参照)。各サンプルをスピンカラムにロードし、そのカラムを 2 mL コレクションチューブに配置します( 材料表を参照)。
- 1分間、全速速(10,000 x g)でサンプルを遠心分離し、流量を廃棄します。
- 各カラムに200 μLの洗浄バッファを追加し、フルスピードで1分間回転し、フロースルーを破棄します( 材料表を参照)。
- 各カラムに200 μLの脱サル化バッファを加え、カラムを室温で15分間放置します( 材料表を参照)。インキュベーション後、カラムを全速速度で1分間回転させ、フロースルーを廃棄します。
- 各カラムに200 μLの洗浄バッファーを追加し、1分間フルスピードでスピンします( 材料表を参照)。
- この手順をもう一度繰り返します。
- 各カラムに46 μLのdH2Oを加え、各カラムを新しい滅菌1.5 mLの単独使用ポリプロピレンチューブに入れる( 材料表を参照)。DNAを溶出させるために2分間、各チューブを回転させます。
- シングルユースポリプロピレンチューブから各スピンカラムを取り外し、廃棄します( 材料表を参照)。DNAは分析の準備が整いました。
ヒトゲノムにおけるCpG遺伝子座におけるDNAメチル化評価用アレイプラットフォーム
- リアルタイムPCR増幅および検出機器でのFFPE QCアッセイを使用したDNA(品質チェック:QC)の品質を評価し、その後のデータ分析はメーカーの指示に従って実行されます( 材料表を参照)。
注意: 推奨される 5.0 より小さい∆Cq 値を持つサンプルは、さらに14. - DNAメチル化の複雑な評価のために配列プラットフォームを用いてサンプルを解析し、ゲノム中の>450,000 CpGサイトのメチル化状態を評価します( 材料表を参照)。アレイ プラットフォームの製品情報シート、データシート、および製品ファイルは15.
注:アッセイは、FFPE材料14のメーカーの指示に従って行われます。 - 配列プラットフォームのデータ分析ツールで、高位および低位の遺伝子座メチル化を、それぞれ 0 ~ 1 の範囲でβ値として計算します ( 材料表を参照)。市販のソフトウェアを使用して( 材料表を参照)、β値を計算します。
- DNAメチル化プラットフォームの複雑な評価のためにアレイプラットフォーム上で生成されたデータをRソフトウェア環境(Rv.2.15.1)にインポートし、メチル化アレイ16,17を分析するためのツールを用いて処理する。
注: データ分析ツールを使用して生成されるヒートマップでは、列は教師なしクラスタリングによって順序付けされますが、行の順序は、上から下への差分メチル化の t 統計量の有意度の減少に基づいています。 - 教師なしクラスタリングの最初の差別化器を使用して、ヒートマップを高メチル化グループと低メチル化グループに分割します。
注: 定量的メチル化特異的 PCR (qMSP) を使用した検証では、プロモーター領域の CpG 島に対するβ値の大きい値に基づいて、qMSP のプライマーおよびプローブ設計に適合性があるため、遺伝子を選択します。
5. 定量的メチル化特異的 PCR (qMSP)
- qMSPの蛍光ベースのリアルタイムPCRのテンプレートとして、ステップ3.3のバイサルファイト修飾DNAを使用して、各遺伝子解析におけるプロモーター領域のメチル化を評価します。
- 96 ウェルリアルタイム PCR 装置を使用して qMSP を実行します ( 資料表を参照)。
- 200 nMフォワードプライマー、200 nMリバースプライマー、および80 nMプローブを用いて、バイサルファイト修飾DNA上の標的遺伝子のプロモーターメチル化状態を確認します。マスターミックスを準備し、16.6 mM (NH4)2SO4、67mM Tris pH 8.8、10 mM β-メルカプトエタノール、10 nMフルオレセイン、各デオキシヌクレオチド三リン酸の0.166 mM、および0.04 U/μlのDNAポリメラーゼ(材料表を参照)を使用してマスターミックスを準備します。すべてのアッセイの各ウェルの最終反応容積は25μLである。
- qMSP のサイクリングは、95 °C で 5 分間、95 °C が 15 s、60 °C が 1 分間、72 °C が 1 分間続きます。
注:標的遺伝子は、より大きなベータ値を有し、プロモーター領域のCpG島に関連し、qMSPのプライマーおよびプローブ設計に適しているという基準に基づいて選択されるべきである。
- 正のメチル化制御としてCpGメチル化(M.SssI)で処理されたヒトゲノムを使用してください( 材料表を参照)。
注:メチル化の最終的な定量は、相対的なメチル化値(RMV)として定義され、βアクチン(ACTB)18の平均Ctと比較して、各メチル化検出複製に対して2-ΔΔCtとして計算される。プライマーとプローブ配列を表1に示します。検出されない反復には 100 の Ct が使用され、0 に近い値は 2–ΔΔCtになります。平均 2–ΔΔCt (RMV) = (2-ΔΔδ Ct_replicate_1 + 2-ΔΔ Ct_replicate_2 + 2–ΔΔ Ct_replicate_3)/318を使用します。
結果
トレーニングコホートにおける胃癌患者48名の特徴は以下の通りです(表2):患者の年齢の中央値は74歳(52~89歳)、コホートには38人の男性(79.2%)、10人の女性(20.8%)が含まれていました。患者は35人(72.9%)原発性胃癌と13人の患者(27.1%)残管胃癌(原発性胃癌:胃内の非転移性悪性腫瘍の最初の発生;残骸胃癌:遠位胃切除術後5年以上発症した残骸胃の癌は、元の切除19の理由に関係なく)。患者は23人(47.9%)リンパ節転移と25人の患者(52.1%)なし。まず、外れ値を識別するために48個のサンプル(トレーニングコホート)をすべて積み込みました(図1A)。2つのサンプルは、他からずれた2つの標準偏差より大きいピークを与え、これらのサンプルは除去された(図1B)。したがって、46個のサンプルを、DNAプロモーターによるハイパーメチル化によってクラスター化した。結果として得られたヒートマップは、高メチル化と低メチル化に基づいて2つのグループに分けられた(図2)。このヒートマップにより、微分メチル化解析で転写開始部位(TSS)の1,500 bp以内の上位50個のプローブを可視化できます。高メチル化基と低メチル化基は、積極的な悪性表現型に関連する臨床病理学的要因が異なった。すなわち、癌の種類(原発性胃癌:PGC)(p=0.01、 オッズ比= 9.09 (1.67-50.00))およびリンパ節転移(陽性)の存在(p = 0.03、オッズ比= 6.82(1.16-40.08))は、臨床病理学的因子が多量共同変量の共変量として使用された場合、有意な独立した予測因子として浮上した( 表3)。最後に、マイクロアレイ解析において、EPB41L3遺伝子20,21(表1に示すプライマーおよびプローブ配列)を同定し、マイクロアレイ解析においてトレーニングコホートを高低メチル化基に体系化することに強く関連付けられることを確認した。qMSPを用いて、試験コホート中のEPB41L3(126サンプル)のマイクロアレイ解析の結果を検証した。試験コホートにおける患者の特徴を表4に示す。PGC組織におけるEPB41L3のMPVは、1変量分析において残胃癌(RGC)のMPVよりも有意に高かった(図3A)。同様に、リンパ節転移を有するサンプル中のRmvは、リンパ節転移を伴わないものよりも有意に高かった(図3B)。このように、DNAメチル化ゲノム全体の解析は、GI悪性腫瘍患者における特定の臨床状態を特徴付けるために特定の遺伝子を同定するのに役立ちます。
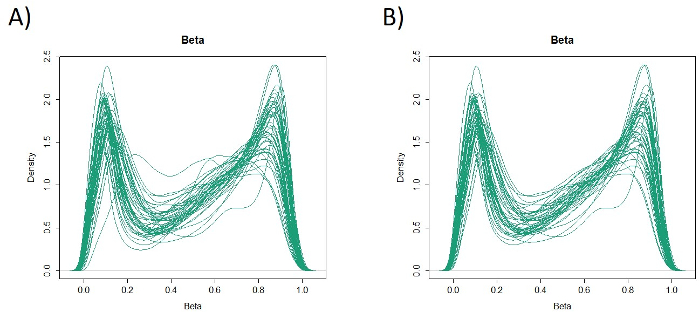
図1:48サンプル(トレーニングコホート)のベータ値。48個のサンプル(トレーニングコホート)をすべて積み込み、外れ値を調べた(A)。2つのサンプルは外れ値であるピークを持ち、これらは取り除かれた(B)。この図の大きなバージョンを表示するには、ここをクリックしてください。
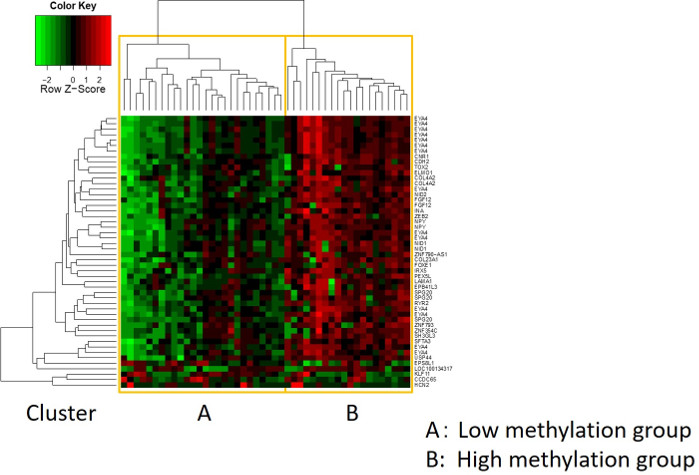
図 2: 結果のヒートマップ。残りの46サンプルは、DNAプロモーターによるハイパーメチル化によってクラスター化した。ヒートマップは、高メチル化基と低メチル化基に分けた。このヒートマップにより、微分メチル化解析で転写開始部位(TSS)の1,500 bp以内の上位50個のプローブを可視化できます。 この図の大きなバージョンを表示するには、ここをクリックしてください。
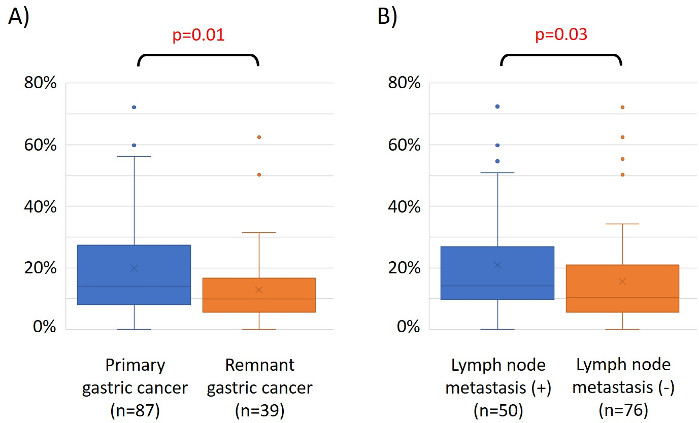
図3:EPB41L3のEPB41L3の相対メチル化値(MV)は、一次胃癌(PGC)対残膜胃癌(RGC)、およびリンパ節転移の有無にかかわらず場合。試験コホート中のEPB41L3(126サンプル)のマイクロアレイ解析の結果をqMSPを用いて検証した。(A) 1変量分析では、PGC組織におけるEPB41L3のRPVは、RGC(p = 0.01)のRPB41L3よりも有意に高かった。(B) 同様に、リンパ節転移を有するサンプル中のRmvは、リンパ節転移のないものよりも有意に高かった(p = 0.03)。 この図の大きなバージョンを表示するには、ここをクリックしてください。
| 遺伝子 | フォワード 5' - 3' | 逆 5' - 3' | プローブ |
| B-アクチン | タグ GGA GTA タット アッグ TTG GGG AAG TT | AAC ACA CAA CaA CaCA CAA ATT CAC | \56-FAM\ TGT GGG GTG \ZEN\ GTG ATGギャグギャグ GTT タグ \3IABkFQ\ |
| EPB41L3 | GGG ATA GTG GGG TTG ACG C | アタ AAA ATC CCG ACG AAC GA | AAA TTC GAA AAA CCG CGC GAC GCC GAA ACC A |
表1:プライマーとプローブ配列。
| 臨床病理学的要因 | 変数 | |
| 年齢 | 74 (52 - 89) * | |
| 性別 | 男性 / 女性 | 38 (79.2%) / 10 (20.8%) |
| 型 | PGC / RGC | 35 (72.9%) / 13 (27.1%) |
| リンパ節転移 | (+) / (-) | 23 (47.9%) / 25 (52.1%) |
| PGC: 原発性胃癌、RGC:残骸胃癌 | ||
| * 中央値(最小最大値) | ||
表2:トレーニングコホートにおける胃癌患者48例の特徴
| P値 | オッズ比 | 95% 信頼区間 | |
| タイプ(PGC) | 0.01 | 9.09 | 1.67 – 50.00 |
| リンパ節転移(+) | 0.03 | 6.82 | 1.16 – 40.08 |
| PGC: 原発性胃癌 | |||
表3:高メチル化基(クラスタB)の予測因子。
| 臨床病理学的要因 | 変数 | |
| 年齢 | 71 (33 - 86) * | |
| 性別 | 男性 / 女性 | 96 (76.2%) / 30 (23.8%) |
| 型 | PGC / RGC | 87 (69.0%) / 39 (31.0%) |
| リンパ節転移 | (+) / (-) | 50 (39.7%) / 76 (60.3%) |
| PGC: 原発性胃癌、RGC:残骸胃癌 | ||
| * 中央値(最小最大値) | ||
表4: 試験コホートにおける胃癌患者126人の特徴.
ディスカッション
DNAメチル化ゲノム全体解析から正確な結果を得るには、3つの重要なステップがあります。第1は、代表的なH&E染色切片に基づく2つの修飾病理医による腫瘍領域のマクロ解剖である。不正確なマクロ解剖は、隣接する非癌性組織との汚染を引き起こす可能性があり、信頼性の低い結果を引き起こす。したがって、慎重なマクロ解剖が必要です。2つ目はDNAの品質評価(品質チェック:QC)です。QC (∆Cq > 5.0) に失敗したサンプルは、質の低いデータを提供する可能性があります。そのため、∆Cq > 5.0 のサンプルを削除し、他のサンプルを使用する必要があります。第3のステップは、β値の計算であり、メチオン信号/合計(メチル化+非メチル化)信号17としてアレイプラットフォームソフトウェアのデータ解析ツールによって決定される。β値の範囲は 0 ~ 1 (または 0%から 100%)この値の主な問題は、その高い不均一性が、メチル化範囲の極端(β = 0またはβ=1)でのサンプル間の分散が17を大幅に減少することを意味するので、その貧弱な統計的特性である。また、サンプルの品質が悪いため、β値は再現可能な二分性ピーク22を示さない可能性があり、そのようなピークのないサンプルはさらなる研究から除外されるべきである。さらに、標的遺伝子は、より大きなベータ値を有し、プロモーター領域におけるCpG島に関連し、qMSPのプライマーおよびプローブ設計に適しているという基準に基づいて選択されるべきである。
ヒトゲノムにおけるCpG遺伝子座におけるDNAメチル化の評価は、特定のゲノム遺伝子座を調査するために、一定量のプローブを用いたマイクロアレイベースの技術を用いて行われる。これは、エピゲノムワイドアソシエーション研究(EWAS)において最も広く使用されている方法であり、その低コスト、少量のDNA必要量、およびサンプル処理時間が著しく短いため、多くの臨床サンプル23のハイスループット処理が可能である。しかし、個々のCpG遺伝子座におけるDNAメチル化の複雑な評価のためのアレイプラットフォームは、エピジェネティックに改変された遺伝子組み換え遺伝子座のプローブの数と特異性によって制限され、ゲノム領域の探索を妨げる。WGBSは一般的に、ゲノムカバレッジ10、11の広いスペクトルのためにゴールドスタンダード法と見なされています。しかしながら、この方法は、多数のサンプル10,11の分析に相当なコストと比較的長い処理時間を有する。したがって、必ずしも実現可能とは限りません。それに比べて、ヒトゲノム中の個々のCpG軌跡におけるDNAメチル化の複雑な評価のためのアレイプラットフォームは、コストおよびゲノムカバレッジの観点から使用することは合理的である。最近、最新のアップグレードされたビーズチップは24を使用する準備が整いました。これらのアッセイは、測定されたCpG部位のほぼ2倍の分析に役立ち、大規模なサンプル集団に対する理想的なゲノムワイドアソシエーション研究(GWAS)を達成することができます。
要約すると、ヒトゲノム中の個々のCpG遺伝子座におけるDNAメチル化を複雑に評価するためのアレイプラットフォームを用いたDNAメチル化ゲノム全体解析は、消化管癌におけるエピジェネティックバイオマーカーに関する重要な情報を提供することができる。WGBSと比較すると、この方法はコスト効率が良く、サンプル処理時間を短縮します。従って、この方法はCpG遺伝子座におけるDNAメチル化の検出方法が、エピジェネティックバイオマーカー研究において広く用いられている可能性が高い。
開示事項
著者らは開示するものは何もない。
謝辞
私たちは、ジョンズホプキンス大学医学部のシドニーキンメル総合がんセンター外科の外科部門のすべてのメンバーに特に感謝しています。我々はまた、亜硫酸塩治療とqMSPの手順に関する寛大な技術的指導のためにクリステン・ロジャースに感謝します。
資料
| Name | Company | Catalog Number | Comments |
| (NH4)2SO4 | Sigma-Aldrich | 14148 | Step 5.2. |
| 10% Sodium dodecyl sulfate (SDS) | Quality Biological | 351-032-721 EA | Step 2.1. |
| 100 % Ethanol | Sigma-Aldrich | 24194 | Step 1.7. |
| ABI StepOnePlus Real-Time PCR System | Applied BioSystems | 4376600 | Step 5.2. 96-well Real-Time PCR instrument |
| CT conversion reagent | Zymo Research | D5001-1 | Step 3.2.3. |
| Deoxynucleotide triphosphate (dNTP) | Invitrogen | 10297-018 | Step 5.2. |
| DEPC-treated water | Quality Biological | 351-068-131 | Step 2.1. |
| Ethylenediaminetetraacetic acid (EDTA) | Corning | 46-034-CL | Step 2.1. |
| EZ DNA Methylation Kit | Zymo Research | D5002 | Step 3.2. |
| Fluorescein | Bio-Rad | #1708780 | Step 5.2. |
| GenomeStudio | omicX | OMICS_00854 | Step 4.3. Data analysis tool for an array platform as a β-value, with a range from 0 to 1 |
| Human genomic DNA | New England Bio Labs | N4002S | Step 5.3. |
| Infinium HD FFPE QC Kit | Illumina | WG-321-1001 | Step 4.1. FFPE QC assay on a real-time PCR amplification |
| Infinium HumanMethylation450 assay | Illumina | WG-314 | Step 4.2. Array platform for complex evaluation of DNA methylation to assess the methylation status of >450,000 CpG sites in the genome |
| LightCycler 480 | Roche | 5015278001 | Step 4.1. |
| M-Binding Buffer | Zymo Research | D5002-3 | Step 3.2.6. |
| M-Desulphonation Buffer | Zymo Research | D5002-5 | Step 3.2.9. |
| M-Dilution Buffer | Zymo Research | D5002-2 | Step 3.2.1. |
| Minfi package | Bioconductor | N/A | Step 4.4. |
| M-Wash Buffer | Zymo Research | D5002-4 | Step 3.2.10. |
| Platinum Taq polymerase | ThermoFisher Scientific | 10966-034 | Step 5.2. |
| Proteinase K | New England Biolabs | P8107S | Step 2.8. |
| Single-use polypropylene (Eppendorf) tube | Eppendorf | 24533495 | Step 2.5.2. |
| Tris hydrochloride (Tris-HCL) 2 M pH 8.8 | Quality Biological | 351-092-101 | Step 2.1. |
| Xylene | Sigma-Aldrich | 214736 | Step 1.3. |
| Zymo Spin 1 Column | Zymo Research | C1003 | Step 3.2.6. |
| β-Mercaptoethanol | Sigma-Aldrich | M3148 | Step 5.2. |
参考文献
- Qiu, J. Epigenetics: unfinished symphony. Nature. 441 (7090), 143-145 (2006).
- Okugawa, Y., Grady, W. M., Goel, A. Epigenetic alterations in colorectal cancer: emerging biomarkers. Gastroenterology. 149 (5), 1204-1225 (2015).
- Ahuja, N., Li, Q., Mohan, A. L., Baylin, S. B., Issa, J. P. Aging and DNA methylation in colorectal mucosa and cancer. Cancer Research. 58 (23), 5489-5494 (1998).
- Hsieh, C. J., et al. Hypermethylation of the p16INK4a promoter in colectomy specimens of patients with long-standing and extensive ulcerative colitis. Cancer Research. 58, 3942-3945 (1998).
- Ushijima, T., Okochi-Takada, E. Aberrant methylations in cancer cells: where do they come from. Cancer Science. 96 (4), 206-211 (2005).
- Coppedè, F. Epigenetic biomarkers of colorectal cancer: Focus on DNA methylation. Cancer Letters. 342 (2), 238-247 (2014).
- Vasilatou, D., Papageorgiou, S. G., Dimitriadis, G., Pappa, V. Epigenetic alterations and microRNAs: new players in the pathogenesis of myelodysplastic syndromes. Epigenetics. 8 (6), 561-570 (2013).
- Ma, X., Wang, Y. W., Zhang, M. Q., Gazdar, A. F. DNA methylation data analysis and its application to cancer research. Epigenomics. 5 (3), 301-316 (2013).
- Morris, T. J., Beck, S. Analysis pipelines and packages for Infinium HumanMethylation450 BeadChip (450k) data. Methods. 72, 3-8 (2015).
- Rauluseviciute, I., Drabløs, F., Rye, M. B. DNA methylation data by sequencing: experimental approaches and recommendations for tools and pipelines for data analysis. Clinical Epigenetics. 11 (1), 193 (2019).
- Cazaly, E. Making sense of the epigenome using data integration approaches. Frontiers in Pharmacology. 10, 126 (2019).
- Leal, A., Sidransky, D., Brait, M. Tissue and cell-free DNA-based epigenomic approaches for cancer detection. Clinical Chemistry. 66 (1), 105-116 (2020).
- Song, W., Ren, J., Wang, W. J., Wang, C. T., Fu, T. Genome-wide methylation and expression profiling identify a novel epigenetic signature in gastrointestinal pan-adenocarcinomas. Epigenomics. 12 (11), (2020).
- Wong, E. M., et al. Tools for translational epigenetic studies involving formalin-fixed paraffin-embedded human tissue: applying the Infinium HumanMethyation450 Beadchip assay to large population-based studies. BMC Research Notes. 8, 543 (2015).
- . Illumina Available from: https://jp.support.illumina.com/downloads/infinium_humanmethylation450_product_files.html (2020)
- Fackler, M. J., et al. Genome-wide methylation analysis identifies genes specific to breast cancer hormone receptor status and risk of recurrence. Cancer Research. 71 (19), 6195-6207 (2011).
- Dedeurwaerder, S., et al. A comprehensive overview of Infinium HumanMethylation450 data processing. Briefings in Bioinformatics. 15 (6), 929-941 (2014).
- Hulbert, A., et al. Early detection of lung cancer using DNA promoter hypermethylation in plasma and sputum. Clinical Cancer Research: An Official Journal of the American Association for Cancer Research. 23 (8), 1998-2005 (2017).
- Shimada, H., Fukagawa, T., Haga, Y., Oba, K. Does remnant gastric cancer really differ from primary gastric cancer? A systematic review of the literature by the Task Force of Japanese Gastric Cancer Association. Gastric Cancer: Official Journal of the International Gastric Cancer Association and the Japanese Gastric Cancer Association. 19 (2), 339-349 (2016).
- Eijsink, J. J., et al. Detection of cervical neoplasia by DNA methylation analysis in cervico-vaginal lavages, a feasibility study. Gynecologic Oncology. 120 (2), 280-283 (2011).
- Sugimoto, K., et al. DNA methylation genome-wide analysis in remnant and primary gastric cancers. Gastric Cancer: Official Journal of the International Gastric Cancer Association and the Japanese Gastric Cancer Association. 22 (6), 1109-1120 (2019).
- Wang, Z., Wu, X., Wang, Y. A framework for analyzing DNA methylation data from Illumina Infinium HumanMethylation450 BeadChip. BMC Bioinformatics. 19, 115 (2018).
- Teh, A. L., et al. Comparison of methyl-capture sequencing vs. Infinium 450K methylation array for methylome analysis in clinical samples. Epigenetics. 11 (1), 36-48 (2016).
- Zhou, W., Laird, P. W., Shen, H. Comprehensive characterization, annotation and innovative use of Infinium DNA methylation BeadChip probes. Nucleic Acids Research. 45 (4), 22 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved