Method Article
Геном-Широкий анализ метилирования ДНК при раке желудочно-кишечного тракта
В этой статье
Резюме
В этом случае мы описываем процедуру геномного анализа метилирования ДНК при раке желудочно-кишечного тракта. Процедура имеет отношение к исследованиям, которые исследуют связь между метилированием генов и факторов, способствующих канцерогенеза при раке желудочно-кишечного тракта.
Аннотация
Метилирование ДНК является важным эпигенетическим изменением, которое является биологически значимым и частым направлением исследований рака. Метилирование ДНК в ширину генома является полезной мерой для обеспечения точного анализа состояния метилирования злокачественных новообразований желудочно-кишечного тракта (ГИ). Учитывая многочисленные потенциальные трансляционные использования анализа метилирования ДНК, практикующие клиницисты и другие новые для исследований метилирования ДНК должны быть в состоянии понять шаг за шагом, как эти генома всей анализы выполняются. Цель этого протокола заключается в предоставлении подробного описания того, как этот метод используется для идентификации биомаркеров при злокачественных новообразованиях GI. Важно отметить, что мы описываем три важных шага, которые необходимы для получения точных результатов в ходе анализа генома в ширину. Ясно и лаконично написано, эти три метода часто не хватает и не заметно для тех, кто новый эпигенетических исследований. Мы использовали 48 образцов злокачественных новообразований GI (рак желудка), чтобы подчеркнуть практически, как геном всей ДНК метилирования анализ может быть выполнен для злокачественных новообразований GI.
Введение
Эпигенетика относится к назойки изменения функции гена без изменения последовательностиДНК 1. Такие изменения могут быть связаны с метилированием ДНК, при котором метиловые группы на основе ДНК могут изменять экспрессию генов через изменения в упаковке хроматина. Развитие и прогрессирование рака может произойти, если этот эффект приводит к изменению экспрессии генов супрессораопухоли 2. Старение и хроническое воспаление являются как причинами рака, так и основными причинами изменений в метилированииДНК у человека 3,4,5. Следовательно, это позволяет использовать метилирование ДНК в качестве биомаркера в диагностике рака, а также в качестве мишени для лечения и профилактики. Для раннего выявления и прогноза рака, метилирование ДНК в настоящее время измеряется в опухоли, крови, мочи истула образцов 6, в то время как деметилирующие агенты в настоящее время используются для лечения лейкемии, такие как миелодиспластическийсиндром 7.
Геномный анализ метилирования ДНК с использованием массивной платформы для комплексной оценки метилирования ДНК в отдельном локусе CpG в геноме человека может быть использован для изучения состояния метилирования более чем 450 000 cpG сайтов вгеномной ДНК 8,9, что позволяет исследовать эпигенетику рака (см. таблицу материалов). Технологии секвенирования всего генома бисульфита (WGBS) изменили наши подходы в областиэпигенетики 10,11. Тем не менее, есть некоторые недостатки технологий с точки зрения существенной стоимости и времени обработки для эпигенетическогоанализа большого количества образцов 10,11. Таким образом, массивная платформа более осуществима для комплексной оценки метилирования ДНК в геноме человека. Доступность подходов для анализа метилирования в ширину генома улучшилась за последние несколько лет и позволяет нам расширить наши знания о том, как метилирование ДНК способствует развитию рака ипрогрессированию 12. Недавние успехи в микроаррей-платформы подходы дают нам обоснование для генома всей метилирования анализа для выявления новых эпигенетической подписи в ракежелудочно-кишечного тракта 13. Цель этого протокола состоит в том, чтобы предоставить подробное описание того, как этот метод используется для идентификации биомаркеров в злокачественных новообразованиях GI.
протокол
Все процедуры, которым следуют, соответствуют этическим стандартам комитета по этике исследований в области человека. Исследование было одобрено Институциональным советом по обзору в больнице Университета Дзюнтендо Сидзуока, и письменное информированное согласие было отменено из-за ретроспективного дизайна.
1. Мыть горки
- Подготовьте 10 мкм необлитаных секций, закрепленных на формалине (FFPE).
- Поместите слайды в стеклянный держатель слайда: используйте около 3-5 крупнейших поперечных слайдов, если ткань минимальна, и больше слайдов не требуется.
- Заполните держатель слайда ксиленом и убедитесь, что все ткани на слайде погружены в воду. Разрешить сидеть в течение 15 минут.
- Через 15 минут вылейте ксилен со слайдами, удерживаемыми наконечником пипетки, чтобы горки не выпадают.
- Налейте больше ксилена до того же уровня, что и раньше. Разрешить сидеть еще 15 минут.
- Через 15 минут вылейте ксилен снова.
- Заполните держатель слайда 100% этанолом (EtOH) и убедитесь, что все ткани на слайде полностью погружены. Разрешить сидеть в течение 3 минут.
- Слейте EtOH, тщательно держа горки. Пополнить до того же уровня с более EtOH. Разрешить сидеть в течение 2 минут.
- Слейте EtOH снова и удалить слайд. Аккуратно поместите их лицом вверх на чистое бумажное полотенце, чтобы высохнуть. Разрешить сидеть в течение 10 минут.
2. Очистка слайдов
- Подготовка пищеварения / лиза буфера с 650 мл дитилпирокарбоната (DEPC)-обработанной водой, 100 мл этилендиаминтетраацетической кислоты (ЭДТА), 50 мл соляного хлорида Триса (Tris-HCL) 2 Мн 8,8 и 200 мл 10% додекилового сульфата натрия (SDS) (см. таблицу материалов).
- Заполните однополетную полипропилевую трубку 1,5 мл буфером пищеварения/лиза (см. таблицу материалов).
- Положите чистый наконечник пипетки в буфер.
- Определить раковые ткани области опухоли наиболее подходящих для макродиссеции в соответствии с соответствующими H и E окрашенных разделе.
ПРИМЕЧАНИЕ: Область опухоли для макродиссекции должна быть определена предпочтительно двумя квалифицированными патологоанатомами. - Macrodissect ткани рака на основе отмеченных Н И E окрашенных разделе.
- Возьмите чистое лезвие бритвы и аккуратно соскребать раковую ткань со слайда, стараясь держать ее в одном куске, чтобы было легче работать.
- Возьмите наконечник пипетки из одной из одноразовых полипропилевых трубок, содержащих буфер, и используйте его для передачи утилизированной ткани в буферный флакон (см. таблицу материалов).
ПРИМЕЧАНИЕ: Влажный кончик должен привлечь ткани, что делает передачу легче.
- Повторите шаг 2.5 с остальными слайдами.
- После того, как все ткани находится в одноискуемой полипропилетовой трубке, используйте наконечник, чтобы убедиться, что ткань полностью погружена и не прилипла к стене трубки.
- Добавьте 20 йл субтилисина связанных с серином protease на флакон и осторожно Флик, чтобы смешать (см. Таблицу материалов).
- Поместите флакон в тепловой блок 55 градусов по Цельсию, по крайней мере 4 ч или на ночь. Убедитесь в том, чтобы слегка вихрь после 2 ч.
3. Бисульфитовое лечение
- В качестве образца используйте 45 МКЛ раствора переваренной ткани.
- Выполните обработку бисульфита с использованием реагентов в комплекте преобразования бисульфита в соответствии с инструкциями производителя (см. таблицу материалов).
- Добавьте 5 МКЛ буфера разбавления в образец ДНК и инкубировать при 37 градусов по Цельсию в течение 15 минут (см. таблицу материалов).
- В то время как образцы инкубируются, подготовь реагент преобразования бисульфита, добавив 750 л dH2O и 210 МКЛ буфера разбавления в одну трубку реагента преобразования КТ (см. Таблицу материалов). Смешайте трубки вихрем в течение 10 минут.
- После инкубации за 15 минут добавьте 100 МКЛ подготовленного реагента преобразования КТ в каждый образец и перемешайте с помощью инверсии.
- Инкубировать образцы в темноте при 50 градусов по Цельсию в течение 12 до 16 ч.
- После инкубации удалите образцы и поместите на лед на 10 минут.
- Добавьте 400 МКЛ связывающего буфера и смешайте каждый образец, протыкаясь вверх и вниз (см. таблицу материалов). Загрузите каждый образец в колонку спина и поместите столбец в трубку для сбора 2 мл (см. Таблицу материалов).
- Центрифуга каждого образца на полной скорости (10 000 х г)в течение 1 мин и отбросить протекаемый поток.
- Добавьте 200 йл буфера стирки к каждому столбецу и вращайтесь на полной скорости в течение 1 мин, отбрасывая протекающий поток (см. таблицу материалов).
- Добавьте к каждому столбец 200 л буфера десульфонации и позвольте столбецу стоять при комнатной температуре в течение 15 минут (см. таблицу материалов). После инкубации вращайте столбцы на полной скорости в течение 1 минуты и отбрасывайте протекание.
- Добавьте 200 мкл буфера стирки к каждой колонке и вращайтесь на полной скорости в течение 1 мин (см. таблицу материалов).
- Повторите этот шаг еще раз.
- Добавьте 46 йл dH2O к каждой колонке и поместите каждую колонку в новую стерильную однопропиленую трубку 1,5 мл мл (см. таблицу материалов). Спин каждой трубки в течение 2 минут, чтобы elute ДНК.
- Удалите каждый столбец спина из одной полипропиленовой трубки и отбросьте (см. таблицу материалов). ДНК готова к анализу.
4. Платформа Array для оценки метилирования ДНК в локусе CpG в геноме человека
- Оцените качество ДНК (проверка качества: КК) с помощью анализа FFPE КК на инструменте усиления и обнаружения ПЦР в режиме реального времени, с последующим анализом данных, выполненным в соответствии с инструкциями производителей (см. таблицу материалов).
ПРИМЕЧАНИЕ: Образцы с ∆Cq значения меньше, чем рекомендуемые 5.0 дополнительно обрабатываются14. - Проанализируйте образцы с помощью массивной платформы для комплексной оценки метилирования ДНК для оценки состояния метилирования участков cpG в геноме (см. таблицу материалов). Информационный бюллетень продукта, лист данных и файлы продуктов для платформы массивадоступны 15.
ПРИМЕЧАНИЕ: Анализ проводится в соответствии с инструкциями производителя для материала FFPE14. - Рассчитайте метилирование с высоким и низким локусом в инструменте анализа данных для платформы массива в качестве β-значения, с диапазоном от 0 до 1, соответственно (см. таблицу материалов). Используйте коммерчески доступное программное обеспечение (см. таблицуматериалов) для β стоимости.
- Импортные данные, генерируемые на платформе массива для комплексной оценки платформы метилирования ДНК в среду программного обеспечения R (R v.2.15.1) и обработать их с помощью инструмента для анализа метилированиямассивов 16,17.
ПРИМЕЧАНИЕ: На тепловой карты, которая генерируется с помощью инструмента анализа данных, столбцы заказываются неконтролируемой кластеризацией, в то время как порядок строк основан на уменьшающихся значениях статистики t для дифференциального метилирования сверху вниз. - Разделите тепловую карту на группы высокого и низкого метилирования, используя первый юбилиатор в неконтролируемом кластеризации.
ПРИМЕЧАНИЕ: Для проверки с количественным метилирования конкретных ПЦР (qMSP), выбрать гены на основе больших β-значение по отношению к CpG островов в регионе промоутер, и из-за их пригодности для грунтовка и зонд дизайн для qMSP.
5. Количественный метилирование-специфический ПЦР (qMSP)
- Используйте бисульфит-модифицированную ДНК из шага 3.3 в качестве шаблона для ПЦР на основе флуоресценции в режиме реального времени для оценки метилирования региона промоутера в каждом генном анализе.
- Выполните qMSP с помощью 96-ну ПЦР в режиме реального времени (см. таблицу материалов).
- Проверьте состояние промоутера метилирования гена-мишени на бисульфит-модифицированной ДНК с использованием 200 нм вперед грунтовки, 200 нм обратной грунтовки, и 80 нМ зондов. Подготовка мастер смесь с 16,6 мМ (NH4)2SO4, 67 мМ Трис рН 8,8, 10 мМм β-меркаптоэтанол, 10 нм флуоресцентаин, 0,166 мМ каждого деоксинуклеотида трифосфата и 0,04 U/l полимеразы ДНК (см. таблицу материалов). Окончательный объем реакции в каждой хорошо для всех анализов составляет 25 мкл.
- Выполните езду на велосипеде по qMSP следующим образом: 95 градусов по Цельсию в течение 5 минут, а затем 55 циклов по 95 градусов по Цельсию в течение 15 с, 60 градусов по Цельсию в течение 1 мин, и 72 КК в течение 1 мин.
ПРИМЕЧАНИЕ: Целевой ген должен быть выбран на основе критериев наличия больших бета-значений, связанных с островами CpG в регионе промоутеров, и подходит для грунтовка и зонда дизайн для qMSP.
- Использование генома человека, обработанного cpG methylase (M.SssI) в качестве положительного контроля метилирования (см. таблицу материалов).
ПРИМЕЧАНИЕ: Окончательная количественная оценка метилирования определяется как относительное значение метилирования (RMV) и рассчитывается как 2- кт для каждой репликации обнаружения метилирования по сравнению со средним Ct для β-Actin (ACTB)18. Последовательности праймера и зонда показаны в таблице 1. Ct 100 используется для незамеченных репликаций, что дает значение 2 -КТ близко к нулю. Используется следующая формула: средний2 - кт (RMV) - (2 -Я Ct_replicate_1 - 2- Я Ct_replicate_2 - 2- Я Ct_replicate_3)/318.
Результаты
Характеристики 48 пациентов с раком желудка в учебной когортетаковы (таблица 2): среднийвозраст пациентов составил 74 года (52-89 лет), а в когорту входили 38 мужчин (79,2%) и 10 женщин (20,8%). Было 35 пациентов (72,9%) с первичным раком желудка и у 13 пациентов (27,1%) с остатками рака желудка (первичный рак желудка: первое появление неметатической злокачественности в желудке; остаточный рак желудка: рак в остатке желудка, который развился более чем через 5 лет после дистальной гастрэктомии, независимо от причины первоначальной ресекции19). Было 23 пациента (47,9%) с метастазами лимфатических узлов и 25 пациентами (52.1%) Без. Во-первых, все 48 образцов (учебная когорта) были загружены для идентификации выбросов(рисунок 1A). Два образца дали пики, которые были больше, чем два стандартных отклонения смещены от других, и эти образцы были удалены(рисунок 1B). Таким образом, 46 образцов были сгруппированы с помощью гиперметилирования ДНК-промоутера. В результате тепловая карта была разделена на две группы на основе высокого и низкого метилирования(рисунок 2). Эта тепловая карта позволяет визуализировать 50 лучших зондов в пределах 1500 б.п. от транскрипционные стартовые площадки (TSS) в дифференциальном анализе метилирования. Высокие и низкие метилирования группы отличаются клиникопатологических факторов, связанных с агрессивным злокачественным фенотипом. То есть, тип рака (первичный рак желудка: PGC) (р 0,01, коэффициент коэффициентов 9,09 (1,67-50,00)) и наличие метастазов лимфатических узлов (положительный) (р 0,03, коэффициент коэффициента 6,82 (1,16-40,08)) стали значительными независимымипрогностическими факторами, когдаклиникопатологические факторы использовались в качестве ковариатов в многовариатном анализе (таблица 3). Наконец, мы определили ген EPB41L320,21 (праймер и зонд последовательности показано в таблице 1), чтобы быть тесно связаны с кодификацией подготовки когорты в высоких и низких метилирования групп в микроаррей анализа. С помощью qMSP были проверены результаты микроаррейного анализа EPB41L3 в когорте испытаний (126 образцов). Характеристики пациентов в когорте теста показаны в таблице 4. RMVs EPB41L3 в тканях PGC были значительно выше, чем у остатков рака желудка (RGC) в унивариатном анализе (р No 0,01) (Рисунок 3A). Аналогичным образом, RMVs в образцах с метастазами лимфатических узлов были значительно выше, чем те, без метастазов лимфатических узлов (р 0,03) (Рисунок 3B). Таким образом, анализ генома метилирования ДНК может помочь нам определить конкретные гены для характеристики определенного клинического статуса у пациентов с злокачественными новообразованиями GI.
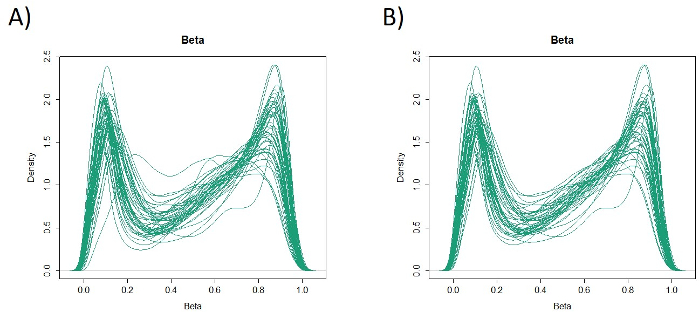
Рисунок 1: Бета-значения в 48 образцах (учебная когорта). Все 48 образцов (учебная когорта) были загружены и выбросы были изучены (A). Два образца имели пики, которые были выбросы, и они были удалены (B). Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
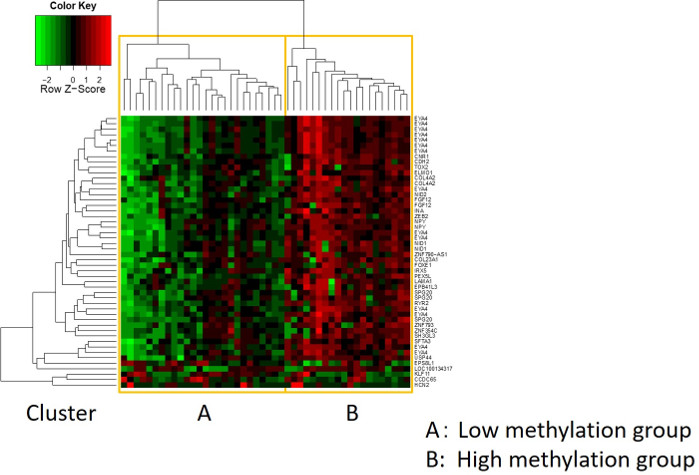
Рисунок 2: В результате тепловая карта. Остальные 46 образцов были сгруппированы с помощью гиперметилирования ДНК-промоутера. Тепловая карта была разделена на высокие и низкие метилирование групп. Эта тепловая карта позволяет визуализировать 50 лучших зондов в пределах 1500 б.п. от транскрипционные стартовые площадки (TSS) в дифференциальном анализе метилирования. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
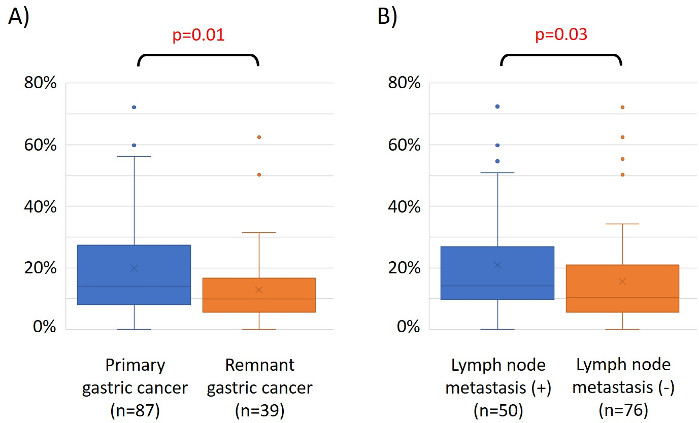
Рисунок 3: Относительные значения метилирования (RMVs) для EPB41L3 в первичном раке желудка (PGC) против остатков рака желудка (RGC), а также в случаях с и без метастазов лимфатических узлов. Результаты микроаррейного анализа EPB41L3 в испытательной когорте (126 образцов) были проверены с помощью qMSP. (A) В унивариатном анализе, RMVs EPB41L3 в тканях PGC были значительно выше, чем в RGC (р 0,01). (B) Аналогичным образом, RMVs в образцах с метастазами лимфатических узлов были значительно выше, чем у тех, у кого нет метастазов лимфатических узлов (р 0,03). Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
| Ген | Вперед 5' - 3' | Обратный 5' - 3' | Зонд |
| B-ACTIN | ТЕГ GGA GTA ТАТ AGG TTG GGG AAG TT | ААК АКА ЦАА ЦАА АКА АЦА ATT CAC | (56-FAM) TGT GGG GTG (ЗЕН) ГТГ ГТГ АТГ КЛЯП ГТТ ТЕГ |
| EPB41L3 | GGG ATA GTG GGG TTG ACG C | АТА ААА УВД CCG ACG AAC GA | ААА ТТК ГАА ААА CCG CGC GAC GCC GAA АКК А |
Таблица 1: Праймер и зонд последовательности.
| Клиникопатологические факторы | Переменные | |
| Возраст | 74 (52 - 89) * | |
| Гендерного | Мужской / женский | 38 (79.2%) / 10 (20.8%) |
| Тип | PGC / RGC | 35 (72.9%) / 13 (27.1%) |
| Метастазы лимфатических узлов | (+) / (-) | 23 (47.9%) / 25 (52.1%) |
| PGC: Первичный рак желудка, RGC: Остаток рака желудка | ||
| Медиана (минимум максимум) | ||
Таблица 2: Характеристики у 48 пациентов с раком желудка в учебной когорте.
| P-значение | Коэффициент коэффициентов | 95% Интервал доверия | |
| Тип (PGC) | 0.01 | 9.09 | 1.67 – 50.00 |
| Метастазы лимфатических узлов (я) | 0.03 | 6.82 | 1.16 – 40.08 |
| PGC: Первичный рак желудка | |||
Таблица 3: Прогностический фактор для группы высокого метилирования (кластер B).
| Клиникопатологические факторы | Переменные | |
| Возраст | 71 (33 - 86) * | |
| Гендерного | Мужской / женский | 96 (76.2%) / 30 (23.8%) |
| Тип | PGC / RGC | 87 (69.0%) / 39 (31.0%) |
| Метастазы лимфатических узлов | (+) / (-) | 50 (39.7%) / 76 (60.3%) |
| PGC: Первичный рак желудка, RGC: Остаток рака желудка | ||
| Медиана (минимум максимум) | ||
Таблица 4: Характеристики у 126 пациентов с раком желудка в когорте теста.
Обсуждение
Есть три важных шага в получении точных результатов анализа генома метилирования ДНК. Во-первых, макродиссеция области опухоли предпочтительно два квалифицированных патологоанатомов на основе репрезентативных H и E окрашенных разделов. Неточная макродиссеция может привести к загрязнению соседних нераковых тканей, что приводит к ненадежным результатам; таким образом, требуется тщательное макродиссемент. Во-вторых, оценка качества ДНК (проверка качества: КК). Образцы, которые не КК (∆Cq и 5.0) может дать низкое качество данных. Таким образом, образцы с ∆Cq йgt; 5.0 должны быть удалены и другие используются. Третьим шагом является расчет β-значения, которое определяется инструментом анализа данных для программного обеспечения платформы массива как метилированный сигнал / общий (метилированный и неметилированный) сигнал17. Значение β колеблется от 0-1 (или 0%-100%), что просто интерпретировать биологически17. Основная проблема с этим значением заключается в его плохих статистических свойствах, так как его высокая гетеросхестичность подразумевает, что дисперсия между образцами в крайностях метилирования диапазона (β No 0 или β No 1) сильноуменьшается 17. Кроме того, из-за низкого качества выборки, β-значения не могут показать воспроизводимыебифазные пики 22, и образцы без таких пиков должны быть исключены из дальнейшего изучения. Кроме того, целевой ген должен быть выбран на основе критериев наличия больших бета-значений, связанных с островами CpG в регионе промоутеров, и подходит для грунтовка и зонда дизайн для qMSP.
Оценка метилирования ДНК в локусе CpG в геноме человека проводится с использованием технологии на основе микроаррей с фиксированным количеством зондов для обследования конкретных геномных локусов. Это наиболее широко используемый метод в эпигеноме всей ассоциации исследований (EWAS) из-за его низкой стоимости, небольшое количество ДНК требуется, и заметно короче время обработки образца, что позволяет высокой пропускной способности обработки многих клиническихобразцов 23. Однако массивная платформа для комплексной оценки метилирования ДНК в отдельном локусе CpG ограничена количеством и специфичностью зондов для эпигенетически измененных локусов, что препятствует исследованию некоторых геномных регионов. WGBS, как правило, рассматривается как золотой стандартный метод из-за его более широкого спектрагеномного охвата 10,11. Тем не менее, этот метод имеет значительные затраты и относительно долгое время обработки для анализабольшого количества образцов 10,11. Таким образом, это не всегда осуществимо. Для сравнения, массивная платформа для комплексной оценки метилирования ДНК в отдельном локусе CpG в геноме человека разумна для использования с точки зрения стоимости и геномного охвата. В последнее время последние обновленные чипы бисера получили готовы использовать24. Эти анализы могут помочь нам проанализировать почти удвоенные измеренные участки CpG, которые могут достичь идеального исследования ассоциации генома (GWAS) для больших популяций образцов.
Таким образом, анализ генома метилирования ДНК с помощью массивной платформы для комплексной оценки метилирования ДНК в отдельном локусе CpG в геноме человека может предоставить важную информацию об эпигенетических биомаркеров при раке желудочно-кишечного тракта. По сравнению с WGBS, этот метод является экономически эффективным и сокращает время обработки образцов. Таким образом, этот метод для обнаружения метилирования ДНК в локусе CpG, вероятно, будет широко использоваться в эпигенетических исследованиях биомаркеров.
Раскрытие информации
Авторов нечего раскрывать.
Благодарности
Мы особенно признательны всем членам отделения хирургии, Сидни Киммел Всеобъемлющего онкологического центра в Университете Джонса Хопкинса школы медицины для полезных дискуссий и технической поддержки. Мы также благодарим Кристен Роджерс за щедрое техническое руководство по процедурам лечения бисульфита и qMSP.
Материалы
| Name | Company | Catalog Number | Comments |
| (NH4)2SO4 | Sigma-Aldrich | 14148 | Step 5.2. |
| 10% Sodium dodecyl sulfate (SDS) | Quality Biological | 351-032-721 EA | Step 2.1. |
| 100 % Ethanol | Sigma-Aldrich | 24194 | Step 1.7. |
| ABI StepOnePlus Real-Time PCR System | Applied BioSystems | 4376600 | Step 5.2. 96-well Real-Time PCR instrument |
| CT conversion reagent | Zymo Research | D5001-1 | Step 3.2.3. |
| Deoxynucleotide triphosphate (dNTP) | Invitrogen | 10297-018 | Step 5.2. |
| DEPC-treated water | Quality Biological | 351-068-131 | Step 2.1. |
| Ethylenediaminetetraacetic acid (EDTA) | Corning | 46-034-CL | Step 2.1. |
| EZ DNA Methylation Kit | Zymo Research | D5002 | Step 3.2. |
| Fluorescein | Bio-Rad | #1708780 | Step 5.2. |
| GenomeStudio | omicX | OMICS_00854 | Step 4.3. Data analysis tool for an array platform as a β-value, with a range from 0 to 1 |
| Human genomic DNA | New England Bio Labs | N4002S | Step 5.3. |
| Infinium HD FFPE QC Kit | Illumina | WG-321-1001 | Step 4.1. FFPE QC assay on a real-time PCR amplification |
| Infinium HumanMethylation450 assay | Illumina | WG-314 | Step 4.2. Array platform for complex evaluation of DNA methylation to assess the methylation status of >450,000 CpG sites in the genome |
| LightCycler 480 | Roche | 5015278001 | Step 4.1. |
| M-Binding Buffer | Zymo Research | D5002-3 | Step 3.2.6. |
| M-Desulphonation Buffer | Zymo Research | D5002-5 | Step 3.2.9. |
| M-Dilution Buffer | Zymo Research | D5002-2 | Step 3.2.1. |
| Minfi package | Bioconductor | N/A | Step 4.4. |
| M-Wash Buffer | Zymo Research | D5002-4 | Step 3.2.10. |
| Platinum Taq polymerase | ThermoFisher Scientific | 10966-034 | Step 5.2. |
| Proteinase K | New England Biolabs | P8107S | Step 2.8. |
| Single-use polypropylene (Eppendorf) tube | Eppendorf | 24533495 | Step 2.5.2. |
| Tris hydrochloride (Tris-HCL) 2 M pH 8.8 | Quality Biological | 351-092-101 | Step 2.1. |
| Xylene | Sigma-Aldrich | 214736 | Step 1.3. |
| Zymo Spin 1 Column | Zymo Research | C1003 | Step 3.2.6. |
| β-Mercaptoethanol | Sigma-Aldrich | M3148 | Step 5.2. |
Ссылки
- Qiu, J. Epigenetics: unfinished symphony. Nature. 441 (7090), 143-145 (2006).
- Okugawa, Y., Grady, W. M., Goel, A. Epigenetic alterations in colorectal cancer: emerging biomarkers. Gastroenterology. 149 (5), 1204-1225 (2015).
- Ahuja, N., Li, Q., Mohan, A. L., Baylin, S. B., Issa, J. P. Aging and DNA methylation in colorectal mucosa and cancer. Cancer Research. 58 (23), 5489-5494 (1998).
- Hsieh, C. J., et al. Hypermethylation of the p16INK4a promoter in colectomy specimens of patients with long-standing and extensive ulcerative colitis. Cancer Research. 58, 3942-3945 (1998).
- Ushijima, T., Okochi-Takada, E. Aberrant methylations in cancer cells: where do they come from. Cancer Science. 96 (4), 206-211 (2005).
- Coppedè, F. Epigenetic biomarkers of colorectal cancer: Focus on DNA methylation. Cancer Letters. 342 (2), 238-247 (2014).
- Vasilatou, D., Papageorgiou, S. G., Dimitriadis, G., Pappa, V. Epigenetic alterations and microRNAs: new players in the pathogenesis of myelodysplastic syndromes. Epigenetics. 8 (6), 561-570 (2013).
- Ma, X., Wang, Y. W., Zhang, M. Q., Gazdar, A. F. DNA methylation data analysis and its application to cancer research. Epigenomics. 5 (3), 301-316 (2013).
- Morris, T. J., Beck, S. Analysis pipelines and packages for Infinium HumanMethylation450 BeadChip (450k) data. Methods. 72, 3-8 (2015).
- Rauluseviciute, I., Drabløs, F., Rye, M. B. DNA methylation data by sequencing: experimental approaches and recommendations for tools and pipelines for data analysis. Clinical Epigenetics. 11 (1), 193 (2019).
- Cazaly, E. Making sense of the epigenome using data integration approaches. Frontiers in Pharmacology. 10, 126 (2019).
- Leal, A., Sidransky, D., Brait, M. Tissue and cell-free DNA-based epigenomic approaches for cancer detection. Clinical Chemistry. 66 (1), 105-116 (2020).
- Song, W., Ren, J., Wang, W. J., Wang, C. T., Fu, T. Genome-wide methylation and expression profiling identify a novel epigenetic signature in gastrointestinal pan-adenocarcinomas. Epigenomics. 12 (11), (2020).
- Wong, E. M., et al. Tools for translational epigenetic studies involving formalin-fixed paraffin-embedded human tissue: applying the Infinium HumanMethyation450 Beadchip assay to large population-based studies. BMC Research Notes. 8, 543 (2015).
- . Illumina Available from: https://jp.support.illumina.com/downloads/infinium_humanmethylation450_product_files.html (2020)
- Fackler, M. J., et al. Genome-wide methylation analysis identifies genes specific to breast cancer hormone receptor status and risk of recurrence. Cancer Research. 71 (19), 6195-6207 (2011).
- Dedeurwaerder, S., et al. A comprehensive overview of Infinium HumanMethylation450 data processing. Briefings in Bioinformatics. 15 (6), 929-941 (2014).
- Hulbert, A., et al. Early detection of lung cancer using DNA promoter hypermethylation in plasma and sputum. Clinical Cancer Research: An Official Journal of the American Association for Cancer Research. 23 (8), 1998-2005 (2017).
- Shimada, H., Fukagawa, T., Haga, Y., Oba, K. Does remnant gastric cancer really differ from primary gastric cancer? A systematic review of the literature by the Task Force of Japanese Gastric Cancer Association. Gastric Cancer: Official Journal of the International Gastric Cancer Association and the Japanese Gastric Cancer Association. 19 (2), 339-349 (2016).
- Eijsink, J. J., et al. Detection of cervical neoplasia by DNA methylation analysis in cervico-vaginal lavages, a feasibility study. Gynecologic Oncology. 120 (2), 280-283 (2011).
- Sugimoto, K., et al. DNA methylation genome-wide analysis in remnant and primary gastric cancers. Gastric Cancer: Official Journal of the International Gastric Cancer Association and the Japanese Gastric Cancer Association. 22 (6), 1109-1120 (2019).
- Wang, Z., Wu, X., Wang, Y. A framework for analyzing DNA methylation data from Illumina Infinium HumanMethylation450 BeadChip. BMC Bioinformatics. 19, 115 (2018).
- Teh, A. L., et al. Comparison of methyl-capture sequencing vs. Infinium 450K methylation array for methylome analysis in clinical samples. Epigenetics. 11 (1), 36-48 (2016).
- Zhou, W., Laird, P. W., Shen, H. Comprehensive characterization, annotation and innovative use of Infinium DNA methylation BeadChip probes. Nucleic Acids Research. 45 (4), 22 (2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены