Method Article
فحص حركية الميرنا في المكان والزمان في الإشريكية القولونية باستخدام الفلورس أحادي الجزيء بلونين في التهجين الموقعي
In This Article
Summary
يصف هذا البروتوكول تطبيق الفلورو جزيء واحد في التهجين الموقعي (smFISH) لقياس حركية المير النووي في الجسم الحي من التوليف والتحلل.
Abstract
الفلور أحادي الجزيء في التهجين الموضعي (smFISH) يسمح بحساب العدد المطلق من mRNAs في الخلايا الفردية. هنا، ونحن وصف تطبيق smFISH لقياس معدلات النسخ وتدهور مرنا في Escherichia القولونية. كما يعتمد smFISH على خلايا ثابتة ، نقوم بأداء smFISH في نقاط زمنية متعددة خلال تجربة الوقت ، أي عندما تمر الخلايا بتغييرات متزامنة عند تحريض أو قمع التعبير الجيني. في كل نقطة زمنية، يتم تمييز المناطق الفرعية من مرناً بشكل طيفي إلى الاستطالة النسخي التحقيقي وإنهاء سابق لأوانه. كما تسمح نتيجة هذا البروتوكول بتحليل التعريب داخل الخلايا للـ mRNAs والتغاير في أرقام نسخ مرنا بين الخلايا. باستخدام هذا البروتوكول العديد من العينات (~ 50) يمكن معالجتها في غضون 8 ساعة ، مثل مقدار الوقت اللازم لبعض العينات. نناقش كيفية تطبيق هذا البروتوكول لدراسة حركية النسخ والتحلل لمختلف mRNAs في الخلايا البكتيرية.
Introduction
تدفق المعلومات الوراثية من الحمض النووي إلى مرنا والبروتين هي واحدة من العمليات الخلوية الأساسية، التي تنظيم مهم للياقة الخلوية1. يتم تحديد عدد mRNAs في خلية بواسطة اثنين من العمليات الحيوية، النسخ، وتدهور مرنا. ومع ذلك، فإن كيفية تنظيم النسخ وتدهور مرنا في الزمان والمكان من خلية واحدة لا تزال غير مفهومة تماما، ويرجع ذلك إلى حد كبير إلى نقص الأساليب التجريبية لقياس حركيتها كميا في الجسم الحي.
يمكن لأساليب تستند إلى mRNAs الإجمالي المستخرجة من مجموعة من الخلايا ، مثل لطخة الشمالية ، RT - PCR ، تسلسل الحمض النووي الريبي ، وmicroarrays التعبير الجيني ، قياس الفرق النسبي في مستويات مرنا وقد استخدمت على نطاق واسع لتحليل معدل استطالة النسخ2،3،4،5 أو معدل تدهور الحمض النووي الريبي6،7. ومع ذلك ، فإنها لا توفر العدد المطلق من mRNAs في الخلية الواحدة ، وبالتالي ، فهي ليست مناسبة لالرقاب معدل بدء النسخ8. كذلك، لأن mRNAs يتم استخراجها من مجموعة من الخلايا، لا يمكن قياس التوزيع المكاني للmRNAs داخل خلية واحدة ومدى تغير أرقام نسخ مرنا بين الخلايا.
الجيل التالي من تسلسل الحمض النووي الريبي على الخلايا الفردية (scRNAseq) يمكن أن يحدد عدد mRNAs لكل خلية في مقياس الجينوم9. ومع ذلك، لا يزال من الصعب استخدام هذه التقنية لقياس حركية النسخ، وذلك بسبب التحديات مع إعداد العينة وارتفاع التكلفة. على وجه الخصوص، تطبيق scRNAseq على البكتيريا كان من الصعب من الناحية الفنية بسبب انخفاض وفرة مرنا10،11.
ويستند التألق أحادي الجزيء في التهجين الموقعي (smFISH) على تهجين المسابر ذات التسمية الفلورية وحيدة التي تقطعت بها السبل والتي تُكمل تسلسلاتها للمر ن ب. م. الهدف من الفائدة12،13. ويماثل مفهوم التهجين الخاص بالتسلسل المفهوم المستخدم في البقعة الشمالية أو RT-PCR، ولكن يتم التهجين في الموقع داخل خلايا ثابتة، للحفاظ على التعريب الأصلي للـ mRNAs. يتم تضخيم إشارة مرنا واحدة باستخدام العديد من المسابير، ~ 20 النيوكليوتيدات (nt) في الطول، والتهجين إلى أجزاء مختلفة من مرنا (الشكل 1A)13. في هذا النهج التحقيق "التبليط" ، وعدد من المسابير اللازمة للكشف عن مرنا واحدة يحدد حدا أقل على طول مرنا التي يمكن أن تكون مقايسة. بدلا من ذلك، قد يكون مرنا من الفائدة من تنصهر نسخ إلى مجموعة غير ترميز من تسلسل مشغل لاك جنبا إلى جنب، بحيث نسخ متعددة من مستوصف اللاكمو المسمى الفلورية التهجين إلى مرنا واحد(الشكل 1B)14.
وقد استخدمت smFISH لتحديد عدد mRNAs لكل خلية في حالة ثابتة (أي عندما يكون التوليف والاضمحلال في التوازن) وتحليل متوسط وتقلب mRNAs بين الخلايا البكتيرية15,16,17. في الآونة الأخيرة، وقد تم تطبيق smFISH لتحديد أرقام مرنا في حالة غير ثابتة، والحق بعد تحريض أو قمع التعبير الجيني في الإشريكية القولونية18،19،20. ثم استخدمت التغيرات الزمنية في أرقام نسخة مرنا المطلقة لحساب معدل بدء النسخ، واستطالته، وإنهائه، وكذلك معدل تدهور مرنا. لهذا التطبيق، يمكن أن تكون إجراءات SMFISH التقليدية مرهقة لأن هناك العديد من العينات، كل منها يمثل نقطة زمنية واحدة، التي تحتاج إلى الذهاب من خلال خطوات تبادل العازلة متعددة (أي، الطرد المركزي والغسيل). هنا، ونحن وصف بروتوكول smFISH، والتي يتم تبسيط خطوات معالجة العينة بشكل كبير من خلال وجود الخلايا الالتزام السطح من غطاء والسوائل عن طريق التعرق مع نظام الترشيح فراغ14،19. باستخدام التعبير عن lacZ في الإشريكية القولونية كمثال، يتم إثبات سير العمل الكامل(الشكل 2)،بما في ذلك تحليل الصور(الشكل 3)مما يؤدي إلى الحركية في الجسم الحي للنسخ (البدء، الاستطالة، وإنهاء) وتدهور الرنا، وتقلب من خلية إلى خلية في التعبير مرنا، وتعريب مرنا. نتوقع أن البروتوكول ينطبق على نطاق واسع على التحقيق في الحركية الجسم الحي وتوطين mRNAs الأخرى في أنواع البكتيريا المختلفة.
Protocol
1. إعداد مسابير سمك البحر
ملاحظة: لتسمية مسابير smFISH مع fluorophore واحد، اتبع بروتوكول قياسي لوضع العلامات الحمض النووي oligonucleotides على أساس الكيمياء استر NHS21.
- تصميم مسابير SMFISH. تقرر ما إذا كان لاستخدام "تبليط" تحقيقات أو "صفيف" تحقيقات(الشكل 1)للجين من الفائدة. راجع قسم المناقشة حول كيفية اتخاذ القرار.
- بالنسبة إلى "التبليط" تحقيقات (الشكل 1A) ، استخدم أداة مصمم التحقيق عبر الإنترنت (على سبيل المثال ، انظر جدول المواد).
- بالنسبة إلى "الصفيف" المسابر(الشكل 1B)، قم بإجراء بحث تسلسل BLAST للتأكد من أن تسلسل التحقيق ليس مكملاً لأي تسلسلات أخرى لمرنا.
- لدراسة النسخ الميرنا lacZ و التحلل الحركية، استخدام مجموعتين من 24 مسبارا، كل مجموعة تغطي الأولى والأخيرة 1 كيلوبايت المناطق من lacZ (3,072 bp)19.
ملاحظة: يشار إلى هذه المجموعات المجسّة فيما يلي باسم "مسبار الـ MRNA 5" و"مسبار مرنا 3" على التوالي. يتم سرد تسلسل هذه المسابر في جدول المواد.
- تسلسل التحقيق النظام كما oligonucleotides الحمض النووي مع رابط أميني C6 في نهاية 5 '. قم بذوبان المسابير الفردية في الماء إلى 1 mM.
- الجمع بين كميات من المجس من تحقيقات ل "5' مسبار مرنا" و "3' مرنا التحقيق" مجموعات. على سبيل المثال، بالنسبة لـ 5'mRNA مسبار تعيين لlacZ،والجمع بين 20 ميكرولتر من كل مسبار (مجموع 24 نوعا من تحقيقات في المجموعة).
- أداء الإيثانول هطولالأمطار 22 من المسابير مجتمعة لإزالة أي تلوثات من الأمينات الأولية والثانوية (مثل تريس، والجليسين، وأملاح الأمونيوم) التي يمكن أن تمنع تفاعل الاقتران. في النهاية، حل بيليه الحمض النووي في 100 ميكرولتر من الماء (تسفر عن ~ 4.5 mM من الحمض النووي في مجموعة مسبار).
ملاحظة: يوصى بهذه الخطوة حتى إذا خضعت المسابير لتنقية غيرlt قياسية من قبل الشركة المصنعة. وقد تعمل عملية تنقية معيارية قائمة على الفلتر بدلاً من هطول الإيثانول بالإضافة إلى ذلك. - اختيار اثنين من الفلوروبوريات متميزة الطيفي مع مويتس استر NHS أحادية الوظائف، بحيث يمكن تسمية 5 'و 3' مرنا 'مجموعات التحقيق بشكل تفاضلي. على سبيل المثال، إعداد Cy5 NHS استر ل 5 'مسابير مرنا و Cy3B NHS استر ل 3 'مسابير مرنا. حل كل نوع من الفلوروفور في DMSO اللامائية إلى النهائي 20 ملغ / مل (~ 25 مل).
- إعداد 0.1 م بيكربونات الصوديوم (درجة الH 8.5) الحق قبل كل رد فعل وضع العلامات. التعرض للهواء لفترة طويلة سوف يقلل من درجة اله PH ويقلل من كفاءة وضع العلامات.
- للتفاعل الاقتران، والجمع بين ما يلي: 15 ميكرولتر من مخزون Cy5 fluorophore (من الخطوة 1.5)، 4 ميكرولتر من 5' مرنا مجموعة مسبار (من الخطوة 1.4)، 75 ميكرولتر من بيكربونات الصوديوم (من الخطوة 1.6)، و 7 ميكرولتر من الماء. التفاف أنبوب مع رقائق الألومنيوم ويهز في درجة حرارة الغرفة لمدة 3-6 ساعة.
ملاحظة: لا يؤدي فترة حضانة أطول بالضرورة إلى زيادة كفاءة وضع العلامات. أيضا، يمكن أن يتم توسيع التفاعل صعودا أو إلى أسفل إذا تم الحفاظ على تركيزات المكونات. - كرر الخطوة المذكورة أعلاه لمجموعة مسبار مرنا 3 وفلوروفور المقابلة (أي Cy3B NHS-ester).
- أداء الإيثانول هطول الأمطار22 لإزالة جزيئات صبغة غير رد فعل. قم بحل بيليه في ~ 50 ميكرولتر من العازلة TE (10m تريس-HCl pH 8.0 مع 1mM EDTA).
- تقدير تركيزات الحمض النووي والفلوروفور باستخدام مطياف الأشعة فوق البنفسجية فيس.
- قياس امتصاص في 260 نانومتر و 559 نانومتر (Cy3B) أو 649 نانومتر (Cy5). إذا كانت العينة مركزة للغاية بحيث لا يمكن أن تعطي قياس دقيق، قم بتخفيف 1 ميكرولتر من العينة إلى 10 ميكرولتر.
- تحويل امتصاص إلى تركيز:
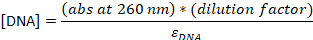
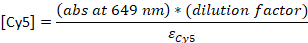
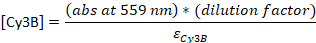
εالحمض النووي = 0.2 μM-1 (للحمض النووي واحد 20-nt تقطعت بهم السبل), εCy5 = 0.25 μM-1, ε وCy 3B = 0.13 μM-1
ملاحظة: [DNA] هو تركيز المجسات الكلية داخل الحل. تركيز المسابير الفردية حوالي 24x أقل. وسيستخدم تركيز المسبر الكلية "كـتركيزات مسبار" من هذه النقطة. إذا كانت النسبة بين [DNA] و [صبغ] هي 1، فقد يتم تخطي الخطوة التالية من HPLC23، ويجب تخفيف العينة إلى 4-5 μM النهائية في مخزن TE المؤقت.
- (مستحسن) تنقية مسابير المسمى من مسابير غير مسمى والأصباغ الحرة باستخدام HPLC.
ملاحظة: على الرغم من أن هذه الخطوة تنقية إضافية سيؤدي إلى فقدان العينة، فإنه مفيد للتطبيقات المتلقين للمعلومات. إزالة مسابير الحمض النووي غير الموصومة ستزيد من إشارة الفلوريس من أهداف مرنا وإزالة الأصباغ غير المنقحة سوف تقلل من الفلورنسي الخلفية.- إعداد HPLC مع عمود C18 التحليلية القياسية، 0.1 M ثلاثية الأبعاد خلات (TEAA) كما العازلة A، acetonitrile كما المخزن المؤقت باء.
- إضافة 1 م TEAA إلى العينة (من الخطوة 1.9) لجعل TEAA 0.1 م.
- تعيين برنامج التدرج على النحو التالي: 0-5 دقيقة مع 0٪ B، 5-35 دقيقة مع تدرج خطي 0-30٪ من B، 35-37 دقيقة مع تدرج خطي 30-100٪ من B، و 37-40 دقيقة مع 0٪ B. الحفاظ على معدل التدفق عند 0.1 مل / دقيقة وسجل الكروماتوجرامات في 260 و 649 نانومتر (للعينات Cy5 المسمى) أو في 260 و 559 نانومتر (للعينات Cy3B المسمى).
- جمع العينة مائل عندما يزيد امتصاص في كل من الحمض النووي وقنوات الفلوروفور.
- تركيز العينة مائل باستخدام مكثف فراغ وإعادة تعليق بيليه في 50-100 μL TE العازلة.
- تحقق من تركيز الحمض النووي والفلوروفور باستخدام مطياف الأشعة فوق البنفسجية -فيس (انظر الخطوة 1.10). تمييع، إذا لزم الأمر، لجعل التركيز النهائي حول 4-5 μM. تخزين المسابير في -20 درجة مئوية
2- إعداد الحلول
- إعداد كمية كبيرة من المياه المعالجة DEPC والمخازن(الجدول 1). هذه الحلول يمكن أن تستمر أكثر من عام في درجة حرارة الغرفة.
- إعداد 4x حل التثبيت وغسيل الحل (الجدول 1).
- إعداد حل ما قبل التهجين و حل التهجين التحقيق (الجدول 1). إعداد حل التهجين التحقيق أثناء الحضانة في الخطوة 5.1 أو الخطوة 6.1 ومن ثم الاحتفاظ الحل في 37 درجة مئوية كونترتوب شاكر لمدة 20-40 دقيقة مع غطاء لتقليل التعرض للضوء.
ملاحظة: تم تحسين تركيزات الفورماميد، SSC، والمسبار لمجموعات مسبار lacZ لتقليل الفلورسيه الخلفية مع تعظيم الإشارة الحقيقية. راجع قسم المناقشة للحصول على تفاصيل حول كيفية تعديل هذه التركيزات للتطبيقات المختلفة.
3. إعداد أغطية والشرائح الزجاجية
- أغطية نظيفة والشرائح الزجاجية.
- ضع غطاءً فرديًا وشرائح في جرة كوبلين باستخدام ملقط. تأكد من أن الشرائح والزلاقات مفصولة ولا تلمس بعضها البعض.
- ملء جرة مع الإيثانول 100٪ وإغلاق الغطاء. وضع الجرة في الماء حمام منظف بالموجات فوق الصوتية و sonicate لمدة 15-20 دقيقة.
ملاحظة: بالنسبة لـ sonicator حمام الماء، فمن المستحسن لإيقاف وظيفة سخان. - صب الايثانول وغسل مع الماء فائقة الخطورة 3-4x. استخدام المياه المتدفقة مباشرة من آلة تنقية المياه.
- صب الماء من جرة وملء مع 70٪ الإيثانول. إغلاق غطاء وأداء سونيكيشن لمدة 15-20 دقيقة وغسل بالماء فائقة السخاء.
- ملء جرة مع الماء فائقة الpure و sonicate لمدة 15-20 دقيقة.
ملاحظة: يمكن الاحتفاظ بالأغطية والشرائح الزجاجية بين عشية وضحاها في جرة كوبلين المليئة بالماء فائق الخطورة. - اتخاذ شريحة أو غطاء من جرة كوبلين باستخدام ملقط نظيفة وضربة الجافة باستخدام الغازN 2. كرر هذا من أجل الشرائح وأغطية الأغطية المتبقية.
- ضع الشرائح المجففة في مربع تخزين نظيف حتى يتم استخدامه في الخطوة 7.5. ضع الأغطية المجففة في صندوق تلميح ماصة فارغ 1000-μL ، والذي سيكون بمثابة "غرفة" في الإجراء المتبقي.
- باستخدام علامة مبيدة للماء، رسم الدوائر على الأغطية بعد ثقوب دائرية في صندوق تلميح ماصة. هذه الدوائر (~ 0.5 سم في القطر) ستكون بمثابة "آبار". انتظر على الأقل 5-10 دقيقة لعلامة أن تكون المجففة تماما.
ملاحظة: دوماً الاحتفاظ غطاء صندوق تلميح مغلق. - تطبيق قطرة 20-μL من 0.1٪ بولي-L-ليسين إلى كل بئر. احتضان لمدة 10-50 دقيقة في درجة حرارة الغرفة.
ملاحظة: ضبط هذا الحجم وفقا لحجم البئر. تأكد من أن الحل يغطي تماما منطقة البئر. لحضانة أطول، يجب الحرص على تجنب التبخر. - بعد حضانة التعرق بولي-L-ليسين دون لمس السطح لأن هذا سوف كشط بولي-ل-ليسين قبالة. ثم تطبيق قطرة (~ 20 ميكرولتر) من المياه DEPC إلى بولي L-ليسين الآبار المعالجة. أغلق غطاء "الغرفة" لمنع التبخر حتى الخطوة 5.1.
4. الوقت بالطبع التجربة و تثبيت العينة
- تنمو الخلايا القولونية في ~ 20 مل ثقافة السائل في قارورة 250 مل. الحفاظ على قارورة في حمام الماء شاكر (30 درجة مئوية) والاستمرار في الاهتزاز. وقف شاكر فقط عند أخذ العينات.
ملاحظة: يتم الحصول على النتائج المقدمة في هذه الورقة من خلايا MG1655 التي تزرع في المتوسط M9 الحد الأدنى تكمل مع 0.2٪ الجلسرين، 0.1٪ الأحماض casamino، و 1 ملغم / لتر الثيامين إلى مرحلة النمو الأسي (OD600~ 0.2). - أضف 250 ميكرولتر من محلول التثبيت 4x في أنبوب فارغ 1.5 مل. كرر وإعداد أنابيب متعددة، بقدر ما نقاط الوقت التي يتعين اتخاذها. تسمية الأنابيب مع أرقام نقطة الوقت والاحتفاظ بها في درجة حرارة الغرفة.
- خذ 750 ميكرولتر من ثقافة الخلايا (OD600~ 0.2) قبل بدء تجربة الوقت بالطبع. إضافة الثقافة إلى أنبوب وضع علامة "الوقت صفر" (من الخطوة 4.2). عكس أنبوب بلطف لخلط الخلايا مع حل التثبيت.
ملاحظة: لا الماصات صعودا وهبوطا لخلط، دوامة، أو "تكون الخام" على الخلايا. تمثل هذه العينة حالة مكبوتة وسوف تستخدم كتحكم لحساب كثافة الفلورنس لـ مرنا واحدة (انظر الخطوة 9.4). - إضافة 0.02-1 mM من isopropyl β-D-1-thiogalactopyranoside (IPTG) إلى الثقافة السائلة للحث على التعبير lacZ. بدء مؤقت عند هذه النقطة (ر = 0 دقيقة) وعينة في فترة زمنية معينة (على سبيل المثال، كل 1 دقيقة) من ذلك الحين فصاعدا. لأخذ العينات، كرر الخطوة 4.3.
- أضف 5 mM orthonitropheynl-β-D-fucopyranoside (ONPF) أو 500 mM جلوكوز24 في وقت معين خلال تجربة الدورة الزمنية (على سبيل المثال في t = 1.5 دقيقة) لقمع التعبير lacZ. بعد إعادة القمع، استمر في أخذ عينات من الثقافات (الخطوة 4.3) لتتبع تدهور الرنا.
ملاحظة: يمكن أيضا أن يتم القمع مع ~ 400 ميكروغرام / مل rifampicin، وهو المانع بدء النسخ25. - للتهيّم، حضن الأنابيب التي تحتوي على خلايا عينة في درجة حرارة الغرفة لمدة 15 دقيقة، تليها حضانة في الثلج لمدة 30 دقيقة.
- لإزالة المثبتات، الطرد المركزي الأنابيب في 4500 س ز لمدة 4 دقائق في درجة حرارة الغرفة. إزالة الماشحة مع ماصة.
ملاحظة: تأكد من التخلص من الفورمالديهايد في حاوية نفايات منفصلة بعد بروتوكول الأمان. - إضافة 1 مل DEPC-PBS وإعادة تعليق الخلايا. كرر الطرد المركزي وإعادة التعليق 2X مرات أكثر.
ملاحظة: الخلايا الثابتة هشة وتحتاج إلى علاج لطيف. بعناية إعادة تعليق بيليه وتجنب فقاعات. - بعد الخطوة غسل النهائي، وإعادة تعليق الخلايا في ~ 30 ميكرولتر DEPC-PBS.
5. Permeabilization من أغشية الخلايا
- تطبيق كل عينة نقطة زمنية على آبار مختلفة على غطاء (~ 30 ميكرولتر في البئر). انتظر 10-30 دقيقة في درجة حرارة الغرفة لخلايا التمسك على السطح. تجنب دمج قطرات السائل بين الآبار.
- لشطف قبالة الخلايا غير المنضمة، التعرق السائل وتطبيق ~ 20 ميكرولتر برنامج تلفزيوني DEPC إلى كل بئر. التعرق دي بي سي برنامج تلفزيوني في غضون بضع دقائق.
- Permeabilize أغشية الخلايا عن طريق تطبيق 15 ميكرولتر من 70٪ الإيثانول إلى كل بئر لمدة 4 دقائق. استلهم الإيثانول بعد 4 دقائق، وتأكد من أن الآبار جافة تماما.
ملاحظة: من المهم الحد من العلاج بالإيثانول لمدة 4 دقائق. العلاج الأطول سوف يؤدي إلى الإفراط في permeabilization. - تطبيق 30 ميكرولتر من محلول الغسيل على كل بئر.
6. التهجين التحقيق
- استلهم محلول الغسيل من كل بئر. تطبيق 30 ميكرولتر من حل ما قبل التهجين على كل بئر. احتضان الغرفة في فرن 37 درجة مئوية لمدة 30 دقيقة.
ملاحظة: إضافة ~ 50 مل من الماء إلى الجزء السفلي من الغرفة لتوفير الرطوبة. - التعرق حل ما قبل التهجين من كل بئر. تطبيق ~ 30 μL من حل التهجين التحقيق لكل بئر. غطي الغرفة بورق الألمنيوم واحتضانها في فرن 37 درجة مئوية لمدة 2 ساعة.
ملاحظة: تأكد من أن حل التهجين المسباري موجود في 37 درجة مئوية من كونترتوب شاكر قبل هذه الخطوة. تجنب دمج السوائل بين الآبار. تطبيق حجم أصغر من الحل على كل بئر، إذا لزم الأمر.
7. بعد التهجين غسل وإعداد للتصوير
- باستخدام ماصة متعددة القنوات، وتطبيق ~ 30 ميكرولتر من محلول الغسيل على كل بئر في كل مرة. التعرق وتكرار 3-5x مرات الغسيل. احتضان الغرفة في الفرن 37 درجة مئوية لمدة 15-30 دقيقة.
- كرر الخطوة 7.1 مرتين أكثر من ذلك.
- غسل كل جيدا مع DEPC- برنامج تلفزيوني 5x مرات. اتبع الطريقة المستخدمة في الخطوة 7.1 ولكن تخطي عملية الحضانة.
- استلهم السائل من غطاء الأغطية. تطبيق 4 ميكرولتر من DEPC-PBS إلى كل بئر.
- باستخدام ملقط، ورفع والوجه غطاء، ووضع بلطف على شريحة زجاجية (من الخطوة 3.2). تجنب الفقاعات.
- ختم حواف الغطاء مع صمغ الأسنان سيليكون.
- انتظر حتى يتم ترسيخ اللثة. يمكن للمرء أن يتوقف هنا ويخزن الشريحة بين عشية وضحاها عند 4 درجة مئوية.
ملاحظة: تشير بروتوكولات سمّية أخرى إلى إضافة الكواشف الكسح الأكسجين (على سبيل المثال، الجلوكوز أوكسيداز / كاتالاز) أو استخدام التجارية المضادة للتلاشي تصاعد المتوسطة14،26 لزيادة قابلية التصوير من الفلوروفورس.
8. التصوير
- للعثور على منطقة ذات أهمية، استخدم النمط المباشر لتصوير التباين في المرحلة. تغيير مجال الرؤية داخل بئر عن طريق المناورة في مرحلة عصا التحكم. اختر منطقة حيث كثافة الخلية هو الأمثل (أي، هناك العديد من الخلايا التي يتم فصل معظمها). ضبط z-focus بحيث تكون صور الخلايا على النقيض من المرحلة في التركيز.
- أخذ لقطات في ترتيب Cy5 (التعرض 4 ق) ، Cy3 (2 ق التعرض) ، وتباين المرحلة (0.2 ق التعرض).
ملاحظة: يتم تصوير جزيئات صبغ Cy3B في قناة Cy3، ويشار إلى الصور على أنها صور Cy3. - كرر الخطوات 8.1-8.2 للحصول على صور من ~ 10 مناطق مختلفة داخل بئر.
- نقل الهدف إلى بئر آخر وكرر الخطوات 8.1-8.3.
- تصدير الصور كملفات TIFF.
- (اختياري) صورة متعددة الألوان الخرز adsorbed على سطح الغطاء في القنوات Cy5 و Cy3 لتحديد التحول المكاني بين القنوات Cy5 و Cy3 لأغراض تسجيل الصور.
- تطبيق ~ 10 ميكرولتر من الخرز الفلورسنت متعددة الألوان (0.2 μm القطر) على سطح غطاء نظيف وانتظر لمدة 10-30 دقيقة. بعد الغسيل مع ~ 50 ميكرولتر من برنامج تلفزيوني، وتطبيق ~ 5 ميكرولتر من برنامج تلفزيوني وساندويتش الأغطية مع شريحة زجاجية. ختم وجبل على المجهر.
- حبات الصورة في كل من قنوات Cy5 و Cy3.
9. تحليل الصور
ملاحظة: يتوفر رمز Matlab المستخدم في هذه الخطوة في موقع GitHub التالي: https://github.com/sjkimlab/Code_Publication/tree/master/JoVE_2020. يحتوي مجلد GitHub على كل ما يلزم لتحليل الصور، بما في ذلك قيم المعلمات لتجزئة الخلايا وتحديد البقعة. يتم شرح الإجراء في هذه الخطوة في النص الرئيسي، ويسمى "FISHworkflow.m".
- افتح أداة تجزئة الخلايا، مثل microbeTracker27 أو روفتي28، وحمّل صور التباين في المرحلة. اختر "إطارات مستقلة" واضغط على زر يسمى "جميع الإطارات" لبدء عملية التجزئة ، والتي يتم من خلالها تحديد الخلايا وحساب معالمها (الشكل 3B،C).
ملاحظة: تتوفر بروتوكولات مفصلة لاستخدام حزم البرامج هذه عبر الإنترنت (على سبيل المثال، oufti.org). - تحميل الصور Cy5 fluorescence في وظيفة spotFinder من microbeTracker أو Oufti، واضغط على "تشغيل" زر لبدء تحديد بقعة وتكديس استنادا إلى 2D غاوسي تركيب(الشكل 3B،C). كرر هذه الخطوة لصور Cy3 الفلورانس لتحليل البقع في قناة Cy3. هذه الخطوة تنتج قائمة من البقع في كل خلية، بما في ذلك كثافتها والإحداثيات.
- (اختياري) تصفية البقع الخافتة (ايجابيات كاذبة) باستخدام عتبة، كما هو موضح في ملف FISHworkflow.m.
ملاحظة: فحص البقع الفلورية في السيطرة السلبية (على سبيل المثال MG1655Δ lacZ)وتحديد عتبة لتصفية الإيجابيات الكاذبة. - للحصول على كثافة بقعة مرنا واحدة، استخدم قائمة من شدة البقعة التي تم قياسها في الوقت صفر (قبل إضافة IPTG)، وتناسب توزيع كثافة البقعة مع نموذج خليط غاوسي مع اثنين من مكونات الخليط. اتخاذ موقف الذروة من السكان غاوسي الأول (الخط الأسود في الشكل 3D، E) كما كثافة بقعة من مرنا واحدة. قم بإجراء هذا للبقع Cy5 وبقع Cy3 بشكل منفصل للحصول على شدة البقعة لـ 5 و 3 'لاك زد مرنا.
ملاحظة: كرر هذا في كل تجربة وقت بالطبع لأن شدة بقعة مرنا واحدة يمكن أن تختلف قليلا في التجارب المختلفة. - قم بتقسيم شدة الفلور في بقعة مع شدة مرنا واحدة (من الخطوة 9.4) للحصول على عدد mRNAs داخل بقعة. سوم كثافة بقعة تطبيع داخل خلية لحساب العدد الإجمالي للميرنا في خلية(الشكل 3F). قم بإجراء هذه الحسابات لـ 5 'و 3' مرنا بشكل منفصل.
- حساب ومؤامرة أرقام الرنا المتوسط لكل خلية في كل نقطة زمنية (على سبيل المثال، الشكل 4B)، وتحليل في الحركية في الجسم الحي من النسخ وتدهور مرنا من التغير الزمني في مستويات الحمض النووي الريبي المتوسط(الشكل 4B).
- للحصول على معدل استطالة النسخ، قم بأداء أقل المربعات المناسبة لخط إلى الارتفاع الأولي في إشارات مرنا 5 و3 'و تحديد اعتراض المستويات القاعدية (الشكل 4B). ويشير الفرق بين عمليات الاعتراض هذه إلى متوسط الوقت الذي يمكن أن يُقلّل به RNAPs من منطقة المسبار 5 إلى منطقة المسبار 3. تقسيم المسافة بين مجموعتين مسبار (2 كيلوبايت) مع هذا الوقت للحصول على متوسط معدل استطالة النسخ.
- للحصول على معدل تحلل مرنا، تناسب دالة اضمحلال أسي، y = A·exp(-t/τ) إلى منطقة الاضمحلال النهائي لإشارات 5'و3'مرنا (على سبيل المثال، الشكل 4B). المعلمة المناسب، τ، هو متوسط عمر مرنا.
- (اختياري) تحليل التباين بين الخلايا في التعبير الجيني (على سبيل المثال، استجابة مستوى الخلية للحث الوارد في الشكل 4C)،استناداً إلى توزيع أرقام الرنا في كل خلية (محسوبة في الخطوة 9.5).
- (اختياري) باستخدام معلومات حول موقع بقعة على طول المحاور الرئيسية والثانوية من خلية (التي تم الحصول عليها من الخطوة 9.2)، وتحليل توطين mRNAs (الشكل 4D،E).
- (اختياري) تحليل التعريب المشترك لـ 5 و3 'mRNAs (الشكل 5) من خلال مقارنة توطين البقع المكتشفة في قنوات Cy5 و Cy3.
- تحميل صور من الخرز متعددة الألوان (الخطوة 8.6) في وظيفة spotFinderF في microbeTracker والحصول على إحداثيات من تينرويدات حبة في القنوات Cy5 و Cy3. استخدام قائمة إحداثيات المركزية لحساب مصفوفة تحويل affine، الذي يعلم كيف يتم تحويل قنوات Cy5 و Cy3 وتناوب فيما يتعلق بعضها البعض29.
- تطبيق مصفوفة تحويل affine إلى صور Cy5 و Cy3 FISH لتحويل صور Cy3 في إحداثيات Cy5. تصنيف إذا كان موضع مشترك مع بقعة أخرى في قناة مختلفة. على سبيل المثال، يعتبر بقعة في قناة Cy5 أن تكون موضعية مع بقعة أخرى في قناة Cy3 إذا كانت المسافة بين 150 نانومتر(الشكل 5).
- تحليل عدد البقع Cy5 تصنف على أنها "موضعية" مع Cy3 البقع في كل نقطة زمنية. أيضا، تحليل شدة البقع المشتركة(الشكل 5).
النتائج
ويبين الشكل 3 الصور التمثيلية من هذا البروتوكول smFISH. حقل كامل من الرؤية (86.7 μm × 66.0 μm باستخدام إعداد المجهر لدينا مفصلة في جدول المواد)يظهر ~ 500 E. الخلايا القولونية المنتشرة في جميع أنحاء الميدان (الشكل 3A). إذا كانت كثافة الخلايا أعلى بكثير مما هو موضح في هذه الصورة، يصبح من الصعب تجزئة الخلايا التلقائية حيث أن خوارزميات التجزئة لا تحدد الخلايا الفردية بشكل موثوق عندما تلمس الخلايا بعضها البعض. يحتاج المرء إلى ضبط تركيز الخلايا ووقت الحضانة المستخدم للالتزام السطحي (الخطوة 5.1) لتحقيق الكثافة المثلى للخلايا في مجال الرؤية.
وينبغي أن تظل مورفولوجيا الخلايا في صور النقيض المرحلة مماثلة لتلك التي من الخلايا الحية لأغراض تجزئة(الشكل 3A-C). إذا كانت الخلايا أكثر من permeabilized، يتغير مورفولوجيا الخلية (مثل "أشباح"؛ الشكل التكميلي 1). في هذه الحالة، قد يقلل المرء من مدة معالجة الإيثانول بنسبة 70٪ في الخطوة 5.3.
قبل التعريفي كان متوسط مستوى التعبير lacZ ~ 0.03 mRNAs في الخلية الواحدة، بما يتفق مع التقارير السابقة15،30. أيضا، توزيع كثافة بقعة مرنا لاكز قبل التعريفي لم تتناسب بشكل جيد مع التوزيع العادي أو توزيع بواسون بسبب وجود بقع مع كثافة عالية(الشكل 3D،E). وهذا يشير إلى أن معظم البقع المكتشفة تحت الدولة lacZ المكبوتة تمثل مرناً واحداً، ولكن مجموعة صغيرة من البقع تحتوي على أكثر من مرناً لاكز. لعزل السكان مع مرنا لاكز واحد، استخدمنا نموذج خليط غاوسي مع اثنين من مكونات الخليط (insets في الشكل 3D،E). ثم، تم اتخاذ متوسط من غاوسي الأول كثافة متوسط بقعة مرنا واحدة (على سبيل المثال، ذروة المنحنى الأسود في الشكل 3D)وتستخدم لتحويل كثافة بقعة إلى عدد من mRNAs، عن أي بقع اكتشفت في تجربة الوقت. لحساب العدد الإجمالي للـ mRNAs داخل الخلية، تم تجميع كثافة البقعة المُطَوَّرَة في كل خلية (الشكل 3F)19.
عندما يكون مستوى التعبير من الميرنا lacZ منخفضة، هناك واحد أو اثنين من بقع لاكنزا محدودة الريبوز مرنا فصل مكاني داخل خلية. وبالتالي، يمكن تحليل صور هذه البقع من قبل 2D غاوسي المناسب لكثافتها والتعريب.
عندما يكون مستوى التعبير مرتفعًا ، بحيث تتداخل البقع مع بعضها البعض داخل الخلية ، لا يقوم التركيب 2D Gaussian بالتكين الكمي الموثوق به. في هذه الحالة، ينبغي حساب مستوى مرنا عن طريق قسمة مجموع، إشارة الفلورس التي طرحت في الخلفية داخل خلية مع متوسط شدة مرناواحدة 19.
عندما يتم تحريض التعبير عن lacZ، وإشارة من 5'lacZ مرنا يزيد أولا وأن من 3'lacZ مرنا يزيد في وقت لاحق lacZ (الشكل 4B). إذا تم قمع التعبير عن lacZ، كل من 5 'و 3'lacZ الإشارات مرنا انخفاض مع بعض التأخير في ما بين (الشكل 4B). lacZ للحصول على معدل استطالة النسخ، فإن ارتفاع إشارات 5 'و3' يتناسب أولاً مع الخطوط (الشكل 4B) ، ويتم أخذ الفرق في تقاطعات x كوقاة لـ RNAPs للسفر المسافة بين منطقتي التحقيق (2000 nt). ويمكن قياس معدل الاستطالة النسخ من كل تجربة الوقت بالطبع ويمكن حساب الانحرافات المعيارية من التكرارات التجريبية. وكان متوسط معدل الاستطالة النسخ 15-30 nt / ث في ظل ظروفنا التجريبية19.
بالإضافة إلى ذلك، تم الحصول على معدل تحلل مرنا (عكس عمر الحمض النووي الريبي) عن طريق تركيب منطقة التحلل مع دالة أسية(الشكل 4B). لدينا بيانات الدورة الزمنية يحتوي على تدهور مرنا أثناء وبعد النسخ31. نحن تناسب النقاط الزمنية بعد 3 'بدأ المرنا في الاضمحلال (ر > 6 دقائق) للتحقيق في تدهور mRNAs صدر. حصلنا على ~ 90 ق كمتوسط عمر إما 5 'أو 3' lacZ مرنا19.
ويمكن حساب معدل بدء النسخ من ميل زيادة الإشارة 5 بعد الحث(الشكل 4B، الأزرق) ، أو من متوسط عدد مرنا في حالة ثابتة (وهو معدل البدء مقسوما على معدل التدهور). وعلاوة على ذلك، يمكن تقدير احتمال إنهاء النسخ السابق لأوانه، إما بأخذ النسبة بين منحدر زيادة الإشارة 3 'مقابل زيادة إشارة5'32 أو بين مستويات ثابتة من 3 'و 5' مناطق مرنا19.
لأن smFISH هو تقنية خلية واحدة ، يمكننا تحليل تقلب الخلايا إلى الخلية في النسخ. على سبيل المثال، يمكن للمرء أن يحلل النسبة المئوية للخلايا التي تعبر عن اللاكز مرنا بعد إضافة IPTG(الشكل 4C). يمكن للمرء أيضا معالجة ما إذا كان تغيير التعريب مرنا بعد التعريفي. لاحظنا أن 5 'و 3' بقع الكارنيا مرنا تتحرك قليلا إلى الخارج، بعيدا عن مركز الخلية(الشكل 4D, E),بما يتفق مع التقرير السابق33.
وأخيرا، يمكن تحليل التعريب المشترك بين 5 'و 3' بقع مرنا أن تكون مفيدة (الشكل 5A). على سبيل المثال، في حالة مكبوتة (الوقت صفر)، حوالي 25٪ من 5'mRNA البقع هي موضعية مع بقعة مرنا 3'. في ر = 1 دقيقة، كما لو أن العديد من loci الجينات لديها 5 'مرنا التوليف، ولكن ليس بعد 3'MRNA التوليف، معظم 5'mRNA البقع هي في حد ذاتها دون 3 'MRNA إشارة (أي، احتمال منخفض من التعريب المشترك). ومع ذلك، عندما يظهر مرنا 3 '(أي، ر = 2 دقيقة)، يزيد احتمال التعريب المشترك (السهم الأرجواني في الشكل 5A،B). هذه النقطة الزمنية، عندما يصبح التعريب المشترك متكرر، يعتمد على معدل الاستطالة النسخ. يمكن استخدام مؤامرة الكثافة 2-D من 5 'و 3' lacZ أرقام مرنا داخل كل بقعة التعريب المشترك الكشف في هذه النقطة الوقت لاستنتاج كثافة RNAPs على الجين lacZ (الشكل 5C). كما ذكرت سابقا19، وأرقام 5 'مرنا في هذه المؤامرة تشير إلى أن معظم loci lacZ لديها أقل من 10 RNAPs على الحمض النووي عندما يتم تحريض التعبير lacZ بواسطة 1 mM IPTG. بالإضافة إلى ذلك، ترتبط أرقام 3 'مرنا في هذه المؤامرة إلى تجميع RNAPs34. حقيقة أن عدد 3 'مرنا 'قريب من واحد يعني أن ما يقرب من واحد فقط RNAP يدخل المنطقة 3 'التحقيق. وهذا يشير إلى أن RNAPs على الجين lacZ يتم فصل مكاني، بدلاً من تشكيل كتلة (أو "قافلة").
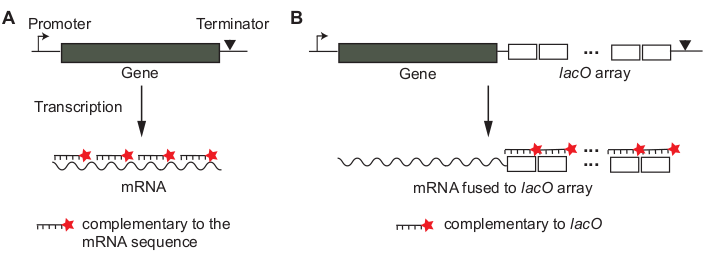
الشكل 1: تصميم مسابير smFISH لـ مرنا من الاهتمام. (أ) طريقة التجانب. يتم اختيار تسلسلات من oligonucleotides الحمض النووي قصيرة (~ 20 BP في الطول) بحيث يمكن أن تغطي مرنا من الفائدة. مسابير أليوغنوكليوتيد تحمل جزيء صبغة فلورية. (B) أسلوب صفيف. مجموعة غير ترميزية من التسلسلات الترادفية (على سبيل المثال، "صفيفال lacO") تنصهر بشكل نسخي إلى مرنا من الفائدة. ويستخدم المسبار المسمى بفلورسنتليس المكمل للوحدة المتكررة (على سبيل المثال، مسبار اللاكوس بطول 17 بريتيش كلبي) لتضخيم إشارة مرنا. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
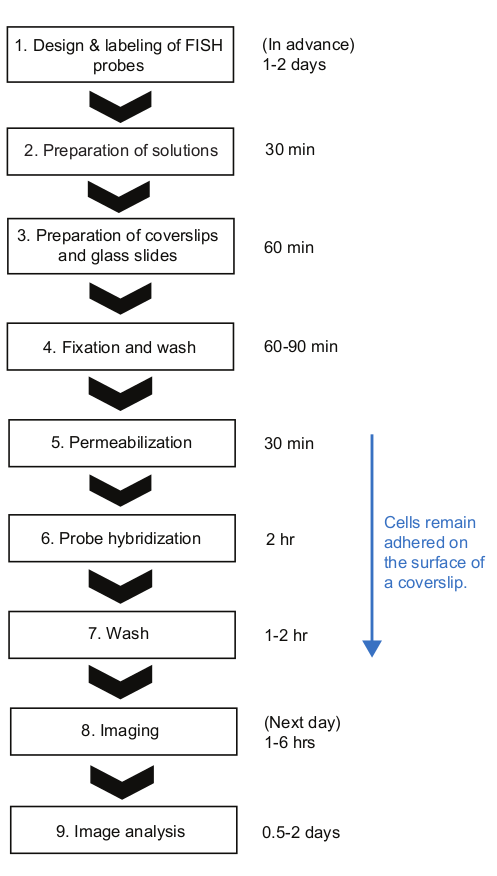
الشكل 2: التخطيطي للإجراءات التجريبية SMFISH والمدة الزمنية لكل خطوة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 3: تحليل صور smFISH. (أ-ج) smFISH صورة المجهر من 5 'لاك ميرنا (أحمر) و 3 'lacZ مرنا (الأخضر) في البرية من نوع E. القولونية (MG1655) نمت في متوسط M9 الحد الأدنى تكمل مع 0.2٪ الجلسرين، 0.1٪ أحماض كازامينو، و 1 ملغم/ لتر الثيامين في 30 درجة مئوية (أ) صورة تمثيلية لعينة من t = 3 دقائق بعد الحث مع 0.05 mM IPTG في t = 0 دقيقة والقمع مع 500 mM الجلوكوز في ر = 1.5 دقيقة. المرحلة النقيض واثنين من الصور fluorescence من Cy5 (ل5'lacZ مرنا، الأحمر) و Cy3 (ل 3' lacZ مرنا، الأخضر) كانت متراكبة مع التلوين الزائفة. lacZ تُظهر الصورة حقلاً كاملاً من 86.7 ميكرومتر × 66.0 ميكرومتر. شريط مقياس، 5 ميكرومتر. (B) التكبير في نسخة من منطقة صغيرة (مربع أصفر) في (A). تظهر الخطوط العريضة للخلية باللون الأبيض، وتظهر بقع الفلوريسين التي تم تحديدها من تحليل الصور بنقاط حمراء. شريط مقياس، 1 ميكرومتر. (C) الكشف عن الخطوط العريضة للخلية والبقع الفلورية تحت حالة تعبير عالية (ر = 4 دقائق بعد الاستقراء مع 1 mM IPTG). شريط مقياس، 1 ميكرومتر. (D-E) توزيعات من 5 'و 3' مرنا بقعة كثافة تقاس قبل إضافة IPTG (الدولة المكبوتة). تظهر المدرجات البيانية مع اثنين من الدالات الغاوسية (الأسود والرمادي) القيم المتوسط من نموذج الخليط الغاوسي. Inset يظهر الكم الكم من مؤامرة من الأرقام العشوائية التي تم إنشاؤها من نماذج خليط غاوسي وقياس تجريبيا كثافة النقطة مرنا (ن = 1040 ل 5'مرنا و 680 ل3 'مرنا). (واو)المعلومات التي تم الحصول عليها لخلية فردية مدببة في لوحة (B). بالنسبة للخلية (i) تم تحديد بقع في قنوات Cy5 و Cy3، وتم تحديد كثافتها (I) والتنسيق على طول المحور القصير والطويل للخلية(d, l)من تركيب 2D Gaussian. بعد أن تم تلخيص كثافة بقعة التطبيع لتحقيق العدد الإجمالي لل 5 'أو 3'mRNAs في هذه الخلية. أيضا، يمكن تحليل التعريب المشترك بين النقاط من قنوات مختلفة كما هو موضح في المثال المبين في الشكل 5. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 4: تحليل حركية في الجسم الحي للنسخ والتحلل مرنا. (أ) صور تخطيطية وتمثيلية لتجارب smFISH ذات لونين تقيس التغيرات في مستويات الـ lacZ mRNA بمرور الوقت. خطوط منقط الأحمر والأخضر تشير إلى Cy5 أو Cy3B المسمى تحقيقات oligonucleotide التي تهجين إلى 1 كيلو بايت طويلة 5 ' و 3 ' مناطق مرنا من لاك مرنا في E. القولونية، على التوالي. كما تظهر تراكبات صورتين من الفلوريسنس مع صورة تباين المرحلة في نقاط زمنية مبينة بعد الحث مع 0.2 mM IPTG في t = 0 دقيقة. تم قمع النسخ مع 500 mM الجلوكوز في ر = 1.5 دقيقة. شريط مقياس، 1 ميكرومتر. وقد تم تعديل هذا الرقم من كيم وآخرون19. (B)5 'و 3' أرقام مرنا lacZ لكل خلية مع مرور الوقت، خلال التجربة الموصوفة في لوحة (A). أشرطة الخطأ هي bootstrapped SEMs. تم تحليل ما لا يقل عن 1200 خلية في كل نقطة زمنية. كان الارتفاع الأولي لإشارات الـ 5 و 3 مرناً مناسبًا لخط (أزرق). وكان الفرق في تقاطع x 1.93 دقيقة، مما أسفر عن متوسط معدل الاستطالة النسخية من 17.3 nt /s. كان الاضمحلال النهائي لإشارات الـ 5 و 3 مرناً مناسبة مع وظيفة اضمحلال أسي (رمادي). تشير معلمات الملاءمة إلى أن متوسط عمر مرنا هو 1.52 دقيقة لـ 5 'مرنا و 1.66 دقيقة لـ 3'مرنا. (C) النسبة المئوية للخلايا التي تحتوي على بقع مرناً لاكية أو أكثر خلال التجربة الموضحة في (A). أشرطة الخطأ هي bootstrapped SEMs. (د)توطين بقعة على محور الخلية القصير. يمكن للمرء أن يحدد حجم قرب بقعة من الغشاء عن طريق تقسيم الموقع على طول المحور القصير (d) مع نصف عرض الخلية (ث). (E) تغيير في توطين بقع مرنا 5 'و 3' lacZ على طول محور الخلايا القصير خلال التجربة الموصوفة في (A). الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
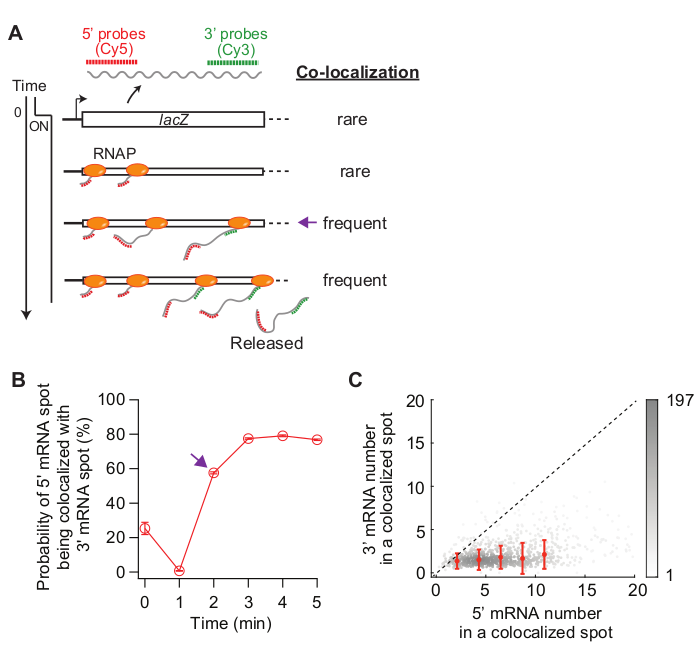
الشكل 5: تحليل التعريب المشترك للبقع 5'و3'mRNA. (A) التخطيطي يظهر توقع المشاركة في التعريب بين 5 'و 3' بقع مرنا بعد الحث. عندما يتم إجراء 3 'مرنا، احتمال وجود بقعة مرنا 5 'يجري المشتركة مع موضعية مع 3 'مرنا نقطة يزيد (السهم الأرجواني). (B) احتمالية التعريب المشترك بعد الحث مع 1 mM IPTG. يشير السهم الأرجواني إلى النقطة الزمنية حيث يصبح احتمال التعريب المشترك متكررًا أولاً وفقًا للمخطط في اللوحة (A). (C) عدد 5 'و 3' lacZ mRNAs داخل بقعة التعريب المشترك الكشف عن في ر = 2 دقيقة بعد التعريفي مع 1 M IPTG (مجموع البقع 841). تمثل النقاط الرمادية بقعًا فردية ذات موضع مشترك، بينما تمثل النقاط الحمراء متوسط البيانات ذات البينة. أشرطة الخطأ هي SEM. يشير ظل الرمادي إلى كثافة النقاط في منطقة معينة من الرسم البياني. الخط المنقط يشير إلى ميل 1. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
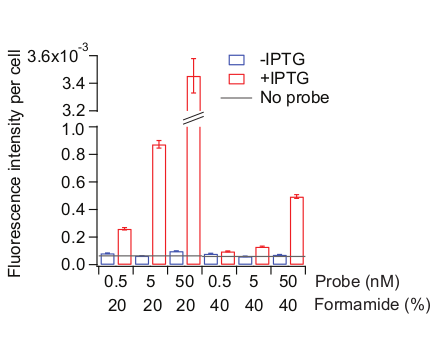
الشكل 6: تحسين حالة التهجين المجسّم. تم استخدام نوعين من العينات: خلايا MG1655 التي نمت كما هو موضح في الشكل 3 وتظل غير مستحثة (زرقاء) أو تعامل مع 0.5 mM IPTG لمدة 20 دقيقة (أحمر). تم إجراء حل التهجين التحقيق مع تركيزات مختلفة من تحقيقات (مجموع 72 Cy5-متقارنة تحقيقات تبليط المنطقة lacZ بأكملها) والفورماميد. كما تم تعديل تركيزات الفورماميد في محلول ما قبل التهجين ومحلول الغسيل، وفقا لذلك. يشير "لا مسبار" (الخط الرمادي) إلى مستوى الفلورس للخلايا المضافة IPTG التي تم علاجها بدون مسابير أثناء خطوة التهجين. تم حساب متوسط كثافة الفلوريسينت التي تم تطبيعها حسب منطقة الخلية (AU) من 300-800 خلية. أشرطة الخطأ هي bootstrapped SEMs. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
الشكل التكميلي 1: مشوهة موروفولولوجيا الخلية بسبب أكثر من permeabilization. تراكب من النقيض المرحلة (مقياس الرمادي)، 5 'lacZ مرنا (Cy5، أحمر)، و3 'lacZ مرنا (Cy3، الأخضر) صور من الخلايا MG1655 5 دقيقة بعد التعريفي مع 1 mM IPTG. (A) مثال يظهر خليط من الخلايا الطبيعية والخلايا المميّزة بشكل مفرط التي تفتقر إلى مورفولوجيا طبيعية (يشار إليها بسهام وردية). (B) مثال يظهر خلايا "شبحي" تتجمع معا. شريط مقياس = 1 μm. الرجاء انقر هنا لتحميل هذا الملف.
| مياه DEPC | ||||
| أضف 0.1% DEPC إلى الماء فائق البور واحتضان الزجاجة (المغطاة) في فرن 37 درجة مئوية بين عشية وضحاها والأوتلاف في اليوم التالي. | ||||
| DEPC PBS (10X) | ||||
| اخلط ما يلي: | ||||
| 80 غرام | NaCl (نهائي 1.37 M) | |||
| 2 غرام | KCl (النهائي 27 mM) | |||
| 14.2 غرام | Na2HPO4 (نهائي 100 mM) | |||
| 2.7 غرام | KH2PO4 (نهائي 20 mM) | |||
| الترابور المياه إلى 1 لتر | ||||
| فلتر (0.22 ميكرومتر) في زجاجة. | ||||
| إضافة 0.1% DEPC واتبع التعليمات للمياه DEPC. | ||||
| لجعل 1X الحل، وتمييع 10 مرات مع المياه DEPC. | ||||
| 1M DEPC فوسفات الصوديوم العازلة، وhH 7.4 | ||||
| اخلط ما يلي: | ||||
| 115 غ | Na2HPO4 | |||
| 22.8 غرام | NaH2PO4 | |||
| البور الفائقة المياه إلى 1 لتر | ||||
| فلتر (0.22 ميكرومتر) في زجاجة. | ||||
| إضافة 0.1% DEPC واتبع التعليمات للمياه DEPC. | ||||
| 4X حل التثبيت (16٪ الفورمالديهايد) | ||||
| 5 مل | 20% فورمالديهايد | |||
| 500 ميكرولتر | مياه DEPC | |||
| 750 ميكرولتر | 1M DEPC فوسفات الصوديوم العازلة، وhH 7.4 | |||
| يُخزن عند 4 درجة مئوية لمدة تصل إلى 2-4 أسابيع. | ||||
| تنبيه: الفورمالديهايد سام. ارتداء القفازات واستخدام غطاء محرك الدخان عند جعل هذا الحل. | ||||
| محلول الغسيل | ||||
| اخلط ما يلي: | ||||
| 10 مل | فورنامايد (نهائي 25%) | |||
| 4 مل | 20X SSC (2X النهائي) | |||
| ملء المياه DEPC إلى 40 مل | ||||
| فلتر (0.22 ميكرومتر) وتخزينها عند 4 درجات مئوية | ||||
| تنبيه: الفورماميد سام. ارتداء القفازات واستخدام غطاء محرك الدخان عند جعل هذا الحل. | ||||
| حل ما قبل التهجين | ||||
| 200 ميكرولتر | فورنامايد (نهائي 20%) | |||
| 100 ميكرولتر | 20X SSC (2X النهائي) | |||
| 10 ميكرولتر | 100X VRC (1X النهائي) | |||
| 25 ميكرولتر | 4 في المائة (ث/الخامس) BSA (نهائي 0.1 في المائة) | |||
| 685 ميكرولتر | مياه DEPC | |||
| ملاحظة: دوامة الأسهم VRC قبل اتخاذ 10 ميكرولتر. | ||||
| تنبيه: فورماميد سام وتيراتوجين معروف. ارتداء القفازات والتعامل معها تحت غطاء محرك الدخان. | ||||
| حل التهجين التحقيق | ||||
| 200 ميكرولتر | فورنامايد (نهائي 20%) | |||
| 100 ميكرولتر | 20X SSC (2X النهائي) | |||
| 10 ميكرولتر | 100X VRC (1X النهائي) | |||
| 25 ميكرولتر | 4 في المائة (ث/الخامس) BSA (نهائي 0.1 في المائة) | |||
| 10 ميكرولتر | 40 ملغم / مل E. القولونية tRNA (النهائي 0.4 ملغ / مل) | |||
| 200 ميكرولتر | 50٪ كبريتات ديكستران (نهائي 10٪) | |||
| x ميكرولتر | 5 'مرنا التحقيق مجموعة (من الخطوة 1.12) إلى 4 nM النهائي. | |||
| y μL | 3'mRNA مجموعة التحقيق (من الخطوة 1.12) إلى 4 nM النهائي. | |||
| - | DEPC المياه لجعل حجم إجمالي 1 مل | |||
| ملاحظة: إضافة سلفات ديكستران الماضي. لأنه لزج جدا، وقطع نهاية تلميح ماصة قبل اتخاذ 200 μL من الأسهم 50٪. بعد إضافة كبريتات dextran، ماصة صعودا وهبوطا لتجانس الحل. | ||||
الجدول 1: وصفات الحلول المستخدمة.
Discussion
هنا، قدمنا بروتوكول smFISH لقياس حركية مرنا في الإشريكية القولونية. في بروتوكولات smFISH المنشورة سابقا للبكتيريا23، تم الاحتفاظ بالخلايا في الأنابيب حتى نهاية البروتوكول ، وهذا هو حتى تكون جاهزة للتصوير. في حين أن له فوائد كثيرة، مثل الحد الأدنى من الربط غير المحدد من المسابير الفلورية على سطح الغطاء23، فمن الصعب اتباع هذه البروتوكولات عندما يكون هناك العديد من العينات من تجربة مسار زمني. أولاً، يحتاج حجم كبير نسبياً من الخلايا (>1 مل) إلى أخذ عينات منه وحتى حصاده قبل التثبيت. ثانياً، تحتاج عينات الخلايا إلى الطرد عدة مرات لتبادل الحلول والغسيل بعد خطوة التهجين. في بروتوكول لدينا، يتم خلط حجم صغير (< 1 مل) من الثقافة مباشرة مع حل تحديد في أنبوب 1.5-مل، مما يساعد على "تجميد" بسرعة حالة الخلية في لحظة أخذ العينات. أيضا، تبقى الخلايا تعلق على السطح طوال العملية، ويمكن تبادل حلول مختلفة بسرعة عن طريق التعرق السوائل مع نظام الترشيح فراغ وتطبيق حل قطرات في وقت واحد مع ماص متعدد القنوات. هذا الاختلاف يجعل لدينا بروتوكول مفيد للغاية عندما يكون هناك عدد كبير من العينات تحتاج إلى معالجة في وقت واحد. باستخدام بروتوكول لدينا، 12-48 عينات يمكن التعامل معها في وقت واحد، ويمكن الانتهاء من الإجراء كامل FISH في غضون ~ 8 ساعات، حول كمية مماثلة من الوقت اللازم للحصول على عينات قليلة(الشكل 2). على الرغم من أننا استخدمنا التعبير عن lacZ في الإشريكية القولونية كمثال، البروتوكول ينطبق على نطاق واسع على الجينات المختلفة والأنواع البكتيرية مع الاعتبارات التي نوقشت أدناه.
بالنسبة للجينات المختلفة ، فإن أول شيء يجب مراعاته هو تحقيقات smFISH. يمكن للمرء أن تصميم تحقيقات oligonucleotide التي بلاط مرنا من الفائدة (الشكل 1A)13. في هذا النهج التحقيق "التبليط"، كل مسبار هو ~ 20 قاعدة طويلة وتسمى مع الفلوروفور في 5 'أو 3' 10000000000000000000000000000000000000000000000000000000000000000000000000000000000000 هذه الاستراتيجية مريحة حيث لا توجد حاجة إلى التلاعب الجيني. وبدلاً من ذلك، يمكن إدراج تكرار تناظري من ~20 بت في الطنين، وهو غريب عن التسلسل الجيني (على سبيل المثال، مجموعة من تسلسل اللاكو في Caulobacter crescentus14)في المنطقة غير المترجمة لجين ذي أهمية، ويستخدم مسبار واحد مكمل لوحدة التكرار لتسمية نهج مرنا ("صفيف"؛ الشكل 1 ب). في كلتا الحالتين، تزين الفلوروفهورات المتعددة مرنا، مما يعطي إشارة الفلورانس المضخمة التي يمكن تمييزها بسهولة عن مسبار واحد غير محدد داخل خلية.
يعتمد اختيار نهج "التبليط" أو "الصفيف" على التحكم السلبي، وهي عينة يتم فيها اختبار الربط غير المحدد للمسبارات لأنها تفتقر إلى الRNA المستهدف. بالنسبة إلى تحقيقات التبليط (الشكل 1A) ، يمكن أن تكون سلالة متحولة بدون جينة ذات فائدة أو حالة ، والتي لا يتم فيها نسخ الجين (على سبيل المثال ، قمع lacZ)بمثابة تحكم سلبي لاختبار الربط غير محدد من المسابير. لsmFISH على أساس مجموعة (الشكل 1B) ، سلالة من نوع البرية تفتقر إلى مجموعة يمكن أن تكون بمثابة سيطرة سلبية لأنه لا يحتوي على مواقع ملزمة للتحقيقات.
قد تعتمد ظروف التهجين المثلى على تسلسلات التحقيق وحتى على اختيار الأصباغ الفلوروفور. نحن الأمثل شرط التهجين لمجموعات مسبار lacZ عن طريق الحفاظ على درجة حرارة التهجين عند 37 درجة مئوية واختبار تركيزات مختلفة من مجموعات التحقيق و formamide في حل التهجين. تميل التركيزات الأعلى من الفورماميد إلى تقليل كل من غير محدد ومحدد ملزم26,35. نوصي بتغيير التهجين وظروف الغسيل بشكل منهجي مع الحفاظ على وقت التهجين ودرجة الحرارة نفسها. كما يصبح الشرط أكثر صرامة، على حد سواء غير محددة والمحددة ملزمة انخفاض(الشكل 6). من المهم العثور على نقطة يبدأ فيها الربط غير المحدد في التسوية تحت عتبة مقبولة دون المساس بـ Binding المحدد. على سبيل المثال، استخدمنا مستوى الإشارة الذي تم الحصول عليه دون أي تحقيقات ("لا تحقيقات") كعتبة(الشكل 6).
ويقتصر أسلوب smFISH ذات اللونين الذي يصف منطقتين منفصلتين من الميرنا على الجينات الطويلة. لقياس معدل استطالة النسخ ، استفدنا من حقيقة أن lacZ طويل (3075 bp) ويمكن أن يسببه IPTG التعبير عنه. عندما يكون الجين قصيرًا ، من الصعب تصميم مجموعتين من مسبار التبليط (بالقرب من 5 'و3' ينتهي) وحل تأخير الوقت بين ظهور مناطق 5 'مقابل 3' مرنا. في هذه الحالة، يمكن للمرء أن عد mRNAs الوليدة في حالة ثابتة بواسطة smFISH وتحليل توزيعها مع نموذج التحليلية التي لديها معدل الاستطالة النسخ كمعلمة المناسب20. أيضا، عندما لا يكون الجين من الفائدة غير قابل للاختصاص، يمكن للمرء أن يعامل الخلايا مع ريفامبيسين في الوقت صفر وقياس التغير الزمني في 5 'و 3' مرنا المناطق الفرعية. ويمكن بعد ذلك التأخير من انخفاض إشارة مرنا 5 إلى أن من 3 'إشارة مرنا يمكن استخدامها لحساب معدل استطالة النسخ كما فعلت سابقا31.
وأخيراً، فإن بروتوكول smFISH متعدد الاستخدامات ويمكن دمجه مع مخططات وضع العلامات الأخرى. سابقا, تم تصور موضع الحمض النووي جنبا إلى جنب مع mRNAs من خلال الجمع بين سمك الحمض النووي مع أي من الحمض النووي FISH14 أو نظام الفلورسنت مراسل المشغل20. ويمكن تصور منتجات البروتين عن طريق أداء المناعةfluorescence جنبا إلى جنب مع سمك مرنا14,36. أيضا، يمكن أن تكون جنبا إلى جنب مع ثلاثية الأبعاد فائقة الدقةالمجهرية 37 لتصور mRNAs في جميع الأبعاد الثلاثة38،39.
Disclosures
ويعلن أصحاب البلاغ أنهما لا يملكان مصالح مالية متنافسة.
Acknowledgements
وقد تم تطوير هذا البروتوكول من قبل س. ك. خلال بحثها ما بعد الدكتوراه في مختبر الدكتور كريستين جاكوبس فاغنر في معهد هوارد هيوز الطبي ومعهد العلوم الميكروبية في جامعة ييل. نشكر الدكتورة جاكوبس فاغنر وأعضاء مختبرها على مختلف المدخلات خلال تطوير الأسلوب ولورا تروير على القراءة النقدية للمخطوطة. س. ك. يعترف بالدعم من برنامج علماء سيرل؛ يعترف ك. ف. بدعم جائزة جيمس جيمس دارس بريبل للأبحاث من جامعة إلينوي.
Materials
| Name | Company | Catalog Number | Comments |
| Bacterial strain | |||
| Escherichia coli MG1655 | |||
| Chemicals, peptides, and others | |||
| Acetonitrile | Sigma-Aldrich | 34851 | UPLC buffer B |
| Ammonium chloride | Fisher Chemical | A661-500 | To make M9 medium |
| Bovine serum albumin (BSA) | Sigma-Aldrich | B2518 | Probe hybridization |
| Calcium chloride | Acros Organics | 349610250 | To make M9 medium |
| Casamino acid | BD Difco | 223050 | To make M9 medium |
| Cy3B NHS ester | GE Healthcare Life Sciences | PA63101 | Fluorophore for FISH probes |
| Cy5 NHS ester | GE Healthcare Life Sciences | PA15101 | Fluorophore for FISH probes |
| DEPC | Sigma-Aldrich | D5758 | |
| Dextran sulfate | Millipore | S4030 | Probe hybridization |
| Dextrose | Fisher Chemical | D16 | To repress the expression of lacZ |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | To dissolve fluorophores |
| E. coli tRNA | Sigma-Aldrich | R1753 | Probe hybridization |
| Ethanol | Decon Laboratories | 2701 | Used in DNA purification, lysis, and cleaning coverslips |
| FISH probes | Biosearch Technologies | Sequences are published in ref#16 | |
| Formaldehyde | Ladd Research Industries | 20295 | Fixation |
| Formamide | American Bio | AB00600 | Probe hybridization, pre-hybridization, and wash |
| Glycerol | Americanbio | AB00751-01000 | To make M9 medium |
| Isopropylthio-β-galactoside (IPTG) | Invitrogen | 15529019 | lacZ induction |
| Magnesium sulfate | Fisher Chemical | M65-500 | To make M9 medium |
| 2-Nitrophenyl β-D-fucopyranoside (ONPF) | Santa Cruz Biotechnology | sc-216258 | lacZ repression |
| Picodent twinsil 22 | Picodent | 1300 1000 | Sealant |
| Poly-L-lysine | Sigma-Aldrich | P8920 | To treat the coverslip surface |
| Potassium chloride | Fisher BioReagents | BP366-500 | To make PBS |
| Potassium phosphate monobasic | Fisher BioReagents | BP362-500 | To make PBS and M9 medium |
| Rifampicin | Sigma-Aldrich | R3501 | To stop transcription initiation |
| Saline-sodium citrate buffer (SSC) | Invitrogen | AM9763 | Probe hybridization, pre-hybridization, and wash |
| sodium bicarbonate | Fisher BioReagents | BP328-500 | Fluorophore-probe conjugation |
| Sodium chloride | Fisher BioReagents | BP358-1 | For DNA purification, PBS and M9 medium |
| Sodium phosphate dibasic | Fisher BioReagents | BP332-500 | To make PBS and M9 medium |
| Sodium phosphate monobasic | Fisher BioReagents | BP329-500 | To make a sodium phosphate buffer |
| Super PAP Pen | Invitrogen | 8899 | Hydrophobic marker for coverslips |
| TetraSpek microspheres | Invitrogen | T7280 | Controls for multi-channel registration |
| Thiamine | Sigma-Aldrich | T1270 | To make M9 medium |
| Triethylammonium acetate | Sigma-Aldrich | 90358 | UPLC buffer A |
| Vanadyl ribonucleoside complex (VRC) | Sigma-Aldrich | 94742 | Probe hybridization and pre-hybridization |
| Equipment | |||
| C18 column | Waters | Acquity BEH C18 column | |
| Countertop centrifuge | Eppendorf | 5425 | |
| Countertop incubator | Eppendorf | Thermomixer F1.5 | |
| Incubator (Oven) | Thermo Scientific | 51030514 | Gravity convection |
| Water purification system | Millipore | Milli-Q Reference | |
| Nanodrop | Thermo Scientific | 2000C | |
| Nitrogen gas | Building | For blow-drying coverslips and glass slides | |
| UPLC | Waters | Acquity UPLC system | |
| Vacuum and aspirator | Building | Aspirator is made of a filtration flask with a side arm. | |
| Vacuum concentrator | Labconco | 7810010 | Centrivap; to dry samples collected from UPLC. |
| Vortexer | Scientific Industries | Genie-2 SI-0236 | |
| Water bath shaker | New Brunswick | Innova 3100 | Critical for time-course experiments |
| Water bath sonicator | VWR | 97043-960 | To clean coverslips and glass slides |
| Tools | |||
| 1.5-mL tubes | Eppendorf | 22431021 | DNA lobind tubes |
| 1000-uL pipette tip box | Denville Scientific | P1126 | An empty box after using all the tips |
| Coplin jar | SPI | 01240-AB | To clean coverslips and glass slides |
| Coverslip | Fisher Scientific | 22-050-230 | 24x60 No1 |
| Filtered pipette tips | Denville Scientific | P1121,P1122,P1126 | SHARP® Precision Barrier Tips |
| Forceps | SPI | K35a | To handle clean coverslips and glass slides |
| Glass slide | Fisher Scientific | 12-544-1 | |
| Gloves | Microflex | MK-296-M | |
| Multichannel pipetter | Eppendorf | 2231300045 | To use in the washing step (#7) |
| Pipette | Gilson | P1000, P200, P20 | |
| Reagent reservoir | MTC Bio | P8025-1S | To use in the washing step (#7) |
| Syringe filter (0.22 um) | Millipore | SLGS033SS | |
| Timer | VWR | 62344-641 | |
| Software and algorithms | |||
| MATLAB | Mathworks | R2013 and up | https://www.mathworks.com |
| MicrobeTracker or Oufti | https://www.github.com/JacobsWagnerLab/MicrobeTracker | ||
| https://oufti.org/ | |||
| Stellaris Probe Designer | Biosearch Technologies | https://www.biosearchtech.com/support/tools/design-software/stellaris-probe-designer | |
| Microscope | |||
| CCD camera | Hamamatsu Photonics | Orca-II-ER | |
| Cy3 filter set | Chroma | 49004 | |
| Cy5 filter set | Chroma | 49006 | |
| Epi-fluorescence microscope | Nikon | Eclipse Ti | For phase-contrast and epi fluorescence |
| Fluorescence excitation source | Lumencor | SOLA-E | |
| Nikon Elements software | Nikon | software that controls the microscope setup | |
| Phase-contrast 100x objective | Nikon | Plan Apochromat (NA 1.45) | |
| Probe sequence | |||
| DNA oligos with C6 amino modification at the 5' end | Biosearch Technologies Inc | ||
| lacZ1 | GTGAATCCGTAATCATGGTC | 5' mRNA | |
| lacZ2 | TCACGACGTTGTAAAACGAC | 5' mRNA | |
| lacZ3 | ATTAAGTTGGGTAACGCCAG | 5' mRNA | |
| lacZ4 | TATTACGCCAGCTGGCGAAA | 5' mRNA | |
| lacZ5 | ATTCAGGCTGCGCAACTGTT | 5' mRNA | |
| lacZ6 | AAACCAGGCAAAGCGCCATT | 5' mRNA | |
| lacZ7 | AGTATCGGCCTCAGGAAGAT | 5' mRNA | |
| lacZ8 | AACCGTGCATCTGCCAGTTT | 5' mRNA | |
| lacZ9 | TAGGTCACGTTGGTGTAGAT | 5' mRNA | |
| lacZ10 | AATGTGAGCGAGTAACAACC | 5' mRNA | |
| lacZ11 | GTAGCCAGCTTTCATCAACA | 5' mRNA | |
| lacZ12 | AATAATTCGCGTCTGGCCTT | 5' mRNA | |
| lacZ13 | AGATGAAACGCCGAGTTAAC | 5' mRNA | |
| lacZ14 | AATTCAGACGGCAAACGACT | 5' mRNA | |
| lacZ15 | TTTCTCCGGCGCGTAAAAAT | 5' mRNA | |
| lacZ16 | ATCTTCCAGATAACTGCCGT | 5' mRNA | |
| lacZ17 | AACGAGACGTCACGGAAAAT | 5' mRNA | |
| lacZ18 | GCTGATTTGTGTAGTCGGTT | 5' mRNA | |
| lacZ19 | TTAAAGCGAGTGGCAACATG | 5' mRNA | |
| lacZ20 | AACTGTTACCCGTAGGTAGT | 5' mRNA | |
| lacZ21 | ATAATTTCACCGCCGAAAGG | 5' mRNA | |
| lacZ22 | TTTCGACGTTCAGACGTAGT | 5' mRNA | |
| lacZ23 | ATAGAGATTCGGGATTTCGG | 5' mRNA | |
| lacZ24 | TTCTGCTTCAATCAGCGTGC | 5' mRNA | |
| lacZ25 | ACCATTTTCAATCCGCACCT | ||
| lacZ26 | TTAACGCCTCGAATCAGCAA | ||
| lacZ27 | ATGCAGAGGATGATGCTCGT | ||
| lacZ28 | TCTGCTCATCCATGACCTGA | ||
| lacZ29 | TTCATCAGCAGGATATCCTG | ||
| lacZ30 | CACGGCGTTAAAGTTGTTCT | ||
| lacZ31 | TGGTTCGGATAATGCGAACA | ||
| lacZ32 | TTCATCCACCACATACAGGC | ||
| lacZ33 | TGCCGTGGGTTTCAATATTG | ||
| lacZ34 | ATCGGTCAGACGATTCATTG | ||
| lacZ35 | TGATCACACTCGGGTGATTA | ||
| lacZ36 | ATACAGCGCGTCGTGATTAG | ||
| lacZ37 | GATCGACAGATTTGATCCAG | ||
| lacZ38 | AAATAATATCGGTGGCCGTG | ||
| lacZ39 | TTTGATGGACCATTTCGGCA | ||
| lacZ40 | TATTCGCAAAGGATCAGCGG | ||
| lacZ41 | AAGACTGTTACCCATCGCGT | ||
| lacZ42 | TGCCAGTATTTAGCGAAACC | ||
| lacZ43 | AAACGGGGATACTGACGAAA | ||
| lacZ44 | TAATCAGCGACTGATCCACC | ||
| lacZ45 | GGGTTGCCGTTTTCATCATA | ||
| lacZ46 | TCGGCGTATCGCCAAAATCA | ||
| lacZ47 | TTCATACAGAACTGGCGATC | ||
| lacZ48 | TGGTGTTTTGCTTCCGTCAG | ||
| lacZ49 | ACGGAACTGGAAAAACTGCT | 3' mRNA | |
| lacZ50 | TATTCGCTGGTCACTTCGAT | 3' mRNA | |
| lacZ51 | GTTATCGCTATGACGGAACA | 3' mRNA | |
| lacZ52 | TTTACCTTGTGGAGCGACAT | 3' mRNA | |
| lacZ53 | GTTCAGGCAGTTCAATCAAC | 3' mRNA | |
| lacZ54 | TTGCACTACGCGTACTGTGA | 3' mRNA | |
| lacZ55 | AGCGTCACACTGAGGTTTTC | 3' mRNA | |
| lacZ56 | ATTTCGCTGGTGGTCAGATG | 3' mRNA | |
| lacZ57 | ACCCAGCTCGATGCAAAAAT | 3' mRNA | |
| lacZ58 | CGGTTAAATTGCCAACGCTT | 3' mRNA | |
| lacZ59 | CTGTGAAAGAAAGCCTGACT | 3' mRNA | |
| lacZ60 | GGCGTCAGCAGTTGTTTTTT | 3' mRNA | |
| lacZ61 | TACGCCAATGTCGTTATCCA | 3' mRNA | |
| lacZ62 | TAAGGTTTTCCCCTGATGCT | 3' mRNA | |
| lacZ63 | ATCAATCCGGTAGGTTTTCC | 3' mRNA | |
| lacZ64 | GTAATCGCCATTTGACCACT | 3' mRNA | |
| lacZ65 | AGTTTTCTTGCGGCCCTAAT | 3' mRNA | |
| lacZ66 | ATGTCTGACAATGGCAGATC | 3' mRNA | |
| lacZ67 | ATAATTCAATTCGCGCGTCC | 3' mRNA | |
| lacZ68 | TGATGTTGAACTGGAAGTCG | 3' mRNA | |
| lacZ69 | TCAGTTGCTGTTGACTGTAG | 3' mRNA | |
| lacZ70 | ATTCAGCCATGTGCCTTCTT | 3' mRNA | |
| lacZ71 | AATCCCCATATGGAAACCGT | 3' mRNA | |
| lacZ72 | AGACCAACTGGTAATGGTAG | 3' mRNA | |
References
- Bervoets, I., Charlier, D. Diversity, versatility and complexity of bacterial gene regulation mechanisms: opportunities and drawbacks for applications in synthetic biology. FEMS Microbiology Reviews. 43 (3), 304-339 (2019).
- Epshtein, V., Nudler, E. Cooperation between RNA polymerase molecules in transcription elongation. Science. 300 (5620), 801-805 (2003).
- Vogel, U., Jensen, K. F. The RNA chain elongation rate in Escherichia coli depends on the growth rate. Journal of Bacteriology. 176 (10), 2807-2813 (1994).
- Tennyson, C. N., Klamut, H. J., Worton, R. G. The human dystrophin gene requires 16 hours to be transcribed and is cotranscriptionally spliced. Nature Genetics. 9, 184 (1995).
- Singh, J., Padgett, R. A. Rates of in situ transcription and splicing in large human genes. Nature Structural & Molecular Biology. 16, 1128 (2009).
- Selinger, D. W., Saxena, R. M., Cheung, K. J., Church, G. M., Rosenow, C. Global RNA Half-Life Analysis in Escherichia coli Reveals Positional Patterns of Transcript Degradation. Genome Research. 13 (2), 216-223 (2003).
- Bernstein, J. A., Khodursky, A. B., Lin, P. -. H., Lin-Chao, S., Cohen, S. N. Global analysis of mRNA decay and abundance in Escherichia coli at single-gene resolution using two-color fluorescent DNA microarrays. Proceedings of the National Academy of Sciences. 99 (15), 9697-9702 (2002).
- Pérez-Ortín, J. E., Medina, D. A., Chávez, S., Moreno, J. What do you mean by transcription rate. BioEssays. 35 (12), 1056-1062 (2013).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6 (5), 377-382 (2009).
- Kuchina, A., et al. Microbial single-cell RNA sequencing by split-pool barcoding. BioRxiv. , 869248 (2019).
- Blattman, S. B., Jiang, W., Oikonomou, P., Tavazoie, S. Prokaryotic single-cell RNA sequencing by in situ combinatorial indexing. Nature Microbiology. , (2020).
- Femino, A., Fay, F., Fogarty, K., Singer, R. Visualization of single RNA transcripts in situ. Science. 280 (5363), 585-590 (1998).
- Raj, A., vanden Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877-879 (2008).
- Montero Llopis, P., et al. Spatial organization of the flow of genetic information in bacteria. Nature. 466 (7302), 77-81 (2010).
- So, L. -. h., et al. General properties of transcriptional time series in Escherichia coli. Nature Genetics. 43 (6), 554-560 (2011).
- Taniguchi, Y., et al. Quantifying E. coli proteome and transcriptome with single-molecule sensitivity in single cells. Science. 329 (5991), 533-538 (2010).
- Jones, D. L., Brewster, R. C., Phillips, R. Promoter architecture dictates cell-to-cell variability in gene expression. Science. 346 (6216), 1533-1536 (2014).
- Iyer, S., Park, B. R., Kim, M. Absolute quantitative measurement of transcriptional kinetic parameters in vivo. Nucleic Acids Research. 44 (18), 142 (2016).
- Kim, S., Beltran, B., Irnov, I., Jacobs-Wagner, C. Long-Distance Cooperative and Antagonistic RNA Polymerase Dynamics via DNA Supercoiling. Cell. 179 (1), 106-119 (2019).
- Wang, M., Zhang, J., Xu, H., Golding, I. Measuring transcription at a single gene copy reveals hidden drivers of bacterial individuality. Nature Microbiology. 4 (12), 2118-2127 (2019).
- Joo, C., Ha, T. . Labeling DNA (or RNA) for single-molecule FRET. 2012 (9), 1005-1008 (2012).
- Sambrook, J., Russell, D. W. . Standard Ethanol Precipitation of DNA in Microcentrifuge Tubes. 2006 (1), 4456 (2006).
- Skinner, S. O., Sepúlveda, L. A., Xu, H., Golding, I. Measuring mRNA copy number in individual Escherichia coli cells using single-molecule fluorescent in situ hybridization. Nature Protocols. 8 (6), 1100-1113 (2013).
- Adesnik, M., Levinthal, C. The synthesis and degradation of lactose operon messenger RNA in E. coli. Cold Spring Harbor Symposia on Quantitative Biology. 35, 451-459 (1970).
- Campbell, E. A., et al. Structural mechanism for rifampicin inhibition of bacterial RNA polymerase. Cell. 104 (6), 901-912 (2001).
- Raj, A., Tyagi, S., Walter, N. G. . Methods in Enzymology. 472, 365-386 (2010).
- Sliusarenko, O., Heinritz, J., Emonet, T., Jacobs-Wagner, C. High-throughput, subpixel precision analysis of bacterial morphogenesis and intracellular spatio-temporal dynamics. Molecular Microbiology. 80 (3), 612-627 (2011).
- Paintdakhi, A., et al. Oufti: an integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Molecular Microbiology. 99 (4), 767-777 (2016).
- Moffitt, J. R., Zhuang, X., Filonov, G. S., Jaffrey, S. R. . Methods in Enzymology. 572, 1-49 (2016).
- Yu, J., Xiao, J., Ren, X., Lao, K., Xie, X. S. Probing gene expression in live cells, one protein molecule at a time. Science. 311 (5767), 1600-1603 (2006).
- Chen, H., Shiroguchi, K., Ge, H., Xie, X. S. Genome-wide study of mRNA degradation and transcript elongation in Escherichia coli. Molecular Systems Biology. 11 (1), 781 (2015).
- Vogel, U., Sørensen, M., Pedersen, S., Jensen, K. F., Kilstrup, M. Decreasing transcription elongation rate in Escherichia Coli exposed to amino acid starvation. Molecular Microbiology. 6 (15), 2191-2200 (1992).
- Yang, S., et al. Transcription and translation contribute to gene locus relocation to the nucleoid periphery in E. coli. Nature Communications. 10 (1), 5131 (2019).
- Zenklusen, D., Larson, D. R., Singer, R. H. Single-RNA counting reveals alternative modes of gene expression in yeast. Nature Structural & Molecular Biology. 15 (12), 1263-1271 (2008).
- Fontenete, S., Guimarães, N., Wengel, J., Azevedo, N. F. Prediction of melting temperatures in fluorescence in situ hybridization (FISH) procedures using thermodynamic models. Critical Reviews in Biotechnology. 36 (3), 566-577 (2016).
- Sepúlveda, L. A., Xu, H., Zhang, J., Wang, M., Golding, I. Measurement of gene regulation in individual cells reveals rapid switching between promoter states. Science. 351 (6278), 1218-1222 (2016).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-Dimensional Super-Resolution Imaging by Stochastic Optical Reconstruction Microscopy. Science. 319 (5864), 810-813 (2008).
- Moffitt, J. R., Pandey, S., Boettiger, A. N., Wang, S., Zhuang, X. Spatial organization shapes the turnover of a bacterial transcriptome. eLife. 5, 13065 (2016).
- Fei, J., et al. Determination of in vivo target search kinetics of regulatory noncoding RNA. Science. 347 (6228), 1371-1374 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved