Method Article
利用两色单分子荧光在原位杂交中Escherichia coli探索大肠杆菌中空间和时间中的 mRNA 动力学
摘要
该协议描述了单分子荧光原位杂交(smFISH)用于测量mRNA合成和降解的体内动力学的应用。
摘要
单分子荧光原位杂交(smFISH)允许计算单个细胞中mRNA的绝对数量。在这里,我们描述了smFISH的应用,以测量在大肠杆菌的转录 率和mRNA降解率。由于 smFISH 基于固定细胞,我们在时间过程实验中在多个时间点执行 smFISH,即当细胞在基因表达诱导或抑制时发生同步变化时。在每个时间点,mRNA的亚区被光谱区分,以探测转录伸长和过早终止。该协议的结果还允许分析mRNA的细胞内定位和mRNA拷贝数中的异质性。使用此协议,可以在 8 小时内处理许多样本 (+50),就像几个样本需要的时间一样。我们讨论如何应用此协议来研究细菌细胞中不同mRNA的转录和降解动力学。
引言
从DNA到mRNA和蛋白质的遗传信息流动是最基本的细胞过程之一,其调节对细胞的健身很重要。细胞中的mRNA数量由两个动态过程(转录和mRNA降解)决定。然而,在单个细胞的时间和空间中如何调节转录和mRNA降解,仍不完全了解,这主要是由于缺乏在体内定量测量其动力学的实验方法。
基于从北方印迹、RT-PCR、RNA测序、基因表达微阵列等细胞群中提取的总mRNA的方法,可以测量mRNA水平的相对差异,并广泛用于分析转录率,,2、3、4、5,34或mRNA降解率6、7。75然而,它们不提供每个细胞的mRNA的绝对数量,因此,它们不适合探测转录起始率8。此外,由于mRNA是从细胞群体中提取的,因此无法测量单个细胞内mRNA的空间分布和mRNA拷贝数的变异性。
对单个细胞(scRNAseq)进行下一代RNA测序,可以量化基因组规模9中每个细胞的mRNA数量。然而,由于样品制备和高成本的挑战,仍然难以使用这种技术来测量转录动力学。特别是,由于mRNA丰度低10,11,scRNAseq在细菌中的应用在技术上是困难的。
单分子荧光原位杂交(smFISH)基于荧光标记单链探头的杂交,其序列与目标mRNA12,13,的补充。序列特异性杂交的概念与北方印迹或RT-PCR中使用的概念类似,但杂交在固定单元内就地完成,以保持mRNA的原生定位。单个mRNA的信号使用许多探针放大,长度为+20核苷酸(nt),杂交到mRNA的不同部分(图1A)13。13在这种"平铺"探针方法中,检测单个mRNA所需的探针数量对可测定的mRNA长度设定了下限。或者,感兴趣的mRNA可以转录地融合到串联Lac运算符序列的非编码数组中,使荧光标记的lacO探针的多个拷贝杂交到单个mRNA(图1B)14。14
smFISH已用于量化每个细胞在稳定状态下(即合成和衰变处于平衡状态时)的mRNA数量,并分析细菌细胞15、16、17,16中mRNA的均值和变异性。最近,smFISH已被应用于量化mRNA数字在非稳定状态,就在诱导或抑制基因表达在大肠杆菌18,19,20。,19,20然后,使用绝对mRNA拷贝数的时间变化来计算转录起始率、伸长率和终止率,以及mRNA降解率。对于此应用程序,传统的 smFISH 过程可能很繁琐,因为有许多样本(每个样本代表一个时间点)需要经过多个缓冲区交换步骤(即离心和洗涤)。在这里,我们描述了一个smFISH协议,其中样品处理步骤被显著简化,使细胞粘附在盖玻片的表面,并吸附液体与真空过滤系统14,19。14,以大肠杆菌中乳酸的表达E. coli为例,演示了完整的工作流程(图2),包括图像分析(图3),产生转录(启动、伸长和终止)和mRNA降解、mRNA表达中的细胞对细胞变异性以及mRNA定位。我们预计,该协议广泛适用于探测体内动力学和定位其他mRNA在各种细菌物种。
研究方案
1. smFISH 探头的准备
注:要用单个荧光素标记smFISH探针,请遵循基于NHS酯化学21的核酸寡核苷酸标签的标准协议。
- 设计 smFISH 探头。决定是使用"平铺"探针还是"阵列"探针(图1)来治疗感兴趣的基因。请参阅 关于 如何做出决定的讨论部分。
- 对于"平铺"探头(图1A),请使用在线探针设计器工具(例如, 参见材料表)。
- 对于"阵列"探针(图1B),执行 BLAST 序列搜索,以确保探针序列不与任何其他 mRNA 序列互补。
- 要研究 lacZ mRNA 转录和降解动力学,请使用两组 24 个探头,每组覆盖 lacZ (3,072 bp) 19 的第一个和最后1kb 区域。
注:以下探针组分别称为"5'mRNA探针"和"3'mRNA探针"。这些探头的顺序列在" 材料表"中。
- 在 5' 端将具有 C6 氨基链接器的 DNA 寡核苷酸排序为 DNA 寡核苷酸。将单个探头溶解在水中至 1 mM。
- 组合"5' mRNA 探针"和"3' mRNA 探针"集的等量探头。例如,对于 lacZ的 5' mRNA 探头组,组合每个探头的 20 μL(该探头中总共 24 种探针)。
- 执行乙醇沉淀22 的组合探针,以去除可抑制结合反应的初级胺和二级胺(如三叶草、甘氨酸和铵盐)的任何污染。最后,将DNA颗粒溶解在100μL水中(在探针中产生±4.5m的DNA)。
注:即使探头经过制造商的标准脱盐纯化,也建议执行此步骤。标准的过滤器净化可以代替乙醇沉淀,并除乙醇沉淀外。 - 选择两个光谱不同的荧光团与单功能NHS酯莫伊蒂,使5' 和3'mRNA探头集可以标记为差分。例如,为 5' mRNA 探头准备 Cy5 NHS 酯,为 3' mRNA 探头准备 Cy3B NHS 酯。将每种类型的荧光团溶解在无水 DMSO 中,最后为 20 mg/mL(±25 mM)。
- 在每次贴标反应前准备0.1 M碳酸氢钠(pH 8.5)。长时间暴露在空气中会降低其pH值并降低标签效率。
- 对于结合反应,结合以下:Cy5氟磷丝15μL(从步骤1.5),4μL的5'mRNA探针组(从步骤1.4),75μL碳酸氢钠(从步骤1.6)和7μL的水。用铝箔包裹管子,在室温下摇动3-6小时。
注:更长的孵育时间并不一定会提高标签效率。此外,如果保持成分的浓度,反应可以放大或缩小。 - 对 3' mRNA 探头集和相应的荧光团(即 Cy3B NHS-酯)重复上述步骤。
- 执行乙醇沉淀22 去除无反应的染料分子。将颗粒溶解在 ±50 μL 的 TE 缓冲液中(10mM Tris-HCl pH 8.0,带 1mM EDTA)。
- 使用UV-Vis光谱仪估计DNA和荧光的浓度。
- 在 260 nm 和 559 nm (Cy3B) 或 649 nm (Cy5) 时测量吸光度。如果样品浓度过重,不能进行精确测量,则将样品的 1 μL 稀释至 10 μL。
- 将吸收度转换为浓度:
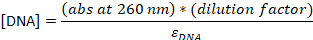
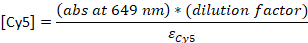
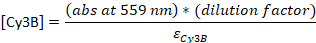
εDNA = 0.2 μM-1(对于 20-nt 单链 DNA),εCy5 =-1 0.25 μM-1,和 εCy3B = 0.13-1μM-1-1
注:[DNA] 是溶液中总探头的浓度。单个探头的浓度大约低24倍。从这一点开始,总探头的浓度将用作"探针浓度"。如果 [DNA] 和 [染料] 之间的比率为 1,则以下 HPLC 步骤可能会跳过23,并且样品应在 TE 缓冲液中稀释为最终 4-5 μM。
- (推荐)使用 HPLC 从未标记的探头和自由染料中净化带标签的探头。
注:虽然此附加纯化步骤会导致样品损失,但它对下游应用是有益的。去除未标记的DNA探针将增加mRNA目标的荧光信号,去除未反应的染料将减少背景荧光。- 将 HPLC 与标准分析 C18 列、0.1 M 三乙醇醋酸 (TEAA) 作为缓冲器 A 和乙酰酸盐作为缓冲器 B 进行准备。
- 将 1 M TEAA 添加到样品中(从步骤 1.9)中,制作 0.1 M TEAA。
- 将梯度程序设置为如下:0-5 分钟(带 0% B;使用 0-30% B 的 5-35 分钟) 35-37 分钟,B 的线性梯度为 30-100%,B 的线性梯度为 37-40 分钟,保持 0.1 mL/min 的流速,将色谱记录在 260 和 649 nm(用于 Cy5 标记样品)或 260 和 559 nm(对于 Cy3B 标记样品)。
- 当DNA和荧光通道的吸收度增加时,收集洗脱样品。
- 使用真空浓缩器浓缩洗样,将颗粒重新悬浮在 50-100 μL TE 缓冲液中。
- 使用UV-Vis光谱仪检查DNA和荧光团的浓度(见步骤1.10)。如有必要,稀释,使最终浓度在4-5μM左右。将探头储存在-20°C
2. 准备解决方案
- 准备大量的DEPC处理的水和缓冲液(表1)。这些解决方案在室温下可以持续一年以上。
- 准备4x固定溶液和洗涤液(表1)。
- 准备预杂交解决方案和探针杂交解决方案(表1)。在步骤 5.1 或步骤 6.1 中孵育期间准备探针杂交溶液,然后在 37 °C 台面摇床中使用盖,将溶液保持 20-40 分钟,以尽量减少暴露在光线下。
注:对乳酸探针组进行了优化, 以最大限度地减少 背景荧光,同时最大化真实信号。有关 如何修改 不同应用的这些浓度的详细信息,请参阅"讨论"部分。
3. 准备盖玻片和玻璃滑梯
- 清洁盖玻片和玻璃滑梯。
- 使用钳子将单独的盖玻片和幻灯片放在科普林罐中。确保盖玻片和幻灯片是分开的,不会相互接触。
- 用100%乙醇填充罐子,然后合上盖子。将罐子放在水浴超声波清洁剂中,并发出15-20分钟的声波。
注:对于水浴声波机,建议关闭加热器功能。 - 倒出乙醇,用超纯水洗3-4倍。使用直接从净水机流出的水。
- 从罐子里倒出水,用70%的乙醇加满。合上盖子,进行15-20分钟的声波处理,用超纯水清洗。
- 用超纯水和声波填充罐子15-20分钟。
注:盖玻片和玻璃滑梯可以放在装满超纯水的科普林罐中过夜。 - 使用干净的钳子从 Coplin 罐中拿出一张幻灯片或盖玻片,然后使用 N2 气体吹干 。对其余幻灯片和盖玻片重复此步骤。
- 将干燥的幻灯片放在干净的储物盒中,直到步骤 7.5 中使用。将干燥的盖玻片放在空的 1,000-μL 移液器尖端盒中,在其余过程中用作"室"。
- 使用疏水标记,在移液器尖端盒的圆形孔后在盖玻片上绘制圆圈。这些圆(直径为±0.5厘米)将作为"井"。等待至少 5-10 分钟,使标记完全干燥。
注:始终关闭尖端盒的盖子。 - 将 20-μL 滴滴 0.1% 聚 L-lysine 涂抹到每一个井中。在室温下孵育10-50分钟。
注:根据井大小调整此体积。确保溶液完全覆盖井区。对于更长的孵育时间,请注意避免蒸发。 - 孵化后吸气多-L-lysine,无需接触表面,因为这将刮掉聚-L-莱辛。然后将一滴DEPC水涂抹到聚L-Lysine处理的井中。关闭"室"的盖子,防止蒸发,直到步骤 5.1。
4. 时间过程实验和样品固定
- 在 ±20 mL 液体培养中,在 250 mL 烧瓶中培养大肠杆菌细胞。将烧瓶留在水浴摇床 (30 °C) 中,继续摇动。仅在取样时停止摇床。
注:本文提供的结果来自MG1655细胞生长在M9最小介质中,辅以0.2%甘油,0.1%的卡萨米诺酸和1毫克/L硫胺到指数生长阶段(OD600±0.2)。 - 在空的 1.5 mL 管中加入 250 μL 的 4x 固定溶液。重复并准备多个管子,与要拍摄的时间点一样多。用时间点编号标记管子,并在室温下保持。
- 在开始时间过程实验之前,服用 750 μL 的细胞培养(OD600±0.2)。将培养添加到标记为"时间零"的管中(从步骤 4.2 开始)。轻轻反转管子,将细胞与固定溶液混合。
注:不要上下移液,以混合、涡流或"粗糙"的细胞。此示例表示压抑状态,并将用作计算单个 mRNA 的荧光强度的控件(参见步骤 9.4)。 - 在液体培养中加入0.02-1 mM的异丙基β-D-1-thiogalactopyranoside(IPTG),以诱导 乳酸 表达。在此点启动计时器(t = 0 分钟),并在特定时间间隔(例如,每 1 分钟)开始采样。对于采样,重复步骤 4.3。
- 在时间过程实验(例如,在 t = 1.5 分钟)的某一时间添加 5 mM 正畸-β-D-fucopyranoside (ONPF) 或 500 mM 葡萄糖24, 以抑制 lacZ 表达。重新抑制后,继续采样培养物(步骤4.3),以跟踪mRNA降解。
注:抑制也可以用+400μg/mL利福平,转录启动抑制剂25。 - 对于固定,在室温下孵育含有采样细胞的管子15分钟,随后在冰中孵育30分钟。
- 要去除固定剂,在室温下以 4,500 x g 离心管 4 分钟。用移液器拆下上流液。
注:请务必按照安全协议将甲醛丢弃在单独的废物容器中。 - 添加 1 mL DEPC-PBS 并重新挂起细胞。重复离心和重新悬浮2次以上。
注:固定细胞易碎,需要温和治疗。小心地重新悬浮颗粒并避免气泡。 - 完成最后洗涤步骤后,在 ±30 μL DEPC-PBS 中重新悬浮细胞。
5. 细胞膜的渗透
- 将每个时间点样品应用于盖玻片上的不同孔(每孔±30 μL)。在室温下等待 10-30 分钟,使细胞粘附在表面上。避免在井间合并液体滴。
- 要冲洗掉未绑定的细胞,吸气液体,并在每口井上涂抹 ±20 μL DEPC PBS。在几分钟内吸气 DEPC PBS。
- 通过将15μL的70%乙醇涂到每孔4分钟,使细胞膜渗透。4分钟后吸气乙醇,并确保油井完全干燥。
注:将乙醇处理限制在4分钟至关重要。较长的治疗将导致过度渗透。 - 将 30 μL 的洗涤液涂抹到每个井上。
6. 探头杂交
- 从每一个井中吸出洗涤液。将 30 μL 的预杂交溶液应用于每一个井。在37°C烤箱中孵育腔室30分钟。
注:在腔室底部加入±50 mL水以提供湿度。 - 从每一个井中吸出预杂交解决方案。将 ±30 μL 的探针杂交溶液涂抹在每一个井上。用铝箔盖住房间,在 37 °C 烤箱中孵育 2 小时。
注:在此步骤之前,请确保探头杂交溶液位于 37°C 台面摇床中。避免液体在井之间合并。如果需要,将较小的溶液量应用于每个井。
7. 杂交后清洗和成像准备
- 使用多通道移液器,一次将 ±30 μL 的洗涤溶液涂抹到每一个井上。吸气并重复3-5次洗涤。在37°C烤箱中孵育腔室15-30分钟。
- 再重复步骤 7.1 两次。
- 用 DEPC-PBS 清洗每个井 5 次。遵循步骤 7.1 中使用的方法,但跳过孵化过程。
- 从盖玻片中吸出液体。将 4 μL 的 DEPC-PBS 涂抹在每一个井上。
- 使用钳子,提起并翻转盖玻片,然后轻轻地将其放在玻璃滑梯上(从步骤 3.2 开始)。避免气泡。
- 用硅胶牙龈密封盖玻片的边缘。
- 等待,直到口香糖凝固。你可以在这里停下来,在4°C下将幻灯片存放过夜。
注:其他smFISH协议建议添加氧气清除试剂(如葡萄糖氧化酶/乳酸酶)或使用商业抗褪色安装介质14,26,26以增加氟磷的光14稳定性。
8. 成像
- 要查找感兴趣的区域,请使用相位对比度成像的实时模式。通过操纵舞台操纵手柄,改变井内的视野。选择一个单元格密度最佳的区域(即,有许多细胞大部分是分离的)。调整 z 焦点,使相位对比度单元格图像处于焦点。
- 按 Cy5(4-s 曝光)、Cy3(2-s 曝光)和相位对比度(0.2 s 曝光)的顺序拍摄快照。
注:Cy3B染料分子在Cy3通道中成像,图像称为Cy3图像。 - 重复步骤 8.1-8.2 以获取井内 ±10 个不同区域的图像。
- 将目标移动到另一个井并重复步骤 8.1-8.3。
- 将图像导出为 TIFF 文件。
- (可选)在 Cy5 和 Cy3 通道的盖玻片表面吸附多色珠,以确定 Cy5 和 Cy3 通道之间的空间变化,以便进行图像注册。
- 在干净的盖玻片表面涂抹 ±10 μL 的多色荧光珠(直径 0.2 μm),等待 10-30 分钟。使用 ±50 μL PBS 洗涤后,涂抹 ±5 μL 的 PBS,用玻璃滑梯夹上盖玻片。密封并安装在显微镜上。
- Cy5 和 Cy3 通道中的图像珠子。
9. 图像分析
注意:此步骤中使用的 Matlab 代码可在以下 GitHub 网站中提供:https://github.com/sjkimlab/Code_Publication/tree/master/JoVE_2020。GitHub 文件夹包含图像分析所需的所有内容,包括用于单元格分段和点识别的参数值。此步骤中的过程在主脚本(称为"FISHworkflow.m")中进一步解释。
- 打开细胞分割工具,如微生物跟踪器27或乌夫蒂28,并加载相位对比度图像。选择"独立帧"并按下名为"所有帧"的按钮开始分段过程,从中识别单元格并计算其轮廓(图 3B,C)。
注:使用这些软件包的详细协议可在线获得(例如,oufti.org)。 - 在微生物跟踪器或 Oufti 的点查找功能中加载 Cy5 荧光图像,然后按"运行"按钮,开始基于 2D 高斯形接头进行点识别和定量(图 3B,C)。对 Cy3 荧光图像重复此步骤以分析 Cy3 通道中的点。此步骤生成每个单元格中的点列表,包括其强度和坐标。
- (可选)使用阈值筛选出暗点(误报),如 FISHworkflow.m 文件中所述。
注:检查负对照中的荧光斑(例如 MG1655 μlacZ),并确定过滤假阳性的阈值。 - 要获得单个 mRNA 的点强度,请使用时间零(在添加 IPTG 之前)测量的点强度列表,并使用高斯混合物模型与两个混合成分进行点强度的分布。以第一个高斯总体的峰值位置(图 3D中的黑线,E)作为单个mRNA的点强度。对 Cy5 点和 Cy3 点分别执行此操作,以获得单个 5' 和 3' lacZ mRNA 的点 强度。
注:在每个时道实验中重复,因为单个 mRNA 的点强度在不同的实验中可能略有不同。 - 将点的荧光强度与单个 mRNA 的强度(从步骤 9.4)进行划分,以获得点内的 mRNA 数。求和细胞内正态点强度,计算细胞中mRNA的总数(图3F)。分别对 5' 和 3' mRNA 执行这些计算。
- 计算和绘制每个细胞在每个时间点(例如图4B)的均值mRNA数,并分析从平均mRNA水平的时间变化中转录和mRNA降解的体内动力学(图4B)。
- 为了获得转录伸长率,请对一条线进行最小时拟合,以初始上升为 5' 和 3' mRNA 信号,并识别基底水平的截距(图 4B)。这些截距之间的差值表示RNAP从5'探针区域到3'探针区域的平均时间。在此时间将两个探针集(2 kb)之间的距离分开,以获得平均转录伸长率。
- 为了获得 mRNA 降解率,请将指数衰减函数 y = A=exp(-t/ω)拟合到 5' 和 3' mRNA 信号的最终衰变区(例如图 4B)。拟合参数 α 是平均 mRNA 寿命。
- (可选)根据每个细胞中mRNA数的分布(在步骤 9.5中计算),分析基因表达中的细胞对细胞变异(例如,图4C所示的诱导细胞级响应)。
- (可选)使用有关沿单元格主轴和次要轴的点位置的信息(从步骤 9.2 获取),分析 mRNA 的定位(图 4D,E)。
- (可选)通过比较Cy5和Cy3通道中检测到的点的定位,分析5'和3'mRNA的共同定位(图5)。Figure 5
- 在微生物跟踪器的位点查找器功能中加载多色珠(步骤8.6)的图像,并在Cy5和Cy3通道中获取珠质的坐标。使用质心坐标列表计算仿射变换矩阵,该矩阵通知 Cy5 和 Cy3 通道如何相互移动和旋转29。
- 将仿射变换矩阵应用于 Cy5 和 Cy3 FISH 图像,以在 Cy5 坐标中转换 Cy3 图像。如果点与不同通道中的另一个点共同本地化,进行分类。例如,如果 Cy5 通道中的点与 Cy3 通道中的另一个点(如果其质心之间的距离小于 150 nm)(图 5)
- 分析每个时间点有多少 Cy5 点被归类为与 Cy3 点"共同本地化"。此外,分析共位点的强度(图5)。
结果
图 3显示了此 smFISH 协议中的代表性图像。使用材料表中详细说明的显微镜设置,一个完整视场(86.7 μm x 66.0 μm)显示了分散在该领域(图3A)的500个大肠杆菌细胞。如果细胞的密度比此图像中显示的高得多,则自动细胞分割变得困难,因为当细胞相互接触时,分割算法无法可靠地识别单个细胞。需要调整细胞的浓度和用于表面粘附的孵育时间(步骤5.1),以实现视场中细胞的最佳密度。
相对比图像中细胞的形态应与活细胞的形态保持可比性(图3A-C)。C如果细胞过度渗透,细胞形态会发生变化(如"幽灵";补充图1。在这种情况下,在步骤 5.3 中,可以缩短 70% 乙醇处理的持续时间。
在诱导之前,每个细胞的平均lacZ表达水平为±0.03mRNA,与之前的报告15、30,一致。此外,由于存在强度高的点(图3D,E),感应前lacZ mRNA点强度的分布与正态分布或泊松分布不配合。这表明,在抑制状态下检测到的大多数斑点代表单个lacZ mRNA,但少量斑点包含多个lacZ mRNA。为了用单个lacZ mRNA 分离种群,我们使用高斯混合物模型与两个混合成分(图3D中的内组,E)。然后,将第一个高斯点的均值视为单个 mRNA 点的均值强度(例如,图 3D中黑色曲线的峰值),用于将点强度转换为 mRNA 的数量,用于在时间过程实验中检测到的任何点。为了计算一个单元中的mRNA总数,每个细胞(图3F)中总结了归化的点强度(图3F)19。
当 lacZ mRNA 的表达水平较低时,在细胞内有一个或两个衍射有限 lacZ mRNA 点在空间上分离。因此,这些斑点的图像可以通过2D高斯拟合来分析其强度和定位。
当表达水平高,使斑点在细胞内相互重叠时,2D 高斯拟合不能进行可靠的定量。在这种情况下,应通过将细胞内的背景减去的荧光信号与单个 mRNA19的均度除以 来计算 mRNA 水平。
当诱导乳酸表达时,5'lacZ mRNAlacZ的信号首先增加,3'lacZ mRNAlacZ的信号随后增加(图4B)。如果lacZ 的表达被抑制,则 5' 和 3' lacZ mRNA 信号之间会减少一些延迟(图 4B)。为了获得转录伸长率,5' 和3' 信号的上升首先与线(图4B)相拟,并且将x-intercept的差异作为RNAP在两个探针区域之间行驶的距离(2,000 nt)的时间。从每个时间过程实验中可以测量转录伸长率,也可以根据实验重复计算标准偏差。在我们的实验条件下,转录伸长的平均速率为15-30 nt/s。19
此外,通过拟合衰变区与指数函数(图4B),获得了mRNA降解率(与平均mRNA寿命的相反)。我们的时间过程数据包含转录31期间和之后mRNA降解。我们适合3'mRNA开始衰变后的时间点(t > 6分钟),以探测释放的mRNA的降解。我们获得了+90s作为平均寿命5'或 3'lacZ mRNA 19。
转录启动速率可以从感应后5'信号增加的斜率(图4B,蓝色)或稳定状态下的平均mRNA数(即起始速率除以降解率)计算。此外,通过采取3'信号增加的斜率与5'信号增加32 的斜率,或3'和5'mRNA区域19的稳态水平之间的比例,可以估计提前转录终止的概率。
由于 smFISH 是一种单细胞技术,我们可以分析转录中的细胞对细胞变异性。例如,可以分析添加 IPTG后表达lacZ mRNA的细胞百分比(图4C)。还可以解决诱导后 mRNA 定位是否发生变化。我们观察到5'和 3'lacZ mRNA斑点稍微向外移动,远离细胞中心(图4D,E),与上一份报告33一致。
最后,对5'和3'mRNA点之间的共定位分析可以提供信息(图5A)。例如,在抑制状态(时间为零)中,大约 25% 的 5' mRNA 点与 3' mRNA 点并位。在 t = 1 分钟时,由于许多基因位点具有 5' mRNA 合成,但尚未进行 3' mRNA 合成,因此大多数 5' mRNA 点本身没有 3' mRNA 信号(即共定位概率低)。但是,当 3' mRNA 出现时(即 t = 2 分钟),共同定位的概率会增加(图5A 中的紫色箭头BB )。这个时间点,当共同定位变得频繁,取决于转录伸长率。在此时间点检测到的每个共定位点中 5' 和 3' lacZ mRNA 数的二维密度图可用于推断 lacZ 基因上的RNAP密度(图5C)。如先前报告19,此图中的5'mRNA数字表明,当1 mM ITG诱导lacZ表达时,大多数乳酸位点在DNA上只有不到10个RNAP。此外,此图中的 3' mRNA 数与 RNAP34的聚类有关。3' mRNA 的数量接近 1,这意味着大约只有一个 RNAP 进入 3' 探针区域。这表明,lacZ基因上的RNAP在空间上是分离的,而不是形成一个聚类(或"三聚")。
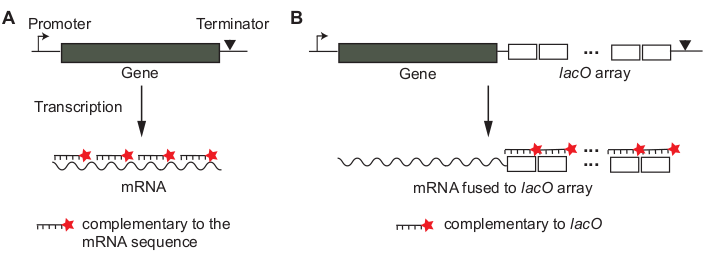
图1:为感兴趣的mRNA设计smFISH探头。 (A) 平铺方法。选择短DNA寡核苷酸序列(长度为±20 bp),以便它们能覆盖感兴趣的mRNA。寡核苷酸探针标有荧光染料分子。(B) 数组方法。串联序列的非编码数组(例如,"lacO 数组")在转录中与感兴趣的 mRNA 融合。荧光标记探针与重复单元的补充(例如,长度为 17 bp 的 lacO 探头)用于放大 mRNA 的信号。 请单击此处查看此图的较大版本。
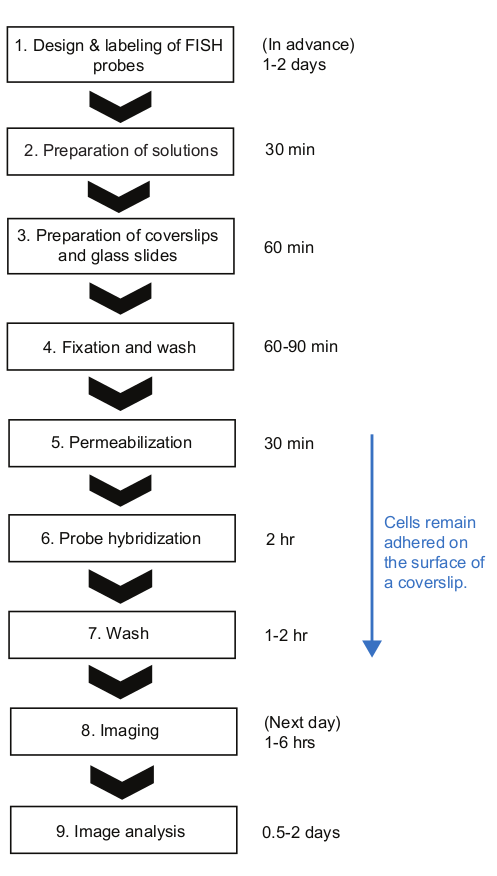
图2:smFISH实验程序的示意图和每一步的时间持续时间。请单击此处查看此图的较大版本。

图3:smFISH图像分析。(A-C) 在 M9 最小介质中生长的野生型大肠杆菌 (MG1655) 中 5' E. coli lacZ mRNA (红色) 和 3' lacZ mRNA (绿色) 的 smFISH 显微镜图像, 辅以 0.2% 甘油, 0.1% 卡萨米诺酸,30 °C时 1 毫克/L 硫胺 ( A ) 30 °C. ( A ) 30 °C . (A) 30 °C 的样品代表图像,感应后 3 分钟,0.05 mM IPTG 在 t = 0 分钟,在 t = 1.5 分钟时抑制 500 mM 葡萄糖。Cy5(5'lacZ mRNA,红色)和Cy3(3'lacZ mRNA,绿色)的相位lacZ对比度和两个荧光图像被伪着色覆盖。 lacZ图像显示整个字段 86.7 μm x 66.0 μm。比例杆,5 μm。(B) 放大缩小字体功能 放大缩小字体功能单元格轮廓以白色显示,从图像分析中识别的荧光点用红点显示。比例杆,1 μm。(C) 在高表达条件下检测细胞轮廓和荧光斑(t = 1 mM IPTG 感应后 4 分钟)。比例杆,1 μm。(D-E)在添加IPTG(抑制状态)之前测量的5'和3'mRNA点强度的分布。直方图显示两个高斯函数(黑色和灰色),其平均值来自高斯混合模型。插入显示从高斯混合物模型生成的随机数的量程-量程图,并进行了实验性测量的 mRNA 点强度(n = 1040 表示 5' mRNA,680 表示 3' mRNA)。(F) 面板(B)中指向单个单元格获得的信息。对于给定的细胞(i),在Cy5和Cy3通道中识别斑点,并且它们的强度(I)和沿细胞的短轴和长轴(d, l)的坐标从2D高斯拟合中量化。在归一化点强度被求和后,产生此细胞中 5' 或 3' mRNA 的总数。此外,可以分析不同通道的点之间的共同定位,如图5 所示。请单击此处查看此图的较大版本。

图4:体内转录和mRNA降解动力学分析。(A) 双色 smFISH 实验的示意图和代表性图像,测量lacZ mRNA 水平随时间的变化。红色和绿色虚线表示Cy5或Cy3B标记的寡核苷酸探针,分别杂交到大肠杆菌中乳Z mRNA的1 kb长5'和3'mRNA区域lacZ。 E. coli还显示两个荧光图像的叠加,在感应后在指示的时间点与相位对比度图像覆盖,在 t = 0 分钟时为 0.2 mM IPTG。转录在 t = 1.5 分钟时用 500 mM 葡萄糖抑制。比例杆,1 μm。这个数字已经修改了从金等人19。(B) 在面板 (A) 中描述的实验中,每个细胞的 5' 和 3' lacZ mRNA 数字会随着时间的推移而增加。错误栏是引导的 SEM。每个时间点至少分析1,200个细胞。5' 和 3' mRNA 信号的初始上升与线(蓝色)拟合。x-截距的差异为1.93分钟,平均转录伸长率为17.3 nt/s。5' 和 3' mRNA 信号的最终衰减与指数衰减函数(灰色)配合。拟合参数表示,5' mRNA 的平均 mRNA 寿命为 1.52 分钟,3' mRNA 的平均 mRNA 寿命为 1.66 分钟。(C) 在(A)中描述的实验中,具有一个或多个乳酸mRNA斑点的细胞的百分比。错误栏是引导的 SEM。(D) 沿着细胞的短轴定位点。可以通过沿短轴(d)与细胞的半宽度(w)分割位置来量化一个点与膜的接近。(E) 在(A)所述实验中,5' 和3' lacZ mRNA点的定位沿细胞短轴的变化。请单击此处查看此图的较大版本。
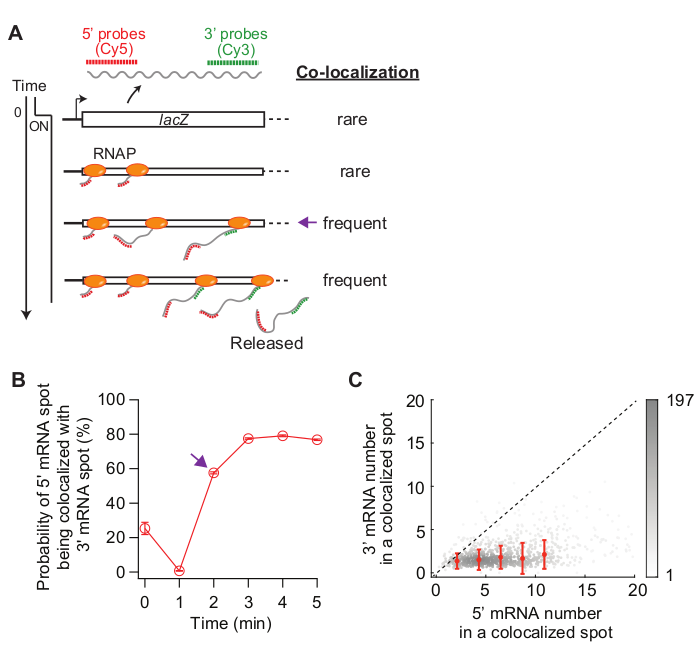
图5:5'和3'mRNA点的共同定位分析。 (A) 导电后显示 5' 和 3' mRNA 点的预期共定位的示意图。当制造 3' mRNA 时,5' mRNA 点与 3' mRNA 点共同局部的概率增加(紫色箭头)。(B) 与1 mM IPTG 感应后共同定位的概率。紫色箭头表示根据面板 (A) 中的示意图,首先共同定位的概率变得频繁的时间点。(C) 在 1 mM IPTG(共 841 个点)感应后 t = 2 分钟内检测到的共定位点内 5' 和 3' lacZ mRNA 的数量。灰点表示单个共本地化点,而红点表示装箱数据的平均值。错误栏是 SEM。灰色阴影表示图形给定区域中的点的密度。虚线表示坡度为 1。 请单击此处查看此图的较大版本。
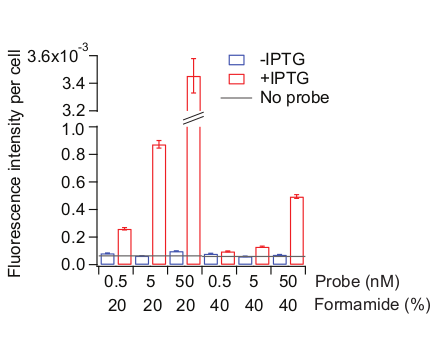
图6:探头杂交条件的优化。 使用了两种样品:MG1655细胞,如图 3所述生长,保持 未引能(蓝色)或用0.5 mM IPTG处理20分钟(红色)。探头杂交溶液采用不同浓度的探针(共72个Cy5结合探头平铺整个 乳酸区 )和甲酰胺。杂交前溶液和洗涤溶液中也相应地调整了甲酰胺浓度。"无探针"(灰色线)表示在杂交步骤中处理无探针的 IPTG 添加细胞的荧光水平。通过300-800个细胞计算,按细胞面积(AU)对平均荧光强度进行规范化。错误栏是引导的 SEM。 请单击此处查看此图的较大版本。
补充图1: 过度渗透导致的扭曲细胞形态。通过 1 mM IPTG 感应 5 分钟后,MG1655 细胞的相位对比度(灰度)、5' lacZ mRNA(Cy5,红色)和 3' lacZ mRNA(Cy3,绿色)图像的叠加。(A) 一个示例,显示正常细胞和过度渗透细胞的混合物,缺乏正常形态(用粉红色箭头表示)。(B) 一个显示"幽灵"细胞聚集在一起的例子。缩放栏 = 1 μm. 请单击此处下载此文件。
| 德普水 | ||||
| 将0.1%的DEPC加入超纯水,在37°C烤箱中孵育瓶子(覆盖),并在第二天进行高压高压。 | ||||
| DEPC PBS (10 倍) | ||||
| 混合以下内容: | ||||
| 80 克 | NaCl (最终 1.37 M) | |||
| 2 克 | KCl (最终 27 mM) | |||
| 14.2 克 | Na2HPO4 (最终 100 mM) | |||
| 2.7 克 | KH2PO4 (最终 20 mM) | |||
| 超纯水至1L | ||||
| 过滤(0.22 μm)放入玻璃瓶中。 | ||||
| 加入 0.1% DEPC,并按照 DEPC 水的指示进行操作。 | ||||
| 要制造 1X 溶液,使用 DEPC 水稀释 10 次。 | ||||
| 1M DEPC 磷酸钠缓冲液,pH 7.4 | ||||
| 混合以下内容: | ||||
| 115 克 | Na2HPO4 | |||
| 22.8 克 | Nah2PO4 | |||
| 超纯水至 1 升 | ||||
| 过滤(0.22 μm)放入玻璃瓶中。 | ||||
| 加入 0.1% DEPC,并按照 DEPC 水的指示进行操作。 | ||||
| 4X 固定溶液(16% 甲醛) | ||||
| 5 mL | 20% 甲醛 | |||
| 500 μL | 德普水 | |||
| 750 μL | 1M DEPC 磷酸钠缓冲液,pH 7.4 | |||
| 在4°C下储存长达2-4周。 | ||||
| 注意:甲醛是有毒的。制作此解决方案时,请戴上手套并使用防烟罩。 | ||||
| 洗涤溶液 | ||||
| 混合以下内容: | ||||
| 10 mL | 甲酰胺(最终 25%) | |||
| 4 mL | 20 倍 SSC(最终 2 倍) | |||
| 将 DEPC 水填充至 40 mL | ||||
| 过滤器(0.22 μm),储存在4°C | ||||
| 注意:甲酰胺是有毒的。制作此解决方案时,请戴上手套并使用防烟罩。 | ||||
| 预杂交解决方案 | ||||
| 200 μL | 甲酰胺(最终 20%) | |||
| 100 μL | 20 倍 SSC(最终 2 倍) | |||
| 10 μL | 100X VRC (最终 1X) | |||
| 25 μL | 4% (w/v) BSA (最终 0.1%) | |||
| 685 μL | 德普水 | |||
| 注:在将 10 μL 从外拿出来之前,涡流 VRC 盘点。 | ||||
| 注意:甲酰胺是有毒的,是已知的畸子。戴上手套,在烟罩下处理。 | ||||
| 探头杂交解决方案 | ||||
| 200 μL | 甲酰胺(最终 20%) | |||
| 100 μL | 20 倍 SSC(最终 2 倍) | |||
| 10 μL | 100X VRC (最终 1X) | |||
| 25 μL | 4% (w/v) BSA (最终 0.1%) | |||
| 10 μL | 40毫克/mL大肠杆菌(最终0.4毫克/千升) | |||
| 200 μL | 50% 硫酸二酯(最终 10%) | |||
| x μL | 5' mRNA 探头组(从步骤 1.12)到最终 4 nM。 | |||
| y μL | 3' mRNA 探头组(从步骤 1.12)到最终 4 nM。 | |||
| - | DEPC 水使总体积 1 mL | |||
| 注:最后添加硫酸二酯。因为它非常粘稠,在从 50% 的库存中拿出 200 μL 之前,先切开移液器尖端的末端。添加硫酸葡萄糖后,上下移液器使溶液均质化。 | ||||
表1:使用的解决方案配方。
讨论
在这里,我们提出了一个smFISH协议,用于测量大肠杆菌中的 mRNA动力学。在先前发布的细菌23的smFISH协议中,细胞被保存在管子里直到协议结束,也就是说,直到它们准备好成像。虽然它有许多优点,如在盖玻片表面23上极少的荧光探针的非特异性结合,但当有许多来自时间过程实验的样本时,很难遵循这些协议。首先,在固定之前,需要对相对大量的细胞(>1 mL)进行采样,甚至收获。其次,细胞样品需要多次离心以交换溶液,并在杂交步骤后进行洗涤。在我们的协议中,少量培养(<1 mL)与1.5 mL管中的固定溶液直接混合,有助于在采样时快速"冻结"细胞状态。此外,在整个过程中,细胞一直附着在表面,通过真空过滤系统吸气液体,用多通道移液器同时应用溶液滴液,可以快速交换不同的溶液。当需要同时处理大量样品时,这种差异使我们的协议非常有利。使用我们的协议,12-48个样本可以同时处理,整个 FISH 过程可以在 +8 小时内完成,大约几个样本需要类似的时间(图 2)。虽然我们以大肠杆菌中的 lacZ 表达为例, 但该协议广泛适用于不同的基因和细菌物种,下面将讨论以下注意事项。
对于不同的基因,首先要考虑的是 smFISH 探针。人们可能设计寡核苷酸探针,将感兴趣的mRNA平铺(图1A)13。13在这种"平铺"探针方法中,每个探头的基座长为 ±20,并在 5' 或 3' 的称从中标有荧光。这种策略很方便,因为不需要基因操作。或者,与基因组序列不一(例如,在考洛菌新月14中)不一的串联重复序列(例如,在Nao细菌新月14中)的串联重复可以插入感兴趣的基因的未翻译区域,并且使用与重复单元互补的单一探针来标记mRNA("数组"方法;图1B.在这两种情况下,多个荧光团都装饰一个mRNA,发出放大的荧光信号,可以很容易地从细胞内非特异性结合的单探头中区分。
是否选择"平铺"或"阵列"方法取决于负对,该样本测试了探针的非特异性结合,因为它缺乏目标mRNA。对于平铺探针(图1A),一种没有感兴趣的基因或条件的突变菌株,其中基因不转录( 例如,压抑lacZ)可以作为检测探针非特异性结合的负对照。对于基于数组的 smFISH(图 1B),缺少阵列的野生类型应变可以用作负控件,因为它不包含探测器的绑定位点。
最佳杂交条件可能取决于探针序列,甚至取决于氟磷染料的选择。我们优化了lacZ 探头集的杂交条件,将杂交温度保持在 37°C,并在杂交溶液中测试不同浓度的探针组和甲酰胺。甲酰胺浓度越高,非特异性和特异性结合物的浓度也容易降低26,35。,35我们建议系统地改变杂交及其洗涤条件,同时保持杂交时间和温度相同。随着条件变得更加严格,非特异性和特定绑定都减少了(图6)。找到非特定绑定开始达到可接受的阈值而不进一步损害特定绑定的点非常重要。例如,我们使用在没有任何探头("无探头")的情况下获得的信号电平作为阈值(图 6)。
双色 smFISH 方法标记 mRNA 的两个独立区域仅限于长基因。为了测量转录伸长率,我们利用了 lacZ 长(3075bp)的事实,其表达可以通过IPTG诱导。当基因短时,很难设计两个平铺探针集(接近 5' 和 3' 端),并解决 5' 与 3' mRNA 区域出现之间的时间延迟。在这种情况下,人们通过 smFISH 可以计算处于稳定状态的新生 mRNA,并分析其分布,分析其分布,该分析模型具有转录伸长率作为拟合参数20。此外,当感兴趣的基因不可被感染时,可以在零时间用利福平治疗细胞,并测量5'和3'mRNA亚区的时间变化。然后,从5'mRNA信号的减少到3'mRNA信号的延迟,就可以像之前31号那样计算转录伸长率。
最后,smFISH 协议是通用的,可以与其他标签方案结合使用。以前,DNA位点与mRNA一起可视化,将mRNA FISH与DNA FISH14或荧光记者-操作系统20相结合。蛋白质产品可以通过执行免疫荧光与mRNA FISH14,36一起可视化。此外,它可以与三维超分辨率显微镜37相结合,在所有三维38,39中可视化mRNA。
披露声明
提交人宣称他们没有相互竞争的经济利益。
致谢
该协议是由S.K.在霍华德·休斯医学研究所和耶鲁大学微生物科学研究所的克里斯汀·雅各布斯-瓦格纳博士实验室进行博士后研究时开发的。我们感谢雅各布斯-瓦格纳博士和她的实验室成员在方法开发过程中提供的各种投入,以及劳拉·特罗耶对手稿的批判性阅读。S.K. 感谢西尔学者计划的支持;K.V.感谢伊利诺伊大学詹姆斯学者预布尔研究奖的支持。
材料
| Name | Company | Catalog Number | Comments |
| Bacterial strain | |||
| Escherichia coli MG1655 | |||
| Chemicals, peptides, and others | |||
| Acetonitrile | Sigma-Aldrich | 34851 | UPLC buffer B |
| Ammonium chloride | Fisher Chemical | A661-500 | To make M9 medium |
| Bovine serum albumin (BSA) | Sigma-Aldrich | B2518 | Probe hybridization |
| Calcium chloride | Acros Organics | 349610250 | To make M9 medium |
| Casamino acid | BD Difco | 223050 | To make M9 medium |
| Cy3B NHS ester | GE Healthcare Life Sciences | PA63101 | Fluorophore for FISH probes |
| Cy5 NHS ester | GE Healthcare Life Sciences | PA15101 | Fluorophore for FISH probes |
| DEPC | Sigma-Aldrich | D5758 | |
| Dextran sulfate | Millipore | S4030 | Probe hybridization |
| Dextrose | Fisher Chemical | D16 | To repress the expression of lacZ |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | To dissolve fluorophores |
| E. coli tRNA | Sigma-Aldrich | R1753 | Probe hybridization |
| Ethanol | Decon Laboratories | 2701 | Used in DNA purification, lysis, and cleaning coverslips |
| FISH probes | Biosearch Technologies | Sequences are published in ref#16 | |
| Formaldehyde | Ladd Research Industries | 20295 | Fixation |
| Formamide | American Bio | AB00600 | Probe hybridization, pre-hybridization, and wash |
| Glycerol | Americanbio | AB00751-01000 | To make M9 medium |
| Isopropylthio-β-galactoside (IPTG) | Invitrogen | 15529019 | lacZ induction |
| Magnesium sulfate | Fisher Chemical | M65-500 | To make M9 medium |
| 2-Nitrophenyl β-D-fucopyranoside (ONPF) | Santa Cruz Biotechnology | sc-216258 | lacZ repression |
| Picodent twinsil 22 | Picodent | 1300 1000 | Sealant |
| Poly-L-lysine | Sigma-Aldrich | P8920 | To treat the coverslip surface |
| Potassium chloride | Fisher BioReagents | BP366-500 | To make PBS |
| Potassium phosphate monobasic | Fisher BioReagents | BP362-500 | To make PBS and M9 medium |
| Rifampicin | Sigma-Aldrich | R3501 | To stop transcription initiation |
| Saline-sodium citrate buffer (SSC) | Invitrogen | AM9763 | Probe hybridization, pre-hybridization, and wash |
| sodium bicarbonate | Fisher BioReagents | BP328-500 | Fluorophore-probe conjugation |
| Sodium chloride | Fisher BioReagents | BP358-1 | For DNA purification, PBS and M9 medium |
| Sodium phosphate dibasic | Fisher BioReagents | BP332-500 | To make PBS and M9 medium |
| Sodium phosphate monobasic | Fisher BioReagents | BP329-500 | To make a sodium phosphate buffer |
| Super PAP Pen | Invitrogen | 8899 | Hydrophobic marker for coverslips |
| TetraSpek microspheres | Invitrogen | T7280 | Controls for multi-channel registration |
| Thiamine | Sigma-Aldrich | T1270 | To make M9 medium |
| Triethylammonium acetate | Sigma-Aldrich | 90358 | UPLC buffer A |
| Vanadyl ribonucleoside complex (VRC) | Sigma-Aldrich | 94742 | Probe hybridization and pre-hybridization |
| Equipment | |||
| C18 column | Waters | Acquity BEH C18 column | |
| Countertop centrifuge | Eppendorf | 5425 | |
| Countertop incubator | Eppendorf | Thermomixer F1.5 | |
| Incubator (Oven) | Thermo Scientific | 51030514 | Gravity convection |
| Water purification system | Millipore | Milli-Q Reference | |
| Nanodrop | Thermo Scientific | 2000C | |
| Nitrogen gas | Building | For blow-drying coverslips and glass slides | |
| UPLC | Waters | Acquity UPLC system | |
| Vacuum and aspirator | Building | Aspirator is made of a filtration flask with a side arm. | |
| Vacuum concentrator | Labconco | 7810010 | Centrivap; to dry samples collected from UPLC. |
| Vortexer | Scientific Industries | Genie-2 SI-0236 | |
| Water bath shaker | New Brunswick | Innova 3100 | Critical for time-course experiments |
| Water bath sonicator | VWR | 97043-960 | To clean coverslips and glass slides |
| Tools | |||
| 1.5-mL tubes | Eppendorf | 22431021 | DNA lobind tubes |
| 1000-uL pipette tip box | Denville Scientific | P1126 | An empty box after using all the tips |
| Coplin jar | SPI | 01240-AB | To clean coverslips and glass slides |
| Coverslip | Fisher Scientific | 22-050-230 | 24x60 No1 |
| Filtered pipette tips | Denville Scientific | P1121,P1122,P1126 | SHARP® Precision Barrier Tips |
| Forceps | SPI | K35a | To handle clean coverslips and glass slides |
| Glass slide | Fisher Scientific | 12-544-1 | |
| Gloves | Microflex | MK-296-M | |
| Multichannel pipetter | Eppendorf | 2231300045 | To use in the washing step (#7) |
| Pipette | Gilson | P1000, P200, P20 | |
| Reagent reservoir | MTC Bio | P8025-1S | To use in the washing step (#7) |
| Syringe filter (0.22 um) | Millipore | SLGS033SS | |
| Timer | VWR | 62344-641 | |
| Software and algorithms | |||
| MATLAB | Mathworks | R2013 and up | https://www.mathworks.com |
| MicrobeTracker or Oufti | https://www.github.com/JacobsWagnerLab/MicrobeTracker | ||
| https://oufti.org/ | |||
| Stellaris Probe Designer | Biosearch Technologies | https://www.biosearchtech.com/support/tools/design-software/stellaris-probe-designer | |
| Microscope | |||
| CCD camera | Hamamatsu Photonics | Orca-II-ER | |
| Cy3 filter set | Chroma | 49004 | |
| Cy5 filter set | Chroma | 49006 | |
| Epi-fluorescence microscope | Nikon | Eclipse Ti | For phase-contrast and epi fluorescence |
| Fluorescence excitation source | Lumencor | SOLA-E | |
| Nikon Elements software | Nikon | software that controls the microscope setup | |
| Phase-contrast 100x objective | Nikon | Plan Apochromat (NA 1.45) | |
| Probe sequence | |||
| DNA oligos with C6 amino modification at the 5' end | Biosearch Technologies Inc | ||
| lacZ1 | GTGAATCCGTAATCATGGTC | 5' mRNA | |
| lacZ2 | TCACGACGTTGTAAAACGAC | 5' mRNA | |
| lacZ3 | ATTAAGTTGGGTAACGCCAG | 5' mRNA | |
| lacZ4 | TATTACGCCAGCTGGCGAAA | 5' mRNA | |
| lacZ5 | ATTCAGGCTGCGCAACTGTT | 5' mRNA | |
| lacZ6 | AAACCAGGCAAAGCGCCATT | 5' mRNA | |
| lacZ7 | AGTATCGGCCTCAGGAAGAT | 5' mRNA | |
| lacZ8 | AACCGTGCATCTGCCAGTTT | 5' mRNA | |
| lacZ9 | TAGGTCACGTTGGTGTAGAT | 5' mRNA | |
| lacZ10 | AATGTGAGCGAGTAACAACC | 5' mRNA | |
| lacZ11 | GTAGCCAGCTTTCATCAACA | 5' mRNA | |
| lacZ12 | AATAATTCGCGTCTGGCCTT | 5' mRNA | |
| lacZ13 | AGATGAAACGCCGAGTTAAC | 5' mRNA | |
| lacZ14 | AATTCAGACGGCAAACGACT | 5' mRNA | |
| lacZ15 | TTTCTCCGGCGCGTAAAAAT | 5' mRNA | |
| lacZ16 | ATCTTCCAGATAACTGCCGT | 5' mRNA | |
| lacZ17 | AACGAGACGTCACGGAAAAT | 5' mRNA | |
| lacZ18 | GCTGATTTGTGTAGTCGGTT | 5' mRNA | |
| lacZ19 | TTAAAGCGAGTGGCAACATG | 5' mRNA | |
| lacZ20 | AACTGTTACCCGTAGGTAGT | 5' mRNA | |
| lacZ21 | ATAATTTCACCGCCGAAAGG | 5' mRNA | |
| lacZ22 | TTTCGACGTTCAGACGTAGT | 5' mRNA | |
| lacZ23 | ATAGAGATTCGGGATTTCGG | 5' mRNA | |
| lacZ24 | TTCTGCTTCAATCAGCGTGC | 5' mRNA | |
| lacZ25 | ACCATTTTCAATCCGCACCT | ||
| lacZ26 | TTAACGCCTCGAATCAGCAA | ||
| lacZ27 | ATGCAGAGGATGATGCTCGT | ||
| lacZ28 | TCTGCTCATCCATGACCTGA | ||
| lacZ29 | TTCATCAGCAGGATATCCTG | ||
| lacZ30 | CACGGCGTTAAAGTTGTTCT | ||
| lacZ31 | TGGTTCGGATAATGCGAACA | ||
| lacZ32 | TTCATCCACCACATACAGGC | ||
| lacZ33 | TGCCGTGGGTTTCAATATTG | ||
| lacZ34 | ATCGGTCAGACGATTCATTG | ||
| lacZ35 | TGATCACACTCGGGTGATTA | ||
| lacZ36 | ATACAGCGCGTCGTGATTAG | ||
| lacZ37 | GATCGACAGATTTGATCCAG | ||
| lacZ38 | AAATAATATCGGTGGCCGTG | ||
| lacZ39 | TTTGATGGACCATTTCGGCA | ||
| lacZ40 | TATTCGCAAAGGATCAGCGG | ||
| lacZ41 | AAGACTGTTACCCATCGCGT | ||
| lacZ42 | TGCCAGTATTTAGCGAAACC | ||
| lacZ43 | AAACGGGGATACTGACGAAA | ||
| lacZ44 | TAATCAGCGACTGATCCACC | ||
| lacZ45 | GGGTTGCCGTTTTCATCATA | ||
| lacZ46 | TCGGCGTATCGCCAAAATCA | ||
| lacZ47 | TTCATACAGAACTGGCGATC | ||
| lacZ48 | TGGTGTTTTGCTTCCGTCAG | ||
| lacZ49 | ACGGAACTGGAAAAACTGCT | 3' mRNA | |
| lacZ50 | TATTCGCTGGTCACTTCGAT | 3' mRNA | |
| lacZ51 | GTTATCGCTATGACGGAACA | 3' mRNA | |
| lacZ52 | TTTACCTTGTGGAGCGACAT | 3' mRNA | |
| lacZ53 | GTTCAGGCAGTTCAATCAAC | 3' mRNA | |
| lacZ54 | TTGCACTACGCGTACTGTGA | 3' mRNA | |
| lacZ55 | AGCGTCACACTGAGGTTTTC | 3' mRNA | |
| lacZ56 | ATTTCGCTGGTGGTCAGATG | 3' mRNA | |
| lacZ57 | ACCCAGCTCGATGCAAAAAT | 3' mRNA | |
| lacZ58 | CGGTTAAATTGCCAACGCTT | 3' mRNA | |
| lacZ59 | CTGTGAAAGAAAGCCTGACT | 3' mRNA | |
| lacZ60 | GGCGTCAGCAGTTGTTTTTT | 3' mRNA | |
| lacZ61 | TACGCCAATGTCGTTATCCA | 3' mRNA | |
| lacZ62 | TAAGGTTTTCCCCTGATGCT | 3' mRNA | |
| lacZ63 | ATCAATCCGGTAGGTTTTCC | 3' mRNA | |
| lacZ64 | GTAATCGCCATTTGACCACT | 3' mRNA | |
| lacZ65 | AGTTTTCTTGCGGCCCTAAT | 3' mRNA | |
| lacZ66 | ATGTCTGACAATGGCAGATC | 3' mRNA | |
| lacZ67 | ATAATTCAATTCGCGCGTCC | 3' mRNA | |
| lacZ68 | TGATGTTGAACTGGAAGTCG | 3' mRNA | |
| lacZ69 | TCAGTTGCTGTTGACTGTAG | 3' mRNA | |
| lacZ70 | ATTCAGCCATGTGCCTTCTT | 3' mRNA | |
| lacZ71 | AATCCCCATATGGAAACCGT | 3' mRNA | |
| lacZ72 | AGACCAACTGGTAATGGTAG | 3' mRNA | |
参考文献
- Bervoets, I., Charlier, D. Diversity, versatility and complexity of bacterial gene regulation mechanisms: opportunities and drawbacks for applications in synthetic biology. FEMS Microbiology Reviews. 43 (3), 304-339 (2019).
- Epshtein, V., Nudler, E. Cooperation between RNA polymerase molecules in transcription elongation. Science. 300 (5620), 801-805 (2003).
- Vogel, U., Jensen, K. F. The RNA chain elongation rate in Escherichia coli depends on the growth rate. Journal of Bacteriology. 176 (10), 2807-2813 (1994).
- Tennyson, C. N., Klamut, H. J., Worton, R. G. The human dystrophin gene requires 16 hours to be transcribed and is cotranscriptionally spliced. Nature Genetics. 9, 184 (1995).
- Singh, J., Padgett, R. A. Rates of in situ transcription and splicing in large human genes. Nature Structural & Molecular Biology. 16, 1128 (2009).
- Selinger, D. W., Saxena, R. M., Cheung, K. J., Church, G. M., Rosenow, C. Global RNA Half-Life Analysis in Escherichia coli Reveals Positional Patterns of Transcript Degradation. Genome Research. 13 (2), 216-223 (2003).
- Bernstein, J. A., Khodursky, A. B., Lin, P. -. H., Lin-Chao, S., Cohen, S. N. Global analysis of mRNA decay and abundance in Escherichia coli at single-gene resolution using two-color fluorescent DNA microarrays. Proceedings of the National Academy of Sciences. 99 (15), 9697-9702 (2002).
- Pérez-Ortín, J. E., Medina, D. A., Chávez, S., Moreno, J. What do you mean by transcription rate. BioEssays. 35 (12), 1056-1062 (2013).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6 (5), 377-382 (2009).
- Kuchina, A., et al. Microbial single-cell RNA sequencing by split-pool barcoding. BioRxiv. , 869248 (2019).
- Blattman, S. B., Jiang, W., Oikonomou, P., Tavazoie, S. Prokaryotic single-cell RNA sequencing by in situ combinatorial indexing. Nature Microbiology. , (2020).
- Femino, A., Fay, F., Fogarty, K., Singer, R. Visualization of single RNA transcripts in situ. Science. 280 (5363), 585-590 (1998).
- Raj, A., vanden Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877-879 (2008).
- Montero Llopis, P., et al. Spatial organization of the flow of genetic information in bacteria. Nature. 466 (7302), 77-81 (2010).
- So, L. -. h., et al. General properties of transcriptional time series in Escherichia coli. Nature Genetics. 43 (6), 554-560 (2011).
- Taniguchi, Y., et al. Quantifying E. coli proteome and transcriptome with single-molecule sensitivity in single cells. Science. 329 (5991), 533-538 (2010).
- Jones, D. L., Brewster, R. C., Phillips, R. Promoter architecture dictates cell-to-cell variability in gene expression. Science. 346 (6216), 1533-1536 (2014).
- Iyer, S., Park, B. R., Kim, M. Absolute quantitative measurement of transcriptional kinetic parameters in vivo. Nucleic Acids Research. 44 (18), 142 (2016).
- Kim, S., Beltran, B., Irnov, I., Jacobs-Wagner, C. Long-Distance Cooperative and Antagonistic RNA Polymerase Dynamics via DNA Supercoiling. Cell. 179 (1), 106-119 (2019).
- Wang, M., Zhang, J., Xu, H., Golding, I. Measuring transcription at a single gene copy reveals hidden drivers of bacterial individuality. Nature Microbiology. 4 (12), 2118-2127 (2019).
- Joo, C., Ha, T. . Labeling DNA (or RNA) for single-molecule FRET. 2012 (9), 1005-1008 (2012).
- Sambrook, J., Russell, D. W. . Standard Ethanol Precipitation of DNA in Microcentrifuge Tubes. 2006 (1), 4456 (2006).
- Skinner, S. O., Sepúlveda, L. A., Xu, H., Golding, I. Measuring mRNA copy number in individual Escherichia coli cells using single-molecule fluorescent in situ hybridization. Nature Protocols. 8 (6), 1100-1113 (2013).
- Adesnik, M., Levinthal, C. The synthesis and degradation of lactose operon messenger RNA in E. coli. Cold Spring Harbor Symposia on Quantitative Biology. 35, 451-459 (1970).
- Campbell, E. A., et al. Structural mechanism for rifampicin inhibition of bacterial RNA polymerase. Cell. 104 (6), 901-912 (2001).
- Raj, A., Tyagi, S., Walter, N. G. . Methods in Enzymology. 472, 365-386 (2010).
- Sliusarenko, O., Heinritz, J., Emonet, T., Jacobs-Wagner, C. High-throughput, subpixel precision analysis of bacterial morphogenesis and intracellular spatio-temporal dynamics. Molecular Microbiology. 80 (3), 612-627 (2011).
- Paintdakhi, A., et al. Oufti: an integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Molecular Microbiology. 99 (4), 767-777 (2016).
- Moffitt, J. R., Zhuang, X., Filonov, G. S., Jaffrey, S. R. . Methods in Enzymology. 572, 1-49 (2016).
- Yu, J., Xiao, J., Ren, X., Lao, K., Xie, X. S. Probing gene expression in live cells, one protein molecule at a time. Science. 311 (5767), 1600-1603 (2006).
- Chen, H., Shiroguchi, K., Ge, H., Xie, X. S. Genome-wide study of mRNA degradation and transcript elongation in Escherichia coli. Molecular Systems Biology. 11 (1), 781 (2015).
- Vogel, U., Sørensen, M., Pedersen, S., Jensen, K. F., Kilstrup, M. Decreasing transcription elongation rate in Escherichia Coli exposed to amino acid starvation. Molecular Microbiology. 6 (15), 2191-2200 (1992).
- Yang, S., et al. Transcription and translation contribute to gene locus relocation to the nucleoid periphery in E. coli. Nature Communications. 10 (1), 5131 (2019).
- Zenklusen, D., Larson, D. R., Singer, R. H. Single-RNA counting reveals alternative modes of gene expression in yeast. Nature Structural & Molecular Biology. 15 (12), 1263-1271 (2008).
- Fontenete, S., Guimarães, N., Wengel, J., Azevedo, N. F. Prediction of melting temperatures in fluorescence in situ hybridization (FISH) procedures using thermodynamic models. Critical Reviews in Biotechnology. 36 (3), 566-577 (2016).
- Sepúlveda, L. A., Xu, H., Zhang, J., Wang, M., Golding, I. Measurement of gene regulation in individual cells reveals rapid switching between promoter states. Science. 351 (6278), 1218-1222 (2016).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-Dimensional Super-Resolution Imaging by Stochastic Optical Reconstruction Microscopy. Science. 319 (5864), 810-813 (2008).
- Moffitt, J. R., Pandey, S., Boettiger, A. N., Wang, S., Zhuang, X. Spatial organization shapes the turnover of a bacterial transcriptome. eLife. 5, 13065 (2016).
- Fei, J., et al. Determination of in vivo target search kinetics of regulatory noncoding RNA. Science. 347 (6228), 1371-1374 (2015).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。