Method Article
Sondando cinética mRNA no espaço e no tempo em Escherichia coli usando fluorescência de molécula única de duas cores na hibridização situ
Neste Artigo
Resumo
Este protocolo descreve uma aplicação de fluorescência de molécula única na hibridização situ (smFISH) para medir a cinética in vivo da síntese e degradação do mRNA.
Resumo
Fluorescência de molécula única na hibridização situ (smFISH) permite contar o número absoluto de mRNAs em células individuais. Aqui, descrevemos uma aplicação de smFISH para medir as taxas de transcrição e degradação do mRNA em Escherichia coli. Como o smFISH é baseado em células fixas, realizamos smFISH em vários pontos de tempo durante um experimento de curso de tempo, ou seja, quando as células estão passando por alterações sincronizadas após a indução ou repressão da expressão genética. Em cada ponto de tempo, sub-regiões de um mRNA são espectralmente distinguidas para o alongamento da transcrição da sonda e término prematuro. O resultado deste protocolo também permite analisar a localização intracelular de mRNAs e heterogeneidade em números de cópias mRNA entre as células. Usando este protocolo muitas amostras (~50) podem ser processadas dentro de 8h, como a quantidade de tempo necessária para apenas algumas amostras. Discutimos como aplicar este protocolo para estudar a transcrição e a cinética de degradação de diferentes mRNAs em células bacterianas.
Introdução
O fluxo de informações genéticas do DNA para o mRNA e da proteína é um dos processos celulares mais fundamentais, cuja regulação é importante para a aptidão celular1. O número de mRNAs em uma célula é determinado por dois processos dinâmicos, transcrição e degradação do mRNA. No entanto, como a transcrição e a degradação do mRNA são reguladas no tempo e no espaço de uma única célula não é completamente compreendida, em grande parte devido à escassez de métodos experimentais para medir quantitativamente sua cinética in vivo.
Métodos baseados em mRNAs totais extraídos de uma população de células, como mancha do Norte, RT-PCR, sequenciamento de RNA e microarrays de expressão genética, podem medir a diferença relativa nos níveis de mRNA e têm sido amplamente utilizados para analisar a taxa de alongamento da transcrição2,,3,,4,5 ou a taxa de degradação do mRNA6,,7. No entanto, eles não fornecem o número absoluto de mRNAs por célula e, portanto, não são adequados para sondar a taxa de iniciação de transcrição8. Além disso, como os mRNAs são extraídos de uma população de células, a distribuição espacial de mRNAs dentro de uma única célula e a variabilidade dos números de cópias mRNA entre as células não podem ser medidas.
O sequenciamento de RNA de última geração em células individuais (scRNAseq) pode quantificar o número de mRNAs por célula em uma escala genômica9. No entanto, ainda é difícil usar essa técnica para medir a cinética de transcrição, devido a desafios com preparação amostral e alto custo. Em particular, a aplicação de scRNAseq a bactérias tem sido tecnicamente difícil devido à baixa abundância de mRNA10,11.
A fluorescência de molécula única na hibridização situ (smFISH) baseia-se na hibridização de sondas de um único fio fluorescente cujas sequências são complementares ao mRNA alvo de interesse12,13. O conceito de hibridização específica de sequência é semelhante ao usado na mancha norte ou RT-PCR, mas a hibridização é feita in situ dentro de células fixas, para preservar a localização nativa de mRNAs. O sinal de um único mRNA é amplificado usando muitas sondas, ~20 nucleotídeos (nt) de comprimento, hibridizando para diferentes partes de um mRNA (Figura 1A)13. Nesta abordagem da sonda "ladrilho", o número de sondas necessárias para detectar um único mRNA estabelece um limite menor no comprimento do mRNA que pode ser avaliado. Alternativamente, o mRNA de interesse pode ser transcritivamente fundido a uma matriz não codificadora de sequências de operadores lac tandem, de tal forma que várias cópias de uma sonda lacO fluorescentemente rotulada hibridizem para um único mRNA(Figura 1B)14.
smFISH tem sido usado para quantificar o número de mRNAs por célula em estado estável (ou seja, quando a síntese e a decadência estão em equilíbrio) e para analisar a média e variabilidade das mRNAs entre as células bacterianas15,,16,,17. Recentemente, o smFISH foi aplicado para quantificar os números de mRNA em estado não estável, logo após a indução ou repressão da expressão genética em E. coli18,,19,20. As alterações temporais nos números absolutos de cópias do mRNA foram então utilizadas para calcular a taxa de iniciação, alongamento e término da transcrição, bem como a taxa de degradação do mRNA. Para esta aplicação, os procedimentos convencionais de smFISH podem ser complicados porque existem muitas amostras, cada uma representando um ponto de tempo, que precisam passar por múltiplas etapas de troca de buffer (ou seja, centrifugação e lavagem). Aqui, descrevemos um protocolo smFISH, no qual as etapas de manuseio da amostra são dramaticamente simplificadas por ter células aderidas à superfície de um deslizamento de cobertura e aspirando líquidos com um sistema de filtragem de vácuo14,19. Utilizando-se como exemplo a expressão de lacZ em E. coli, demonstra-se o fluxo de trabalho completo (Figura 2),incluindo a análise de imagem(Figura 3) produzindo a cinética in vivo da transcrição (iniciação, alongamento e terminação) e a degradação mRNA, variabilidade célula-celular na expressão mRNA e localização mRNA. Prevemos que o protocolo é amplamente aplicável à sonda cinética in vivo e localização de outros mRNAs em várias espécies de bactérias.
Protocolo
1. Preparação de sondas smFISH
NOTA: Para rotular as sondas smFISH com um único fluoróforo, siga um protocolo padrão para rotular oligonucleotídeos de ácido nucleico com base na química do ér NHS21.
- Projete sondas smFISH. Decida se deve usar sondas de "ladrilho" ou sondas "array"(Figura 1) para o gene de interesse. Veja a seção Discussão sobre como tomar a decisão.
- Para sondas de "ladrilho"(Figura 1A),use uma ferramenta de designer de sonda on-line (por exemplo, consulte Tabela de Materiais).
- Para sondas "array"(Figura 1B),execute uma pesquisa de sequência BLAST para garantir que a sequência do teste não seja complementar a nenhuma outra sequência de mRNA.
- Para estudar a transcrição de lacZ mRNA e a cinética de degradação, utilize dois conjuntos de 24 sondas, cada conjunto cobrindo a primeira e última região de 1 kb de lacZ (3.072 bp)19.
NOTA: Estes conjuntos de sondas são, doravante, chamados de "sonda mRNA de 5" e "sonda mRNA de 3", respectivamente. As sequências dessas sondas estão listadas na Tabela de Materiais.
- Ordem sequências de sonda como oligonucleotídeos de DNA com um linker de amino C6 no final de 5'. Dissolva sondas individuais na água para 1 mM.
- Combine quantidades equimolar de sondas para conjuntos de sonda "5" mRNA e "3" mRNA. Por exemplo, para o conjunto de sondas de 5' mRNA para lacZ,combine 20 μL de cada sonda (total de 24 tipos de sondas no conjunto).
- Realize a precipitação de etanol22 das sondas combinadas para remover quaisquer contaminações de aminas primárias e secundárias (como tris, glicina e sais de amônio) que possam inibir a reação de conjugação. No final, dissolva a pelota de DNA em 100 μL de água (rendendo ~4,5 mM de DNA em um conjunto de sondas).
NOTA: Esta etapa é recomendada mesmo que as sondas passaram por uma purificação de desalto padrão pelo fabricante. Uma purificação padrão baseada em filtro pode funcionar no lugar e, além da precipitação do etanol. - Escolha dois fluoroforos espectralmente distintos com um éster de sisário NHS monofuncional, de modo que os conjuntos de sondas de 5' e 3' mRNA possam ser rotulados diferencialmente. Por exemplo, prepare o ér Cy5 NHS para sondas de 5' mRNA e o éster Cy3B NHS para sondas de 3' mRNA. Dissolva cada tipo de fluoroforos em DMSO a 20 mg/mL final (~25 mM).
- Prepare 0,1 M de bicarbonato de sódio (pH 8,5) logo antes de cada reação de rotulagem. A exposição ao ar por muito tempo diminuirá seu pH e reduzirá a eficiência de rotulagem.
- Para a reação de conjugação, combine o seguinte: 15 μL do estoque de fluoróforo Cy5 (a partir da etapa 1,5), 4 μL de 5' mRNA conjunto de sonda (a partir da etapa 1.4), 75 μL de bicarbonato de sódio (a partir da etapa 1.6) e 7 μL de água. Enrole o tubo com papel alumínio e agite à temperatura ambiente por 3-6 h.
NOTA: A incubação mais longa não resulta necessariamente em maior eficiência de rotulagem. Além disso, a reação pode ser ampliada para cima ou para baixo se as concentrações dos componentes forem mantidas. - Repita o passo acima para o conjunto de sondas de 3' mRNA e o fluoróforo correspondente (ou seja, Cy3B NHS-éster).
- Realize a precipitação de etanol22 para remover moléculas de corante não reagidas. Dissolva a pelota em ~50 μL de tampão TE (10mM Tris-HCl pH 8.0 com 1mM EDTA).
- Estimar as concentrações de DNA e fluoróforo usando um espectrômetro UV-Vis.
- Meça a absorvância em 260 nm e 559 nm (Cy3B) ou 649 nm (Cy5). Se a amostra estiver muito concentrada para produzir uma medição precisa, dilui 1 μL da amostra para 10 μL.
- Converta a absorvância para a concentração:
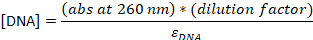
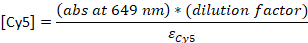
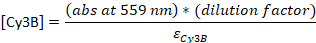
ε DNA = 0,2 μM-1 (para DNA de 20 nt single-stranded), εCy5 = 0,25 μM-1, e εCy3B = 0,13 μM-1
NOTA: [DNA] é a concentração de sondas totais dentro da solução. A concentração de sondas individuais é cerca de 24x menor. A concentração total de sondas será usada como "concentrações de sonda" a partir deste ponto. Se a razão entre [DNA] e [corante] for de 1, a seguinte etapa hplc pode ser ignorada23, e a amostra deve ser diluída para 4-5 μM final no buffer TE.
- (Recomendado) Purifique as sondas rotuladas de sondas sem rótulo e corantes livres usando HPLC.
NOTA: Embora esta etapa adicional de purificação leve à perda da amostra, é benéfica para as aplicações a jusante. A remoção de sondas de DNA não rotuladas aumentará o sinal de fluorescência dos alvos mRNA e a remoção de corantes não redigidos reduzirá a fluorescência de fundo.- Prepare o HPLC com uma coluna C18 analítica padrão, acetato de trietilamônio de 0,1 M (TEAA) como tampão A e acetonitrilo como tampão B.
- Adicione 1 M TEAA à amostra (a partir do passo 1.9) para fazer 0,1 M TEAA.
- Defina o programa de gradiente da seguinte forma: 0-5 min com 0% B, 5-35 min com um gradiente linear de 0-30% de B, 35-37 min com um gradiente linear de 30-100% de B, e 37-40 min com 0% B. Mantenha a taxa de fluxo em 0,1 mL/min e grave cromatogramas em 260 e 649 nm (para as amostras rotuladas cy5) ou a 260 e 559 nm (para as amostras registradas de Cy3B).
- Colete a amostra elucida quando a absorvância aumenta nos canais de DNA e fluorforeiro.
- Concentre a amostra elucida usando um concentrador de vácuo e suspenda a pelota em 50-100 μL TE buffer.
- Verifique a concentração de DNA e fluoróforo usando um espectrômetro UV-Vis (ver Passo 1.10). Diluir, se necessário, para fazer concentração final em torno de 4-5 μM. Armazene as sondas a -20 °C
2. Preparação de soluções
- Prepare um grande volume de água tratada e tampões tratados com DEPC(Tabela 1). Essas soluções podem durar mais de um ano em temperatura ambiente.
- Prepare a solução de fixação 4x e a solução de lavagem(Tabela 1).
- Prepare a solução de pré-hibridização e teste de hibridização(Tabela 1). Prepare a solução de hibridização da sonda durante a incubação na Etapa 5.1 ou etapa 6.1 e, em seguida, mantenha a solução em um agitador de bancada de 37 °C por 20-40 min com uma tampa para minimizar a exposição à luz.
NOTA: As concentrações de formamida, SSC e sonda foram otimizadas para os conjuntos de sonda lacZ para minimizar a fluorescência de fundo, maximizando o sinal real. Consulte a seção Discussão para obter detalhes sobre como modificar essas concentrações para diferentes aplicações.
3. Preparação de tampas e lâminas de vidro
- Tampas limpas e lâminas de vidro.
- Coloque tampas individuais e slides em um frasco de Coplin usando fórceps. Certifique-se de que as tampas e slides estão separados e não se tocando.
- Encha o pote com 100% de etanol e feche a tampa. Coloque o frasco em um limpador ultrassônico de banho de água e sonicato por 15-20 min.
NOTA: Para o sonicador de banho de água, recomenda-se desligar a função do aquecedor. - Despeje o etanol e lave com água ultrauso 3-4x. Use água que flui diretamente da máquina de purificação de água.
- Despeje a água do frasco e encha-a com 70% de etanol. Feche a tampa e realize a sônica por 15-20 min e lave com água ultrapura.
- Encha o frasco com água ultrauso e sonicato por 15-20 min.
NOTA: Deslizamentos de tampas e lâminas de vidro podem ser mantidos durante a noite no frasco de Coplin cheio de água ultrapura. - Pegue um slide ou uma mancha de cobertura do frasco coplin usando fórceps limpos e seque-o usando gás N2. Repita isso para os slides e deslizamentos restantes.
- Coloque os slides secos em uma caixa de armazenamento limpa até usar na Etapa 7.5. Coloque as tampas secas em uma caixa de ponta de pipeta vazia de 1.000 μL, que servirá como uma "câmara" no procedimento restante.
- Usando um marcador hidrofóbico, desenhe círculos nas tampas seguindo furos circulares na caixa de ponta da pipeta. Esses círculos (~0,5 cm de diâmetro) servirão como "poços". Espere pelo menos 5-10 min para que o marcador seja completamente seco.
NOTA: Mantenha sempre a tampa da caixa de ponta fechada. - Aplique uma queda de 20-μL de 0,1% de poli-L-lysine em cada poço. Incubar por 10-50 min em temperatura ambiente.
NOTA: Ajuste este volume de acordo com o tamanho do poço. Certifique-se de que a solução cobre completamente a área do poço. Para uma maior incubação, tenha cuidado para evitar a evaporação. - Após a incubação aspirar poli-L-lysine sem tocar na superfície, pois isso raspará a poli-L-lysina fora. Em seguida, aplique uma gota (~20 μL) de água DEPC nos poços tratados de poli-L-lysina. Feche a tampa da "câmara" para evitar a evaporação até o passo 5.1.
4. Experimento de curso de tempo e fixação de amostras
- Cresça células E. coli em cultura líquida de ~20 mL em um frasco de 250 mL. Mantenha o frasco em um agitador de banho de água (30 °C) e continue tremendo. Pare o agitador somente ao colher amostras.
NOTA: Os resultados apresentados neste artigo são obtidos de células MG1655 cultivadas em M9 médio mínimo suplementado com 0,2% glicerol, 0,1% casaminoácidos e 1 mg/L de tiamina para uma fase de crescimento exponencial (OD600~0,2). - Adicione 250 μL da solução de fixação 4x em um tubo vazio de 1,5 mL. Repita e prepare vários tubos, tanto quanto os pontos de tempo a serem tomados. Rotule os tubos com números de ponto de tempo e mantenha-os em temperatura ambiente.
- Tome 750 μL de cultura celular (OD600~0,2) antes de iniciar um experimento de curso de tempo. Adicione a cultura a um tubo marcado para "tempo zero" (a partir do passo 4.2). Inverta o tubo suavemente para misturar células com a solução de fixação.
NOTA: Não encobrindo escote para misturar, vórtice ou "ser áspero" nas células. Esta amostra representa o estado reprimido e será usada como controle para calcular a intensidade de fluorescência de um único mRNA (ver Passo 9.4). - Adicione 0,02-1 mM de isopropílico β-D-1-thiogalactopyranoside (IPTG) à cultura líquida para induzir a expressão lacZ. Inicie um temporizador neste ponto (t = 0 min) e amostra em um determinado intervalo de tempo (por exemplo, a cada 1 minuto) a partir de então. Para amostragem, repita o passo 4.3.
- Adicione 5 mM orthonitropheynl-β-D-fucopyranoside (ONPF) ou 500 mM de glicose24 em um determinado momento durante o experimento do curso de tempo (por exemplo, em t = 1,5 min) para reprimir a expressão lacZ. Após a repressão, continue a amostrar as culturas (Passo 4.3) para acompanhar a degradação do mRNA.
NOTA: A repressão também pode ser feita com ~400 μg/mL rifampicina, um inibidor de iniciação de transcrição25. - Para fixação, incubar os tubos contendo células amostradas à temperatura ambiente por 15 minutos, seguido de incubação no gelo por 30 minutos.
- Para remover fixativos, centrifugar os tubos a 4.500 x g por 4 min a temperatura ambiente. Remova o supernatante com uma pipeta.
NOTA: Certifique-se de descartar formaldeído em um recipiente de resíduos separado seguindo o protocolo de segurança. - Adicione 1 mL DEPC-PBS e suspenda as células. Repita a centrifugação e a re-suspensão 2x mais vezes.
NOTA: As células fixas são frágeis e precisam de tratamento suave. Suspenda cuidadosamente a pelota e evite bolhas. - Após a etapa final de lavagem, suspenda as células em ~30 μL DEPC-PBS.
5. Permeabilização das membranas celulares
- Aplique cada amostra de ponto de tempo em diferentes poços no deslizamento de tampas (~30 μL por poço). Aguarde de 10 a 30 minutos à temperatura ambiente para que as células aderam na superfície. Evite a fusão das gotas líquidas entre os poços.
- Para enxaguar células sem entrada, aspire o líquido e aplique ~20 μL DEPC PBS em cada poço. Aspirar DEPC PBS dentro de alguns minutos.
- Permeabilize as membranas celulares aplicando 15 μL de 70% de etanol em cada poço por 4 min. Aspire o etanol depois dos 4 minutos, e certifique-se de que os poços estão completamente secos.
NOTA: É fundamental limitar o tratamento do etanol por 4 minutos. Um tratamento mais longo resultará em superemmenesorização. - Aplique 30 μL da solução de lavagem a cada poço.
6. Hibridização da sonda
- Aspire a solução de lavagem de cada poço. Aplique 30 μL da solução de pré-hibridização a cada poço. Incubar a câmara no forno de 37 °C por 30 min.
NOTA: Adicione ~50 mL de água ao fundo da câmara para fornecer umidade. - Aspire a solução de pré-hibridização de cada poço. Aplique ~30 μL da solução de hibridização da sonda a cada poço. Cubra a câmara com papel alumínio e incubar no forno de 37 °C por 2h.
NOTA: Certifique-se de que a solução de hibridização da sonda está no agitador de bancada de 37 °C antes desta etapa. Evite a fusão de líquidos entre os poços. Aplique um volume menor da solução a cada poço, se necessário.
7. Lavagem pós-hibridização e preparação para imagem
- Usando uma pipeta multicanal, aplique ~30 μL da solução de lavagem para cada poço de uma só vez. Aspire e repita 3-5x vezes de lavagem. Incubar a câmara no forno de 37 °C por 15-30 min.
- Repita o passo 7.1 mais duas vezes.
- Lave cada poço com DEPC-PBS 5x vezes. Siga o método utilizado na Etapa 7.1, mas pule o processo de incubação.
- Aspire o líquido da tampa. Aplique 4 μL de DEPC-PBS a cada poço.
- Usando fórceps, levante e vire a tampa e coloque-a suavemente sobre um escorregador de vidro (da Etapa 3.2). Evite bolhas.
- Sele as bordas da tampa com goma de silicone.
- Espere até a gengiva se solidificar. Pode-se fazer uma pausa aqui e armazenar o slide durante a noite a 4 °C.
NOTA: Outros protocolos smFISH sugerem adicionar reagentes de limpeza de oxigênio (por exemplo, glicose oxidase/catalase) ou usar um meio comercial de montagem anti-fade14,26 para aumentar a fotoestabilidade dos fluoroforos.
8. Imagem
- Para encontrar uma área de interesse, use o modo ao vivo de imagem de contraste de fase. Mude o campo de visão dentro de um poço manobrando o joystick do palco. Escolha uma área onde a densidade celular é ótima (ou seja, há muitas células que são principalmente separadas). Ajuste o foco z de modo que as imagens celulares de contraste de fase estejam em foco.
- Tire instantâneos na ordem de Cy5 (exposição de 4 s), Cy3 (exposição de 2 s) e contraste de fase (exposição de 0,2 s).
NOTA: As moléculas de corante Cy3B são imagens no canal Cy3, e as imagens são referidas como imagens Cy3. - Repita as etapas 8.1-8.2 para adquirir imagens de ~10 áreas diferentes dentro de um poço.
- Mova o objetivo para outro poço e repita as etapas 8.1-8.3.
- Exportar imagens como arquivos TIFF.
- (Opcional) Contas multicoloridos de imagem adsorvidas na superfície de deslizamento de cobertura nos canais Cy5 e Cy3 para determinar a mudança espacial entre os canais Cy5 e Cy3 para fins de registro de imagem.
- Aplique ~10 μL de contas fluorescentes multicoloridas (0,2 μm de diâmetro) em uma superfície de deslizamento de cobertura limpa e espere por 10-30 min. Depois de lavar com ~50 μL de PBS, aplique ~5 μL de PBS e sanduiche o deslizamento com um slide de vidro. Selar e montar no microscópio.
- Contas de imagem nos canais Cy5 e Cy3.
9. Análise de imagem
NOTA: O código Matlab usado nesta etapa está disponível no seguinte site do GitHub: https://github.com/sjkimlab/Code_Publication/tree/master/JoVE_2020. A pasta GitHub contém tudo o que é necessário para a análise de imagens, incluindo valores de parâmetros para segmentação celular e identificação de manchas. O procedimento nesta etapa é ainda explicado no script mestre, chamado "FISHworkflow.m".
- Abra uma ferramenta de segmentação celular, como microbeTracker27 ou Oufti28,e imagens de contraste de fase de carga. Escolha "Quadros independentes" e pressione um botão chamado "Todos os quadros" para iniciar o processo de segmentação, a partir do qual as células são identificadas e seus contornos são calculados(Figura 3B,C).
NOTA: Protocolos detalhados para o uso desses pacotes de software estão disponíveis online (por exemplo, oufti.org). - Carregue imagens de fluorescência Cy5 na função spotFinder de micróbiosTracker ou Oufti, e pressione o botão "Executar" para iniciar a identificação e quantificação do local com base no encaixe gaussiano 2D(Figura 3B,C). Repita esta etapa para imagens de fluorescência Cy3 analisarem manchas no canal Cy3. Esta etapa produz uma lista de pontos em cada célula, incluindo suas intensidades e coordenadas.
- (Opcional) Filtrar pontos fracos (falsos positivos) usando um limiar, como explicado no arquivo FISHworkflow.m.
NOTA: Examine as manchas fluorescentes no controle negativo (por exemplo, MG1655 ΔlacZ) e determine o limiar para filtrar falsos positivos. - Para obter a intensidade spot de um único mRNA, use uma lista de intensidades spot medidas no tempo zero (antes de adicionar IPTG), e encaixe na distribuição de intensidades spot com um modelo de mistura gaussiana com dois componentes de mistura. Tome a posição máxima da primeira população gaussiana (linha preta na Figura 3D,E) como a intensidade spot de um único mRNA. Execute isso para as vagas cy5 e Cy3 separadamente para obter a intensidade spot de um único mRNA de 5' e 3' lacZ.
NOTA: Repita isso em todos os experimentos de tempo- curso porque a intensidade spot de um único mRNA pode variar ligeiramente em experimentos diferentes. - Divida a intensidade de fluorescência de um ponto com a intensidade de um único mRNA (a partir do passo 9.4) para obter o número de mRNAs dentro de um local. Somam intensidades de manchas normalizadas dentro de uma célula para calcular o número total de mRNA em uma célula (Figura 3F). Realize estes cálculos para 5' e 3' mRNA separadamente.
- Calcular e traçar os números médios de mRNA por célula em cada ponto de tempo (por exemplo, Figura 4B), e analisar a cinética in vivo da transcrição e da degradação do mRNA a partir da mudança temporal nos níveis médios de mRNA(Figura 4B).
- Para obter a taxa de alongamento da transcrição, realize um encaixe de linha menor para o aumento inicial em sinais de mRNA de 5' e 3' e identifique interceptações para os níveis basais(Figura 4B). A diferença entre essas interceptações indica o tempo médio para os RNAPs viajarem da região da sonda de 5' para a região da sonda de 3'. Divida a distância entre dois conjuntos de sondas (2 kb) com este tempo para obter a taxa média de alongamento da transcrição.
- Para obter a taxa de degradação do mRNA, encaixe uma função exponencial de decaimento, y = A·exp(-t/τ) à região de decadência final dos sinais de decadência de 5' e 3' (por exemplo, Figura 4B). O parâmetro de montagem, τ, é a vida média do mRNA.
- (Opcional) Analise a variação célula-célula na expressão genética (por exemplo, a resposta de nível celular à indução mostrada na Figura 4C), com base na distribuição dos números de mRNA em cada célula (calculada na Etapa 9.5).
- (Opcional) Utilizando informações sobre localização spot ao longo dos eixos principais e menores de uma célula (obtidas a partir da Etapa 9.2), analise a localização de mRNAs (Figura 4D,E).
- (Opcional) Analisar a co-localização de 5' e 3' mRNAs (Figura 5)comparando a localização das manchas detectadas nos canais Cy5 e Cy3.
- Carregue imagens de contas multicoloridas (Passo 8.6) na função spotFinderF no microesferaTracker e obtenha coordenadas de quartos de contas nos canais Cy5 e Cy3. Use a lista de coordenadas centóides para calcular a matriz de transformação afim, que informa como os canais Cy5 e Cy3 são deslocados e girados em relação uns aos outros29.
- Aplique a matriz de transformação afim às imagens Cy5 e Cy3 FISH para converter imagens Cy3 na coordenada Cy5. Classifique se uma vaga é co-localizada com outro ponto em um canal diferente. Por exemplo, uma vaga no canal Cy5 é considerada co-localizada com outra vaga no canal Cy3 se a distância entre seus centroides for inferior a 150 nm(Figura 5).
- Analise quantas manchas de Cy5 são classificadas como "co-localizadas" com pontos Cy3 em cada ponto de tempo. Além disso, analise a intensidade dos pontos co-localizados(Figura 5).
Resultados
A Figura 3 mostra imagens representativas deste protocolo smFISH. Um campo de visão completo (86,7 μm x 66,0 μm usando nossa configuração de microscopia detalhada em Tabela de Materiais) mostra ~500 células E. coli dispersas por todo o campo(Figura 3A). Se a densidade das células é muito maior do que a mostrada nesta imagem, a segmentação automática de células torna-se difícil, pois os algoritmos de segmentação não identificam de forma confiável células individuais quando as células se tocam. É preciso ajustar a concentração das células e o tempo de incubação utilizado para a adesão à superfície (Passo 5.1) para alcançar a densidade ideal das células no campo de visão.
A morfologia das células nas imagens de contraste de fase deve permanecer comparável à das células vivas para fins de segmentação(Figura 3A-C). Se as células são super-permeabilizadas, a morfologia celular muda (como "fantasmas"; Figura Suplementar 1). Nesse caso, pode-se reduzir a duração do tratamento de 70% do etanol na Etapa 5.3.
Antes da indução, o nível médio de expressão lacZ era de ~0,03 mRNAs por célula, consistente com os relatórios anteriores15,,30. Além disso, a distribuição das intensidades de manchas de lacZ mRNA antes da indução não se encaixava bem com uma distribuição normal ou uma distribuição de Poisson devido à presença de manchas com alta intensidade(Figura 3D,E). Isso sugere que a maioria das manchas detectadas sob o estado reprimido representam um único lacZ mRNA, mas uma pequena população de manchas contém mais de um mRNA lacZ. Para isolar a população com um único lacZ mRNA, utilizou-se um modelo de mistura gaussiana com dois componentes de mistura (insets na Figura 3D,E). Em seguida, a média do primeiro gaussiano foi tomada como a intensidade média de um único ponto mRNA (por exemplo, o pico da curva negra na Figura 3D) e usada para converter a intensidade spot para o número de mRNAs, para quaisquer pontos detectados no experimento time-curso. Para calcular o número total de mRNAs dentro de uma célula, as intensidades de manchas normalizadas foram somadas em cada célula (Figura 3F)19.
Quando o nível de expressão de lacZ mRNA é baixo, há um ou dois pontos de mRNA lacZ limitados por difração espacialmente separados dentro de uma célula. Assim, as imagens dessas manchas podem ser analisadas por encaixe gaussiano 2D para sua intensidade e localização.
Quando o nível de expressão é alto, de tal forma que as manchas se sobrepõem umas às outras dentro de uma célula, o encaixe gaussiano 2D não faz quantificação confiável. Nesse caso, o nível de mRNA deve ser calculado dividindo o sinal total de fluorescência subtraída em fundo dentro de uma célula com a intensidade média de um único mRNA19.
Quando a expressão de lacZ é induzida, o sinal de 5' lacZ mRNA aumenta primeiro e o de 3' lacZ mRNA aumenta mais tarde(Figura 4B). Se a expressão de lacZ for reprimida, os sinais de mRNA de 5' e 3' lacZ diminuem com algum atraso no meio(Figura 4B). Para obter a taxa de alongamento da transcrição, o aumento dos sinais de 5' e 3' são primeiro adequados com linhas(Figura 4B), e a diferença nas interceptações x são tomadas como o tempo para os RNAPs percorrerem a distância entre duas regiões de sonda (2.000 nt). A taxa de alongamento da transcrição pode ser medida a partir de cada experimento de curso-tempo e desvios padrão podem ser calculados a partir de duplicatas experimentais. A taxa média de alongamento da transcrição foi de 15-30 nt/s em nossas condições experimentais19.
Além disso, a taxa de degradação do mRNA (inverso da vida média do mRNA) foi obtida mediante a adequação da região de decaimento com uma função exponencial(Figura 4B). Nossos dados do curso de tempo contêm degradação do mRNA durante e após a transcrição31. Encaixamos os pontos de tempo depois que 3' mRNA começou a decair (t > 6 min) para sondar a degradação de mRNAs lançados. Obtivemos ~90 s como uma vida média de 5' ou 3' lacZ mRNA19.
A taxa de iniciação da transcrição pode ser calculada a partir da inclinação do aumento do sinal de 5' após a indução (Figura 4B, azul), ou do número médio de mRNA em estado estável (que é a taxa de iniciação dividida pela taxa de degradação). Além disso, a probabilidade de rescisão de transcrição prematura pode ser estimada, seja tomando a razão entre o aumento de sinal de inclinação de 3' versus o de 5' sinal aumentode 32 ou entre os níveis de estado estável de 3' e 5' mRNA regiões19.
Como o SMFISH é uma técnica unicelular, podemos analisar a variabilidade célula-celular na transcrição. Por exemplo, pode-se analisar a porcentagem de células expressando lacZ mRNA após a adição do IPTG(Figura 4C). Pode-se também abordar se a localização do mRNA muda após a indução. Observamos que as manchas de mRNA de 5' e 3' lacZ movem-se ligeiramente para fora, longe do centro da célula (Figura 4D,E), consistentes com um relatório anterior33.
Por fim, a análise da co-localização entre as manchas de 5' e 3' mRNA pode ser informativa (Figura 5A). Por exemplo, no estado reprimido (tempo zero), cerca de 25% das vagas de 5' mRNA são co-localizadas com um ponto de 3' mRNA. Em t = 1 min, como muitos gene loci têm síntese de 5' mRNA, mas ainda não 3' mRNA síntese, a maioria dos pontos de 5' mRNA são por si só sem sinal de 3' mRNA (ou seja, baixa probabilidade de co-localização). No entanto, quando o mRNA de 3' aparece (i.e., t = 2 min), a probabilidade de co-localização aumenta (seta roxa na Figura 5A,B). Neste momento, quando a co-localização se torna frequente, depende da taxa de alongamento da transcrição. O gráfico de densidade 2D de 5' e 3' números de mRNA lacZ dentro de cada ponto de co-localização detectado neste momento pode ser usado para inferir a densidade de RNAPs no gene lacZ (Figura 5C). Como relatado anteriormente19, os números de 5' mRNA nesta trama indicam que a maioria dos lacZ loci têm menos de 10 RNAPs no DNA quando a expressão lacZ é induzida por 1 mM IPTG. Além disso, os números de 3' mRNA nesta trama estão relacionados com o agrupamento de RNAPs34. O fato de que o número de 3' mRNA é próximo de um significa que aproximadamente apenas um RNAP entra na região da sonda de 3'. Isso sugere que os RNAPs no gene lacZ são espacialmente separados, em vez de formar um cluster (ou "comboio").
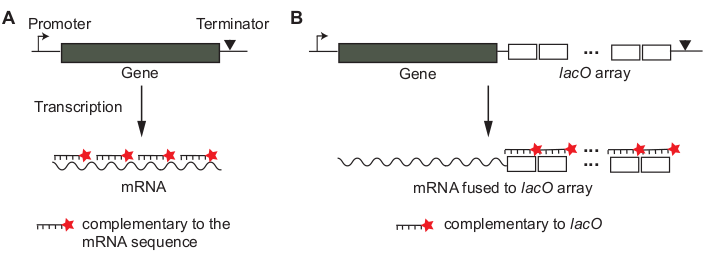
Figura 1: Projeto de sondas smFISH para um mRNA de interesse. UmAmétodo de revestimento. Sequências de oligonucleotídeos de DNA curtos (~20 bp de comprimento) são escolhidas para que possam cobrir o mRNA de interesse. As sondas de oligonucleotídeos são rotuladas com uma molécula de corante fluorescente. (B) Um método de matriz. Uma matriz não codificadora de sequências tandem (por exemplo, "matriz lacO") é transcritivamente fundida ao mRNA de interesse. Sonda fluorescentemente rotulada complementar à unidade de repetição (por exemplo, sonda lacO de 17 bp de comprimento) é usada para amplificar o sinal de um mRNA. Clique aqui para ver uma versão maior desta figura.
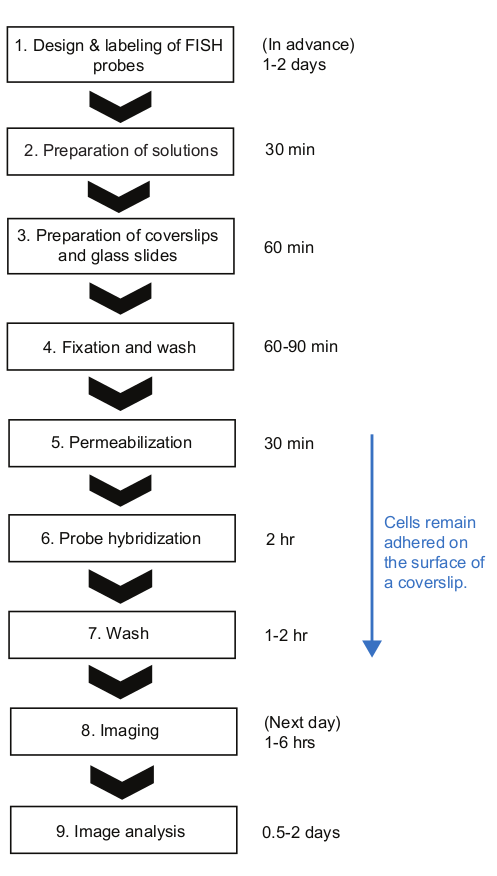
Figura 2: Esquema de procedimento experimental smFISH e duração do tempo de cada etapa. Clique aqui para ver uma versão maior desta figura.

Figura 3: análise de imagem smFISH. (A-C) imagem de microscopia smFISH de 5' lacZ mRNA (vermelho) e 3' lacZ mRNA (verde) em E. coli (MG1655) cultivada em M9 médio mínimo suplementado com 0,2% de glicerol, 0,1% de casaminoácidos, e 1 mg/L de tiamina a 30 °C. (A) Imagem representativa de uma amostra de t = 3 min após indução com 0,05 mM IPTG em t = 0 min e repressão com glicose de 500 mM em t = 1,5 min. O contraste de fase e duas imagens de fluorescência de Cy5 (para 5' lacZ mRNA, vermelho) e Cy3 (para 3' lacZ mRNA, verde) foram sobrepostos com pseudo-coloração. A imagem mostra um campo inteiro de 86,7 μm x 66,0 μm. Barra de escala, 5 μm. (B) Versão zoom-in de uma pequena região (caixa amarela) em (A). Contornos celulares são mostrados em branco, e manchas de fluorescência identificadas a partir da análise de imagem são mostradas com pontos vermelhos. Barra de escala, 1 μm. (C) Detecção de contornos celulares e manchas fluorescentes sob uma condição de alta expressão (t = 4 min após indução com 1 mM IPTG). Barra de escala, 1 μm. (D-E) Distribuições de intensidades de manchas de 5' e 3' mRNA medidas antes de adicionar IPTG (o estado reprimido). Os histogramas são mostrados com duas funções gaussianas (preto e cinza) cujos valores médios são do modelo de mistura gaussiana. O inset mostra um gráfico quântico de números aleatórios gerados a partir dos modelos de mistura gaussiana e intensidades de manchas de mRNA medida experimentalmente (n = 1040 para 5' mRNA e 680 para 3' mRNA). (F) Informações obtidas para uma célula individual apontada no painel (B). Para uma determinada célula (i), as manchas foram identificadas nos canais Cy5 e Cy3, e sua intensidade (I) e coordenada ao longo do eixo curto e longo de uma célula (d, l) foram quantificadas a partir do encaixe gaussiano 2D. Após a normalização, as intensidades das manchas foram somadas para produzir o número total de 5' ou 3' mRNAs nesta célula. Além disso, a co-localização entre pontos de diferentes canais pode ser analisada como no exemplo mostrado na Figura 5. Clique aqui para ver uma versão maior desta figura.

Figura 4: Análise da cinética in vivo da transcrição e degradação do mRNA. (A) Imagens esquemáticas e representativas de experimentos de smFISH de duas cores que medem mudanças nos níveis de mRNA lacZ ao longo do tempo. Linhas pontilhadas vermelhas e verdes indicam sondas oligonucleotídeos rotuladas cy5 ou Cy3B que hibridizam as regiões de 1 kb-long de 5' e 3' mRNA de lacZ mRNA em E. coli,respectivamente. Também são mostradas sobreposições de duas imagens de fluorescência com uma imagem de contraste de fase em pontos de tempo indicados após indução com IPTG de 0,2 mM em t = 0 min. A transcrição foi reprimida com 500 mM de glicose em t = 1,5 min. Barra de escala, 1 μm. O valor foi modificado a partir de Kim et al19. (B) 5' e 3' lacZ mRNA números por célula ao longo do tempo, durante o experimento descrito no painel (A). As barras de erro são SEMs bootstrapped. Pelo menos 1.200 células foram analisadas por ponto de tempo. A subida inicial dos sinais de 5' e 3' mRNA foi adequada com uma linha (azul). A diferença nas interceptações x foi de 1,93 min, rendendo a taxa média de alongamento da transcrição de 17,3 nt/s. A decadência final dos sinais de 5' e 3' mRNA foi encaixada com uma função de decadência exponencial (cinza). Os parâmetros de ajuste indicam que a vida útil média do mRNA é de 1,52 min para 5' mRNA e 1,66 min para 3' mRNA. (C) Percentual de células com uma ou mais manchas de lacZ mRNA durante o experimento descrito em (A). As barras de erro são SEMs bootstrapped. (D) Localização de um ponto ao longo do eixo curto de uma célula. Pode-se quantificar a proximidade de um ponto com a membrana dividindo a localização ao longo do eixo curto(d)com meia largura da célula(w). (E) Mudança na localização de manchas de mRNA de 5' e 3' lacZ ao longo do eixo curto das células durante o experimento descrito em (A). Clique aqui para ver uma versão maior desta figura.
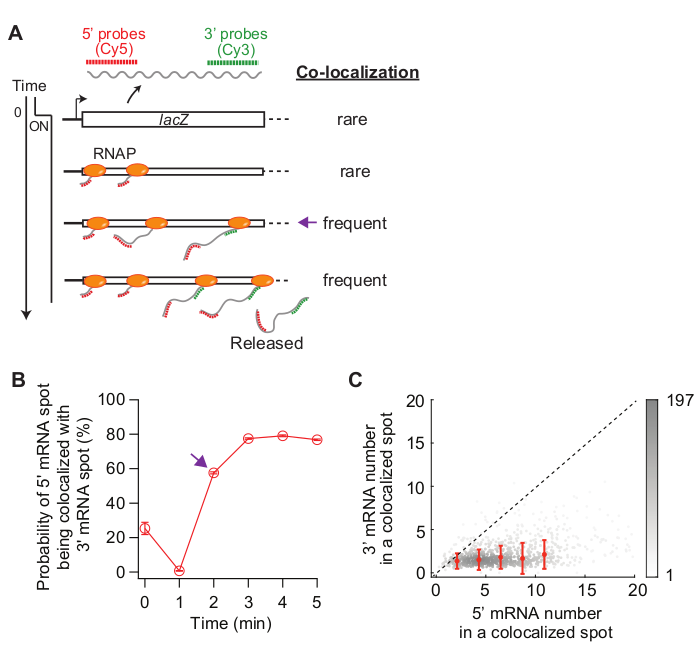
Figura 5: Análise de co-localização de 5' e 3' mRNA spots. (A) Esquema mostrando a co-localização esperada entre 5' e 3' mRNA pontos após indução. Quando o mRNA de 3' é feito, a probabilidade de um ponto de 5' mRNA ser co-localizado com um ponto de 3' mRNA aumenta (seta roxa). (B) A probabilidade de co-localização após indução com 1 mM IPTG. A seta roxa indica o ponto de tempo onde a probabilidade de co-localização se torna frequente de acordo com o esquema no painel (A). (C) O número de mRNAs de 5' e 3' lacZ dentro de um ponto de co-localização detectado em t = 2 min após indução com 1 mM IPTG (total de 841 pontos). Os pontos cinzentos representam pontos co-localizados individuais, enquanto os pontos vermelhos representam a média de dados binados. As barras de erro são SEM. A tonalidade de cinza indica a densidade de pontos em uma determinada área do gráfico. A linha pontilhada indica uma inclinação de 1. Clique aqui para ver uma versão maior desta figura.
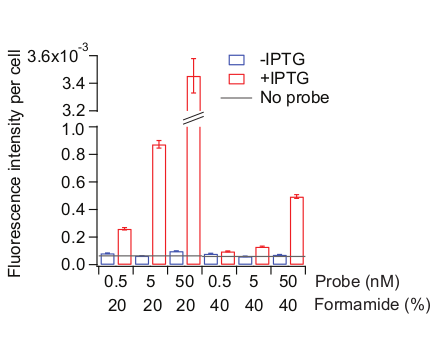
Figura 6: Otimização da condição de hibridização da sonda. Foram utilizadas duas amostras: células MG1655 cultivadas como descritas na Figura 3 e permanecem sem induzida (azul) ou tratadas com IPTG de 0,5 mM por 20 min (vermelho). A solução de hibridização da sonda foi feita com diferentes concentrações de sondas (total de 72 sondas conjugadas cy5 que inguram toda a região lacZ) e de formamida. As concentrações de formamida também foram ajustadas na solução de pré-hibridização e na solução de lavagem, em conformidade. "Nenhuma sonda" (linha cinza) indica o nível de fluorescência das células adicionadas ao IPTG tratadas sem sondas durante a etapa de hibridização. A intensidade média de fluorescência normalizada pela área celular (UA) foi calculada a partir de 300-800 células. As barras de erro são SEMs bootstrapped. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Morfologias celulares distorcidas devido à supermenabilização. Sobreposição de contraste de fase (escala cinza), 5' lacZ mRNA (Cy5, vermelho) e imagens de 3' lacZ mRNA (Cy3, verde) de células MG1655 5 min após a indução com 1 mM IPTG. (A) Um exemplo mostrando mistura de células normais e células excessivamente permeabilizadas sem morfologia normal (indicada com setas rosa). (B) Um exemplo mostrando células "fantasmagóricas" agrupadas. Barra de escala = 1 μm. Clique aqui para baixar este arquivo.
| Água DEPC | ||||
| Adicione 0,1% DEPC à água ultrauso e incubar a garrafa (coberta) no forno de 37°C durante a noite e autoclave no dia seguinte. | ||||
| DEPC PBS (10X) | ||||
| Misture o seguinte: | ||||
| 80 g | NaCl (final 1,37 M) | |||
| 2 g | KCl (final de 27 mM) | |||
| 14,2 g | Na2HPO4 (final de 100 mM) | |||
| 2,7 g | KH2PO4 (final de 20 mM) | |||
| Água ultrauso para 1L | ||||
| Filtro (0,22 μm) em uma garrafa de vidro. | ||||
| Adicione 0,1% DEPC e siga a instrução para água DEPC. | ||||
| Para fazer a solução 1X, diluir 10 vezes com água DEPC. | ||||
| 1M depc tampão fosfato de sódio, pH 7.4 | ||||
| Misture o seguinte: | ||||
| 115 g | Na2HPO4 | |||
| 22,8 g | NaH2PO4 | |||
| Água ultrauso para 1 L | ||||
| Filtro (0,22 μm) em uma garrafa de vidro. | ||||
| Adicione 0,1% DEPC e siga a instrução para água DEPC. | ||||
| Solução de fixação 4X (16% formaldeído) | ||||
| 5 mL | 20% de formaldeído | |||
| 500 μL | Água DEPC | |||
| 750 μL | 1M depc tampão fosfato de sódio, pH 7.4 | |||
| Armazene a 4 °C por até 2-4 semanas. | ||||
| ATENÇÃO: O formaldeído é tóxico. Use luvas e use um capuz de fumaça ao fazer esta solução. | ||||
| Solução de lavagem | ||||
| Misture o seguinte: | ||||
| 10 mL | Formamida (25% final) | |||
| 4 mL | 20X SSC (final 2X) | |||
| Encha água DEPC a 40 mL | ||||
| Filtrar (0,22 μm) e armazenar a 4 °C | ||||
| ATENÇÃO: A formamida é tóxica. Use luvas e use um capuz de fumaça ao fazer esta solução. | ||||
| Solução de pré-hibridização | ||||
| 200 μL | Formamida (20% final) | |||
| 100 μL | 20X SSC (final 2X) | |||
| 10 μL | 100X VRC (final 1X) | |||
| 25 μL | 4% (w/v) BSA (final 0,1%) | |||
| 685 μL | Água DEPC | |||
| NOTA: Vortex as ações vrc antes de tirar 10 μL para fora. | ||||
| ATENÇÃO: A formamida é tóxica e um teratogênio conhecido. Use luvas e manuseie-o sob um capô de fumaça. | ||||
| Solução de hibridização da sonda | ||||
| 200 μL | Formamida (20% final) | |||
| 100 μL | 20X SSC (final 2X) | |||
| 10 μL | 100X VRC (final 1X) | |||
| 25 μL | 4% (w/v) BSA (final 0,1%) | |||
| 10 μL | 40 mg/mL E. coli tRNA (final 0,4 mg/mL) | |||
| 200 μL | 50% de sulfato de dextran (10% final) | |||
| x μL | Conjunto de sondas mRNA de 5' (do passo 1.12) até o final de 4 nM. | |||
| y μL | 3' mRNA conjunto de sonda (da etapa 1.12) até o final de 4 nM. | |||
| - | Água DEPC para fazer o volume total de 1 mL | |||
| NOTA: Adicione por último o sulfato dextran. Por ser muito viscoso, corte a ponta de uma ponta de pipeta antes de tirar 200 μL do estoque de 50%. Depois de adicionar sulfato dextran, pipeta para cima e para baixo para homogeneizar a solução. | ||||
Tabela 1: Receitas das soluções utilizadas.
Discussão
Aqui, apresentamos um protocolo smFISH para medir cinética mRNA em E. coli. Nos protocolos smFISH publicados anteriormente para bactérias23, as células foram mantidas nos tubos até o final do protocolo, ou seja, até que estejam prontas para a imagem. Embora tenha muitos benefícios, como a ligação mínima e inespecífica de sondas fluorescentes na superfície do deslizamento de cobertura23,é difícil seguir esses protocolos quando há muitas amostras de um experimento de curso de tempo. Primeiro, um volume relativamente grande de células (>1 mL) precisa ser amostrado e até mesmo colhido antes da fixação. Em segundo lugar, as amostras de células precisam ser centrifugadas várias vezes para trocar soluções e lavar após a etapa de hibridização. Em nosso protocolo, um pequeno volume (<1 mL) de cultura é diretamente misturado com uma solução de fixação em um tubo de 1,5 mL, ajudando a "congelar" rapidamente o estado celular no momento da amostragem. Além disso, as células permanecem presas à superfície durante todo o procedimento, e diferentes soluções podem ser trocadas rapidamente aspirando líquidos com um sistema de filtragem a vácuo e aplicando gotas de solução de uma só vez com uma pipeta multicanal. Essa diferença torna nosso protocolo altamente vantajoso quando um grande número de amostras precisa ser processado de uma só vez. Usando nosso protocolo, 12-48 amostras podem ser manuseadas simultaneamente e todo o procedimento FISH pode ser concluído dentro de ~8 horas, cerca de um tempo semelhante necessário para algumas amostras(Figura 2). Embora tenhamos usado como exemplo a expressão de lacZ em E. coli, o protocolo é amplamente aplicável a diferentes genes e espécies bacterianas com considerações discutidas abaixo.
Para diferentes genes, a primeira coisa a considerar são sondas smFISH. Pode-se projetar sondas oligonucleotídeos que ladrilho o mRNA de interesse(Figura 1A)13. Nesta abordagem da sonda "ladrilho", cada sonda tem ~20 de comprimento base e é rotulada com um fluoróforo no terminus de 5' ou 3'. Esta estratégia é conveniente, pois nenhuma manipulação genética é necessária. Alternativamente, uma repetição tandem de ~20 bp sequência, estranha à sequência genômica (por exemplo, uma matriz de sequência lacO em Caulobacter crescentus14), pode ser inserida na região não traduzida de um gene de interesse e uma única sonda complementar à unidade de repetição é usada para rotular a abordagem mRNA ("array"; Figura 1B). Em ambos os casos, vários fluoroforos decoram um mRNA, dando sinal de fluorescência amplificada que pode ser facilmente diferenciado de uma única sonda inespecificamente ligada dentro de uma célula.
Se escolher abordagens de "ladrilho" ou "matriz" depende do controle negativo, uma amostra onde a ligação inespecífica de sondas é testada porque não tem o mRNA alvo. Para sondas de ladrilho(Figura 1A), uma cepa mutante sem o gene de interesse ou uma condição, na qual o gene não é transcrito (por exemplo, a repressão da lacZ) pode servir como um controle negativo para testar a ligação inespecífica de sondas. Para o smFISH baseado em matriz (Figura 1B),uma cepa de tipo selvagem sem a matriz pode servir como um controle negativo porque não contém locais de vinculação para as sondas.
As condições ideais de hibridização podem depender das sequências da sonda e até mesmo da escolha de corantes fluoróforos. Otimizamos a condição de hibridização para conjuntos de sondas lacZ mantendo a temperatura de hibridização a 37 °C e testando diferentes concentrações de conjuntos de sondas e formamida na solução de hibridização. Maiores concentrações de formamida tendem a reduzir tanto a vinculação inespecífica quanto específica26,35. Recomendamos mudar sistematicamente a hibridização e suas condições de lavagem, mantendo o tempo e a temperatura da hibridização os mesmos. À medida que a condição se torna mais rigorosa, tanto a diminuição vinculante não específica quanto específica(Figura 6). É importante encontrar um ponto em que a vinculação inespecífica comece a bater abaixo de um limiar aceitável sem comprometer ainda mais a vinculação específica. Por exemplo, usamos o nível de sinal obtido sem quaisquer sondas ("sem sondas") como limiar(Figura 6).
O método smFISH de duas cores rotulando duas regiões separadas de um mRNA está limitado a genes longos. Para medir a taxa de alongamento da transcrição, aproveitamos o fato de que lacZ é longo (3075 bp) e sua expressão pode ser induzida pelo IPTG. Quando um gene é curto, é difícil projetar dois conjuntos de sondas de ladrilhos (perto de extremidades de 5' e 3') e resolver o atraso de tempo entre as aparências de 5' vs. 3' mRNA regiões. Neste caso, pode-se contar mRNAs nascentes em estado estável por smFISH e analisar sua distribuição com um modelo analítico que tem a taxa de alongamento de transcrição como parâmetrode montagem 20. Além disso, quando um gene de interesse não é indutor, pode-se tratar células com rifampicina no momento zero e medir a mudança temporal nas sub-regiões de 5' e 3' mRNA. O atraso da diminuição do sinal de 5' mRNA para o de 3' mRNA pode então ser usado para calcular a taxa de alongamento da transcrição como feito anteriormente31.
Finalmente, o protocolo smFISH é versátil e pode ser combinado com outros esquemas de rotulagem. Anteriormente, o lócus de DNA foi visualizado juntamente com mRNAs, combinando mRNA FISH com o DNA FISH14 ou o sistema fluorescente reporter-operator20. Os produtos proteicos podem ser visualizados pela realização de imunofluorescência juntamente com mRNA FISH14,36. Além disso, pode ser combinado com microscopia tridimensional de super-resolução37 para visualizar mRNAs nas três dimensões38,39.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Este protocolo foi desenvolvido pela S.K. durante sua pesquisa de pós-doutorado no laboratório da Dra. Agradecemos ao Dr. Jacobs-Wagner e seus membros do laboratório por várias entradas durante o desenvolvimento do método e Laura Troyer pela leitura crítica do manuscrito. S.K. reconhece apoio do Programa De Estudiosos searle; K.V. reconhece o apoio do James Scholar Preble Research Award da Universidade de Illinois.
Materiais
| Name | Company | Catalog Number | Comments |
| Bacterial strain | |||
| Escherichia coli MG1655 | |||
| Chemicals, peptides, and others | |||
| Acetonitrile | Sigma-Aldrich | 34851 | UPLC buffer B |
| Ammonium chloride | Fisher Chemical | A661-500 | To make M9 medium |
| Bovine serum albumin (BSA) | Sigma-Aldrich | B2518 | Probe hybridization |
| Calcium chloride | Acros Organics | 349610250 | To make M9 medium |
| Casamino acid | BD Difco | 223050 | To make M9 medium |
| Cy3B NHS ester | GE Healthcare Life Sciences | PA63101 | Fluorophore for FISH probes |
| Cy5 NHS ester | GE Healthcare Life Sciences | PA15101 | Fluorophore for FISH probes |
| DEPC | Sigma-Aldrich | D5758 | |
| Dextran sulfate | Millipore | S4030 | Probe hybridization |
| Dextrose | Fisher Chemical | D16 | To repress the expression of lacZ |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | To dissolve fluorophores |
| E. coli tRNA | Sigma-Aldrich | R1753 | Probe hybridization |
| Ethanol | Decon Laboratories | 2701 | Used in DNA purification, lysis, and cleaning coverslips |
| FISH probes | Biosearch Technologies | Sequences are published in ref#16 | |
| Formaldehyde | Ladd Research Industries | 20295 | Fixation |
| Formamide | American Bio | AB00600 | Probe hybridization, pre-hybridization, and wash |
| Glycerol | Americanbio | AB00751-01000 | To make M9 medium |
| Isopropylthio-β-galactoside (IPTG) | Invitrogen | 15529019 | lacZ induction |
| Magnesium sulfate | Fisher Chemical | M65-500 | To make M9 medium |
| 2-Nitrophenyl β-D-fucopyranoside (ONPF) | Santa Cruz Biotechnology | sc-216258 | lacZ repression |
| Picodent twinsil 22 | Picodent | 1300 1000 | Sealant |
| Poly-L-lysine | Sigma-Aldrich | P8920 | To treat the coverslip surface |
| Potassium chloride | Fisher BioReagents | BP366-500 | To make PBS |
| Potassium phosphate monobasic | Fisher BioReagents | BP362-500 | To make PBS and M9 medium |
| Rifampicin | Sigma-Aldrich | R3501 | To stop transcription initiation |
| Saline-sodium citrate buffer (SSC) | Invitrogen | AM9763 | Probe hybridization, pre-hybridization, and wash |
| sodium bicarbonate | Fisher BioReagents | BP328-500 | Fluorophore-probe conjugation |
| Sodium chloride | Fisher BioReagents | BP358-1 | For DNA purification, PBS and M9 medium |
| Sodium phosphate dibasic | Fisher BioReagents | BP332-500 | To make PBS and M9 medium |
| Sodium phosphate monobasic | Fisher BioReagents | BP329-500 | To make a sodium phosphate buffer |
| Super PAP Pen | Invitrogen | 8899 | Hydrophobic marker for coverslips |
| TetraSpek microspheres | Invitrogen | T7280 | Controls for multi-channel registration |
| Thiamine | Sigma-Aldrich | T1270 | To make M9 medium |
| Triethylammonium acetate | Sigma-Aldrich | 90358 | UPLC buffer A |
| Vanadyl ribonucleoside complex (VRC) | Sigma-Aldrich | 94742 | Probe hybridization and pre-hybridization |
| Equipment | |||
| C18 column | Waters | Acquity BEH C18 column | |
| Countertop centrifuge | Eppendorf | 5425 | |
| Countertop incubator | Eppendorf | Thermomixer F1.5 | |
| Incubator (Oven) | Thermo Scientific | 51030514 | Gravity convection |
| Water purification system | Millipore | Milli-Q Reference | |
| Nanodrop | Thermo Scientific | 2000C | |
| Nitrogen gas | Building | For blow-drying coverslips and glass slides | |
| UPLC | Waters | Acquity UPLC system | |
| Vacuum and aspirator | Building | Aspirator is made of a filtration flask with a side arm. | |
| Vacuum concentrator | Labconco | 7810010 | Centrivap; to dry samples collected from UPLC. |
| Vortexer | Scientific Industries | Genie-2 SI-0236 | |
| Water bath shaker | New Brunswick | Innova 3100 | Critical for time-course experiments |
| Water bath sonicator | VWR | 97043-960 | To clean coverslips and glass slides |
| Tools | |||
| 1.5-mL tubes | Eppendorf | 22431021 | DNA lobind tubes |
| 1000-uL pipette tip box | Denville Scientific | P1126 | An empty box after using all the tips |
| Coplin jar | SPI | 01240-AB | To clean coverslips and glass slides |
| Coverslip | Fisher Scientific | 22-050-230 | 24x60 No1 |
| Filtered pipette tips | Denville Scientific | P1121,P1122,P1126 | SHARP® Precision Barrier Tips |
| Forceps | SPI | K35a | To handle clean coverslips and glass slides |
| Glass slide | Fisher Scientific | 12-544-1 | |
| Gloves | Microflex | MK-296-M | |
| Multichannel pipetter | Eppendorf | 2231300045 | To use in the washing step (#7) |
| Pipette | Gilson | P1000, P200, P20 | |
| Reagent reservoir | MTC Bio | P8025-1S | To use in the washing step (#7) |
| Syringe filter (0.22 um) | Millipore | SLGS033SS | |
| Timer | VWR | 62344-641 | |
| Software and algorithms | |||
| MATLAB | Mathworks | R2013 and up | https://www.mathworks.com |
| MicrobeTracker or Oufti | https://www.github.com/JacobsWagnerLab/MicrobeTracker | ||
| https://oufti.org/ | |||
| Stellaris Probe Designer | Biosearch Technologies | https://www.biosearchtech.com/support/tools/design-software/stellaris-probe-designer | |
| Microscope | |||
| CCD camera | Hamamatsu Photonics | Orca-II-ER | |
| Cy3 filter set | Chroma | 49004 | |
| Cy5 filter set | Chroma | 49006 | |
| Epi-fluorescence microscope | Nikon | Eclipse Ti | For phase-contrast and epi fluorescence |
| Fluorescence excitation source | Lumencor | SOLA-E | |
| Nikon Elements software | Nikon | software that controls the microscope setup | |
| Phase-contrast 100x objective | Nikon | Plan Apochromat (NA 1.45) | |
| Probe sequence | |||
| DNA oligos with C6 amino modification at the 5' end | Biosearch Technologies Inc | ||
| lacZ1 | GTGAATCCGTAATCATGGTC | 5' mRNA | |
| lacZ2 | TCACGACGTTGTAAAACGAC | 5' mRNA | |
| lacZ3 | ATTAAGTTGGGTAACGCCAG | 5' mRNA | |
| lacZ4 | TATTACGCCAGCTGGCGAAA | 5' mRNA | |
| lacZ5 | ATTCAGGCTGCGCAACTGTT | 5' mRNA | |
| lacZ6 | AAACCAGGCAAAGCGCCATT | 5' mRNA | |
| lacZ7 | AGTATCGGCCTCAGGAAGAT | 5' mRNA | |
| lacZ8 | AACCGTGCATCTGCCAGTTT | 5' mRNA | |
| lacZ9 | TAGGTCACGTTGGTGTAGAT | 5' mRNA | |
| lacZ10 | AATGTGAGCGAGTAACAACC | 5' mRNA | |
| lacZ11 | GTAGCCAGCTTTCATCAACA | 5' mRNA | |
| lacZ12 | AATAATTCGCGTCTGGCCTT | 5' mRNA | |
| lacZ13 | AGATGAAACGCCGAGTTAAC | 5' mRNA | |
| lacZ14 | AATTCAGACGGCAAACGACT | 5' mRNA | |
| lacZ15 | TTTCTCCGGCGCGTAAAAAT | 5' mRNA | |
| lacZ16 | ATCTTCCAGATAACTGCCGT | 5' mRNA | |
| lacZ17 | AACGAGACGTCACGGAAAAT | 5' mRNA | |
| lacZ18 | GCTGATTTGTGTAGTCGGTT | 5' mRNA | |
| lacZ19 | TTAAAGCGAGTGGCAACATG | 5' mRNA | |
| lacZ20 | AACTGTTACCCGTAGGTAGT | 5' mRNA | |
| lacZ21 | ATAATTTCACCGCCGAAAGG | 5' mRNA | |
| lacZ22 | TTTCGACGTTCAGACGTAGT | 5' mRNA | |
| lacZ23 | ATAGAGATTCGGGATTTCGG | 5' mRNA | |
| lacZ24 | TTCTGCTTCAATCAGCGTGC | 5' mRNA | |
| lacZ25 | ACCATTTTCAATCCGCACCT | ||
| lacZ26 | TTAACGCCTCGAATCAGCAA | ||
| lacZ27 | ATGCAGAGGATGATGCTCGT | ||
| lacZ28 | TCTGCTCATCCATGACCTGA | ||
| lacZ29 | TTCATCAGCAGGATATCCTG | ||
| lacZ30 | CACGGCGTTAAAGTTGTTCT | ||
| lacZ31 | TGGTTCGGATAATGCGAACA | ||
| lacZ32 | TTCATCCACCACATACAGGC | ||
| lacZ33 | TGCCGTGGGTTTCAATATTG | ||
| lacZ34 | ATCGGTCAGACGATTCATTG | ||
| lacZ35 | TGATCACACTCGGGTGATTA | ||
| lacZ36 | ATACAGCGCGTCGTGATTAG | ||
| lacZ37 | GATCGACAGATTTGATCCAG | ||
| lacZ38 | AAATAATATCGGTGGCCGTG | ||
| lacZ39 | TTTGATGGACCATTTCGGCA | ||
| lacZ40 | TATTCGCAAAGGATCAGCGG | ||
| lacZ41 | AAGACTGTTACCCATCGCGT | ||
| lacZ42 | TGCCAGTATTTAGCGAAACC | ||
| lacZ43 | AAACGGGGATACTGACGAAA | ||
| lacZ44 | TAATCAGCGACTGATCCACC | ||
| lacZ45 | GGGTTGCCGTTTTCATCATA | ||
| lacZ46 | TCGGCGTATCGCCAAAATCA | ||
| lacZ47 | TTCATACAGAACTGGCGATC | ||
| lacZ48 | TGGTGTTTTGCTTCCGTCAG | ||
| lacZ49 | ACGGAACTGGAAAAACTGCT | 3' mRNA | |
| lacZ50 | TATTCGCTGGTCACTTCGAT | 3' mRNA | |
| lacZ51 | GTTATCGCTATGACGGAACA | 3' mRNA | |
| lacZ52 | TTTACCTTGTGGAGCGACAT | 3' mRNA | |
| lacZ53 | GTTCAGGCAGTTCAATCAAC | 3' mRNA | |
| lacZ54 | TTGCACTACGCGTACTGTGA | 3' mRNA | |
| lacZ55 | AGCGTCACACTGAGGTTTTC | 3' mRNA | |
| lacZ56 | ATTTCGCTGGTGGTCAGATG | 3' mRNA | |
| lacZ57 | ACCCAGCTCGATGCAAAAAT | 3' mRNA | |
| lacZ58 | CGGTTAAATTGCCAACGCTT | 3' mRNA | |
| lacZ59 | CTGTGAAAGAAAGCCTGACT | 3' mRNA | |
| lacZ60 | GGCGTCAGCAGTTGTTTTTT | 3' mRNA | |
| lacZ61 | TACGCCAATGTCGTTATCCA | 3' mRNA | |
| lacZ62 | TAAGGTTTTCCCCTGATGCT | 3' mRNA | |
| lacZ63 | ATCAATCCGGTAGGTTTTCC | 3' mRNA | |
| lacZ64 | GTAATCGCCATTTGACCACT | 3' mRNA | |
| lacZ65 | AGTTTTCTTGCGGCCCTAAT | 3' mRNA | |
| lacZ66 | ATGTCTGACAATGGCAGATC | 3' mRNA | |
| lacZ67 | ATAATTCAATTCGCGCGTCC | 3' mRNA | |
| lacZ68 | TGATGTTGAACTGGAAGTCG | 3' mRNA | |
| lacZ69 | TCAGTTGCTGTTGACTGTAG | 3' mRNA | |
| lacZ70 | ATTCAGCCATGTGCCTTCTT | 3' mRNA | |
| lacZ71 | AATCCCCATATGGAAACCGT | 3' mRNA | |
| lacZ72 | AGACCAACTGGTAATGGTAG | 3' mRNA | |
Referências
- Bervoets, I., Charlier, D. Diversity, versatility and complexity of bacterial gene regulation mechanisms: opportunities and drawbacks for applications in synthetic biology. FEMS Microbiology Reviews. 43 (3), 304-339 (2019).
- Epshtein, V., Nudler, E. Cooperation between RNA polymerase molecules in transcription elongation. Science. 300 (5620), 801-805 (2003).
- Vogel, U., Jensen, K. F. The RNA chain elongation rate in Escherichia coli depends on the growth rate. Journal of Bacteriology. 176 (10), 2807-2813 (1994).
- Tennyson, C. N., Klamut, H. J., Worton, R. G. The human dystrophin gene requires 16 hours to be transcribed and is cotranscriptionally spliced. Nature Genetics. 9, 184 (1995).
- Singh, J., Padgett, R. A. Rates of in situ transcription and splicing in large human genes. Nature Structural & Molecular Biology. 16, 1128 (2009).
- Selinger, D. W., Saxena, R. M., Cheung, K. J., Church, G. M., Rosenow, C. Global RNA Half-Life Analysis in Escherichia coli Reveals Positional Patterns of Transcript Degradation. Genome Research. 13 (2), 216-223 (2003).
- Bernstein, J. A., Khodursky, A. B., Lin, P. -. H., Lin-Chao, S., Cohen, S. N. Global analysis of mRNA decay and abundance in Escherichia coli at single-gene resolution using two-color fluorescent DNA microarrays. Proceedings of the National Academy of Sciences. 99 (15), 9697-9702 (2002).
- Pérez-Ortín, J. E., Medina, D. A., Chávez, S., Moreno, J. What do you mean by transcription rate. BioEssays. 35 (12), 1056-1062 (2013).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6 (5), 377-382 (2009).
- Kuchina, A., et al. Microbial single-cell RNA sequencing by split-pool barcoding. BioRxiv. , 869248 (2019).
- Blattman, S. B., Jiang, W., Oikonomou, P., Tavazoie, S. Prokaryotic single-cell RNA sequencing by in situ combinatorial indexing. Nature Microbiology. , (2020).
- Femino, A., Fay, F., Fogarty, K., Singer, R. Visualization of single RNA transcripts in situ. Science. 280 (5363), 585-590 (1998).
- Raj, A., vanden Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877-879 (2008).
- Montero Llopis, P., et al. Spatial organization of the flow of genetic information in bacteria. Nature. 466 (7302), 77-81 (2010).
- So, L. -. h., et al. General properties of transcriptional time series in Escherichia coli. Nature Genetics. 43 (6), 554-560 (2011).
- Taniguchi, Y., et al. Quantifying E. coli proteome and transcriptome with single-molecule sensitivity in single cells. Science. 329 (5991), 533-538 (2010).
- Jones, D. L., Brewster, R. C., Phillips, R. Promoter architecture dictates cell-to-cell variability in gene expression. Science. 346 (6216), 1533-1536 (2014).
- Iyer, S., Park, B. R., Kim, M. Absolute quantitative measurement of transcriptional kinetic parameters in vivo. Nucleic Acids Research. 44 (18), 142 (2016).
- Kim, S., Beltran, B., Irnov, I., Jacobs-Wagner, C. Long-Distance Cooperative and Antagonistic RNA Polymerase Dynamics via DNA Supercoiling. Cell. 179 (1), 106-119 (2019).
- Wang, M., Zhang, J., Xu, H., Golding, I. Measuring transcription at a single gene copy reveals hidden drivers of bacterial individuality. Nature Microbiology. 4 (12), 2118-2127 (2019).
- Joo, C., Ha, T. . Labeling DNA (or RNA) for single-molecule FRET. 2012 (9), 1005-1008 (2012).
- Sambrook, J., Russell, D. W. . Standard Ethanol Precipitation of DNA in Microcentrifuge Tubes. 2006 (1), 4456 (2006).
- Skinner, S. O., Sepúlveda, L. A., Xu, H., Golding, I. Measuring mRNA copy number in individual Escherichia coli cells using single-molecule fluorescent in situ hybridization. Nature Protocols. 8 (6), 1100-1113 (2013).
- Adesnik, M., Levinthal, C. The synthesis and degradation of lactose operon messenger RNA in E. coli. Cold Spring Harbor Symposia on Quantitative Biology. 35, 451-459 (1970).
- Campbell, E. A., et al. Structural mechanism for rifampicin inhibition of bacterial RNA polymerase. Cell. 104 (6), 901-912 (2001).
- Raj, A., Tyagi, S., Walter, N. G. . Methods in Enzymology. 472, 365-386 (2010).
- Sliusarenko, O., Heinritz, J., Emonet, T., Jacobs-Wagner, C. High-throughput, subpixel precision analysis of bacterial morphogenesis and intracellular spatio-temporal dynamics. Molecular Microbiology. 80 (3), 612-627 (2011).
- Paintdakhi, A., et al. Oufti: an integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Molecular Microbiology. 99 (4), 767-777 (2016).
- Moffitt, J. R., Zhuang, X., Filonov, G. S., Jaffrey, S. R. . Methods in Enzymology. 572, 1-49 (2016).
- Yu, J., Xiao, J., Ren, X., Lao, K., Xie, X. S. Probing gene expression in live cells, one protein molecule at a time. Science. 311 (5767), 1600-1603 (2006).
- Chen, H., Shiroguchi, K., Ge, H., Xie, X. S. Genome-wide study of mRNA degradation and transcript elongation in Escherichia coli. Molecular Systems Biology. 11 (1), 781 (2015).
- Vogel, U., Sørensen, M., Pedersen, S., Jensen, K. F., Kilstrup, M. Decreasing transcription elongation rate in Escherichia Coli exposed to amino acid starvation. Molecular Microbiology. 6 (15), 2191-2200 (1992).
- Yang, S., et al. Transcription and translation contribute to gene locus relocation to the nucleoid periphery in E. coli. Nature Communications. 10 (1), 5131 (2019).
- Zenklusen, D., Larson, D. R., Singer, R. H. Single-RNA counting reveals alternative modes of gene expression in yeast. Nature Structural & Molecular Biology. 15 (12), 1263-1271 (2008).
- Fontenete, S., Guimarães, N., Wengel, J., Azevedo, N. F. Prediction of melting temperatures in fluorescence in situ hybridization (FISH) procedures using thermodynamic models. Critical Reviews in Biotechnology. 36 (3), 566-577 (2016).
- Sepúlveda, L. A., Xu, H., Zhang, J., Wang, M., Golding, I. Measurement of gene regulation in individual cells reveals rapid switching between promoter states. Science. 351 (6278), 1218-1222 (2016).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-Dimensional Super-Resolution Imaging by Stochastic Optical Reconstruction Microscopy. Science. 319 (5864), 810-813 (2008).
- Moffitt, J. R., Pandey, S., Boettiger, A. N., Wang, S., Zhuang, X. Spatial organization shapes the turnover of a bacterial transcriptome. eLife. 5, 13065 (2016).
- Fei, J., et al. Determination of in vivo target search kinetics of regulatory noncoding RNA. Science. 347 (6228), 1371-1374 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados