Method Article
Situ Hibridizasyonda İki Renkli Tek Moleküllü Floresan Kullanarak Escherichia coli'de Uzay ve Zamanda MRNA KinetikLerini Sondalama
Bu Makalede
Özet
Bu protokol, mRNA sentezi ve bozulmasının in vivo kinetiklerini ölçmek için yerinde hibridizasyonda (smFISH) tek moleküllü floresan uygulamasını açıklar.
Özet
Yerinde hibridizasyonda tek moleküllü floresan (smFISH) tek tek hücrelerdeki mutlak mRNA sayısını saymaya olanak sağlar. Burada, Escherichia colitranskripsiyon ve mRNA bozulma oranlarını ölçmek için smFISH bir uygulama açıklar. smFISH sabit hücrelere dayandığı için, bir zaman dersi deneyi sırasında, yani hücrelerin gen ekspresyonunun indüksiyonu veya baskısı üzerine senkronize değişiklikler emildiğinde birden fazla zaman noktasında smFISH gerçekleştiriyoruz. Her zaman noktasında, bir mRNA'nın alt bölgeleri transkripsiyon uzamasını ve erken sonlandırmayı araştırmak için ayırt edilir. Bu protokolün sonucu aynı zamanda hücreler arasında mRNA kopya sayılarında mRNA'ların hücre içi lokalizasyonu ve heterojeniteanalizine de olanak sağlar. Bu protokol kullanılarak birçok örnek (~50) sadece birkaç örnek için gereken süre gibi 8 saat içinde işlenebilir. Bakteri hücrelerindeki farklı mRNA'ların transkripsiyon ve bozunma kinetiğini incelemek için bu protokolün nasıl uygulanacağı tartışılmaktadır.
Giriş
DNA'dan mRNA ve proteine genetik bilgi akışı, regülasyonu hücresel fitness1için önemli olan en temel hücresel süreçlerden biridir. Hücredeki mRNA sayısı transkripsiyon ve mRNA bozulması olmak üzere iki dinamik süreçle belirlenir. Ancak, transkripsiyon ve mRNA bozulmasının tek bir hücrenin zaman ve uzayda nasıl düzenlendiği, büyük ölçüde in vivo daki kinetiklerini nicel olarak ölçmek için deneysel yöntemlerin yetersizliğinden dolayı tam olarak anlaşılamamıştır.
Kuzey leke, RT-PCR, RNA dizilemesi ve gen ekspresyonu mikrodizileri gibi bir hücre popülasyonundan elde edilen toplam mRNA'lara dayalı yöntemler, mRNA düzeylerindeki bağıl farkı ölçebilir ve transkripsiyonuzamaoranını 2,3,4,5 veya mRNA bozulma oranı6,7olarak analiz etmek için yaygın olarak kullanılmıştır. Ancak, hücre başına mRNA mutlak sayısını sağlamaz ve bu nedenle, transkripsiyon başlatma oranını araştırmak için uygun değildir8. Ayrıca, mRNA'lar bir hücre popülasyonundan çıkarıldığı için, mRNA'ların tek bir hücre içindeki mekansal dağılımı ve mRNA kopya numaralarının hücreler arasındaki değişkenliği ölçülemememaktadır.
Tek tek hücrelerde yeni nesil RNA dizilimi (scRNAseq) genomik ölçekte hücre başına mRNA sayısını ölçebilir9. Ancak, örnek hazırlama ve yüksek maliyet ile ilgili zorluklar nedeniyle transkripsiyon kinetik ölçmek için bu tekniği kullanmak zor olmaya devam etmektedir. Özellikle, bakterilere scRNaAseq uygulaması düşük mRNA bolluğu nedeniyle teknik olarak zor olmuştur10,11.
Yerinde hibridizasyonda tek moleküllü floresan (smFISH) floresan etiketli tek iplikli probların melezleştirilmesine dayanır ve dizileri ilgialanının hedef mRNA'sına tamamlayıcıdır12,13. Diziye özgü hibridizasyon kavramı Kuzey blot veya RT-PCR kullanılan benzer, ancak melezleme sabit hücreler içinde yerinde yapılır, mRNA'ların yerel yerelleştirme korumak için. Tek bir mRNA'nın sinyali, bir mRNA'nın farklı kısımlarına hibridize edilebilen, ~20 nükleotit (nt) uzunluğunda birçok prob kullanılarak yükseltilir (Şekil 1A)13. Bu "döşeme" sondası yaklaşımında, tek bir mRNA'yı algılamak için gereken prob sayısı, tazyiklebilen mRNA uzunluğuna daha düşük bir sınır belirler. Alternatif olarak, ilgi mRNA transkripsiyonel tandem Lac operatör dizileri olmayan bir kodlama dizi erimiş olabilir, bir floresan etiketli lacO prob birden fazla kopya tek bir mRNA hibridize gibi (Şekil 1B)14.
smFISH sabit durumda hücre başına mRNA sayısını ölçmek için kullanılmıştır (yani, sentez ve çürüme dengede olduğunda) ve bakteri hücreleri arasında mRNA ortalama ve değişkenlik analiz etmek için15,16,17. Son zamanlarda, smFISH sabit olmayan durumda mRNA numaraları ölçmek için uygulanmıştır, sağ e. coligen ekspresyonu indüksiyon veya baskı sonra18,19,20. Daha sonra mutlak mRNA kopya numaralarındaki zamansal değişiklikler transkripsiyon başlatma, uzama ve sonlandırma oranının yanı sıra mRNA bozulma oranını hesaplamak için kullanıldı. Bu uygulama için, geleneksel smFISH prosedürleri hantal olabilir, çünkü her biri bir zaman noktasını temsil eden, birden fazla arabellek değişim adımlarından geçmesi gereken (yani santrifüj ve yıkama) birçok örnek vardır. Burada, örnek işleme adımlarının, hücrelerin bir kapak kayması yüzeyine yapıştırılarak ve vakum filtrasyon sistemi 14,19ile sıvıları azarlayarak önemli ölçüde basitleştirildiği bir smFISHprotokolünütanımlıyoruz. E. coli'deki lacZ ifadesini örnek olarak kullanarak, transkripsiyon (inisiyasyon, uzama ve sonlandırma) in vivo kinetiği (inisiyasyon, uzama ve sonlandırma) ve mRNA bozunması, mRNA ekspresyonunda hücreden hücreye değişkenlik ve mRNA lokalizasyonu içeren görüntü analizi(Şekil 3)dahil olmak üzere, tam iş akışı(Şekil 2)gösterilmiştir. Protokolün, çeşitli bakteri türlerinde in vivo kinetik ve diğer mRNA'ların lokalizasyonu için yaygın olarak uygulanacağını tahmin ediyoruz.
Protokol
1. SMFISH problarının hazırlanması
NOT: SMFISH problarını tek bir florofor ile etiketlemek için, NHS ester kimyası21'edayalı nükleik asit oligonükleotidlerin etiketletilmesi için standart bir protokol izleyin.
- Tasarım smFISH probları. İlgi geni için "fayans" probları mı yoksa "dizi" probları mı(Şekil 1)kullanacağınıza karar verin. Kararın nasıl verilebildiğini anlatan Tartışma bölümüne bakın.
- "Döşeme" probları için(Şekil 1A),çevrimiçi bir sonda tasarımcısı aracı kullanın (örn. Malzeme Tablosu'nabakın).
- "Dizi" sondaları(Şekil 1B)için, sonda sırasının diğer mRNA dizilerine tamamlayıcı olmadığından emin olmak için blast sırasını gerçekleştirin.
- lacZ mRNA transkripsiyon ve bozulma kinetik çalışma için, 24 problar, her bir set lacZ (3.072 bp)19ilk ve son 1 kb bölgeleri kapsayan iki set kullanın.
NOT: Bu sonda setleri, bundan sonra sırasıyla "5' mRNA sondası" ve "3' mRNA sondası" olarak adlandırılır. Bu probların dizileri Malzeme Tablosu'ndalistelenmiştir.
- 5' sonunda bir C6 amino bağlayıcı ile DNA oligonükleotidolarak Sipariş sonda dizileri. Suda ayrı ayrı probları 1 mM'ye çözün.
- "5' mRNA prob" ve "3' mRNA prob" kümeleri için equimolar miktarda probları birleştirin. Örneğin, lacZiçin ayarlanan 5' mRNA prob için, her probun 20 μL'sini (kümedeki toplam 24 prob türü) birleştirir.
- Konjugasyon reaksiyonunu inhibe eden primer ve sekonder aminlerin (Tris, glisin ve amonyum tuzları gibi) kontaminasyonunu gidermek için kombine probların22'sini etanol çökeltme gerçekleştirin. Sonunda, DNA peletini 100 μL suda çözün (bir sonda setinde ~4,5 mM DNA elde edin).
NOT: Problar üretici tarafından standart tuzdan arındırma geçirmiş olsa bile bu adım önerilir. Standart filtre tabanlı arınma, etanol yağışının yerine ve ek olarak çalışabilir. - Tek fonksiyonlu NHS ester moiety ile iki spectrally farklı floropores seçin, böyle 5' ve 3' mRNA prob setleri farklı olarak etiketlenebilir. Örneğin, Cy5 NHS esterini 5' mRNA probları için, Cy3B NHS esterini ise 3' mRNA probları için hazırlayın. Susuz DMSO'daki her bir florofor türünü 20 mg/mL 'ye (~25 mM) kadar eritin.
- Her etiketleme reaksiyonundan hemen önce 0,1 M sodyum bikarbonat (pH 8,5) hazırlayın. Uzun süre havaya maruz kalmak pH'ını düşürür ve etiketleme verimliliğini azaltır.
- Konjugasyon reaksiyonu için aşağıdakileri birleştirin: Cy5 florofor stokunun 15 0L'si (Adım 1,5'ten), 4'l 5' mRNA prob seti (Adım 1.4'ten), 75 μL sodyum bikarbonat (Adım 1.6'dan itibaren) ve 7°L su. Alüminyum folyo ile tüp sarın ve 3-6 saat oda sıcaklığında sallayın.
NOT: Daha uzun kuluçka mutlaka daha fazla etiketleme verimliliği neden olmaz. Ayrıca, bileşenlerin konsantrasyonları korunursa reaksiyon yukarı veya aşağı ölçeklendirilebilir. - 3' mRNA prob seti ve buna karşılık gelen florofor (yani Cy3B NHS-ester) için yukarıdaki adımı tekrarlayın.
- Reaksiyona girilmemiş boya moleküllerini çıkarmak için etanol çökeltisi22 yapın. Pelleti ~ 50 μL TE tampon (10mM Tris-HCl pH 8.0 1mM EDTA ile) eritin.
- UV-Vis spektrometresi kullanarak DNA ve florofor konsantrasyonlarını tahmin edin.
- Emiciliği 260 nm ve 559 nm (Cy3B) veya 649 nm (Cy5) olarak ölçün. Numune doğru bir ölçüm elde edemeyecek kadar konsantre yse, numunenin 1 00L'sini 10 μL'ye seyreltin.
- Emiciliği konsantrasyona dönüştürün:
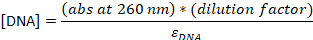
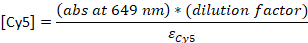
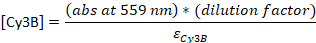
εDNA = 0.2 μM-1 (20-nt tek iplikçikli DNA için), εCy5 = 0.25 μM-1ve εCy3B = 0.13 μM-1
NOT: [DNA] çözelti içinde toplam probların konsantrasyonudur. Bireysel probların konsantrasyonu yaklaşık 24 kat daha düşüktür. Toplam probların konsantrasyonu bu noktadan itibaren "sonda konsantrasyonları" olarak kullanılacaktır. [DNA] ve [boya] arasındaki oran 1 ise, aşağıdaki HPLC adımı23atlanabilir ve örnek TE tamponunda son 4-5 μM'ye seyreltilmelidir.
- (Tavsiye edilir) Etiketli probları etiketlenmemiş problardan ve serbest boyalardan HPLC kullanarak arındırın.
NOT: Bu ek arıtma adımı numune kaybına yol açsa da, akış aşağı uygulamaları için yararlıdır. Etiketlenmemiş DNA sondalarının çıkarılması mRNA hedeflerinden gelen floresan sinyalini artıracak ve tepkisiz boyaların çıkarılması arka plan floresansını azaltacaktır.- Standart analitik C18 sütunu ile HPLC hazırlayın, tampon A olarak 0.1 M triethylamonnium asetat (TEAA), tampon B olarak asetonitril.
- 0,1 M TEAA yapmak için örneğe (Adım 1,9'dan) 1 M TEAA ekleyin.
- Degrade programını aşağıdaki gibi ayarlayın: 0-5 dk %0 B, 5-35 dk %0-30 doğrusal degrade b, 35-37 dk %30-100 doğrusal degradeli B ve 37-40 dk %0 B. Akış hızını 0,1 mL/dk'da tutun ve 260 ve 649 nm (Cy5 etiketli numuneler için) veya 260 ve 559 nm (Cy3B etiketli numuneler için) de kromatogramları kaydedin.
- Hem DNA hem de florofor kanallarında absorbans arttığında eluted örnek toplamak.
- Eluted numuneyi vakum konsantratörü kullanarak yoğunlaştırın ve 50-100 μL TE tamponunda peleti yeniden askıya alın.
- UV-Vis spektrometresi kullanarak DNA ve florofor konsantrasyonunun kontrol edin (Bkz. Adım 1.10). Seyreltik, gerekirse, 4-5 μM civarında son konsantrasyon yapmak için.
2. Çözümlerin hazırlanması
- Depc ile işlenmiş su ve tamponlar büyük hacimli hazırlayın(Tablo 1). Bu çözümler oda sıcaklığında bir yıldan fazla sürebilir.
- 4x sabitleme çözeltisi ve yıkama çözeltisi hazırlayın (Tablo 1).
- Ön hibridizasyon çözümve prob hibridizasyon çözeltisi(Tablo 1)hazırlayın. Adım 5.1 veya Step 6.1'de kuluçka sırasında prob hibridizasyon çözeltisini hazırlayın ve ışığa maruz kalmamayı en aza indirmek için çözeltiyi 20-40 dakika boyunca 37 °C'lik bir tezgah sallayıcıda saklayın.
NOT: Formamid, SSC ve probu konsantrasyonları, gerçek sinyali maksimize ederken arka plan floresansını en aza indirmek için lacZ prob setleri için optimize edildi. Bu konsantrasyonların farklı uygulamalar için nasıl değiştirilebildiğini hakkında ayrıntılar için Tartışma bölümüne bakın.
3. Kapak ve cam kaydırakların hazırlanması
- Kapakları ve cam slaytları temizleyin.
- Tek tek kapakları ve slaytları bir Coplin kavanozuna forceps kullanarak yerleştirin. Kapak ve slaytların birbirinden ayrıldığından ve birbirine dokunmadığından emin olun.
- Kavanozu %100 etanolile doldurun ve kapağıkapatın. Bir su banyosu ultrasonik temizleyici ve sonicate 15-20 dakika için kavanoz yerleştirin.
NOT: Su banyosu sonicator için ısıtıcı işlevini kapatmak için tavsiye edilir. - Etanol dökün ve ultra saf su ile yıkayın 3-4x. Doğrudan su arıtma makinesinden akan suyu kullanın.
- Kavanozdan su dökün ve% 70 etanol ile doldurun. Kapağıkapatın ve 15-20 dakika sonication gerçekleştirmek ve ultrasaf su ile yıkayın.
- 15-20 dakika ultrasaf su ve sonicate ile kavanoz doldurun.
NOT: Kapaklar ve cam kaydıralar bir gecede ultra saf su ile dolu Coplin kavanozunda saklanabilir. - Coplin kavanozundan temiz çifenekler kullanarak bir kaydırak veya kapak çıktın ve N2 gazı kullanarak üfleyin. Kalan slaytlar ve kapaklar için bunu tekrarlayın.
- Kurutulmuş slaytları Adım 7.5'te kullanıma kadar temiz bir saklama kutusuna yerleştirin. Kurutulmuş kapakları, kalan işlemde "hazne" görevi gören 1.000 μL'lik boş bir pipet ucu kutusuna yerleştirin.
- Hidrofobik bir marker kullanarak, pipet ucu kutusundaki dairesel delikleri takiben kapaklara daireler çizin. Bu daireler (çapı ~ 0.5 cm) "kuyu" olarak hizmet edecektir. Markertamamen kurutulması için en az 5-10 dakika bekleyin.
NOT: Bahşiş kutusunun kapağını her zaman kapalı tutun. - Her kuyuya %0,1 poli-L-l-lizin 20-l damla uygulayın. Oda sıcaklığında 10-50 dakika kuluçka.
NOT: Bu hacmi kuyu büyüklüğüne göre ayarlayın. Çözümün kuyu alanını tamamen kapsadığından emin olun. Daha uzun kuluçka için, buharlaşma önlemek için dikkatli olun. - Kuluçkadan sonra yüzeye dokunmadan poli-L-lizin aspirasyon poli-L-lizin kazıyacak gibi. Daha sonra poli-L-lizin işlenmiş kuyulara bir damla (~20°L) DEPC suyu uygulayın. Adım 5.1 kadar buharlaşmayı önlemek için "hazne" kapağını kapatın.
4. Zaman-ders deneyi ve örnek fiksasyonu
- 250 mL'lik bir şişede ~20 mL sıvı kültüründe E. coli hücrelerini büyütün. Şişeyi su banyosu shaker'da (30 °C) tutun ve sallamaya devam edin. Sadece numune alırken çalkalayıcıyı durdurun.
NOT: Bu yazıda sunulan sonuçlar MG1655 hücreleri M9 minimal ortamda% 0.2 gliserol ile takviye olarak yetiştirilen elde edilir, 0.1% casamino asitler, ve 1 mg / L tiamin üstel büyüme fazı (OD600~ 0.2). - Boş bir 1,5 mL tüpe 4x sabitleme çözeltisinin 250 μL'sini ekleyin. Tekrarlayın ve alınacak zaman noktaları kadar birden fazla tüp hazırlayın. Tüpleri zaman noktası numaralarıyla etiketleyin ve oda sıcaklığında tutun.
- Bir zaman kursu deneyine başlamadan önce 750 μL hücre kültürü (OD600~0,2) alın. Kültürü "sıfır zaman" (Adım 4.2'den) için işaretlenmiş bir tüpe ekleyin. Hücreleri sabitleme solüsyonuyla karıştırmak için tüpü nazikçe ters çevirin.
NOT: Hücrelerde karıştırmak, girdap lamak veya "kaba olmak" için pipetleri yukarı ve aşağı yapmayın. Bu örnek bastırılmış durumu temsil eder ve tek bir mRNA'nın floresan yoğunluğunu hesaplamak için denetim olarak kullanılır (Bkz. Adım 9.4). - LacZ ekspresyonu indüklemek için sıvı kültürüne 0,02-1 mM izopropil β-D-1-thiogalactopyranoside (IPTG) ekleyin. Bu noktadabir zamanlayıcı başlatın (t = 0 dk) ve belirli bir zaman aralığında (örneğin, her 1 dakika) örnek o andan itibaren. Örnekleme için Adım 4.3'u tekrarlayın.
- LacZ ekspresyonunu bastırmak için zaman-kurs deneyi sırasında belirli bir zamanda (örn. t = 1,5 dk) 5 mM ortonitropheynl-β-D-fucopyranoside (ONPF) veya 500 mM glikoz24 ekleyin. Yeniden baskıdan sonra, mRNA bozulmasını izlemek için kültürleri (Adım 4.3) örneklemeye devam edin.
NOT: Baskı da ~ 400 μg/mL rifampisin, bir transkripsiyon inisiyasyon inhibitörü25ile yapılabilir. - Fiksasyon için, oda sıcaklığında örneklenmiş hücreleri içeren tüpleri 15 dakika boyunca kuluçkaya yatırın, ardından 30 dakika buzda kuluçka.
- Fiksatifleri gidermek için tüpleri 4500 x g'de oda sıcaklığında 4 dk santrifüj edin. Bir pipet ile supernatant çıkarın.
NOT: Güvenlik protokolünü izleyerek formaldehiti ayrı bir atık kabına attığından emin olun. - 1 mL DEPC-PBS ekleyin ve hücreleri yeniden askıya alın. Tekrar santrifüj ve yeniden süspansiyon 2x daha fazla kez.
NOT: Sabit hücreler kırılgandır ve nazik bir tedaviye ihtiyaç duyarlar. Dikkatle pelet yeniden askıya ve kabarcıklar kaçının. - Son yıkama adımından sonra ~ 30 μL DEPC-PBS'deki hücreleri yeniden askıya alın.
5. Hücre zarlarının permeabilizasyonu
- Kapak kayma (~ 30 μL kuyu başına) farklı kuyulara her zaman noktası örnek uygulayın. Hücrelerin yüzeye yapışması için oda sıcaklığında 10-30 dakika bekleyin. Kuyular arasında sıvı damla birleştirme kaçının.
- Bağlanmamış hücreleri durulamak için sıvıyı aspire edin ve her kuyuya ~20°L DEPC PBS uygulayın. Birkaç dakika içinde DEPC PBS aspire.
- 4 dakika boyunca her kuyuya %70 etanol 15 μL uygulayarak hücre zarlarını permeabilize edin. 4 dakika sonra etanol aspire ve kuyular tamamen kuru olduğundan emin olun.
NOT: Etanol tedavisinin 4 dakika süreyle sınırlandırılması çok önemlidir. Daha uzun tedavi aşırı permeabilizasyon alacaktır. - Yıkama çözeltisinin 30 μL'sini her kuyuya uygulayın.
6. Prob hibridizasyonu
- Yıkama çözeltisini her kuyudan aspire edin. Her kuyuya 30 μL ön hibridizasyon çözeltisi uygulayın. 37 °C fırında 30 dk için hazne kuluçkaya yatırın.
NOT: Nem sağlamak için haznenin dibine ~50 mL su ekleyin. - Her kuyudan ön hibridizasyon çözümaspire. Prob hibridizasyon çözeltisinin ~30 μL'sini her kuyuya uygulayın. 37 °C fırında 2 saat boyunca odayı alüminyum folyo ile kaplayın ve kuluçkaya yatırın.
NOT: Bu adımdan önce prob hibridizasyon çözeltisinin 37 °C tezgah sallayıcıda olduğundan emin olun. Kuyular arasında sıvıların birleştirilmesinden kaçının. Gerekirse her kuyuya çözeltinin daha küçük bir hacmi uygulayın.
7. Hibritizasyon sonrası yıkama ve görüntüleme ye hazırlık
- Çok kanallı bir pipet kullanarak, yıkama çözeltisinin ~30 μL'sini her bir kuyuya aynı anda uygulayın. Aspire ve yıkama 3-5x kez tekrarlayın. 37 °C fırında 15-30 dk için hazneyi kuluçkaya yatırın.
- Adım 7.1'i iki kez daha tekrarlayın.
- DePC-PBS ile her bir kuyuda 5x kez yıkayın. Adım 7.1'de kullanılan yöntemi izleyin, ancak kuluçka işlemini atlayın.
- Örtüden sıvıyı aspire edin. Her kuyuya 4 μL DEPC-PBS uygulayın.
- Forseps kullanarak, kapağı kaldırın ve çevirin ve yavaşça bir cam slayt üzerine yerleştirin (Adım 3.2). Kabarcıklar kaçının.
- Silikon diş sakız ile kapak kenarları mühür.
- Sakız katılanına kadar bekleyin. Burada duraklatmak ve 4 °C'de bir gecede slayt saklayabilirsiniz.
NOT: Diğer smFISH protokolleri oksijen atma reaktifleri eklemeyi (örneğin, glikoz oksidaz/katalaz) veya floroforların fotostabilitesini artırmak için ticari bir anti-fade montaj ortamı14,26 kullanarak öneririz.
8. Görüntüleme
- İlgi çekici bir alan bulmak için faz kontrast görüntülemesinin canlı modunu kullanın. Sahne joystick manevra tarafından bir kuyu içinde görüş alanını değiştirin. Hücre yoğunluğunun en uygun olduğu alanı seçin (örneğin, çoğunlukla ayrılmış birçok hücre vardır). Faz kontrastlı hücre görüntüleri odakta olacak şekilde z-odaklama ayarlayın.
- Cy5 (4-s pozlama), Cy3 (2-s pozlama) ve faz kontrastı (0,2's pozlama) sırasına göre anlık görüntüler alın.
NOT: Cy3B boya molekülleri Cy3 kanalında ve görüntülercy3 görüntüleri olarak adlandırılır. - Bir kuyu içinde ~10 farklı alanların görüntülerini elde etmek için Adımları 8.1-8.2'yi yineleyin.
- Hedefi başka bir kuyuya taşıyın ve Steps 8.1-8.3'u tekrarlayın.
- Görüntüleri TIFF dosyaları olarak dışa aktarın.
- (İsteğe bağlı) Görüntü kayıt amacıyla Cy5 ve Cy3 kanalları arasındaki mekansal kaymayı belirlemek için Cy5 ve Cy3 kanallarında kapak yüzeyinde adsorbe edilen görüntü çok renkli boncuklar.
- Temiz bir kapak yüzeyi üzerinde ~10 μL çok renkli floresan boncuk (0,2 μm çapında) uygulayın ve 10-30 dakika bekleyin. ~50 μL PBS ile yıkandıktan sonra ~ 5 μL PBS uygulayın ve kapağı cam bir kaydırakla sandviç uygulayın. Mikroskobu kapatın ve monte edin.
- Hem Cy5 hem de Cy3 kanallarında görüntü boncukları.
9. Görüntü analizi
NOT: Bu adımda kullanılan Matlab kodu aşağıdaki GitHub web sitesinde mevcuttur: https://github.com/sjkimlab/Code_Publication/tree/master/JoVE_2020. GitHub klasörü, hücre segmentasyonu ve nokta tanımlama için parametre değerleri de dahil olmak üzere görüntü çözümlemesi için gereken her şeyi içerir. Bu adımdaki yordam, "FISHworkflow.m" adlı ana komut dosyasında daha da açıklanmıştır.
- MicrobeTracker27 veya Oufti28gibi bir hücre segmentasyon aracını açın ve faz kontrast görüntülerini yükleyin. "Bağımsız çerçeveler" seçeneğini belirleyin ve hücrelerin tanımlandığı ve konturlarıhesaplanan segmentasyon işlemini başlatmak için "Tüm çerçeveler" adlı düğmeye basın(Şekil 3B,C).
NOT: Bu yazılım paketlerini kullanmak için ayrıntılı protokoller çevrimiçi olarak mevcuttur (örneğin, oufti.org). - MicrobeTracker veya Oufti spotFinder fonksiyonu nda Cy5 floresan görüntüleri yükleyin ve 2D Gaussian montaj dayalı nokta tanımlama ve nicelik başlamak için "Çalıştır" düğmesine basın (Şekil 3B,C). Cy3 kanalındaki noktaları analiz etmek için Cy3 floresan görüntüleri için bu adımı tekrarlayın. Bu adım, yoğunlukları ve koordinatları da dahil olmak üzere her hücredeki noktalar bir listesini üretir.
- (İsteğe bağlı) FISHworkflow.m dosyasında açıklandığı gibi, bir eşik kullanarak loş noktaları (yanlış pozitifler) filtreleyin.
NOT: Negatif kontroldeki floresan noktaları inceleyin (örneğin MG1655 ΔlacZ)ve yanlış pozitifleri filtrelemek için eşiği belirleyin. - Tek bir mRNA'nın nokta yoğunluğunu elde etmek için, sıfır zamanda ölçülen nokta yoğunluklarının bir listesini kullanın (IPTG eklemeden önce) ve iki karışım bileşenine sahip Bir Gauss karışım modeliyle nokta yoğunluklarının dağılımına uyun. Tek bir mRNA'nın nokta yoğunluğu olarak ilk Gauss popülasyonunun (Şekil 3D,E'dekisiyah çizgi) en yüksek konumunu alın. Tek bir 5' ve 3'lacZ mRNA nokta yoğunluğu elde etmek lacZ için ayrı ayrı Cy5 noktalar ve Cy3 noktalar için bunu gerçekleştirin.
NOT: Tek bir mRNA'nın nokta yoğunluğu farklı deneylerde biraz değişebildiği için bunu her zaman denemesinde tekrarlayın. - Bir noktanın floresan yoğunluğunu tek bir mRNA'nın yoğunluğuyla (Adım 9.4'ten) bölüp bir nokta içindeki mRNA sayısını elde edin. Bir hücredeki toplam mRNA sayısını hesaplamak için bir hücre içindeki normalleştirilmiş nokta yoğunluklarını toplamı(Şekil 3F). Bu hesaplamaları 5' ve 3' mRNA için ayrı ayrı gerçekleştirin.
- Her zaman noktasında (şekil 4B)hücre başına ortalama mRNA sayılarını hesaplayın ve çizin ve transkripsiyon ve mRNA bozulmasının ortalama mRNA düzeylerindeki zamansal değişimden in vivo kinetiklerini analiz edin(Şekil 4B).
- Transkripsiyon uzama oranını elde etmek için, 5' ve 3' mRNA sinyallerindeki ilk artışa bir çizginin en az kareler uydurmasını gerçekleştirin ve bazal seviyelere kesitleri belirleyin(Şekil 4B). Bu kesişmeler arasındaki fark, RNAP'ların 5' sonda bölgesinden 3' sonda bölgesine ortalama süreyi gösterir. Transkripsiyon uzama sının ortalama oranını elde etmek için bu süreyle iki sonda kümesi (2 kb) arasındaki mesafeyi bölün.
- mRNA bozunma oranını elde etmek için, üstel bozunma fonksiyonu olan y = A·exp(-t/τ) 5' ve 3' mRNA sinyallerinin son bozunma bölgesine (örneğin, Şekil 4B)sığdırın. Montaj parametresi, τ, ortalama mRNA ömrüdür.
- (İsteğe bağlı) Gen ekspresyonundaki hücreden hücreye değişimi (örneğin, Şekil 4C'degösterilen indüksiyona hücre düzeyinde yanıt), her hücredeki mRNA numaralarının dağılımına göre analiz edin (Adım 9.5'te hesaplanır).
- (İsteğe bağlı) Bir hücrenin ana ve küçük eksenleri boyunca nokta konumu hakkında bilgi kullanarak (Adım 9.2'den elde edilen), mRNA'ların lokalizasyonunu analiz edin (Şekil 4D,E).
- (İsteğe bağlı) Cy5 ve Cy3 kanallarında saptanan noktaların lokalizasyonunu karşılaştırarak 5' ve 3' mRNA'ların(Şekil 5)ortak lokalizasyonunu analiz edin.
- MicrobeTracker spotFinderF fonksiyonu çok renkli boncuk (Adım 8.6) görüntüleri yük ve Cy5 ve Cy3 kanallarında boncuk centroids koordinatlarını elde. Cy5 ve Cy3 kanallarının birbirine göre nasıl kaydırıldığı ve döndürüldüğünü bildiren affine dönüşüm matrisini hesaplamak için merkezoid koordinatlar listesini kullanın29.
- Cy5 koordinatındaki Cy3 görüntülerini dönüştürmek için cy5 ve Cy3 FISH görüntülerine affine dönüştürme matrisini uygulayın. Bir nokta farklı bir kanaldaki başka bir noktayla birlikte yerelleştirilmişse sınıflandırın. Örneğin, merkezsoidleri arasındaki mesafe 150 nm'den azsa Cy5 kanalındaki bir noktanın Cy3 kanalındaki başka bir noktayla birlikte lokalize olduğu kabul edilir(Şekil 5).
- Her zaman noktasında Cy3 noktaları ile "eş lokalize" olarak sınıflandırılan kaç Cy5 noktalarını analiz edin. Ayrıca, eş lokalize noktaların yoğunluğunu analiz edin (Şekil 5).
Sonuçlar
Şekil 3 bu smFISH protokolünden temsili görüntüler gösterir. Tam görüş alanı (86.7 μm x 66.0 μm Malzeme Tablosu'ndaayrıntılı olarak yer alan mikroskopi kurulumumuzu kullanarak) ~500 E. coli hücresinin alana dağılmış olduğunu gösterir(Şekil 3A). Hücrelerin yoğunluğu bu resimde gösterilenden çok daha yüksekse, segmentasyon algoritmaları hücreler birbirine dokunduğunda hücreleri güvenilir bir şekilde tanımlamadığı için otomatik hücre segmentasyonu zorlaşır. Bir görüş alanında hücrelerin en uygun yoğunluğunu elde etmek için yüzey yapışma (Adım 5.1) için kullanılan hücre konsantrasyonu ve kuluçka süresi ayarlamak gerekir.
Faz kontrast ı simgelerdeki hücrelerin morfolojisi segmentasyon amacıyla canlı hücrelerinkiyle karşılaştırılabilir kalmalıdır(Şekil 3A-C). Hücreler aşırı permeabilize ise, hücre morfolojisi değişir ("hayaletler" gibi; Ek Şekil 1). Bu durumda, bir Adım 5.3% etanol tedavisinin süresini azaltabilir.
İndüksiyon dan önce ortalama lacZ ifade düzeyi hücre başına ~0,03 mRNA idi, önceki raporlarla tutarlı15,30. Ayrıca, indüksiyon öncesi lacZ mRNA nokta yoğunluklarının dağılımı, yüksek yoğunluklu lekelerin varlığı nedeniyle normal bir dağılım aveya Poisson dağılımına uymadı (Şekil 3D,E). Bu, bastırılmış durum altında algılanan lekelerin çoğunun tek bir lacZ mRNA'yı temsil ettiğini, ancak küçük bir leke popülasyonunun birden fazla lacZ mRNA içerdiğini göstermektedir. Nüfusu tek bir lacZ mRNA ile izole etmek için, iki karışım bileşeni (Şekil 3D,E'dekiinsets) içeren bir Gaussian karışım modeli kullandık. Daha sonra, ilk Gaussian ortalaması tek bir mRNA noktasının ortalama yoğunluğu olarak alınmıştır (örneğin, Şekil 3D'dekisiyah eğrinin zirvesi) ve zaman denemesinde tespit edilen noktalar için nokta yoğunluğunu mRNA sayısına dönüştürmek için kullanılmıştır. Bir hücreiçindeki toplam mRNA sayısını hesaplamak için, normalleştirilmiş nokta yoğunlukları her hücrede özetlenmiştir (Şekil 3F)19.
lacZ mRNA'nın ifade düzeyi düşük olduğunda, bir hücre içinde uzamsal olarak ayrılmış bir veya iki kırınım sınırlı lakZ mRNA noktaları vardır. Bu nedenle, bu noktalar görüntüleri yoğunluk ve lokalizasyon için 2D Gaussian uydurma tarafından analiz edilebilir.
İfade düzeyi yüksek olduğunda, noktalar bir hücre içinde birbiriyle çakıştığında, 2D Gaussian uydurma güvenilir niceleme yapmaz. Bu durumda, mRNA düzeyi, tek bir mRNA19ortalama yoğunluğu ile bir hücre içinde toplam, arka plan çıkarılan floresan sinyali bölerek hesaplanmalıdır.
LacZ ekspresyonu indüklendiğinde, 5'in lacZ mRNA sinyali önce artar ve 3' lacZ mRNA daha sonra artar (Şekil 4B). lacZ ifadesi bastırılırsa, hem 5' hem de 3' lacZ mRNA sinyalleri arasında bir gecikme ile azalır (Şekil 4B). Transkripsiyon uzama oranını elde etmek için, 5' ve 3' sinyallerin yükselişi ilk olarak çizgilerle(Şekil 4B)uyar ve x-keseleri arasındaki fark, IKI prob bölgesi (2.000 nt) arasındaki mesafeyi kat etme süresi olarak alınır. Transkripsiyon uzama oranı her zaman denemesinden ölçülebilir ve standart sapmalar deneysel kopyalardan hesaplanabilir. Transkripsiyon uzamasının ortalama oranı deneysel koşullarımız altında 15-30 nt/s idi19.
Ayrıca, çürüme bölgesinin üstel bir fonksiyonla donatılması ile mRNA bozulma oranı (ortalama mRNA ömrünün tersi) elde edilmiştir (Şekil 4B). Zaman kursu verilerimiz transkripsiyon sırasında ve sonrasında mRNA bozulması içerir31. 3' mRNA çürümeye başladıktan sonra zaman noktalarına uyuyoruz (t > 6 dk) serbest bırakılan mRNA'ların bozulmasını araştırmak için. Biz 5' veya 3' lacZ mRNA19ortalama ömrü olarak ~ 90 s elde etti.
Transkripsiyon başlatma oranı indüksiyon sonrası 5' sinyal artışı nın eğiminden(Şekil 4B, mavi) veya sabit durumdaki ortalama mRNA sayısından (bozulma oranına göre bölünen başlatma oranıdır) hesaplanabilir. Ayrıca, erken transkripsiyon sonlandırma olasılığı tahmin edilebilir, ya 3'ün sinyal artışı nın eğimi arasındaki oran 3' sinyal artışı32'ye veya 3' ile 5' mRNA bölgeleri arasındaki sabit durum seviyeleri arasında19olarak tahmin edilebilir.
SMFISH tek hücreli bir teknik olduğu için transkripsiyonda hücreden hücreye değişkenliği analiz edebiliriz. Örneğin, IPTG eklendikten sonra lacZ mRNA ifade eden hücrelerin yüzdesini analiz edebilirsiniz (Şekil 4C). Ayrıca, indüksiyondan sonra mRNA yerelleştirmenin değişip değişmediğini de ele alabilirsiniz. 5' ve 3' lacZ mRNA lekelerinin hücrenin merkezinden biraz dışa doğru hareket ettiğini gözlemledik (Şekil 4D,E),önceki bir raporla uyumlu33.
Son olarak, 5' ve 3' mRNA noktaları arasında eş-lokalizasyon analizi bilgilendirici olabilir (Şekil 5A). Örneğin, bastırılmış durumda (sıfır zaman), 5' mRNA noktalarının yaklaşık %25'i 3' mRNA noktasıyla birlikte lokalize edilir. t = 1 dk, birçok gen loci 5 ' mRNA sentezi var, ama henüz 3' mRNA sentezi, 5 'mRNA lekeleri çoğu kendileri tarafından 3' mRNA sinyal (yani, ko-lokalizasyon düşük olasılık) olmadan vardır. Ancak, 3' mRNA göründüğünde (yani, t = 2 dk), birlikte yerelleştirme olasılığı artar (Şekil 5A'dakimor ok ,B). Bu zaman noktası, ortak yerelleştirme sık hale geldiğinde, transkripsiyon uzama oranına bağlıdır. Bu noktada tespit edilen her bir ko-lokalizasyon noktasında ki 5' ve 3' lacZ mRNA numaralarının 2-Yoğunluk çizimi, lacZ genindeki RNAP yoğunluğunu saptamak için kullanılabilir (Şekil 5C). Daha önce bildirildiği gibi19, Bu çizimde 5 ' mRNA numaraları lacZ ekspresyonu 1 mM IPTG tarafından indüklenen zaman LACZ loci en DNA üzerinde 10'dan az RNAPs olduğunu göstermektedir. Ayrıca, bu çizimdeki 3' mRNA numaraları RNA'larınkümeleme 34ile ilgilidir. 3' mRNA sayısının bire yakın olması, 3'ün sonda bölgesine kabaca sadece bir RNAP girdiği anlamına gelir. Bu, lacZ genindeki RNAP'ların bir küme (veya "konvoy" oluşturmak yerine) mekansal olarak ayrıldığını göstermektedir.
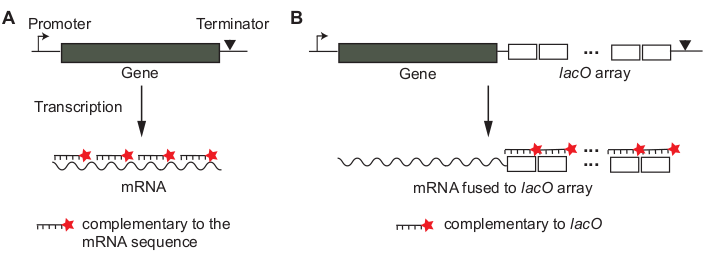
Şekil 1: SMFISH problarının bir mRNA için tasarlanması. (A) Fayans yöntemi. Kısa DNA oligonükleotid dizileri (~20 bp uzunluğunda) seçiliyor, böylece ilgi nin mRNA'sını kapsayabilirler. Oligonükleotid problar floresan boya molekülü ile etiketlenmiştir. (B) Bir dizi yöntemi. Tandem dizilerinin kodlamayan bir dizisi (örneğin,"lacO dizisi") transkripsiyonel olarak ilgi çeken mRNA'ya yapıştırılır. Bir mRNA sinyalini yükseltmek için floresan olarak etiketlenmiş prob tekrar ünitesine (örn. 17 bp uzunluğunda ki lacO prob) kullanılır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
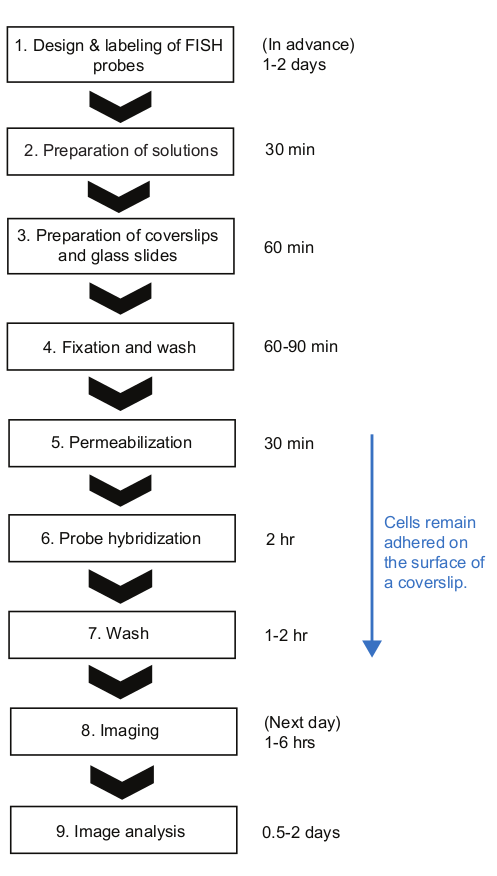
Şekil 2: SMFISH deneysel prosedür şematik ve her adımın zaman süresi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 3: smFISH görüntü analizi. (A-C) smFISH mikroskobu görüntüsü 5' lacZ mRNA (kırmızı) ve 3' lacZ mRNA (yeşil) vahşi tip E. coli (MG1655) M9 minimal ortamda yetiştirilen 0.2% gliserol ile takviye, 0.1% casamino asitler, ve 1 mg / L tiamin 30 °C. (A) t = 3 dk bir örnek ten temsili bir görüntü t = 0 mM IPTG ile t = 0 dk ve baskı ile 500 mM glikoz ile t = 1,5 dk. Faz kontrastı ve Cy5 (5' lacZ mRNA, kırmızı için) ve Cy3 'in iki floresan görüntüsü (3' lacZ mRNA için, yeşil) sözde boyama ile kaplandı. Görüntü 86.7 μm x 66.0 μm'lik bir alanın tamamını gösterir. Ölçek çubuğu, 5 μm. (B) (A) içinde küçük bir bölgenin (sarı kutu) yakınlaştırma sürümü. Hücre anahatları beyaz, ve görüntü analizinden tanımlanan floresan noktalar kırmızı noktalarla gösterilir. Ölçek çubuğu, 1 μm. (C) Yüksek ekspresyon altında hücre anahatları ve floresan lekelerin saptanması (t = 4 dk indüksiyon sonrası 1 mM IPTG). Ölçek çubuğu, 1 μm. (D-E) IPTG (bastırılmış durum) eklemeden önce ölçülen 5' ve 3' mRNA nokta yoğunluklarının dağılımları. Histogramlar, ortalama değerleri Gauss karışım modeline ait olan iki Gauss fonksiyonu (siyah ve gri) ile gösterilir. Inset, Gausskarışım modellerinden ve deneysel olarak ölçülen mRNA nokta yoğunluklarından (5' mRNA için n = 1040 ve 3' mRNA için 680) oluşturulan rastgele sayıların nicel-nicel çizimini gösterir. (F) Panel (B) işaretli tek bir hücre için elde edilen bilgiler. Belirli bir hücre için (i), noktalar Cy5 ve Cy3 kanallarında tespit edildi ve bunların yoğunluğu (I) ve bir hücrenin kısa ve uzun ekseni boyunca koordinat(d, l) 2D Gaussian uydurmadan ölçüldü. Normalleşme nokta yoğunlukları bu hücredeki toplam 5' veya 3' mRNA'yı ortaya çıkarmak için toplandı. Ayrıca, farklı kanallardan noktalar arasındaki birlikte yerelleştirme Şekil 5'tegösterilen örnekte olduğu gibi analiz edilebilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 4: Transkripsiyon ve mRNA bozulmasının in vivo kinetik analizi. (A) Zaman içinde lacZ mRNA düzeylerindeki değişiklikleri ölçen iki renkli smFISH deneylerinin şematik ve temsili görüntüleri. Kırmızı ve yeşil noktalı çizgiler, Sırasıyla E. coli'deki lacZ mRNA'nın 1-kb uzunluğunda5' ve 3' mRNA bölgelerine melezlenen Cy5 veya Cy3B etiketli oligonükleotid problarını gösterir. Ayrıca t = 0 dk 0.2 mM IPTG ile indüksiyon sonrası belirtilen zaman noktalarında faz kontrast görüntü ile iki floresan görüntü bindirmeleri gösterilir. Transkripsiyon t = 1.5 dk 500 mM glikoz ile bastırıldı. Ölçek çubuğu, 1 μm. Rakam Kim ve ark19değiştirilmiştir. (B) 5' ve 3' lakZ mRNA sayıları zaman içinde hücre başına, panelde açıklanan deney sırasında (A). Hata çubukları önyüklemeli SEM'lerdir. Zaman başına en az 1.200 hücre analiz edildi. 5' ve 3' mRNA sinyallerinin ilk yükselişi bir çizgi (mavi) ile uyumluydu. X-intercepts farkı 1.93 dk, transkripsiyon uzama ortalama oranı 17.3 nt / s verim. 5' ve 3' mRNA sinyallerinin son çürümesi üstel bozunma fonksiyonuna (gri) uyuyordu. Uyum parametreleri ortalama mRNA ömrünün 5' mRNA için 1,52 dk, 3' mRNA için 1,66 dk olduğunu göstermektedir. (C) (A) de açıklanan deney sırasında bir veya daha fazla lakZ mRNA lekeli hücrelerin yüzdesi. Hata çubukları önyüklemeli SEM'lerdir. (D) Bir noktanın hücrenin kısa ekseni boyunca yerelleştirilmesi. Bir noktanın zara olan yakınlığını, kısa eksen boyunca(d)hücrenin yarım genişliğiyle (w)bölerek ölçebilirsiniz. (E) (A) de açıklanan deney sırasında hücrelerin kısa ekseni boyunca 5' ve 3' lakZ mRNA noktalarının lokalizasyonundaki değişim. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
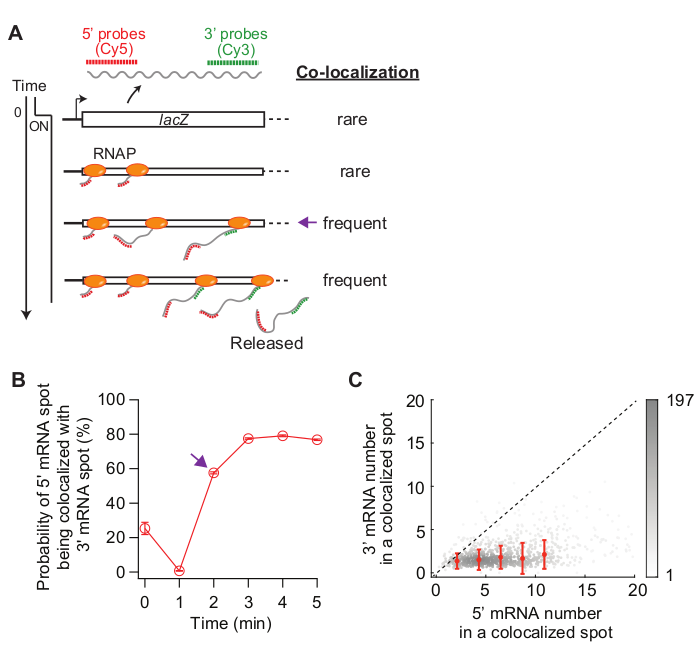
Şekil 5: 5' ve 3' mRNA noktalarının eş lokalizasyonunun analizi. (A) Şema indüksiyon sonrası 5' ve 3' mRNA noktaları arasında beklenen ko-lokalizasyonu gösterir. 3' mRNA yapıldığında, 5' mRNA noktasının 3' mRNA nokta artar (mor ok) ile birlikte lokalize olma olasılığı. (B) 1 mM IPTG ile indüksiyon sonrası birlikte lokalizasyon olasılığı. Mor ok, panel (A) şemasına göre eş yerelleştirme olasılığının ilk sıklıkta olduğu zaman noktasını gösterir. (C) 1 mM IPTG (toplam 841 nokta) ile indüksiyon sonrası t = 2 dk saptanan bir ko-lokalizasyon noktasında bulunan 5' ve 3' lacZ mRNA sayısı. Gri noktalar tek tek eş yerelleştirilmiş noktaları temsil ederken, kırmızı noktalar binned verilerin ortalamasını temsil eder. Hata çubukları SEM'dir. Grinin gölgesi grafiğin belirli bir alanındaki noktaların yoğunluğunu gösterir. Noktalı çizgi 1 eğimi gösterir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
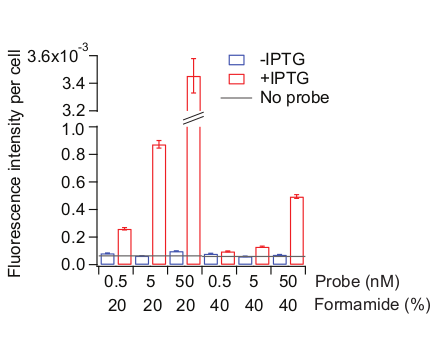
Şekil 6: Prob hibridizasyon koşulunun optimizasyonu. İki tür örnek kullanıldı: Şekil 3'te açıklandığı gibi yetiştirilen mg1655 hücreleri indüklenmemiş (mavi) veya 20 dakika boyunca 0,5 mM IPTG ile tedavi edildi .. Prob hibridizasyon çözeltisi farklı konsantrasyonlarda problar (toplam 72 Cy5 konjuge problar tüm lacZ bölge fayans) ve formamid ile yapıldı. Formamid konsantrasyonları da pre-hibridizasyon çözeltisi ve yıkama çözeltisi, buna göre ayarlandı. "Prob yok" (gri çizgi) hibritleştirme adımı sırasında prob olmadan tedavi edilen IPTG eklenen hücrelerin floresan düzeyini gösterir. Hücre alanı (AÜ) tarafından normalleştirilen ortalama floresan yoğunluğu 300-800 hücreden hesaplandı. Hata çubukları önyüklemeli SEM'lerdir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Ek Şekil 1: Aşırı permeabilizasyona bağlı bozuk hücre morfolojileri. Faz kontrastı (gri skala), 5' lacZ mRNA (Cy5, kırmızı) ve 3' lacZ mRNA (Cy3, yeşil) MG1655 hücrelerinin 1 mM IPTG ile indüksiyondan 5 dk sonra görüntülerinin yer kaplaması. (A) Normal morfolojisi olmayan normal hücrelerin ve aşırı permeabilize hücrelerin karışımını gösteren bir örnek (pembe oklarla gösterilir). (B) "hayalet" hücrelerin bir araya toplanmış gösteren bir örnek. Ölçek çubuğu = 1 μm. Bu dosyayı indirmek için lütfen buraya tıklayınız.
| DEPC Su | ||||
| Ultra saf suya %0,1 DEPC ekleyin ve şişeyi (kapalı) bir gecede 37°C fırında ve ertesi gün otoklavda kuluçkaya yatırın. | ||||
| DEPC PBS (10X) | ||||
| Aşağıdakileri karıştırın: | ||||
| 80 g | NaCl (son 1.37 M) | |||
| 2 g | KCl (son 27 mM) | |||
| 14.2 g | Na2HPO4 (son 100 mM) | |||
| 2.7 g | KH2PO4 (son 20 mM) | |||
| 1L'ye ultra saf su | ||||
| Cam şişeye (0,22 μm) süzün. | ||||
| %0,1 DEPC ekleyin ve DEPC suyu yönergesini uygulayın. | ||||
| 1X çözeltisi yapmak için DEPC suyu ile 10 kez seyreltin. | ||||
| 1M DEPC sodyum fosfat tamponu, pH 7.4 | ||||
| Aşağıdakileri karıştırın: | ||||
| 115 g | Na2HPO4 | |||
| 22.8 g | NaH2PO4 | |||
| Ultra saf su 1 L | ||||
| Cam şişeye (0,22 μm) süzün. | ||||
| %0,1 DEPC ekleyin ve DEPC suyu yönergesini uygulayın. | ||||
| 4X sabitleme çözeltisi (%16 formaldehit) | ||||
| 5 mL | %20 formaldehit | |||
| 500 μL | DEPC su | |||
| 750 μL | 1M DEPC sodyum fosfat tamponu, pH 7.4 | |||
| 4 °C'de 2-4 haftakadar saklayın. | ||||
| DİkKAT: Formaldehit toksiktir. Bu çözümü yaparken eldiven giyin ve duman kaputu kullanın. | ||||
| Yıkama çözeltisi | ||||
| Aşağıdakileri karıştırın: | ||||
| 10 mL | Formamid (%25) | |||
| 4 mL | 20X SSC (son 2X) | |||
| DEPC suyunu 40 mL'ye doldurun | ||||
| Filtre (0,22 μm) ve 4 °C'de saklayın | ||||
| DİkKAT: Formamide toksiktir. Bu çözümü yaparken eldiven giyin ve duman kaputu kullanın. | ||||
| Ön hibridizasyon çözümü | ||||
| 200 μL | Formamid (%20) | |||
| 100 μL | 20X SSC (son 2X) | |||
| 10 μL | 100X VRC (son 1X) | |||
| 25 μL | %4 (w/v) BSA (%0.1) | |||
| 685 μL | DEPC su | |||
| NOT: 10 μL çıkarmadan önce VRC stoğunu girdap. | ||||
| DİkKAT: Formamid toksik ve bilinen bir teratojendir. Eldiven giyin ve duman kaputunun altında tut. | ||||
| Prob hibridizasyon çözümü | ||||
| 200 μL | Formamid (%20) | |||
| 100 μL | 20X SSC (son 2X) | |||
| 10 μL | 100X VRC (son 1X) | |||
| 25 μL | %4 (w/v) BSA (%0.1) | |||
| 10 μL | 40 mg/mL E. coli tRNA (son 0.4 mg/mL) | |||
| 200 μL | %50 dekstran sülfat (%10) | |||
| x μL | 5' mRNA sonda seti (Adım 1.12'den son 4 nM'ye kadar). | |||
| y μL | 3' mRNA sonda seti (Adım 1.12'den son 4 nM'ye kadar). | |||
| - | DEPC su toplam hacmi 1 mL yapmak | |||
| NOT: Son dextran sülfat ekleyin. Çok viskoz olduğundan, %50'lik stoktan 200 μL çıkarmadan önce pipet ucunu kesin. Dekstran sülfat ekledikten sonra, pipet yukarı ve aşağı çözeltihomojeize etmek. | ||||
Tablo 1: Kullanılan çözeltilerin tarifleri.
Tartışmalar
Burada, E. colimRNA kinetik ölçmek için bir smFISH protokolü sundu. Daha önce yayınlanan bakteriler için smFISH protokollerinde23, hücreler protokolün sonuna kadar tüplerde tutuldu, yani görüntülemeye hazır olana kadar. Kapak yüzeyi23'tefloresan probların minimum spesifik olmayan bağlanması gibi birçok faydası olsa da, bir zaman kursu deneyinden çok sayıda örnek olduğunda bu protokolleri takip etmek zordur. İlk olarak, nispeten büyük miktarda hücre (>1 mL) saptanmadan önce örneklenmeli ve hatta hasat edilmelidir. İkinci olarak, hücre örneklerinin çözüm değişimi ve hibridizasyon adımından sonra yıkanması için birden çok kez santrifüj edilmesi gerekir. Protokolümüzde, küçük bir kültür hacmi (<1 mL) doğrudan 1,5 mL'lik bir tüpteki sabitleme çözeltisi ile karıştırılır ve örnekleme anında hücre durumunun hızla "dondurulmasına" yardımcı olur. Ayrıca, hücreler işlem boyunca yüzeye bağlı kalır ve farklı çözeltiler vakum filtrasyon sistemi ile sıvıların aspire edilmesi ve çok kanallı pipet ile aynı anda çözelti damlaları uygulanarak hızlı bir şekilde değiştirilebilir. Bu fark, çok sayıda numunenin aynı anda işlenmesi gerektiğinde protokolümüzü son derece avantajlı hale getirir. Protokolümüzü kullanarak, 12-48 numune aynı anda işlenebilir ve tüm FISH prosedürü ~8 saat içinde, yaklaşık birkaç örnek için gerekli olan benzer bir süre içinde tamamlanabilir(Şekil 2). E. coli'deki lacZ ifadesini örnek olarak kullanmamıza rağmen, protokol aşağıda tartışılan hususlar ile farklı genler ve bakteri türleri için yaygın olarak uygulanmaktadır.
Farklı genler için göz önünde bulundurulması gereken ilk şey smFISH sondalarıdır. İlginin mRNA'sını çinileyen oligonükleotid problar tasarlanabilir (Şekil 1A)13. Bu "fayans" sonda yaklaşımında, her prob ~20 baz uzunluğundadır ve 5' veya 3' terminusta bir florofor ile etiketlenir. Hiçbir genetik manipülasyon gerekli olduğu gibi bu strateji uygundur. Alternatif olarak, genomik diziye yabancı olan ~20 bp dizisinin bir tandem tekrarı (örneğin, Caulobacter crescentus14'tekibir dizi lako dizisi), ilgi çekici bir genin çevrilmemiş bölgesine eklenebilir ve tekrar birimine tamamlayıcı tek bir prob mRNA ("dizi" yaklaşımı) etiketlemek için kullanılır; Şekil 1B). Her iki durumda da, birden fazla florofor bir mRNA süsler, kolayca bir hücre içinde özel olarak bağlı olmayan tek bir prob ayırt edilebilir amplifiye floresan sinyali veren.
"Döşeme" veya "dizi" yaklaşımlarının seçilip seçilemeyeceği negatif denetime bağlıdır, hedef mRNA'dan yoksun olduğu için probların spesifik olmayan bağlanmasının test edildiği bir örnek. Fayans probları için(Şekil 1A),genin yazılmaması (örneğin, lacZ'ninbastırılması) ilgi geni veya bir durum olmayan mutant bir tür, probların spesifik olmayan bağlanmasını test etmek için negatif bir kontrol görevi görebilir. Dizi tabanlı smFISH(Şekil 1B)için, diziden yoksun bir yabani tür, problar için bağlayıcı siteler içermediğinden negatif denetim olarak kullanılabilir.
Optimum hibridizasyon koşulları sonda dizilerine ve hatta florofor boyaseçimine bağlı olabilir. LacZ prob setleri için hibridizasyon koşulunu 37 °C'de tutarak ve hibridizasyon çözeltisinde farklı prob setleri ve formamid konsantrasyonlarını test ederek optimize ettik. Formamid yüksek konsantrasyonları hem nonspesifik hem de spesifik bağlayıcılığı azaltmak eğilimindedir26,35. Melezleme süresini ve sıcaklığını aynı tutarken, melezleşme ve yıkama koşullarını sistematik olarak değiştirmenizi öneririz. Durum daha sıkı hale geldikçe, hem spesifik hem de spesifik bağlama azalması(Şekil 6). Spesifik olmayan bağlamanın, belirli bağlamadan daha fazla ödün vermeden kabul edilebilir bir eşiğin altına vurmaya başladığı bir nokta bulmak önemlidir. Örneğin, herhangi bir sonda olmadan elde edilen sinyal düzeyini ("prob yok") eşik olarak kullandık (Şekil 6).
Bir mRNA'nın iki ayrı bölgesini etiketleyen iki renkli smFISH yöntemi uzun genlerle sınırlıdır. Transkripsiyon uzama oranını ölçmek için, lacZ'ın uzun (3075 bp) olduğu ve ifadesinin IPTG tarafından indüklenebildiği gerçeğinden yararlandık. Bir gen kısa olduğunda, iki döşeme sondası seti (5' ve 3' uçlara yakın) tasarlamak ve 5' ile 3' mRNA bölgelerinin görünümleri arasındaki zaman gecikmesini çözmek zordur. Bu durumda, smFISH tarafından sürekli durumda yeni doğan mRNA'ları sayabilir ve transkripsiyon uzama oranı uygun bir parametre20olan analitik bir model ile dağılımlarını analiz edebilir. Ayrıca, ilgi bir gen indüklenebilir olmadığında, bir zaman sıfır rifampisin ile hücreleri tedavi edebilir ve 5've 3'mRNA alt bölgelerinde zamansal değişim ölçmek. 5' mRNA sinyalinin düşürülmesinden 3' mRNA sinyaline gecikme, daha önce31'deyapıldığı gibi transkripsiyon uzama oranını hesaplamak için kullanılabilir.
Son olarak, smFISH protokolü çok yönlüdür ve diğer etiketleme şemaları ile birleştirilebilir. Daha önce, DNA lokusu mRNA FISH ile DNA FISH14 veya floresan muhabir-operatör sistemi20ile birleştirilerek mRNA ile birlikte görselleştirildi. Protein ürünleri mRNA FISH14,36ile birlikte immünororesans gerçekleştirerek görselleştirilebilir. Ayrıca, üç boyutlu süper çözünürlüklü mikroskopi ile kombine edilebilir37 her üç boyutta mRNA görselleştirmek için38,39.
Açıklamalar
Yazarlar hiçbir rakip mali çıkarları olduğunu beyan.
Teşekkürler
Bu protokol S.K. tarafından Dr. Christine Jacobs-Wagner'in Howard Hughes Tıp Enstitüsü'ndeki laboratuvarında ve Yale Üniversitesi Mikrobiyal Bilimler Enstitüsü'ndeki doktora sonrası araştırması sırasında geliştirilmiştir. Dr. Jacobs-Wagner ve laboratuvar üyelerine yöntem geliştirme sırasında ki çeşitli girdiler için ve Laura Troyer'a makalenin eleştirel okuması için teşekkür ederiz. S.K. Searle Scholars Programı'nın desteğini kabul eder; K.V. Illinois Üniversitesi'nden James Scholar Preble Araştırma Ödülü'nün desteğini kabul eder.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Bacterial strain | |||
| Escherichia coli MG1655 | |||
| Chemicals, peptides, and others | |||
| Acetonitrile | Sigma-Aldrich | 34851 | UPLC buffer B |
| Ammonium chloride | Fisher Chemical | A661-500 | To make M9 medium |
| Bovine serum albumin (BSA) | Sigma-Aldrich | B2518 | Probe hybridization |
| Calcium chloride | Acros Organics | 349610250 | To make M9 medium |
| Casamino acid | BD Difco | 223050 | To make M9 medium |
| Cy3B NHS ester | GE Healthcare Life Sciences | PA63101 | Fluorophore for FISH probes |
| Cy5 NHS ester | GE Healthcare Life Sciences | PA15101 | Fluorophore for FISH probes |
| DEPC | Sigma-Aldrich | D5758 | |
| Dextran sulfate | Millipore | S4030 | Probe hybridization |
| Dextrose | Fisher Chemical | D16 | To repress the expression of lacZ |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | To dissolve fluorophores |
| E. coli tRNA | Sigma-Aldrich | R1753 | Probe hybridization |
| Ethanol | Decon Laboratories | 2701 | Used in DNA purification, lysis, and cleaning coverslips |
| FISH probes | Biosearch Technologies | Sequences are published in ref#16 | |
| Formaldehyde | Ladd Research Industries | 20295 | Fixation |
| Formamide | American Bio | AB00600 | Probe hybridization, pre-hybridization, and wash |
| Glycerol | Americanbio | AB00751-01000 | To make M9 medium |
| Isopropylthio-β-galactoside (IPTG) | Invitrogen | 15529019 | lacZ induction |
| Magnesium sulfate | Fisher Chemical | M65-500 | To make M9 medium |
| 2-Nitrophenyl β-D-fucopyranoside (ONPF) | Santa Cruz Biotechnology | sc-216258 | lacZ repression |
| Picodent twinsil 22 | Picodent | 1300 1000 | Sealant |
| Poly-L-lysine | Sigma-Aldrich | P8920 | To treat the coverslip surface |
| Potassium chloride | Fisher BioReagents | BP366-500 | To make PBS |
| Potassium phosphate monobasic | Fisher BioReagents | BP362-500 | To make PBS and M9 medium |
| Rifampicin | Sigma-Aldrich | R3501 | To stop transcription initiation |
| Saline-sodium citrate buffer (SSC) | Invitrogen | AM9763 | Probe hybridization, pre-hybridization, and wash |
| sodium bicarbonate | Fisher BioReagents | BP328-500 | Fluorophore-probe conjugation |
| Sodium chloride | Fisher BioReagents | BP358-1 | For DNA purification, PBS and M9 medium |
| Sodium phosphate dibasic | Fisher BioReagents | BP332-500 | To make PBS and M9 medium |
| Sodium phosphate monobasic | Fisher BioReagents | BP329-500 | To make a sodium phosphate buffer |
| Super PAP Pen | Invitrogen | 8899 | Hydrophobic marker for coverslips |
| TetraSpek microspheres | Invitrogen | T7280 | Controls for multi-channel registration |
| Thiamine | Sigma-Aldrich | T1270 | To make M9 medium |
| Triethylammonium acetate | Sigma-Aldrich | 90358 | UPLC buffer A |
| Vanadyl ribonucleoside complex (VRC) | Sigma-Aldrich | 94742 | Probe hybridization and pre-hybridization |
| Equipment | |||
| C18 column | Waters | Acquity BEH C18 column | |
| Countertop centrifuge | Eppendorf | 5425 | |
| Countertop incubator | Eppendorf | Thermomixer F1.5 | |
| Incubator (Oven) | Thermo Scientific | 51030514 | Gravity convection |
| Water purification system | Millipore | Milli-Q Reference | |
| Nanodrop | Thermo Scientific | 2000C | |
| Nitrogen gas | Building | For blow-drying coverslips and glass slides | |
| UPLC | Waters | Acquity UPLC system | |
| Vacuum and aspirator | Building | Aspirator is made of a filtration flask with a side arm. | |
| Vacuum concentrator | Labconco | 7810010 | Centrivap; to dry samples collected from UPLC. |
| Vortexer | Scientific Industries | Genie-2 SI-0236 | |
| Water bath shaker | New Brunswick | Innova 3100 | Critical for time-course experiments |
| Water bath sonicator | VWR | 97043-960 | To clean coverslips and glass slides |
| Tools | |||
| 1.5-mL tubes | Eppendorf | 22431021 | DNA lobind tubes |
| 1000-uL pipette tip box | Denville Scientific | P1126 | An empty box after using all the tips |
| Coplin jar | SPI | 01240-AB | To clean coverslips and glass slides |
| Coverslip | Fisher Scientific | 22-050-230 | 24x60 No1 |
| Filtered pipette tips | Denville Scientific | P1121,P1122,P1126 | SHARP® Precision Barrier Tips |
| Forceps | SPI | K35a | To handle clean coverslips and glass slides |
| Glass slide | Fisher Scientific | 12-544-1 | |
| Gloves | Microflex | MK-296-M | |
| Multichannel pipetter | Eppendorf | 2231300045 | To use in the washing step (#7) |
| Pipette | Gilson | P1000, P200, P20 | |
| Reagent reservoir | MTC Bio | P8025-1S | To use in the washing step (#7) |
| Syringe filter (0.22 um) | Millipore | SLGS033SS | |
| Timer | VWR | 62344-641 | |
| Software and algorithms | |||
| MATLAB | Mathworks | R2013 and up | https://www.mathworks.com |
| MicrobeTracker or Oufti | https://www.github.com/JacobsWagnerLab/MicrobeTracker | ||
| https://oufti.org/ | |||
| Stellaris Probe Designer | Biosearch Technologies | https://www.biosearchtech.com/support/tools/design-software/stellaris-probe-designer | |
| Microscope | |||
| CCD camera | Hamamatsu Photonics | Orca-II-ER | |
| Cy3 filter set | Chroma | 49004 | |
| Cy5 filter set | Chroma | 49006 | |
| Epi-fluorescence microscope | Nikon | Eclipse Ti | For phase-contrast and epi fluorescence |
| Fluorescence excitation source | Lumencor | SOLA-E | |
| Nikon Elements software | Nikon | software that controls the microscope setup | |
| Phase-contrast 100x objective | Nikon | Plan Apochromat (NA 1.45) | |
| Probe sequence | |||
| DNA oligos with C6 amino modification at the 5' end | Biosearch Technologies Inc | ||
| lacZ1 | GTGAATCCGTAATCATGGTC | 5' mRNA | |
| lacZ2 | TCACGACGTTGTAAAACGAC | 5' mRNA | |
| lacZ3 | ATTAAGTTGGGTAACGCCAG | 5' mRNA | |
| lacZ4 | TATTACGCCAGCTGGCGAAA | 5' mRNA | |
| lacZ5 | ATTCAGGCTGCGCAACTGTT | 5' mRNA | |
| lacZ6 | AAACCAGGCAAAGCGCCATT | 5' mRNA | |
| lacZ7 | AGTATCGGCCTCAGGAAGAT | 5' mRNA | |
| lacZ8 | AACCGTGCATCTGCCAGTTT | 5' mRNA | |
| lacZ9 | TAGGTCACGTTGGTGTAGAT | 5' mRNA | |
| lacZ10 | AATGTGAGCGAGTAACAACC | 5' mRNA | |
| lacZ11 | GTAGCCAGCTTTCATCAACA | 5' mRNA | |
| lacZ12 | AATAATTCGCGTCTGGCCTT | 5' mRNA | |
| lacZ13 | AGATGAAACGCCGAGTTAAC | 5' mRNA | |
| lacZ14 | AATTCAGACGGCAAACGACT | 5' mRNA | |
| lacZ15 | TTTCTCCGGCGCGTAAAAAT | 5' mRNA | |
| lacZ16 | ATCTTCCAGATAACTGCCGT | 5' mRNA | |
| lacZ17 | AACGAGACGTCACGGAAAAT | 5' mRNA | |
| lacZ18 | GCTGATTTGTGTAGTCGGTT | 5' mRNA | |
| lacZ19 | TTAAAGCGAGTGGCAACATG | 5' mRNA | |
| lacZ20 | AACTGTTACCCGTAGGTAGT | 5' mRNA | |
| lacZ21 | ATAATTTCACCGCCGAAAGG | 5' mRNA | |
| lacZ22 | TTTCGACGTTCAGACGTAGT | 5' mRNA | |
| lacZ23 | ATAGAGATTCGGGATTTCGG | 5' mRNA | |
| lacZ24 | TTCTGCTTCAATCAGCGTGC | 5' mRNA | |
| lacZ25 | ACCATTTTCAATCCGCACCT | ||
| lacZ26 | TTAACGCCTCGAATCAGCAA | ||
| lacZ27 | ATGCAGAGGATGATGCTCGT | ||
| lacZ28 | TCTGCTCATCCATGACCTGA | ||
| lacZ29 | TTCATCAGCAGGATATCCTG | ||
| lacZ30 | CACGGCGTTAAAGTTGTTCT | ||
| lacZ31 | TGGTTCGGATAATGCGAACA | ||
| lacZ32 | TTCATCCACCACATACAGGC | ||
| lacZ33 | TGCCGTGGGTTTCAATATTG | ||
| lacZ34 | ATCGGTCAGACGATTCATTG | ||
| lacZ35 | TGATCACACTCGGGTGATTA | ||
| lacZ36 | ATACAGCGCGTCGTGATTAG | ||
| lacZ37 | GATCGACAGATTTGATCCAG | ||
| lacZ38 | AAATAATATCGGTGGCCGTG | ||
| lacZ39 | TTTGATGGACCATTTCGGCA | ||
| lacZ40 | TATTCGCAAAGGATCAGCGG | ||
| lacZ41 | AAGACTGTTACCCATCGCGT | ||
| lacZ42 | TGCCAGTATTTAGCGAAACC | ||
| lacZ43 | AAACGGGGATACTGACGAAA | ||
| lacZ44 | TAATCAGCGACTGATCCACC | ||
| lacZ45 | GGGTTGCCGTTTTCATCATA | ||
| lacZ46 | TCGGCGTATCGCCAAAATCA | ||
| lacZ47 | TTCATACAGAACTGGCGATC | ||
| lacZ48 | TGGTGTTTTGCTTCCGTCAG | ||
| lacZ49 | ACGGAACTGGAAAAACTGCT | 3' mRNA | |
| lacZ50 | TATTCGCTGGTCACTTCGAT | 3' mRNA | |
| lacZ51 | GTTATCGCTATGACGGAACA | 3' mRNA | |
| lacZ52 | TTTACCTTGTGGAGCGACAT | 3' mRNA | |
| lacZ53 | GTTCAGGCAGTTCAATCAAC | 3' mRNA | |
| lacZ54 | TTGCACTACGCGTACTGTGA | 3' mRNA | |
| lacZ55 | AGCGTCACACTGAGGTTTTC | 3' mRNA | |
| lacZ56 | ATTTCGCTGGTGGTCAGATG | 3' mRNA | |
| lacZ57 | ACCCAGCTCGATGCAAAAAT | 3' mRNA | |
| lacZ58 | CGGTTAAATTGCCAACGCTT | 3' mRNA | |
| lacZ59 | CTGTGAAAGAAAGCCTGACT | 3' mRNA | |
| lacZ60 | GGCGTCAGCAGTTGTTTTTT | 3' mRNA | |
| lacZ61 | TACGCCAATGTCGTTATCCA | 3' mRNA | |
| lacZ62 | TAAGGTTTTCCCCTGATGCT | 3' mRNA | |
| lacZ63 | ATCAATCCGGTAGGTTTTCC | 3' mRNA | |
| lacZ64 | GTAATCGCCATTTGACCACT | 3' mRNA | |
| lacZ65 | AGTTTTCTTGCGGCCCTAAT | 3' mRNA | |
| lacZ66 | ATGTCTGACAATGGCAGATC | 3' mRNA | |
| lacZ67 | ATAATTCAATTCGCGCGTCC | 3' mRNA | |
| lacZ68 | TGATGTTGAACTGGAAGTCG | 3' mRNA | |
| lacZ69 | TCAGTTGCTGTTGACTGTAG | 3' mRNA | |
| lacZ70 | ATTCAGCCATGTGCCTTCTT | 3' mRNA | |
| lacZ71 | AATCCCCATATGGAAACCGT | 3' mRNA | |
| lacZ72 | AGACCAACTGGTAATGGTAG | 3' mRNA | |
Referanslar
- Bervoets, I., Charlier, D. Diversity, versatility and complexity of bacterial gene regulation mechanisms: opportunities and drawbacks for applications in synthetic biology. FEMS Microbiology Reviews. 43 (3), 304-339 (2019).
- Epshtein, V., Nudler, E. Cooperation between RNA polymerase molecules in transcription elongation. Science. 300 (5620), 801-805 (2003).
- Vogel, U., Jensen, K. F. The RNA chain elongation rate in Escherichia coli depends on the growth rate. Journal of Bacteriology. 176 (10), 2807-2813 (1994).
- Tennyson, C. N., Klamut, H. J., Worton, R. G. The human dystrophin gene requires 16 hours to be transcribed and is cotranscriptionally spliced. Nature Genetics. 9, 184 (1995).
- Singh, J., Padgett, R. A. Rates of in situ transcription and splicing in large human genes. Nature Structural & Molecular Biology. 16, 1128 (2009).
- Selinger, D. W., Saxena, R. M., Cheung, K. J., Church, G. M., Rosenow, C. Global RNA Half-Life Analysis in Escherichia coli Reveals Positional Patterns of Transcript Degradation. Genome Research. 13 (2), 216-223 (2003).
- Bernstein, J. A., Khodursky, A. B., Lin, P. -. H., Lin-Chao, S., Cohen, S. N. Global analysis of mRNA decay and abundance in Escherichia coli at single-gene resolution using two-color fluorescent DNA microarrays. Proceedings of the National Academy of Sciences. 99 (15), 9697-9702 (2002).
- Pérez-Ortín, J. E., Medina, D. A., Chávez, S., Moreno, J. What do you mean by transcription rate. BioEssays. 35 (12), 1056-1062 (2013).
- Tang, F., et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6 (5), 377-382 (2009).
- Kuchina, A., et al. Microbial single-cell RNA sequencing by split-pool barcoding. BioRxiv. , 869248 (2019).
- Blattman, S. B., Jiang, W., Oikonomou, P., Tavazoie, S. Prokaryotic single-cell RNA sequencing by in situ combinatorial indexing. Nature Microbiology. , (2020).
- Femino, A., Fay, F., Fogarty, K., Singer, R. Visualization of single RNA transcripts in situ. Science. 280 (5363), 585-590 (1998).
- Raj, A., vanden Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877-879 (2008).
- Montero Llopis, P., et al. Spatial organization of the flow of genetic information in bacteria. Nature. 466 (7302), 77-81 (2010).
- So, L. -. h., et al. General properties of transcriptional time series in Escherichia coli. Nature Genetics. 43 (6), 554-560 (2011).
- Taniguchi, Y., et al. Quantifying E. coli proteome and transcriptome with single-molecule sensitivity in single cells. Science. 329 (5991), 533-538 (2010).
- Jones, D. L., Brewster, R. C., Phillips, R. Promoter architecture dictates cell-to-cell variability in gene expression. Science. 346 (6216), 1533-1536 (2014).
- Iyer, S., Park, B. R., Kim, M. Absolute quantitative measurement of transcriptional kinetic parameters in vivo. Nucleic Acids Research. 44 (18), 142 (2016).
- Kim, S., Beltran, B., Irnov, I., Jacobs-Wagner, C. Long-Distance Cooperative and Antagonistic RNA Polymerase Dynamics via DNA Supercoiling. Cell. 179 (1), 106-119 (2019).
- Wang, M., Zhang, J., Xu, H., Golding, I. Measuring transcription at a single gene copy reveals hidden drivers of bacterial individuality. Nature Microbiology. 4 (12), 2118-2127 (2019).
- Joo, C., Ha, T. . Labeling DNA (or RNA) for single-molecule FRET. 2012 (9), 1005-1008 (2012).
- Sambrook, J., Russell, D. W. . Standard Ethanol Precipitation of DNA in Microcentrifuge Tubes. 2006 (1), 4456 (2006).
- Skinner, S. O., Sepúlveda, L. A., Xu, H., Golding, I. Measuring mRNA copy number in individual Escherichia coli cells using single-molecule fluorescent in situ hybridization. Nature Protocols. 8 (6), 1100-1113 (2013).
- Adesnik, M., Levinthal, C. The synthesis and degradation of lactose operon messenger RNA in E. coli. Cold Spring Harbor Symposia on Quantitative Biology. 35, 451-459 (1970).
- Campbell, E. A., et al. Structural mechanism for rifampicin inhibition of bacterial RNA polymerase. Cell. 104 (6), 901-912 (2001).
- Raj, A., Tyagi, S., Walter, N. G. . Methods in Enzymology. 472, 365-386 (2010).
- Sliusarenko, O., Heinritz, J., Emonet, T., Jacobs-Wagner, C. High-throughput, subpixel precision analysis of bacterial morphogenesis and intracellular spatio-temporal dynamics. Molecular Microbiology. 80 (3), 612-627 (2011).
- Paintdakhi, A., et al. Oufti: an integrated software package for high-accuracy, high-throughput quantitative microscopy analysis. Molecular Microbiology. 99 (4), 767-777 (2016).
- Moffitt, J. R., Zhuang, X., Filonov, G. S., Jaffrey, S. R. . Methods in Enzymology. 572, 1-49 (2016).
- Yu, J., Xiao, J., Ren, X., Lao, K., Xie, X. S. Probing gene expression in live cells, one protein molecule at a time. Science. 311 (5767), 1600-1603 (2006).
- Chen, H., Shiroguchi, K., Ge, H., Xie, X. S. Genome-wide study of mRNA degradation and transcript elongation in Escherichia coli. Molecular Systems Biology. 11 (1), 781 (2015).
- Vogel, U., Sørensen, M., Pedersen, S., Jensen, K. F., Kilstrup, M. Decreasing transcription elongation rate in Escherichia Coli exposed to amino acid starvation. Molecular Microbiology. 6 (15), 2191-2200 (1992).
- Yang, S., et al. Transcription and translation contribute to gene locus relocation to the nucleoid periphery in E. coli. Nature Communications. 10 (1), 5131 (2019).
- Zenklusen, D., Larson, D. R., Singer, R. H. Single-RNA counting reveals alternative modes of gene expression in yeast. Nature Structural & Molecular Biology. 15 (12), 1263-1271 (2008).
- Fontenete, S., Guimarães, N., Wengel, J., Azevedo, N. F. Prediction of melting temperatures in fluorescence in situ hybridization (FISH) procedures using thermodynamic models. Critical Reviews in Biotechnology. 36 (3), 566-577 (2016).
- Sepúlveda, L. A., Xu, H., Zhang, J., Wang, M., Golding, I. Measurement of gene regulation in individual cells reveals rapid switching between promoter states. Science. 351 (6278), 1218-1222 (2016).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-Dimensional Super-Resolution Imaging by Stochastic Optical Reconstruction Microscopy. Science. 319 (5864), 810-813 (2008).
- Moffitt, J. R., Pandey, S., Boettiger, A. N., Wang, S., Zhuang, X. Spatial organization shapes the turnover of a bacterial transcriptome. eLife. 5, 13065 (2016).
- Fei, J., et al. Determination of in vivo target search kinetics of regulatory noncoding RNA. Science. 347 (6228), 1371-1374 (2015).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır