A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
عزل الخلايا البطانية الشبكية للجيل التالي
In This Article
Summary
يصف هذا البروتوكول طريقة لعزل الخلايا البطانية الشبكية بعد الولادة للفئران المحسنة لإنتاجية الخلايا ونقاوتها وقابليتها للحياة. هذه الخلايا مناسبة لنهج تسلسل الجيل التالي.
Abstract
أدت التحسينات الأخيرة في تسلسل الجيل التالي إلى تطوير معرفة الباحثين بالبيولوجيا الجزيئية والخلوية ، مع العديد من الدراسات التي كشفت عن نماذج جديدة في بيولوجيا الأوعية الدموية. يتطلب تطبيق هذه الأساليب على نماذج تطور الأوعية الدموية تحسين تقنيات عزل الخلايا من الأنسجة الجنينية وما بعد الولادة. يجب أن يكون كل من إنتاجية الخلية وقابليتها للحياة والنقاء الأقصى للحصول على نتائج دقيقة وقابلة للتكرار من مناهج التسلسل من الجيل التالي. يستخدم الباحثون نموذج الأوعية الدموية لشبكية العين للفأر الوليدي لدراسة آليات تطور الأوعية الدموية. استخدم الباحثون هذا النموذج للتحقيق في آليات تكوين الأوعية الدموية وتحديد مصير الشرايين الوريدية أثناء تكوين الأوعية الدموية ونضجها. يتطلب تطبيق تقنيات تسلسل الجيل التالي لدراسة نموذج تطور الأوعية الدموية في شبكية العين تحسين طريقة لعزل الخلايا البطانية الشبكية التي تزيد من إنتاجية الخلايا وصلاحيتها ونقاوتها. يصف هذا البروتوكول طريقة لعزل أنسجة شبكية الفئران والهضم والتنقية باستخدام فرز الخلايا المنشط بالفلورة (FACS). تشير النتائج إلى أن مجموعة الخلايا البطانية CD31 + / CD45 المنقاة ب FACS غنية للغاية للتعبير الجيني للخلايا البطانية ولا تظهر أي تغيير في الصلاحية لمدة 60 دقيقة بعد FACS. وشملت النتائج التمثيلية لنهج تسلسل الجيل التالي على الخلايا البطانية المعزولة باستخدام هذه الطريقة، بما في ذلك تسلسل الحمض النووي الريبي السائب وتسلسل الحمض النووي الريبي أحادي الخلية، مما يدل على أن هذه الطريقة لعزل الخلايا البطانية في شبكية العين متوافقة مع تطبيقات تسلسل الجيل التالي. ستسمح هذه الطريقة لعزل الخلايا البطانية في شبكية العين بتقنيات تسلسل متقدمة للكشف عن آليات جديدة لتطور الأوعية الدموية.
Introduction
إن القدرة الإنتاجية العالية لتسلسل الأحماض النووية عبر مناهج التسلسل من الجيل التالي قد عززت بشكل كبير معرفة الباحثين بالبيولوجيا الجزيئية والخلوية. تشمل هذه التقنيات المتقدمة تسلسل الحمض النووي الريبي للنسخ الكامل ، وتسلسل الحمض النووي للمناطق المستهدفة لتحديد تعدد أشكال النوكليوتيدات المفردة (SNPs) ، وتسلسل الحمض النووي لعوامل النسخ المرتبطة في تسلسل الترسيب المناعي للكروماتين (ChIP) ، أو مناطق الكروماتين المفتوحة في الفحص لتسلسل الكروماتين الذي يمكن الوصول إليه بواسطة Transposase (ATAC) ، وتسلسل الحمض النووي الريبي أحادي الخلية1 . في بيولوجيا الأوعية الدموية ، سمحت هذه التطورات للباحثين بتوضيح الآليات المعقدة للتطور والمرض ، إلى جانب تمييز أنماط التعبير الجيني على طول سلسلة متصلة من الأنماط الظاهرية المختلفة 2,3. يمكن للتجارب المستقبلية تحديد الآليات المعقدة بشكل أكبر من خلال الجمع بين تسلسل الجيل التالي والنماذج المقيمة لتطور الأوعية الدموية ، ولكن يجب أن تكون طرق تحضير العينات متوافقة مع تقنيات التسلسل المتقدمة.
تعتمد جودة ودقة وقابلية استنساخ مناهج التسلسل من الجيل التالي على طريقة تحضير العينة. عند عزل مجموعة فرعية من الخلايا أو توليد معلقات أحادية الخلية من الأنسجة ، فإن طرق الهضم والتنقية المثلى ضرورية لزيادة عدد الخلايا وصلاحيتها ونقاوتها 4,5. يتطلب ذلك توازنا في طريقة الهضم: الهضم القوي ضروري لتحرير الخلايا من الأنسجة والحصول على خلايا كافية لنهج المصب ، لكن صلاحية الخلية ستتأثر سلبا إذا كان الهضم قويا جدا 6,7. بالإضافة إلى ذلك ، يعد نقاء عدد الخلايا ضروريا للحصول على نتائج قوية وتحليل دقيق للبيانات ، والتي يمكن تحقيقها من خلال FACS. هذا يسلط الضوء على أهمية تحسين طرق عزل الخلايا لتطبيق تسلسل الجيل التالي على النماذج الراسخة لتطور الأوعية الدموية.
نموذج مميز جيدا للتحقيق في تطور الأوعية الدموية هو نموذج تطوير الأوعية الدموية في شبكية العين. يتطور الأوعية الدموية الشبكية للفأر بعد الولادة في ضفيرة سطحية ثنائية الأبعاد ، مع ظهور نشوء وعائي أولي من العصب البصري في يوم ما بعد الولادة (P) 3 ، وجبهة وعائية مع خلايا ساق وطرف ونضوج أولي للأوعية مرئية عند P6 ، ونضوج الضفيرة الوعائية مرئية بعد P9 8,9. أثناء إعادة تشكيل الضفيرة الوعائية الأولية ، تخضع الخلايا البطانية لمواصفات نحو الأنماط الظاهرية الشريانية والشعيرية والوريدية في أوعية مختلفة لتوليد شبكة الدورة الدموية10,11. لذلك ، تسمح هذه الطريقة للباحثين بتصور تكوين الضفيرة الوعائية الوعائية والمواصفات البطانية الشريانية الوريدية والنضج في نقاط زمنية مختلفة أثناء التطور9. بالإضافة إلى ذلك ، يوفر هذا النموذج طريقة للتحقيق في آثار التلاعب المعدل وراثيا على تكوين الأوعية الدموية وتطور الضفيرة الوعائية ، والتي تم تطبيقها للتحقيق في تطور الأوعية الدموية ، والتشوهات الشريانية الوريدية ، والأوعية الدموية الجديدة التي يسببها الأكسجين12،13،14،15،16 . من أجل الجمع بين نهج تسلسل الجيل التالي مع نموذج تطوير الأوعية الدموية في شبكية العين ، من الضروري وجود بروتوكول محسن لعزل الخلايا البطانية من أنسجة الشبكية.
يصف هذا البروتوكول طريقة محسنة لهضم أنسجة الشبكية من الفئران في P6 لزيادة إنتاجية الخلايا ونقاوتها وقابليتها للحياة. يتم عزل أنسجة الشبكية من الفئران P6 ، ويتم هضمها لمدة 20 دقيقة ، وصبغها المناعي ل CD31 و CD45 ، وتنقيتها من خلال FACS لعزل تعليق خلية واحدة من الخلايا البطانية في حوالي 2.5 ساعة (الشكل 1 أ). تم العثور على هذه الخلايا البطانية للحفاظ على قابلية عالية للحياة لمدة 60 دقيقة بعد العزل17 ، مما يسمح للمكتبة الاستعدادات لطرق تسلسل الجيل التالي. بالإضافة إلى ذلك ، يتم توفير نتائج تمثيلية لبوابة FACS ونتائج مراقبة الجودة من طريقتين منفصلتين لتسلسل الجيل التالي باستخدام بروتوكول العزل هذا: تسلسل الحمض النووي الريبي الكامل للنسخ وتسلسل الحمض النووي الريبي أحادي الخلية. تسمح هذه الطريقة باستخدام مناهج تسلسل الجيل التالي جنبا إلى جنب مع نموذج الأوعية الدموية في شبكية العين لتوضيح آليات جديدة لتطور الأوعية الدموية.
Protocol
وافقت لجان رعاية واستخدام الحيوان المؤسسية في جامعة ييل وجامعة فيرجينيا على جميع التجارب على الحيوانات المدرجة في هذا البروتوكول.
1. الحصول على عيون الفأر لعزل الشبكية
- قم بإعداد 1x PBS بارد وأضف 500 ميكرولتر إلى كل بئر من لوحة 48 بئر.
- القتل الرحيم للفئران حديثي الولادة في اليوم السادس بعد الولادة (P6) وفقا للمبادئ التوجيهية المؤسسية المعتمدة. في هذه التجربة ، يتم القتل الرحيم لحوالي 4-8 فئران حديثي الولادة في P6 عن طريق استنشاق الأيزوفلوران لمدة ثلاث دقائق على الأقل بعد توقف التنفس ، يليه قطع الرأس.
- إزالة العينين من كل من الفئران. قطع الجلد والغشاء فوق العين عن طريق قطع عمودي على الجفن باستخدام مقص التشريح. بعد ذلك ، استخدم الملقط للضغط برفق فوق وأسفل العين بحيث تتحرك العين خارج التجويف.
- قرصة بعناية تحت العين مع ملقط وقطع العصب البصري الذي يبقي العين متصلة. بعد ذلك ، ضع كل عين في صفيحة 48 بئرا في برنامج تلفزيوني بارد 1x محضر حتى الانتهاء من الحصاد.
ملاحظة: استخدم بئرا واحدا لكل ماوس، مع وجود كلتا العينين من ماوس واحد في نفس البئر.
- قرصة بعناية تحت العين مع ملقط وقطع العصب البصري الذي يبقي العين متصلة. بعد ذلك ، ضع كل عين في صفيحة 48 بئرا في برنامج تلفزيوني بارد 1x محضر حتى الانتهاء من الحصاد.
2. عزل أنسجة شبكية الفأر
- املأ طبق بتري ، المبطن بوسادة تشريح في الأسفل ، ب 500 ميكرولتر من 1x PBS بارد الجليد لغمر العينين ووضعه تحت مجهر تشريح مضبوط على تكبير 4.0x.
- العينين في وسادة التشريح باستخدام ماصة نقل ذات طرف عريض حتى لا تتلف العينين.
ملاحظة: تأكد من تغطية العينين بالكامل ببرنامج تلفزيوني.
- العينين في وسادة التشريح باستخدام ماصة نقل ذات طرف عريض حتى لا تتلف العينين.
- باستخدام ملقطين تشريح دقيقين ، أمسك العصب البصري بأحد الملقط لتحقيق الاستقرار ، ومع الملقط الثاني يخترق ثقبا عبر الغرفة الأمامية حيث تتصل القرنية والصلبة (الشكل 1 ب). تمزيق الثقب في دائرة حوالي 75 ٪ من الطريق حول القرنية.
- بينما لا تزال تمسك العصب البصري بأحد الملقط ، استخدم الملقط الثاني لتمزيق الصلبة برفق من أنسجة الشبكية.
- على النحو الوارد أعلاه ، استخدم الملقط الثاني لتمزيق الجسم الزجاجي لأنسجة الشبكية برفق.
- مرة أخرى ، باستخدام الملقط الثاني ، قم بإزالة العدسة وأوعية الضفيرة الهيالويد برفق والتي لم تؤتي ثمارها عند إزالة الجسم الزجاجي. للقيام بذلك ، قم بالوصول إلى شبكية العين باستخدام ملقط مفتوح حتى تلمس شبكية العين تقريبا ، وأغلق الملقط للاستيلاء على العدسة وأوعية الضفيرة الهيالويد ، واسحب الملقط من شبكية العين. يمكن تكرار ذلك حتى تتم إزالة جميع الأوعية.
ملاحظة: تشبه الضفيرة الهيالويد بنية واضحة تشبه الويب.
- املأ أنابيب الطرد المركزي الدقيقة سعة 2 مل ب 500 ميكرولتر من 1x PBS المثلج وضع شبكية العين في الأنابيب. اعزل جميع شبكية العين عن العينين وضعها في برنامج تلفزيوني بارد قبل المضي قدما.
ملاحظة: استخدم أنبوبا واحدا لكل ماوس، مع شبكيتي عين من نفس الماوس تتشاركان أنبوبا. إذا تمزق نسيج الشبكية ، فقم بنقله إلى قطع متعددة. يجب أن يستغرق الوقت الإجمالي لتشريح أنسجة الشبكية لفضلات الفئران 15-60 دقيقة ، بدءا من القتل الرحيم وينتهي بعزل أنسجة الشبكية النهائي.
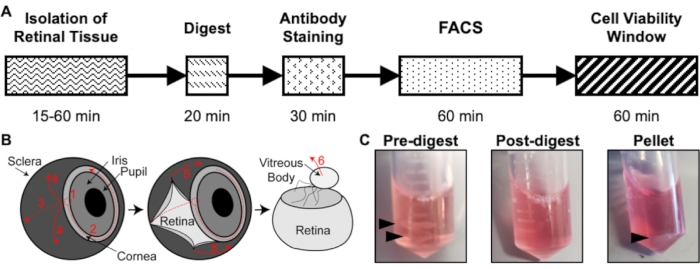
الشكل 1: نظرة عامة على بروتوكول العزل. (أ) رسم تخطيطي للجدول الزمني للعزل مع الوقت المقدر لكل خطوة: عزل أنسجة الشبكية ، والهضم ، وتلطيخ الأجسام المضادة ، و FACS ، ونافذة صلاحية الخلية. (ب) دليل خطوة بخطوة لعزل أنسجة الشبكية عن العين ، مع خطوات تشريح مرقمة: 1) ثقب القرنية ، 2) القرنية المسيل للدموع ، 3) الصلبة المسيلة للدموع ، 4) إزالة الصلبة من الشبكية ، 5) إزالة الصلبة والأنسجة المتصلة من الشبكية ، 6) إزالة الجسم الزجاجي والأوعية الزجاجية من الشبكية. (ج) صور تمثيلية لأنسجة الشبكية خلال خطوات الهضم المختلفة: ما قبل الهضم ، ما بعد الهضم ، بيليه (الأسهم السوداء تسلط الضوء على أنسجة الشبكية أو حبيبات الخلية). أعيد نشره بإذن من Chavkin et al. نشر في S. Karger AG ، بازل17. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
3. هضم أنسجة الشبكية في تعليق خلية واحدة
- تحضير محلول هضم 500 ميكرولتر لكل شبكيتين معزولتين. أضف FBS وكولاجيناز من النوع الثاني إلى DMEM إلى تركيز نهائي بنسبة 10٪ FBS و 1 مجم / مل كولاجيناز من النوع الثاني. اخلطي المحلول ودفئيه إلى 37 درجة مئوية باستخدام حمام مائي.
- قم بإزالة PBS الزائد من أنابيب الطرد المركزي الدقيقة سعة 2 مل عن طريق سحب العينات بعناية. اترك ما يكفي من PBS لتغطية أنسجة الشبكية بالكامل ، حوالي 100 ميكرولتر في كل أنبوب.
- أضف 500 ميكرولتر من محلول الهضم إلى كل أنبوب به نسيج شبكي (الشكل 1 ج).
- استخدم ماصة P1000 وطرف ماصة لتحريك أنسجة الشبكية لأعلى ولأسفل في محلول الهضم خمس مرات.
- احتضن خليط الهضم في حمام مائي على حرارة 37 درجة مئوية لمدة 20 دقيقة.
- استخدم ماصة P1000 وطرف ماصة لماصة خليط الهضم لأعلى ولأسفل كل 5 دقائق. بعد الحضانة ، يذوب نسيج الشبكية في تعليق خلية واحدة ، لذلك يجب أن يكون خليط الهضم غائما (الشكل 1C).
4. عد الخلايا
- ضع جهاز طرد مركزي منضدية عند 4 درجات مئوية وضع أنابيب خليط الهضم بالداخل. حبيبات خليط الهضم عن طريق الطرد المركزي في 375 × غرام لمدة 5 دقائق.
- قم بإزالة محلول الهضم الطافي بعناية عن طريق سحب العينات. لا تزعج بيليه الخلية (الشكل 1C). أعد تعليق الحبيبات في 500 ميكرولتر من الثلج البارد 1x PBS عن طريق الخلط برفق لأعلى ولأسفل مع الماصة.
- عد الخلايا باستخدام مقياس الدم تحت المجهر.
ملاحظة: يبلغ عدد الخلايا حوالي 1 × 106 خلايا لكل شبكيتين. - تأكد من إعادة تعليق الخلايا بشكل صحيح عن طريق الخلط اللطيف ، ثم توزيع 20 ميكرولتر من تعليق الخلية في ثلاثة أنابيب للتحكم في تلطيخ (الأنبوب 1: التحكم في IgG ، الأنبوب 2: التحكم في CD31 ، والأنبوب 3: التحكم CD45) ، كما هو موضح ومستخدم سابقا17,18.
- ضع جهاز طرد مركزي منضدية عند 4 درجات مئوية وضع أنابيب تحتوي على خلايا بالداخل. حبيبات الخلايا عن طريق الطرد المركزي في 375 × غرام لمدة 5 دقائق.
5. المناعية الخلايا مع الأجسام المضادة
- تحضير 100 ميكرولتر من المخزن المؤقت للتلطيخ لكل شبكيتين ملطختين بالإضافة إلى أربعة أنابيب تحكم. أضف FBS و HEPES و D-Glucose إلى محلول HBSS إلى تركيز نهائي قدره 10٪ FBS و 10 mM HEPES و 1 مجم / مل D-Glucose.
- الحصول على الأجسام المضادة المترافقة بالفلورسنت ضد CD31 و CD45. أضف الأجسام المضادة بتخفيف 1: 100 في المخزن المؤقت للتلطيخ (الأنبوب 1: الجسم المضاد IgG فقط ، الأنبوب 2: الجسم المضاد CD31 فقط ، والأنبوب 3: الجسم المضاد CD45 فقط). أضف 1 ميكرولتر من الجسم المضاد لكل 100 ميكرولتر من محلول التلوين للحصول على تركيز نهائي للجسم المضاد يبلغ 2 ميكروغرام / مل.
- قم بإزالة PBS بعناية من حبيبات الخلية المغسولة عن طريق سحب العينات.
- أعد تعليق الحبيبات في 100 ميكرولتر من محلول تلطيخ الأجسام المضادة لكل 0.5 × 106 خلايا.
- احتضان المعلق أحادي الخلية بمحلول تلطيخ الأجسام المضادة لمدة 30 دقيقة على الثلج في الظلام. اضغط على الأنابيب كل 10 دقائق لخلط الخلايا برفق.
6. الاستعداد لفرز الخلايا المنشط بالتألق
- ضع جهاز طرد مركزي منضدية عند 4 درجات مئوية وضع الأنابيب التي تحتوي على خلايا بالداخل. خلايا بيليه بالطرد المركزي عند 375 × جم لمدة 5 دقائق.
- قم بإزالة المادة الطافية وأعد تعليق هذه الخلايا في 500 ميكرولتر من 1x PBS لغسلها.
- ضع جهاز طرد مركزي منضدية عند 4 درجات مئوية وضع الأنابيب التي تحتوي على خلايا بالداخل. خلايا بيليه بالطرد المركزي عند 375 × جم لمدة 5 دقائق
- قم بإعداد 1.5 مل من المخزن المؤقت FACS (1٪ FBS في 1x PBS).
- قم بإزالة المادة الطافية وأعد تعليق حبيبات الخلية في 300 ميكرولتر من FACS Buffer عن طريق الخلط برفق مع ماصة.
- يضاف يوديد البروبيديوم (PI) إلى أنابيب العينة التي تحتوي على مخزن FACS المؤقت إلى تركيز نهائي قدره 0.5 ميكروغرام / مل. يستخدم PI كعلامة جدوى.
- انقل ودمج معلقات الخلية في أنابيب اختبار سعة 5 مل من خلال غطاء مصفاة خلوية. يجب أن يحتوي غطاء مصفاة الخلية على مرشح 35 ميكرومتر ، ويمكن سحب معلقات الخلية مباشرة على الفلتر.
- احتفظ بأنابيب FACS على الجليد في الظلام وانقلها إلى فارز الخلايا.
7. إعداد أداة نظام مراقبة الأصول الميدانية
- قم بتركيب فوهة 100 ميكرومتر في جهاز FACS.
ملاحظة: تقلل فوهة الحجم هذه من حجم التجميع وتزيد من كثافة الخلايا المعزولة. - قم بإعداد 250 ميكرولتر من 1x PBS في أنبوب طرد مركزي دقيق سعة 1.5 مل كأنبوب تجميع ليتم تثبيته في أداة FACS.
8. عزل الخلايا البطانية القابلة للحياة عبر FACS
- قم بتشغيل أداة FACS والكمبيوتر. انقر على أيقونة برنامج FACS لفتح برنامج FACS على الكمبيوتر لتشغيل وتشغيل أداة FACS. إجراء اختبارات مراقبة الجودة الروتينية.
- قم بتحميل العينات الملطخة بالتحكم في أداة FACS لضبط المحاور. ابدأ بخلايا الأجسام المضادة IgG فقط لتوليد وضبط FSC و SSC ، ثم الأجسام المضادة CD31 فقط لتوليد وضبط CD31 ، ثم الأجسام المضادة CD45 فقط لتوليد وضبط CD45. تسجيل البيانات من عينات التحكم.
- قم بتحميل خلايا العينة الملطخة في أداة FACS وقم بتشغيل العينة.
- قم ببوابة الخلايا بناء على معلمات التشتت الأمامي والتشتت الجانبي (FSC-A و SSC-A ، على التوالي) (الشكل 2 أ).
ملاحظة: يتم استخدام معلمات FSC-A و SSC-A لتحديد الخلايا بناء على الحجم والكثافة والدقة وخصائص السطح ومعامل الانكسار. - خلايا البوابة بواسطة FSC-A و FSC-H لتحديد الخلايا المزدوجة وجمع الخلايا المفردة فقط (الشكل 2 أ).
ملاحظة: يتم استخدام معلمات FSC-A و FSC-H لتحديد خلايا مفردة بناء على مبدأ أنه أثناء التشتت الأمامي ، ستكون ثنائيات الخلية عبارة عن قطرات تحتوي على نسبة مساحة إلى ارتفاع أكبر. - خلايا البوابة بواسطة PI و SSC-A لتحديد الخلايا القابلة للحياة (الشكل 2 أ).
ملاحظة: ستكون الخلايا القابلة للحياة سلبية PI. - اصنع بوابة CD31 + / CD45 مع عناصر تحكم وخلايا بوابة بواسطة CD31 و CD45 (الشكل 2 أ ، ب).
- أدخل أنبوب تجميع مع 250 ميكرولتر من المخزن المؤقت FACS.
- ابدأ في فرز الخلايا التي تكون CD31 إيجابية / CD45 سلبية في أنبوب التجميع المثبت.
- احتفظ بأنابيب التجميع على الجليد لمزيد من التحليل. يمكن معالجة العينات لتطبيقات التسلسل من الجيل التالي في هذه الخطوة1.
9. إجراء فحص الجدوى
- امزج 10 ميكرولتر من الخلايا مع 10 ميكرولتر من محلول تريبان الأزرق 0.4٪ لتقييم صلاحية الخلية.
- عد الخلايا القابلة للحياة عن طريق سحب الخليط الملون على شريحة مقياس الدم. ضع الشريحة تحت المجهر للحصول على عدد دقيق من الجدوى. عد الخلايا الزرقاء على أنها غير قابلة للحياة وعد الخلايا الواضحة على أنها قابلة للحياة.
- اقسم عدد الخلايا القابلة للحياة على إجمالي عدد الخلايا لحساب النسبة المئوية للصلاحية.
10. إجراء فحص التعبير الجيني
- الحصول على 10000-20000 خلية لكل عينة وإجراء عزل الحمض النووي الريبي لتحليل التعبير الجيني للخلايا البطانية لشبكية العين. يتم إجراء عزل الحمض النووي الريبي باستخدام مجموعة متاحة تجاريا تستخدم ترسيب الحمض النووي الريبي القائم على الإيثانول ، ثم أعمدة الطرد المركزي والأغشية لغسل وتخفيف الحمض النووي الريبي المنقى من محللة الخلية. يمكن معالجة العينات لبعض تطبيقات التسلسل من الجيل التالي في هذه الخطوة1.
- تحويل الحمض النووي الريبي إلى cDNA باستخدام إنزيم النسخ العكسي والكواشف الداعمة19.
- تحديد كمي باستخدام صبغة ربط الحمض النووي وآلة PCR الكمية (qPCR)20. قم بتحميل عينات cDNA في لوحة qPCR بسعة 384 بئرا مع مواد أولية لتضخيم الجينات ذات الأهمية. يجب أن يصل حجم التفاعل إلى 10 ميكرولتر ، بما في ذلك صبغة ربط الحمض النووي ، والتي سيتم استخدامها لتحديد تضخيم الجينات.
- استخدم البادئات الأمامية والخلفية CD31 و VE-Cadherin و CD45 و β-actin لقياس التعبير الجيني.
- استخدم العينة لتطبيقات تسلسل الجيل التالي1.
النتائج
يؤدي هضم أنسجة الشبكية والتلطيخ المناعي ل CD31 و CD45 إلى مجموعة محددة من الخلايا البطانية CD31 + / CD45 بعد بوابة الخلايا والخلايا المفردة والقدرة على البقاء (الشكل 2 أ). مطلوب تلطيخ مناعي CD45 للقضاء على خلايا CD31 + / CD45 + ، والتي تشمل الصفائح الدموية وبعض الكريات البيض21. يج?...
Discussion
يصف هذا البروتوكول طريقة لعزل الخلايا البطانية من أنسجة شبكية الفئران بعد الولادة والتي تم تحسينها للحصول على عدد خلايا مرتفع ونقاوة وقابلية للحياة. يتم الحصول على نقاء الخلية عن طريق عزل FACS لمجموعات الخلايا البطانية من معلق الخلية الواحدة المهضوم بواسطة CD31 + / CD45- التلوين المناعي. يتم قيا...
Disclosures
ليس لدى المؤلفين إفصاحات ذات صلة.
Acknowledgements
شكرا لمرفق قياس التدفق الخلوي بجامعة ييل ، والمرفق الأساسي لقياس التدفق الخلوي بجامعة فيرجينيا ، ومركز ييل للتحليل الجينومي ، وجامعة فيرجينيا لتحليل الجينوم والتكنولوجيا الأساسية على جهودهم وخبراتهم ونصائحهم في المساهمة في التجارب المقدمة. تم تمويل هذه الدراسة من خلال منح المعاهد الوطنية للصحة إلى N.W.C. (T32 HL007224 ، T32 HL007284) ، SC (T32 HL007284) ، KW (R01 HL142650) ، و K.K.H. (R01 HL146056 ، R01 DK118728 ، UH3 EB025765).
Materials
| Name | Company | Catalog Number | Comments |
| 2 mL Eppendorf safe-lock tubes | USA Scientific | 4036-3352 | |
| 5 ml Falcon Test Tubes with Cell Strainer Snap Cap | Corning | 352235 | |
| 60 mm Non TC-treated Culture Dish | Corning | 430589 | |
| APC Rat Anti-Mouse CD31 | BD Biosciences | 551262 | |
| APC Rat IgG2a κ Isotype Control | BD Biosciences | 553932 | |
| BD FACSChorus Software | BD Biosciences | FACSCHORUS | |
| BD FACSMelody Cell Sorter | BD Biosciences | FACSMELODY | |
| Collagenase Type II | Sigma-Aldrich | 234115 | |
| Costar 48-well Clear TC-treated Multiple Well Plates, Individually Wrapped, Sterile | Corning | 3548 | |
| D-Glucose | Gibco | A2494001 | |
| Disposable Graduated Transfer Pipettes | Fisher Scientific | 12-711-9AM | |
| Dissecting Pan Wax | Carolina | 629100 | |
| Dissection scissors | Fine Science Tools | 14085-08 | |
| Dissection Stereo Microscope M165 FC | Leica | M165FC | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-052 | |
| Dulbecco’s Phosphate Buffered Saline (PBS) | Gibco | 14190144 | |
| Eppendorf Flex-Tubes Microcentrifuge Tubes 1.5 mL | Sigma-Aldrich | 22364120 | |
| Fetal Bovine Serum (FBS) | Gemini Bio | 100-106 | |
| Fine dissection forceps | Fine Science Tools | 11250-00 | |
| Hank's Buffered Salt Solution (HBSS) | Gibco | 14175095 | |
| HEPES (1M) | Gibco | 15630130 | |
| iScript cDNA Synthesis Kit | Bio-Rad | 1708890 | |
| Isoflurane, USP | Covetrus | 11695067772 | |
| Isotemp General Purpose Deluxe Water Bath | Fisher Scientific | FSGPD20 | |
| Primer: ActB_Forward: 5’- agagggaaatcgtgcgtgac -3’ | Integrated DNA Technologies | N/A | |
| Primer: ActB_Reverse: 5’- caatagtgatgacctggccgt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Forward: 5’- gagcccaatcacgtttcagttt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Reverse: 5’- tccttcctgcttcttgctagct -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Forward: 5’- gggttgttctgtgccttgtt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Reverse: 5’- ctggacggacacagttagca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Forward: 5’- tcctctgcatcctcactatcaca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Reverse: 5’- gtaagtgaccaactgctcgtgaat -3’ | Integrated DNA Technologies | N/A | |
| Propidium iodide | Sigma-Aldrich | P4864 | |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| Sorvall Legend Micro 21R Centrifuge, Refrigerated | ThermoFisher | 75002477 | |
| SYBR-Green iTaq Universal SYBR Green Supermix | Bio-Rad | 172-5120 | |
| Trypan Blue Solution | ThermoFisher | 15250061 | |
| V450 Rat Anti-Mouse CD45 | BD Biosciences | 560501 | |
| V450 Rat IgG2b, κ Isotype Control | BD Biosciences | 560457 |
References
- Slatko, B. E., Gardner, A. F., Ausubel, F. M. Overview of next-generation sequencing technologies. Current Protocols in Molecular Biology. 122 (1), 59 (2018).
- Chavkin, N. W., Hirschi, K. K. Single cell analysis in vascular biology. Frontiers in Cardiovascular Medicine. 7, 42 (2020).
- Ma, F., Hernandez, G. E., Romay, M., Iruela-Arispe, M. L. Single-cell RNA sequencing to study vascular diversity and function. Current Opinion in Hematology. 28 (3), 221-229 (2021).
- Potter, A. S., Potter, S. S., S, Dissociation of tissues for single-cell analysis. Methods in Molecular Biology. 1926, 55-62 (2019).
- Braga, F. A. V., Miragaia, R. J. Tissue handling and dissociation for single-cell RNA-seq. Single Cell Methods: Methods in Molecular Biology. 1979, 9-21 (2019).
- Brink, S. C., et al. Single-cell sequencing reveals dissociatin-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Skulska, K., Wegrzyn, A. S., Chelmonska-Soyta, A., Chodaczek, G. Impact of tissue enzymatic digestion on analysis of immune cells in mouse reproductive mucosa with a focus on gammadelta T cells. Journal of Immunological Methods. 474, 112665 (2019).
- Connolly, S., Hores, T., Smith, L., D'Amore, P. Characterization of vascular development in the mouse retina. Microvascular Research. 36 (3), 275-290 (1988).
- Crist, A., Young, C., Meadows, S. Characterization of arteriovenous identity in the developing neonate mouse retina. Gene Expression Patterns: GEP. 23-24, 22-31 (2017).
- dela Paz, N. G., D'Amore, P. A. Arterial versus venous endothelial cells. Cell and Tissue Research. 335 (1), 5-16 (2009).
- Fang, J. S., Hirschi, K. K. Molecular regulation of arteriovenous endothelial cell specification. F1000Res. 8, (2019).
- Smith, L. E., et al. Oxygen-induced retinopathy in the mouse. Investigative Ophthalmology and Visual Science. 35 (1), 101-111 (1994).
- Pitulescu, M. E., Schmidt, I., Benedito, R., Adams, R. H. Inducible gene targeting in the neonatal vasculature and analysis of retinal angiogenesis in mice. Nature Protocols. 5 (9), 1518-1534 (2010).
- Ruiz, S., et al. A mouse model of hereditary hemorrhagic telangiectasia generated by transmammary-delivered immunoblocking of BMP9 and BMP10. Scientific Reports. 6, 37366 (2016).
- Fang, J. S., et al. Shear-induced Notch-Cx37-p27 axis arrests endothelial cell cycle to enable arterial specification. Nature Communication. 8 (1), 2149 (2017).
- Ola, R., et al. SMAD4 prevents flow induced arterial-venous malformations by inhibiting Casein Kinase 2. Circulation. 138 (21), 2379-2394 (2018).
- Chavkin, N. W., Walsh, K., Hirschi, K. K. Isolation of highly purified and viable retinal endothelial cells. Journal of Vascular Research. 58 (1), 49-57 (2020).
- Hulspas, R., O'Gorman, M. R. G., Wood, B. L., Gratama, J. W., Sutherland, D. R. Considerations for the control of background fluorescence in clinical flow cytometry. Cytometry PartB: Clinical Cytometry. 76 (6), 355-364 (2009).
- Bachman, J. Reverse-transcription PCR (RT-PCR). Methods in Enzymology. 530, 67-74 (2013).
- Green, M. R., Sambrook, J. Quantification of RNA by real-time Reverse Transcription-Polymerase Chain Reaction (RT-PCR). Cold Spring Harbour Protocols. 2018 (10), (2018).
- Liu, L., Shi, G. P. CD31: beyond a marker for endothelial cells. Cardiovascular Research. 94 (1), 3-5 (2012).
- Zarkada, G., et al. Specialized endothelial tip cells guide neuroretina vascularization and blood-retina-barrier formation. Developmental Cell. 56 (15), 2237-2251 (2021).
- Su, X., Sorenson, C. M., Sheibani, N. Isolation and characterization of murine retinal endothelial cells. Molecular Vision. 9, 171-178 (2003).
- Benedito, R., et al. The notch ligands Dll4 and Jagged1 have opposing effects on angiogenesis. Cell. 137 (6), 1124-1135 (2009).
- Daneman, R., et al. Wnt/beta-catenin signaling is required for CNS, but not non-CNS, angiogenesis. Proceedings of the National Academy of Sciences of the United States of America. 106 (2), 641-646 (2009).
- Okabe, K., et al. Neurons limit angiogenesis by titrating VEGF in retina. Cell. 159 (3), 584-596 (2014).
- Crist, A. M., Lee, A. R., Patel, N. R., Westhoff, D. E., Meadows, S. M. Vascular deficiency of Smad4 causes arteriovenous malformations: a mouse model of Hereditary Hemorrhagic Telangiectasia. Angiogenesis. 21 (2), 363-380 (2018).
- Kim, Y. H., Choe, S. W., Chae, M. Y., Hong, S., Oh, S. P. SMAD4 deficiency leads to development of arteriovenous malformations in neonatal and adult mice. Journal of the American Heart Association. 7 (21), 009514 (2018).
- Ma, W., et al. Absence of TGFbeta signaling in retinal microglia induces retinal degeneration and exacerbates choroidal neovascularization. eLife. 8, 42049 (2019).
- Luo, W., et al. Arterialization requires the timely suppression of cell growth. Nature. 589 (7842), 437-441 (2021).
- Lawson, N. D., Vogel, A. M., Weinstein, B. M. sonic hedgehog and vascular endothelial growth factor act upstream of the Notch pathway during arterial endothelial differentiation. Developmental Cell. 3 (1), 127-136 (2002).
- Larrivee, B., et al. ALK1 signaling inhibits angiogenesis by cooperating with the Notch pathway. Developmental Cell. 22 (3), 489-500 (2012).
- Wythe, J. D., et al. ETS factors regulate Vegf-dependent arterial specification. Developmental Cell. 26 (1), 45-58 (2013).
- Davis, D. M., Purvis, J. E. Computational analysis of signaling patterns in single cells. Seminars in Cell and Developmental Biology. , 35-43 (2015).
- Gaudet, S., Miller-Jensen, K. Redefining signaling pathways with an expanding single-cell toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14 (11), 1083-1086 (2017).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32 (4), 381-386 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved