需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
小鼠视网膜内皮细胞分离用于二代测序
摘要
该协议描述了一种分离鼠出生后视网膜内皮细胞的方法,该方法针对细胞产量,纯度和活力进行了优化。这些细胞适用于下一代测序方法。
摘要
下一代测序的最新进展提高了研究人员对分子和细胞生物学的认识,几项研究揭示了血管生物学的新范式。将这些方法应用于血管发育模型需要优化胚胎和出生后组织的细胞分离技术。细胞产量、活力和纯度都需要最大化,才能从下一代测序方法中获得准确且可重现的结果。研究人员使用新生小鼠视网膜血管化模型来研究血管发育的机制。研究人员使用该模型来研究血管形成和成熟过程中血管生成和动脉静脉命运规格的机制。应用下一代测序技术研究视网膜血管发育模型需要优化一种分离视网膜内皮细胞的方法,以最大限度地提高细胞产量、活力和纯度。该协议描述了使用荧光激活细胞分选(FACS)进行小鼠视网膜组织分离,消化和纯化的方法。结果表明,FACS纯化的CD31 + / CD45-内皮细胞群高度富集内皮细胞基因表达,并且在FACS后60分钟内活力没有变化。其中包括使用该方法分离的内皮细胞的下一代测序方法的代表性结果,包括批量RNA测序和单细胞RNA测序,表明这种用于视网膜内皮细胞分离的方法与下一代测序应用兼容。这种视网膜内皮细胞分离方法将允许先进的测序技术来揭示血管发育的新机制。
引言
通过下一代测序方法对核酸进行测序的高通量能力极大地提高了研究人员对分子和细胞生物学的认识。这些先进技术包括全转录组 RNA 测序、靶区 DNA 测序以鉴定单核苷酸多态性 (SNP)、染色质免疫沉淀 (ChIP) 测序中结合转录因子的 DNA 测序,或转座酶可及染色质 (ATAC) 测序测定中的开放染色质区域,以及单细胞 RNA 测序1.在血管生物学中,这些进展使研究人员能够阐明复杂的发育和疾病机制,以及沿着不同表型的连续统一体区分基因表达模式2,3。未来的实验可以通过将下一代测序与评估的血管发育模型相结合来进一步定义复杂的机制,但样品制备方法需要与先进的测序技术兼容。
二代测序方法的质量、准确性和重现性取决于样品制备方法。当分离细胞亚群或从组织中产生单细胞悬液时,最佳的消化和纯化方法对于最大化细胞群的细胞数量、活力和纯度至关重要4,5。这需要消化方法的平衡:强消化对于从组织中释放细胞并获得足够的细胞用于下游方法是必要的,但如果消化太强,细胞活力将受到负面影响6,7。此外,细胞群的纯度对于可靠的结果和准确的数据分析是必要的,这可以通过FACS完成。这突出了优化细胞分离方法以将二代测序应用于已建立的血管发育模型的重要性。
用于研究血管发育的一个特征良好的模型是鼠视网膜血管发育模型。鼠视网膜脉管系统在出生后二维浅丛中发育,出生后第 (P)3 天从视神经可见初始血管生成萌芽,在 P6 处可见血管生成前部,有柄和尖端细胞,初始血管成熟可见,P9 8,9 后可见血管丛成熟。在初始血管丛的重塑过程中,内皮细胞在不同血管中经历对动脉、毛细血管和静脉表型的规范,以产生循环网络10,11。因此,该方法使研究人员能够在发育过程中的不同时间点可视化血管生成血管丛形成和内皮动脉 - 静脉规格和成熟9。此外,该模型提供了一种研究转基因操作对血管生成和血管丛发育影响的方法,该方法已应用于研究血管发育、动脉静脉畸形和氧诱导的新生血管形成12,13,14,15,16.为了将下一代测序方法与小鼠视网膜血管发育模型相结合,需要一种优化的方案,用于从视网膜组织中分离内皮细胞。
该协议描述了一种优化的方法,用于在P6下消化小鼠的视网膜组织,以最大限度地提高细胞产量,纯度和活力。从P6小鼠中分离视网膜组织,消化20分钟,对CD31和CD45进行免疫染色,并通过FACS纯化以在约2.5小时内分离内皮细胞的单细胞悬液(图1A)。发现这些内皮细胞在分离后60分钟内保持高活力17,允许为下一代测序方法进行文库制备。此外,还提供了使用该分离方案的两种独立的下一代测序方法的FACS门控和质量控制结果的代表性结果:全转录组RNA测序和单细胞RNA测序。该方法允许将下一代测序方法与视网膜血管化模型结合使用,以阐明血管发育的新机制。
研究方案
耶鲁大学和弗吉尼亚大学的机构动物护理和使用委员会批准了本协议中列出的所有动物实验。
1.获取小鼠眼睛进行视网膜隔离
- 准备 1x 冰冷的 PBS,并向 48 孔板的每个孔中加入 500 μL。
- 根据批准的机构指南,在出生后第六天(P6)对新生小鼠实施安乐死。对于该实验,大约4-8只新生小鼠在呼吸停止后通过异氟醚吸入在 P6处安乐死至少三分钟,然后斩首。
- 从每只小鼠身上取下眼睛。使用解剖剪刀垂直于眼睑切割眼睛上的皮肤和膜。然后,用镊子轻轻地按压眼睛上方和下方,使眼睛移出眼窝。
- 用镊子小心地捏住眼睛下方,并切断保持眼睛附着的视神经。然后,将每只眼睛放在准备好的1x冰冷PBS中的48孔板中,直到完成收获。
注意:每只鼠标使用一个孔,两只眼睛来自同一孔中的一只鼠标。
- 用镊子小心地捏住眼睛下方,并切断保持眼睛附着的视神经。然后,将每只眼睛放在准备好的1x冰冷PBS中的48孔板中,直到完成收获。
2.分离小鼠视网膜组织
- 用 500 μL 1x 冰冷的 PBS 填充底部衬有解剖垫的培养皿,以浸没眼睛并将其置于设置为 4.0 倍放大倍率的解剖显微镜下。
- 使用具有宽尖端的转移移液管将眼睛悬浮在解剖垫中,以免损坏眼睛。
注意:确保眼睛完全被PBS覆盖。
- 使用具有宽尖端的转移移液管将眼睛悬浮在解剖垫中,以免损坏眼睛。
- 使用两个细夹钳,用其中一个镊子固定视神经以稳定,并用第二个镊子在角膜和巩膜连接的前房中刺穿一个孔(图1B)。在角膜周围约75%处将孔撕成一个圆圈。
- 在仍然用其中一个镊子握住视神经的同时,使用第二个镊子轻轻地将巩膜从视网膜组织上撕下。
- 如上所述,使用第二个镊子轻轻撕下视网膜组织的玻璃体。
- 再次,使用第二个镊子,轻轻地取出晶状体和透明体丛血管,这些血管在玻璃体被移除时没有脱落。为此,用张开的镊子伸入视网膜,直到几乎接触视网膜,关闭镊子以抓住晶状体和透明体丛血管,然后将镊子从视网膜中拉出。可以重复此操作,直到所有血管都被移除。
注意:透明体丛类似于透明的网状结构。
- 用 500 μL 1x 冰冷的 PBS 填充 2 mL 微量离心管,并将视网膜放入管中。将所有视网膜与眼睛隔离,并将它们放入冰冷的PBS中,然后再继续。
注意:每只小鼠使用一个管子,同一鼠标的两个视网膜共用一个管子。如果视网膜组织撕裂,请将其分多块转移。一窝小鼠的视网膜组织解剖总时间应为15-60分钟,从安乐死开始,以最终的视网膜组织分离结束。
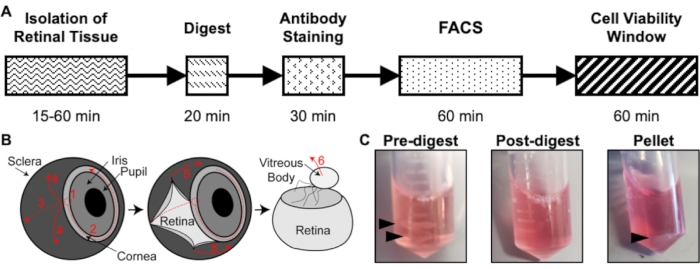
图 1:隔离方案概述。 (A)分离时间表示意图,每个步骤的估计时间:视网膜组织的分离,消化,抗体染色,FACS和细胞活力窗口。(B)从眼睛中分离视网膜组织的分步指南,带有编号的解剖步骤:1)刺穿角膜,2)撕裂角膜,3)巩膜泪,4)从视网膜上去除巩膜,5)进一步从视网膜上去除巩膜和连接组织,6)从视网膜上去除玻璃体和玻璃体血管。(C)各种消化步骤中视网膜组织的代表性图像:消化前,消化后,沉淀(黑色箭头突出显示视网膜组织或细胞沉淀)。经Chavkin等人许可转载,发表于巴塞尔17号的S. Karger AG。请点击此处查看此图的大图。
3. 将视网膜组织消化成单细胞悬液
- 为每分离出两个视网膜准备 500 μL 消化溶液。将FBS和II型胶原酶添加到DMEM中,终浓度为10%FBS和1 mg/mL II型胶原酶。混合并使用水浴将溶液加热至37°C。
- 小心移液,从 2 mL 微量离心管中去除多余的 PBS。留下足够的PBS以完全覆盖视网膜组织,每个管中约100μL。
- 向每个带有视网膜组织的管中加入 500 μL 消化溶液(图 1C)。
- 使用 P1000 移液器和移液器吸头在消化液中上下移液视网膜组织五次。
- 将消化混合物在37°C水浴中孵育20分钟。
- 使用 P1000 移液器和移液器吸头每 5 分钟上下移液消化混合物。孵育后,视网膜组织已溶解成单细胞悬浮液,因此消化混合物应为浑浊(图1C)。
4. 计数单元格
- 将台式离心机置于4°C,并将消化混合物管放入其中。通过以375× g 离心5分钟来沉淀消化混合物。
- 通过移液小心地除去上清液消化溶液。不要打扰细胞沉淀(图1C)。用移液器轻轻上下混合,将沉淀重新悬浮在 500 μL 冰冷的 1x PBS 中。
- 在显微镜下用血细胞计数器计数细胞。
注意:细胞计数约为每两个视网膜 1 x 106 个 细胞。 - 确保通过温和混合将细胞正确重悬,然后将 20 μL 细胞悬液等分到三个管中进行对照染色(管 1:IgG 对照、管 2:CD31 对照和管 3:CD45 对照),如前所述和使用17,18。
- 将台式离心机置于4°C,并将含有细胞的试管置于其中。通过以375× g 离心5分钟来沉淀细胞。
5. 用抗体对细胞进行免疫染色
- 每两个染色的视网膜加上四个对照管准备 100 μL 染色缓冲液。将FBS、HEPES和D-葡萄糖添加到HBSS缓冲液中,终浓度为10%FBS、10 mM HEPES和1 mg/mL D-葡萄糖。
- 获得针对 CD31 和 CD45 的荧光偶联抗体。以 1:100 稀释度将抗体加入染色缓冲液中(试管 1:仅 IgG 抗体,试管 2:仅 CD31 抗体,试管 3:仅 CD45 抗体)。每 100 μL 染色缓冲液加入 1 μL 抗体,最终抗体浓度为 2 μg/mL。
- 通过移液小心地从洗涤的细胞沉淀中取出PBS。
- 将沉淀重悬于每 0.5 x 106 个细胞的 100 μL 抗体染色溶液中。
- 将单细胞悬液与抗体染色溶液在黑暗的冰上孵育30分钟。每10分钟轻敲一次试管以轻轻混合细胞。
6. 准备荧光激活细胞分选
- 将台式离心机置于4°C,并将含有细胞的管放入其中。通过以375× g 离心5分钟来沉淀细胞。
- 取出上清液并将这些细胞重悬于 500 μL 1x PBS 中洗涤。
- 将台式离心机置于4°C,并将含有细胞的管放入其中。通过以375× g 离心5分钟沉淀细胞
- 准备 1.5 mL FACS 缓冲液(1x PBS 中的 1% FBS)。
- 除去上清液,用移液器轻轻混合,将细胞沉淀重悬于300μLFACS缓冲液中。
- 向含有FACS缓冲液的样品管中加入碘化丙啶(PI),最终浓度为0.5 μg/mL。PI用作生存能力标记。
- 通过细胞过滤器卡扣帽将细胞悬液转移并合并到 5 mL 试管中。细胞过滤器卡扣帽应包含一个35μm过滤器,细胞悬浮液可以直接移液到过滤器上。
- 将FACS管放在黑暗中的冰上,并将其转移到细胞分选机中。
7. 设置FACS仪器
- 将 100 μm 喷嘴安装到 FACS 仪器中。
注意:此尺寸喷嘴可最大限度地减少收集体积并最大限度地提高分离的细胞密度。 - 将 250 μL 1x PBS 制备到 1.5 mL 微量离心管中作为收集管,以安装到 FACS 仪器中。
8. 通过 FACS 分离 活的内皮细胞
- 打开 FACS 仪器和计算机。单击FACS软件图标以打开计算机上的FACS软件以运行和操作FACS仪器。执行常规质量控制测试。
- 将对照染色的样品加载到 FACS 仪器中以调整轴。从仅IgG抗体细胞产生和调节FSC和SSC开始,然后CD31抗体仅产生和调节CD31,然后CD45抗体仅产生和调节CD45。记录对照样品中的数据。
- 将染色的样品池加载到FACS仪器中并运行样品。
- 根据前向散射和侧向散射(分别为FSC-A和SSC-A)参数对细胞进行门控(图2A)。
注意:FSC-A 和 SSC-A 参数用于根据尺寸、密度、粒度、表面属性和折射率选择细胞。 - 通过FSC-A和FSC-H门控细胞以识别细胞双联体并仅收集单个细胞(图2A)。
注意:FSC-A 和 FSC-H 参数用于选择单个细胞,其原理是在前向散射期间,细胞双峰将是含有更大面积高度比的液滴。 - 通过PI和SSC-A对细胞进行门控,以鉴定活细胞(图2A)。
注意:活细胞将是PI阴性的。 - 通过CD31和CD45制作带有对照和门控细胞的CD31 + / CD45-门(图2A,B)。
- 插入装有 250 μL FACS 缓冲液的收集管。
- 开始将CD31阳性/CD45阴性的细胞分选到已安装的收集管中。
- 将收集管放在冰上以进行进一步分析。在此步骤1 中,可以处理样品用于下一代测序应用。
9. 进行活性测定
- 将 10 μL 细胞与 10 μL 0.4% 台盼蓝溶液混合,以评估细胞活力。
- 通过将染色的混合物移液到血细胞计数器载玻片上来计数活细胞。将载玻片放在显微镜下以获得准确的活性计数。将蓝色细胞计数为无活力,将透明细胞计数为可存活细胞。
- 将活细胞计数除以总细胞计数以计算活力百分比。
10. 进行基因表达测定
- 每个样品获得10,000-20,000个细胞并进行RNA分离以分析鼠视网膜内皮细胞基因表达。使用市售试剂盒进行RNA分离,该试剂盒使用基于乙醇的RNA沉淀,然后离心和膜柱从细胞裂解物中洗涤和洗脱纯化的RNA。在此步骤1 中,可以处理样品以进行一些二代测序应用。
- 使用逆转录酶和支持试剂将RNA转化为cDNA19。
- 使用DNA结合染料和定量PCR(qPCR)机20进行定量。将cDNA样品加载到带有引物的384孔qPCR板中,以扩增感兴趣的基因。反应体积总计应为 10 μL,包括 DNA 结合染料,该染料将用于定量基因扩增。
- 使用 CD31、VE-钙粘蛋白、CD45 和 β-肌动蛋白正向和反向引物来测量基因表达。
- 将该样本用于二代测序应用1.
结果
视网膜组织的消化和CD31和CD45的免疫染色导致在对细胞,单细胞和活力进行门控后可识别的CD31 + / CD45-内皮细胞群(图2A)。需要CD45免疫染色以消除CD31 + / CD45 +细胞,其中包括血小板和一些白细胞21。应对每个实验进行对照,以显示抗体特异性并指导门控策略(图2B)。这个百分比相对较低,约占消化单细胞悬液中所有细胞的0.5%-1.0%。
讨论
该协议描述了一种从出生后鼠视网膜组织中分离内皮细胞的方法,该方法已针对高细胞数量,纯度和活力进行了优化。通过 CD31+/CD45-免疫染色从消化的单细胞悬液中分离内皮细胞群的 FACS 获得细胞纯度。在台盼蓝染色和 qPCR 对 CD31、CD45 和 VE-钙粘蛋白的基因表达测定中量化分离质量(尽管 VE-钙粘蛋白未用于免疫染色)。该方案中的关键步骤是视网膜组织分离和组织消化。应快速进行视网膜组织分?...
披露声明
作者没有相关披露。
致谢
感谢耶鲁大学流式细胞术设施、弗吉尼亚大学流式细胞术核心设施、耶鲁大学基因组分析中心和弗吉尼亚大学基因组分析和技术核心,感谢他们在为所介绍的实验做出贡献方面的努力、专业知识和建议。这项研究由NIH资助N.W.C.(T32 HL007224,T32 HL007284),SC(T32 HL007284),K.W.(R01 HL142650)和K.K.H.(R01 HL146056,R01 DK118728,UH3 EB025765)。
材料
| Name | Company | Catalog Number | Comments |
| 2 mL Eppendorf safe-lock tubes | USA Scientific | 4036-3352 | |
| 5 ml Falcon Test Tubes with Cell Strainer Snap Cap | Corning | 352235 | |
| 60 mm Non TC-treated Culture Dish | Corning | 430589 | |
| APC Rat Anti-Mouse CD31 | BD Biosciences | 551262 | |
| APC Rat IgG2a κ Isotype Control | BD Biosciences | 553932 | |
| BD FACSChorus Software | BD Biosciences | FACSCHORUS | |
| BD FACSMelody Cell Sorter | BD Biosciences | FACSMELODY | |
| Collagenase Type II | Sigma-Aldrich | 234115 | |
| Costar 48-well Clear TC-treated Multiple Well Plates, Individually Wrapped, Sterile | Corning | 3548 | |
| D-Glucose | Gibco | A2494001 | |
| Disposable Graduated Transfer Pipettes | Fisher Scientific | 12-711-9AM | |
| Dissecting Pan Wax | Carolina | 629100 | |
| Dissection scissors | Fine Science Tools | 14085-08 | |
| Dissection Stereo Microscope M165 FC | Leica | M165FC | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-052 | |
| Dulbecco’s Phosphate Buffered Saline (PBS) | Gibco | 14190144 | |
| Eppendorf Flex-Tubes Microcentrifuge Tubes 1.5 mL | Sigma-Aldrich | 22364120 | |
| Fetal Bovine Serum (FBS) | Gemini Bio | 100-106 | |
| Fine dissection forceps | Fine Science Tools | 11250-00 | |
| Hank's Buffered Salt Solution (HBSS) | Gibco | 14175095 | |
| HEPES (1M) | Gibco | 15630130 | |
| iScript cDNA Synthesis Kit | Bio-Rad | 1708890 | |
| Isoflurane, USP | Covetrus | 11695067772 | |
| Isotemp General Purpose Deluxe Water Bath | Fisher Scientific | FSGPD20 | |
| Primer: ActB_Forward: 5’- agagggaaatcgtgcgtgac -3’ | Integrated DNA Technologies | N/A | |
| Primer: ActB_Reverse: 5’- caatagtgatgacctggccgt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Forward: 5’- gagcccaatcacgtttcagttt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Reverse: 5’- tccttcctgcttcttgctagct -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Forward: 5’- gggttgttctgtgccttgtt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Reverse: 5’- ctggacggacacagttagca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Forward: 5’- tcctctgcatcctcactatcaca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Reverse: 5’- gtaagtgaccaactgctcgtgaat -3’ | Integrated DNA Technologies | N/A | |
| Propidium iodide | Sigma-Aldrich | P4864 | |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| Sorvall Legend Micro 21R Centrifuge, Refrigerated | ThermoFisher | 75002477 | |
| SYBR-Green iTaq Universal SYBR Green Supermix | Bio-Rad | 172-5120 | |
| Trypan Blue Solution | ThermoFisher | 15250061 | |
| V450 Rat Anti-Mouse CD45 | BD Biosciences | 560501 | |
| V450 Rat IgG2b, κ Isotype Control | BD Biosciences | 560457 |
参考文献
- Slatko, B. E., Gardner, A. F., Ausubel, F. M. Overview of next-generation sequencing technologies. Current Protocols in Molecular Biology. 122 (1), 59 (2018).
- Chavkin, N. W., Hirschi, K. K. Single cell analysis in vascular biology. Frontiers in Cardiovascular Medicine. 7, 42 (2020).
- Ma, F., Hernandez, G. E., Romay, M., Iruela-Arispe, M. L. Single-cell RNA sequencing to study vascular diversity and function. Current Opinion in Hematology. 28 (3), 221-229 (2021).
- Potter, A. S., Potter, S. S., S, Dissociation of tissues for single-cell analysis. Methods in Molecular Biology. 1926, 55-62 (2019).
- Braga, F. A. V., Miragaia, R. J. Tissue handling and dissociation for single-cell RNA-seq. Single Cell Methods: Methods in Molecular Biology. 1979, 9-21 (2019).
- Brink, S. C., et al. Single-cell sequencing reveals dissociatin-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Skulska, K., Wegrzyn, A. S., Chelmonska-Soyta, A., Chodaczek, G. Impact of tissue enzymatic digestion on analysis of immune cells in mouse reproductive mucosa with a focus on gammadelta T cells. Journal of Immunological Methods. 474, 112665 (2019).
- Connolly, S., Hores, T., Smith, L., D'Amore, P. Characterization of vascular development in the mouse retina. Microvascular Research. 36 (3), 275-290 (1988).
- Crist, A., Young, C., Meadows, S. Characterization of arteriovenous identity in the developing neonate mouse retina. Gene Expression Patterns: GEP. 23-24, 22-31 (2017).
- dela Paz, N. G., D'Amore, P. A. Arterial versus venous endothelial cells. Cell and Tissue Research. 335 (1), 5-16 (2009).
- Fang, J. S., Hirschi, K. K. Molecular regulation of arteriovenous endothelial cell specification. F1000Res. 8, (2019).
- Smith, L. E., et al. Oxygen-induced retinopathy in the mouse. Investigative Ophthalmology and Visual Science. 35 (1), 101-111 (1994).
- Pitulescu, M. E., Schmidt, I., Benedito, R., Adams, R. H. Inducible gene targeting in the neonatal vasculature and analysis of retinal angiogenesis in mice. Nature Protocols. 5 (9), 1518-1534 (2010).
- Ruiz, S., et al. A mouse model of hereditary hemorrhagic telangiectasia generated by transmammary-delivered immunoblocking of BMP9 and BMP10. Scientific Reports. 6, 37366 (2016).
- Fang, J. S., et al. Shear-induced Notch-Cx37-p27 axis arrests endothelial cell cycle to enable arterial specification. Nature Communication. 8 (1), 2149 (2017).
- Ola, R., et al. SMAD4 prevents flow induced arterial-venous malformations by inhibiting Casein Kinase 2. Circulation. 138 (21), 2379-2394 (2018).
- Chavkin, N. W., Walsh, K., Hirschi, K. K. Isolation of highly purified and viable retinal endothelial cells. Journal of Vascular Research. 58 (1), 49-57 (2020).
- Hulspas, R., O'Gorman, M. R. G., Wood, B. L., Gratama, J. W., Sutherland, D. R. Considerations for the control of background fluorescence in clinical flow cytometry. Cytometry PartB: Clinical Cytometry. 76 (6), 355-364 (2009).
- Bachman, J. Reverse-transcription PCR (RT-PCR). Methods in Enzymology. 530, 67-74 (2013).
- Green, M. R., Sambrook, J. Quantification of RNA by real-time Reverse Transcription-Polymerase Chain Reaction (RT-PCR). Cold Spring Harbour Protocols. 2018 (10), (2018).
- Liu, L., Shi, G. P. CD31: beyond a marker for endothelial cells. Cardiovascular Research. 94 (1), 3-5 (2012).
- Zarkada, G., et al. Specialized endothelial tip cells guide neuroretina vascularization and blood-retina-barrier formation. Developmental Cell. 56 (15), 2237-2251 (2021).
- Su, X., Sorenson, C. M., Sheibani, N. Isolation and characterization of murine retinal endothelial cells. Molecular Vision. 9, 171-178 (2003).
- Benedito, R., et al. The notch ligands Dll4 and Jagged1 have opposing effects on angiogenesis. Cell. 137 (6), 1124-1135 (2009).
- Daneman, R., et al. Wnt/beta-catenin signaling is required for CNS, but not non-CNS, angiogenesis. Proceedings of the National Academy of Sciences of the United States of America. 106 (2), 641-646 (2009).
- Okabe, K., et al. Neurons limit angiogenesis by titrating VEGF in retina. Cell. 159 (3), 584-596 (2014).
- Crist, A. M., Lee, A. R., Patel, N. R., Westhoff, D. E., Meadows, S. M. Vascular deficiency of Smad4 causes arteriovenous malformations: a mouse model of Hereditary Hemorrhagic Telangiectasia. Angiogenesis. 21 (2), 363-380 (2018).
- Kim, Y. H., Choe, S. W., Chae, M. Y., Hong, S., Oh, S. P. SMAD4 deficiency leads to development of arteriovenous malformations in neonatal and adult mice. Journal of the American Heart Association. 7 (21), 009514 (2018).
- Ma, W., et al. Absence of TGFbeta signaling in retinal microglia induces retinal degeneration and exacerbates choroidal neovascularization. eLife. 8, 42049 (2019).
- Luo, W., et al. Arterialization requires the timely suppression of cell growth. Nature. 589 (7842), 437-441 (2021).
- Lawson, N. D., Vogel, A. M., Weinstein, B. M. sonic hedgehog and vascular endothelial growth factor act upstream of the Notch pathway during arterial endothelial differentiation. Developmental Cell. 3 (1), 127-136 (2002).
- Larrivee, B., et al. ALK1 signaling inhibits angiogenesis by cooperating with the Notch pathway. Developmental Cell. 22 (3), 489-500 (2012).
- Wythe, J. D., et al. ETS factors regulate Vegf-dependent arterial specification. Developmental Cell. 26 (1), 45-58 (2013).
- Davis, D. M., Purvis, J. E. Computational analysis of signaling patterns in single cells. Seminars in Cell and Developmental Biology. , 35-43 (2015).
- Gaudet, S., Miller-Jensen, K. Redefining signaling pathways with an expanding single-cell toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14 (11), 1083-1086 (2017).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32 (4), 381-386 (2014).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。