Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Isolierung von retinen retinalen Endothelzellen für die Sequenzierung der nächsten Generation
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt eine Methode zur Isolierung von murinen postnatalen retinalen Endothelzellen, die für Zellertrag, Reinheit und Lebensfähigkeit optimiert sind. Diese Zellen eignen sich für Next-Generation-Sequenzierungsansätze.
Zusammenfassung
Jüngste Verbesserungen in der Next-Generation-Sequenzierung haben das Wissen der Forscher über Molekular- und Zellbiologie erweitert, wobei mehrere Studien neue Paradigmen in der vaskulären Biologie aufdecken. Die Anwendung dieser Methoden auf Modelle der vaskulären Entwicklung erfordert die Optimierung von Zellisolierungstechniken aus embryonalen und postnatalen Geweben. Zellausbeute, Lebensfähigkeit und Reinheit müssen alle maximal sein, um genaue und reproduzierbare Ergebnisse aus Sequenzierungsansätzen der nächsten Generation zu erhalten. Das retinale Vaskularisationsmodell der neonatalen Maus wird von Forschern verwendet, um Mechanismen der vaskulären Entwicklung zu untersuchen. Forscher haben dieses Modell verwendet, um Mechanismen der Angiogenese und der arteriell-venösen Schicksalsspezifikation während der Blutgefäßbildung und -reifung zu untersuchen. Die Anwendung von Next-Generation-Sequenzierungstechniken zur Untersuchung des retinalen vaskulären Entwicklungsmodells erfordert die Optimierung einer Methode zur Isolierung von retinalen Endothelzellen, die die Zellausbeute, Lebensfähigkeit und Reinheit maximiert. Dieses Protokoll beschreibt eine Methode zur Isolierung, Verdauung und Reinigung von retinem retinalem Gewebe unter Verwendung von Fluoreszenz-aktivierter Zellsortierung (FACS). Die Ergebnisse deuten darauf hin, dass die FACS-gereinigte CD31+/CD45-Endothelzellpopulation für die Genexpression von Endothelzellen stark angereichert ist und keine Veränderung der Lebensfähigkeit für 60 Minuten nach FACS aufweist. Enthalten sind repräsentative Ergebnisse von Next-Generation-Sequenzierungsansätzen an Endothelzellen, die mit dieser Methode isoliert wurden, einschließlich Bulk-RNA-Sequenzierung und Einzelzell-RNA-Sequenzierung, die zeigen, dass diese Methode zur retinalen Endothelzellisolierung mit Next-Generation-Sequenzierungsanwendungen kompatibel ist. Diese Methode der retinalen Endothelzellisolierung wird fortschrittliche Sequenzierungstechniken ermöglichen, um neue Mechanismen der vaskulären Entwicklung aufzudecken.
Einleitung
Die hohe Durchsatzkapazität der Sequenzierung von Nukleinsäuren über Next-Generation-Sequencing-Ansätze hat das Wissen der Forscher über Molekular- und Zellbiologie erheblich erweitert. Diese fortschrittlichen Techniken umfassen die Sequenzierung der gesamten Transkriptom-RNA, die DNA-Sequenzierung von Zielregionen zur Identifizierung von Einzelnukleotid-Polymorphismen (SNPs), die DNA-Sequenzierung gebundener Transkriptionsfaktoren bei der Chromatin-Immunpräzipitation (ChIP)-Sequenzierung oder offene Chromatinregionen in der ATAC-Sequenzierung (Assay for Transposase-Accessible Chromatin) und die Einzelzell-RNA-Sequenzierung1 . In der Gefäßbiologie haben diese Fortschritte es den Forschern ermöglicht, komplizierte Mechanismen der Entwicklung und Krankheit aufzuklären und Genexpressionsmuster entlang eines Kontinuums unterschiedlicher Phänotypen zu unterscheiden 2,3. Zukünftige Experimente können komplexe Mechanismen weiter definieren, indem sie die Sequenzierung der nächsten Generation mit evaluierten Modellen der vaskulären Entwicklung kombinieren, aber die Methoden zur Probenvorbereitung müssen mit den fortschrittlichen Sequenzierungstechniken kompatibel sein.
Die Qualität, Genauigkeit und Reproduzierbarkeit von Next-Generation-Sequenzierungsansätzen hängt von der Methode der Probenvorbereitung ab. Bei der Isolierung einer Untergruppe von Zellen oder der Erzeugung einzelliger Suspensionen aus Geweben sind optimale Verdauungs- und Reinigungsmethoden unerlässlich, um die Zellzahl, Lebensfähigkeit und Reinheit der Zellpopulation zu maximieren 4,5. Dies erfordert ein Gleichgewicht in der Verdauungsmethode: Eine starke Verdauung ist notwendig, um Zellen aus dem Gewebe zu befreien und genügend Zellen für nachgeschaltete Ansätze zu erhalten, aber die Zelllebensfähigkeit wird negativ beeinflusst, wenn die Verdauung zu stark ist 6,7. Darüber hinaus ist die Reinheit der Zellpopulation für robuste Ergebnisse und eine genaue Analyse der Daten erforderlich, die durch FACS erreicht werden kann. Dies unterstreicht die Bedeutung der Optimierung von Zellisolationsmethoden, um die Next-Generation-Sequenzierung auf etablierte Modelle der vaskulären Entwicklung anzuwenden.
Ein gut charakterisiertes Modell zur Untersuchung der vaskulären Entwicklung ist das murine retinale vaskuläre Entwicklungsmodell. Das murine retinale Gefäßsystem entwickelt sich postnatal in einem zweidimensionalen oberflächlichen Plexus mit anfänglichem angiogenem Sprießen aus dem Sehnerv, sichtbar am postnatalen Tag (P)3, angiogener Front mit Stiel- und Spitzenzellen und anfänglicher Gefäßreifung sichtbar bei P6 und Reifung des vaskulären Plexus sichtbar nach P9 8,9. Während des Umbaus des anfänglichen vaskulären Plexus werden Endothelzellen in verschiedenen Gefäßen in Richtung arterieller, kapillarer und venöser Phänotypen spezifiziert, um ein Kreislaufnetzwerkzu erzeugen 10,11. Daher ermöglicht diese Methode den Forschern, die angiogene Gefäßplexusbildung und die endotheliale arteriell-venöse Spezifikation und Reifung zu verschiedenen Zeitpunkten während der Entwicklung sichtbar zu machen9. Darüber hinaus bietet dieses Modell eine Methode zur Untersuchung der Auswirkungen transgener Manipulation auf die Angiogenese und die Entwicklung des vaskulären Plexus, die zur Untersuchung von Gefäßentwicklung, arteriell-venösen Fehlbildungen und sauerstoffinduzierter Neovaskularisation eingesetzt wurde 12,13,14,15,16 . Um Next-Generation-Sequenzierungsansätze mit dem murinen retinalen vaskulären Entwicklungsmodell zu kombinieren, ist ein optimiertes Protokoll für die Isolierung von Endothelzellen aus Netzhautgewebe notwendig.
Dieses Protokoll beschreibt eine optimierte Methode zur Verdauung von Netzhautgewebe von Mäusen bei P6, um die Zellausbeute, Reinheit und Lebensfähigkeit zu maximieren. Netzhautgewebe wird aus P6-Mäusen isoliert, für 20 min verdaut, für CD31 und CD45 immungefärbt und durch FACS gereinigt, um eine Einzelzellsuspension von Endothelzellen in etwa 2,5 h zu isolieren (Abbildung 1A). Es wurde festgestellt, dass diese Endothelzellen 60 Minuten nach der Isolierung eine hohe Lebensfähigkeit aufrechterhalten17, was Bibliothekspräparate für Sequenzierungsmethoden der nächsten Generation ermöglicht. Darüber hinaus werden repräsentative Ergebnisse für FACS-Gating- und Qualitätskontrollergebnisse aus zwei separaten Next-Generation-Sequenzierungsmethoden unter Verwendung dieses Isolationsprotokolls bereitgestellt: die gesamte Transkriptom-RNA-Sequenzierung und die Einzelzell-RNA-Sequenzierung. Diese Methode ermöglicht es, Next-Generation-Sequenzierungsansätze in Verbindung mit dem retinalen Vaskularisationsmodell zu verwenden, um neue Mechanismen der vaskulären Entwicklung aufzuklären.
Protokoll
Die Institutional Animal Care and Use Committees der Yale University und der University of Virginia genehmigten alle in diesem Protokoll aufgeführten Tierversuche.
1. Besorgen Sie sich Mausaugen für die Netzhautisolierung
- Bereiten Sie 1x eiskaltes PBS vor und geben Sie 500 μL in jede Vertiefung einer 48-Well-Platte.
- Euthanasieren Sie neugeborene Mäuse am sechsten postnatalen Tag (P6) gemäß genehmigten institutionellen Richtlinien. Für dieses Experiment werden Würfe von etwa 4-8 neonatalen Mäusen bei P6 durch Isofluran-Inhalation für mindestens drei Minuten nach dem Atemstillstand eingeschläfert, gefolgt von einer Enthauptung.
- Entfernen Sie die Augen von jeder der Mäuse. Schneiden Sie die Haut und die Membran über dem Auge ab, indem Sie mit einer Sezierschere senkrecht zum Augenlid schneiden. Verwenden Sie dann eine Pinzette, um sanft über und unter das Auge zu drücken, so dass sich das Auge aus der Augenhöhle bewegt.
- Kneifen Sie vorsichtig mit der Pinzette unter das Auge und schneiden Sie den Sehnerv, der das Auge befestigt hält. Legen Sie dann jedes Auge in die 48-Well-Platte in das vorbereitete 1x eiskalte PBS, bis die Ernte beendet ist.
HINWEIS: Verwenden Sie eine Vertiefung pro Maus, wobei sich beide Augen einer einzelnen Maus im selben Vertiefung befinden.
- Kneifen Sie vorsichtig mit der Pinzette unter das Auge und schneiden Sie den Sehnerv, der das Auge befestigt hält. Legen Sie dann jedes Auge in die 48-Well-Platte in das vorbereitete 1x eiskalte PBS, bis die Ernte beendet ist.
2. Netzhautgewebe der Maus isolieren
- Füllen Sie eine Petrischale, die mit einem Präparierpad auf der Unterseite ausgekleidet ist, mit 500 μL 1x eiskaltem PBS, um die Augen einzutauchen, und legen Sie sie unter ein Dissektionsmikroskop, das auf 4,0-fache Vergrößerung eingestellt ist.
- Hängen Sie die Augen mit einer Transferpipette mit breiter Spitze in das Präparierpad, um die Augen nicht zu beschädigen.
HINWEIS: Stellen Sie sicher, dass die Augen vollständig mit PBS bedeckt sind.
- Hängen Sie die Augen mit einer Transferpipette mit breiter Spitze in das Präparierpad, um die Augen nicht zu beschädigen.
- Halten Sie den Sehnerv mit einer der Pinzetten zur Stabilisierung mit zwei feinen Dissektionszangen fest und stechen Sie mit der zweiten Pinzette ein Loch durch die Vorderkammer, wo sich Hornhaut und Sklera verbinden (Abbildung 1B). Reißen Sie das Loch in einem Kreis etwa 75% des Weges um die Hornhaut.
- Während Sie den Sehnerv noch mit einer der Pinzetten halten, verwenden Sie die zweite Pinzette, um die Sklera sanft vom Netzhautgewebe abzureißen.
- Verwenden Sie wie oben die zweite Pinzette, um den Glaskörper des Netzhautgewebes vorsichtig abzureißen.
- Entfernen Sie mit der zweiten Pinzette vorsichtig die Linsen- und Hyaloidplexusgefäße, die sich nicht gelöst haben, als der Glaskörper entfernt wurde. Um dies zu tun, greifen Sie mit einer offenen Pinzette in die Netzhaut, bis Sie fast die Netzhaut berühren, schließen Sie die Pinzette, um die Linsen- und Hyaloidplexusgefäße zu greifen, und ziehen Sie die Pinzette aus der Netzhaut heraus. Dies kann wiederholt werden, bis alle Gefäße entfernt sind.
HINWEIS: Der Plexus hyaloideus ähnelt einer klaren, netzartigen Struktur.
- Füllen Sie 2 mL Mikrozentrifugenröhrchen mit 500 μL 1x eiskaltem PBS und legen Sie die Netzhaut in die Röhrchen. Isolieren Sie alle Netzhäute von den Augen und legen Sie sie in eiskaltes PBS, bevor Sie fortfahren.
HINWEIS: Verwenden Sie eine Röhre pro Maus, wobei sich zwei Netzhäute derselben Maus eine Röhre teilen. Wenn Netzhautgewebe gerissen ist, übertragen Sie es in mehrere Stücke. Die Gesamtzeit der Netzhautgewebedissektion für einen Wurf von Mäusen sollte 15-60 Minuten dauern, beginnend mit der Euthanasie und endend mit der endgültigen Netzhautgewebeisolierung.
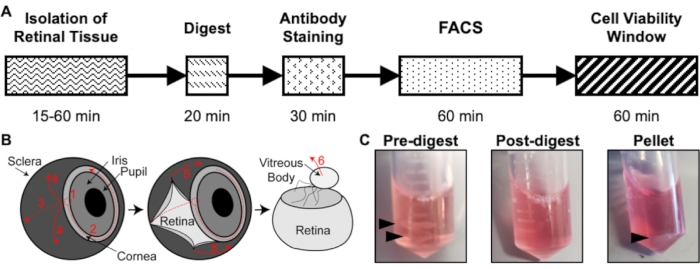
Abbildung 1: Überblick über das Isolationsprotokoll. (A) Schematische Darstellung des Zeitplans für die Isolierung mit einer geschätzten Zeit für jeden Schritt: Isolierung von Netzhautgewebe, Verdauung, Antikörperfärbung, FACS und Zelllebensfähigkeitsfenster. (B) Schritt-für-Schritt-Anleitung zur Isolierung von Netzhautgewebe aus dem Auge, mit nummerierten Dissektionsschritten: 1) Hornhaut durchstechen, 2) Hornhaut reißen, 3) Sklera reißen, 4) Sklera aus der Netzhaut entfernen, 5) Sklera und Bindegewebe aus der Netzhaut entfernen, 6) den Glaskörper und die Glaskörpergefäße von der Netzhaut entfernen. (C) Repräsentative Bilder von Netzhautgewebe während verschiedener Verdauungsschritte: Pre-digest, Post-digest, Pellet (schwarze Pfeile markieren Netzhautgewebe oder Zellpellet). Wiederveröffentlichung mit freundlicher Genehmigung von Chavkin et al. veröffentlicht in S. Karger AG, Basel17. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
3. Netzhautgewebe zu einer einzelligen Suspension verdauen
- Bereiten Sie 500 μL Aufschlusslösung für jeweils zwei isolierte Netzhäute vor. Fügen Sie FBS und Kollagenase Typ II zu DMEM zu einer Endkonzentration von 10% FBS und 1 mg / ml Kollagenase Typ II hinzu. Die Lösung wird mit einem Wasserbad gemischt und auf 37 °C erwärmt.
- Entfernen Sie überschüssiges PBS aus den 2-ml-Mikrozentrifugenröhrchen durch vorsichtiges Pipettieren. Lassen Sie genügend PBS, um das Netzhautgewebe vollständig zu bedecken, etwa 100 μL in jedem Röhrchen.
- Geben Sie 500 μL der Verdauungslösung in jedes Röhrchen mit Netzhautgewebe (Abbildung 1C).
- Verwenden Sie einen P1000-Pipettierer und eine Pipettenspitze, um das Netzhautgewebe in der Verdauungslösung fünfmal auf und ab zu pipettieren.
- Die Aufschlussmischung in einem 37 °C warmen Wasserbad für 20 min inkubieren.
- Verwenden Sie einen P1000-Pipettierer und eine Pipettenspitze, um die Aufschlussmischung alle 5 Minuten auf und ab zu pipettieren. Nach der Inkubation hat sich das Netzhautgewebe in eine einzellige Suspension aufgelöst, so dass die Verdauungsmischung trüb sein sollte (Abbildung 1C).
4. Zellen zählen
- Stellen Sie eine Tischzentrifuge auf 4 °C ein und legen Sie die Aufschlussmischröhrchen hinein. Pelletieren Sie die Aufschlussmischung durch Zentrifugieren bei 375 x g für 5 min.
- Die überstehende Verdauungslösung vorsichtig durch Pipettieren entfernen. Stören Sie das Zellpellet nicht (Abbildung 1C). Suspendieren Sie das Pellet in 500 μL eiskaltem 1x PBS durch vorsichtiges Auf- und Abmischen mit der Pipette.
- Zählen Sie die Zellen mit einem Hämozytometer unter einem Mikroskop.
HINWEIS: Die Zellzahl beträgt ungefähr 1 x 106 Zellen pro zwei Netzhäute. - Stellen Sie sicher, dass die Zellen durch sanftes Mischen ordnungsgemäß resuspendiert werden, und aliquot dann 20 μL Zellsuspension in drei Röhrchen zur Kontrolle der Färbung (Röhrchen 1: IgG-Kontrolle, Röhrchen 2: CD31-Kontrolle und Röhrchen 3: CD45-Kontrolle), wie zuvor beschrieben und verwendet17,18.
- Stellen Sie eine Tischzentrifuge auf 4 °C ein und legen Sie Röhrchen mit Zellen hinein. Pelletieren Sie die Zellen durch Zentrifugieren bei 375 x g für 5 min.
5. Immunfärben Sie die Zellen mit Antikörpern
- Bereiten Sie 100 μL Färbepuffer pro zwei gefärbte Netzhäute plus vier Kontrollröhrchen vor. Fügen Sie FBS, HEPES und D-Glucose zu HBSS-Puffer auf eine Endkonzentration von 10% FBS, 10 mM HEPES und 1 mg / ml D-Glucose hinzu.
- Erhalten Sie fluoreszenzkonjugierte Antikörper gegen CD31 und CD45. Geben Sie die Antikörper in einer Verdünnung von 1:100 in den Färbepuffer (Röhrchen 1: Nur IgG-Antikörper, Röhrchen 2: nur CD31-Antikörper und Röhrchen 3: nur CD45-Antikörper). Fügen Sie 1 μL Antikörper pro 100 μL des Färbepuffers hinzu, um eine endgültige Antikörperkonzentration von 2 μg / ml zu erhalten.
- Entfernen Sie das PBS vorsichtig durch Pipettieren aus dem gewaschenen Zellpellet.
- Resuspendieren Sie das Pellet in 100 μL Antikörperfärbelösung pro 0,5 x 106 Zellen.
- Inkubieren Sie die Einzelzellsuspension mit der Antikörperfärbelösung für 30 min auf Eis im Dunkeln. Klopfen Sie alle 10 Minuten auf die Röhrchen, um die Zellen vorsichtig zu mischen.
6. Vorbereitung auf fluoreszenzaktivierte Zellsortierung
- Stellen Sie eine Tischzentrifuge auf 4 °C ein und legen Sie die Röhrchen mit den Zellen hinein. Pelletzellen durch Zentrifugation bei 375 x g für 5 min.
- Entfernen Sie den Überstand und resuspendieren Sie diese Zellen in 500 μL 1x PBS zum Waschen.
- Stellen Sie eine Tischzentrifuge auf 4 °C ein und legen Sie die Röhrchen mit den Zellen hinein. Pelletzellen durch Zentrifugation bei 375 x g für 5 min
- Bereiten Sie 1,5 ml FACS-Puffer vor (1% FBS in 1x PBS).
- Entfernen Sie den Überstand und resuspendieren Sie das Zellpellet in 300 μL FACS-Puffer durch vorsichtiges Mischen mit einem Pipettor.
- Geben Sie Propidiumiodid (PI) zu den Probenröhrchen, die den FACS-Puffer enthalten, bis zu einer Endkonzentration von 0,5 μg/ml. PI wird als Lebensfähigkeitsmarker verwendet.
- Die Zellsuspensionen werden durch eine Schnappkappe des Zellsiebs in 5 ml Reagenzgläser überführt und kombiniert. Die Schnappkappe des Zellsiebs sollte einen 35-μm-Filter enthalten, und die Zellsuspensionen können direkt auf den Filter pipettiert werden.
- Halten Sie die FACS-Röhrchen im Dunkeln auf Eis und übertragen Sie sie in den Zellsortierer.
7. Einrichtung des FACS-Instruments
- Installieren Sie eine 100-μm-Düse in ein FACS-Gerät.
HINWEIS: Diese Größendüse minimiert das Sammelvolumen und maximiert die isolierte Zelldichte. - Bereiten Sie 250 μL 1x PBS in ein 1,5-ml-Mikrozentrifugenröhrchen als Sammelröhrchen vor, das in das FACS-Gerät eingebaut wird.
8. Isolieren Sie lebensfähige Endothelzellen über FACS
- Schalten Sie das FACS-Instrument und den Computer ein. Klicken Sie auf das FACS-Softwaresymbol, um die FACS-Software auf dem Computer zu öffnen, um das FACS-Gerät auszuführen und zu bedienen. Führen Sie routinemäßige Qualitätskontrolltests durch.
- Legen Sie die kontrollgefärbten Proben in das FACS-Gerät, um die Achsen anzupassen. Beginnen Sie mit reinen IgG-Antikörperzellen, um FSC und SSC zu erzeugen und anzupassen, dann CD31-Antikörper nur, um CD31 zu erzeugen und anzupassen, und dann CD45-Antikörper nur, um CD45 zu erzeugen und anzupassen. Zeichnen Sie Daten aus Kontrollproben auf.
- Laden Sie gefärbte Probenzellen in das FACS-Gerät und führen Sie die Probe aus.
- Gattern Sie die Zellen basierend auf den Parametern Vorwärtsstreuung und Seitenstreuung (FSC-A bzw. SSC-A) (Abbildung 2A).
HINWEIS: Die Parameter FSC-A und SSC-A werden verwendet, um Zellen basierend auf Größe, Dichte, Granularität, Oberflächeneigenschaften und Brechungsindex auszuwählen. - Gate-Zellen mittels FSC-A und FSC-H, um Zelldubletten zu identifizieren und nur einzelne Zellen zu sammeln (Abbildung 2A).
HINWEIS: Die Parameter FSC-A und FSC-H werden verwendet, um einzelne Zellen nach dem Prinzip auszuwählen, dass Zelldubletten während der Vorwärtsstreuung Tröpfchen mit einem größeren Verhältnis von Fläche zu Höhe sind. - Gate-Zellen mittels PI und SSC-A zur Identifizierung lebensfähiger Zellen (Abbildung 2A).
HINWEIS: Lebensfähige Zellen sind PI-negativ. - Erstellen Sie ein CD31+/CD45-Gate mit Bedienelementen und Gate-Zellen von CD31 und CD45 (Abbildung 2A,B).
- Führen Sie ein Sammelröhrchen mit 250 μL FACS-Puffer ein.
- Sortieren Sie Zellen, die CD31-positiv/CD45-negativ sind, in das installierte Sammelrohr.
- Bewahren Sie die Sammelröhrchen zur weiteren Analyse auf Eis auf. In diesem Schritt1 können Proben für Sequenzierungsanwendungen der nächsten Generation verarbeitet werden.
9. Rentabilitätstest durchführen
- Mischen Sie 10 μL Zellen mit 10 μL 0,4% Trypanblau-Lösung, um die Zelllebensfähigkeit zu beurteilen.
- Zählen Sie lebensfähige Zellen, indem Sie die gefärbte Mischung auf einen Hämozytometerobjektträger pipettieren. Legen Sie den Objektträger unter ein Mikroskop, um genaue Lebensfähigkeitswerte zu erhalten. Zählen Sie blaue Zellen als nicht lebensfähig und zählen Sie klare Zellen als lebensfähig.
- Teilen Sie die Anzahl der lebensfähigen Zellen durch die Gesamtzahl der Zelle, um die prozentuale Lebensfähigkeit zu berechnen.
10. Genexpressionstest durchführen
- Erhalten Sie 10.000-20.000 Zellen pro Probe und führen Sie eine RNA-Isolierung durch, um die Genexpression von retinalen retinalen Endothelzellen zu analysieren. Die RNA-Isolierung wird mit einem kommerziell erhältlichen Kit durchgeführt, das ethanolbasierte RNA-Fällung und dann Zentrifugations- und Membransäulen verwendet, um gereinigte RNA aus Zelllysat zu waschen und zu eluieren. In diesem Schritt1 können Proben für einige Sequenzierungsanwendungen der nächsten Generation verarbeitet werden.
- Wandeln Sie die RNA in cDNA um, indem Sie das Enzym der reversen Transkriptase und unterstützende Reagenzienverwenden 19.
- Quantifizierung mit einem DNA-bindenden Farbstoff und einer quantitativen PCR (qPCR)20. Laden Sie cDNA-Proben in eine 384-Well-qPCR-Platte mit Primern, um Gene von Interesse zu amplifizieren. Das Reaktionsvolumen sollte 10 μL betragen, einschließlich DNA-bindender Farbstoff, der zur Quantifizierung der Genamplifikation verwendet wird.
- Verwenden Sie CD31, VE-Cadherin, CD45 und β-Aktin-Vorwärts- und Rückwärtsprimer, um die Genexpression zu messen.
- Verwenden Sie das Beispiel für Sequenzierungsanwendungen der nächsten Generation1.
Ergebnisse
Die Verdauung von Netzhautgewebe und die Immunfärbung für CD31 und CD45 führt zu einer identifizierbaren Population von CD31+/CD45-Endothelzellen nach Gating für Zellen, einzelne Zellen und Lebensfähigkeit (Abbildung 2A). Eine CD45-Immunfärbung ist erforderlich, um CD31+/CD45+-Zellen zu eliminieren, zu denen Blutplättchen und einige Leukozyten gehören21. Für jedes Experiment sollten Kontrollen durchgeführt werden, um die Antikörperspezifität und die Gating...
Diskussion
Dieses Protokoll beschreibt eine Methode zur Isolierung von Endothelzellen aus postnatalem murinem Netzhautgewebe, die für eine hohe Zellzahl, Reinheit und Lebensfähigkeit optimiert wurde. Die Zellreinheit wird durch FACS-Isolierung von Endothelzellpopulationen aus der verdauten Einzelzellsuspension durch CD31+/CD45-Immunfärbung erreicht. Die Qualität der Isolierung wird in Assays auf Lebensfähigkeit durch Trypanblau-Färbung und Genexpression mittels qPCR für CD31, CD45 und VE-Cadherin quantifiziert (obwohl VE-Cad...
Offenlegungen
Die Autoren haben keine relevanten Angaben.
Danksagungen
Vielen Dank an die Yale Flow Cytometry Facility, die University of Virginia Flow Cytometry Core Facility, das Yale Center for Genomic Analysis und den University of Virginia Genome Analysis and Technology Core für ihre Bemühungen, ihr Fachwissen und ihre Beratung bei der Durchführung der vorgestellten Experimente. Diese Studie wurde durch NIH-Zuschüsse an N.W.C. (T32 HL007224, T32 HL007284), S.C. (T32 HL007284), K.W. (R01 HL142650) und K.K.H. (R01 HL146056, R01 DK118728, UH3 EB025765) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| 2 mL Eppendorf safe-lock tubes | USA Scientific | 4036-3352 | |
| 5 ml Falcon Test Tubes with Cell Strainer Snap Cap | Corning | 352235 | |
| 60 mm Non TC-treated Culture Dish | Corning | 430589 | |
| APC Rat Anti-Mouse CD31 | BD Biosciences | 551262 | |
| APC Rat IgG2a κ Isotype Control | BD Biosciences | 553932 | |
| BD FACSChorus Software | BD Biosciences | FACSCHORUS | |
| BD FACSMelody Cell Sorter | BD Biosciences | FACSMELODY | |
| Collagenase Type II | Sigma-Aldrich | 234115 | |
| Costar 48-well Clear TC-treated Multiple Well Plates, Individually Wrapped, Sterile | Corning | 3548 | |
| D-Glucose | Gibco | A2494001 | |
| Disposable Graduated Transfer Pipettes | Fisher Scientific | 12-711-9AM | |
| Dissecting Pan Wax | Carolina | 629100 | |
| Dissection scissors | Fine Science Tools | 14085-08 | |
| Dissection Stereo Microscope M165 FC | Leica | M165FC | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-052 | |
| Dulbecco’s Phosphate Buffered Saline (PBS) | Gibco | 14190144 | |
| Eppendorf Flex-Tubes Microcentrifuge Tubes 1.5 mL | Sigma-Aldrich | 22364120 | |
| Fetal Bovine Serum (FBS) | Gemini Bio | 100-106 | |
| Fine dissection forceps | Fine Science Tools | 11250-00 | |
| Hank's Buffered Salt Solution (HBSS) | Gibco | 14175095 | |
| HEPES (1M) | Gibco | 15630130 | |
| iScript cDNA Synthesis Kit | Bio-Rad | 1708890 | |
| Isoflurane, USP | Covetrus | 11695067772 | |
| Isotemp General Purpose Deluxe Water Bath | Fisher Scientific | FSGPD20 | |
| Primer: ActB_Forward: 5’- agagggaaatcgtgcgtgac -3’ | Integrated DNA Technologies | N/A | |
| Primer: ActB_Reverse: 5’- caatagtgatgacctggccgt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Forward: 5’- gagcccaatcacgtttcagttt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Reverse: 5’- tccttcctgcttcttgctagct -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Forward: 5’- gggttgttctgtgccttgtt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Reverse: 5’- ctggacggacacagttagca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Forward: 5’- tcctctgcatcctcactatcaca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Reverse: 5’- gtaagtgaccaactgctcgtgaat -3’ | Integrated DNA Technologies | N/A | |
| Propidium iodide | Sigma-Aldrich | P4864 | |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| Sorvall Legend Micro 21R Centrifuge, Refrigerated | ThermoFisher | 75002477 | |
| SYBR-Green iTaq Universal SYBR Green Supermix | Bio-Rad | 172-5120 | |
| Trypan Blue Solution | ThermoFisher | 15250061 | |
| V450 Rat Anti-Mouse CD45 | BD Biosciences | 560501 | |
| V450 Rat IgG2b, κ Isotype Control | BD Biosciences | 560457 |
Referenzen
- Slatko, B. E., Gardner, A. F., Ausubel, F. M. Overview of next-generation sequencing technologies. Current Protocols in Molecular Biology. 122 (1), 59 (2018).
- Chavkin, N. W., Hirschi, K. K. Single cell analysis in vascular biology. Frontiers in Cardiovascular Medicine. 7, 42 (2020).
- Ma, F., Hernandez, G. E., Romay, M., Iruela-Arispe, M. L. Single-cell RNA sequencing to study vascular diversity and function. Current Opinion in Hematology. 28 (3), 221-229 (2021).
- Potter, A. S., Potter, S. S., S, Dissociation of tissues for single-cell analysis. Methods in Molecular Biology. 1926, 55-62 (2019).
- Braga, F. A. V., Miragaia, R. J. Tissue handling and dissociation for single-cell RNA-seq. Single Cell Methods: Methods in Molecular Biology. 1979, 9-21 (2019).
- Brink, S. C., et al. Single-cell sequencing reveals dissociatin-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Skulska, K., Wegrzyn, A. S., Chelmonska-Soyta, A., Chodaczek, G. Impact of tissue enzymatic digestion on analysis of immune cells in mouse reproductive mucosa with a focus on gammadelta T cells. Journal of Immunological Methods. 474, 112665 (2019).
- Connolly, S., Hores, T., Smith, L., D'Amore, P. Characterization of vascular development in the mouse retina. Microvascular Research. 36 (3), 275-290 (1988).
- Crist, A., Young, C., Meadows, S. Characterization of arteriovenous identity in the developing neonate mouse retina. Gene Expression Patterns: GEP. 23-24, 22-31 (2017).
- dela Paz, N. G., D'Amore, P. A. Arterial versus venous endothelial cells. Cell and Tissue Research. 335 (1), 5-16 (2009).
- Fang, J. S., Hirschi, K. K. Molecular regulation of arteriovenous endothelial cell specification. F1000Res. 8, (2019).
- Smith, L. E., et al. Oxygen-induced retinopathy in the mouse. Investigative Ophthalmology and Visual Science. 35 (1), 101-111 (1994).
- Pitulescu, M. E., Schmidt, I., Benedito, R., Adams, R. H. Inducible gene targeting in the neonatal vasculature and analysis of retinal angiogenesis in mice. Nature Protocols. 5 (9), 1518-1534 (2010).
- Ruiz, S., et al. A mouse model of hereditary hemorrhagic telangiectasia generated by transmammary-delivered immunoblocking of BMP9 and BMP10. Scientific Reports. 6, 37366 (2016).
- Fang, J. S., et al. Shear-induced Notch-Cx37-p27 axis arrests endothelial cell cycle to enable arterial specification. Nature Communication. 8 (1), 2149 (2017).
- Ola, R., et al. SMAD4 prevents flow induced arterial-venous malformations by inhibiting Casein Kinase 2. Circulation. 138 (21), 2379-2394 (2018).
- Chavkin, N. W., Walsh, K., Hirschi, K. K. Isolation of highly purified and viable retinal endothelial cells. Journal of Vascular Research. 58 (1), 49-57 (2020).
- Hulspas, R., O'Gorman, M. R. G., Wood, B. L., Gratama, J. W., Sutherland, D. R. Considerations for the control of background fluorescence in clinical flow cytometry. Cytometry PartB: Clinical Cytometry. 76 (6), 355-364 (2009).
- Bachman, J. Reverse-transcription PCR (RT-PCR). Methods in Enzymology. 530, 67-74 (2013).
- Green, M. R., Sambrook, J. Quantification of RNA by real-time Reverse Transcription-Polymerase Chain Reaction (RT-PCR). Cold Spring Harbour Protocols. 2018 (10), (2018).
- Liu, L., Shi, G. P. CD31: beyond a marker for endothelial cells. Cardiovascular Research. 94 (1), 3-5 (2012).
- Zarkada, G., et al. Specialized endothelial tip cells guide neuroretina vascularization and blood-retina-barrier formation. Developmental Cell. 56 (15), 2237-2251 (2021).
- Su, X., Sorenson, C. M., Sheibani, N. Isolation and characterization of murine retinal endothelial cells. Molecular Vision. 9, 171-178 (2003).
- Benedito, R., et al. The notch ligands Dll4 and Jagged1 have opposing effects on angiogenesis. Cell. 137 (6), 1124-1135 (2009).
- Daneman, R., et al. Wnt/beta-catenin signaling is required for CNS, but not non-CNS, angiogenesis. Proceedings of the National Academy of Sciences of the United States of America. 106 (2), 641-646 (2009).
- Okabe, K., et al. Neurons limit angiogenesis by titrating VEGF in retina. Cell. 159 (3), 584-596 (2014).
- Crist, A. M., Lee, A. R., Patel, N. R., Westhoff, D. E., Meadows, S. M. Vascular deficiency of Smad4 causes arteriovenous malformations: a mouse model of Hereditary Hemorrhagic Telangiectasia. Angiogenesis. 21 (2), 363-380 (2018).
- Kim, Y. H., Choe, S. W., Chae, M. Y., Hong, S., Oh, S. P. SMAD4 deficiency leads to development of arteriovenous malformations in neonatal and adult mice. Journal of the American Heart Association. 7 (21), 009514 (2018).
- Ma, W., et al. Absence of TGFbeta signaling in retinal microglia induces retinal degeneration and exacerbates choroidal neovascularization. eLife. 8, 42049 (2019).
- Luo, W., et al. Arterialization requires the timely suppression of cell growth. Nature. 589 (7842), 437-441 (2021).
- Lawson, N. D., Vogel, A. M., Weinstein, B. M. sonic hedgehog and vascular endothelial growth factor act upstream of the Notch pathway during arterial endothelial differentiation. Developmental Cell. 3 (1), 127-136 (2002).
- Larrivee, B., et al. ALK1 signaling inhibits angiogenesis by cooperating with the Notch pathway. Developmental Cell. 22 (3), 489-500 (2012).
- Wythe, J. D., et al. ETS factors regulate Vegf-dependent arterial specification. Developmental Cell. 26 (1), 45-58 (2013).
- Davis, D. M., Purvis, J. E. Computational analysis of signaling patterns in single cells. Seminars in Cell and Developmental Biology. , 35-43 (2015).
- Gaudet, S., Miller-Jensen, K. Redefining signaling pathways with an expanding single-cell toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14 (11), 1083-1086 (2017).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32 (4), 381-386 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten