A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
خط أنابيب للتحقيق في الهياكل ومسارات الإشارات لمستقبلات سفينغوزين 1-فوسفات
In This Article
Summary
يمارس S1P آثاره الفسيولوجية المتنوعة من خلال الفصيلة الفرعية لمستقبلات S1P (S1PRs). هنا ، يتم وصف خط أنابيب لشرح هياكل ووظيفة S1PRs.
Abstract
الليزوفوسفاتيدات (LPLs) هي دهون نشطة بيولوجيا تشمل سفينغوزين 1-فوسفات (S1P) ، وحمض الليسوفوسفاتيديك ، إلخ. S1P ، وهو منتج استقلابي من الدهون السفينغولية في غشاء الخلية ، هو واحد من أفضل LPLs المميزة التي تنظم مجموعة متنوعة من الاستجابات الفسيولوجية الخلوية عبر مسارات الإشارات بوساطة مستقبلات سفينغوزين 1-فوسفات (S1PRs). هذا ينطوي على أن نظام إشارات S1P-S1PRs هو هدف علاجي محتمل ملحوظ للاضطرابات ، بما في ذلك التصلب المتعدد (MS) ، واضطرابات المناعة الذاتية ، والسرطان ، والالتهابات ، وحتى COVID-19. تتكون S1PRs ، وهي مجموعة فرعية صغيرة من عائلة مستقبلات البروتين المقترن من الفئة A G (GPCR) ، من خمسة أنواع فرعية: S1PR1 و S1PR2 و S1PR3 و S1PR4 و S1PR5. ومع ذلك ، فإن الافتقار إلى المعلومات الهيكلية التفصيلية يعوق اكتشاف الأدوية التي تستهدف S1PRs. هنا ، طبقنا طريقة المجهر الإلكتروني المبرد لحل بنية مجمع S1P-S1PRs ، وأوضحنا آلية التنشيط ، والتعرف الانتقائي على الأدوية ، واقتران بروتين G باستخدام الفحوصات الوظيفية القائمة على الخلايا. يمكن أيضا دراسة مستقبلات الليزوفوسفوليبيد الأخرى (LPLRs) و GPCRs باستخدام هذه الاستراتيجية.
Introduction
Sphingosine-1-phosphate (S1P) ، وهو منتج استقلابي من الدهون السفينغولية في غشاء الخلية ، هو جزيء إشارات ليسوفوسفاتيدي في كل مكان يتضمن أنشطة بيولوجية مختلفة ، بما في ذلك الاتجار بالخلايا الليمفاوية ، وتطور الأوعية الدموية ، والسلامة البطانية ، ومعدل ضربات القلب1،2،3. يمارس S1P آثاره الفسيولوجية المتنوعة من خلال خمسة أنواع فرعية من مستقبلات S1P (S1PRs 1-5) ؛ تم العثور على S1PRs في مجموعة متنوعة من الأنسجة وتظهر تفضيلات فريدة لبروتينات G المصب 4,5. يقترن S1PR1 في المقام الأول ببروتين Gi ، الذي يمنع لاحقا إنتاج cAMP ؛ يقترن S1PR2 و S1PR3 ب Gi و Gq و G12/13 ، و S1PR4 و S1PR5 ينقلان الإشارة من خلال Gi و G12/136.
تعد إشارات S1P-S1PR هدفا علاجيا حاسما لأمراض متعددة ، بما في ذلك اضطرابات المناعة الذاتية7 والالتهاب8 والسرطان9 وحتى COVID-1910. في عام 2010 ، تم ترخيص fingolimod (FTY720) كأول دواء في فئته يستهدف S1PRs لعلاج التصلب المتعدد الانتكاسي (MS)11. ومع ذلك ، فهو قادر على الارتباط بجميع S1PRs باستثناء S1PR2 ، في حين أن الارتباط غير المحدد ب S1PR3 يؤدي إلى وذمة القشرة الدماغية ، وانقباض الأوعية الدموية والشعب الهوائية ، والتسرب الظهاري الرئوي12. كاستراتيجية بديلة لزيادة الانتقائية العلاجية ، تم إنتاج روابط خاصة بالنوع الفرعي للمستقبلات. تمت الموافقة على Siponimod (BAF312) في عام 2019 لعلاج التصلب المتعدد الانتكاسي13 ؛ يستهدف بشكل فعال S1PR1 و S1PR5 ، في حين أنه ليس لديه تقارب مع S1PR3 ، مما يدل على آثار جانبية أقل في الممارسة السريرية14. في عام 2020 ، أذنت إدارة الغذاء والدواء الأمريكية ب ozanimod لعلاج مرض التصلب العصبي المتعدد15. وقد أفيد أن أوزانيمود يحمل انتقائية أكبر بمقدار 25 ضعفا ل S1PR1 مقارنة ب S1PR516. والجدير بالذكر أنه في سياق جائحة COVID-19 الحالية ، تم اكتشاف أن الأدوية الناهضة التي تستهدف S1PRs يمكن استخدامها لعلاج COVID-19 باستخدام تقنيات العلاج المناعي17. بالمقارنة مع فينجوليمود ، أظهر أوزانيمود تفوقا في خفض الأعراض لدى مرضى COVID-19 ويخضع الآن للتجارب السريرية10. إن فهم الأساس الهيكلي ووظيفة S1PRs يضع أساسا مهما لتطوير دواء يستهدف بشكل انتقائي S1PRs18.
تستخدم العديد من التقنيات للتحقيق في المعلومات الهيكلية للجزيئات الحيوية الكبيرة ، بما في ذلك علم البلورات بالأشعة السينية ، والرنين المغناطيسي النووي (NMR) ، والمجهر الإلكتروني (EM). اعتبارا من مارس 2022 ، هناك أكثر من 180،000 هيكل مودع في بنك بيانات البروتين (PDB) ، وقد تم حل معظمها عن طريق علم البلورات بالأشعة السينية. ومع ذلك ، مع أول بنية دقة شبه ذرية ل TPRV1 (دقة 3.4 Å) التي أبلغ عنها Yifan Cheng و David Julius في 201319 ، أصبح المجهر الإلكتروني المبرد (cryo-EM) تقنية سائدة لهياكل البروتين ، وكان العدد الإجمالي لهياكل EM PDB أكثر من 10000. تتمثل مجالات الاختراق الحاسمة في تطوير كاميرات جديدة للتصوير تعرف باسم كاميرات الكشف المباشر عن الإلكترونات وخوارزميات معالجة الصور الجديدة. أحدثت Cryo-EM ثورة في بيولوجيا البنية واكتشاف الأدوية القائمة على البنية في العقد الماضي20. نظرا لأن فهم كيفية قيام المجمعات الجزيئية الكبيرة بأدوارها المعقدة في الخلية الحية هو موضوع مركزي في العلوم البيولوجية ، فإن cryo-EM لديه القدرة على الكشف عن تكوينات المجمعات الجزيئية الديناميكية ، خاصة بالنسبة للبروتينات عبر الغشاء21. مستقبلات G-protein المقترنة (GPCRs) هي أكبر عائلة فائقة من البروتينات عبر الغشاء وأهداف أكثر من 30٪ من المستحضرات الصيدلانية المسوقة حاليا22. ساهم تطوير cryo-EM في انفجار الهياكل عالية الدقة لمجمعات بروتين GPCR-G ، مما مكن من التحديد الهيكلي للأهداف "المستعصية" التي لا تزال غير متاحة للتحليل البلوري بالأشعة السينية في تصميم الأدوية23. وبالتالي ، يوفر تطبيق cryo-EM فرصة لتحديد البنية ثلاثية الأبعاد ل GPCRs في ظروف قريبة من الأصل بالقرب من الدقة الذرية24. إن التقدم في مجال التبريد الكهرومغناطيسي يجعل من الممكن تصور الأسس الميكانيكية لتحفيز GPCR أو تثبيطه ، والاستفادة بشكل أكبر في الكشف عن مواقع الربط الجديدة لإنشاء الأدوية المستهدفة GPCR25.
بالاعتماد على الخطوات الهائلة لتقنية cryo-EM ، حددنا هياكل مجمعات إشارات S1PR1 و S1PR3 و S1PR5-Gi المؤلمة مؤخرا26,27. في البشر ، توجد S1PRs في أنسجة مختلفة وتظهر تفضيلات فريدة لبروتينات G 4,5 في المراحل النهائية. يقترن S1PR1 في المقام الأول ببروتين Gi ، الذي يمنع لاحقا إنتاج أحادي فوسفات الأدينوسين الدوري 3 ′ ، 5 ′ (cAMP). S1PR3 و S1PR5 قادران أيضا على الاقتران مع Gi 6,28. نظرا لأن تنشيط مستقبلات Gi-pair يقلل من إنتاج cAMP29 ، فقد تم إدخال فحص cAMP لتثبيط Gi لقياس تأثيرات تثبيط cAMP لالتقاط التعديلات الوظيفية26,27. باستخدام نسخة متحولة من Photinus pyralis luciferase حيث تم إدخال بروتين ملزم ل cAMP ، يوفر فحص cAMP هذا طريقة بسيطة وموثوقة لمراقبة نشاط GPCR من خلال التغيرات في تركيز cAMPداخل الخلايا 30. وهو اختبار وظيفي حساس وغير مشع ويمكن تطبيقه لمراقبة الإشارات النهائية في الوقت الحقيقي لمجموعة واسعة من GPCRs لأغراض اكتشاف الأدوية31.
هنا ، يتم تقديم ملخص للطرق الحاسمة في حل طرق التنشيط والتعرف على الأدوية ل S1PRs ، بما في ذلك في المقام الأول التلاعب بالتبريد EM وفحص cAMP المثبط Gi. تهدف هذه المقالة إلى توفير إرشادات تجريبية شاملة لمزيد من الاستكشافات في هياكل ووظائف GPCRs.
Protocol
1. تنقية مركب البروتين S1PRs-G
- لتنقية مركب البروتين S1PRs-G البشري ، قم باستنساخ cDNAs من S1PR1 التي تفتقر إلى بقايا C-terminal 338-382 ، والنوع البري S1PR3 ، و S1PR5 المبتور مع 345-398 في المحطة C ، والنوع البري Gi1 في ناقل pFastBac1 و cDNAs من النوع البري Gβ1 و Gγ2 في ناقل pFastBacdual (جدول المواد).
ملاحظة: تحتوي جميع التركيبات الخاصة ب S1PRs أيضا على تسلسل إشارة الهيماجلوتينين (HA) متبوعا بعلامة Flag epitope في المحطة N وعلامة 10x الخاصة به عند C-terminus. أيضا ، تم إدخال تسلسل الحمض النووي الاصطناعي (جدول المواد) لترجمة الليزوزيم T4 (T4L) في المحطة N من S1PRs من أجل تسهيل التعبير عن المستقبلات وتنقيتها. - تحضير الفيروس البقعي الذي يشفر S1PRs و Gi1 و Gβ1γ2
- أضف المتجهات المؤتلفة إلى 50 ميكرولتر من الإشريكية القولونية المختصة DH5α (E. coli) المخزنة في أنبوب 1.5 مل عند -80 درجة مئوية ، واحتضنها على الجليد لمدة 30 دقيقة.
- قم بصدمة الخلايا بالحرارة عند 42 درجة مئوية لمدة 90 ثانية ، وانقلها إلى الجليد على الفور ، وقم بتبريدها لمدة 2 دقيقة.
- رج الأنبوب عند 37 درجة مئوية لمدة 3-5 ساعات بعد تناول 300 ميكرولتر من وسط مرق الليسوجيني (LB). لوحة 100 ميكرولتر من الخلايا إلى لوحة أجار LB وحضانت عند 37 درجة مئوية عن طريق البقاء في الظلام لمدة 48 ساعة.
- قم بتلقيح المستعمرة البيضاء إلى 5 مل من وسط LB يحتوي على 50 ميكروغرام / مل كاناميسين ، و 10 ميكروغرام / مل تتراسيكلين ، و 7 ميكروغرام / مل جنتاميسين ، والاستزراع عند 37 درجة مئوية لمدة 16 ساعة.
- اعزل الباكميد المؤتلف باستخدام مجموعة البلازميد miniprep (جدول المواد) باتباع تعليمات الشركة المصنعة ، لإنتاج الفيروس البقعي P0.
ملاحظة: قبل الاستخدام ، تم تحليل bacmid النقي بواسطة PCR باستخدام pUC / M13 التمهيدي الأمامي والخلفي. بالنسبة ل PCR ، عدد الدورات = 30 دورة ، ودرجة حرارة الانصهار = 58 درجة مئوية ، ووقت التمديد = 1 دقيقة لكل 1 كيلو بايت. - تحضير الفيروس البقعي P0 كما هو موضح في بروتوكول سابق32.
- قم بزراعة خلايا sf9 (وسط ESF921) في ألواح من ستة آبار وتحقق من أن الخلايا في مرحلة السجل (1.0-1.5 × 106 خلايا / مل).
- تمييع 8 ميكرولتر من كاشف نقل الفيروس البقعي في 100 ميكرولتر من وسط غريس، واحتضان لمدة 30 دقيقة في درجة حرارة الغرفة. تمييع 10 ميكروغرام من باكميد المؤتلف في 100 ميكرولتر من وسط غريس، وتخلط بلطف. امزج الباكميد المخفف مع كاشف نقل الفيروس البقعي المخفف ، واخلطه بلطف ، واحتضنه لمدة 30 دقيقة في درجة حرارة الغرفة.
- يضاف الخليط (الباكميد والكاشف) على الخلايا (الخطوة 1.2.6.1) ، ويطحنها على درجة حرارة 27 درجة مئوية لمدة 3 ساعات.
- قم بإزالة وسط Grace واستبدله ب 2 مل من وسط زراعة الخلايا ESF921. قم بطلاء الألواح ذات الستة آبار عند 27 درجة مئوية واجمع وسط زراعة الخلايا ESF921 بعد 5 أيام من النقل.
- جهاز طرد مركزي عند 500 × جم عند 4 درجات مئوية لمدة 10 دقائق لإزالة الخلايا والحطام. انقل السوبرناتانت إلى أنابيب 2 مل. هذا هو مخزون الفيروس البقعي P0.
- عزل مخزون فيروس P1
- انقل 30 مل من خلايا sf9 إلى زجاجة مخروطية ، وزرع عند 27 درجة مئوية مع اهتزاز عند 270 دورة في الدقيقة ، وتحقق من أن الخلايا تصل إلى مرحلة السجل (1.0-1.5 × 106 خلايا / مل).
- أضف 2 مل من مخزون فيروس P0 إلى الزجاجة ورجه عند 270 دورة في الدقيقة لمدة 4 أيام عند 27 درجة مئوية.
- انقل الخلايا إلى أنبوب 50 مل ، وأجهزة طرد مركزي عند 1800 × جم لمدة 10 دقائق عند 4 درجات مئوية لإزالة الخلايا والحطام ، ونقل السوبرنات إلى أنابيب 50 مل. هذا هو مخزون الفيروس البقعي P1.
- تضخيم مخزون الفيروسات البقعية
- كرر الخطوة 1.2.7 باستخدام 50 مل من خلايا sf9 في مرحلة السجل (1.0-1.5 × 106 خلايا/مل) و 1 مل من مخزون P1.
- قم بتخزين مخزون الفيروس البقعي P2 الناتج عند 4 درجات مئوية ، مما يحميه من الضوء.
ملاحظة: لا تضخم الفيروس البقعي إلى أجل غير مسمى ، حيث تم إنتاج الطفرات الضارة مع كل مقطع.
- التعبير عن مركب البروتين S1PRs-G
- استزراع خلايا الحشرات sf9 للوصول إلى كثافة 2.5 × 106 خلايا / مل ، وتشارك في العدوى مع الفيروس البقعي P2 الذي يشفر S1PRs و Gi1 و Gβ1γ2 بنسبة حجم 1: 2: 1 ، والاستزراع مرة أخرى عند 27 درجة مئوية لمدة 48 ساعة.
- جمع الخلايا عن طريق الطرد المركزي في 700 × غرام عند 4 درجات مئوية لمدة 15 دقيقة ، وتجميدها في النيتروجين السائل ، وتخزينها في -80 درجة مئوية للاستخدام.
- تنقية البروتين
- قم بإذابة حبيبات الخلية التي تم الحصول عليها في الخطوة 1.3 في درجة حرارة الغرفة ، ثم أعد تعليقها في مخزن مؤقت للتحلل (20 mM HEPES pH 7.5 ، 50 mM NaCl ، 5 mM MgCl 2 ، 5 mM CaCl2) مع 100 ميكروغرام / مل بنزاميدين ، 100 ميكروغرام / مل ليوبيبتين ، 100 ميكروغرام / مل أبروتينين ، 25 مللي أمبيريز / مل ، وناهض 10 ميكرومتر. حرك تعليق الخلية في درجة حرارة الغرفة لمدة 2 ساعة للحث على تكوين مركب البروتين S1PRs-G.
ملاحظة: Apyrase هو ATP ثنائي فوسفوهيدرولاز. إنه يحفز إزالة فوسفات غاما من ATP وفوسفات بيتا من ADP. - انقل المحلول إلى الأنابيب ، وأجهزة الطرد المركزي عند 70000 × g لمدة 10 دقائق ، وقم بإزالة supernatant بعناية. أعد تعليق الكريات في المخزن المؤقت القابل للذوبان (20 mM HEPES pH 7.5 ، 100 mM NaCl ، 5 mM MgCl 2 ، 5 mM CaCl2 ، 0.5٪ (w / v) LMNG ، 0.1٪ (w / v) CHS ، 1٪ (w / v) cholate sodium ، 10٪ (v / v) glycerol).
- انقل التعليق إلى كوب Dounce وقم بتجانس الكريات بالكامل. أضف ناهض 10 ميكرومتر ، و 4 ملغ من scFv16 ، و 100 ميكروغرام / مل بنزاميدين ، و 100 ميكروغرام / مل ليوبيبتين ، و 100 ميكروغرام / مل أبروتينين ، و 25 مللي أمبيريز / مل إلى التعليق ، ويحرك عند 4 درجات مئوية لمدة 2 ساعة.
ملاحظة: خطوات تجانس الكريات ضرورية لإنتاج مركب البروتين GPCR-G. - انقل المحلول إلى الأنابيب ، وأجهزة الطرد المركزي عند 100000 × g لمدة 30 دقيقة عند 4 درجات مئوية.
- قم بموازنة راتنج العلم مسبقا مع المخزن المؤقت للغسيل (20 mM HEPES pH 7.5 ، 100 mM NaCl ، 5 mM MgCl 2 ، 5 mM CaCl2 ، ناهض 10 μM ، 0.0375٪ (w / v) LMNG ، 0.0125٪ (w / v) GDN ، 0.01٪ (w / v) CHS).
- انقل السوبرناتانت إلى الأنابيب ، واحتضنها مع راتنج العلم عند 4 درجات مئوية لمدة 2 ساعة.
- قم بتحميل الخليط أعلاه على العمود الزجاجي واغسل العمود ب 50 مل من الغسيل العازل المكمل ب 100 ميكروغرام / مل بنزاميدين ، و 100 ميكروغرام / مل ليوبيبتين ، و 100 ميكروغرام / مل من الأبروتينين.
- قم بإخراج العمود ب 10 مل من المخزن المؤقت للإزالة الذي يحتوي على 20 mM HEPES pH 7.5 ، 100 mM NaCl ، 5 mM EDTA ، 200 ميكروغرام / مل ببتيد العلم ، ناهض 10 ميكرومتر ، 0.0375٪ (w / v) LMNG ، 0.0125٪ (w / v) GDN ، 0.01٪ (w / v) CHS ، 100 ميكروغرام / مل بنزاميدين ، 100 ميكروغرام / مل ليوبيبتين ، و 100 ميكروغرام / مل أبروتينين.
- اجمع مركب البروتين S1PRs-G وركزه على 1 مل باستخدام مكثف قطع 100 كيلو دالتون عند 1300 × جم عند 4 درجات مئوية. قم بالتصفية من خلال مرشح 0.22 ميكرومتر ، وأجهزة الطرد المركزي عند 13000 × g لمدة 10 دقائق عند 4 درجات مئوية لإزالة الركام.
- قم بتحميل مركب البروتين S1PRs-G على عمود ترشيح هلام كروماتوغرافي استبعاد الحجم (SEC) متوازن مسبقا مع المخزن المؤقت SEC الذي يحتوي على 20 mM HEPES pH 7.5 و 100 mM NaCl وناهض 10 μM و 100 Μm TCEP و 0.00375٪ (w/v) LMNG و 0.00125٪ (w/v) GDN و 0.001٪ (w/v) CHS بمعدل تدفق 0.5 مل / دقيقة عند 4 درجات مئوية.
- اجمع كسور الذروة وركز باستخدام مكثف قطع 100 كيلو دال أمبير عند 1300 × جم عند 4 درجات مئوية للتبريد-EM.
- قم بإذابة حبيبات الخلية التي تم الحصول عليها في الخطوة 1.3 في درجة حرارة الغرفة ، ثم أعد تعليقها في مخزن مؤقت للتحلل (20 mM HEPES pH 7.5 ، 50 mM NaCl ، 5 mM MgCl 2 ، 5 mM CaCl2) مع 100 ميكروغرام / مل بنزاميدين ، 100 ميكروغرام / مل ليوبيبتين ، 100 ميكروغرام / مل أبروتينين ، 25 مللي أمبيريز / مل ، وناهض 10 ميكرومتر. حرك تعليق الخلية في درجة حرارة الغرفة لمدة 2 ساعة للحث على تكوين مركب البروتين S1PRs-G.
2. المجهر الإلكتروني لحل بنية S1PRs
- جمع البيانات
- لإعداد شبكات cryo-EM ، امسك شبكات 300-mesh Au R1.2 / 1.3 لمدة 10 ثوان وتفريغ التوهج لمدة 60 ثانية عند 15 مللي أمبير باستخدام نظام تنظيف تفريغ التوهج.
- قم بإجراء تزجيج العينة كما هو موضح سابقا33,34. في وحدة التحكم في الغطس المتجمد، اضبط درجة الحرارة على 4 درجات مئوية والرطوبة النسبية على 100٪ لبيئة عمل الغرفة. استخدم قوة اللطخة لمدة 0 ، ووقت الانتظار من 0 ثانية ، ووقت اللطخة من 2 أو 3 ثانية ، ووقت التصريف من 0 ثانية. وعادة ما يتطلب فقط 3 ميكرولتر من العينة بتركيزات 5-10 ملغم/مل للتزجيج المفرد.
- قم بقص الشبكات وتحميلها في تجميع الشبكة التلقائي ، وتحميل تجميع الشبكة التلقائي في Nanocab ، وتحميل Nanocab في المجهر بواسطة المحمل التلقائي كما هو موضح سابقا35. جودة عينة الشاشة مع برنامج EPU234. عادة ، كانت البيانات التي تم جمعها في المناطق ذات سمك الجليد المناسب أفضل.
- جمع بيانات cryo-EM كما هو موضح بالتفصيل سابقا35. بالنسبة لمستقبل S1P ، اضبط إزاحة إلغاء التركيز البؤري بين -1.0 ميكرومتر و -1.8 ميكرومتر مع جرعة إلكترون التعرض من 50-65 e-/Å2. بالنسبة لمجمعات S1PR1-Gi ، قم بجمع مكدس الأفلام تلقائيا باستخدام برنامج EPU2 في وضع العد مع كاشف K2 في وقت تعرض إجمالي يبلغ 2 ثانية ، ومعدل تسجيل يبلغ خمسة إطارات خام في الثانية ، وجرعة إجمالية تبلغ 56 e-/Å2 لإنتاج 35 إطارا لكل مكدس.
ملاحظة: عادة ما تكون هناك حاجة إلى أكثر من 5000 فيلم لإعادة بناء بنية المستقبلات.
- معالجة البيانات باستخدام مزيج من RELION36 و cryoSPARC37 للحصول على خريطة مثالية لكثافة cryo-EM. استخدم RELION-3.1_gpu_ompi4 لمعالجة البيانات في البداية والتي تنطوي على عمليات مماثلة كما هو موضح سابقا34.
- في محطة نظام Linux، أدخل الدليل الأصل لدليل تخزين البيانات.
- أدخل الأمر reon في المحطة الطرفية لفتح واجهة المستخدم الرسومية RELION (GUI).
ملاحظة: إذا كانت هذه هي المرة الأولى التي يتم فيها فتح واجهة المستخدم الرسومية RELION في هذا الدليل ، فستظهر نافذة مطالبة ؛ انقر فوق نعم. - انقر فوق استيراد في شريط الوظائف على الجانب الأيمن من واجهة المستخدم الرسومية RELION لاستيراد البيانات الخام إلى RELION.
- في الخيار أفلام/ميكروفونات، حدد نعم لاستيراد أفلام/ميكروغرافات خام؟، وأدخل مسار البيانات في حقل ملفات الإدخال الخام (يوصى باستخدام أحرف البدل)، وحدد نعم ل هل هذه الأفلام متعددة الإطارات؟. أدخل حجم البكسل للأفلام في حقل حجم البكسل (Angstrom)، وجهد تشغيل المجهر (بالكيلوفولت) في حقل الجهد (kV)، والانحراف الكروي للمجهر في حقل الانحراف الكروي (mm). هذه هي المعلمات التي تم تسجيلها في وقت جمع البيانات.
- في الخيار قيد التشغيل ، قم بتعديل اسم قائمة الانتظار وفقا للخادم الذي يعمل عليه البرنامج (تحتاج الوظائف الأخرى أيضا إلى تعديل هذه المعلمة). اترك المعلمات الأخرى عند القيم الافتراضية التي تم تعيينها بواسطة RELION. أخيرا ، عند اكتشاف كافة المعلمات بشكل صحيح ، انقر فوق تشغيل! لتشغيل البرنامج.
- استخدم وظيفة تصحيح الحركة لمحاذاة جميع الإطارات38.
- في خيار الإدخال / الإخراج ، انقر فوق استعراض واختر إخراج وظيفة الاستيراد المسماة movies.star كإدخال لملف STAR لأفلام الإدخال. أدخل الجرعة لكل إطار في حقل الجرعة لكل إطار (e/A2) الذي يساوي الجرعة الإجمالية مقسوما على عدد الإطارات. حدد لا لحفظ مجموع أطياف الطاقة؟.
- في خيار الحركة، أدخل 250 للعامل B، و5.5 لعدد التصحيحات X، Y، و2 لإطارات المجموعة (تأكد من جرعة المجموعة >3). إذا لم يتم الرجوع إلى البيانات خلال فترة التجميع ، فستكون هناك حاجة إلى صورة مرجعية مكتسبة يمكن الحصول عليها عن طريق الحصول على مساحة شبكة فارغة. حدد لا لاستخدام تنفيذ RELION الخاص؟ وأدخل الدليل الذي يحتوي على الملف القابل للتنفيذ الخاص ب MOTIONCOR2 39في الحقل القابل للتنفيذ MOTIONCOR2.
- في الخيار قيد التشغيل ، اختر MPI المناسب ورقم مؤشر الترابط وفقا لقوة الحوسبة للخادم ؛ هنا ، تم استخدام MPI = 8 والمواضيع = 3.
- استخدم وظيفة تقدير CTF لتعديل صورة cryo-EM للعينة المزججة40. في خيار الإدخال / الإخراج ، انقر فوق استعراض واختر إخراج Motion cor المسمى micrographs.star المصحح كإدخال لملف STAR لأفلام الإدخال. في خيار Gctf، حدد نعم ل UseGctf بدلا من ذلك؟.
- استخدم دالة تحديد المجموعة الفرعية لإزالة الصور المجهرية بقيمة rlnCtfMaxResolutin >4.
- في خيار الإدخال / الإخراج ، انقر فوق تصفح الموجود على يمين OR حدد من micrographs.star واختر إخراج CtfFind المسمى micrographs_ctf.star كمدخل. في خيار المجموعة الفرعية، حدد نعم للتحديد استنادا إلى قيم بيانات التعريف؟ وأدخل 4 لقيمة الحد الأقصى لبيانات التعريف لإزالة البيانات التالفة.
- الانتقاء اليدوي: اختر بعض الصور يدويا للاختيار والتصنيف الأولي.
- في خيار الإدخال / الإخراج ، انقر فوق استعراض واختر micrographs_selected.star من دليل التحديد السابق (الخطوة 2.2.6) كإدخال.
- انقر على تشغيل! (نافذة تنبثق). انقر فوق ملف في الزاوية العلوية اليسرى من النافذة الجديدة وانقر فوق عكس التحديد لإلغاء تحديد جميع الصور. حدد مربع التحديد في أقصى اليسار في الصف المقابل لكل إدخال وانقر فوق اختيار للتحقق من الصور وتحديد ~ 500 صورة جيدة. انقر فوق ملف > حفظ التحديد لحفظ الصور المحددة وإغلاق النافذة.
- الانتقاء التلقائي: تعد حزم برامج التقاط الجسيمات الآلية مفيدة وقوية41.
- في خيار الإدخال / الإخراج ، انقر فوق استعراض إلى يمين الميكروجرافات المدخلة للاختيار التلقائي واختر micrographs_selected.star من دليل ManualPick السابق (الخطوة 2.2.7) كمدخل. يتم استخدام خوارزمية Laplacian-of-Gaussian في البداية ، لذا حدد نعم ل OR: استخدم Laplacian-of-Gaussian.
- في خيار Laplacian ، اضبط Min.diameter لمرشح LoG (A) على 80 و Max.diameter لمرشح LoG (A) على 130. في خيار الانتقاء التلقائي، اضبط الحد الأدنى لمسافة الجسيمات (A) على 65، وحدد نعم لاستخدام تسريع وحدة معالجة الرسومات إذا كان من الممكن الوصول إلى وحدة معالجة الرسومات.
- استخراج الجسيمات للخطوات التالية.
- في خيار الإدخال / الإخراج ، انقر فوق استعراض على يمين ملف STAR المجهري واختر micrographs_selected.star من الخطوة 2.2.6. انقر فوق استعراض على يمين إحداثيات الإدخال واختر cords_suffix_autopick.star من الخطوة 2.2.8.
- في خيار استخراج، حدد نعم لجسيمات إعادة القياس واضبط الحجم المعاد قياسه (بيكسلات) على 128 لتسريع الخطوات اللاحقة.
- تصنيف 2D للتصنيف الأولي للجسيمات
- في خيار الإدخال/الإخراج، انقر فوق استعراض على يمين ملف STAR لإدخال الصور واختر particles.star من الخطوة 2.2.9. في خيار التحسين، اضبط عدد الفئات على 100 وقطر القناع (A) على 140.
- في خيار الحساب، اضبط عدد الجسيمات المجمعة على 10، وأدخل الدليل الموجود على محرك أقراص محلي سريع (على سبيل المثال، محرك أقراص SSD) في حقل نسخ الجسيمات إلى دليل الخدش، وحدد نعم لاستخدام تسريع GPU؟ للحصول على سرعة معالجة أسرع.
- اختيار مجموعة فرعية لاختيار نتائج 2D جيدة كقوالب لاختيار الجسيمات
- في خيار الإدخال / الإخراج ، انقر فوق استعراض إلى يمين تحديد الفئات من model.star واختر إخراج الخطوة 2.2.10 المسمى run_it025_model.star كمدخل. انقر على تشغيل!. في النافذة المنبثقة، حدد فرز الصور وعكس الفرز؟ وانقر على عرض!.
- اختر نتائج 2D تمثيلية جيدة كمرجع لوظيفة الانتقاء التلقائي.
ملاحظة: يتم وضع النتائج المحددة في مربع باللون الأحمر. يتم عرض نتائج تصنيف 2D الجيدة والسيئة لاحقا. - انقر بزر الماوس الأيمن وحدد حفظ الفئات المحددة.
- استخدم القالب للجولة الثانية من الانتقاء التلقائي. في خيار الإدخال / الإخراج ، انقر فوق تصفح على يمين الميكروجرافات المدخلة للاختيار التلقائي واختر micrographs_selected.star من الخطوة 2.2.6. انقر فوق استعراض على يمين المراجع ثنائية الأبعاد ، واختر class_averages.star من الخطوة 2.2.11 ، وحدد لا ل OR: استخدم Laplacian-of-Gaussian.
- قم بإجراء استخراج الجسيمات مرة أخرى باستخدام coord_suffix_autopick.star من الخطوة 2.2.12 و micrographs_selected.star من الخطوة 2.2.6.
- قم بإجراء تصنيف ثنائي الأبعاد مرة أخرى باستخدام particles.star من الخطوة 2.2.13.
- قم بإجراء تحديد مجموعة فرعية مرة أخرى باستخدام run_it025_optimiser.star من الخطوة 2.2.14.
ملاحظة: يجب اختيار جميع الصور 2D ذات الخطوط الواضحة والأشكال الصحيحة. - قم بإجراء استخراج الجسيمات على النحو التالي. في خيار الإدخال/الإخراج ، انقر فوق استعراض على يمين ملف STAR المجهري، واختر micrographs_selected.star من الخطوة 2.2.6، وحدد نعم ل OR إعادة استخراج الجسيمات المكررة؟. انقر فوق استعراض على يمين ملف STAR للجسيمات المكررة واختر particles.star من الخطوة 2.2.15.
- نموذج أولي ثلاثي الأبعاد وإنشاء خريطة مرجعية: في خيار الإدخال / الإخراج ، انقر فوق تصفح إلى يمين ملف STAR لإدخال الصور واختر particles.star من الخطوة 2.2.16. اضبط عدد الفئات على 1، وقطر القناع (A) على 140 في خيار التحسين .
- تصنيف ثلاثي الأبعاد وإنشاء خريطة ثلاثية الأبعاد أولية: في خيار الإدخال / الإخراج ، انقر فوق تصفح إلى يمين ملف STAR لإدخال الصور واختر particles.star من الخطوة 2.2.16. انقر فوق استعراض على يمين الخريطة المرجعية واختر initial_model.mrc من الخطوة 2.2.17. اضبط عدد الفئات على 4-6، وقطر القناع (A) على 140 في خيار التحسين .
- إنشاء قناع: حدد خريطة (خرائط) 3D جيدة من الخطوة 2.2.17 كإدخال في خيار الإدخال / الإخراج . اضبط عتبة الربط الأولي على 0.05 (ضبط استنادا إلى الإخراج)، وقم بتوسيع الخريطة الثنائية التي يبلغ عدد وحدات البكسل هذه إلى 3، وأضف حافة ناعمة من هذا العدد الكبير من وحدات البكسل إلى 8 في خيار القناع .
- استخدم cryoSPARC للمعالجة التالية.
- قم بإنشاء مساحة عمل جديدة وانقر على منشئ الوظائف للوظيفة الأولى.
- لاستيراد مكدس الجسيمات، أدخل مسار الجسيمات (الخطوة 2.2.16) في حقل مسار تعريف الجسيمات ومسار الفيلم (الخطوة 2.2.16) في حقل مسار بيانات الجسيمات .
ملاحظة: المعلمات تسريع الجهد (kV) ، الانحراف الكروي (mm) ، وحجم البكسل (Angstrom) هي نفسها كما كانت من قبل. قيمة تباين السعة (الكسر ) هي 0.1. - استيراد وحدات تخزين ثلاثية الأبعاد عن طريق إدخال مسار أفضل وحدة تخزين ثلاثية الأبعاد في الخطوة 2.2.18 في مسار بيانات وحدة التخزين وتحديد خريطة لنوع وحدة التخزين التي يتم استيرادها.
- قم باستيراد قناع عن طريق إدخال مسار القناع (الخطوة 2.2.19) في مسار بيانات وحدة التخزين وتحديد قناع لنوع وحدة التخزين التي يتم استيرادها.
- التنقيح غير الموحد: خذ مخرجات الخطوات 2-2-22 و2-2-23 و2-2-24 كمدخلات.
ملاحظة: هذه الوظيفة مفيدة جدا لبروتينات الغشاء.- اسحب مخرجات الخطوة 2.2.22 (imported_particles) كمدخلات لجسيمات الصقل غير المنتظم (الجسيمات)، ومخرجات الخطوة 2.2.23 (imported_volume_1) كمدخلات لحجم (حجم) الصقل غير الموحد، ومخرجات الخطوة 2.2.24 ( imported_mask_1) كمدخلات لقناع (قناع) الصقل غير الموحد.
ملاحظة: في بعض الأحيان يمكن تحقيق نتائج أفضل بدون قناع. - انقر فوق قائمة الانتظار لبدء المعالجة.
- اسحب مخرجات الخطوة 2.2.22 (imported_particles) كمدخلات لجسيمات الصقل غير المنتظم (الجسيمات)، ومخرجات الخطوة 2.2.23 (imported_volume_1) كمدخلات لحجم (حجم) الصقل غير الموحد، ومخرجات الخطوة 2.2.24 ( imported_mask_1) كمدخلات لقناع (قناع) الصقل غير الموحد.
- قم بتشغيل الخطوات 2.2.18-2.2.25 للحصول على نتائج أفضل. من خلال سلسلة المعالجة المذكورة أعلاه ، يمكن الحصول على خريطة S1PR-Gi 3D جيدة الدقة.
3. S1PRs-Gi بوساطة اختبار تثبيط cAMP
ملاحظة: تم تقسيم تجربة تثبيط cAMP بوساطة S1PRs-Gi إلى عدة أجزاء، وفيما يلي إجراءات تجريبية مفصلة. يظهر المبدأ التجريبي والعملية التجريبية العامة في شكل مخطط انسيابي في الشكل 1.
- بناء البلازميدات
- قم باستنساخ cDNAs من النوع البري S1PR1 و S1PR3 و S1PR5 في pcDNA3.1+ المتجه مع تسلسل إشارة HA متبوعا بعلامة علم في المحطة N (جدول المواد).
ملاحظة: تم إنشاء طفرات لجميع المستقبلات باستخدام مجموعة الطفرات (جدول المواد).
- قم باستنساخ cDNAs من النوع البري S1PR1 و S1PR3 و S1PR5 في pcDNA3.1+ المتجه مع تسلسل إشارة HA متبوعا بعلامة علم في المحطة N (جدول المواد).
- تحضير البلازميد
- أضف ناقلات pcDNA3.1+ المؤتلفة أو بلازميد المستشعر (جدول المواد) إلى 50 ميكرولتر من الإشريكية القولونية المختصة DH5α (E. coli) المخزنة في أنبوب 1.5 مل عند -80 درجة مئوية بشكل منفصل ، واحتضنها على الثلج لمدة 30 دقيقة. قم بصدمة الخلايا بالحرارة عند 42 درجة مئوية لمدة 90 ثانية ، وانقلها إلى الجليد على الفور ، وقم بتبريدها لمدة 2 دقيقة.
ملاحظة: يعبر بلازميد المستشعر الذي توفره مجموعة مقايسة cAMP المثبطة ل Gi (جدول المواد) عن جين luciferase معدل يندمج مع مجال ربط cAMP ويزيد من نشاط التلألؤ عند ربط cAMP. - هز الأنبوب عند 37 درجة مئوية لمدة 1 ساعة بعد تناول 300 ميكرولتر من وسط مرق الليسوجيني (LB). لوحة 100 ميكرولتر من الخلايا إلى لوحة أجار LB وحضانت عند 37 درجة مئوية عن طريق البقاء في الظلام لمدة 48 ساعة. قم بتلقيح المستعمرة البيضاء إلى 5 مل من وسط LB يحتوي على 100 ميكروغرام / مل أمبيسلين ، والاستزراع عند 37 درجة مئوية لمدة 16 ساعة.
- عزل الحمض النووي باستخدام مجموعة البلازميد miniprep (جدول المواد) باتباع تعليمات الشركة المصنعة ؛ كان البلازميد بتركيز أكثر من 350 نانوغرام / ميكرولتر بقيمة A260 / A280 بين 1.7 و 1.9.
- أضف ناقلات pcDNA3.1+ المؤتلفة أو بلازميد المستشعر (جدول المواد) إلى 50 ميكرولتر من الإشريكية القولونية المختصة DH5α (E. coli) المخزنة في أنبوب 1.5 مل عند -80 درجة مئوية بشكل منفصل ، واحتضنها على الثلج لمدة 30 دقيقة. قم بصدمة الخلايا بالحرارة عند 42 درجة مئوية لمدة 90 ثانية ، وانقلها إلى الجليد على الفور ، وقم بتبريدها لمدة 2 دقيقة.
- زراعة الخلايا
- قم بطبق خلايا CHO-K1 في طبق بتري 10 سم ، واستزرعها في حاضنة 37 درجة مئوية مع 5٪ CO2 ، وحصادها عندما تكون الطبقة الأحادية عند التقاء 80٪ -90٪.
- استنشق وسط نمو خلايا CHO-K1 ، وأضف 4 مل من 0.05٪ من التربسين-EDTA المسخن مسبقا عند 37 درجة مئوية على طبق بتري بلطف ، واحتضنه لمدة 15 ثانية. ثم أضف 4 مل من وسط النمو يتكون من F12 متوسط + 10٪ FBS.
- قم بإزاحة الخلايا من سطح طبق بتري عن طريق التمايل بلطف والنقر على جانب طبق بتري. املأ أنبوبا مخروطيا بتعليق الخلية. يحرك ويماصة ببطء لإزالة كتل الخلايا بلطف.
- خلايا الطرد المركزي في 250 × غرام لمدة 5 دقائق في درجة حرارة الغرفة ، وشفط supernatant ، وإنعاش مع 3 مل من PBS. كرر هذه الخطوة.
- حدد عدد الخلايا باستخدام مقياس الدم، وخلايا أجهزة الطرد المركزي عند 250 × جم لمدة 5 دقائق في درجة حرارة الغرفة.
- قم بشفط PBS المخزن المؤقت وإعادة تعليق خلايا CHO-K1 مع 3 مل من وسط النمو الذي يتكون من F12 المتوسطة و 10٪ FBS.
- أضف 2 مل من وسط النمو المكون من F12 و 10٪ FBS في كل بئر من صفيحة البئر الستة ، وبذور 150 ميكرولتر من تعليق الخلايا في كل بئر للحفاظ على الخلايا بكثافة 1.5 × 105 خلايا / مل. احتضن الصفيحة المكونة من ستة آبار في حاضنة زراعة الأنسجة 37 درجة مئوية مع 5٪ CO2 لمدة 24 ساعة تقريبا.
- النقل العابر
- تمييع 2 ميكروغرام من الحمض النووي (الخطوة 3.2.3) إلى 200 ميكرولتر من المخزن المؤقت لكاشف النقل (جدول المواد). اخلطي عن طريق الدوامة لمدة 10 ثوان والدوران لفترة وجيزة قبل الاستخدام.
ملاحظة: هنا ، يحتوي 2 ميكروغرام من الحمض النووي على 0.5 ميكروغرام من ناقل المستقبل (S1PR1 أو S1PR3 أو S1PR5) و 1.5 ميكروغرام من بلازميد المستشعر. - أضف 4 ميكرولتر من كاشف النقل (جدول المواد) ، دوامة لمدة 10 ثوان ، وتدور لفترة وجيزة قبل الاستخدام. احتضان لمدة 15 دقيقة في درجة حرارة الغرفة.
- أسقط ببطء 200 ميكرولتر من مزيج النقل في كل بئر (يحتوي على خلايا CHO-K1) لتوزيعها بالتساوي. رج الطبق المكون من ستة آبار بلطف لضمان الخلط الدقيق.
- استبدل وسط النقل بعد 4-6 ساعات بوسط نمو الخلية المكون من F12 متوسط + 10٪ FBS ، وأعد اللوحة المكونة من ستة آبار إلى الحاضنة.
- خلايا الحصاد 24-48 ساعة بعد النقل.
- هضم خلايا CHO-K1 على البئر مع 500 ميكرولتر من 0.05 ٪ التربسين-EDTA (المسخنة مسبقا عند 37 درجة مئوية) لمدة 15 ثانية وإضافة 1 مل من وسط النمو يتكون من F12 المتوسطة + 10 ٪ FBS. قم بإزاحة الخلايا من سطح البئر عن طريق الهزاز والنقر بلطف على جانب البئر.
- انقل تعليق الخلية إلى أنبوب مخروطي ، وأجهزة طرد مركزي عند 250 × g لمدة 5 دقائق في درجة حرارة الغرفة. صب قبالة supernatant وحصاد الخلايا المنقولة.
ملاحظة: قبل تحديد إشارة التألق ، تحقق من مستويات التعبير عن سطح الخلية للمستقبلات بواسطة ELISA كما هو موضح سابقا26.
- تمييع 2 ميكروغرام من الحمض النووي (الخطوة 3.2.3) إلى 200 ميكرولتر من المخزن المؤقت لكاشف النقل (جدول المواد). اخلطي عن طريق الدوامة لمدة 10 ثوان والدوران لفترة وجيزة قبل الاستخدام.
- التوازن مع ملح D-Luciferin-potassium (جدول المواد)
- الخلايا التي تم حصادها (24-48 ساعة بعد النقل) مع 3 مل من المخزن المؤقت للفحص على الفور (أي محلول الملح المتوازن من هانك (HBSS) الذي يحتوي على 10 mM HEPES pH 7.4) ، مع تخفيف إضافي بنسبة 3٪ v / v لملح D-Luciferin-potassium.
- أضف 90 ميكرولتر من تعليق الخلايا لكل بئر من صفيحة 96 بئرا باستخدام ماصة متعددة القنوات واخلطها بلطف.
- احتضان لمدة 40 دقيقة في درجة حرارة الغرفة.
- تحديد إشارة التألق
- قم بإعداد محاليل مخزون 10 mM مسبقا من Siponimod المذاب في DMSO وقم بإجراء تخفيف تسلسلي باستخدام مخزن HBSS المخزن المؤقت الذي يحتوي على 25 ميكرومتر فورسكولين قبل تحفيز الليجند.
ملاحظة: باستثناء المجموعة الضابطة بدون رباط ، فإن الباقي له نطاق تدرج تركيز 10-11-10-5 مول / لتر. - تحفيز مع 10 ميكرولتر (لكل بئر) من محلول ناهض بتركيزات مختلفة لمدة 30 دقيقة.
- عد إشارات التلألؤ على قارئ microplate باستخدام معلمات البرامج المرتبطة (جدول المواد) على النحو التالي. حدد التلألؤ لطريقة الكشف، ونقطة النهاية لنوع القراءة، وألياف التلألؤ لنوع البصريات. اضبط كسب البصريات على 255.
ملاحظة: تم تكرار كل قياس في ثلاث تجارب مستقلة على الأقل، كل منها في ثلاث مرات. - احصل على قيم إشارة التألق، واستورد البيانات إلى برنامج جدول بيانات، وقم بمعالجة البيانات باستخدام دالة استجابة الجرعة غير الخطية (ملاءمة المنحنى).
- قم بإعداد محاليل مخزون 10 mM مسبقا من Siponimod المذاب في DMSO وقم بإجراء تخفيف تسلسلي باستخدام مخزن HBSS المخزن المؤقت الذي يحتوي على 25 ميكرومتر فورسكولين قبل تحفيز الليجند.
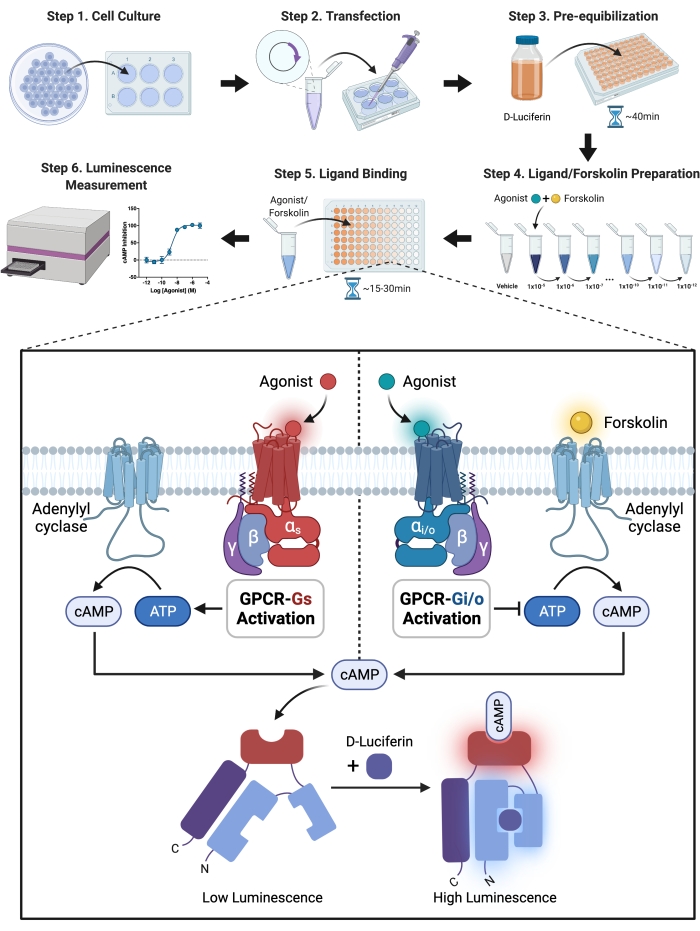
الشكل 1: رسم تخطيطي للتجربة. دليل تفصيلي تدريجي للإعداد التجريبي والتنفيذ. باختصار ، تم التعبير عن المستقبل و luciferase المعدل بشكل عابر في خلايا CHO-K1 عن طريق نقل المستقبل وبلازميد المستشعر إلى الخلايا ذات كاشف النقل. تم تعليق الخلايا في محلول HBSS مع ملح D-Luciferin-potassium ، ركيزة luciferase ، وزرعت في صفيحة 96 بئرا بعد 24 ساعة. للسماح بالنفاذية في الخلايا ، يجب أن يكون D-luciferin متوازنا مسبقا مع الخلايا. يحول الإنزيم التأكسدي luciferase luciferin إلى oxyluciferin وينبعث منه الضوء. من ناحية أخرى ، يولد luciferase المعدل الضوء عبر تفاعل كيميائي فقط عندما يرتبط ب cAMP ، وشدة الضوء لها ارتباط إيجابي بمستويات cAMP في الخلايا. تم تنظيم مستويات cAMP باستخدام GPCR الذي تم تنشيطه بواسطة ناهض. خفضت المستقبلات المقترنة ب Gi مستويات cAMP ، مما استلزم إضافة فورسكولين لتنشيط سيكلاز الأدينيليل في تجربة cAMP المثبطة ل Gi. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
النتائج
قبل تجميد عينة مركب S1PRs-Gi ، يجب فصل العينة النقية بواسطة كروماتوغرافيا استبعاد الحجم (SEC) وتحليلها باستخدام كروماتوغرافيا ترشيح الهلام. يوضح الشكل 2 مجمع S1PR3-Gi كمثال. عادة ما كان الجزء الأقصى من مركب بروتين GPCR-G المتجانس يقع عند ~ 10.5 مل من كروماتوغرافيا استبعاد الحجم (
Discussion
يصف هذا البروتوكول خط أنابيب أساسي لتحديد هياكل S1PRs بواسطة cryo-EM وقياس فعالية تنشيط S1PRs بواسطة اختبار تثبيط cAMP بوساطة Gi. بعض الخطوات حاسمة لنجاح التجربة.
لتنقية مجمع S1PRs-Gi ، يجب إيلاء المزيد من الاهتمام لجودة الفيروس وصحة خلايا sf9 . يتم تقليل التعبير عن المستقبل بشكل كبير ف...
Disclosures
المؤلفون ليس لديهم تضارب في المصالح.
Acknowledgements
تم جمع بيانات مجمع S1PRs-Gi في مركز غرب الصين Cryo-EM في جامعة سيتشوان ومركز Cryo-EM في الجامعة الجنوبية للعلوم والتكنولوجيا (SUSTech) ومعالجتها في مركز Duyu للحوسبة عالية الأداء في جامعة سيتشوان. تم دعم هذا العمل من قبل مؤسسة العلوم الطبيعية الصينية (32100965 إلى L.C. ، 32100988 إلى W.Y. ، 31972916 إلى Z.S.) وصندوق أبحاث ما بعد الدكتوراه بدوام كامل من جامعة سيتشوان (2021SCU12003 إلى L.C.)
Materials
| Name | Company | Catalog Number | Comments |
| 0.05% trypsin-EDTA | GIBCO | Cat# 25300054 | |
| 0.22 µM filter | Thermo Fisher Scientific | Cat# 42213-PS | |
| 100 kDa cut-off concentrator | Thermo Fisher Scientific | Cat# 88533 | |
| 6-well plate | Corning | Cat# 43016 | |
| 96-well plate | Corning | Cat# 3917 | |
| Aprotinin | Sigma-Aldrich | Cat# 9087-70-1 | |
| Apyrase | NEB | Cat# M0398S | |
| Baculovirus transfection reagent | Thermo Fisher Scientific | Cat# 10362100 | For the preparation of P0 baculovirus |
| Benzamidine | Sigma-Aldrich | Cat# B6506 | |
| CHO-K1 | ATCC | N/A | |
| CHS | Sigma-Aldrich | Cat# C6512 | |
| CryoSPARC | Punjani, A., et al.,2017 | https://cryosparc.com/ | |
| DH5α competent E.coli | Thermo Fisher Scientific | Cat# EC0112 | |
| D-Luciferin-Potassium Salt | Sigma- Aldrich | Cat# 50227 | |
| DMSO | Sigma- Aldrich | Cat# D2438 | |
| EDTA | Thermo Fisher Scientific | Cat# S311-500 | |
| ESF921 cell culture medium | Expression Systems | Cat# 96-001 | |
| Excel | microsoft | N/A | |
| F12 medium | Invitrogen | Cat# 11765 | |
| FBS | Cell Box | Cat# SAG-01U-02 | |
| Flag resin | Sigma- Aldrich | Cat# A4596 | |
| Forskolin | APExBIO | Cat# B1421 | |
| Gctf | Zhang, 2016 | https://www.mrc-lmb.cam.ac.uk/kzhang/Gctf/ | |
| GDN | Anatrace | Cat# GDN101 | |
| Gel filtration column | GE healthcare | Cat# 28990944 | |
| Gen5 3.11 | BIO-TEK | N/A | |
| Gentamicin | Solarbio | Cat# L1312 | |
| GloSensor cAMP assay kit | Promega | Cat# E1291 | Gi-inhibition cAMP assay kit |
| GloSensor plasmid | Promega | Cat# E2301 | Sensor plasmid |
| Grace’s medium | GIBCO | Cat# 11595030 | |
| GraphPad Prism 8 | Graphpad | N/A | |
| HBSS | Thermo Fisher Scientific | Cat# 88284 | |
| HEPES | Sigma- Aldrich | Cat# H4034 | |
| jetPRIME Reagent | Polyplus Transfection | Cat# 114-15 | transfection reagent |
| Janamycin | Solarbio | Cat# K1030 | |
| LB medium | Invitrogen | Cat# 12780052 | |
| Leupeptin | Sigma-Aldrich | Cat# L2884 | |
| LMNG | Anatrace | Cat# NG310 | |
| MotionCor2 | (Zheng et al., 2017) | https://emcore.ucsf.edu/ucsf-software | |
| NanoCab | Thermo Fisher Scientific | Cat# 1121822 | |
| PBS | Invitrogen | Cat# 14190-144 | |
| pcDNA3.1-HA-FLAG-S1PRs | GenScript | N/A | |
| pFastBac1-Gαi | GenScript | N/A | |
| pFastBac1-HA-FLAG-T4L-S1PRs-His10 | GenScript | N/A | |
| pFastBacdual-Gβ1γ2 | GenScript | N/A | |
| PureLink HiPure Plasmid Miniprep Kit | Invitrogen | Cat# K210003 | For the preparation of plasmids and P0 baculovirus |
| Q5 site-Directed Mutagenesis kit | NEB | Cat# E0554S | For the preparation of plasmids |
| Quantifoil | Quantifoil | Cat# 251448 | |
| RELION-3.1 | (Zivanov et al., 2018) | https://www2.mrc-lmb.cam.ac.uk/relion | |
| S1PRs cDNA | addgene | N/A | |
| scFv16 | Invitrogen | Cat# 703976 | |
| Sf9 | Expression Systems | N/A | |
| Siponimod | Selleck | Cat# S7179 | |
| sodium cholate | Sigma-Aldrich | Cat# C1254 | |
| Synergy H1 microplate reader | BIO-TEK | N/A | |
| Synthetic T4L DNA (sequence) | N/A | N/A | Aacatcttcgagatgctgcgcatcgacgaagg cctgcgtctcaagatttacaagaataccgaagg ttattacacgattggcatcggccacctcctgaca aagagcccatcactcaacgctgccaagtctga actggacaaagccattggtcgcaacaccaac ggtgtcattacaaaggacgaggcggagaaac tcttcaaccaagatgtagatgcggctgtccgtgg catcctgcgtaatgccaagttgaagcccgtgt atgactcccttgatgctgttcgccgtgcagcctt gatcaacatggttttccaaatgggtgagaccgg agtggctggttttacgaactccctgcgcatgctcc agcagaagcgctgggacgaggccgcagtga atttggctaaatctcgctggtacaatcagacacc taaccgtgccaagcgtgtcatcactaccttccg tactggaacttgggacgcttac |
| TCEP | Thermo Fisher Scientific | Cat# 77720 | |
| Tetracycline | Solarbio | Cat# T8180 | |
| Vitrobot Mark IV | Thermo Fisher Scientific | N/A |
References
- Verstockt, B., et al. Sphingosine 1-phosphate modulation and immune cell trafficking in inflammatory bowel disease. Nature Reviews: Gastroenterology & Hepatology. , 1-16 (2022).
- Rosen, H., Stevens, R. C., Hanson, M., Roberts, E., Oldstone, M. B. Sphingosine-1-phosphate and its receptors: structure, signaling, and influence. Annual Review of Biochemistry. 82, 637-662 (2013).
- Cartier, A., Hla, T. Sphingosine 1-phosphate: Lipid signaling in pathology and therapy. Science. 366 (6463), 5551 (2019).
- Jozefczuk, E., Guzik, T. J., Siedlinski, M. Significance of sphingosine-1-phosphate in cardiovascular physiology and pathology. Pharmacological Research. 156, 104793 (2020).
- Kihara, Y., Maceyka, M., Spiegel, S., Chun, J. Lysophospholipid receptor nomenclature review: IUPHAR Review 8. British Journal of Pharmacology. 171 (15), 3575-3594 (2014).
- Bryan, A. M., Del Poeta, M. Sphingosine-1-phosphate receptors and innate immunity. Cellular Microbiology. 20 (5), 12836 (2018).
- Pelletier, D., Hafler, D. A. Fingolimod for multiple sclerosis. New England Journal of Medicine. 366 (4), 339-347 (2012).
- Obinata, H., Hla, T. Sphingosine 1-phosphate and inflammation. International Immunology. 31 (9), 617-625 (2019).
- Pyne, N. J., Pyne, S. Sphingosine 1-phosphate and cancer. Nature Reviews: Cancer. 10 (7), 489-503 (2010).
- Abu-Farha, M., et al. The role of lipid metabolism in COVID-19 virus infection and as a drug target. International Journal of Molecular Sciences. 21 (10), 3544 (2020).
- Chun, J., Kihara, Y., Jonnalagadda, D., Blaho, V. A. Fingolimod: lessons learned and new opportunities for treating Multiple Sclerosis and other disorders. Annual Review of Pharmacology and Toxicology. 59, 149-170 (2019).
- Murakami, A., et al. Sphingosine 1-phosphate (S1P) regulates vascular contraction via S1P3 receptor: investigation based on a new S1P3 receptor antagonist. Molecular Pharmacology. 77 (4), 704-713 (2010).
- Cao, L., et al. Siponimod for multiple sclerosis. Cochrane Database of Systematic Reviews. 11, (2021).
- Scott, L. J. Siponimod: a review in secondary progressive Multiple Sclerosis. CNS Drugs. 34 (11), 1191-1200 (2020).
- Lamb, Y. N. Ozanimod: first approval. Drugs. 80 (8), 841-848 (2020).
- Scott, F. L., et al. Ozanimod (RPC1063) is a potent sphingosine-1-phosphate receptor-1 (S1P1) and receptor-5 (S1P5) agonist with autoimmune disease-modifying activity. British Journal of Pharmacology. 173 (11), 1778-1792 (2016).
- McGowan, E. M., Haddadi, N., Nassif, N. T., Lin, Y. Targeting the SphK-S1P-SIPR pathway as a potential therapeutic approach for COVID-19. International Journal of Molecular Sciences. 21 (19), 7189 (2020).
- O'Sullivan, C., Dev, K. K. The structure and function of the S1P1 receptor. Trends in Pharmacological Sciences. 34 (7), 401-412 (2013).
- Liao, M., Cao, E., Julius, D., Cheng, Y. Structure of the TRPV1 ion channel determined by electron cryo-microscopy. Nature. 504 (7478), 107-112 (2013).
- Bai, X. C., McMullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- Murata, K., Wolf, M. Cryo-electron microscopy for structural analysis of dynamic biological macromolecules. Biochimica et Biophysica Acta General Subjects. 1862 (2), 324-334 (2018).
- Zhang, M., et al. Cryo-EM structure of an activated GPCR-G protein complex in lipid nanodiscs. Nature Structural & Molecular Biology. 28 (3), 258-267 (2021).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews: Drug Discovery. 17 (7), 471-492 (2018).
- Ishchenko, A., Gati, C., Cherezov, V. Structural biology of G protein-coupled receptors: new opportunities from XFELs and cryoEM. Current Opinion in Structural Biology. 51, 44-52 (2018).
- Yang, D., et al. G protein-coupled receptors: structure- and function-based drug discovery. Signal Transduction and Targeted Therapy. 6 (1), 7 (2021).
- Yuan, Y., et al. Structures of signaling complexes of lipid receptors S1PR1 and S1PR5 reveal mechanisms of activation and drug recognition. Cell Research. 31 (12), 1263-1274 (2021).
- Zhao, C., et al. Structural insights into sphingosine-1-phosphate recognition and ligand selectivity of S1PR3-Gi signaling complexes. Cell Research. 32 (2), 218-221 (2022).
- Xu, Z., et al. Structural basis of sphingosine-1-phosphate receptor 1 activation and biased agonism. Nature Chemical Biology. 18, 281-288 (2022).
- Liu, Y. F., Ghahremani, M. H., Rasenick, M. M., Jakobs, K. H., Albert, P. R. Stimulation of cAMP synthesis by Gi-coupled receptors upon ablation of distinct Galphai protein expression. Gi subtype specificity of the 5-HT1A receptor. Journal of Biological Chemistry. 274 (23), 16444-16450 (1999).
- Buccioni, M., et al. Innovative functional cAMP assay for studying G protein-coupled receptors: application to the pharmacological characterization of GPR17. Purinergic Signalling. 7 (4), 463-468 (2011).
- Wang, F. I., Ding, G., Ng, G. S., Dixon, S. J., Chidiac, P. Luciferase-based GloSensor cAMP assay: Temperature optimization and application to cell-based kinetic studies. Methods. , (2021).
- Audet, M., et al. Small-scale approach for precrystallization screening in GPCR X-ray crystallography. Nature Protocols. 15 (1), 144-160 (2020).
- Sgro, G. G., Costa, T. R. D. Cryo-EM grid preparation of membrane protein samples for single particle analysis. Frontiers in Molecular Biosciences. 5, 74 (2018).
- White, J. B. R., et al. Single particle cryo-electron microscopy: from sample to structure. Journal of Visualized Experiments. (171), e62415 (2021).
- Thompson, R. F., Iadanza, M. G., Hesketh, E. L., Rawson, S., Ranson, N. A. Collection, pre-processing and on-the-fly analysis of data for high-resolution, single-particle cryo-electron microscopy. Nature Protocols. 14 (1), 100-118 (2019).
- Fernandez-Leiro, R., Scheres, S. H. W. A pipeline approach to single-particle processing in RELION. Acta Crystallographica Section D. 73 (6), 496-502 (2017).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14 (3), 290-296 (2017).
- Brilot, A. F., et al. Beam-induced motion of vitrified specimen on holey carbon film. Journal of Structural Biology. 177 (3), 630-637 (2012).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14 (4), 331-332 (2017).
- Zhang, K. Gctf: Real-time CTF determination and correction. Journal of Structural Biology. 193 (1), 1-12 (2016).
- Scheres, S. H. Semi-automated selection of cryo-EM particles in RELION-1.3. Journal of Structural Biology. 189 (2), 114-122 (2015).
- Liu, S., et al. Differential activation mechanisms of lipid GPCRs by lysophosphatidic acid and sphingosine 1-phosphate. Nature Communications. 13 (1), 731 (2022).
- Duan, J., et al. Cryo-EM structure of an activated VIP1 receptor-G protein complex revealed by a NanoBiT tethering strategy. Nature Communications. 11 (1), 4121 (2020).
- DiIorio, M. C., Kulczyk, A. W. A robust single-particle cryo-electron microscopy (cryo-EM) processing workflow with cryoSPARC, RELION, and Scipion. Journal of Visualized Experiments. (179), e63387 (2022).
- Pradelles, P., Grassi, J., Chabardes, D., Guiso, N. Enzyme immunoassays of adenosine cyclic 3',5'-monophosphate and guanosine cyclic 3',5'-monophosphate using acetylcholinesterase. Analytical Chemistry. 61 (5), 447-453 (1989).
- Jiang, L. I., et al. Use of a cAMP BRET sensor to characterize a novel regulation of cAMP by the sphingosine 1-phosphate/G13 pathway. Journal of Biological Chemistry. 282 (14), 10576-10584 (2007).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved