Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Конвейер для исследования структур и сигнальных путей рецепторов сфингозин-1-фосфата
В этой статье
Резюме
S1P оказывает свое разнообразное физиологическое воздействие через подсемейство рецепторов S1P (S1PR). Здесь описан конвейер для объяснения структур и функций S1PR.
Аннотация
Лизофосфолипиды (LPL) являются биологически активными липидами, которые включают сфингозин-1-фосфат (S1P), лизофосфатидную кислоту и т. Д. S1P, метаболический продукт сфинголипидов в клеточной мембране, является одним из наиболее характерных LPL, который регулирует различные клеточные физиологические реакции через сигнальные пути, опосредованные рецепторами сфингозин-1-фосфата (S1PR). Это означает, что сигнальная система S1P-S1PR является замечательной потенциальной терапевтической мишенью для расстройств, включая рассеянный склероз (РС), аутоиммунные расстройства, рак, воспаление и даже COVID-19. S1PR, небольшое подмножество семейства рецепторов, связанных с G-белком класса A (GPCR), состоит из пяти подтипов: S1PR1, S1PR2, S1PR3, S1PR4 и S1PR5. Однако отсутствие подробной структурной информации препятствует открытию лекарств, нацеленных на S1PR. Здесь мы применили метод криоэлектронной микроскопии для решения структуры комплекса S1P-S1PR и выяснили механизм активации, селективного распознавания лекарств и связи G-белка с помощью клеточных функциональных анализов. Другие лизофосфолипидные рецепторы (LPLR) и GPCR также могут быть изучены с использованием этой стратегии.
Введение
Сфингозин-1-фосфат (S1P), метаболический продукт сфинголипидов в клеточной мембране, представляет собой вездесущую лизофосфатидную сигнальную молекулу, которая включает в себя различные биологические активности, включая торговлю лимфоцитами, развитие сосудов, эндотелиальную целостность и частоту сердечных сокращений 1,2,3. S1P оказывает свое разнообразное физиологическое воздействие через пять подтипов рецепторов S1P (S1PRs 1-5); S1PR встречаются в различных тканях и демонстрируют уникальные предпочтения для последующих G-белков 4,5. S1PR1 в первую очередь связан с белком Gi, который впоследствии ингибирует выработку цАМФ; S1PR2 и S1PR3 соединены с Gi, Gq и G12/13, а S1PR4 и S1PR5 передают сигнал через Gi и G12/136.
Передача сигналов S1P-S1PR является критически важной терапевтической мишенью для нескольких заболеваний, включая аутоиммунные расстройства7, воспаление8, рак9 и даже COVID-1910. В 2010 году финголимод (FTY720) был лицензирован как первый в своем классе препарат, нацеленный на S1PR для лечения рецидивирующего рассеянного склероза (РС)11. Тем не менее, он способен связываться со всеми S1PR, кроме S1PR2, в то время как неспецифическое связывание с S1PR3 приводит к отеку коры головного мозга, сужению сосудов и бронхов и утечке эпителия легких12. В качестве альтернативной стратегии повышения терапевтической селективности были получены подтип-специфические лиганды для рецептора. Сипонимод (BAF312) был одобрен в 2019 году для лечения рецидивирующего рассеянного склероза13; он эффективно нацелен на S1PR1 и S1PR5, тогда как он не имеет сродства к S1PR3, проявляя меньше побочных эффектов в клинической практике14. В 2020 году Управление по санитарному надзору за качеством пищевых продуктов и медикаментов США разрешило озанимод для терапииРС 15. Сообщалось, что озанимод обладает в 25 раз большей селективностью для S1PR1, чем для S1PR516. Примечательно, что в контексте нынешней пандемии COVID-19 было обнаружено, что агонистические препараты, нацеленные на S1PR, могут быть использованы для лечения COVID-19 с использованием методов иммуномодулирующей терапии17. По сравнению с финголимодом, озанимод показал превосходство в снижении симптомов у пациентов с COVID-19 и в настоящее время проходит клинические испытания10. Понимание структурной основы и функции S1PR закладывает значительную основу для разработки препарата, который избирательно нацелен на S1PRs18.
Многие методы используются для исследования структурной информации биомакромолекул, включая рентгеновскую кристаллографию, ядерный магнитный резонанс (ЯМР) и электронную микроскопию (ЭМ). По состоянию на март 2022 года в банке данных белка (PDB) хранится более 180 000 структур, и большинство из них были разрешены с помощью рентгеновской кристаллографии. Тем не менее, с первой структурой TPRV1 с почти атомным разрешением (разрешение 3,4 Å), о которой сообщили Ифань Чэн и Дэвид Джулиус в 2013году 19, криоэлектронная микроскопия (крио-ЭМ) стала основным методом для белковых структур, и общее количество структур EM PDB составило более 10 000. Важнейшими областями прорыва являются разработка новых камер для визуализации, известных как камеры прямого обнаружения электронов, и новые алгоритмы обработки изображений. Крио-ЭМ произвел революцию в структурной биологии и структурно-ориентированном открытии лекарств за последнее десятилетие20. Поскольку понимание того, как макромолекулярные комплексы выполняют свои сложные роли в живой клетке, является центральной темой в биологических науках, крио-ЭМ имеет потенциал для выявления конформаций динамических молекулярных комплексов, особенно для трансмембранных белков21. Рецепторы, связанные с G-белком (GPCR), являются крупнейшим надсемейством трансмембранных белков и мишенью для более чем 30% продаваемых в настоящее время фармацевтических препаратов22. Разработка крио-ЭМ способствовала всплеску структур с высоким разрешением белковых комплексов GPCR-G, что позволяет определять структуры для «трудноизлечимых» мишеней, которые все еще недоступны для рентгеновского кристаллографического анализа в конструкции лекарственного средства23. Следовательно, крио-ЭМ-приложение дает возможность определить трехмерную структуру GPCR в почти нативных условиях при близком к атомному разрешению24. Достижения в области крио-ЭМ позволяют визуализировать механистические основы стимуляции или ингибирования GPCR, а также дополнительную пользу в раскрытии новых сайтов связывания для создания GPCR-таргетных лекарств25.
Опираясь на огромные успехи крио-ЭМ технологии, мы недавно идентифицировали структуры агонизированных сигнальных комплексов S1PR1-, S1PR3- и S1PR5-Gi26,27. У людей S1PR обнаруживаются в различных тканях и демонстрируют уникальные предпочтения в отношении последующих G-белков 4,5. S1PR1 в основном связан с белком Gi, который впоследствии ингибирует выработку 3',5'-циклического аденозинмонофосфата (цАМФ). S1PR3 и S1PR5 также способны сцепляться с Gi 6,28. Поскольку активация Gi-связанных рецепторов снижает выработку цАМФ29, был введен анализ Ги-ингибирования цАМФ для измерения эффектов ингибирования цАМФ для захвата функциональных изменений 26,27. Используя мутантную версию люциферазы Photinus pyralis, в которую был вставлен цАМФ-связывающий фрагмент белка, этот анализ цАМФ предлагает простой и надежный метод мониторинга активности GPCR через изменения внутриклеточной концентрации цАМФ30. Он представляет собой чувствительный и нерадиоактивный функциональный анализ и может применяться для мониторинга нисходящей сигнализации в режиме реального времени широкого спектра GPCR для целей обнаружения лекарств31.
Здесь приводится краткое изложение критических методов разрешения режимов активации и распознавания лекарств S1PR, в первую очередь включая крио-ЭМ-манипуляции и анализ Ги-ингибирования цАМФ. Целью этой статьи является предоставление всестороннего экспериментального руководства для дальнейших исследований структур и функций GPCR.
протокол
1. Очистка белкового комплекса S1PRs-G
- Чтобы очистить человеческий белковый комплекс S1PRs-G, клонируют cDNAs S1PR1, лишенные C-концевых остатков 338-382, S1PR3 дикого типа S1PR3, S1PR5, усеченные с 345-398 на C-конце, и Дикий тип Gi1 в вектор pFastBac1 и cDNAs дикого типа Gβ1 и Gγ2 в вектор pFastBacdual (Таблица материалов).
ПРИМЕЧАНИЕ: Все конструкции для S1RR также содержат последовательность сигналов гемагглютинина (HA), за которой следует эпитопная метка Flag на N-конце и 10-кратная его метка на C-конце. Кроме того, синтетическая последовательность ДНК (Таблица материалов) для трансляции лизоцима Т4 (T4L) была вставлена в N-конец S1PR, чтобы облегчить экспрессию и очистку рецепторов. - Препарат бакуловируса, кодирующего S1PRs, Gi1 и Gβ1γ2
- Добавьте рекомбинантные векторы к 50 мкл dh5α-компетентной кишечной палочки (E. coli), хранящейся в пробирке объемом 1,5 мл при -80 °C, и инкубируйте на льду в течение 30 мин.
- Тепловой удар по ячейкам при 42 °C в течение 90 с, немедленно перенесите их на лед и охладите в течение 2 мин.
- Встряхните пробирку при 37 °C в течение 3-5 ч после добавления 300 мкл среды лизогенного бульона (LB). Пластину 100 мкл клеток к пластине агара LB и инкубируют при 37 °C, выдерживая в темноте в течение 48 ч.
- Инокулируют белую колонию в 5 мл LB-среды, содержащей 50 мкг/мл канамицина, 10 мкг/мл тетрациклина и 7 мкг/мл гентамицина, и культивируют при 37 °C в течение 16 ч.
- Изолируйте рекомбинантный бакмид с помощью плазмидного минипрепа (Таблица материалов) в соответствии с инструкциями производителя для получения бакуловируса P0.
ПРИМЕЧАНИЕ: Перед применением очищенный бакмид анализировали методом ПЦР с помощью прямых и обратных праймеров pUC/M13. Для ПЦР число циклов = 30 циклов, температура плавления = 58 °C, а время продления = 1 мин на 1 Кб. - Готовят панкуловирус P0, как описано в более раннем протоколе32.
- Культивируйте клетки sf9 (среда ESF921) в шестилуночных пластинах и убедитесь, что клетки находятся в фазе log (1,0-1,5 x 106 клеток/мл).
- Развести 8 мкл бакуловирусного трансфекционного реагента в 100 мкл среды Грейс и инкубировать в течение 30 мин при комнатной температуре. Разбавьте 10 мкг рекомбинантного бакмида в 100 мкл среды Грейс и аккуратно перемешайте. Разбавленный бакмид соединить с разбавленным реагентом для трансфекции бакуловируса, аккуратно перемешать и инкубировать в течение 30 мин при комнатной температуре.
- Добавьте смесь (бакмид и реагент) на клетки (этап 1.2.6.1) и нанесите на них пластины при температуре 27 °C в течение 3 ч.
- Удалите среду Grace и замените ее 2 мл клеточной культуральной среды ESF921. Обложите пластины с шестью лунками при 27 °C и соберите среду для культивирования клеток ESF921 через 5 дней после трансфекции.
- Центрифуга при 500 х г при 4 °C в течение 10 мин для удаления клеток и мусора. Переведите супернатант в пробирки по 2 мл. Это запас бакуловируса P0.
- Изолируйте запасы вирусов P1
- Переложите 30 мл клеток sf9 в коническую бутылку, культивируйте при 27 °C с встряхиванием при 270 об/мин и убедитесь, что клетки достигают логарифмической фазы (1,0-1,5 x 106 клеток/мл).
- Добавьте в бутылку 2 мл вирусного бульона P0 и встряхните при 270 об/мин в течение 4 дней при 27 °C.
- Переложите ячейки в трубку объемом 50 мл, центрифугу при 1 800 х г в течение 10 мин при 4 °C для удаления клеток и мусора и перенесите супернатант в трубки объемом 50 мл. Это запас бакуловируса P1.
- Усиление бакуловирусного поголовья
- Повторите шаг 1.2.7, используя 50 мл ячеек sf9 в фазе журнала (1,0-1,5 x 106 ячеек/мл) и 1 мл запаса P1.
- Храните полученный запас бакуловируса Р2 при 4 °C, защищая его от света.
ПРИМЕЧАНИЕ: Не усиливайте бакуловирус бесконечно, так как вредные мутанты производились с каждым проходом.
- Экспрессия белкового комплекса S1PRs-G
- Культивируйте клетки насекомых sf9 до плотности 2,5 х 106 клеток/мл, совместно заражайте с бакуловирусом P2, кодирующим S1PRs, Gi1 и Gβ1γ2 в объемном соотношении 1:2:1, и снова культивируйте при 27 °C в течение 48 ч.
- Соберите клетки путем центрифугирования при 700 х г при 4 °C в течение 15 мин, заморозьте их в жидком азоте и храните при -80 °C для использования.
- Очистка белка
- Разморозьте клеточную гранулу, полученную на стадии 1,3, при комнатной температуре, а затем повторно суспендировали ее в буфере лизиса (20 мМ HEPES pH 7,5, 50 мМ NaCl, 5 мМ MgCl2, 5 мМ CaCl2), дополненном 100 мкг/мл бензамидина, 100 мкг/мл лейпептина, 100 мкг/мл апротинина, 25 мЕд/мл апиразы и 10 мкМ агониста. Перемешайте клеточную суспензию при комнатной температуре в течение 2 ч, чтобы индуцировать образование белкового комплекса S1PRs-G.
ПРИМЕЧАНИЕ: Апираза представляет собой АТФ-дифосфогидролазу. Он катализирует удаление гамма-фосфата из АТФ и бета-фосфата из АДФ. - Переложите раствор в пробирки, центрифугируйте при 70 000 х г в течение 10 мин и осторожно удалите супернатант. Повторно суспендировать гранулы в солюбилизирующий буфер (20 мМ HEPES pH 7,5, 100 мМ NaCl, 5 мМMgCl2, 5 мМ CaCl2, 0,5% (мас./об.) ЛМНГ, 0,1% (мас./об.) CHS, 1% (мас./об.) холата натрия, 10% (v/v) глицерина).
- Переложите суспензию на стекло Dounce и полностью гомогенизируйте гранулу. Добавьте к суспензии 10 мкМ агонист, 4 мг scFv16, 100 мкг/мл бензамидина, 100 мкг/мл лейпептина, 100 мкг/мл апротинина и 25 мЕд/мл апиразы и перемешайте при 4 °C в течение 2 ч.
ПРИМЕЧАНИЕ: Этапы гомогенизации гранул имеют решающее значение для производства белкового комплекса GPCR-G. - Переложите раствор в пробирки и центрифугу при 100 000 х г в течение 30 мин при 4 °C.
- Предварительно уравновешивайте флаговую смолу с помощью промывочного буфера (20 мМ HEPES pH 7,5, 100 мМ NaCl, 5 мМ MgCl2, 5 мМ CaCl2, агонист 10 мкМ, 0,0375% (мас./об.) LMNG, 0,0125% (мас./об.) GDN, 0,01% (мас./об.) CHS).
- Переложите надосадочное вещество в трубки и инкубируйте с флаг-смолой при 4 °C в течение 2 ч.
- Загрузите вышеуказанную смесь на стеклянную колонну и промыть колонну 50 мл промывочного буфера, дополненного 100 мкг/мл бензамидина, 100 мкг/мл лейпептина и 100 мкг/мл апротинина.
- Элюируют колонку с 10 мл буфера элюирования, содержащего 20 мМ HEPES pH 7,5, 100 мМ NaCl, 5 мМ ЭДТА, 200 мкг/мл флаг-пептида, 10 мкМ агониста, 0,0375% (мас./об.) LMNG, 0,0125% (мас./об.) GDN, 0,01% (мас./об.) CHS, 100 мкг/мл бензамидина, 100 мкг/мл лейпептина и 100 мкг/мл апротинина.
- Соберите белковый комплекс S1PRs-G и сконцентрируйте до 1 мл с помощью отсечной концентраторной установки 100 кДа при 1 300 х г при 4 °C. Фильтровать через фильтр 0,22 мкМ и центрифугу при 13 000 х г в течение 10 мин при 4 °C для удаления агрегатов.
- Загрузите белковый комплекс S1PRs-G на фильтрующую колонну геля с исключением размера (SEC), предварительно уравновешенную буфером SEC, содержащим 20 мМ HEPES pH 7,5, 100 мМ NaCl, агонист 10 мкМ, 100 мкм TCEP, 0,00375% (мас./об.) LMNG, 0,00125% (мас./об.) GDN и 0,001% (мас./об.) CHS при скорости потока 0,5 мл/мин при 4 °C.
- Соберите пиковые фракции и концентрат с помощью отсечной концентраторной установки 100 кДа при 1 300 х г при 4 °C для крио-ЭМ.
- Разморозьте клеточную гранулу, полученную на стадии 1,3, при комнатной температуре, а затем повторно суспендировали ее в буфере лизиса (20 мМ HEPES pH 7,5, 50 мМ NaCl, 5 мМ MgCl2, 5 мМ CaCl2), дополненном 100 мкг/мл бензамидина, 100 мкг/мл лейпептина, 100 мкг/мл апротинина, 25 мЕд/мл апиразы и 10 мкМ агониста. Перемешайте клеточную суспензию при комнатной температуре в течение 2 ч, чтобы индуцировать образование белкового комплекса S1PRs-G.
2. Электронная микроскопия для разрешения структуры S1PR
- Сбор данных
- Для подготовки крио-ЭМ-сеток держите 300-сетчатые сетки Au R1.2/1.3 в течение 10 с и тлеющий разряд в течение 60 с при 15 мА с помощью системы очистки тлеющего разряда.
- Выполняют витрификацию образцов, как описано ранее 33,34. В консоли погружной заморозки установите температуру на 4 °C и относительную влажность на 100% для рабочей среды камеры. Используйте силу пятна в течение 0, время ожидания 0 с, время блотирования 2 или 3 с и время слива 0 с. Обычно требуется только 3 мкл образца в концентрациях 5-10 мг/мл для одиночной витрификации.
- Обрезайте и загружайте сетки в сборку автоматической сетки, загружайте сборку автоматической сетки в Nanocab и загружайте Nanocab в микроскоп с помощью автозагрузчика, как описано ранее35. Качество образца экрана с программным обеспечением EPU234. Обычно данные, собранные в районах подходящей толщины льда, были лучше.
- Соберите крио-ЭМ данные, как подробно описано ранее35. Для рецептора S1P установите смещение дефокуса между -1,0 мкм и -1,8 мкм с экспозиционной электронной дозой 50-65 e-/Å2. Для комплексов S1PR1-Gi автоматически собирать стек фильмов с помощью программного обеспечения EPU2 в режиме подсчета с помощью детектора K2 при общем времени экспозиции 2 с, скорости записи пяти необработанных кадров в секунду и общей дозе 56 e-/Å2 для получения 35 кадров на стек.
ПРИМЕЧАНИЕ: Обычно для восстановления структуры рецептора требуется более 5000 фильмов.
- Обработка данных с использованием комбинации RELION36 и криоСПАРК37 для получения идеальной крио-ЭМ карты плотности. Используйте RELION-3.1_gpu_ompi4 для первоначальной обработки данных, которая включает в себя операции, аналогичные описанным ранее34.
- В системном терминале Linux введите родительский каталог каталога хранилища данных.
- Введите команду relion в терминале, чтобы открыть графический интерфейс пользователя RELION.
ПРИМЕЧАНИЕ: Если это первый раз, когда RELION GUI был открыт в этом каталоге, появится окно приглашения; нажмите кнопку Да. - Нажмите «Импорт» на панели функций в правой части графического интерфейса RELION, чтобы импортировать необработанные данные в RELION.
- В параметре «Фильмы/микрофоны» выберите «Да» для параметра «Импортировать необработанные фильмы/микроснимки?», введите путь к данным в поле «Необработанные входные файлы» (рекомендуется использовать подстановочные знаки) и выберите «Да» для параметра «Являются ли эти многокадровые фильмы?». Введите размер пикселя фильмов в поле «Размер пикселя» (Ангстрем), рабочее напряжение микроскопа (в кВ) в поле «Напряжение» (кВ) и сферическую аберрацию микроскопа в поле «Сферическая аберрация» (мм). Это параметры, которые были записаны в момент сбора данных.
- В параметре Выполняется измените имя очереди в соответствии с сервером, на котором запущена программа (другие функции также должны изменить этот параметр). Оставьте остальные параметры в значениях по умолчанию, установленных RELION. Наконец, когда все параметры определены правильно, нажмите кнопку ВЫПОЛНИТЬ! , чтобы запустить программу.
- Используйте функцию коррекции движения для выравнивания всех кадров38.
- В опции ввода-вывода нажмите «Обзор» и выберите выход функции импорта с именем movies.star в качестве входных данных файла Input movies STAR. Введите дозу на кадр в поле Доза на кадр (e/A2), которое равно общей дозе, деленной на количество кадров. Выберите Нет для параметра Сохранить сумму спектров мощности?.
- В параметре «Движение» введите 250 для B-фактора, 5,5 для «Количество патчей X,Y» и 2 для групповых кадров (обеспечьте дозу группового >3). Если данные не были связаны с усилением в течение периода сбора, необходимо изображение с привязкой к коэффициенту усиления, которое можно получить, получив пустую область сетки. Выберите Нет для использования собственная реализация RELION? и введите каталог, содержащий исполняемый файл MOTIONCOR239, в исполняемое поле MOTIONCOR2 .
- В параметре Выполняется выберите соответствующий MPI и номер потока в соответствии с вычислительной мощностью сервера; здесь использовались MPI = 8 и threads = 3.
- Используйте функцию оценки CTF для модуляции крио-ЭМ изображения остеклованного образца40. В опции ввода-вывода нажмите «Обзор» и выберите выход Motion cor с именем corrected micrographs.star в качестве входных данных файла Input movies STAR. В параметре Gctf выберите Да для UseGctf?.
- Используйте функцию выбора подмножеств для удаления микроснимков со значением rlnCtfMaxResolutin >4.
- В опции ввода-вывода нажмите «Обзор», расположенную справа от ИЛИ выберите из micrographs.star , и выберите выход CtfFind с именем micrographs_ctf.star в качестве входных данных. В параметре Подмножество выберите Да для параметра Выбрать на основе значений метаданных? и введите 4 в качестве максимального значения метаданных , чтобы удалить неверные данные.
- Ручной отбор: выберите некоторые изображения вручную для предварительного выбора и классификации.
- В опции ввода-вывода нажмите «Обзор» и выберите micrographs_selected.star из предыдущего каталога Select (шаг 2.2.6) в качестве входных данных.
- Нажмите на RUN! (появится окно). Нажмите «Файл» в левом верхнем углу нового окна и нажмите « Инвертировать выделение », чтобы отменить выбор всех изображений. Проверьте крайний левый флажок выбора в соответствующей строке каждой записи и нажмите на выбор , чтобы проверить изображения и выбрать ~ 500 хороших изображений. Нажмите «Файл > сохранить», чтобы сохранить выбранные изображения и закрыть окно.
- Автоподбор: пакеты программного обеспечения для автоматического сбора частиц являются полезными и мощными41.
- В опции ввода-вывода нажмите «Обзор» справа от «Входных микроснимков» для автовыбора и выберите micrographs_selected.star из предыдущего каталога ManualPick (шаг 2.2.7) в качестве входных данных. В начале используется алгоритм Лапласиана Гаусса, поэтому выберите Да для OR: используйте Лапласиан-оф-Гаусс.
- В параметре Laplacian установите минимальный диаметр для фильтра LoG (A) равным 80 и максимальный диаметр для фильтра LoG (A) равным 130. В параметре Автопикирование установите для параметра Минимальное расстояние между частицами (A) значение 65 и выберите Да для параметра Использовать ускорение графического процессора, если доступ к графическому процессору возможен.
- Извлеките частицы для следующих шагов.
- В опции ввода-вывода нажмите «Обзор» справа от файла MICROGRAPH STAR и выберите micrographs_selected.star из шага 2.2.6. Нажмите «Обзор» справа от входных координат и выберите cords_suffix_autopick.star из шага 2.2.8.
- В параметре «Извлечь » выберите «Да » для параметра «Изменить масштабирование частиц » и установите для параметра «Повторно масштабируемый размер (пиксели)» значение 128 , чтобы ускорить последующие шаги.
- 2D классификация для предварительной классификации частиц
- В опции ввода-вывода нажмите «Обзор» справа от файла STAR входных изображений и выберите particles.star из шага 2.2.9. В параметре Оптимизация установите для параметра Количество классов значение 100, а для параметра Диаметр маски (A) — значение 140.
- В параметре Вычисления установите для параметра Количество частиц пула значение 10, введите каталог, который находится на быстром локальном диске (например, SSD-диске), в поле Копировать частицу в скретч-каталог и выберите Да для использования ускорения графического процессора? для более высокой скорости обработки.
- Выбор подмножества для выбора хороших 2D-результатов в качестве шаблонов для выбора частиц
- В опции ввода-вывода нажмите кнопку Обзор справа от Выберите классы из model.star и выберите выходные данные Шага 2.2.10 с именем run_it025_model.star в качестве входных данных. Нажмите на КНОПКУ ВЫПОЛНИТЬ!. Во всплывающем окне установите флажки Сортировать изображения на и Обратная сортировка? и нажмите На Дисплей!.
- Выберите хорошие репрезентативные 2D-результаты в качестве ссылки на функцию автоподбора .
ПРИМЕЧАНИЕ: Выбранные результаты выделены красным цветом. Хорошие и плохие результаты 2D-классификации показаны позже. - Щелкните правой кнопкой мыши и выберите Сохранить выбранные классы.
- Используйте шаблон для второго раунда автоподбора. В опции ввода-вывода нажмите «Обзор» справа от «Входных микроснимков» для автовыбора и выберите micrographs_selected.star из шага 2.2.6. Нажмите «Обзор» справа от 2D-ссылок, выберите class_averages.star из шага 2.2.11 и выберите «Нет для ИЛИ: используйте Laplacian-of-Gaussian».
- Снова выполните извлечение частиц, используя coord_suffix_autopick.star из Шага 2.2.12 и micrographs_selected.star из Шага 2.2.6.
- Снова выполните 2D-классификацию, используя particles.star из шага 2.2.13.
- Снова выполните выделение подмножества с помощью run_it025_optimiser.star из шага 2.2.14.
ПРИМЕЧАНИЕ: Все 2D-изображения с четкими контурами и правильными формами должны быть выбраны. - Выполните извлечение частиц следующим образом. В опции ввода/вывода нажмите кнопку Обзор справа от файла MICROGRAPH STAR, выберите micrographs_selected.star из шага 2.2.6 и выберите Да для ИЛИ повторного извлечения рафинированных частиц?. Нажмите на Обзор справа от файла Refined particles STAR и выберите particles.star из Шага 2.2.15.
- 3D-начальная модель и генерация эталонной карты: В опции ввода-вывода нажмите «Обзор» справа от файла STAR «Входные изображения» и выберите particles.star из шага 2.2.16. Установите для параметра Количество классов значение 1, а для параметра Оптимизация — значение Диаметр маски (A) — 140.
- 3D-классификация и создание предварительной 3D-карты: В опции ввода-вывода нажмите «Обзор» справа от файла STAR «Входные изображения» и выберите particles.star из шага 2.2.16. Нажмите кнопку Обзор справа от Справочной карты и выберите initial_model.mrc из Шага 2.2.17. Установите для параметра «Количество классов» значение 4–6, а для параметра «Оптимизация» — значение «Диаметр маски (A)».
- Генерация маски: выберите хорошие 3D-карты из шага 2.2.17 в качестве входных данных в опции ввода-вывода. Установите порог начальной бинаризации равным 0,05 (настройте в зависимости от выходных данных), увеличьте двоичное сопоставление этого количества пикселей до 3 и добавьте мягкий край этого количества пикселей в 8 в параметре «Маска».
- Используйте cryoSPARC для следующей обработки.
- Создайте новую рабочую область и щелкните Конструктор заданий для первого задания.
- Чтобы импортировать стек частиц, введите путь частицы (шаг 2.2.16) в поле мета-путь частицы и путь фильма (шаг 2.2.16) в поле путь данных частицы .
ПРИМЕЧАНИЕ: Параметры Ускоряющее напряжение (кВ), Сферическая аберрация (мм) и Размер пикселя (Ангстрем) такие же, как и раньше. Значение амплитудного контраста (дроби) равно 0,1. - Импортируйте 3D-тома, введя путь к лучшему 3D-тому на шаге 2.2.18 в пути к данным тома и выбрав Map в поле Тип импортируемого тома.
- Импортируйте маску, введя путь к маске (шаг 2.2.19) в путь к данным тома и выбрав Маска в поле Тип импортируемого тома.
- Неравномерное уточнение: Возьмите результаты этапов 2.2.22, 2.2.23 и 2.2.24 в качестве входных данных.
ПРИМЕЧАНИЕ: Эта функция очень полезна для мембранных белков.- Перетащите выход Шага 2.2.22 (imported_particles) в качестве входа частиц (частиц) Неоднородного Уточнения, выход Шага 2.2.23 (imported_volume_1) как вход объема (объема) Неравномерного Уточнения и выход Шага 2.2.24 (imported_mask_1) в качестве входа маски (маски) Неравномерного Уточнения.
ПРИМЕЧАНИЕ: Иногда лучшие результаты могут быть достигнуты без маски. - Щелкните Очередь , чтобы начать обработку.
- Перетащите выход Шага 2.2.22 (imported_particles) в качестве входа частиц (частиц) Неоднородного Уточнения, выход Шага 2.2.23 (imported_volume_1) как вход объема (объема) Неравномерного Уточнения и выход Шага 2.2.24 (imported_mask_1) в качестве входа маски (маски) Неравномерного Уточнения.
- Выполните шаги 2.2.18-2.2.25 для достижения лучших результатов. Благодаря вышеуказанной серии обработки можно получить карту S1PR-Gi 3D с хорошим разрешением.
3. S1PRs-Gi опосредованный анализ ингибирования цАМФ
ПРИМЕЧАНИЕ: Эксперимент по ингибированию цАМФ, опосредованный S1PRs-Gi, был разделен на несколько частей, и ниже приведены подробные экспериментальные процедуры. Экспериментальный принцип и общий экспериментальный процесс показаны в виде блок-схемы на рисунке 1.
- Построение плазмид
- Субклонирование cDNAs диких типов S1PR1, S1PR3 и S1PR5 в вектор pcDNA3.1+ с последовательностью сигналов HA, за которой следует тег Flag на N-конце (Таблица материалов).
ПРИМЕЧАНИЕ: Мутации для всех рецепторов были сгенерированы с помощью набора мутагенеза (Таблица материалов).
- Субклонирование cDNAs диких типов S1PR1, S1PR3 и S1PR5 в вектор pcDNA3.1+ с последовательностью сигналов HA, за которой следует тег Flag на N-конце (Таблица материалов).
- Получение плазмиды
- Добавьте рекомбинантные векторы pcDNA3.1+ или плазмиду датчика (Таблица материалов) к 50 мкл dh5α-компетентной кишечной палочки (E. coli), хранящейся в пробирке объемом 1,5 мл при -80 °C отдельно, и инкубируйте на льду в течение 30 мин. Тепловой удар по ячейкам при 42 °C в течение 90 с, немедленно перенесите их на лед и охладите в течение 2 мин.
ПРИМЕЧАНИЕ: Сенсорная плазмида, обеспечиваемая набором для анализа ги-ингибирования цАМФ (Таблица материалов), экспрессирует модифицированный ген люциферазы, слитый с цАМФ-связывающим доменом, и увеличивает люминесцентную активность при связывании цАМФ. - Встряхните пробирку при 37 °C в течение 1 ч после добавления 300 мкл среды лизогенного бульона (LB). Пластину 100 мкл клеток к агаровой пластине LB и инкубируют при 37 °C, выдерживая в темноте в течение 48 ч. Инокулируют белую колонию в 5 мл LB-среды, содержащей 100 мкг/мл ампициллина, и культивируют при 37 °C в течение 16 ч.
- Изолировать ДНК с помощью плазмидного минипрепа (Таблица материалов) в соответствии с инструкциями производителя; плазмида находилась в концентрации более 350 нг/мкл со значением А260/А280 между 1,7 и 1,9.
- Добавьте рекомбинантные векторы pcDNA3.1+ или плазмиду датчика (Таблица материалов) к 50 мкл dh5α-компетентной кишечной палочки (E. coli), хранящейся в пробирке объемом 1,5 мл при -80 °C отдельно, и инкубируйте на льду в течение 30 мин. Тепловой удар по ячейкам при 42 °C в течение 90 с, немедленно перенесите их на лед и охладите в течение 2 мин.
- Клеточная культура
- Поместите клетки CHO-K1 в чашку Петри размером 10 см, культивируйте их в инкубаторе с температурой 37 ° C с 5% CO2 и соберите их, когда монослой находится на слиянии 80-90%.
- Аспирируйте питательную среду клеток CHO-K1, аккуратно добавьте 4 мл 0,05% трипсина-ЭДТА, предварительно нагретого при 37 °C, на чашку Петри и инкубируйте в течение 15 с. Затем добавляют 4 мл питательной среды, состоящей из среды F12 + 10% FBS.
- Вытесните клетки с поверхности чашки Петри, осторожно покачиваясь и постукивая по боковой стороне чашки Петри. Наполните коническую трубку клеточной суспензией. Медленно перемешайте и медленно выньчите, чтобы аккуратно удалить комочки клеток.
- Центрифужные ячейки при 250 х г в течение 5 мин при комнатной температуре, аспирируют супернатант и реанимируют 3 мл PBS. Повторите этот шаг.
- Определяют номер ячейки с помощью гемацитометра и центрифужных ячеек при 250 х г в течение 5 мин при комнатной температуре.
- Аспират PBS буферные и повторноуспендированные клетки CHO-K1 с 3 мл питательной среды, состоящей из среды F12 и 10% FBS.
- Добавьте 2 мл питательной среды, состоящей из среды F12 и 10% FBS, в каждую лунку шестилуночной пластины и посейте 150 мкл клеточной суспензии в каждую лунку, чтобы сохранить ячейки при плотности 1,5 х 105 клеток/мл. Инкубируют шестилуночную пластину в инкубаторе культуры тканей при температуре 37 °C с 5% CO2 в течение примерно 24 ч.
- Транзиторная трансфекция
- Разбавить 2 мкг ДНК (Стадия 3.2.3) в 200 мкл буфера трансфекционного реагента (Таблица материалов). Перемешайте путем вихря в течение 10 с и ненадолго вращаясь перед использованием.
ПРИМЕЧАНИЕ: Здесь 2 мкг ДНК содержат 0,5 мкг рецепторного вектора (S1PR1, S1PR3 или S1PR5) и 1,5 мкг плазмиды датчика. - Добавьте 4 мкл трансфекционного реагента (Таблица материалов), вихрь в течение 10 с и ненадолго вращайте перед использованием. Инкубировать в течение 15 мин при комнатной температуре.
- Медленно капните 200 мкл трансфекционной смеси в каждую лунку (содержащую клетки CHO-K1), чтобы распределить равномерно. Аккуратно встряхните шестилуночную пластину, чтобы обеспечить тщательное перемешивание.
- Замените трансфекционную среду через 4-6 ч средой роста клеток, состоящей из среды F12 + 10% FBS, и верните шестилуночную пластину в инкубатор.
- Сбор клеток через 24-48 ч после трансфекции.
- Переварить клетки CHO-K1 на лунке с 500 мкл 0,05% трипсина-ЭДТА (предварительно нагретого при 37 °C) в течение 15 с и добавить 1 мл питательной среды, состоящей из среды F12 + 10% FBS. Выбивайте клетки с поверхности скважины, раскачивая и осторожно постукивая по боковой части скважины.
- Переложите клеточную суспензию в коническую трубку и центрифугу при 250 х г в течение 5 мин при комнатной температуре. Вылейте супернатант и соберите трансфектированные клетки.
ПРИМЕЧАНИЕ: Перед определением флуоресцентного сигнала проверьте уровни экспрессии рецепторов на клеточной поверхности с помощью ИФА, как описано ранее26.
- Разбавить 2 мкг ДНК (Стадия 3.2.3) в 200 мкл буфера трансфекционного реагента (Таблица материалов). Перемешайте путем вихря в течение 10 с и ненадолго вращаясь перед использованием.
- Уравновешивание солью D-люциферин-калия (Таблица материалов)
- Суспендировать собранные клетки (24-48 ч после трансфекции) с 3 мл буфера анализа немедленно (т.е. сбалансированный солевой раствор Хэнка (HBSS), содержащий 10 мМ HEPES pH 7,4), с дополнительным 3% v/v разбавления D-люциферин-калиевой соли.
- Добавьте 90 мкл клеточной суспензии на лунку 96-луночной пластины с помощью многоканальной пипетки и аккуратно перемешайте.
- Инкубировать в течение 40 мин при комнатной температуре.
- Определение флуоресцентного сигнала
- Заранее готовят 10 мМ стоковых растворов Сипонимода, растворенных в ДМСО, и производят серийное разбавление с использованием буфера HBSS, содержащего 25 мкМ форсколина перед стимуляцией лиганда.
ПРИМЕЧАНИЕ: За исключением контрольной группы без лиганда, остальные имеют диапазон градиента концентрации 10-11-10-5 моль/л. - Стимулируют 10 мкл (на лунку) раствора агониста в различных концентрациях в течение 30 мин.
- Подсчитайте люминесцентные сигналы на считывателе микропластин с помощью соответствующих параметров программного обеспечения (Таблица материалов) следующим образом. Выберите Люминесценция для метода обнаружения, Конечная точка для типа считывания и Люминесцентное волокно для типа оптики. Установите коэффициент усиления оптики равным 255.
ПРИМЕЧАНИЕ: Каждое измерение повторялось по меньшей мере в трех независимых экспериментах, каждый в трех экземплярах. - Получение значений флуоресцентного сигнала, импорт данных в программу для работы с электронными таблицами и обработка данных с помощью функции нелинейной регрессии (подгонки кривой) «доза-реакция».
- Заранее готовят 10 мМ стоковых растворов Сипонимода, растворенных в ДМСО, и производят серийное разбавление с использованием буфера HBSS, содержащего 25 мкМ форсколина перед стимуляцией лиганда.
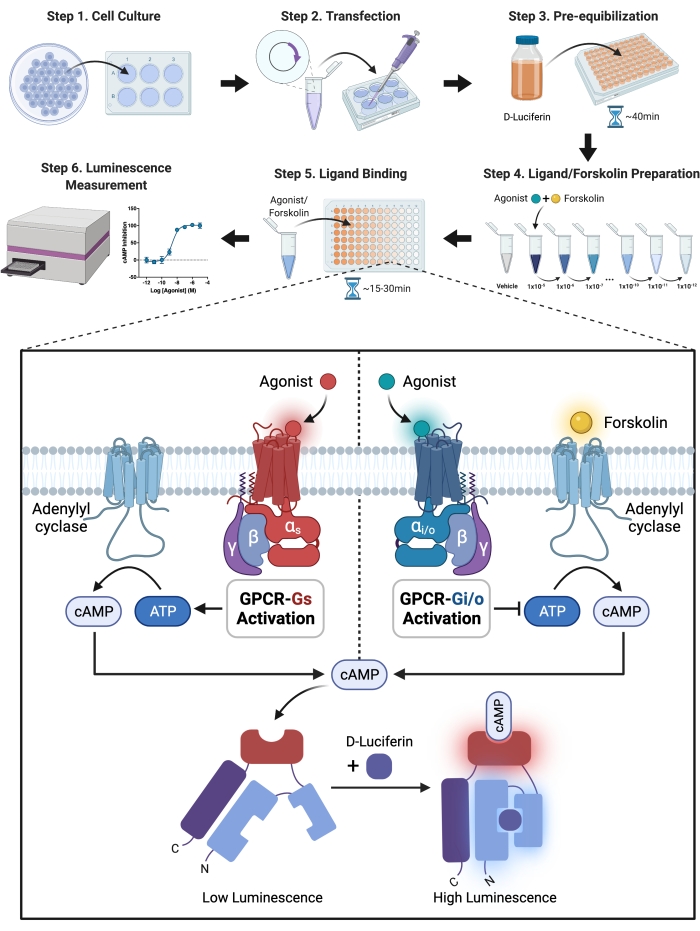
Рисунок 1: Схематическая иллюстрация эксперимента. Подробное пошаговое руководство по экспериментальной настройке и выполнению. Короче говоря, рецептор и модифицированная люцифераза были временно совместно экспрессированы в клетках CHO-K1 путем трансфекции рецептора и сенсорной плазмиды в клетки с помощью трансфекционного реагента. Клетки суспендировали в растворе HBSS с D-люциферин-калиевой солью, субстратом люциферазы, и засевали в 96-луночную пластину через 24 ч. Чтобы обеспечить проникновение в клетки, D-люциферин должен быть предварительно уравновешен с клетками. Окислительный фермент люцифераза превращает люциферин в оксилюциферин и излучает свет. Модифицированная люцифераза, с другой стороны, генерирует свет посредством химической реакции только при связывании с цАМФ, а интенсивность света имеет положительную связь с уровнями цАМФ в клетках. Уровни цАМФ регулировались с помощью GPCR, активированного агонистом. Gi-связанные рецепторы снижали уровни цАМФ, что требовало добавления форсколина для активации аденилилциклазы в эксперименте с Gi-ингибированием цАМФ. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Результаты
Перед замораживанием образца комплекса S1PRs-Gi очищенный образец необходимо отделить с помощью размерно-эксклюзионной хроматографии (SEC) и проанализировать с помощью гель-фильтрационной хроматографии. На рисунке 2 показан комплекс S1PR3-Gi в качестве примера. Пиковая фракци...
Обсуждение
Этот протокол описывает первичный конвейер для определения структур S1RR с помощью крио-ЭМ и измерения эффективности активации S1RR с помощью Gi-опосредованного анализа ингибирования цАМФ. Некоторые шаги имеют решающее значение для успеха эксперимента.
Для очистки комплек?...
Раскрытие информации
У авторов нет конфликта интересов.
Благодарности
Данные комплекса S1PRs-Gi были собраны в Западно-Китайском крио-ЭМ-центре в Сычуаньском университете и Центре крио-ЭМ в Южном университете науки и техники (SUSTech) и обработаны в Центре высокопроизводительных вычислений Duyu в Сычуаньском университете. Эта работа была поддержана Фондом естественных наук Китая (32100965 в Лос-Анджелесе, 32100988 в W.Y., 31972916 в Z.S.) и Фондом постдокторских исследований Сычуаньского университета (2021SCU12003 в Лос-Анджелесе).
Материалы
| Name | Company | Catalog Number | Comments |
| 0.05% trypsin-EDTA | GIBCO | Cat# 25300054 | |
| 0.22 µM filter | Thermo Fisher Scientific | Cat# 42213-PS | |
| 100 kDa cut-off concentrator | Thermo Fisher Scientific | Cat# 88533 | |
| 6-well plate | Corning | Cat# 43016 | |
| 96-well plate | Corning | Cat# 3917 | |
| Aprotinin | Sigma-Aldrich | Cat# 9087-70-1 | |
| Apyrase | NEB | Cat# M0398S | |
| Baculovirus transfection reagent | Thermo Fisher Scientific | Cat# 10362100 | For the preparation of P0 baculovirus |
| Benzamidine | Sigma-Aldrich | Cat# B6506 | |
| CHO-K1 | ATCC | N/A | |
| CHS | Sigma-Aldrich | Cat# C6512 | |
| CryoSPARC | Punjani, A., et al.,2017 | https://cryosparc.com/ | |
| DH5α competent E.coli | Thermo Fisher Scientific | Cat# EC0112 | |
| D-Luciferin-Potassium Salt | Sigma- Aldrich | Cat# 50227 | |
| DMSO | Sigma- Aldrich | Cat# D2438 | |
| EDTA | Thermo Fisher Scientific | Cat# S311-500 | |
| ESF921 cell culture medium | Expression Systems | Cat# 96-001 | |
| Excel | microsoft | N/A | |
| F12 medium | Invitrogen | Cat# 11765 | |
| FBS | Cell Box | Cat# SAG-01U-02 | |
| Flag resin | Sigma- Aldrich | Cat# A4596 | |
| Forskolin | APExBIO | Cat# B1421 | |
| Gctf | Zhang, 2016 | https://www.mrc-lmb.cam.ac.uk/kzhang/Gctf/ | |
| GDN | Anatrace | Cat# GDN101 | |
| Gel filtration column | GE healthcare | Cat# 28990944 | |
| Gen5 3.11 | BIO-TEK | N/A | |
| Gentamicin | Solarbio | Cat# L1312 | |
| GloSensor cAMP assay kit | Promega | Cat# E1291 | Gi-inhibition cAMP assay kit |
| GloSensor plasmid | Promega | Cat# E2301 | Sensor plasmid |
| Grace’s medium | GIBCO | Cat# 11595030 | |
| GraphPad Prism 8 | Graphpad | N/A | |
| HBSS | Thermo Fisher Scientific | Cat# 88284 | |
| HEPES | Sigma- Aldrich | Cat# H4034 | |
| jetPRIME Reagent | Polyplus Transfection | Cat# 114-15 | transfection reagent |
| Janamycin | Solarbio | Cat# K1030 | |
| LB medium | Invitrogen | Cat# 12780052 | |
| Leupeptin | Sigma-Aldrich | Cat# L2884 | |
| LMNG | Anatrace | Cat# NG310 | |
| MotionCor2 | (Zheng et al., 2017) | https://emcore.ucsf.edu/ucsf-software | |
| NanoCab | Thermo Fisher Scientific | Cat# 1121822 | |
| PBS | Invitrogen | Cat# 14190-144 | |
| pcDNA3.1-HA-FLAG-S1PRs | GenScript | N/A | |
| pFastBac1-Gαi | GenScript | N/A | |
| pFastBac1-HA-FLAG-T4L-S1PRs-His10 | GenScript | N/A | |
| pFastBacdual-Gβ1γ2 | GenScript | N/A | |
| PureLink HiPure Plasmid Miniprep Kit | Invitrogen | Cat# K210003 | For the preparation of plasmids and P0 baculovirus |
| Q5 site-Directed Mutagenesis kit | NEB | Cat# E0554S | For the preparation of plasmids |
| Quantifoil | Quantifoil | Cat# 251448 | |
| RELION-3.1 | (Zivanov et al., 2018) | https://www2.mrc-lmb.cam.ac.uk/relion | |
| S1PRs cDNA | addgene | N/A | |
| scFv16 | Invitrogen | Cat# 703976 | |
| Sf9 | Expression Systems | N/A | |
| Siponimod | Selleck | Cat# S7179 | |
| sodium cholate | Sigma-Aldrich | Cat# C1254 | |
| Synergy H1 microplate reader | BIO-TEK | N/A | |
| Synthetic T4L DNA (sequence) | N/A | N/A | Aacatcttcgagatgctgcgcatcgacgaagg cctgcgtctcaagatttacaagaataccgaagg ttattacacgattggcatcggccacctcctgaca aagagcccatcactcaacgctgccaagtctga actggacaaagccattggtcgcaacaccaac ggtgtcattacaaaggacgaggcggagaaac tcttcaaccaagatgtagatgcggctgtccgtgg catcctgcgtaatgccaagttgaagcccgtgt atgactcccttgatgctgttcgccgtgcagcctt gatcaacatggttttccaaatgggtgagaccgg agtggctggttttacgaactccctgcgcatgctcc agcagaagcgctgggacgaggccgcagtga atttggctaaatctcgctggtacaatcagacacc taaccgtgccaagcgtgtcatcactaccttccg tactggaacttgggacgcttac |
| TCEP | Thermo Fisher Scientific | Cat# 77720 | |
| Tetracycline | Solarbio | Cat# T8180 | |
| Vitrobot Mark IV | Thermo Fisher Scientific | N/A |
Ссылки
- Verstockt, B., et al. Sphingosine 1-phosphate modulation and immune cell trafficking in inflammatory bowel disease. Nature Reviews: Gastroenterology & Hepatology. , 1-16 (2022).
- Rosen, H., Stevens, R. C., Hanson, M., Roberts, E., Oldstone, M. B. Sphingosine-1-phosphate and its receptors: structure, signaling, and influence. Annual Review of Biochemistry. 82, 637-662 (2013).
- Cartier, A., Hla, T. Sphingosine 1-phosphate: Lipid signaling in pathology and therapy. Science. 366 (6463), 5551 (2019).
- Jozefczuk, E., Guzik, T. J., Siedlinski, M. Significance of sphingosine-1-phosphate in cardiovascular physiology and pathology. Pharmacological Research. 156, 104793 (2020).
- Kihara, Y., Maceyka, M., Spiegel, S., Chun, J. Lysophospholipid receptor nomenclature review: IUPHAR Review 8. British Journal of Pharmacology. 171 (15), 3575-3594 (2014).
- Bryan, A. M., Del Poeta, M. Sphingosine-1-phosphate receptors and innate immunity. Cellular Microbiology. 20 (5), 12836 (2018).
- Pelletier, D., Hafler, D. A. Fingolimod for multiple sclerosis. New England Journal of Medicine. 366 (4), 339-347 (2012).
- Obinata, H., Hla, T. Sphingosine 1-phosphate and inflammation. International Immunology. 31 (9), 617-625 (2019).
- Pyne, N. J., Pyne, S. Sphingosine 1-phosphate and cancer. Nature Reviews: Cancer. 10 (7), 489-503 (2010).
- Abu-Farha, M., et al. The role of lipid metabolism in COVID-19 virus infection and as a drug target. International Journal of Molecular Sciences. 21 (10), 3544 (2020).
- Chun, J., Kihara, Y., Jonnalagadda, D., Blaho, V. A. Fingolimod: lessons learned and new opportunities for treating Multiple Sclerosis and other disorders. Annual Review of Pharmacology and Toxicology. 59, 149-170 (2019).
- Murakami, A., et al. Sphingosine 1-phosphate (S1P) regulates vascular contraction via S1P3 receptor: investigation based on a new S1P3 receptor antagonist. Molecular Pharmacology. 77 (4), 704-713 (2010).
- Cao, L., et al. Siponimod for multiple sclerosis. Cochrane Database of Systematic Reviews. 11, (2021).
- Scott, L. J. Siponimod: a review in secondary progressive Multiple Sclerosis. CNS Drugs. 34 (11), 1191-1200 (2020).
- Lamb, Y. N. Ozanimod: first approval. Drugs. 80 (8), 841-848 (2020).
- Scott, F. L., et al. Ozanimod (RPC1063) is a potent sphingosine-1-phosphate receptor-1 (S1P1) and receptor-5 (S1P5) agonist with autoimmune disease-modifying activity. British Journal of Pharmacology. 173 (11), 1778-1792 (2016).
- McGowan, E. M., Haddadi, N., Nassif, N. T., Lin, Y. Targeting the SphK-S1P-SIPR pathway as a potential therapeutic approach for COVID-19. International Journal of Molecular Sciences. 21 (19), 7189 (2020).
- O'Sullivan, C., Dev, K. K. The structure and function of the S1P1 receptor. Trends in Pharmacological Sciences. 34 (7), 401-412 (2013).
- Liao, M., Cao, E., Julius, D., Cheng, Y. Structure of the TRPV1 ion channel determined by electron cryo-microscopy. Nature. 504 (7478), 107-112 (2013).
- Bai, X. C., McMullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- Murata, K., Wolf, M. Cryo-electron microscopy for structural analysis of dynamic biological macromolecules. Biochimica et Biophysica Acta General Subjects. 1862 (2), 324-334 (2018).
- Zhang, M., et al. Cryo-EM structure of an activated GPCR-G protein complex in lipid nanodiscs. Nature Structural & Molecular Biology. 28 (3), 258-267 (2021).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews: Drug Discovery. 17 (7), 471-492 (2018).
- Ishchenko, A., Gati, C., Cherezov, V. Structural biology of G protein-coupled receptors: new opportunities from XFELs and cryoEM. Current Opinion in Structural Biology. 51, 44-52 (2018).
- Yang, D., et al. G protein-coupled receptors: structure- and function-based drug discovery. Signal Transduction and Targeted Therapy. 6 (1), 7 (2021).
- Yuan, Y., et al. Structures of signaling complexes of lipid receptors S1PR1 and S1PR5 reveal mechanisms of activation and drug recognition. Cell Research. 31 (12), 1263-1274 (2021).
- Zhao, C., et al. Structural insights into sphingosine-1-phosphate recognition and ligand selectivity of S1PR3-Gi signaling complexes. Cell Research. 32 (2), 218-221 (2022).
- Xu, Z., et al. Structural basis of sphingosine-1-phosphate receptor 1 activation and biased agonism. Nature Chemical Biology. 18, 281-288 (2022).
- Liu, Y. F., Ghahremani, M. H., Rasenick, M. M., Jakobs, K. H., Albert, P. R. Stimulation of cAMP synthesis by Gi-coupled receptors upon ablation of distinct Galphai protein expression. Gi subtype specificity of the 5-HT1A receptor. Journal of Biological Chemistry. 274 (23), 16444-16450 (1999).
- Buccioni, M., et al. Innovative functional cAMP assay for studying G protein-coupled receptors: application to the pharmacological characterization of GPR17. Purinergic Signalling. 7 (4), 463-468 (2011).
- Wang, F. I., Ding, G., Ng, G. S., Dixon, S. J., Chidiac, P. Luciferase-based GloSensor cAMP assay: Temperature optimization and application to cell-based kinetic studies. Methods. , (2021).
- Audet, M., et al. Small-scale approach for precrystallization screening in GPCR X-ray crystallography. Nature Protocols. 15 (1), 144-160 (2020).
- Sgro, G. G., Costa, T. R. D. Cryo-EM grid preparation of membrane protein samples for single particle analysis. Frontiers in Molecular Biosciences. 5, 74 (2018).
- White, J. B. R., et al. Single particle cryo-electron microscopy: from sample to structure. Journal of Visualized Experiments. (171), e62415 (2021).
- Thompson, R. F., Iadanza, M. G., Hesketh, E. L., Rawson, S., Ranson, N. A. Collection, pre-processing and on-the-fly analysis of data for high-resolution, single-particle cryo-electron microscopy. Nature Protocols. 14 (1), 100-118 (2019).
- Fernandez-Leiro, R., Scheres, S. H. W. A pipeline approach to single-particle processing in RELION. Acta Crystallographica Section D. 73 (6), 496-502 (2017).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14 (3), 290-296 (2017).
- Brilot, A. F., et al. Beam-induced motion of vitrified specimen on holey carbon film. Journal of Structural Biology. 177 (3), 630-637 (2012).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14 (4), 331-332 (2017).
- Zhang, K. Gctf: Real-time CTF determination and correction. Journal of Structural Biology. 193 (1), 1-12 (2016).
- Scheres, S. H. Semi-automated selection of cryo-EM particles in RELION-1.3. Journal of Structural Biology. 189 (2), 114-122 (2015).
- Liu, S., et al. Differential activation mechanisms of lipid GPCRs by lysophosphatidic acid and sphingosine 1-phosphate. Nature Communications. 13 (1), 731 (2022).
- Duan, J., et al. Cryo-EM structure of an activated VIP1 receptor-G protein complex revealed by a NanoBiT tethering strategy. Nature Communications. 11 (1), 4121 (2020).
- DiIorio, M. C., Kulczyk, A. W. A robust single-particle cryo-electron microscopy (cryo-EM) processing workflow with cryoSPARC, RELION, and Scipion. Journal of Visualized Experiments. (179), e63387 (2022).
- Pradelles, P., Grassi, J., Chabardes, D., Guiso, N. Enzyme immunoassays of adenosine cyclic 3',5'-monophosphate and guanosine cyclic 3',5'-monophosphate using acetylcholinesterase. Analytical Chemistry. 61 (5), 447-453 (1989).
- Jiang, L. I., et al. Use of a cAMP BRET sensor to characterize a novel regulation of cAMP by the sphingosine 1-phosphate/G13 pathway. Journal of Biological Chemistry. 282 (14), 10576-10584 (2007).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены