需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
研究鞘氨醇1-磷酸受体结构和信号通路的管道
摘要
S1P通过S1P受体(S1PRs)亚家族发挥其多样化的生理作用。这里描述了一个管道来阐述S1PR的结构和功能。
摘要
溶血磷脂(LPL)是生物活性脂质,包括鞘氨醇1-磷酸(S1P),溶血磷脂酸等。S1P是细胞膜中鞘脂的代谢产物,是表征最好的LPL之一, 通过 鞘氨醇1-磷酸受体(S1PRs)介导的信号通路调节各种细胞生理反应。这意味着S1P-S1PR信号系统是疾病(包括多发性硬化症(MS),自身免疫性疾病,癌症,炎症甚至COVID-19)的显着潜在治疗靶点。S1PR是A类G蛋白偶联受体(GPCR)家族的一小部分,由五种亚型组成:S1PR1,S1PR2,S1PR3,S1PR4和S1PR5。然而,缺乏详细的结构信息阻碍了靶向S1PR的药物发现。在这里,我们应用冷冻电子显微镜方法求解了S1P-S1PR复合物的结构,并利用基于细胞的功能测定阐明了活化,选择性药物识别和G蛋白偶联的机制。其他溶酶磷脂受体(LPLR)和GPCR也可以使用这种策略进行研究。
引言
鞘氨醇-1-磷酸(S1P)是细胞膜中鞘脂的代谢产物,是一种无处不在的溶血磷脂信号分子,涉及各种生物活性,包括淋巴细胞运输,血管发育,内皮完整性和心率1,2,3。S1P通过五种S1P受体亚型(S1PRs 1-5)发挥其多样化的生理作用;S1PR存在于各种组织中,对下游G蛋白表现出独特的偏好4,5。S1PR1主要与Gi蛋白偶联,随后抑制cAMP的产生;S1PR2 和 S1PR3 与 Gi、Gq 和 G12/13 耦合,S1PR4 和 S1PR5 通过 Gi 和 G12/136 转换信号。
S1P-S1PR信号传导是多种疾病的关键治疗靶点,包括自身免疫性疾病7,炎症8,癌症9,甚至COVID-1910。2010年,芬戈莫德(FTY720)被批准为靶向S1PR治疗复发性多发性硬化症(MS)11的同类药物。然而,它能够与除S1PR2以外的所有S1PR结合,而与S1PR3的非特异性结合导致大脑皮层水肿,血管和支气管收缩以及肺上皮渗漏12。作为增加治疗选择性的替代策略,已经产生了受体的亚型特异性配体。西波尼莫德(BAF312)于2019年被批准用于复发性MS治疗13;它有效地靶向S1PR1和S1PR5,而它对S1PR3没有亲和力,在临床实践中表现出较少的副作用14。2020年,美国食品和药物管理局授权奥扎尼莫特用于MS治疗15。据报道,臭氧莫德对S1PR1的选择性比S1PR516高25倍。值得注意的是,在当前COVID-19大流行的背景下,已经发现靶向S1PR的激动剂药物可以通过使用免疫调节治疗技术来治疗COVID-19 17。与芬戈莫德相比,奥扎尼莫德在降低COVID-19患者的症状方面显示出优越性,目前正在进行临床试验10。了解S1PR的结构基础和功能为开发选择性靶向S1PR18的药物奠定了重要基础。
许多技术用于研究生物大分子的结构信息,包括X射线晶体学,核磁共振(NMR)和电子显微镜(EM)。截至2022年3月,蛋白质数据库(PDB)上沉积了超过180,000个结构,其中大多数已通过X射线晶体学解析。然而,随着程轶凡和David Julius在2013年19日报道了TPRV1的第一个近原子分辨率结构(3.4 Å分辨率),冷冻电子显微镜(cryo-EM)已成为蛋白质结构的主流技术,EM PDB结构的总数超过10,000个。关键的突破领域是开发新的成像相机,称为直接电子检测相机和新的图像处理算法。在过去的十年中,冷冻电镜彻底改变了结构生物学和基于结构的药物发现20.由于了解大分子复合物如何在活细胞中履行其复杂的作用是生物科学的中心主题,冷冻电镜有可能揭示动态分子复合物的构象,特别是对于跨膜蛋白21。G蛋白偶联受体(GPCRs)是跨膜蛋白的最大超家族,也是目前上市药物中30%以上的靶标22。冷冻电镜的发展促成了GPCR-G蛋白复合物的高分辨率结构的爆发,从而能够确定药物设计23中X射线晶体学分析仍然无法获得的"难处理"靶标的结构。因此,冷冻电镜应用提供了在接近原子分辨率24的情况下确定近天然条件下GPCR三维结构的机会。冷冻电镜的进步使得可视化GPCR刺激或抑制的机制基础成为可能,并进一步有利于揭示GPCR靶向药物创造的新型结合位点25。
依靠冷冻电镜技术的巨大进步,我们最近确定了激动的S1PR1-,S1PR3-和S1PR5-Gi信号复合物的结构26,27。在人类中,S1PR存在于各种组织中,对下游G蛋白表现出独特的偏好4,5。S1PR1主要与Gi蛋白偶联,随后抑制3',5'-环磷酸腺苷(cAMP)的产生。S1PR3 和 S1PR5 也能够与 Gi6,28 耦合。由于Gi偶联受体激活降低了cAMP29的产生,因此引入了Gi抑制cAMP测定法来测量捕获功能改变26,27的cAMP抑制作用。使用萤 火虫 荧光素酶的突变版本,其中插入了cAMP结合蛋白部分,该cAMP测定提供了一种简单可靠的方法,用于通过细胞内cAMP浓度30的变化来监测GPCR活性。它是一种灵敏的非放射性功能测定,可用于监测各种GPCR的实时下游信号传导,用于药物发现目的31.
这里总结了解决S1PR的活化和药物识别模式的关键方法,主要包括冷冻电镜操作和Gi抑制cAMP测定。本文旨在为进一步探索GPCR的结构和功能提供全面的实验指导。
研究方案
1. S1PRs-G蛋白复合物的纯化
- 为了纯化人S1PRs-G蛋白复合物,将缺乏C端残基338-382的S1PR1的C1PR1的cDNA、在C末端用345-398截断的野生型S1PR5和野生型Gi1克隆到pFastBac1载体中,将野生型Gβ1和Gγ2的cDNA克隆到pFastBacdual载体中(材料表)。
注意:S1PR的所有构建体还包含血凝素(HA)信号序列,后跟N端的Flag表位标签和C端的10倍他的标签。此外,将用于翻译T4溶菌酶(T4L)的合成DNA序列(材料表)插入S1PR的N端,以促进受体表达和纯化。 - 编码S1PR、Gi1和Gβ1γ2的杆状病毒的制备
- 将重组载体加入50μL储存在-80°C的1.5mL管中的DH5α主管 大肠杆菌(大肠杆菌) 中,并在冰上孵育30分钟。
- 在42°C下热冲击细胞90秒,立即将它们转移到冰上,并冷却2分钟。
- 在补充300μL溶血汤(LB)培养基后,在37°C下摇动管3-5小时。将100μL细胞板铺板至LB琼脂平板上,并在37°C下通过保持黑暗48小时来孵育。
- 将白色菌落接种到含有50μg/ mL卡那霉素,10μg/ mL四环素和7μg/ mL庆大霉素的5mLLB培养基中,并在37°C下培养16小时。
- 按照制造商的说明,使用质粒mimprep试剂盒(材料表)分离重组杆粒,以产生P0杆状病毒。
注意:在使用之前,用pUC / M13正向和反向引物通过PCR分析纯化的杆粒。对于PCR,循环次数= 30个循环,熔解温度= 58°C,延伸时间= 每1 Kb1分钟。 - 按照先前方案32中所述制备P0杆状病毒。
- 在六孔板中培养 sf9 细胞(ESF921培养基),并验证细胞是否处于对数期(1.0-1.5 x 106 个细胞/ mL)。
- 在100μLGrace培养基中稀释8μL杆状病毒转染试剂,并在室温下孵育30分钟。在100μLGrace培养基中稀释10μg重组杆粒,并轻轻混合。将稀释的杆粒与稀释的杆状病毒转染试剂混合,轻轻混合,并在室温下孵育30分钟。
- 将混合物(杆粒和试剂)加入细胞(步骤1.2.6.1),并在27°C下平板3小时。
- 取出Grace的培养基,用2 mL ESF921细胞培养基代替。在27°C下接种六孔板,并在转染后5天后收集ESF921细胞培养基。
- 在4°C下以500× g 离心10分钟以除去细胞和碎屑。将上清液转移到2 mL管中。这是P0杆状病毒的股票。
- 分离 P1 病毒库存
- 将30mL sf9 细胞转移到锥形瓶中,在27°C下培养,以270rpm振荡,并验证细胞达到对数阶段(1.0-1.5×106 个细胞/ mL)。
- 向瓶中加入2 mL P0病毒储备液,并在27°C下以270rpm摇动4天。
- 将细胞转移到50mL管中,在4°C下以1,800× g 离心10分钟以除去细胞和碎屑,并将上清液转移到50mL管中。这是P1杆状病毒库存。
- 扩增杆状病毒储液
- 在对数期(1.0-1.5 x 106个细胞/mL)中使用50 mL sf9细胞和1 mL P1储备液重复步骤1.2.7。
- 将产生的P2杆状病毒储备储存在4°C,保护其免受光照。
注意:不要无限期地扩增杆状病毒,因为每次传代都会产生有害的突变体。
- S1PRs-G蛋白复合物的表达
- 培养 sf9 昆虫细胞以达到2.5×106 个细胞/ mL密度,与编码S1PR,Gi1和Gβ1γ2的P2杆状病毒以1:2:1的体积比共同感染,并在27°C下再次培养48小时。
- 通过在4°C下以700× g 离心15分钟来收集细胞,将它们冷冻在液氮中,并将其储存在-80°C下使用。
- 蛋白质纯化
- 在室温下解冻步骤1.3中获得的细胞沉淀,然后将其重悬于裂解缓冲液(20mM HEPES pH 7.5,50mM NaCl,5mMMgCl2,5mM CaCl2),其中补充有100μg/mL苯甲脒,100μg/mL亮肽,100μg/mL抑肽酶,25mU/ mL吡咯烷酮和10μM激动剂。在室温下搅拌细胞悬浮液2小时以诱导S1PRs-G蛋白复合物的形成。
注意:阿比拉斯是一种ATP二磷酸水解酶。它催化从ATP中去除γ磷酸盐和从ADP中除去β磷酸盐。 - 将溶液转移到管中,以70,000× g 离心10分钟,并小心地除去上清液。将沉淀重悬于增溶缓冲液(20 mM HEPES pH 7.5,100 mM氯化钠,5 mM氯化镁2,5mM氯化钙2,0.5%(w / v)改性活液,0.1%(w / v)CHS,1%(w / v)胆酸钠,10%(v / v)甘油)中。
- 将悬浮液转移到玻璃道恩斯中,并使颗粒完全均质化。向悬浮液中加入10μM激动剂,4mg scFv16,100μg/ mL苯甲脒,100μg/ mL亮肽酶,100μg/ mL抑肽酶和25mU / mL吡咯烷酮,并在4°C下搅拌2小时。
注意:匀浆颗粒的步骤对于GPCR-G蛋白复合物的生产至关重要。 - 将溶液转移到管中,并在4°C下以100,000× g 离心30分钟。
- 用洗涤缓冲液(20 mM HEPES pH 7.5,100 mM 氯化钠,5 mM 氯化镁2,5 mM 钙氯化2,10 μM 激动剂,0.0375% (w/v) 改性活化液、0.0125% (w/v) GDN、0.01% (w/v) CHS)预平衡标志树脂。
- 将上清液转移到管中,并与标志树脂在4°C下孵育2小时。
- 将上述混合物上样到玻璃柱上,并用50 mL洗涤缓冲液洗涤柱,其中补充有100μg/ mL苯甲脒,100μg/ mL亮蛋白和100μg/ mL抑肽酶。
- 用含有 20 mM HEPES pH 7.5、100 mM 氯化钠、5 mM EDTA、200 μg/mL 标志肽、10 μM 激动剂、0.0375% (w/v) LMNG、0.0125% (w/v) GDN、0.01% (w/v) CHS、100 μg/mL 苯甲脒、100 μg/mL 低聚肽和 100 μg/mL 抑肽的洗脱缓冲液洗脱色谱柱。
- 收集 S1PRs-G 蛋白复合物,并使用 100 kDa 截止浓缩器在 4 °C 下以 1,300 x g 浓度浓缩至 1 mL。 通过0.22μM过滤器过滤,并在4°C下以13,000× g 离心10分钟以除去聚集体。
- 将 S1PRs-G 蛋白复合物加载到尺寸排阻色谱 (SEC) 凝胶过滤柱上,该柱与含有 20 mM HEPES pH 7.5、100 mM 氯化钠、10 μM 激动剂、10 μM TCEP、0.00375% (w/v) LMNG、0.00125% (w/v) GDN 和 0.001% (w/v) CHS 的 SEC 缓冲液在 4 °C 下以 0.5 mL/min 的流速进行预平衡。
- 收集峰级分,并使用100 kDa临界浓缩器在4°C下以1,300 x g 浓度进行冷冻电镜浓缩。
- 在室温下解冻步骤1.3中获得的细胞沉淀,然后将其重悬于裂解缓冲液(20mM HEPES pH 7.5,50mM NaCl,5mMMgCl2,5mM CaCl2),其中补充有100μg/mL苯甲脒,100μg/mL亮肽,100μg/mL抑肽酶,25mU/ mL吡咯烷酮和10μM激动剂。在室温下搅拌细胞悬浮液2小时以诱导S1PRs-G蛋白复合物的形成。
2. 通过电子显微镜解决S1PR结构
- 数据采集
- 要准备冷冻电镜网格,请使用辉光放电清洁系统将300目Au R1.2 / 1.3网格保持10秒,并在15 mA下进行辉光放电60 s。
- 如前所述进行样品玻璃化33,34。在暴冻控制台中,将腔室工作环境的温度设置为4°C,相对湿度设置为100%。使用印迹力为0,等待时间为0 s,印迹时间为2或3 s,排水时间为0 s。它通常只需要3μL样品,浓度为5-10mg / mL即可进行单次玻璃化。
- 将网格剪切并加载到自动网格组件中,将自动网格组件加载到Nanocab中,并通过自动加载器将Nanocab加载到显微镜中,如前所述35。使用 EPU2 软件筛选样品质量34.通常,在合适的冰层厚度区域收集的数据更好。
- 收集冷冻电镜数据,如前面35所述。对于S1P受体,将散焦偏移设置在-1.0μm和-1.8μm之间,暴露电子剂量为50-65 e-/ Å2。对于 S1PR1-Gi 复合物,使用 EPU2 软件在计数模式下使用 K2 检测器自动收集电影堆栈,总曝光时间为 2 秒,记录速率为每秒 5 个原始帧,总剂量为 56 e-/Å2 ,每堆栈生成 35 帧。
注意:通常需要超过5,000部电影来重建受体结构。
- 使用 RELION 组合处理数据36 和冷冻冷冻37 以获得理想的冷冻电镜密度图。使用 RELION-3.1_gpu_ompi4最初处理数据,这涉及前面描述的类似操作34.
- 在 Linux 系统终端中,输入数据存储目录的父目录。
- 在终端中输入 相关命令以打开相关图形用户界面 (GUI)。
注意:如果这是第一次在此目录中打开RELION GUI,将弹出一个提示窗口;单击" 是"。 - 单击 RELION GUI 右侧功能栏中的" 导入 ",将原始数据导入到 RELION 中。
- 在"影片/麦克风"选项中,为"导入原始影片/显微照片?"选择"是",在"原始输入文件"字段中输入数据路径(建议使用通配符),然后为"这些是多帧影片吗?"选择"是"。在像素大小(埃)字段中输入电影的像素大小,在电压(kV)字段中输入显微镜的工作电压(以kV为单位),在球面像差(mm)字段中输入显微镜的球面像差。这些是在数据收集时记录的参数。
- 在 "正在运行 "选项中,根据运行程序的服务器修改 队列名称 (其他函数也需要修改此参数)。将其他参数保留为 RELION 设置的默认值。最后,当正确检测到所有参数时,单击 RUN! 以运行该程序。
- 使用 运动校正 功能对所有帧38 进行对齐。
- 在 I/O 选项中,单击"浏览",然后选择名为 movies.star 的导入函数的输出作为输入电影 STAR 文件的输入。在每帧剂量(e/ A2)字段中输入每帧剂量,该字段等于总剂量除以帧数。为"保存功率谱之和?"选择"否"。
- 在"运动"选项中,为 B 因子输入 250,为面片数 X,Y 输入 5.5,为组帧输入 2(确保组 >3)。 如果在采集期间数据没有增益参考,则需要增益参考图像,该图像可以通过获取空白网格区域获得。选择"否使用 RELION 自己的实现?" ,然后在 MOTIONCOR2 可执行文件字段中输入包含 MOTIONCOR239 可执行文件的目录。
- 在 "正在运行 "选项中,根据服务器的计算能力选择合适的 MPI 和线程编号;这里使用了 MPI = 8 和线程 = 3。
- 使用CTF估计函数调制玻璃化标本40的冷冻电镜图像。在 I/O 选项中,单击"浏览",然后选择名为校正显微照片.star 的运动的输出作为输入电影 STAR 文件的输入。在"Gctf"选项中,为"使用"改为"是"。
- 使用 子集选择 函数删除值为 rlnCtfMax 溶血素 >4 的显微照片。
- 在 I/O 选项中,单击位于"或从显微照片.star 中选择"右侧的"浏览",然后选择名为 micrographs_ctf.star 的 CtfFind 的输出作为输入。在"子集"选项中,为"基于元数据值选择?" 选择"选择",并为"删除错误数据的最大元数据值"输入 4。
- 手动拾取:手动拾取一些图像以进行初步拾取和分类。
- 在 I/O 选项中,单击"浏览",然后从上一个"选择目录"(步骤 2.2.6)中选择"micrographs_selected.star"作为输入。
- 单击 "运行!" (弹出一个窗口)。单击新窗口左上角的" 文件 ",然后单击" 反转选择 "以取消所有图像的选择。选中每个条目对应行中最左侧的选择框,然后单击 Pick 以检查图像并选择 ~500 张好图像。单击 文件>保存选择 以保存所选图像并关闭窗口。
- 自动拾取:自动颗粒拾取软件包有用且功能强大41.
- 在 I/O 选项中,单击"输入显微照片"右侧的"浏览"以进行自动选择,然后从上一个 ManualPick 目录(步骤 2.2.7)中选择"micrographs_selected.star"作为输入。开始时使用高斯的拉普拉斯算法,因此请为 OR 选择"是":使用高斯的拉普拉斯算法。
- 在"拉普拉斯"选项中,将 LoG 滤波器 (A) 的最小直径设置为 80,将 LoG 滤波器 (A) 的最大直径设置为 130。在"自动选取"选项中,将"最小粒子间距离 (A)"设置为 65,并为"如果可以访问 GPU,则使用 GPU 加速"选择"是"。
- 提取颗粒以进行后续步骤。
- 在 I/O 选项中,单击显微照片 STAR 文件右侧的"浏览",然后从步骤 2.2.6 中选择micrographs_selected.star。单击输入坐标右侧的浏览,然后从步骤 2.2.8 中选择cords_suffix_autopick.star。
- 在"提取"选项中,为"重新缩放粒子"选择"是",并将"重新缩放大小(像素)"设置为 128 以加快后续步骤。
- 用于颗粒初步分类的 2D 分类
- 在 I/O 选项中,单击输入图像 STAR 文件右侧的"浏览",然后从步骤 2.2.9 中选择粒子.star。在"优化"选项中,将"类数"设置为 100,将"掩模直径 (A)"设置为 140。
- 在"计算"选项中,将"池化粒子数"设置为 10,在"将粒子复制到暂存目录"字段中输入位于快速本地驱动器(例如 SSD 驱动器)上的目录,然后为"使用 GPU 加速?" 选择"是"以获得更快的处理速度。
- 子集选择,用于选择良好的 2D 结果作为模板来选择粒子
- 在 I/O 选项中,单击"从 model.star 选择类"右侧的"浏览",然后选择名为 run_it025_model.star 的步骤 2.2.10 的输出作为输入。点击运行!。在弹出窗口中,选中"打开对图像进行排序"和"反向排序?",然后单击"显示!"。
- 选择具有良好代表性的 2D 结果作为自动拾取功能的参考。
注意:所选结果以红色框显示。好的和坏的 2D 分类结果将在后面显示。 - 右键单击并选择保存 所选类。
- 将模板用于第二轮自动选取。在 I/O 选项中,单击"输入显微照片"右侧的"浏览"以进行自动选择,然后从步骤 2.2.6 中选择micrographs_selected.star。单击 2D 参照右侧的"浏览",从步骤 2.2.11 中选择"class_averages"星形,然后为"OR:使用拉普拉斯高西亚n"选择"否"。
- 使用步骤 2.2.12中的coord_suffix_autopick.star 和步骤2.2.6 中的micrographs_selected.star 再次执行粒子提取。
- 使用步骤 2.2.13 中的粒子.star 再次执行 2D 分类。
- 使用步骤 2.2.14 中的 run_it025_optimiser.star 再次执行子集选择。
注意:需要选择所有具有清晰轮廓和正确形状的2D图像。 - 按如下方式进行颗粒提取。在 I/O 选项中,单击显微照片 STAR 文件右侧的"浏览",选择步骤 2.2.6 中的micrographs_selected.star,然后为"是"或"重新提取精制颗粒?"。单击"精炼粒子 STAR 文件"右侧的"浏览",然后从步骤 2.2.15 中选择粒子.star。
- 3D 初始模型并生成参考图:在 I/O 选项中,单击输入图像 STAR 文件右侧的"浏览",然后从步骤 2.2.16 中选择粒子.star。在"优化"选项中,将"类数"设置为 1,并将"掩模直径 (A)"设置为 140。
- 3D 分类并生成初步的 3D 地图:在 I/O 选项中,单击输入图像 STAR 文件右侧的"浏览",然后从步骤 2.2.16 中选择粒子.star。单击参考地图右侧的浏览,然后选择步骤 2.2.17 中的initial_model.mrc。在"优化"选项中,将"类数"设置为 4-6,将"掩模直径 (A)"设置为 140。
- 掩码生成:在 I/O 选项中选择步骤 2.2.17 中的良好 3D 地图作为输入。将"初始二值化阈值"设置为 0.05(根据输出进行调整),将"将这么多像素的二进制映射扩展到 3",并在"蒙版"选项中将这么多像素的软边添加到 8。
- 使用冷冻冷冻冷冻处理。
- 创建一个新的工作区,然后单击第一个作业的" 作业生成器 "。
- 要导入粒子堆栈,请在"粒子元路径"字段中输入 粒子路径 (步骤 2.2.16),在" 粒子数据 路径"字段中输入影片路径(步骤 2.2.16)。
注: 参数加速电压 (kV)、球面像差 (mm) 和 像素大小(埃) 与之前相同。 振幅对比度 (分数)值为 0.1。 - 通过在步骤 2.2.18 的"体积"数据路径中输入最佳 3D 体积的路径,然后为要导入的体积类型选择"映射"来导入 3D 体积。
- 通过在卷数据路径中输入掩码路径(步骤 2.2.19),然后为要导入的卷的类型选择掩码来导入掩码。
- 非均匀细化:将步骤 2.2.22、2.2.23 和 2.2.24 的输出作为输入。
注意:此功能对于膜蛋白非常有用。- 将步骤 2.2.22 (imported_particles) 的输出作为非均匀细化 粒子(粒子)的输入,将步骤 2.2.23 (imported_volume_1) 的输出作为非均匀细化 体积(体积)的输入,将步骤 2.2.24 (imported_mask_1) 的输出作为非均匀细化 掩码(掩码)的输入。
注意:有时,在没有掩模的情况下可以取得更好的效果。 - 单击" 队列 "开始处理。
- 将步骤 2.2.22 (imported_particles) 的输出作为非均匀细化 粒子(粒子)的输入,将步骤 2.2.23 (imported_volume_1) 的输出作为非均匀细化 体积(体积)的输入,将步骤 2.2.24 (imported_mask_1) 的输出作为非均匀细化 掩码(掩码)的输入。
- 运行步骤 2.2.18-2.2.25 以获得更好的结果。通过以上一系列处理,可以获得分辨率不错的S1PR-Gi 3D地图。
3. S1PRs-Gi 介导的 cAMP 抑制试验
注:将S1PR-Gi介导的cAMP抑制实验分为几个部分,以下是详细的实验步骤。实验原理和一般实验过程如图1所示,流程图为图 1。
- 质粒构建
- 将野生型 S1PR1、S1PR3 和 S1PR5 的 cDNA 亚克隆到载体 pcDNA3.1+ 中,并带有 HA 信号序列,后跟 N 末端的标记(材料表)。
注意:所有受体的突变都是通过使用诱变试剂盒产生的(材料表)。
- 将野生型 S1PR1、S1PR3 和 S1PR5 的 cDNA 亚克隆到载体 pcDNA3.1+ 中,并带有 HA 信号序列,后跟 N 末端的标记(材料表)。
- 质粒的制备
- 将重组pcDNA3.1 +载体或传感器质粒(材料表)分别加入到50μL在-80°C下储存在1.5mL管中的DH5α合格 大肠杆菌(大肠杆菌) 中,并在冰上孵育30分钟。在42°C下热冲击细胞90秒,立即将它们转移到冰上,并冷却2分钟。
注意:Gi抑制cAMP测定试剂盒(材料表)提供的传感器质粒表达与cAMP结合结构域融合的修饰荧光素酶基因,并在cAMP结合时增加发光活性。 - 在补充300μL溶血汤(LB)培养基后,在37°C下摇动管1小时。将100μL细胞板置于LB琼脂平板上,并在37°C下保持黑暗48小时。将白菌落接种到含有100μg/mL氨苄青霉素的5mLLB培养基中,并在37°C下培养16小时。
- 按照制造商的说明,使用质粒小型制备试剂盒(材料表)分离DNA;质粒的浓度超过350 ng / μL,A260 / A280 的值在1.7和1.9之间。
- 将重组pcDNA3.1 +载体或传感器质粒(材料表)分别加入到50μL在-80°C下储存在1.5mL管中的DH5α合格 大肠杆菌(大肠杆菌) 中,并在冰上孵育30分钟。在42°C下热冲击细胞90秒,立即将它们转移到冰上,并冷却2分钟。
- 细胞培养
- 将CHO-K1细胞置于10厘米培养皿中,在具有5%CO2的37°C培养箱中培养,并在单层在80%-90%汇合时收获它们。
- 吸出CHO-K1细胞生长培养基,将4mL在37°C下预热的0.05%胰蛋白酶-EDTA轻轻加入培养皿中,并孵育15秒。然后,加入4 mL由F12培养基+ 10%FBS组成的生长培养基。
- 通过轻轻摇曳并敲击培养皿的侧面,将细胞从培养皿表面移开。用细胞悬浮液填充锥形管。缓慢搅拌并移液,以轻轻去除细胞团块。
- 在室温下以250× g 离心细胞5分钟,吸出上清液,并用3mL PBS复苏。重复此步骤。
- 用血细胞计数器测定细胞数,并在室温下以250× g 离心细胞5分钟。
- 吸出PBS缓冲液并用3 mL由F12培养基和10%FBS组成的生长培养基重悬CHO-K1细胞。
- 将2mL由F12培养基和10%FBS组成的生长培养基加入到六孔板的每个孔中,并将150μL细胞悬浮液接种到每个孔中,以使细胞保持在1.5×105 个细胞/ mL的密度。将六孔板在具有5%CO2 的37°C组织培养箱中孵育约24小时。
- 瞬态转染
- 将2μgDNA(步骤3.2.3)稀释到200μL转染试剂缓冲液中(材料表)。通过涡旋混合10秒,并在使用前短暂旋转。
注意:在这里,2微克DNA含有0.5微克受体载体(S1PR1,S1PR3或S1PR5)和1.5微克传感器质粒。 - 加入4μL转染试剂(材料表),涡旋10秒,并在使用前短暂旋转。在室温下孵育15分钟。
- 将200μL转染混合物缓慢滴入每个孔(含有CHO-K1细胞)中以均匀分布。轻轻摇动六孔板,确保充分混合。
- 4-6小时后用F12培养基+ 10%FBS组成的细胞生长培养基替换转染培养基,并将六孔板返回到培养箱中。
- 转染后24-48小时收获细胞。
- 用500μL 0.05%胰蛋白酶-EDTA(在37°C下预热)在孔上消化CHO-K1细胞15秒,并添加1mL由F12培养基+ 10%FBS组成的生长培养基。通过摇动并轻轻敲击孔的侧面将细胞从孔表面移开。
- 将细胞悬浮液转移到锥形管中,并在室温下以250× g 离心5分钟。倒出上清液并收获转染的细胞。
注意:在荧光信号测定之前,通过ELISA检查受体的细胞表面表达水平,如前所述26。
- 将2μgDNA(步骤3.2.3)稀释到200μL转染试剂缓冲液中(材料表)。通过涡旋混合10秒,并在使用前短暂旋转。
- 与D-荧光素钾盐的平衡(材料表)
- 立即用3mL测定缓冲液(即含有10mM HEPES pH 7.4的汉克平衡盐溶液(HBSS))悬浮收获的细胞(转染后24-48小时),另外3%v / v稀释D-荧光素 - 钾盐。
- 使用多通道移液管在96孔板的每孔中加入90μL细胞悬浮液,并轻轻混合。
- 在室温下孵育40分钟。
- 荧光信号测定
- 预先制备溶解在DMSO中的10mM西波尼莫德储备溶液,并在配体刺激前使用含有25μM毛喉素的HBSS缓冲液进行连续稀释。
注意:除无配体的对照组外,其余组的浓度梯度范围为10-11-10-5 mol/L。 - 用不同浓度的10μL(每孔)激动剂溶液刺激30分钟。
- 使用相关软件(材料表)参数计算酶标仪上的发光信号,如下所示。选择" 发光 "作为 检测方法,选择"端点" 作为 读取类型,选择 "发光光纤 "作为 "光学类型"。将 光学增益 设置为 255。
注意:每次测量在至少三个独立的实验中重复,每个实验一式三份。 - 获取荧光信号的值,将数据导入电子表格程序,并使用非线性回归(曲线拟合)剂量响应函数处理数据。
- 预先制备溶解在DMSO中的10mM西波尼莫德储备溶液,并在配体刺激前使用含有25μM毛喉素的HBSS缓冲液进行连续稀释。
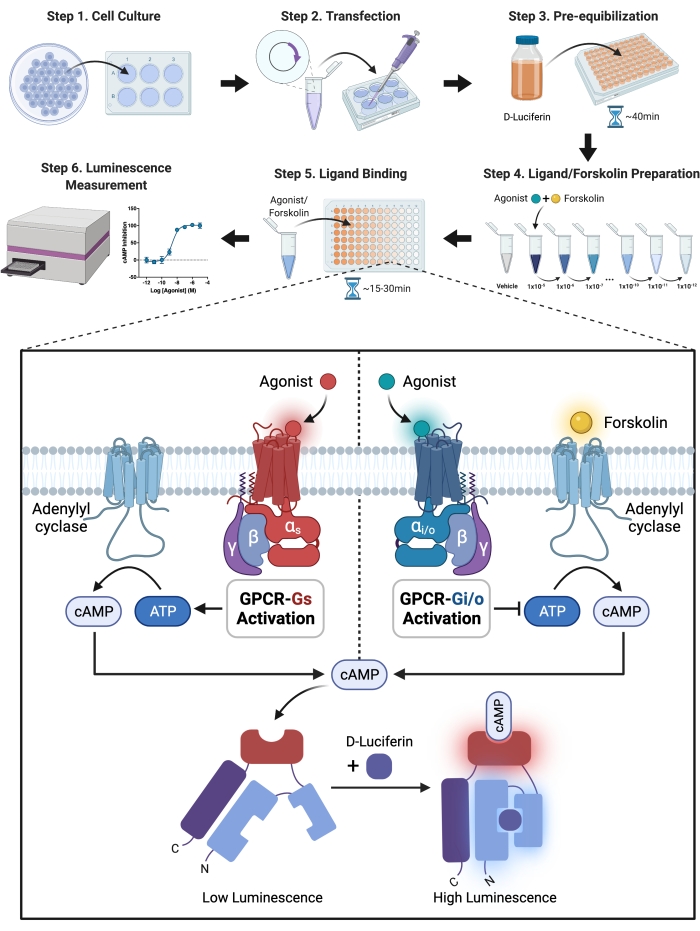
图1:实验示意图。 实验设置和执行的详细分步指南。简而言之,受体和修饰的荧光素酶通过用转染试剂将受体和传感器质粒转染到细胞中,在CHO-K1细胞中短暂共表达。将细胞悬浮在含有D-荧光素 -钾盐(荧光素酶底物)的HBSS溶液中,并在24小时后接种到96孔板中。为了允许渗透到细胞中,D-荧光素必须与细胞预先平衡。氧化酶荧光素酶将荧光素转化为氧化荧光素并发光。另一方面,修饰的荧光素酶仅在与cAMP结合时才 通过 化学反应产生光,并且光的强度与细胞中的cAMP水平呈正相关。cAMP水平由激动剂激活的GPCR调节。Gi偶联受体降低了cAMP的水平,需要在Gi抑制cAMP实验中添加毛喉素来激活腺苷环化酶。 请点击此处查看此图的大图。
结果
在冷冻S1PRs-Gi复合物的样品之前,需要通过尺寸排阻色谱(SEC)分离纯化的样品,并用凝胶过滤色谱进行分析。 图2 显示了以S1PR3-Gi复合物为例。均相GPCR-G蛋白复合物的峰分数通常位于大小排阻色谱的~10.5mL(图2A)。S1PR3-Gi配合物的SDS页面分析(图2B)揭示了对应于S1PR3,Gαi,Gβ和scFv16的理论条带的四个条带,因此表明该配...
讨论
该协议描述了通过冷冻电镜测定S1PR结构的主要管道,并通过Gi介导的cAMP抑制测定法测量S1PR的活化效力。一些步骤对实验的成功至关重要。
为了纯化S1PR-Gi复合物,病毒的质量和 sf9 细胞的健康应该更加关注。受体的表达在较差 的sf9 细胞中显着降低。通过测量 sf9 细胞的直径来评估其健康状况。健康的 sf9 细胞的直径约为15μm。S1PRs-Gi复合物的产量受?...
披露声明
作者没有利益冲突。
致谢
S1PR-Gi复合物的数据在四川大学华西冷冻电镜中心和南方科技大学低温电镜中心采集,并在四川大学杜玉高性能计算中心进行处理。本研究由中国自然科学基金(32100965至LC.C.,32100988至W.Y.,31972916至Z.S.)和四川大学专职博士后研究基金(2021SCU12003至L.C.)资助。
材料
| Name | Company | Catalog Number | Comments |
| 0.05% trypsin-EDTA | GIBCO | Cat# 25300054 | |
| 0.22 µM filter | Thermo Fisher Scientific | Cat# 42213-PS | |
| 100 kDa cut-off concentrator | Thermo Fisher Scientific | Cat# 88533 | |
| 6-well plate | Corning | Cat# 43016 | |
| 96-well plate | Corning | Cat# 3917 | |
| Aprotinin | Sigma-Aldrich | Cat# 9087-70-1 | |
| Apyrase | NEB | Cat# M0398S | |
| Baculovirus transfection reagent | Thermo Fisher Scientific | Cat# 10362100 | For the preparation of P0 baculovirus |
| Benzamidine | Sigma-Aldrich | Cat# B6506 | |
| CHO-K1 | ATCC | N/A | |
| CHS | Sigma-Aldrich | Cat# C6512 | |
| CryoSPARC | Punjani, A., et al.,2017 | https://cryosparc.com/ | |
| DH5α competent E.coli | Thermo Fisher Scientific | Cat# EC0112 | |
| D-Luciferin-Potassium Salt | Sigma- Aldrich | Cat# 50227 | |
| DMSO | Sigma- Aldrich | Cat# D2438 | |
| EDTA | Thermo Fisher Scientific | Cat# S311-500 | |
| ESF921 cell culture medium | Expression Systems | Cat# 96-001 | |
| Excel | microsoft | N/A | |
| F12 medium | Invitrogen | Cat# 11765 | |
| FBS | Cell Box | Cat# SAG-01U-02 | |
| Flag resin | Sigma- Aldrich | Cat# A4596 | |
| Forskolin | APExBIO | Cat# B1421 | |
| Gctf | Zhang, 2016 | https://www.mrc-lmb.cam.ac.uk/kzhang/Gctf/ | |
| GDN | Anatrace | Cat# GDN101 | |
| Gel filtration column | GE healthcare | Cat# 28990944 | |
| Gen5 3.11 | BIO-TEK | N/A | |
| Gentamicin | Solarbio | Cat# L1312 | |
| GloSensor cAMP assay kit | Promega | Cat# E1291 | Gi-inhibition cAMP assay kit |
| GloSensor plasmid | Promega | Cat# E2301 | Sensor plasmid |
| Grace’s medium | GIBCO | Cat# 11595030 | |
| GraphPad Prism 8 | Graphpad | N/A | |
| HBSS | Thermo Fisher Scientific | Cat# 88284 | |
| HEPES | Sigma- Aldrich | Cat# H4034 | |
| jetPRIME Reagent | Polyplus Transfection | Cat# 114-15 | transfection reagent |
| Janamycin | Solarbio | Cat# K1030 | |
| LB medium | Invitrogen | Cat# 12780052 | |
| Leupeptin | Sigma-Aldrich | Cat# L2884 | |
| LMNG | Anatrace | Cat# NG310 | |
| MotionCor2 | (Zheng et al., 2017) | https://emcore.ucsf.edu/ucsf-software | |
| NanoCab | Thermo Fisher Scientific | Cat# 1121822 | |
| PBS | Invitrogen | Cat# 14190-144 | |
| pcDNA3.1-HA-FLAG-S1PRs | GenScript | N/A | |
| pFastBac1-Gαi | GenScript | N/A | |
| pFastBac1-HA-FLAG-T4L-S1PRs-His10 | GenScript | N/A | |
| pFastBacdual-Gβ1γ2 | GenScript | N/A | |
| PureLink HiPure Plasmid Miniprep Kit | Invitrogen | Cat# K210003 | For the preparation of plasmids and P0 baculovirus |
| Q5 site-Directed Mutagenesis kit | NEB | Cat# E0554S | For the preparation of plasmids |
| Quantifoil | Quantifoil | Cat# 251448 | |
| RELION-3.1 | (Zivanov et al., 2018) | https://www2.mrc-lmb.cam.ac.uk/relion | |
| S1PRs cDNA | addgene | N/A | |
| scFv16 | Invitrogen | Cat# 703976 | |
| Sf9 | Expression Systems | N/A | |
| Siponimod | Selleck | Cat# S7179 | |
| sodium cholate | Sigma-Aldrich | Cat# C1254 | |
| Synergy H1 microplate reader | BIO-TEK | N/A | |
| Synthetic T4L DNA (sequence) | N/A | N/A | Aacatcttcgagatgctgcgcatcgacgaagg cctgcgtctcaagatttacaagaataccgaagg ttattacacgattggcatcggccacctcctgaca aagagcccatcactcaacgctgccaagtctga actggacaaagccattggtcgcaacaccaac ggtgtcattacaaaggacgaggcggagaaac tcttcaaccaagatgtagatgcggctgtccgtgg catcctgcgtaatgccaagttgaagcccgtgt atgactcccttgatgctgttcgccgtgcagcctt gatcaacatggttttccaaatgggtgagaccgg agtggctggttttacgaactccctgcgcatgctcc agcagaagcgctgggacgaggccgcagtga atttggctaaatctcgctggtacaatcagacacc taaccgtgccaagcgtgtcatcactaccttccg tactggaacttgggacgcttac |
| TCEP | Thermo Fisher Scientific | Cat# 77720 | |
| Tetracycline | Solarbio | Cat# T8180 | |
| Vitrobot Mark IV | Thermo Fisher Scientific | N/A |
参考文献
- Verstockt, B., et al. Sphingosine 1-phosphate modulation and immune cell trafficking in inflammatory bowel disease. Nature Reviews: Gastroenterology & Hepatology. , 1-16 (2022).
- Rosen, H., Stevens, R. C., Hanson, M., Roberts, E., Oldstone, M. B. Sphingosine-1-phosphate and its receptors: structure, signaling, and influence. Annual Review of Biochemistry. 82, 637-662 (2013).
- Cartier, A., Hla, T. Sphingosine 1-phosphate: Lipid signaling in pathology and therapy. Science. 366 (6463), 5551 (2019).
- Jozefczuk, E., Guzik, T. J., Siedlinski, M. Significance of sphingosine-1-phosphate in cardiovascular physiology and pathology. Pharmacological Research. 156, 104793 (2020).
- Kihara, Y., Maceyka, M., Spiegel, S., Chun, J. Lysophospholipid receptor nomenclature review: IUPHAR Review 8. British Journal of Pharmacology. 171 (15), 3575-3594 (2014).
- Bryan, A. M., Del Poeta, M. Sphingosine-1-phosphate receptors and innate immunity. Cellular Microbiology. 20 (5), 12836 (2018).
- Pelletier, D., Hafler, D. A. Fingolimod for multiple sclerosis. New England Journal of Medicine. 366 (4), 339-347 (2012).
- Obinata, H., Hla, T. Sphingosine 1-phosphate and inflammation. International Immunology. 31 (9), 617-625 (2019).
- Pyne, N. J., Pyne, S. Sphingosine 1-phosphate and cancer. Nature Reviews: Cancer. 10 (7), 489-503 (2010).
- Abu-Farha, M., et al. The role of lipid metabolism in COVID-19 virus infection and as a drug target. International Journal of Molecular Sciences. 21 (10), 3544 (2020).
- Chun, J., Kihara, Y., Jonnalagadda, D., Blaho, V. A. Fingolimod: lessons learned and new opportunities for treating Multiple Sclerosis and other disorders. Annual Review of Pharmacology and Toxicology. 59, 149-170 (2019).
- Murakami, A., et al. Sphingosine 1-phosphate (S1P) regulates vascular contraction via S1P3 receptor: investigation based on a new S1P3 receptor antagonist. Molecular Pharmacology. 77 (4), 704-713 (2010).
- Cao, L., et al. Siponimod for multiple sclerosis. Cochrane Database of Systematic Reviews. 11, (2021).
- Scott, L. J. Siponimod: a review in secondary progressive Multiple Sclerosis. CNS Drugs. 34 (11), 1191-1200 (2020).
- Lamb, Y. N. Ozanimod: first approval. Drugs. 80 (8), 841-848 (2020).
- Scott, F. L., et al. Ozanimod (RPC1063) is a potent sphingosine-1-phosphate receptor-1 (S1P1) and receptor-5 (S1P5) agonist with autoimmune disease-modifying activity. British Journal of Pharmacology. 173 (11), 1778-1792 (2016).
- McGowan, E. M., Haddadi, N., Nassif, N. T., Lin, Y. Targeting the SphK-S1P-SIPR pathway as a potential therapeutic approach for COVID-19. International Journal of Molecular Sciences. 21 (19), 7189 (2020).
- O'Sullivan, C., Dev, K. K. The structure and function of the S1P1 receptor. Trends in Pharmacological Sciences. 34 (7), 401-412 (2013).
- Liao, M., Cao, E., Julius, D., Cheng, Y. Structure of the TRPV1 ion channel determined by electron cryo-microscopy. Nature. 504 (7478), 107-112 (2013).
- Bai, X. C., McMullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- Murata, K., Wolf, M. Cryo-electron microscopy for structural analysis of dynamic biological macromolecules. Biochimica et Biophysica Acta General Subjects. 1862 (2), 324-334 (2018).
- Zhang, M., et al. Cryo-EM structure of an activated GPCR-G protein complex in lipid nanodiscs. Nature Structural & Molecular Biology. 28 (3), 258-267 (2021).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews: Drug Discovery. 17 (7), 471-492 (2018).
- Ishchenko, A., Gati, C., Cherezov, V. Structural biology of G protein-coupled receptors: new opportunities from XFELs and cryoEM. Current Opinion in Structural Biology. 51, 44-52 (2018).
- Yang, D., et al. G protein-coupled receptors: structure- and function-based drug discovery. Signal Transduction and Targeted Therapy. 6 (1), 7 (2021).
- Yuan, Y., et al. Structures of signaling complexes of lipid receptors S1PR1 and S1PR5 reveal mechanisms of activation and drug recognition. Cell Research. 31 (12), 1263-1274 (2021).
- Zhao, C., et al. Structural insights into sphingosine-1-phosphate recognition and ligand selectivity of S1PR3-Gi signaling complexes. Cell Research. 32 (2), 218-221 (2022).
- Xu, Z., et al. Structural basis of sphingosine-1-phosphate receptor 1 activation and biased agonism. Nature Chemical Biology. 18, 281-288 (2022).
- Liu, Y. F., Ghahremani, M. H., Rasenick, M. M., Jakobs, K. H., Albert, P. R. Stimulation of cAMP synthesis by Gi-coupled receptors upon ablation of distinct Galphai protein expression. Gi subtype specificity of the 5-HT1A receptor. Journal of Biological Chemistry. 274 (23), 16444-16450 (1999).
- Buccioni, M., et al. Innovative functional cAMP assay for studying G protein-coupled receptors: application to the pharmacological characterization of GPR17. Purinergic Signalling. 7 (4), 463-468 (2011).
- Wang, F. I., Ding, G., Ng, G. S., Dixon, S. J., Chidiac, P. Luciferase-based GloSensor cAMP assay: Temperature optimization and application to cell-based kinetic studies. Methods. , (2021).
- Audet, M., et al. Small-scale approach for precrystallization screening in GPCR X-ray crystallography. Nature Protocols. 15 (1), 144-160 (2020).
- Sgro, G. G., Costa, T. R. D. Cryo-EM grid preparation of membrane protein samples for single particle analysis. Frontiers in Molecular Biosciences. 5, 74 (2018).
- White, J. B. R., et al. Single particle cryo-electron microscopy: from sample to structure. Journal of Visualized Experiments. (171), e62415 (2021).
- Thompson, R. F., Iadanza, M. G., Hesketh, E. L., Rawson, S., Ranson, N. A. Collection, pre-processing and on-the-fly analysis of data for high-resolution, single-particle cryo-electron microscopy. Nature Protocols. 14 (1), 100-118 (2019).
- Fernandez-Leiro, R., Scheres, S. H. W. A pipeline approach to single-particle processing in RELION. Acta Crystallographica Section D. 73 (6), 496-502 (2017).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14 (3), 290-296 (2017).
- Brilot, A. F., et al. Beam-induced motion of vitrified specimen on holey carbon film. Journal of Structural Biology. 177 (3), 630-637 (2012).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14 (4), 331-332 (2017).
- Zhang, K. Gctf: Real-time CTF determination and correction. Journal of Structural Biology. 193 (1), 1-12 (2016).
- Scheres, S. H. Semi-automated selection of cryo-EM particles in RELION-1.3. Journal of Structural Biology. 189 (2), 114-122 (2015).
- Liu, S., et al. Differential activation mechanisms of lipid GPCRs by lysophosphatidic acid and sphingosine 1-phosphate. Nature Communications. 13 (1), 731 (2022).
- Duan, J., et al. Cryo-EM structure of an activated VIP1 receptor-G protein complex revealed by a NanoBiT tethering strategy. Nature Communications. 11 (1), 4121 (2020).
- DiIorio, M. C., Kulczyk, A. W. A robust single-particle cryo-electron microscopy (cryo-EM) processing workflow with cryoSPARC, RELION, and Scipion. Journal of Visualized Experiments. (179), e63387 (2022).
- Pradelles, P., Grassi, J., Chabardes, D., Guiso, N. Enzyme immunoassays of adenosine cyclic 3',5'-monophosphate and guanosine cyclic 3',5'-monophosphate using acetylcholinesterase. Analytical Chemistry. 61 (5), 447-453 (1989).
- Jiang, L. I., et al. Use of a cAMP BRET sensor to characterize a novel regulation of cAMP by the sphingosine 1-phosphate/G13 pathway. Journal of Biological Chemistry. 282 (14), 10576-10584 (2007).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。