A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
اتباع ديناميات المتغيرات الهيكلية في المجموعات السكانية المتطورة تجريبيا
In This Article
Summary
لقد طورنا طريقة فعالة من حيث التكلفة لمتابعة ديناميكيات أليل تعدد أشكال النوكليوتيدات غير المفردة والتي يمكن تكييفها بسهولة مع أرشيفات التطور التجريبية المجمدة. اقترنت تقنية تفاعل البوليميراز المتسلسل الثلاثي مع الرحلان الكهربائي الشعري المتوازي الآلي لتحديد التردد النسبي لأليل الإدخال على مدار التطور التجريبي.
Abstract
من المعروف الآن أن المتغيرات الهيكلية (SVs) (أي الحذف والإدراج والازدواجية والانقلابات) تلعب دورا مهما في تباين النمط الظاهري ، وبالتالي في عمليات مثل تحديد المرض أو التكيف مع بيئة جديدة. ومع ذلك ، فإن متغيرات النوكليوتيدات المفردة تحظى باهتمام أكبر بكثير من SVs ، ربما لأنها أسهل في اكتشافها ، ومن السهل التنبؤ بآثارها المظهرية. وقد أدى تطوير تكنولوجيات التسلسل العميق القصير والطويل القراءة إلى تحسين الكشف عن التسلسل العميق، ولكن التحديد الكمي لتواترها من بيانات التسلسل المجمع (poolseq) لا يزال معقدا ومكلفا من الناحية التقنية.
هنا ، نقدم طريقة بسيطة وغير مكلفة إلى حد ما ، والتي تسمح للباحثين بمتابعة ديناميات تردد أليل SV. كمثال على التطبيق ، نتبع تكرار إدخال تسلسل الإدراج (IS) في مجموعات التطور التجريبي للبكتيريا. تعتمد هذه الطريقة على تصميم ثلاثة توائم من الاشعال حول حدود المتغير الهيكلي ، بحيث تختلف الأمبليكونات الناتجة عن تضخيم النوع البري (WT) والأليلات المشتقة في الحجم بنسبة 5٪ على الأقل ، وأن كفاءة تضخيمها متشابهة. ثم يتم تحديد كمية كل amplicon عن طريق الكهربائي الشعري المتوازي وتطبيعها إلى منحنى المعايرة. يمكن توسيع هذه الطريقة بسهولة لتشمل التحديد الكمي لتواتر المتغيرات الهيكلية الأخرى (الحذف والازدواجية والانقلاب) ونهج التجميع التسلسلي للسكان الطبيعيين ، بما في ذلك مجموعات مسببات الأمراض داخل المريض.
Introduction
المتغيرات الهيكلية (SVs) هي تغييرات في التسلسل الجيني ، تؤثر بشكل عام على 50 نقطة أساس أو أكثر. الفئات الأربع من SVs الموصوفة هي عمليات إدراج كبيرة وعمليات حذف كبيرة وانعكاسات وازدواجية. حتى وقت قريب ، تم تكريس المزيد من الاهتمام لمتغيرات النوكليوتيدات المفردة (SNVs) مقارنة بالمتغيرات الهيكلية ، من حيث تأثيراتها المظهرية ودورها كمحددات وراثية للمرض ، أو مساهمتها في التكيف. ربما يرجع ذلك إلى أنه من الأسهل اكتشاف SNVs والتنبؤ بآثارها المظهرية. ومع ذلك ، فإن تقنيات التسلسل العميق قصيرة وطويلة القراءة قد حسنت بقوة اكتشاف SVs ، على الأقل في الجينومات الفردية أو النسيلية1. في موازاة ذلك ، تم توصيف آثارها المظهرية بشكل أفضل ، وتم توثيق العديد من الأمثلة على آثارها كمحددات وراثية للأمراض البشرية 2,3 أو التكيف مع بيئة جديدة4.
عمليات الحذف والإدراج ، غالبا بسبب إدخالات العناصر الجينية المتنقلة (MGE) ، أكثر تعطيلا بكثير من تعدد أشكال النوكليوتيدات المفردة (SNPs) وتؤدي إلى طفرات إزاحة الإطار وتعديلات بنية البروتين. يؤدي الحذف وإدخال MGE داخل الجينات دائما تقريبا إلى تعطيل الجينات ، ويمكن أن يؤدي الإدراج في المناطق غير المشفرة إلى قمع أو تعبير تأسيسي للجينات المجاورة عندما تحتوي تسلسلات الإدراج (ISs) على تسلسلات المروج أو الإنهاء5. في حين أن خروج الجينات الأساسية يؤدي إلى آثار ضارة واضحة على اللياقة البكتيرية ، فإن فقدان الجينات غير الأساسية مفيد في بعض الحالات. وعلى الرغم من التكاليف المتأصلة في الازدواجية، فإنها يمكن أن تكون مفيدة أيضا، وتشارك في التكيف لأنها تؤدي إلى تغيير في جرعة الجينات؛ يمكن أن تكون الزيادة في نشاط بروتين معين مفيدة اعتمادا على الظروف6.
عادة ما تبدأ مجموعات التطور التجريبي الميكروبي بالحيوانات المستنسخة. هذا الغياب الأولي للتنوع الجيني ، جنبا إلى جنب مع خاصية "البيئة المغلقة" لأنابيب الاختبار ، يؤدي إلى إمكانات محدودة للغاية للتطور عن طريق اكتساب الجينات من خلال نقل الجينات الأفقية وإعادة التركيب. في هذه الظروف المحددة ، تكون المساهمة في تكييف عمليات الحذف والازدواجية وإدخال MGE داخل الجينوم مهمة بشكل خاص ؛ غالبا ما تتكيف البكتيريا من خلال طفرات فقدان الوظيفة (ويرجع ذلك أساسا إلى عمليات الحذف أو إدخال MGE) ، مما يؤثر على الجينات غير المفيدة في البيئات الاصطناعية المستقرة ، الغنية بالمغذياتفي كثير من الأحيان ، 7. في أطول تجربة لتطور الإشريكية القولونية ، كانت عمليات إدخال IS150 متكررة بشكل خاص بين المجموعات السكانية التي تطورت بعد 50000 جيل ، حيث تمثل عناصر IS 35٪ من الطفرات التي تصل إلى تردد عال في السكان الذين يحتفظون بمعدل طفرة نقطة أسلافهم8.
دراسات التطور وإعادة التسلسل تجمع بين التطور التجريبي وتقنيات التسلسل من الجيل التالي (NGS) للتحقيق في كيفية تكيف البكتيريا ، على المستويين الظاهري والجينومي ، مع الظروف والضغوط البيئية المختلفة ، مثل مصادر الكربون والطاقة المختلفة والمضادات الحيوية والإجهاد التناضحي9،10،11 . تحصل هذه الدراسات عادة على معلومات جينومية عن المجموعات السكانية المتطورة أو الحيوانات المستنسخة فقط عند نقطة النهاية التجريبية ، وفي بعض الحالات ، في عدد من النقاط الزمنية الوسيطة12،13،14. توفر هذه البيانات نظرة ثاقبة للجينات والمسارات المشاركة في التكيف مع بيئة معينة ، ولكنها نادرا ما تسمح للباحثين بمتابعة ديناميكيات الأليلات الناشئة والكاسحة بمرور الوقت.
تتمثل إحدى الطرق التي يجب اتباعها في هذه الديناميكيات في اختيار عدد محدود من الأليلات المنفصلة ذات الأهمية (بسبب وظيفة الجينات التي تؤثر عليها ، لأنها تكتسح بالتوازي في مجموعات سكانية مستقلة ، وما إلى ذلك) واستخدام تسلسل amplicon لتحديد نسبة الأليل ، وتجميع العديد من النقاط الزمنية في نفس تشغيل التسلسل15. تم استخدام هذه الطريقة بنجاح لمتابعة ديناميكيات المتغيرات صغيرة الحجم (SNPs أو 1 bp indels) في16 و17 مجموعة تجريبية من الميكروبات. ومع ذلك ، في حالة إدخالات indels أو MGE الأكبر حجما ، فإن اختلاف حجم الأمبليكونات يؤدي إلى اختلافات كفاءة تفاعل البوليميراز المتسلسل ، مما يشوه العلاقة بين نسب القراءة والأليل . في بعض الحالات ، يكون فرق الحجم بين الأليلين أعلى من الطول الكلاسيكي للأخمبليون. هنا ، قمنا بإقران تقنية تفاعل البوليميراز المتسلسل الثلاثي مع الرحلان الكهربائي الشعري المتوازي الآلي لتحديد التردد النسبي لأليل الإدخال بناء على تمييز الحجم. يسمح هذا النهج باستغلال النقاط الزمنية التجريبية غير المستغلة لتحديد ديناميات أليل طافر ناشئ ومتابعة تواتره للتثبيت أو الفقد ، بطريقة فعالة من حيث التكلفة. طبقنا هذه الطريقة لتتبع الأليلات الطافرة الناشئة ، التي تحورت من خلال إدخال IS10 ، مما يوفر للنمط الجيني المتحور نمطا ظاهريا مفرط التحور.
تتطلب هذه الطريقة أليلين مستهدفين باختلاف ≥5٪ في الحجم. أولا ، تم تصميم ثلاثة توائم أولية لإنتاج شظايا متشابهة الحجم ، والتي تشترك في التمهيدي المشترك. ثانيا ، يتم تحسين ظروف تفاعل البوليميراز المتسلسل ، ويتم إنتاج منحنى معايرة باستخدام مزيج من النوع البري (WT) والحمض النووي الطافر. أخيرا ، يتم تضخيم العينات بواسطة تفاعل البوليميراز المتسلسل ، ويتم قياس التردد النسبي لكل أليل عن طريق الرحلان الكهربائي الشعري الكمي المتوازي.
Protocol
يتطلب إعداد هذا البروتوكول معرفة دقيقة بنقطة الإدراج أو الحذف أو الانعكاس أو الازدواجية داخل تسلسل الأجداد. عادة ما يتم الحصول على هذه المعلومات عن طريق تسلسل الجينوم الكامل (WGS) لعينات النقطة النهائية أو الوسيطة. في البروتوكول التالي ، يتم إعطاء المبدأ العام لحالة طفرة الإدراج لكل خطوة ، جنبا إلى جنب مع حالة تمثيلية حيث يتم اتباع تكرار إدخال IS10 في جين mutS في مجموعة تطور تجريبية من E. coli. في هذه الفئة من السكان، حددت WGS لمجتمع نقطة النهاية إدخال 1,329 bp IS10 بين المواضع 2,463 و 2,471، مما أدى إلى تكرار موقع الإدراج هذا. تنطبق هذه الطريقة على أنواع SV الثلاثة الأخرى ، ويتم إعطاء خصوصيات كل حالة في المناقشة.
1. تصميم الاشعال الثلاثي
- استخدم ممارسات تصميم التمهيدي الكلاسيكية (18-24 نقطة أساس ، 40٪ -60٪ محتوى GC ، ابدأ / تنتهي بأزواج G / C حيثما أمكن ، فرق Tm < 5 درجات مئوية) لإنتاج البادئات FW1 و RV1. صمم مواد أولية لتضخيم أمبليكون قصير على أليل WT حول موقع إدخال الأليل الطافر (الشكل 1).
ملاحظة: يمكن أن يتراوح حجم amplicon من 100 bp إلى ما يصل إلى 3000 bp ، بما يتماشى مع سلم حجم الحمض النووي المستخدم في الرحلان الكهربائي الشعري. في هذا المثال ، تم تضخيم amplicon 155 bp. يمنع حجم الشظية الصغير المختار هنا التضخيم خارج الهدف لتسلسل إدخال IS10 بأكمله (انظر القسم 2). - صمم برايمر أمامي ثان FW2 ضمن تسلسل الإدراج ، لإنتاج أمبليكون ثان أكبر أو أصغر بنسبة 5٪ تقريبا من أمبليكون WT (الشكل 1). هذا الاختلاف في الحجم بنسبة 5٪ هو الحد الأدنى لفرق الحجم الذي يمكن لجهاز الرحلان الكهربائي الشعري المتوازي تمييزه بشكل موثوق. لذلك ، قم بتصميم الاشعال بحيث يكون للمضخمين اختلاف في الحجم ، وهو أعلى ولكن أقرب ما يمكن إلى العتبة النسبية.
ملاحظة: تأكد من تقليل فرق Tm وتشكيل dimer التمهيدي. في هذا المثال ، تم تصميم التمهيدي الأمامي الثاني لإنتاج 226 bp amplicon ، 71 bp أكبر من WT amplicon. في هذا المثال التمثيلي ، تكون تسلسلات التمهيدي كما يلي:
FW1:AAAGCATTTCCCGAACGCC
RV1: GCGATAAATCCACTCCAGCGCC
FW2: AGTTCGCTTAGGCATGGAAG
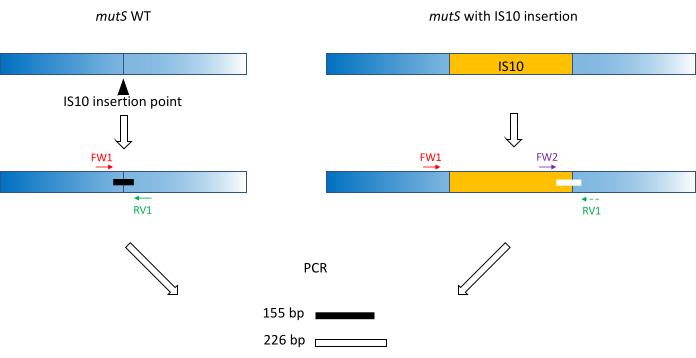
الشكل 1: مخطط تصميم التمهيدي الثلاثي على إدخال mutS WT وإدخال mutS IS10 الطافر. يمثل المثلث الأسود موقع إدخال IS10 في جين mutS. جين WT باللون الأزرق ، و IS10 باللون البرتقالي. تشير البادئات FW1 و RV1 إلى موقع الإدراج IS10 وتنتج مضخم 155 نقطة أساس WT. ينتج التمهيدي RV1 والتمهيدي داخل IS10 FW2 أمبليكون ثان 226 bp. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
2. تحسين ظروف PCR
- تنمو ثقافة بين عشية وضحاها من WT الثابتة واستنساخ أليل متحولة.
- استخراج الحمض النووي باستخدام أي مجموعة.
- تحديد كمية الحمض النووي.
- تحضير عينة من الحمض النووي عن طريق تخفيف استخراج WT والحمض النووي الطافر إلى 5 نانوغرام / ميكرولتر. امزج عينتي الحمض النووي بنسبة 50/50.
- تضخيم 10 نانوغرام من عينات الحمض النووي الثلاثة (WT ، متحولة ، مزيج 50/50) باستخدام مزيج رئيسي PCR جاهز للاستخدام 2x ، و 0.5 μM FW1 التمهيدي ، و 1 μM RV1 التمهيدي ، و 0.5 μM FW2 التمهيدي في حجم تفاعل 20 ميكرولتر. استخدم جل الأغاروز بنسبة 2٪ لترحيل منتج PCR عن طريق الرحلان الكهربائي الكلاسيكي ، وتحديد ظروف PCR المثلى.
ملاحظة: يجب تقليل أوقات الاستطالة لمنع تكوين أمبليكون FW1 و RV1 على الأليل الطافر. يجب ضبط درجة حرارة التلدين لتقليل التضخيم المتحيز للأليلات والتضخيم غير المحدد.- لمتابعة البرنامج في هذا المثال ، استخدم الإعدادات التالية: 98 درجة مئوية لمدة 10 ثوان ، متبوعة ب 25 دورة من 98 درجة مئوية لمدة 1 ثانية ، 58 درجة مئوية لمدة 15 ثانية ، 72 درجة مئوية لمدة 8 ثوان ، وخطوة استطالة نهائية عند 72 درجة مئوية لمدة 1 دقيقة.
ملاحظة: تم تقليل وقت الاستطالة إلى 8 ثوان لمنع تضخيم منتج >1000 bp من التمهيدي الأمامي والتمهيدي RV1 على الأليل الطافر (mutS مع إدخال IS10).
- لمتابعة البرنامج في هذا المثال ، استخدم الإعدادات التالية: 98 درجة مئوية لمدة 10 ثوان ، متبوعة ب 25 دورة من 98 درجة مئوية لمدة 1 ثانية ، 58 درجة مئوية لمدة 15 ثانية ، 72 درجة مئوية لمدة 8 ثوان ، وخطوة استطالة نهائية عند 72 درجة مئوية لمدة 1 دقيقة.
3. منحنى المعايرة
- امزج عينتي الحمض النووي ، WT و mutant ، بنسب 10/90 و 25/75 و 40/60 و 50/50 و 60/40 و 75/25 و 90/10.
ملاحظة: يتم تحضير النسخ المتماثلة البيولوجية من مزارع بكتيرية مستقلة بين عشية وضحاها. - تضخيم باستخدام ظروف PCR المحسنة (انظر القسم 2).
- تحديد كمية منتج الأمبليكون.
- تمييع منتجات PCR إلى 0.1 نانوغرام / ميكرولتر.
- إعداد أداة الكهربائي الشعري المتوازي.
- امزج الجل الطازج والصبغة (مجموعة التحليل الكمي NGS (22 أو 33 أو 55) ؛ جزء HS NGS 1-6000 نقطة أساس لهذا المثال).
ملاحظة: انظر دليل شظية الرحلان الكهربائي الشعري المتوازي HS NGS للحصول على تعليمات مفصلة (انظر جدول المواد).
- امزج الجل الطازج والصبغة (مجموعة التحليل الكمي NGS (22 أو 33 أو 55) ؛ جزء HS NGS 1-6000 نقطة أساس لهذا المثال).
- استبدل محلول التخزين الشعري والمخزن المؤقت للمدخل ، وضع لوحة الشطف العازلة في مواقع الأدراج الصحيحة لأداة الرحلان الكهربائي الشعري المتوازي.
- أضف 2 ميكرولتر من علامة مخفف HS إلى 22 ميكرولتر من كل عينة مخففة في لوحة 96 بئرا.
- أضف سلم حجم (سلم حجم الحمض النووي ؛ النطاق 1-6000 نقطة أساس) من مجموعة التحليل الكمي HS NGS إلى بئر واحد من لوحة 96 بئرا.
- ضع لوحة 96 بئرا في الدرج الصحيح لأداة الرحلان الكهربائي الشعري المتوازي ، وحدد تشغيل على برنامج أداة الرحلان الكهربائي الشعري المتوازي.
- تحليل النتائج باستخدام برنامج تحليل البيانات ، الذي يكتشف ويحدد كل قمة من سلم الحجم ، ويخصص كل قمة من العينات لحجمها الفعلي المعروف.
- عند استخدام مجموعة كمية ، استخدم البرنامج لتحديد كمية الحمض النووي لكل جزء من خلال دمج المنطقة تحت الذروة ، كما هو الحال في تحليل بيانات الكروماتوغرافيا. مرة أخرى ، قارن العينات بكميات معروفة في المعايير لتحديد كل قمة من عينة ، وحساب النسب بين القمم المختلفة المكتشفة في العينات.
- بناء منحنى معايرة (الشكل 2) ، يربط النسبة المعروفة للأليل الطافر (مزيج الحمض النووي) بتلك المقاسة باستخدام أداة الرحلان الكهربائي الشعري المتوازي. يسمح منحنى المعايرة هذا بتقييم موثوقية الطريقة وتصحيحها لتحيز التضخيم الطفيف.
4. إعداد عينة
- تنمو عينات النقطة الزمنية بين عشية وضحاها في الظروف القياسية.
- استخراج الحمض النووي.
- تحديد كمية الحمض النووي.
- تضخيم العينات باستخدام ظروف تفاعل البوليميراز المتسلسل المحسنة (انظر القسم 2).
- قم بتشغيل العينات في أداة الرحلان الكهربائي الشعري المتوازي (انظر الخطوات 3.5-3.10).
5. قياس كمية الأليل
- استخراج كميات الأليل الطافرة من بيانات أداة الرحلان الكهربائي الشعري المتوازي مع البرنامج ، وحساب النسب الفعلية عن طريق رسم هذه القيم على منحنى المعايرة.
النتائج
باستخدام الحمض النووي المستخرج من استنساخ الأسلاف واستنساخ مفرط الحركة المعزول من مجموعة S2.11 عند الجيل 1000 ، أنشأنا منحنى المعايرة الموضح في الشكل 2. تم ربط النسب الطافرة الفعلية من خلطات الحمض النووي المعدة في المختبر والمقاسة بواسطة أداة الرحلان الكهربائي الشعري المتواز...
Discussion
هنا ، اقترحنا طريقة فعالة من حيث التكلفة تسمح باتباع ديناميكيات أليلات SV التكيفية الناشئة في مجموعات التطور التجريبية. تجمع هذه الطريقة بين تقنيات تفاعل البوليميراز المتسلسل الكلاسيكية والرحلان الكهربائي الشعري المتوازي الآلي ، مما يسمح بتحديد الكميات النسبية لألينتين. بمجرد إعداده ، ?...
Disclosures
ليس لدى المؤلفين أي تضارب في المصالح للكشف عنه.
Acknowledgements
تم دعم هذا العمل من قبل ERC HGTCODONUSE (ERC-2015-CoG-682819) إلى S.B. تم إنتاج البيانات المستخدمة في هذا العمل (جزئيا) من خلال المرافق الفنية GenSeq التابعة لمعهد علوم التطور في مونبلييه بدعم من LabEx CeMEB ، وهو برنامج ANR "استثمارات المستقبل" (ANR-10-LABX-04-01).
Materials
| Name | Company | Catalog Number | Comments |
| 96 Well Skirted PCR Plate | 4titude | 4Ti - 0740 | PCR |
| Agarose molecular biology grade | Eurogentec | EP-0010-05 | Agarose gel electrophoresis |
| Agilent DNF-474 HS NGS Fragment Kit Quick Guide for the Fragment Analyzer Systems | Agilent | PDF instruction guide | |
| Buffer TBE | Panreac appliChem | A4228,5000Pc | Agarose gel electrophoresis |
| Calibrated Disposable Inoculating Loops and Needles | LABELIANS | 8175CSR40H | Bacterial culture |
| Dneasy Blood and Tissue Kit | Qiagen | 69506 | DNA extraction |
| Electrophoresis power supply | Amilabo | ST606T | Agarose gel electrophoresis |
| Fragment Analyzer Automated CE System | Agilent | Parallel capillary electrophoresis | |
| Fragment DNA Ladder | Agilent | DNF-396, range 1-6000bp | Parallel capillary electrophoresis |
| GENTAMICIN SULFATE SALT BIOREAGENT | Sigma-Aldrich | G1264-1G | Bacterial culture |
| High Sensitivity diluent marker | Agilent | DNF-373 | Parallel capillary electrophoresis |
| High Sensitivity NGS quantitative analysis kit | Agilent | DNF-474 | Parallel capillary electrophoresis |
| Ladder quick load 1 kb plus DNA ladder | NEB | N0469S | Agarose gel electrophoresis |
| LB Broth, VegitoneNutriSelect Plus | Millipore | 28713 | Bacterial culture |
| Master Mix PCR High Fidelity Phusion Flash | Thermo Fisher Scientific | F548L | PCR |
| Primers | Eurogentec | PCR | |
| Prosize data analysis software v.4 | Agilent | V.4 | Parallel capillary electrophoresis |
| Qubit assays | Invitrogen | MAN0010876 | DNA quantification |
| Qubit dsDNA HS Assay Kit | LIFE TECHNOLOGIES SAS | Q32854 | DNA quantification |
| Thermocycler | Eppendorf | Ep gradients | PCR |
| UVbox, eBOX VX5 | Vilber Lourmat | Agarose gel electrophoresis visualisation | |
| Water for injectable preparation | Aguettant | PROAMP | PCR |
References
- Mahmoud, M., et al. Structural variant calling: the long and the short of it. Genome Biology. 20 (1), 246 (2019).
- Bragg, D. C., et al. Disease onset in X-linked dystonia-parkinsonism correlates with expansion of a hexameric repeat within an SVA retrotransposon in TAF1. Proceedings of the National Academy of Sciences. 114 (51), 11020-11028 (2017).
- Stransky, N., Cerami, E., Schalm, S., Kim, J. L., Lengauer, C. The landscape of kinase fusions in cancer. Nature Communications. 5, 4846 (2014).
- Tenaillon, O., et al. The molecular diversity of adaptive convergence. Science. 335 (6067), 457-461 (2012).
- Vandecraen, J., Chandler, M., Aertsen, A., Van Houdt, R. The impact of insertion sequences on bacterial genome plasticity and adaptability. Critical Reviews in Microbiology. 43 (6), 709-730 (2017).
- Andersson, D. I., Gene Hughes, D. amplification and adaptive evolution in bacteria. Annual Review of Genetics. 43, 167-195 (2009).
- Bailey, S. F., Bataillon, T. Can the experimental evolution programme help us elucidate the genetic basis of adaptation in nature. Molecular Ecology. 25 (1), 203-218 (2016).
- Consuegra, J., et al. Insertion-sequence-mediated mutations both promote and constrain evolvability during a long-term experiment with bacteria. Nature Communications. 12 (1), 980 (2021).
- Burch, C. L., Romanchuk, A., Kelly, M., Wu, Y., Jones, C. D. Genome-wide determination of barriers to horizontal gene transfer. bioRxiv. , (2022).
- Slomka, S., et al. Experimental evolution of Bacillus subtilis reveals the evolutionary dynamics of horizontal gene transfer and suggests adaptive and neutral effects. Genetics. 216 (2), 543-558 (2020).
- Choudhury, D., Saini, S. Evolution of Escherichia coli in different carbon environments for 2,000 generations. Journal of Evolutionary Biology. 32 (12), 1331-1341 (2019).
- Tenaillon, O., et al. Tempo and mode of genome evolution in a 50,000-generation experiment. Nature. 536 (7615), 165-170 (2016).
- Behringer, M. G., et al. Escherichiacoli cultures maintain stable subpopulation structure during long-term evolution. Proceedings of the National Academy of Sciences. 115 (20), 4642-4650 (2018).
- Voordeckers, K., et al. Adaptation to high ethanol reveals complex evolutionary pathways. PLoS Genetics. 11 (11), 1005635 (2015).
- Levy, S. F., et al. Quantitative evolutionary dynamics using high-resolution lineage tracking. Nature. 519 (7542), 181-186 (2015).
- Bruger, E. L., Marx, C. J. A decade of genome sequencing has revolutionized studies of experimental evolution. Current Opinion in Microbiology. 45, 149-155 (2018).
- Grubaugh, N. D., et al. An amplicon-based sequencing framework for accurately measuring intrahost virus diversity using PrimalSeq and iVar. Genome Biology. 20 (1), 8 (2019).
- Bedhomme, S., et al. Evolutionary changes after translational challenges imposed by horizontal gene transfer. Genome Biology and Evolution. 11 (3), 814-831 (2019).
- Tenaillon, O., Toupance, B., Le Nagard, H., Taddei, F., Godelle, B. Mutators, population size, adaptive landscape and the adaptation of asexual populations of bacteria. Genetics. 152 (2), 485-493 (1999).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved