Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Deneysel Olarak Evrimleşmiş Popülasyonlarda Yapısal Varyantların Dinamiklerini Takip Etmek
Bu Makalede
Özet
Deneysel evrim dondurulmuş arşivlerine kolayca uyarlanabilen tek nükleotid olmayan polimorfizm alel dinamiklerini takip etmek için uygun maliyetli bir yöntem geliştirdik. Üçlü PCR tekniği, deneysel evrim boyunca bir yerleştirme alelinin göreceli frekansını ölçmek için otomatik paralel kılcal elektroforez ile birleştirildi.
Özet
Yapısal varyantların (SV'ler) (yani, silmeler, eklemeler, duplikasyonlar ve inversiyonlar) artık fenotipik varyasyonda ve dolayısıyla hastalık belirleme veya yeni bir ortama adaptasyon gibi süreçlerde önemli bir rol oynadığı bilinmektedir. Bununla birlikte, tek nükleotid varyantları SV'lerden çok daha fazla dikkat çekmektedir, çünkü muhtemelen tespit edilmeleri daha kolaydır ve fenotipik etkilerinin tahmin edilmesi daha kolaydır. Kısa ve uzun okunan derin dizileme teknolojilerinin geliştirilmesi, SV'lerin algılanmasını güçlü bir şekilde geliştirmiştir, ancak havuzlanmış sıralama (poolseq) verilerinden frekanslarının ölçülmesi hala teknik olarak karmaşık ve pahalıdır.
Burada, araştırmacıların SV alel frekansının dinamiklerini takip etmelerini sağlayan oldukça basit ve ucuz bir yöntem sunuyoruz. Bir uygulama örneği olarak, bakterilerin deneysel evrim popülasyonlarında bir ekleme dizisi (IS) ekleme sıklığını takip ediyoruz. Bu yöntem, yapısal varyant sınırlarının etrafındaki primer üçlülerinin tasarımına dayanır, böylece vahşi tip (WT) ve türetilmiş alellerin amplifikasyonu ile üretilen amplikonların boyut olarak en az% 5 oranında farklılık göstermesi ve amplifikasyon verimliliklerinin benzer olması gerekir. Her amplikonun miktarı daha sonra paralel kılcal elektroforez ile belirlenir ve bir kalibrasyon eğrisine normalleştirilir. Bu yöntem, diğer yapısal varyantların (delesyonlar, duplikasyonlar ve inversiyonlar) sıklığının nicelleştirilmesine ve hasta içi patojen popülasyonları da dahil olmak üzere doğal popülasyonların havuz-seq yaklaşımlarına kolayca genişletilebilir.
Giriş
Yapısal varyantlar (SV'ler), genellikle 50 bp veya daha fazlasını etkileyen genomik sekansın değişiklikleridir. Açıklanan SV'lerin dört kategorisi büyük eklemeler, büyük silmeler, ters çevirmeler ve çoğaltmalardır. Yakın zamana kadar, tek nükleotid varyantlarına (SNV'ler), fenotipik etkileri ve hastalığın genetik belirleyicileri olarak rolleri veya adaptasyona katkıları açısından yapısal varyantlardan daha fazla dikkat çekilmiştir. Bunun nedeni muhtemelen SNV'leri tespit etmenin ve fenotipik etkilerini tahmin etmenin daha kolay olmasıdır. Bununla birlikte, kısa ve uzun okunan derin dizileme teknolojileri, en azından tek bir bireyde veya klonal genomlardaSV'lerin tespitini güçlü bir şekilde geliştirmiştir 1. Buna paralel olarak, fenotipik etkileri daha iyi karakterize edilmiş ve insan hastalığının genetik belirleyicileri 2,3 veya yeni bir çevreye adaptasyon4 olarak ima edildiklerine dair birçok örnek belgelenmiştir.
Genellikle mobil genetik element (MGE) yerleştirmelerine bağlı olarak delesyonlar ve eklemeler, tek nükleotid polimorfizmlerinden (SNP'ler) çok daha yıkıcıdır ve çerçeve kayması mutasyonlarına ve protein yapısı modifikasyonlarına yol açar. Genlerdeki delesyonlar ve MGE yerleştirmeleri hemen hemen her zaman gen inaktivasyonu ile sonuçlanır ve kodlamayan bölgelere yapılan eklemeler, ekleme dizileri (IS'ler) promotör veya sonlandırma dizileriiçerdiğinde bitişik genlerin baskılanmasına veya kurucu ekspresyonuna yol açabilir 5. Esansiyel genlerin nakavtlanması, bakteriyel uygunluk üzerinde açık zararlı etkilere yol açarken, esansiyel olmayan genlerin kaybı bazı durumlarda faydalıdır. Doğal maliyetlerine rağmen, duplikasyonlar da avantajlı olabilir ve gen dozajında bir değişikliğe yol açtıkları için adaptasyona katılabilirler; Belirli bir proteinin aktivitesindeki bir artış, koşullara bağlı olarak avantajlı olabilir6.
Mikrobiyal deneysel evrim popülasyonları genellikle klonlarla başlar. Genetik çeşitliliğin bu başlangıçtaki yokluğu, test tüplerinin "kapalı çevre" özelliği ile birleştiğinde, yatay gen transferi ve rekombinasyonu yoluyla gen kazanımı yoluyla çok sınırlı bir evrim potansiyeline yol açar. Bu spesifik koşullarda, delesyonların, duplikasyonların ve intragenomik MGE yerleştirilmesinin adaptasyonuna katkısı özellikle önemlidir; bakteriler genellikle fonksiyon kaybı mutasyonları yoluyla adapte olurlar (esas olarak delesyonlar veya MGE eklemeleri nedeniyle), kararlı, genellikle besin açısından zengin, monokültür yapay ortamlarda yararlı olmayan genleri etkiler7. En uzun süredir devam eden E. coli evrim deneyinde, IS150 yerleştirmeleri, 50.000 kuşaktan sonra evrimleşen popülasyonlar arasında özellikle sık görülür ve IS elementleri, atalarının nokta mutasyon oranını koruyan popülasyonlarda yüksek frekansa ulaşan mutasyonların% 35'ini temsil eder8.
Bakterilerin fenotipik ve genomik seviyelerde, farklı karbon ve enerji kaynakları, antibiyotikler ve ozmotik stres gibi farklı çevresel koşullara ve streslere nasıl adapte olduklarını araştırmak için deneysel evrimi ve yeni nesil dizileme (NGS) teknolojilerini birleştiren evrimleşme ve yeniden sıralama çalışmaları 9,10,11 . Bu çalışmalar tipik olarak evrimleşmiş popülasyonlar veya klonlar hakkında genomik bilgiyi yalnızca deneysel bitiş noktasında ve bazı durumlarda 12,13,14 numaralı ara zaman noktalarında elde eder. Bu veriler, belirli bir ortama adaptasyonda yer alan genler ve yollar hakkında fikir verir, ancak nadiren araştırmacıların zaman içinde ortaya çıkan ve süpürülen de novo alellerin dinamiklerini takip etmelerine izin verir.
Bu dinamikleri takip etmek için bir yaklaşım, sınırlı sayıda ayırıcı alel seçmek (etkiledikleri genlerin işlevi nedeniyle, bağımsız popülasyonlarda paralel olarak süpürüldükleri için, vb.) ve alel oranını ölçmek için amplikon dizilimini kullanmak, aynı dizileme koşusunda birçok zaman noktasını bir araya getirmektir15. Bu yöntem, deneysel16 ve doğal17 mikrop popülasyonunda küçük boyutlu varyantların (SNP'ler veya 1 bp indel) dinamiklerini takip etmek için başarıyla kullanılmıştır. Bununla birlikte, daha büyük indeller veya MGE yerleştirmeleri durumunda, amplikonların boyut farkı, okuma ve alel oranları arasındaki ilişkiyi bozan PCR verimlilik farklılıklarını indükler. Bazı durumlarda, iki alel arasındaki boyut farkı, amplikonun klasik uzunluğundan daha üstündür. Burada, boyut ayrımına dayalı bir yerleştirme alelinin göreceli frekansını ölçmek için üçlü bir PCR tekniğini otomatik paralel kılcal elektroforez ile birleştirdik. Bu yaklaşım, ortaya çıkan bir mutant alelin dinamiklerini belirlemek ve fiksasyon veya kayıp sıklığını uygun maliyetli bir şekilde takip etmek için az kullanılan deneysel zaman noktalarının kullanılmasına izin verir. Bu yöntemi, bir IS10 yerleştirme yoluyla mutasyona uğramış ve mutasyona uğramış genotipe hipermutator bir fenotip sağlayan ortaya çıkan mutS- alellerini izlemek için uyguladık.
Bu yöntem, boyut olarak% ≥5'lik bir farka sahip iki hedef alel gerektirir. İlk olarak, astar üçlüleri, ortak bir astarı paylaşan benzer büyüklükteki parçalar üretmek üzere tasarlanmıştır. İkincisi, PCR koşulları optimize edilir ve vahşi tip (WT) ve mutant gDNA karışımları kullanılarak bir kalibrasyon eğrisi üretilir. Son olarak, örnekler PCR ile güçlendirilir ve her alelin göreceli frekansı paralel kantitatif kılcal elektroforez ile ölçülür.
Protokol
Bu protokolün kurulması, atasal dizi içindeki ekleme, silme, ters çevirme veya çoğaltma noktası hakkında kesin bilgi gerektirir. Bu bilgi genellikle uç veya ara nokta örneklerinin tüm genom dizilimi (WGS) ile elde edilir. Aşağıdaki protokolde, her adım için bir yerleştirme mutasyonu vakası için genel prensip, deneysel bir E. coli popülasyonunda mutS genindeki bir IS10 yerleştirme sıklığının izlendiği temsili bir vaka ile birlikte verilmiştir. Bu popülasyonda, uç nokta popülasyonunun WGS'si, 2.463 ve 2.471 pozisyonları arasına 1.329 bp IS10'un yerleştirilmesini tanımladı ve bu ekleme bölgesinin çoğaltılmasıyla sonuçlandı. Bu yöntem diğer üç SV tipi için geçerlidir ve her bir vakanın özellikleri tartışmada verilmiştir.
1. Üçlü astarların tasarımı
- FW1 ve RV1 astarları üretmek için klasik astar tasarım uygulamalarını (18-24 bp, %40-%60 GC içeriği, mümkün olduğunda G/C çiftleriyle başlangıç/bitiş, Tm farkı < 5 °C) kullanın. WT alelinde mutant alel yerleştirme bölgesi etrafındaki kısa bir amplikonu yükseltmek için primerler tasarlayın (Şekil 1).
NOT: Amplikon boyutu, kılcal elektroforezde kullanılan DNA boyutu merdivenine paralel olarak 100 bp ila 3.000 bp arasında değişebilir. Bu örnekte, 155 bp amplikon yükseltilmiştir. Burada seçilen küçük parça boyutu, tüm IS10 yerleştirme dizisinin hedef dışı amplifikasyonunu önler (bkz. bölüm 2). - WT amplikondan yaklaşık %5 daha büyük veya daha küçük ikinci bir amplikon üretmek için yerleştirme sırası içinde ikinci bir ileri astar FW2 tasarlayın (Şekil 1). Bu %5'lik boyut farkı, paralel kılcal elektroforez cihazının güvenilir bir şekilde ayırt edebileceği minimum boyut farkıdır. Bu nedenle, primerleri, iki amplikonun boyut olarak yukarıda ancak göreceli eşiğe mümkün olduğunca yakın olan bir boyut farkına sahip olacak şekilde tasarlayın.
NOT: Tm farkını ve astar dimer oluşumunu en aza indirdiğinizden emin olun. Bu örnekte, ikinci bir ileri astar, WT amplikonundan 71 bp daha büyük olan 226 bp amplikon üretmek üzere tasarlanmıştır. Bu temsili örnekte, astar dizileri aşağıdaki gibidir:
FW1:AAAGCATTTCGCCGAACGCC
RV1: GCGATAAATCCACTCCAGCGCC
FW2: AGTTCGCTTAGGCATGGAAG
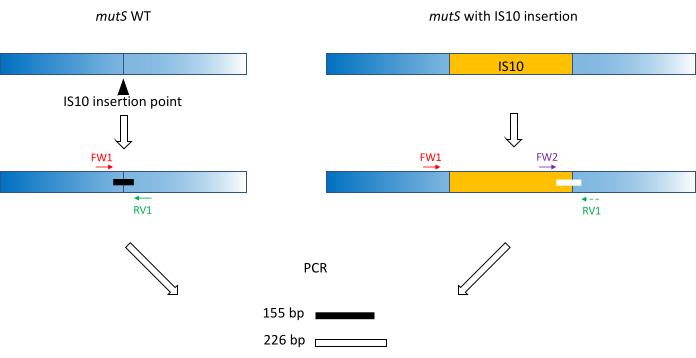
Şekil 1: mutS WT geni ve mutS IS10 eklenmesi üzerine üçlü astar tasarımının şeması. Siyah üçgen, mutS genindeki IS10 yerleştirme bölgesini temsil eder. WT geni mavi, IS10 ise turuncu renktedir. Astarlar FW1 ve RV1, IS10 yerleştirme bölgesini işaretler ve 155 bp WT amplikon üretir. RV1 astarı ve IS10 içi astar FW2, ikinci bir 226 bp amplikon üretir. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
2. PCR koşullarının optimizasyonu
- Bir gecede sabit WT ve mutant alel klonlarından oluşan bir kültür yetiştirin.
- Herhangi bir kit kullanarak DNA'yı çıkarın.
- DNA'yı sayısallaştırın.
- WT ve mutant DNA'nın ekstraksiyonunu 5 ng / μL'ye seyrelterek DNA örneğini hazırlayın İki DNA örneğini 50/50 oranında karıştırın.
- 20 μL reaksiyon hacminde 2x kullanıma hazır PCR ana karışımı, 0,5 μM FW1 astar, 1 μM RV1 astar ve 0,5 μM FW2 astarı kullanarak üç DNA örneğinin (WT, mutant, 50/50 karışımı) 10 ng'sini yükseltin. PCR ürününü klasik elektroforez ile migrasyon yapmak ve optimal PCR koşullarını belirlemek için %2'lik bir agaroz jeli kullanın.
NOT: Mutant alel üzerinde FW1 ve RV1 amplikon oluşumunu önlemek için uzama süreleri en aza indirilmelidir. Tavlama sıcaklığı, alellerin önyargılı amplifikasyonunu ve spesifik olmayan amplifikasyonu en aza indirgemek için ayarlanmalıdır.- Bu örnekteki programı takip etmek için aşağıdaki ayarları kullanın: 10 s için 98 °C, ardından 1 s için 98 °C'lik 25 döngü, 15 s için 58 °C, 8 s için 72 °C ve 1 dakika boyunca 72 °C'de son bir uzama adımı.
NOT: Uzama süresi, >1.000 bp ürünün ileri astardan ve RV1 astarının mutant alel (IS10 yerleştirmeli mutS ) üzerindeki amplifikasyonunu önlemek için 8 sn'ye düşürülmüştür.
- Bu örnekteki programı takip etmek için aşağıdaki ayarları kullanın: 10 s için 98 °C, ardından 1 s için 98 °C'lik 25 döngü, 15 s için 58 °C, 8 s için 72 °C ve 1 dakika boyunca 72 °C'de son bir uzama adımı.
3. Kalibrasyon eğrisi
- İki DNA örneğini, WT ve mutant, 10/90, 25/75, 40/60, 50/50, 60/40, 75/25 ve 90/10 oranlarında karıştırın.
NOT: Biyolojik replikalar bağımsız gece bakteri kültürlerinden hazırlanır. - Optimize edilmiş PCR koşullarını kullanarak güçlendirin (bkz. bölüm 2).
- Amplikon ürününü sayısallaştırın.
- PCR ürünlerini 0,1 ng/μL'ye seyreltin.
- Paralel kılcal elektroforez cihazını hazırlayın.
- Taze jel ve boyayı karıştırın (NGS kantitatif analiz kiti (22, 33 veya 55); HS NGS fragmanı bu örnek için 1-6.000 bp).
NOT: Ayrıntılı talimatlar için paralel kılcal elektroforez HS NGS parça kılavuzuna bakın ( Malzeme Tablosuna bakınız).
- Taze jel ve boyayı karıştırın (NGS kantitatif analiz kiti (22, 33 veya 55); HS NGS fragmanı bu örnek için 1-6.000 bp).
- Kılcal saklama solüsyonunu ve giriş tamponunu değiştirin ve durulama tampon plakasını paralel kılcal elektroforez cihazının doğru çekmece yerlerine yerleştirin.
- 96 delikli bir plakada seyreltilmiş her numunenin 22 μL'sine 2 μL HS seyreltici işaretleyici ekleyin.
- HS NGS kantitatif analiz kitinden 96 delikli plakanın bir kuyucuğuna bir boyut merdiveni (DNA boyutu merdiveni; aralık 1-6.000 bp) ekleyin.
- 96 delikli plakayı paralel kılcal elektroforez cihazının doğru çekmecesine yerleştirin ve paralel kılcal elektroforez cihazı yazılımında Çalıştır'ı seçin.
- Boyut merdiveninin her bir tepe noktasını algılayan ve tanımlayan, örneklerin her bir tepe noktasını bilinen gerçek boyutlarına atayan veri analiz yazılımını kullanarak sonuçları analiz edin.
- Kantitatif bir kit kullanırken, kromatografi veri analizinde olduğu gibi, alanı zirvenin altına entegre ederek her bir parçanın DNA miktarını belirlemek için yazılımı kullanın. Yine, bir numunedeki her bir tepe noktasını ölçmek için numuneleri standartlarda bilinen miktarlarla karşılaştırın ve numunelerde tespit edilen farklı pikler arasındaki oranları hesaplayın.
- Mutant alelin (DNA karışımı) bilinen oranını paralel kılcal elektroforez cihazı kullanılarak ölçülene bağlayan bir kalibrasyon eğrisi oluşturun (Şekil 2). Bu kalibrasyon eğrisi, metodun güvenilirliğinin küçük amplifikasyon yanlılığı için değerlendirilmesini ve düzeltilmesini sağlar.
4. Numune hazırlama
- Zaman noktası örneklerini standart koşullarda bir gecede büyütün.
- DNA'yı çıkarın.
- DNA'yı sayısallaştırın.
- Optimize edilmiş PCR koşullarını kullanarak numuneleri güçlendirin (bkz. bölüm 2).
- Numuneleri paralel kılcal elektroforez cihazında çalıştırın (bkz. adım 3.5-3.10).
5. Alel ölçümü
- Yazılım ile paralel kılcal elektroforez cihazı verilerinden mutant alel miktarlarını çıkarın ve bu değerleri kalibrasyon eğrisinde çizerek gerçek oranları hesaplayın.
Sonuçlar
Bir atasal klondan ekstrakte edilen DNA'yı ve 1000. nesildeki S2.11 popülasyonundan izole edilen bir hipermutatör klonu kullanarak, Şekil 2'de gösterilen kalibrasyon eğrisini belirledik. Laboratuvarda hazırlanan DNA karışımlarından elde edilen ve paralel kılcal elektroforez cihazı ile ölçülen gerçek mutant oranlar, 0.9705'lik bir R2 ile 1.0706 eğiminin doğrusal bir ilişkisi ile bağlandı. Ek olarak, biyolojik replikalar arasında iyi bir anlaşma vardı; Stand...
Tartışmalar
Burada, deneysel evrim popülasyonlarında ortaya çıkan adaptif SV alellerinin dinamiklerinin izlenmesini sağlayan uygun maliyetli bir yöntem önerdik. Bu yöntem, klasik PCR tekniklerini ve otomatik paralel kılcal elektroforezi birleştirerek, iki alelin göreceli miktarlarının belirlenmesini sağlar. Bir kez kurulduktan sonra, birçok numunedeki alel oranlarının paralel olarak ölçülmesine izin verir ve WGS'den çok daha ucuzdur. Bu yöntem, SNP dışı mutasyonlar için amplikon dizilemesine eşdeğer ve b?...
Açıklamalar
Yazarların açıklayacak çıkar çatışmaları yoktur.
Teşekkürler
Bu çalışma ERC HGTCODONUSE (ERC-2015-CoG-682819) tarafından S.B.'ye desteklenmiştir. Bu çalışmada kullanılan veriler, bir ANR "Investissements d'avenir" programı olan LabEx CeMEB'in (ANR-10-LABX-04-01) desteğiyle Institut des Sciences de l'Evolution de Montpellier'in GenSeq teknik tesisleri aracılığıyla (kısmen) üretilmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 96 Well Skirted PCR Plate | 4titude | 4Ti - 0740 | PCR |
| Agarose molecular biology grade | Eurogentec | EP-0010-05 | Agarose gel electrophoresis |
| Agilent DNF-474 HS NGS Fragment Kit Quick Guide for the Fragment Analyzer Systems | Agilent | PDF instruction guide | |
| Buffer TBE | Panreac appliChem | A4228,5000Pc | Agarose gel electrophoresis |
| Calibrated Disposable Inoculating Loops and Needles | LABELIANS | 8175CSR40H | Bacterial culture |
| Dneasy Blood and Tissue Kit | Qiagen | 69506 | DNA extraction |
| Electrophoresis power supply | Amilabo | ST606T | Agarose gel electrophoresis |
| Fragment Analyzer Automated CE System | Agilent | Parallel capillary electrophoresis | |
| Fragment DNA Ladder | Agilent | DNF-396, range 1-6000bp | Parallel capillary electrophoresis |
| GENTAMICIN SULFATE SALT BIOREAGENT | Sigma-Aldrich | G1264-1G | Bacterial culture |
| High Sensitivity diluent marker | Agilent | DNF-373 | Parallel capillary electrophoresis |
| High Sensitivity NGS quantitative analysis kit | Agilent | DNF-474 | Parallel capillary electrophoresis |
| Ladder quick load 1 kb plus DNA ladder | NEB | N0469S | Agarose gel electrophoresis |
| LB Broth, VegitoneNutriSelect Plus | Millipore | 28713 | Bacterial culture |
| Master Mix PCR High Fidelity Phusion Flash | Thermo Fisher Scientific | F548L | PCR |
| Primers | Eurogentec | PCR | |
| Prosize data analysis software v.4 | Agilent | V.4 | Parallel capillary electrophoresis |
| Qubit assays | Invitrogen | MAN0010876 | DNA quantification |
| Qubit dsDNA HS Assay Kit | LIFE TECHNOLOGIES SAS | Q32854 | DNA quantification |
| Thermocycler | Eppendorf | Ep gradients | PCR |
| UVbox, eBOX VX5 | Vilber Lourmat | Agarose gel electrophoresis visualisation | |
| Water for injectable preparation | Aguettant | PROAMP | PCR |
Referanslar
- Mahmoud, M., et al. Structural variant calling: the long and the short of it. Genome Biology. 20 (1), 246 (2019).
- Bragg, D. C., et al. Disease onset in X-linked dystonia-parkinsonism correlates with expansion of a hexameric repeat within an SVA retrotransposon in TAF1. Proceedings of the National Academy of Sciences. 114 (51), 11020-11028 (2017).
- Stransky, N., Cerami, E., Schalm, S., Kim, J. L., Lengauer, C. The landscape of kinase fusions in cancer. Nature Communications. 5, 4846 (2014).
- Tenaillon, O., et al. The molecular diversity of adaptive convergence. Science. 335 (6067), 457-461 (2012).
- Vandecraen, J., Chandler, M., Aertsen, A., Van Houdt, R. The impact of insertion sequences on bacterial genome plasticity and adaptability. Critical Reviews in Microbiology. 43 (6), 709-730 (2017).
- Andersson, D. I., Gene Hughes, D. amplification and adaptive evolution in bacteria. Annual Review of Genetics. 43, 167-195 (2009).
- Bailey, S. F., Bataillon, T. Can the experimental evolution programme help us elucidate the genetic basis of adaptation in nature. Molecular Ecology. 25 (1), 203-218 (2016).
- Consuegra, J., et al. Insertion-sequence-mediated mutations both promote and constrain evolvability during a long-term experiment with bacteria. Nature Communications. 12 (1), 980 (2021).
- Burch, C. L., Romanchuk, A., Kelly, M., Wu, Y., Jones, C. D. Genome-wide determination of barriers to horizontal gene transfer. bioRxiv. , (2022).
- Slomka, S., et al. Experimental evolution of Bacillus subtilis reveals the evolutionary dynamics of horizontal gene transfer and suggests adaptive and neutral effects. Genetics. 216 (2), 543-558 (2020).
- Choudhury, D., Saini, S. Evolution of Escherichia coli in different carbon environments for 2,000 generations. Journal of Evolutionary Biology. 32 (12), 1331-1341 (2019).
- Tenaillon, O., et al. Tempo and mode of genome evolution in a 50,000-generation experiment. Nature. 536 (7615), 165-170 (2016).
- Behringer, M. G., et al. Escherichiacoli cultures maintain stable subpopulation structure during long-term evolution. Proceedings of the National Academy of Sciences. 115 (20), 4642-4650 (2018).
- Voordeckers, K., et al. Adaptation to high ethanol reveals complex evolutionary pathways. PLoS Genetics. 11 (11), 1005635 (2015).
- Levy, S. F., et al. Quantitative evolutionary dynamics using high-resolution lineage tracking. Nature. 519 (7542), 181-186 (2015).
- Bruger, E. L., Marx, C. J. A decade of genome sequencing has revolutionized studies of experimental evolution. Current Opinion in Microbiology. 45, 149-155 (2018).
- Grubaugh, N. D., et al. An amplicon-based sequencing framework for accurately measuring intrahost virus diversity using PrimalSeq and iVar. Genome Biology. 20 (1), 8 (2019).
- Bedhomme, S., et al. Evolutionary changes after translational challenges imposed by horizontal gene transfer. Genome Biology and Evolution. 11 (3), 814-831 (2019).
- Tenaillon, O., Toupance, B., Le Nagard, H., Taddei, F., Godelle, B. Mutators, population size, adaptive landscape and the adaptation of asexual populations of bacteria. Genetics. 152 (2), 485-493 (1999).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır