需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
二维和三维活细胞成像的DNA损伤反应蛋白
摘要
该协议描述了一个可视化的DNA双链断裂以及其定位在有丝分裂过程中DNA损伤激活的信号蛋白的方法。
摘要
双链断裂(DSBs)是最有害的DNA病变的细胞可能会遇到。如果未修复,DNA双链断裂港口的巨大潜力产生突变和染色体畸变1。为了防止这种创伤催化基因组的不稳定性,这是至关重要的,检测DNA双链断裂的细胞,激活DNA损伤反应(DDR),并修复DNA。解除武装,复员和重返社会工作的刺激时,通过触发细胞周期阻滞,以便维修发生或强制的细胞发生凋亡,以维持基因组的完整性。 DSB修复发生的主要机制通过非同源末端连接(NHEJ)和同源重组修复(HRR)(评论2例 )。有许多的蛋白质,其活动必须精确精心策划的解除武装,复员和重返社会正常运行。这里,我们描述了一种方法,2 - 和3 - 维(D)的可视化这些蛋白质之一,53BP1。
p53结合蛋白1(53BP1)定位于地区DNA双链断裂的修饰组蛋白3,4结合,形成病灶在5-15分钟内5。组蛋白修饰和招聘53BP1和其他DDR蛋白DSB网站被认为是染色质的结构重排,以方便周边地区的损失,并有助于DNA修复6。除了 直接参与修复,额外的角色被描述为53BP1在解除武装,复员和重返社会,如调节内-S检验点,G2 / M检验点,并激活下游DDR蛋白7-9。最近,人们发现,53BP1没有形成病灶在有丝分裂过程中DNA损伤,而不是等待细胞进入G1前定位附近的DNA双链断裂6。如53BP1 DDR蛋白质已被发现与有丝分裂的结构(如着丝粒)相关联的进展通过有丝分裂10期间。
在这个协议中,我们描述了使用2 - 和3-D的活细胞成像的可视化53BP1灶形成的DNA损伤剂喜树碱(CPT),以及53BP1在有丝分裂过程中的行为。喜树碱是拓扑异构酶I抑制剂,主要引起DNA双链断裂DNA复制过程中。要做到这一点,我们使用先前描述的53BP1 mCherry荧光融合蛋白结构的53BP1的蛋白质结构域的能够结合DNA双链断裂11。此外,我们使用组蛋白H2B-GFP的荧光融合蛋白的构建,能够监控整个细胞周期,但在特定的染色质的动态,在有丝分裂12。活细胞成像技术在多个方面,是一个很好的工具,以加深我们对DDR在真核细胞中的蛋白质的功能。
研究方案
A.细胞制备
- 正常人体主要的成纤维细胞(GM02270)卡瑞尔细胞储存库,卡姆登,新泽西州,并与hTERT 6永生。细胞生长和扩大在蜂星6-cm培养皿在媒体(4毫升)组成的MEM培养基与20%的胎牛血清(GIBCO),的non-essential/essential氨基酸,维生素,丙酮酸钠,和青霉素/链霉素(HyClone公司)。
- 人胚胎肾293(HEK293)细胞,从美国典型培养物保藏中心获得的,并在蜂星6-cm培养皿中生长和扩大。中,细胞保持在介质(4毫升)中组成的DMEM培养液与10%牛胎儿血清,非必需氨基酸,L-谷氨酰胺和青霉素/链霉素。
- 53BP1 mCherry(N-MYC-53BP1 WT pLPC的普罗Addgene质粒19836)(pCLNR H2BG; Addgene质粒17735)和H2B-GFP融合基因的结构也表达嘌呤霉素或G418抗性基因,分别转(fibrobla针)或转染(HEK293)使用SuperFect(Qiagene)进入细胞,并保持在药物选择。荧光定期检查维护。
- 图像采集前24-48小时,将细胞用胰蛋白酶消化成单细胞悬液,并以较低的密度接种到的3.5厘米FluroDish玻璃底板。
B.显微镜设置和图像采集
该协议被开发使用的蔡司细胞观察SD旋转盘共聚焦显微镜配备了一个AxioObserver Z1架,双通道Yokagawa CSU-X1A 5000旋转的磁盘单元,配光QuantEM 512SC EMCCD相机,HXP 120C光纤为基础的照明灯,4激光器(一Lasos 100mW的多行,氩[458,488,514纳米,50 mW的405 nm二极管,40 mW的561 nm二极管,和30 mW的635 nm二极管),AOTF,一个Pecon的的XL multiS1阶段孵化体系,蔡司孵化模块(模块S O 2,CO 2 TempModule模块S,S,供热机组XL小号)和前个月XY torized阶段与一个NanoScanZ的压电Z插入。为了尽量减少的活细胞成像的球面像差,而在水性介质中,一个C-复消色差63x/1.20水/更正物镜和蔡司Immersol W浸没流体(具有折射率n = 1.334)的支持。对于2通道共焦成像中,RQFT 405/488/568/647分色镜和BP525/50(绿色)和BP629/62(红色)排放过滤器被使用。所用的系统软件与AV4 Multi-channel/Z/T,快速成像,生理学,MosaiX,标记和查找,双摄像头,里面4D,自动对焦和3-D反褶积模块:蔡司Axiovision(版本4.8.2.0)。
- 确保运行,CO 2气体的CO 2孵化系统的模块。如果在显微镜防振空气表支持的,打开空气供给(或N 2气体)的空气表。
- 打开显微镜支架的电源开关,旋转式磁盘装置,数码相机,孵化模块,HXP照明灯,电动载物台,氩气激光,和计算机。
- 经过1分钟的热身时间,将点火钥匙的氩离子激光器为“开”。
- 在蔡司激光控制面板中,打开要使用的激光线的开关。
- 拨动开关切换为“待机”,“激光运行”,的Lasos氩离子激光器控制器,调光控制器的最佳水平( 即正下方的绿色指示灯会变成红色)。
- 启动AxioVision软件。 注 :是专门针对特定的显微镜和组件,它控制的窗口和下拉菜单,可自定义的用户界面AxioVision。因此,每个系统都有可能是唯一的接口。因此,在随后的步骤中提供软件操作的一般说明,而不是特定的软件窗口,标签和/或下拉菜单的方向。
- 成像前约1小时,在软件中,定位控制的孵化器和上部腔室的加热和镜台板的开启。把温度设置为37℃。打开的CO 2的控制和设置在5%的水平。
- 选择用于成像的物镜。在这项研究中,63x/1.20 NA C-复消色差水/更正物镜使用注 :对于这样的镜头,需要浸没介质的折射率类似的水(蔡司Immersol W浸没流体)。
- 如果在一个长的时间内将被采样的多个单独的位置,某些应用足够量的液浸介质中,以确保它从一个位置到另一个进行。
- 放置在舞台上的菜带来物镜成与底部接触。
- 在显微镜控制或软件,直接发出的光目镜,选择“适当的广角过滤器的设置荧光信号的利息(这项研究中,”红“过滤器设置53BP1和”绿色荧光蛋白“过滤器设置为H2B )。/ P>
- 查看通过眼透镜,图像聚焦,细胞中找到一个合适的领域。
- 使用软件或显微镜控制,直接发射的光远离共焦旋转圆盘单元到端口的目镜。
- 在软件中,打开相应的激光(这项研究中,561纳米激光53BP1和H2B的氩离子激光器的488 nm线)。
- 对于每个通道,调整的激光的强度的声光可调谐滤波器(AOTF)控制通过调节到适当的水平。
- 选择适当的的分色镜(RQFT 405/488/568/647)和发射滤波器(BP62分之62953BP1和BP五十○分之五百二十五的的H2B)。打开快门纺丝盘单元。
- 选择“实时”窗口,显示当前的视野。
- 在软件中,打开“相机”控制,选择相机使用(如果是双相机系统),并设定曝光时间约为100毫秒。调整%和EM获得其他埃森。 注:53BP1 mCherry结构显得有些暗淡。我们发现它有利于提高EM增益。
- 在软件中,打开控制的共聚焦转盘单位和调整转盘的速度进入相机的曝光时间,捕捉到一个合适的图像( 例如 〜100毫秒)。单击“设置”锁定的变化。
- 打开“多维收购”。选择通道“选项卡,加载/选择合适的渠道。在这项研究中,我们将使用红色荧光蛋白(561 nm激光激发和BP 629/62过滤器的排放)和GFP(488 nm激光激发和BP525/50过滤器的发射线)定义的通道。
- 为了确保图像配准中,一个共同的分色镜(RQFT 405/488/568/647)用于两条通道。设置软件的“自动对焦”。 注意 :工具→设置编辑器调整的MDA设置。确保有足够的激光功率设置(“足够的激光功率”是指设置,让您激发适当的荧光基团,同时最大限度地减少光漂白)。
- 在“MDA”窗口中,选择“Z-Stack的标签。选择“Z-堆栈在当前的焦点位置”。设置范围〜10微米的Z-Stack,然后选择“最佳”的一些步骤,以确保奈奎斯特采样到Z 注意:确切的Z-Stack的大小将取决于你的细胞的高度。相应的调整。
- 23。单击“开始”,产生的Z-Stack的图像进行分析,以确保设置适合你调查( 例如在这项研究中的,这是非常重要的,整个核)。
- 选择“T”(时间)选项卡。对于喜树碱我们的实验中,我们设置了成像时间点之间的时间间隔为5分钟和1小时的控制(非处理的细胞)视频会议期间的整体。 注意:尽量减少光漂白细胞,实验应该被配置为使得在AOTF激光成像的时间点之间的空白。
- F多点成像几个细胞的菜,在MDA“窗口中,选择”位置“选项卡。确保“应用”设定时间点的每个位置前/后“被选中。 ,选择“Mark_Find”。使用“现场”视图,将周围的菜,并选择合适的视野。
- 多维收购菜单中单击“开始”,开始进行实验,并记录控制视频(未经处理的细胞)。
- 控制视频后,添加适当的处理(在这种情况下,10μM喜树碱)。我们记录了实验(药物处理的细胞)的视频在5-10分钟的时间间隔为2-4小时。 注:要考虑的是速度,一个给定的效果,治疗后出现的一个重要问题。在视频中可以看出,53BP1灶开始〜5分钟后,加入CPT。虽然一个相对容易的过程,手动添加药物的菜和重新校准的显微镜,确实需要时间。设计实验时,必须考虑到这。
- 为了监测米itosis,我们的间隔设为7.5分钟,并记录为4-5小时(特定的设置取决于使用的细胞系和其细胞周期的长度)。可能需要进行调整,在较长的录音,以防止光漂白。
C.图像处理与分析
Volocity软件(珀金埃尔默),该协议的开发利用。获得这些数据(AxioVision)所使用的软件也有能力来处理和分析图像。我们鼓励用户利用该软件提供给他们,并查阅相关文献,对他们的应用程序。
- 打开Volocity软件。创建并命名一个新的库,并导入你的视频文件。
- 要查看的文件,我们通常会发现使用“扩展聚焦方式”设定有极大的帮助。这叠加的Z-Stack片,此协议,使我们能够可视化53BP1灶在不同领域的核。
- 在必要时调整的影片。 VOlocity配备了一系列的工具,以提高采集到的图像质量。很多时间通过确保在显微镜上的设置是合适的,可以被保存在编辑以后。通常情况下,它是有帮助,反卷积您的图像,调整亮度/对比度。实验的基础上,你的特定的编辑需求会有所不同。
- 添加一个相对时间戳和比例尺。
- 要查看单元格中的3-D,切换到“3-D不透明度”设置。这允许在空间的3-D渲染细胞的旋转,从而提供利益的结构的细胞内的多个角度。 注:这是有助益的可视化细胞在不同的平面中,以确定结构感兴趣的旅行。例如,在“扩展聚焦方式”设定,它是很难辨别53BP1不,其实,游离在有丝分裂过程中从DNA。然而,这是显而易见的,在3-D。
- 动画和静止图像可以导出成多种文件类型,根据用户的喜好。 I>
D.代表性的成果
响应对CPT 53BP1灶形成的一个例子的图1中所示。中华映管灶的细胞暴露在5-10分钟范围内,并保持这些病灶在整个持续时间的记录。正如在图2中所示,53BP1解离染色质在有丝分裂的发病,冷凝染色体周围形成薄膜的雾度。末期发生有丝分裂结束,53BP1再次聚集成不同的病灶。虽然HEK293细胞没有接触到CPT,但它们形成丰富的,自发的53BP1所产生的内源性DNA损伤的修复病灶。这一观察结果使我们得出结论,53BP1没有形成病灶早期有丝分裂过程中,在与以前的报告显示,类似的效果后,细胞暴露于电离辐射和radiomimetic的药物5。
从图1“SRC =”/ files/ftp_upload/4251/4251fig1.jpg“/>
图1。喜树碱(10μM)引起DNA双链断裂和向培养基中添加药物后的30分钟内,在骑自行车的成纤维细胞的53BP1灶形成。
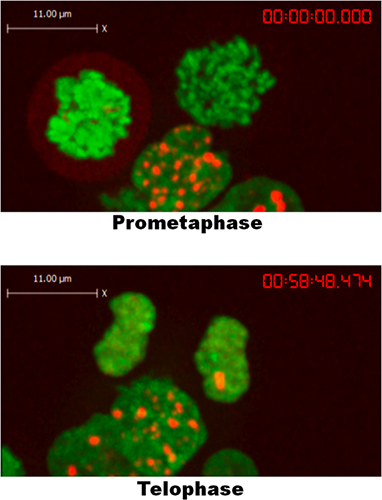
图2。,53BP1并不构成灶在HEK293细胞中有丝分裂过程中,直到telophase/G1。
讨论
维持基因组的完整性,细胞的生存是至关重要的。如果保存的基因组会导致过早衰老,癌变,死亡8。有浓厚的兴趣,挑剔的DDR功能,源于其基础和临床研究的重要性。多年来,以帮助在细胞如何检测和修复DNA损伤的研究已开发了许多技术。传统的方法,如免疫细胞化学和西方墨点法领域的支柱,尽管最近的技术进步已经使日益复杂的方法发展。活细胞成像,在该协议中详细说明,使我们?...
披露声明
没有利益冲突的声明。
致谢
支持部分由R01NS064593和R21ES016636(KV)。显微镜进行VCU - 神经生物学与解剖学显微镜设备,部分支持,资金从NIH-NINDS中心的核心授予5P30NS047463的。旋转盘共聚焦显微镜购买的与NIH-NCRR的奖(1S10RR027957)。
材料
| Name | Company | Catalog Number | Comments |
| 产品 | 公司 | ||
| 陕西蜂星的培养皿 | 格雷纳生物一 | ||
| FluroDish玻璃底菜 | 世界精密仪器公司 | ||
| MEM媒体 | GIBCO | ||
| 非必需氨基酸 | GIBCO | ||
| 氨基酸 | GIBCO | ||
| 维生素 | GIBCO | ||
| 丙酮酸钠 | Invitrogen公司 | ||
| 青霉素/链霉素 | 胎牛血清 | ||
| 胎牛血清 | GIBCO | ||
| N-MYC-53BP1 WT pLPC的普罗; 质粒19836 | Addgene | ||
| pCLNR H2BG质粒17735 | Addgene | ||
| SuperFect | Qiagen公司 | ||
| 蔡司观察SD细胞成像系统 | 蔡司 | ||
| AxioVision(版本4.8.2) | 蔡司 | ||
| 蔡司Immersol W油 | 蔡司 | ||
| Volocity软件(版本6.0) | 珀金埃尔默 |
参考文献
- Botuyan, M. V., Lee, J., Ward, I. M., Kim, J. E., Thompson, J. R., Chen, J., Mer, G. Structural basis for the methylation state-specific recognition of histone H4-K20 by 53BP1 and Crb2 in DNA repair. Cell. 127, 1361-1373 (2006).
- Dimitrova, N., Chen, Y. C., Spector, D. L., de Lange, T. 53BP1 promotes non-homologous end joining of telomeres by increasing chromatin mobility. Nature. 456, 524-528 (2008).
- Feuerhahn, S., Egly, J. M. Tools to study DNA repair: what's in the box. Trends Genet. 24, 467-474 (2008).
- Giunta, S., Belotserkovskaya, R., Jackson, S. P. DNA damage signaling in response to double-strand breaks during mitosis. J. Cell Biol. 190, 197-207 (2010).
- Giunta, S., Jackson, S. P. Give me a break, but not in mitosis: the mitotic DNA damage response marks DNA double-strand breaks with early signaling events. Cell Cycle. 10, 1215-1221 (2011).
- Golding, S. E., Morgan, R. N., Adams, B. R., Hawkins, A. J., Povirk, L. F., Valerie, K. Pro-survival AKT and ERK signaling from EGFR and mutant EGFRvIII enhances DNA double-strand break repair in human glioma cells. Cancer Biol. Ther. 8, 730-738 (2009).
- Huyen, Y., Zgheib, O., Ditullio, R. A., Gorgoulis, V. G., Zacharatos, P., Petty, T. J., Sheston, E. A., Mellert, H. S., Stavridi, E. S., Halazonetis, T. D. Methylated lysine 79 of histone H3 targets 53BP1 to DNA double-strand breaks. Nature. 432, 406-411 (2004).
- Jackson, S. P., Bartek, J. The DNA-damage response in human biology and disease. Nature. 461, 1071-1078 (2009).
- Kanda, T., Sullivan, K. F., Wahl, G. M. Histone-GFP fusion protein enables sensitive analysis of chromosome dynamics in living mammalian cells. Curr. Biol. 8, 377-385 (1998).
- Massignani, M., Canton, I., Sun, T., Hearnden, V., Macneil, S., Blanazs, A., Armes, S. P., Lewis, A., Battaglia, G. Enhanced fluorescence imaging of live cells by effective cytosolic delivery of probes. PLoS One. 5, e10459 (2010).
- Nakamura, K., Sakai, W., Kawamoto, T., Bree, R. T., Lowndes, N. F., Takeda, S., Taniguchi, Y. Genetic dissection of vertebrate 53BP1: a major role in non-homologous end joining of DNA double strand breaks. DNA Repair (Amst). 5, 741-749 (2006).
- Schultz, L. B., Chehab, N. H., Malikzay, A., Halazonetis, T. D. p53 binding protein 1 (53BP1) is an early participant in the cellular response to DNA double-strand breaks. J. Cell. Biol. 151, 1381-1390 (2000).
- Valerie, K., Povirk, L. F. Regulation and mechanisms of mammalian double-strand break repair. Oncogene. 22, 5792-5812 (2003).
- Wang, B., Matsuoka, S., Carpenter, P. B., Elledge, S. J. 53BP1, a mediator of the DNA damage checkpoint. Science. 298, 1435-1438 (2002).
- Ward, I. M., Minn, K., Jorda, K. G., Chen, J. Accumulation of checkpoint protein 53BP1 at DNA breaks involves its binding to phosphorylated histone H2AX. J. Biol. Chem. 278, 19579-19582 (2003).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。