È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Due-e tridimensionale Live Cell Imaging di proteine danno al DNA di risposta
In questo articolo
Riepilogo
Questo protocollo descrive un metodo per la visualizzazione di un DNA a doppio filamento proteina pausa segnalazione attivato in risposta al danno del DNA e la sua localizzazione durante la mitosi.
Abstract
Rotture del doppio filamento (DSB) sono il DNA più deleteria lesioni una cella può incontrare. Se lasciato non riparati, DSB potenziale grande porto per generare mutazioni e aberrazioni cromosomiche 1. Per evitare questo trauma da catalizzare l'instabilità genomica, è fondamentale per le cellule per individuare DSB, attivare la risposta al danno al DNA (DDR), e riparare il DNA. Quando stimolato, il DDR lavora per preservare l'integrità genomica innescando arresto del ciclo cellulare per consentire la riparazione abbia luogo o forzare la cellula a subire apoptosi. I meccanismi predominanti di riparazione DSB avvenire attraverso nonhomologous end-joining (NHEJ) e ricombinazione omologa riparazione (HRR) (recensione in 2). Ci sono molte proteine la cui attività deve essere orchestrato con precisione per la DDR per funzionare correttamente. Qui, si descrive un metodo per 2 - e 3-dimensionale (D) visualizzazione di una di queste proteine, 53BP1.
Il p53-binding protein 1 (53BP1) localizza alle aree diDSB legandosi agli istoni modificati 3,4, la formazione di focolai entro 5-15 minuti 5. Le modifiche degli istoni e di assunzione di proteine e di altri 53BP1 DDR a siti DSB si ritiene di facilitare la riorganizzazione strutturale della cromatina intorno alle aree di danno e contribuire alla riparazione del DNA 6. Al di là di partecipazione diretta nella riparazione, altri ruoli sono stati descritti per 53BP1 nella Ddr, quali la regolazione di un intra-S checkpoint, un G2 / M checkpoint, e l'attivazione di proteine a valle DDR 7-9. Recentemente, è stato scoperto che 53BP1 non forma foci in risposta al danno del DNA indotto durante la mitosi, invece attesa cellule a entrare G1 prima localizzazione per la vicinanza DSBs 6. Proteine DDR come 53BP1 sono stati trovati per associare strutture mitotiche (come cinetocori) durante la progressione attraverso la mitosi 10.
In questo protocollo si descrive l'uso di 2 - e 3-D imaging cellule vive per visualizzarela formazione di foci 53BP1 in risposta al danno del DNA agente camptotecina (CPT), così come il comportamento 53BP1 durante la mitosi. Camptotecina è un inibitore della topoisomerasi I che causa principalmente DSBs durante la replicazione del DNA. Per fare questo, abbiamo utilizzato un precedentemente descritto 53BP1-mCherry proteina di fusione fluorescente costruire costituito da un dominio 53BP1 proteina in grado di legare DSB 11. Inoltre, abbiamo utilizzato un istone H2B-proteina di fusione GFP fluorescente costrutto in grado di monitorare la dinamica della cromatina durante il ciclo cellulare, ma in particolare durante la mitosi 12. Imaging cellulare Live in molteplici dimensioni è un ottimo strumento per approfondire la nostra comprensione della funzione delle proteine DDR nelle cellule eucariotiche.
Protocollo
A. Preparazione cellulare
- Normali fibroblasti umani primari (GM02270) sono stati ottenuti da Repository cella Coriell, Camden, New Jersey, e immortalato con hTERT 6. Le cellule sono state coltivate ed espanse in Cellstar 6 centimetri piatti a mezzi (4 ml) costituiti MEM supplementato con 20% siero fetale bovino (GIBCO), non-essential/essential aminoacidi, vitamine, sodio piruvato, e penicillina / streptomicina (HyClone ).
- Rene embrionale umano 293 (HEK293) cellule sono state ottenute dalla American Type Culture Collection e coltivate ed espanse in Cellstar 6 centimetri piatti. Le cellule sono state mantenute in mezzi (4 ml) costituito da DMEM supplementato con 10% siero bovino fetale, aminoacidi non essenziali, L-glutammina, e penicillina / streptomicina.
- 53BP1 mCherry-(N-Myc-53BP1 PLPC WT-Puro; Addgene plasmide 19.836) e H2B-GFP (pCLNR-H2BG; Addgene plasmide 17735) gene di fusione costrutti anche esprimere puromicina o geni di resistenza G418, rispettivamente, sono state trasdotte (fibroblam) o transfettate (HEK293) utilizzando Superfect (Qiagene) nelle cellule e mantenuto sotto la selezione dei farmaci. Manutenzione di fluorescenza è stata verificata periodicamente.
- 24-48 ore prima acquisizione dell'immagine, le cellule sono state tripsinizzate in una singola cella di sospensione e seminate ad una densità bassa su 3,5 centimetri lastre FluroDish fondo di vetro.
B. Configurazione Microscopio e acquisizione di immagini
Questo protocollo è stato sviluppato utilizzando il cellulare Zeiss Observer microscopio confocale disco SD filatura dotata di uno stand AxioObserver Z1, un doppio canale Yokagawa CSU-X1A unità disco 5000 la filatura, 2 fotometrici QuantEM 512SC telecamere EMCCD, un 120C HXP a base di fibre illuminatore, 4 laser (un Lasos 100mW multi-linea Argon [458, 488, 514 nm], 50 mW 405 nm diodo, 40 mW 561 nm diodo, e 30 mW 635 nm diodo), AOTF, un Pecon XL sistema multiS1 fase di incubazione, incubazione Zeiss moduli (Modulo S O 2, CO 2 Modulo S, S TempModule, Riscaldamento unità XL S) e un Priore motorized fase XY con un inserto NanoScanZ piezo Z. Per minimizzare le aberrazioni sferiche mentre le cellule vive di imaging supportato in un mezzo acquoso, un C-Apochromat 63x/1.20 Acqua / Corr lente obiettivo e Zeiss Immersol fluido immersione W (con un indice di rifrazione n = 1,334) sono stati utilizzati. Per 2 canali imaging confocale, una RQFT 405/488/568/647 specchio dicroico e BP525/50 (verde) e BP629/62 (rosso) sono stati utilizzati filtri di emissione. Il software di sistema utilizzato era Zeiss AxioVision (ver. 4.8.2.0) con AV4 Multi-channel/Z/T, Fast Imaging, Fisiologia, Mosaix, Mark & Find, doppia fotocamera, interna 4D, autofocus e 3-D moduli di deconvoluzione.
- Verificare che la CO2 di gas è in esecuzione per il CO 2 modulo del sistema di incubazione. Se il microscopio è supportato da un antivibrante tavolo aria, accendere l'alimentazione dell'aria (o N 2 gas) per la tabella dell'aria.
- Accendere l'alimentazione per il basamento del microscopio, unità disco rotante, macchine fotografiche, i moduli di incubazione, illuminatore HXP, stadio motorizzato, Argonlaser, e il computer.
- Dopo 1 minuto di tempo di riscaldamento, ruotare la chiave di accensione per il laser Argon su "On".
- Sul pannello di controllo laser Zeiss, attivare gli interruttori per le linee laser da utilizzare.
- Spegnere l'interruttore a bilico per il controller Lasos Argon laser di "stand-by" a "run laser" e regolare il regolatore della luce al livello ottimale (appena al di sotto del punto in cui l'indicatore verde diventa rosso).
- Avviare il software AxioVision. Nota: l'interfaccia utente per AxioVision può essere personalizzato con menu finestre e pull-down che sono specifici per il microscopio particolare e dei componenti che lo controlla. Come tale, ogni sistema ha un'interfaccia potenzialmente unico. Pertanto, le istruzioni generiche per la manipolazione del software sono forniti in fasi successive, piuttosto che indicazioni per le finestre di software specifici, le schede e / o menu a discesa.
- Circa 1 ora prima di imaging, nel software, individuare i controlli per l'incubatrice eaccendere il riscaldamento per la camera superiore e la piastra stadio. Impostare la temperatura a 37 ° C. Accendere la CO 2 di controllo e impostare il livello del 5%.
- Selezionare la lente dell'obiettivo per l'imaging. In questo studio, un NA 63x/1.20 C-Apochromat acqua / Corr lente obiettivo è stato utilizzato. Nota: Per questo obiettivo, un mezzo immersione con un indice di rifrazione simile all'acqua (Zeiss Immersol fluido immersione W) è necessario.
- Se più posizioni sono separati da campionare un lungo periodo di tempo, essere certi di applicare una quantità sufficiente di medie immersione per assicurare che esso venga realizzato da una posizione all'altra.
- Posizionare il piatto sulla scena e portare la lente obiettivo, a contatto con il fondo.
- Utilizzando sia i controlli microscopio o il software, la luce emessa per gli oculari e selezionare l'widefield filtro impostato per il segnale fluorescente di interesse (per questo studio, "Red" filtro impostato per 53BP1 e un "GFP" filtro impostato per H2B ).
- Guarda attraverso le lenti oculari, fuoco l'immagine, e individuare un opportuno campo delle cellule.
- Utilizzando il software o controlli microscopio, la luce emessa dalla oculari alla porta con l'unità confocale disco che gira.
- Nel software, accendere il laser appropriato (per questo studio, il laser 561 nm per 53BP1 e la 488 linea nm del laser Argon per H2B).
- Per ciascun canale, regolare l'intensità del laser regolando il acusto-ottico sintonizzabile filtro (AOTF) controllo ad un livello appropriato.
- Selezionare lo specchio dicroico appropriato (RQFT 405/488/568/647) e filtri di emissione (BP 629/62 per 53BP1 e Bp 525/50 per H2B). Aprire l'otturatore all'unità disco rotante.
- Selezionare la finestra "Live" per visualizzare il campo di vista corrente.
- Nel software, aprire il controllo "Camera", selezionare la telecamera da utilizzare (se un sistema di doppia fotocamera) e impostare il tempo di esposizione di circa 100 ms. Regolare il% e EM guadagnare più ncaNota rio:. L'53BP1-mCherry costrutto appare un po 'debole. Abbiamo trovato utile per aumentare il guadagno EM.
- Nel software, aprire il controllo per l'unità confocale disco rotante e regolare la velocità di rotazione del disco inserendo il tempo di esposizione della fotocamera che è stato impostato per catturare un'immagine adatta (ad esempio ~ 100 ms). Fare clic su "Set" per bloccare il cambiamento.
- Aprite "multi-dimensionale acquisizione". Selezionare la scheda di canale e caricare / selezionare i canali appropriati. Per questo studio, stiamo usando i canali definiti per DsRed (561 nm di eccitazione laser e BP 629/62 filtro per le emissioni) e GFP (488 nm linea laser per l'eccitazione e BP525/50 filtro per le emissioni).
- Per garantire la registrazione dell'immagine, uno specchio dicroico comune (RQFT 405/488/568/647) è stato usato per entrambi i canali. Impostare il software per "Autofocus". Nota: Vai a Strumenti → Impostazioni editor per modificare le impostazioni MDA. Assicurarsi che la potenza del laser adeguata è impostato ("laser di potenza adeguata" si intende un'impostazione che consenteper eccitare il fluoroforo appropriato minimizzando foto-sbiancamento).
- Nella finestra MDA, selezionare la scheda z-stack. Selezionare "Z-stack in posizione del focus corrente". Impostare l'intervallo di ~ 10 micron Z-Stack e scegliere "ottimale" per il numero di misure per garantire il campionamento di Nyquist con Z. Nota: la dimensione esatta della Z-Stack dipenderà l'altezza delle vostre cellule. Regolare di conseguenza.
- 23. Fare clic su "Start" e analizzare il risultato z-serie di immagini per verificare che le impostazioni siano appropriate per quello che si sta indagando (ad esempio, in questo studio, è stato importante per l'immagine del nucleo intero).
- Selezionare l'opzione "T" (tempo) tab. Per il nostro esperimento con camptotecina, abbiamo impostato l'intervallo tra timepoints imaging per 5 min e la durata complessiva della sessione per 1 ora per un controllo (non trattati cellule) video. Nota: per minimizzare fotometabolismo delle cellule, l'esperimento deve essere configurato in modo tale che l'AOTF sbozzati il laser tra i punti di tempo di imaging.
- Fo più punti di imaging di cellule diverse nel piatto, nella finestra MDA, selezionare la scheda posizione. Assicurarsi che "'impostazione prima / dopo il punto di tempo' Applica per posizione" sia selezionata. Selezionare "Mark_Find". Utilizzo della vista "Live", spostare il piatto intorno e selezionare i campi appropriati di vista.
- Fare clic su "Start" nella multidimensionale acquisizione menu per avviare l'esperimento e registrare un video di controllo (di cellule non trattate).
- Dopo il controllo video, aggiungere il trattamento appropriato (in questo caso, camptotecina 10 pM). Abbiamo registrato sperimentale (farmaco cellule trattate) video ad intervalli 5-10 minuti per 2-4 ore. Nota: Un aspetto importante da considerare è la velocità con cui un dato effetto si verifica dopo il trattamento. Come si vede nel video, 53BP1 foci cominciano a da ~ 5 min dopo l'aggiunta della CPT. Anche se un processo relativamente semplice, l'aggiunta di farmaco per il piatto manualmente e ri-calibrazione del microscopio ci vuole tempo. È necessario tenere conto di questo quando si progetta esperimenti.
- Per il monitoraggio mitosis, abbiamo impostato l'intervallo di 7,5 min e registrati per 4-5 ore (le impostazioni specifiche dipendono dalla linea cellulare utilizzato e la lunghezza del suo ciclo cellulare). Rettifiche di valore potrebbe essere necessario essere fatto per evitare che foto-sbiancamento durante registrazioni più lunghe.
C. Image Processing and Analysis
Questo protocollo è stato sviluppato utilizzando il software Volocity (PerkinElmer). Il software utilizzato per acquisire questi dati (AxioVision) ha anche la capacità di elaborare e analizzare le immagini. Gli utenti sono incoraggiati a utilizzare il software a loro disposizione, e consultare la documentazione del caso per quanto riguarda la loro applicazione.
- Aprire il software Volocity. Creare e nominare una nuova libreria, e importare i file video.
- Per visualizzare i file, di solito si trovano più utile usare la "Extended Focus" impostazione. Questo sovrappone i z-stack fette e, per questo protocollo, ci permette di visualizzare 53BP1 focolai in diverse zone del nucleo.
- Regolare i video, se necessario. Volocity è dotato di una serie di strumenti per migliorare la qualità delle immagini acquisite. Garantendo le impostazioni del microscopio erano appropriati, molto tempo possono essere salvati in modifica in seguito. Spesso, è utile deconvolve le immagini, e regolare la luminosità / contrasto. Le vostre esigenze di editing specifici può variare in base dell'esperimento.
- Aggiungi un timbro relativo tempo e barra di scala.
- Per visualizzare le cellule in 3-D, passare alla "3-D opacità" impostazione. Questo consente la rotazione delle cellule resi 3-D nello spazio, fornendo così molteplici prospettive di strutture di interesse all'interno delle cellule. Nota: E 'utile visualizzare le cellule su piani diversi per determinare dove le strutture di viaggio interesse. Per esempio, nel "avanzata Focus" impostazione è difficile discernere che il 53BP1 fa, infatti, dissocia dal DNA durante la mitosi. Tuttavia, questo è evidente in 3-D.
- Filmati e immagini fisse possono essere esportati in una varietà di tipi di file in base alle preferenze dell'utente. i>
D. Rappresentante Risultati
Un esempio di formazione di foci 53BP1 in risposta al CPT è mostrato in Figura 1. Cellule esposte a forma foci CPT entro 5-10 minuti, e mantenere questi focolai tutta la durata della registrazione. Come mostrato in figura 2, 53BP1 dissocia dalla cromatina allo scoppio della mitosi, formando una nebbia sottile intorno cromosomi condensazione. Come telofase si verifica e la mitosi giunge al termine, 53BP1 ancora una volta aggregati in focolai distinti. Mentre le cellule HEK293 non sono stati esposti a CPT, hanno comunque formato abbondante, foci di riparazione spontanea 53BP1 generato dal danno al DNA endogeno. Questa osservazione ci ha permesso di concludere che 53BP1 non forma fuochi durante la mitosi presto, in linea con una precedente relazione che mostra un effetto simile dopo l'esposizione delle cellule a radiazioni ionizzanti e la droga radiomimetici 5.
ure 1 "src =" / files/ftp_upload/4251/4251fig1.jpg "/>
Figura 1. Camptotecina (10 pM) e provoca DSBs 53BP1 formazione foci in bicicletta fibroblasti entro 30 minuti di aggiunta del farmaco al mezzo.
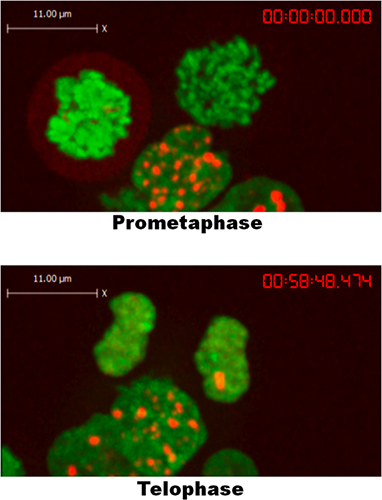
Figura 2. 53BP1 non formare foci durante la mitosi fino telophase/G1 in cellule HEK293.
Discussione
Mantenimento dell'integrità genomica è fondamentale per la sopravvivenza delle cellule. La mancata conservare i risultati del genoma in invecchiamento precoce, carcinogenesi o morte 8. C'è forte interesse a discernere come funziona il DDR, derivanti dalla sua importanza per la ricerca di base e clinica. Molte tecniche sono state sviluppate nel corso degli anni per aiutare nello studio di come le cellule di rilevare e riparare i danni del DNA. I metodi tradizionali, quali immunocitochimica e Western...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Supportato in parte da R01NS064593 e R21ES016636 (KV). Microscopia è stata eseguita presso il VCU - Dipartimento di Neurobiologia e Microscopia strumento Anatomia, sostenuta, in parte, con i fondi NIH NINDS-Centro nucleo concessione 5P30NS047463. Il microscopio confocale disco rotante è stato acquistato con un NIH-NCRR premio (1S10RR027957).
Materiali
| Name | Company | Catalog Number | Comments |
| Prodotto | Azienda | ||
| Cellstar cultura piatti | Greiner Bio-One | ||
| FluroDish piatti di vetro con fondo | Mondo Strumenti di precisione, Inc. | ||
| MEM supporti | GIBCO | ||
| Aminoacidi non essenziali | GIBCO | ||
| Amminoacidi | GIBCO | ||
| Vitamine | GIBCO | ||
| Sodio piruvato | Invitrogen | ||
| Penicillina / streptomicina | HyClone | ||
| Siero fetale bovino | GIBCO | ||
| N-Myc-53BP1 PLPC WT-Puro; plasmide 19836 | Addgene | ||
| pCLNR-H2BG; plasmide 17735 | Addgene | ||
| Superfect | Qiagen | ||
| Zeiss cellulare Observer SD Imaging sistema | Zeiss | ||
| AxioVision (versione 4.8.2) | Zeiss | ||
| Zeiss Immersol W Olio | Zeiss | ||
| Volocity software (versione 6.0) | PerkinElmer |
Riferimenti
- Botuyan, M. V., Lee, J., Ward, I. M., Kim, J. E., Thompson, J. R., Chen, J., Mer, G. Structural basis for the methylation state-specific recognition of histone H4-K20 by 53BP1 and Crb2 in DNA repair. Cell. 127, 1361-1373 (2006).
- Dimitrova, N., Chen, Y. C., Spector, D. L., de Lange, T. 53BP1 promotes non-homologous end joining of telomeres by increasing chromatin mobility. Nature. 456, 524-528 (2008).
- Feuerhahn, S., Egly, J. M. Tools to study DNA repair: what's in the box. Trends Genet. 24, 467-474 (2008).
- Giunta, S., Belotserkovskaya, R., Jackson, S. P. DNA damage signaling in response to double-strand breaks during mitosis. J. Cell Biol. 190, 197-207 (2010).
- Giunta, S., Jackson, S. P. Give me a break, but not in mitosis: the mitotic DNA damage response marks DNA double-strand breaks with early signaling events. Cell Cycle. 10, 1215-1221 (2011).
- Golding, S. E., Morgan, R. N., Adams, B. R., Hawkins, A. J., Povirk, L. F., Valerie, K. Pro-survival AKT and ERK signaling from EGFR and mutant EGFRvIII enhances DNA double-strand break repair in human glioma cells. Cancer Biol. Ther. 8, 730-738 (2009).
- Huyen, Y., Zgheib, O., Ditullio, R. A., Gorgoulis, V. G., Zacharatos, P., Petty, T. J., Sheston, E. A., Mellert, H. S., Stavridi, E. S., Halazonetis, T. D. Methylated lysine 79 of histone H3 targets 53BP1 to DNA double-strand breaks. Nature. 432, 406-411 (2004).
- Jackson, S. P., Bartek, J. The DNA-damage response in human biology and disease. Nature. 461, 1071-1078 (2009).
- Kanda, T., Sullivan, K. F., Wahl, G. M. Histone-GFP fusion protein enables sensitive analysis of chromosome dynamics in living mammalian cells. Curr. Biol. 8, 377-385 (1998).
- Massignani, M., Canton, I., Sun, T., Hearnden, V., Macneil, S., Blanazs, A., Armes, S. P., Lewis, A., Battaglia, G. Enhanced fluorescence imaging of live cells by effective cytosolic delivery of probes. PLoS One. 5, e10459 (2010).
- Nakamura, K., Sakai, W., Kawamoto, T., Bree, R. T., Lowndes, N. F., Takeda, S., Taniguchi, Y. Genetic dissection of vertebrate 53BP1: a major role in non-homologous end joining of DNA double strand breaks. DNA Repair (Amst). 5, 741-749 (2006).
- Schultz, L. B., Chehab, N. H., Malikzay, A., Halazonetis, T. D. p53 binding protein 1 (53BP1) is an early participant in the cellular response to DNA double-strand breaks. J. Cell. Biol. 151, 1381-1390 (2000).
- Valerie, K., Povirk, L. F. Regulation and mechanisms of mammalian double-strand break repair. Oncogene. 22, 5792-5812 (2003).
- Wang, B., Matsuoka, S., Carpenter, P. B., Elledge, S. J. 53BP1, a mediator of the DNA damage checkpoint. Science. 298, 1435-1438 (2002).
- Ward, I. M., Minn, K., Jorda, K. G., Chen, J. Accumulation of checkpoint protein 53BP1 at DNA breaks involves its binding to phosphorylated histone H2AX. J. Biol. Chem. 278, 19579-19582 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon