需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
高速超分辨速度显微镜在活原纤毛中的应用
摘要
最近, 我们映射了三维 (3D) 的运输路线的空间位置的各种蛋白质转运内原纤毛的活细胞。本文详细介绍了新应用于活原纤毛的3D 超分辨率荧光成像方法的实验装置、生物样品的处理过程和数据分析。
摘要
主要纤毛是在许多真核细胞的表面上的 microtubule-based 突起, 它包含了在细胞运动和信号中具有重要作用的独特的蛋白质补充。由于纤毛无法合成自己的蛋白质, 近200独特的睫状蛋白需要在胞和原发纤毛之间进行贩运。然而, 这仍然是一个技术上的挑战, 以地图三维 (3D) 的运输途径的这些蛋白质在活原发纤毛的位置, 由于目前现有的技术的局限性。为了克服这一挑战, 最近我们开发并使用了一个高速虚拟3D 超分辨率显微术, 称为单点边缘激发 sub-diffraction (速度) 显微镜, 以确定3D 空间位置的传输路径胞浆和细胞膜蛋白都是活细胞的主要纤毛。在本文中, 我们将演示的详细设置速度显微镜, 细胞表达荧光蛋白标记的睫状蛋白, real-time 分子跟踪个体蛋白在活纤毛和成就3D 空间概率密度图谱的传输路线的睫状蛋白。
引言
自 1873年, 由于来自目标12的光线衍射, 传统光学显微镜的分辨率被认为仅限于大约 200 nm。目前, 超分辨率光显微镜技术打破了这一限制, 并允许捕捉动态图像与 sub-diffraction (< 200 nm) 的决议。该技术一般分为两大类: 受激发射损耗 (STED) 显微镜的方法, 它产生 sub-diffraction 光照体积由于非线性光学响应的荧光在样本3;和光光显微镜 (手掌) 和随机光学重建显微镜 (风暴) 的超分辨率技术, 利用数学功能, 本地化质心的荧光, 然后重建这些质心要形成超分辨率图像4,5。目前, 由于相对简单的光学设置, 棕榈和风暴是广泛使用的只激活一个小子集的荧光在每个帧的一个长视频的生物准备。这使得更精确的定位由2D 高斯拟合的荧光点, 称为点扩散函数 (聚砜), 荧光标记的蛋白质在每个帧的视频。每个荧光标记的分子的2D 位置, 然后可以叠加在一个单一的成像平面上产生一个超分辨率的图像的生物准备1,2。虽然这些分子的本地化, 超分辨率的方法, 以显微镜肯定革命性的生物样品的成像是如何进行的, 仍然有挑战要克服。例如, 风暴和棕榈可以达到他们的最佳空间分辨率后, 固定的生物样品, 从而提出了静态表示的荧光标记蛋白, 这是一个类似的限制电子显微镜。此外, 为了实现在活细胞中每个荧光标记的蛋白质的高空间分辨率, 样品必须成像在非常长的帧, 不能捕捉蛋白质动力学。因此, 有必要克服这些主要的技术障碍。
为了获得一个高时空分辨率, 它非常适合在活细胞中检测快速移动的蛋白质或 rna, 我们在实验室中开发了超分辨速度显微镜 (图 1)6,7,8. 速度显微镜的几个主要技术进步使我们能够成功地追踪小分子、蛋白质、mRNA 和病毒通过本机核孔复合体 (npc) 的 nucleocytoplasmic 传输,6,7,8. 简要地, 以下速度显微镜的特点将用于跟踪快速移动的大分子通过微米旋转对称结构在活细胞, 例如 npc 和主要纤毛: (1) 倾斜或垂直照明, 使在焦平面的小衍射极限体积内的单分子激发 (图 1);(2) 倾斜的聚砜可以极大地避免焦荧光, 从而提高信噪比。(3) 光照中的 100-500 kW/cm2的光学密度允许数千个光子从单个荧光中收集, 并具有快速检测速度 (> 500 Hz)。(4) 快速检测速度还大大降低了分子空间定位错误 (< 10 nm) 在确定活细胞中移动荧光分子的空间轨迹时, 因为分子扩散是主要因素之一造成移动分子分子定位的缺陷。(5) 良好的2D 到3D 转换算法使我们能够为 NPC 或主要纤毛中的分子提供3D 空间概率密度地图。值得注意的是, 我们在笛卡尔和圆柱坐标系之间的转换过程被用来生成3D 空间概率密度图而不是3D 分子跟踪 (图 2)。此前, 电子显微镜数据显示, NPC9、10和主纤毛11都具有旋转对称结构。原则上, 随机扩散分子通过 NPC 或初级纤毛也应该有旋转对称分布。如图 2所示, 大量随机扩散的分子在气缸内将产生旋转对称分布, 在剖面图, 在 NPC, 进一步导致一个近似一致的空间在相邻的两个环之间的每个非常小的子区域中分布 (图 2E)。这种均匀分布导致圆柱系统中θ维数的空间分布是恒定的。然后, 3D 坐标 (r、x、θ) 可以简化为2D 坐标 (r、x、常量)。实际上, 我们在笛卡尔和圆柱系统之间的转换过程是从 2D (x, Y) 到 2D (R, x, 常数)。常数θ, 指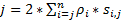 的是空间密度p在图 2E中, 是通过使用等式A来计算的。
的是空间密度p在图 2E中, 是通过使用等式A来计算的。
最终, 分子跟踪在生物研究中有着广泛的应用, 因此, 将开发大量的技术来填充特定的生物龛位12、13、14是很自然的。速度显微镜就是如此。以前, 当与3D 变换算法结合时, 这项技术被开发来解决通过 npc 传递分子的3D 传输路线, 一个 sub-diffraction-sized 和旋转对称的生物结构6。本文中, 原发纤毛也被证明是优秀的模型细胞器。主要纤毛是圆柱形的, 天线状的细胞器 (~ 125 nm 半径), 该项目从大多数哺乳动物的表面15,16,17。它们负责接收外部信号, 并传送通常与生长和新陈代谢相关的细胞内反应15,16。因此, 结构蛋白的通量、跨膜受体的再循环以及细胞内信使的传递是原发性纤毛的重要责任。在主要纤毛和细胞体之间的接合处是一种临界选择性屏障, 称为过渡区或正向, 所有这些蛋白传输必须发生11,18,19, 20。除了 intraflagellar 的浇注功能, 至少有两个传输过程, 即传输和被动扩散, 被认为是负责蛋白质的移动通过这个区域16,21,22. 从人类健康的角度来看, 失去初级纤毛和随后对下游信号的放松管制是许多癌症的特点。此外, 许多遗传性疾病, 如 Bardet Biedl 综合征和多囊肾病, 都与有缺陷的蛋白质转运有关23。sub-diffraction 极限大小和选择性蛋白转运的复杂过程, 使原纤毛成为这项技术的主要目标。在本方法中, 我们将展示一个睫状跨膜蛋白的跟踪, 生长抑素受体 3 (SSTR3)24, 在外部标有 Alexa 氟647和 IFT, IFT2025的成分, 用一个融合的 GFP 分子标记。
研究方案
1. NIH-3T3 细胞制备速度显微镜从库存
- 在实验前1.5 周, 在37° c 解冻的情况下, 从冷冻库中恢复 NIH-3T3 细胞的新鲜培养, 并将细胞转化为25厘米2细胞培养烧瓶, 3 毫升 Dulbecco 的改良鹰培养基 (DMEM) 补充110毫克/毫升丙酮酸钠, 2 毫米谷氨酰胺, 10% 胎牛血清, 1% 青霉素/链霉素。
- 在 5% CO2孵化器中孵育37° c 的细胞。
- 分裂细胞在 80% 70-100, 大约每两天, 至少在实验日前三次, 以确保细胞周期的均匀性。处理细胞与0.25% 胰蛋白酶2分钟在37° c, 抽吸胰蛋白酶和替换它与2毫升媒介。将介质反复移出细胞簇, 去除所需的细胞数量, 使培养基总体积恢复到3毫升。
注意: NIH-3T3 以前是基因工程的, 以表达 NPHP-4, 一种蛋白质, 本地化到26, 融合在 C 总站到 mCherry。mCherry 是一种荧光, 可以在561纳米光照的照射下, 定量地定位其选择性屏障, 使原纤毛处于定向。 - 实验前两天, 将细胞放入35毫米的玻璃底盘中, 60-70% 70-100, 1.5 毫升的培养基为1.1 步, 并将细胞返回孵化室。
- 在实验的前一天, 化学染的细胞与所需的质粒。混合 500-1000 ng 所需的质粒 (见下面的注意) 在 1:2. 5 的比例与转染试剂在0.25 毫升的减少血清培养基中的30毫米玻璃底碟中的35分钟, 用0.25 毫升的质粒取代它转染试剂混合物加上额外的1.25 毫升的减少血清介质没有抗生素。减少血清培养基的目的是促进成功转染, 诱导原发性纤毛生长, 以及保持细胞存活足够长的时间进行实验。第二天将细胞返回孵化室进行实验。
注意: 当执行分子跟踪 IFT20, 一个质粒含有基因改造 IFT20 融合在其 C 总站到 GFP 是使用25。在执行分子跟踪 SSTR3, 一个质粒含有基因改造 SSTR3 融合在其 N 总站的受体肽 (AP) 域和 C 总站到 GFP 是使用22。除 SSTR3 结构外, 含有生物素连接比拉的质粒必须 co-expressed, 必须用10µM 生物素补充转染培养基。然后, 比拉将生物素附着在 ER 的新合成的 AP-SSTR3-GFP 分子的 AP 域上。Alexa647 共轭到三的四生物素结合位点上的亲和, 平均, 然后可以补充的媒体之前成像荧光标签的 AP-SSTR3-GFP 分子在外部表面的细胞22,27.该方法采用 GFP 和 AlexaFluor647;然而, 其他的荧光探针可以使用, 如果他们具有类似的高光稳定性和量子产量。 - 如果使用外部标记的 SSTR3 结构, 在实验前将培养基从玻璃底盘中取出1小时, 用1毫升磷酸盐缓冲盐水 (PBS) 洗涤5次, 并添加1毫升的还原血清培养基, 辅以1µM Alexa647 共轭亲和.
- 试验前不超过15分钟, 从玻璃底盘中取出培养基, 用1毫升 PBS 清洗转染和标记的细胞5次。
- 放置1毫升的成像缓冲器 (20 毫米 HEPES, 110 毫米 KOAc, 5 毫米 NaOAc, 2 毫米 MgOAc, 1 毫米 EGTA, pH 7.3) 在玻璃底盘。
注意: 在成像缓冲区中, 单元格的生存时间不超过3小时。因此, 每道菜只进行2小时的实验。
2. 速度显微镜
注: 速度显微镜设置包括一个倒置荧光显微镜配备了 1.4 NA 100×油浸泡物镜目标, 35 兆瓦 633 nm 氦氖激光器, 50 兆瓦固态 488 nm 和 561 nm 激光器, 片上乘法增益电荷耦合器件摄像机和用于数据采集和处理的显微镜软件包 (图 1)。对于单个通道成像, GFP、mCherry 和 Alexa647 分别为 488 nm、561 nm 或 633 nm 激光器所激发。对于单分子跟踪, 单点光照用于跟踪单个荧光标记的分子。对于荧光成像, 将凹透镜放置在激光光照路径中, 以将光束扩展到均匀的光照场。荧光发射是收集相同的目标, 过滤的一个分色过滤器 (405/488/561/635) 和一个发射过滤器 (405/488/561/635), 并与上述 CCD 相机的运行在500赫兹为单分子跟踪或2赫兹荧光成像。
- 将玻璃底板粘贴到显微镜的舞台上, 并找到一个适当表达所需构造的单元。一旦找到合适的细胞, 将原纤毛底部的 NPHP4-mCherry 点与成像平面上与激光单点照明对应的位置对准。
- 如果使用数字显微镜软件包 (参见材料表), 则在 "焦点控制" 窗口的 "相机" 选项卡中使用 "快照" 功能捕获 NPHP4-mCherry 和 IFT20-GFP 或 AP-SSTR3-GFP 的荧光图像。
注意: 这些图像将作为后续的单个分子位置的参考。 - 一旦获得了参考图像, 局部减少了标记的单一分子的浓度。照片漂白的1兆瓦激光照明二十年代或直到荧光强度接近的背景荧光。
注: 当精确浓度可控时, 使用 0.1-1 nM 标记的单分子。 - 为单分子跟踪准备, 减少激光照射功率〜0.15 兆瓦的单分子标记的 GFP 或〜0.5 兆瓦的分子标记的 Alexa647。
- 一旦激光功率和成像参数设置, 最大增益和强化和 2 ms 帧率, 为单分子成像, 从事适当的照明激光和记录 non-photobleached, 标记的单一分子, 因为它们被运输通过点击 "焦点控制" 窗口 "相机" 标签中的 "流" 按钮, 通过 photobleached 区域。
注: 不超过2分钟的视频应该被抓获, 以尽量减少的影响, 睫状漂移到一个微不足道的水平。 - 在捕获单分子视频后, 使用2D 高斯拟合算法处理视频, 如洛伊盖尔实验室的一瞥, 它精确地本地化了每个分子在一个包含感兴趣的区域的激发的质心。
- 选择所有的单一分子位置的精确 < 10 nm 和纠正的纤毛中心的基础上分布的单分子位置装有2D 高斯函数。
注: 使用2D 到3D 变换算法, IFT20-GFP 和 AP-SSTR3 路线的3D 传输路线分别在睫状纤毛或睫状膜上清晰显示。
3. 2D 到3D 转换
- 在纤毛中, 一旦收集了几千个定位的过渡分子 (信噪比 > 11), 选择纤毛的长轴作为 X 维度。制作位置的 Y 维度直方图, 并以 10 nm 增量获取 bin 总和。
注意: 2D 到3D 转换可以通过手工或任何软件或编程语言进行评估。作者成功地实现了在 Matlab 和 Python 2.7 中的转换。
结果
本节演示了从在原纤毛的 SSTR3 上执行速度显微镜获得的数据, 以研究由 15 nm 外部链接器连接到 Alexa647 (图 3a) 的传输路径。它为验证3D 变换算法的双重目的服务。Alexa647 应只标记主纤毛的外部表面, 因此, 3D 运输路线应在该位置显示高密度的运输路线。NIH-3T3 稳定表达 NPHP3-mCherry, 必须转染 AP-SSTR3-GFP 和孵化根据上述协议与亲和共轭 Alexa647。...
讨论
该协议描述了速度显微镜在初级纤毛的应用, 这是一种高度依赖高效蛋白质传输的细胞信号器官。速度显微镜可以提供高分辨率 (< 10 nm) 的位置为荧光标记分子, 因为他们通过单点照明中心的中间。此前它已被应用于研究通过 NPC 的蛋白质贩运6,7,8。然而, 它可以扩展到研究通过任何 sub-diffraction 细胞腔的贩运。该技术比其他高分?...
披露声明
作者声明没有利益冲突。
致谢
我们感谢 Dr. 克里斯汀 Verhey (密歇根大学, 安阿伯) 和 Dr. 格雷戈里 Pazour (马萨诸塞大学医学院) 提供一些质粒。该项目得到了国家卫生研究院 (NIH GM097037、GM116204 和 GM122552 W.Y.) 的资助。
材料
| Name | Company | Catalog Number | Comments |
| 25 cm2 tissue culture dish | Corning | VV-01936-00 | |
| Penicillin/streptomycin | ThermoFisher | 15140122 | |
| Fetal bovine serum | ThermoFisher | 10438018 | |
| DMEM | ThermoFisher | 10566-016 | |
| OPTIMEM | ThermoFisher | 31985062 | |
| Trypsin | ThermoFisher | 25300054 | |
| Phosphate buffered saline | Sigma-Aldrich | P3813-1PAK | |
| Transit LT1 | Mirus | MIR 2300 | |
| 35 mm glass bottom dish | MatTek | P35GCOL-0-14-C | |
| AlexaFluor 647-conjugated streptavidin | ThermoFisher | S21374 | |
| Biotin | Sigma-Aldrich | B4501-100MG | |
| 633 nm He-Ne laser | Melles Griot | 25-LHP-928-249 | |
| 561 nm solid state laser | Coherent | OBIS 561-50 LS | |
| 488 nm solid state laser | Coherent | 1185053 | |
| Inverted fluorescence microscope | Olympus | IX81 | |
| 1.4-NA 100× oil-immersion apochromatic objective | Olympus | UPLSAPO 100× | |
| On-chip multiplication gain charge-coupled-device camera | Roper Scientific | Cascade 128+ | |
| Dichroic filter | Semrock | Di01- R405/488/561/635-25x36 | |
| Emission filter | Semrock | NF01-405/488/561/635-25X5.0 | |
| Slidebook 6.0 | Intelligent Imaging Innovations | digital microscopy software |
参考文献
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annu Rev Biochem. 78, 993-1016 (2009).
- Leung, B. O., Chou, K. C. Review of super-resolution fluorescence microscopy for biology. Appl Spectrosc. 65, 967-980 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440, 935-939 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313, 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3, 793-796 (2006).
- Ma, J., Yang, W. Three-dimensional distribution of transient interactions in the nuclear pore complex obtained from single-molecule snapshots. Proc Natl Acad Sci USA. 107, 7305-7310 (2010).
- Ma, J., Goryaynov, A., Sarma, A., Yang, W. Self-regulated viscous channel in the nuclear pore complex. Proc Natl Acad Sci USA. 109, 7326-7331 (2012).
- Ma, J., et al. High-resolution three-dimensional mapping of mRNA export through the nuclear pore. Nat Comm. 4, (2013).
- Akey, C. W., Radermacher, M. Architecture of the Xenopus nuclear pore complex revealed by three-dimensional cryo-electron microscopy. J Cell Biol. 122, 1-19 (1993).
- Akey, C. W. Interactions and structure of the nuclear pore complex revealed by cryo-electron microscopy. J Cell Biol. 109, 955-970 (1989).
- Czarnecki, P. G., Shah, J. V. The ciliary transition zone: from morphology and molecules to medicine. Trends Cell Biol. 22, 201-210 (2012).
- Elf, J., Li, G. -. W., Xie, X. S. Probing transcription factor dynamics at the single-molecule level in a living cell. Science. 316, 1191-1194 (2007).
- Anzalone, A., Annibale, P., Gratton, E. 3D orbital tracking in a modified two-photon microscope: an application to the tracking of intracellular vesicles. J Vis Exp. , (2014).
- Ritter, J. G., Veith, R., Veenendaal, A., Siebrasse, J. P., Kubitscheck, U. Light sheet microscopy for single molecule tracking in living tissue. PloS one. 5, 11639 (2010).
- Marshall, W. F., Nonaka, S. Cilia: tuning in to the cell's antenna. Curr Biol. 16, 604-614 (2006).
- Scholey, J. M., Anderson, K. V. Intraflagellar transport and cilium-based signaling. Cell. 125, 439-442 (2006).
- Yang, T. T., et al. Superresolution pattern recognition reveals the architectural map of the ciliary transition zone. Sci Rep. 5, 14096 (2015).
- Craige, B., et al. CEP290 tethers flagellar transition zone microtubules to the membrane and regulates flagellar protein content. J Cell Biol. 190, 927-940 (2010).
- Kee, H. L., et al. A size-exclusion permeability barrier and nucleoporins characterize a ciliary pore complex that regulates transport into cilia. Nat Cell Biol. 14, 431-437 (2012).
- Najafi, M., Maza, N. A., Calvert, P. D. Steric volume exclusion sets soluble protein concentrations in photoreceptor sensory cilia. Proc Natl Acad Sci USA. 109, 203-208 (2012).
- Nachury, M. V., Seeley, E. S., Jin, H. Trafficking to the ciliary membrane: how to get across the periciliary diffusion barrier. Annu Rev Cell Dev Biol. 26, 59-87 (2010).
- Ye, F., et al. Single molecule imaging reveals a major role for diffusion in the exploration of ciliary space by signaling receptors. Elife. 2, 00654 (2013).
- Ross, A. J., et al. Disruption of Bardet-Biedl syndrome ciliary proteins perturbs planar cell polarity in vertebrates. Nat Genetics. 37, 1135-1140 (2005).
- Handel, M., et al. Selective targeting of somatostatin receptor 3 to neuronal cilia. Neuroscience. 89, 909-926 (1999).
- Follit, J. A., Tuft, R. A., Fogarty, K. E., Pazour, G. J. The intraflagellar transport protein IFT20 is associated with the Golgi complex and is required for cilia assembly. Mol Biol Cell. 17, 3781-3792 (2006).
- Awata, J., et al. NPHP4 controls ciliary trafficking of membrane proteins and large soluble proteins at the transition zone. J Cell Sci. 127, 4714-4727 (2014).
- Howarth, M., Ting, A. Y. Imaging proteins in live mammalian cells with biotin ligase and monovalent streptavidin. Nat Protoc. 3, 534-545 (2008).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy. Science. 319, 810-813 (2008).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。