A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
יישום של מיקרוסקופיית מהירות רזולוציה סופר מהיר ב ריסי העיקרי בשידור חי
In This Article
Summary
לאחרונה אנחנו ממופה המיקומים מרחבי תלת מימדי (3D) של נתיבי תחבורה חלבונים שונים translocating בתוך ראשי cilia בתאים חיים. כאן זה פרטים נייר את הגדרת הניסוי, התהליך של דגימות ביולוגיות, הניתוחים נתונים עבור קרינה פלואורסצנטית הרזולוציה סופר 3D הדמיה הגישה לאחרונה המוחלת בחיים cilia העיקרי.
Abstract
ריסי העיקרי הוא מבוסס microtubule בליטה על פני השטח של התאים האיקריוטים רבים והוא מכיל מהווה השלמה ייחודי של חלבונים שהפונקציה אנושות תא תנועתיות, איתות. מאז cilia אינם מסוגלים לסנתז חלבון משלהם, כמעט 200 חלבונים ciliary ייחודי צריך להיות הנסחר בין ציטוזול cilia העיקרי. עם זאת, זה עדיין אתגר טכני כדי למפות תלת מימדי (3D) מיקומים של תחבורה מסלולים עבור חלבונים אלה, חי ראשי cilia בגלל מגבלות הקיימת כיום טכניקות. לכבוש את האתגר, לאחרונה יש שפותחה ואנו המועסקים של מיקרוסקופ וירטואלי 3D super-resolution במהירות גבוהה, כינה מיקרוסקופ תת עקיפה (מהירות) נקודה אחת קצה-עירור, כדי לקבוע את המיקום המרחבי התלת-ממד של נתיבים לתחבורה שניהם cytosolic, קרום חלבונים ב- cilia העיקרי של תאים חיים. במאמר זה נדגים את ההתקנה מפורט של מהירות מיקרוסקופ, ההכנה של תאים המבטאים חלבונים ciliary זריחה-חלבון-שכותרתו ', את מעקב בזמן אמת אחר מולקולה בודדת של חלבונים בודדים ב ריסי בשידור חי, את ההישג של מפות צפיפות ההסתברות מרחבית תלת-ממד של נתיבי תחבורה ciliary חלבונים.
Introduction
מאז כאמור על ידי ארנסט אבה בשנת 1873, הרזולוציה של מיקרוסקופ אור קונבנציונלי יש כבר האמין להיות מוגבל כ 200 ננומטר עקב שבירת האור אובייקטיבית1,2. כיום, סופר-רזולוציה מיקרוסקופ אור טכניקות לשבור מגבלה זו ולאפשר לכידת תמונות דינאמית ברזולוציה תת עקיפה (< 200 ננומטר). הטכניקות בדרך כלל מתחלקים לשתי קטגוריות רחבות: גירוי פליטה דלדול (STED) מיקרוסקופיה המבוסס על גישות, המניבות עקיפה תת נפח תאורה עקב תגובת אופטית לא-לינאריות fluorophores דגימות3; מיקרוסקופ אור photoactivated (דקל), מיקרוסקופיה סטוכסטי שחזור אופטי (סערה)-המבוסס על שיטות זיהוי סופר, אשר מנצלים את פונקציות מתמטיות, ולהתאימם לעגה של centroids של fluorophores ואז לשקם את centroids האלה כדי ליצור סופר רזולוציה תמונות4,5. כיום, עקב הגדרת אופטי מסובכת יחסית, סערה ודקלים בהרחבה מועסקים על ידי הפעלת רק קבוצת משנה קטנה של fluorophores בכל מסגרת של סרטון ארוך של תכשיר ביולוגי. זה מאפשר עבור ההתאמה מדויקת יותר על ידי התאמה לפי עקומת גאוס 2D של הנקודה פלורסנט, כינה את הפונקציה הליין (PSF), שכותרתו fluorescently חלבונים בכל מסגרת של הוידאו. המיקום 2D של כל מולקולה עם התווית fluorescently ואז יכול להיות יונחו על מטוס הדמיה בודד כדי לייצר תמונת סופר רזולוציה של הכנה ביולוגי1,2. בזמן לוקליזציה מולקולה בודדת אלה, גישות סופר-רזולוציה מיקרוסקופיה בהחלט מהפכה כיצד בוצעה הדמיה של דגימות ביולוגיות, עדיין ישנם אתגרים להתגבר. לדוגמה, סערה ודקל ניתן להשיג החלטות המרחבי בצורה הטובה ביותר שלהם לאחר קיבוע של דגימות ביולוגיות, ובכך להציג ייצוג סטטי של החלבונים עם התווית fluorescently, אשר היא מגבלה דומה של מיקרוסקופ אלקטרונים. בנוסף, להשיג רזולוציה מרחבית גבוהה עבור כל חלבון הנקרא fluorescently בתאים חיים, דוגמאות חייב לדימות-framerates רב אשר אינם מסוגלים ללכוד dynamics חלבון. לכן, יש צורך להתגבר על מכשולים טכניים הראשי אלה.
כדי להשיג רזולוציה גבוהה ייתכן כי הוא מתאים היטב עבור זיהוי מהיר-המעבר חלבונים או RNAs בתאים חיים, פיתחנו רזולוציה סופר מהירות מיקרוסקופ ב שלנו מעבדה (איור 1)6,7, 8. ההתקדמות הטכנית מרכזיים במיקרוסקופ מהירות בעבר אפשרו לנו לעקוב בהצלחה nucleocytoplasmic התחבורה של מולקולות קטנות, חלבונים, mRNA ווירוסים דרך מקורית גרעיני הנקבוביות מתחמי (NPCs)6, 7 , 8. בקצרה, התכונות הבאות של מהירות מיקרוסקופ ישמש למעקב מהיר-המעבר מקרומולקולות דרך מבנים סימטריים rotationally מיקרומטר תת בתאים חיים, כגון NPCs cilia ראשי: (1) נוטה או תאורה אנכי PSF מאפשר את עירור של מולקולות יחיד בתוך אמצעי אחסון קטן עקיפה הגבלה ב מישור מוקד (איור 1); (2) PSF נוטה ניתן במידה רבה למנוע out-of-להתמקד פלורסצנטיות, ובכך לשפר את יחס אות לרעש. (3) הצפיפות האופטית של 100-500 קילוואט / ס מ2 ההארה PSF מאפשר אלפי פוטונים להיות שנאספו מ- fluorophores אחד עם זיהוי מהר למהירויות (> 500 Hz). (4) המהיר זיהוי מהירות מקטינה באופן משמעותי גם את השגיאה לוקליזציה המרחבי מולקולה בודדת (< 10 ננומטר) בקביעת את מסלולי המרחבי של מולקולות פלורסנט לגור תאים חיים, כי פעפוע הוא אחד הגורמים העיקריים גורם פגמים של מולקולה בודדת לוקליזציה להעברת מולקולות. (5) להקציע 2D ל 3D המרה אלגוריתמים מאפשרים לנו לספק מפות צפיפות ההסתברות מרחבית תלת-ממד של נתיבי תחבורה עבור מולקולות של NPC או את ריסי העיקרי. ראוי לציין כי תהליך ההמרה בין את קרטזי לבין מערכת תיאום גלילי משמש ליצירת צפיפות ההסתברות מרחבית תלת-ממד מפה ולא תלת-ממד יחיד מולקולה מעקב (איור 2). בעבר, מיקרוסקופ אלקטרונים נתונים נחשפו9,NPC10 ו ריסי הראשי11 לשניהם יש מבנה סימטרי rotationally. בעקרון, באופן אקראי לשדר המולקולות דרך NPC או ריסי הראשי צריך גם הפצות rotationally סימטרי. כפי שמוצג באיור2, מספר גבוה של אקראי לשדר מולקולות בתוך הגליל ליצור הפצות סימטרית rotationally-תצוגת חתך הרוחב עד כדי כך ב NPC, נוסף והתוצאה אחידה כ מרחבית הפצה בכל אזור משנה קטן מאוד בין שתי השכנות טבעות (איור 2E). התפלגות אחידה זה מוביל התפוצה המרחבית לאורך ממד θ באמצעות במערכת צירים היא קבועה. לאחר מכן ניתן לפשט את קואורדינטות תלת-ממד (R, X, θ באמצעות) להיות הקואורדינטות 2D (R, X, קבוע). למעשה, תהליך ההמרה בין קרטזי את המערכות גלילי הוא מ- 2D (X, Y) 2D (R, X, קבוע). Θ באמצעות מתמדת, מתייחס הצפיפות המרחבי p ב- איור 2E, מחושב באמצעות המשוואה A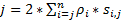 .
.
בסופו של דבר, מולקולה בודדת המעקב מצא יישום רחבות במחקר ביולוגי, לכן טבעי כי שפע של טכניקות יתפתחו למלא נישות ביולוגי מסוים12,13,14. כזה הוא המקרה עם מיקרוסקופ מהירות. בעבר, כאשר בשילוב עם אלגוריתם שינוי תלת-ממד, טכניקה זו פותחה כדי לפתור נתיבי תחבורה תלת-ממדי של מולקולות דרך NPCs, משנה-diffraction בגודל rotationally סימטרי ביולוגי ומבנה6הם מגיעים. בנייר זה, מוצגים cilia הראשי להיות דגם מעולה organelles גם כן. Cilia הראשי הם גלילי, כמו אנטנה organelles (~ 125 nm radius) המקרינים מפני השטח של ביותר בתרבית של תאים15,16,17. הם אחראים על קבלת אותות חיצוניים והעברת מענה תאיים הקשורים בדרך כלל צמיחה חילוף החומרים15,16. לכן, שטף של חלבונים מבניים, מיחזור של רצפטורים transmembrane, והעברת שליחי תאיים האחריות חיוני של cilia העיקרי. בצומת בין של cilia העיקרי לגוף התא היא מכשול סלקטיביות קריטיים, המכונה אזור המעבר או צ', שדרכו כל העברה חלבון זו חייבת להתבצע11,18,19, 20. בנוסף הפונקציה חסימה של למתחם, נחשבים לפחות שני תהליכים התחבורה, תחבורה intraflagellar, דיפוזיה פסיבית, להיות אחראי על התנועה של חלבון דרך זה אזור16,21, 22. מבחינת בריאות האדם, האובדן של cilia העיקרי, ורשות עוקבות של איתות במורד הזרם היא תכונה של סרטן רבים. בנוסף, מחלות גנטיות רבות, כגון תסמונת Bardet-Biedl, מחלת כליות פוליציסטיות, משויכים חלבון פגום תחבורה23. עקיפה תת מגבלת גודל והן את תהליך מורכב של חלבונים סלקטיבי התחבורה באמצעות למתחם להפוך cilia העיקרי זה ליעד עיקרי עבור טכניקה זו. בנייר זה שיטות, נדגים את המעקב אחר טראנסממברנלי ciliary, קולטני סומטוסטטין 3 (SSTR3)24, תווית חיצונית עם אלקסה עבור חיל הים 647, רכיב של: אי, IFT2025, המסומנת מולקולה GFP מאוחה.
Protocol
1. NIH-3T3 תא הכנה מהירות מיקרוסקופיה של מניות
- 1.5 שבועות מראש הניסוי, להתאושש תרבות טריים של תאים NIH-3T3 מניה קפוא על ידי מפשיר ב 37 מעלות צלזיוס והעברת התאים אל בקבוק התרבות התא של2 25 ס מ עם 3 מ"ל בינוני (DMEM ששינה הנשר של Dulbecco) בתוספת 110 מ"ג/מ"ל פירובט נתרן 2 מ מ גלוטמין, סרום שור עוברית 10%, 1% פניצילין/סטרפטומיצין.
- דגירה התאים ב- 37 מעלות צלזיוס ב חממה2 CO 5%.
- לפצל תאים ב- 80% confluency, על כל יומיים, לפחות שלוש פעמים לפני יום ניסיוני כדי להבטיח ההומוגניות של מחזור התא. Trypsinize תאים עם 0.25% טריפסין למשך 2 דקות ב 37 מעלות צלזיוס, תשאף טריפסין ולהחליף אותו עם 2 מ"ל של מדיום. פיפטה המדיום שוב ושוב כדי לשבור את התא אשכולות, להסיר את מספר התאים הרצויים ולהביא את הנפח הכולל של המדיה בחזרה עד 3 מ.
הערה: NIH-3T3 היו בעבר מהונדסים גנטית כדי להביע את NPHP-4, חלבון זה רגישה ל ט ז26, התמזגו התחנה הסופית C כדי mCherry. mCherry הוא fluorophore אשר יכול להיות שמחים עם תאורה nm 561 באופן כמותי בתרגום המכשול סלקטיביות ט ז, אוריינט cilia את ראשי. - יומיים לפני הניסוי, צלחת התאים 35 מ מ זכוכית תחתון דיש-60-70% confluency 1.5 מ של המדיום אותה כשלב 1.1 וחוזרים התאים החממה.
- יום אחד לפני הניסוי, מבחינה כימית transfect התאים עם פלסמיד הרצוי. לערבב 500-1000 ng של פלסמיד הרצויה (ראו הערה למטה) על יחס 1:2.5 עם ריאגנט תרביות תאים ב- 0.25 mL של מדיה מופחת סרום ללא אנטיביוטיקה במשך 30 דקות לשאוב מדיה מ- 35 מ מ זכוכית צלחת התחתון והחלף אותו פלסמיד 0.25 mL / תרביות תאים ריאגנט מיקס פלוס של mL 1.25 נוספת של מדיה מופחת סרום ללא אנטיביוטיקה. מדיה מופחת סרום משרת את המטרה להקל על תרביות תאים מוצלחת, גרימת cilia העיקרי לצמיחה, וכן להשאיר את התאים בחיים מספיק זמן כדי לבצע את הניסוי. לחזור תאים החממה לניסוי. למחרת היום.
הערה: בעת ביצוע המעקב מולקולה אחת של IFT20, פלסמיד המכיל ש-ift20 מהונדס גנטית התמזגו ב לתחנתו C ל GFP הוא בשימוש25. בעת ביצוע יחיד מולקולה מעקב של SSTR3, פלסמיד המכיל SSTR3 מהונדס גנטית התמזגו ב לתחנתו N לתחום פפטיד (AP) מקבל והוא טרמינוס C ל GFP בשימוש22. בנוסף הבונה SSTR3, פלסמיד המכיל את ליגאז ביוטין בירה חייבת להתבטא במשותף, התקשורת תרביות תאים חייב להיות בתוספת 10 מיקרומטר ביוטין. בירה מכן מצרף ביוטין לתחום AP של מולקולות AP-SSTR3-GFP לאחרונה מסונתז ברמה של המיון. Alexa647 מצומדת של האתרים מחייב ביוטין ארבע על streptavidin, שלושה בממוצע, ייתכן ואז ניתן להשלים את התקשורת לפני הדמיה לתווית fluorescently AP-SSTR3-GFP מולקולות על פני השטח החיצוני של התא22,27 . GFP, AlexaFluor647 נמצאים בשימוש בשיטה זו; עם זאת, אחרים הגששים פלורסנט יכול לשמש אם יש להם באופן דומה צילום-ביציבות גבוהה קוונטית תשואות. - אם באמצעות תווית חיצונית SSTR3 הבונה, הסר התקשורת מתחתית הכוס דיש h 1 לפני הניסוי, לשטוף את התא 5 פעמים עם 1 מ ל תמיסת באגירה פוספט (PBS), ולהוסיף 1 מ"ל של מדיה מופחת סרום בתוספת 1 מיקרומטר Alexa647 מצומדת streptavidin.
- לא יותר מ- 15 דקות לפני הניסוי, להסיר מדיה מן המנה התחתון זכוכית ולשטוף את התאים transfected ומתויגים 5 פעמים עם 1 מ"ל ל- PBS.
- מקום 1 מ"ל של מאגר הדמיה (20 מ מ HEPES, 110 מ מ KOAc, 5 מ מ NaOAc, 2 מ מ. MgOAc, 1 מ"מ EGTA, pH 7.3) המנה קרקעית זכוכית.
הערה: במאגר הדמיה, תאים הם קיימא לא יותר מ-3 שעות. לכן, רק 2 h של ניסויים מבוצעים על כל מנה.
2. מהירות מיקרוסקופ
הערה: ההגדרה של מיקרוסקופ מהירות כולל מיקרוסקופ פלורסצנטיות הפוכה מצויד 1.4-נה 100 × שמן-טבילה apochromatic מטרה, עם 35 mW 633 ננומטר הוא-Ne לייזר, 50 mW מצב מוצק 488 ננומטר, 561-nm לייזרים, רווח כפל על שבב תשלום-בשילוב-התקן מצלמה, חבילת תוכנה מיקרוסקופ רכישת נתונים ועיבוד (איור 1). עבור ערוץ נפרד הדמיה, שמחים GFP, mCherry ו- Alexa647 על ידי 488 ננומטר, 561 ננומטר, או לייזרים nm 633, בהתאמה. לצורך מעקב מולקולה בודדת, תאורה בנקודה אחת משמשת למעקב אחר מולקולות בודדות עם התווית fluorescently. עבור הדמיה epifluorescence, עדשה קעורה ממוקם בנתיב תאורה לייזר כדי להרחיב את הקרן לתוך שדה אחיד של תאורה. פליטת קרינה פלואורסצנטית שנאספו על ידי אותה מטרה, מסוננת על-ידי מסנן ודיקרואיק זוהר (405/488 561 635) והן של מסנן פליטה (405/488 561 635) הינם עם תמונה עם מצלמת CCD לעיל פועלים ב 500 הרץ למעקב מולקולה בודדת או 2 הרץ עבור הדמיה epifluorescence.
- מוספית צלחת התחתון זכוכית לשלב של המיקרוסקופ ולאתר תא כראוי מביע המבנה הרצוי. ברגע תא מתאים נמצאה, יישר את המקום NPHP4-mCherry בבסיס cilia ראשי עם המיקום על המטוס הדמיה המתאים להארה בנקודה אחת של הלייזר.
- ללכוד את התמונה epifluorescence של NPHP4-mCherry IFT20-GFP או AP-SSTR3-GFP באמצעות הפונקציה "Snap" בכרטיסיה "המצלמה" החלון "המוקד בקרות" אם באמצעות חבילת התוכנה מיקרוסקופ דיגיטלי (ראה טבלה של חומרים).
הערה: התמונות האלה תפעל כנקודת התייחסות המיקומים הבאים מולקולה בודדת. - ברגע הדימויים הפניה מתקבלים, באופן מקומי להפחית את ריכוז מולקולות יחיד עם תוויות. צילום-אקונומיקה למתחם עם תאורה לייזר mW 1 20 s או עד עוצמת קרינה פלואורסצנטית הוא קרוב לזה של רקע זריחה.
הערה: כאשר ריכוז מדויק יכול להיות נשלט, 0.1-1 ננומטר שכותרתו מולקולות יחיד משמשים. - כדי להתכונן מעקב מולקולה בודדת, להפחית את עוצמת תאורה הלייזר ~0.15 mW עבור מולקולות יחיד עם GFP תוויות או ~0.5 mW עבור מולקולות המסומנת Alexa647.
- ברגע את עוצמת הלייזר והדמיה פרמטרים מוגדרים, רווח מרבי, התעצמות, 2 מילי-שניות מסגרת שיעור, עבור הדמיה מולקולה בודדת, לעסוק הלייזר תאורה המתאימה ולהקליט הלא-photobleached, שכותרתו מולקולות יחיד כפי הם מועברים דרך אזור photobleached למתחם על-ידי לחיצה על לחצן "זרם" בכרטיסיה "המצלמה" חלון "המוקד שולט".
הערה: לא יותר מ 2 דקות של וידאו צריך להילכד כדי למזער את ההשפעות של סחיפה ciliary לרמה זניחה. - לאחר לכידת וידאו מולקולה בודדת, לעבד את קטעי וידאו באמצעות אלגוריתם 2D התאמה לפי עקומת גאוס, כגון מבט חטוף על ידי המעבדה גלס, אשר רגישה בדיוק את centroid של עירור כל מולקולה אחת של PSF באזור המקיף של עניין (AOI).
- בחר כל המיקומים מולקולה בודדת עם דיוק < 10 ננומטר ולתקן במרכז cilia בהתבסס על התפלגות של מולקולה בודדת מיקומים מצויד פונקציה גאוסיאנית 2D.
הערה: באמצעות 2D שינוי תלת-ממד בעזרת אלגוריתם, נתיבי תחבורה תלת-ממד של נתיבי IFT20-GFP ו- AP-SSTR3 מוצגים באופן ברור ciliary קרום axonemal או ciliary, בהתאמה.
3. 2D ל 3D המרה
- לאחר מספר localizations אלף עבור הם מגיעים מולקולות (אות לרעש יחס > 11) ב- ריסי נאספים, בחר לציר הארוך של ריסי הממד X. ליצור היסטוגרמה ממד Y של המיקומים והשג את הסכומים סל ב- nm 10 דרגות.
הערה: 2D ל 3D המרה עשוי להיות מוערך על ידי היד או כל תוכנה או שפת תכנות. המחברים יישמו בהצלחה את הטרנספורמציה Matlab והן 2.7 פייתון.
תוצאות
סעיף זה מדגים את הנתונים המתקבלים ביצוע מהירות מיקרוסקופיה-למתחם של cilia העיקרי ללמוד תוואי התחבורה SSTR3 מחובר באמצעות ~ 15 ננומטר מקשר חיצוני כדי Alexa647 (איור 3א). היא מגישה את המטרה הכפולה של אימות האלגוריתם שינוי תלת-ממדית. Alexa647 צריך רק תווית המשטח הח...
Discussion
פרוטוקול זה מתאר את היישום של מהירות מיקרוסקופ ריסי העיקרי, אברון איתות סלולרי זה מאוד מסתמך על תחבורה יעילה חלבון. מיקרוסקופ מהירות יכול לספק ברזולוציה גבוהה (< 10 ננומטר) מיקומים עבור מולקולות עם התווית fluorescently כפי שהם עוברים ההארה בנקודה אחת שבמרכזה למתחם. בעבר זה הוחל ללמוד החלבון סחר דר...
Disclosures
המחברים מצהירים שאין ניגודי אינטרסים.
Acknowledgements
אנו מודים ד ר קריסטן Verhey (אוניברסיטת מישיגן, אן ארבור) ואת ד ר גרגורי Pazour (בית הספר לרפואה אוניברסיטת מסצ'וסטס) על מתן כמה פלסמידים. הפרויקט נתמך על ידי מענקים המכון הלאומי לבריאות (NIH GM097037, GM116204 ו- GM122552 כדי W.Y.).
Materials
| Name | Company | Catalog Number | Comments |
| 25 cm2 tissue culture dish | Corning | VV-01936-00 | |
| Penicillin/streptomycin | ThermoFisher | 15140122 | |
| Fetal bovine serum | ThermoFisher | 10438018 | |
| DMEM | ThermoFisher | 10566-016 | |
| OPTIMEM | ThermoFisher | 31985062 | |
| Trypsin | ThermoFisher | 25300054 | |
| Phosphate buffered saline | Sigma-Aldrich | P3813-1PAK | |
| Transit LT1 | Mirus | MIR 2300 | |
| 35 mm glass bottom dish | MatTek | P35GCOL-0-14-C | |
| AlexaFluor 647-conjugated streptavidin | ThermoFisher | S21374 | |
| Biotin | Sigma-Aldrich | B4501-100MG | |
| 633 nm He-Ne laser | Melles Griot | 25-LHP-928-249 | |
| 561 nm solid state laser | Coherent | OBIS 561-50 LS | |
| 488 nm solid state laser | Coherent | 1185053 | |
| Inverted fluorescence microscope | Olympus | IX81 | |
| 1.4-NA 100× oil-immersion apochromatic objective | Olympus | UPLSAPO 100× | |
| On-chip multiplication gain charge-coupled-device camera | Roper Scientific | Cascade 128+ | |
| Dichroic filter | Semrock | Di01- R405/488/561/635-25x36 | |
| Emission filter | Semrock | NF01-405/488/561/635-25X5.0 | |
| Slidebook 6.0 | Intelligent Imaging Innovations | digital microscopy software |
References
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annu Rev Biochem. 78, 993-1016 (2009).
- Leung, B. O., Chou, K. C. Review of super-resolution fluorescence microscopy for biology. Appl Spectrosc. 65, 967-980 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440, 935-939 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313, 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3, 793-796 (2006).
- Ma, J., Yang, W. Three-dimensional distribution of transient interactions in the nuclear pore complex obtained from single-molecule snapshots. Proc Natl Acad Sci USA. 107, 7305-7310 (2010).
- Ma, J., Goryaynov, A., Sarma, A., Yang, W. Self-regulated viscous channel in the nuclear pore complex. Proc Natl Acad Sci USA. 109, 7326-7331 (2012).
- Ma, J., et al. High-resolution three-dimensional mapping of mRNA export through the nuclear pore. Nat Comm. 4, (2013).
- Akey, C. W., Radermacher, M. Architecture of the Xenopus nuclear pore complex revealed by three-dimensional cryo-electron microscopy. J Cell Biol. 122, 1-19 (1993).
- Akey, C. W. Interactions and structure of the nuclear pore complex revealed by cryo-electron microscopy. J Cell Biol. 109, 955-970 (1989).
- Czarnecki, P. G., Shah, J. V. The ciliary transition zone: from morphology and molecules to medicine. Trends Cell Biol. 22, 201-210 (2012).
- Elf, J., Li, G. -. W., Xie, X. S. Probing transcription factor dynamics at the single-molecule level in a living cell. Science. 316, 1191-1194 (2007).
- Anzalone, A., Annibale, P., Gratton, E. 3D orbital tracking in a modified two-photon microscope: an application to the tracking of intracellular vesicles. J Vis Exp. , (2014).
- Ritter, J. G., Veith, R., Veenendaal, A., Siebrasse, J. P., Kubitscheck, U. Light sheet microscopy for single molecule tracking in living tissue. PloS one. 5, 11639 (2010).
- Marshall, W. F., Nonaka, S. Cilia: tuning in to the cell's antenna. Curr Biol. 16, 604-614 (2006).
- Scholey, J. M., Anderson, K. V. Intraflagellar transport and cilium-based signaling. Cell. 125, 439-442 (2006).
- Yang, T. T., et al. Superresolution pattern recognition reveals the architectural map of the ciliary transition zone. Sci Rep. 5, 14096 (2015).
- Craige, B., et al. CEP290 tethers flagellar transition zone microtubules to the membrane and regulates flagellar protein content. J Cell Biol. 190, 927-940 (2010).
- Kee, H. L., et al. A size-exclusion permeability barrier and nucleoporins characterize a ciliary pore complex that regulates transport into cilia. Nat Cell Biol. 14, 431-437 (2012).
- Najafi, M., Maza, N. A., Calvert, P. D. Steric volume exclusion sets soluble protein concentrations in photoreceptor sensory cilia. Proc Natl Acad Sci USA. 109, 203-208 (2012).
- Nachury, M. V., Seeley, E. S., Jin, H. Trafficking to the ciliary membrane: how to get across the periciliary diffusion barrier. Annu Rev Cell Dev Biol. 26, 59-87 (2010).
- Ye, F., et al. Single molecule imaging reveals a major role for diffusion in the exploration of ciliary space by signaling receptors. Elife. 2, 00654 (2013).
- Ross, A. J., et al. Disruption of Bardet-Biedl syndrome ciliary proteins perturbs planar cell polarity in vertebrates. Nat Genetics. 37, 1135-1140 (2005).
- Handel, M., et al. Selective targeting of somatostatin receptor 3 to neuronal cilia. Neuroscience. 89, 909-926 (1999).
- Follit, J. A., Tuft, R. A., Fogarty, K. E., Pazour, G. J. The intraflagellar transport protein IFT20 is associated with the Golgi complex and is required for cilia assembly. Mol Biol Cell. 17, 3781-3792 (2006).
- Awata, J., et al. NPHP4 controls ciliary trafficking of membrane proteins and large soluble proteins at the transition zone. J Cell Sci. 127, 4714-4727 (2014).
- Howarth, M., Ting, A. Y. Imaging proteins in live mammalian cells with biotin ligase and monovalent streptavidin. Nat Protoc. 3, 534-545 (2008).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy. Science. 319, 810-813 (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved