Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Aplicación de la microscopía de resolución súper alta velocidad velocidad en vivo cilio primario
En este artículo
Resumen
Recientemente nos asignan la ubicación espacial (3D) tridimensional de rutas de transporte para diversas proteínas translocación dentro de cilios primarios en células vivas. Aquí en este detalles de papel la configuración experimental, el proceso de muestras biológicas y el análisis de datos para la fluorescencia de super-resolución 3D imagen enfoque recién aplicados en viven cilios primarios.
Resumen
El cilio primario es una protrusión de microtubule-basado en la superficie de muchas células eucariotas y contiene un complemento único de proteínas esa función crítica en la motilidad celular y señalización. Cilios son incapaces de sintetizar su propia proteína, cerca de 200 proteínas ciliares únicas necesitan ser objeto de trata entre el citosol y los cilios primarios. Sin embargo, sigue siendo un desafío técnico para asignar lugares de (3D) tridimensionales de vías de transporte de estas proteínas en vivo cilios primarios debido a las limitaciones existentes en la actualidad técnicas. Para conquistar el reto, recientemente hemos desarrollado y emplearon un microscopio de alta velocidad super-resolución 3D virtual, llamado microscopia de la secundario-difracción (velocidad) de solo punto borde-excitación, para determinar la ubicación espacial 3D de vías de transporte para tanto citosólica y proteínas de la membrana de los cilios primarios de células vivas. En este artículo, le mostraremos la configuración detallada de la microscopia de la velocidad, la preparación de las células que expresan proteínas ciliares marcado con la proteína de la fluorescencia, el seguimiento en tiempo real de una sola molécula de proteínas individuales en vivo cilio y el logro de de densidad de probabilidad espacial 3D mapas de rutas de transporte ciliar proteínas.
Introducción
Como afirma Ernst Abbe en 1873, la resolución de la microscopía de luz convencional ha sido cree que limitada a aproximadamente 200 nm debido a la difracción de la luz del objetivo1,2. En la actualidad, técnicas de microscopía de superresolución rompen esta limitación y permiten la captura de imágenes dinámicas con la resolución de la secundario-difracción (< 200 nm). Las técnicas generalmente caen en dos amplias categorías: estimuló la emisión (STED) el agotamiento basado en la microscopia enfoques, generan volumen de iluminación sub-difracción debido a la respuesta óptica no lineal de fluoróforos en muestras3; y microscopia ligera fotoactivado (PALM) y microscopía óptica estocástica de la reconstrucción (tormenta)-basado en técnicas de súper-resolución, que utilizan funciones matemáticas para localizar los centroides de fluoróforos y luego reconstituir estos centroides para formar imágenes de súper-resolución4,5. Actualmente, debido a la configuración óptica relativamente sin complicaciones, Palma y tormenta son ampliamente empleados por activar sólo un pequeño subconjunto de fluoróforos en cada fotograma de un video largo de una preparación biológica. Esto permite la localización más exacta por ajuste gaussiano 2D del spot fluorescente, denominada la función de extensión de punto (PSF), de proteínas marcada con fluorescencia en cada fotograma del vídeo. La ubicación 2D de cada molécula marcada con fluorescencia puede entonces se superponen en un solo plano de proyección de imagen para producir una imagen de súper-resolución de la preparación biológica1,2. Mientras estos localización de una sola molécula, enfoques de super-resolución microscopía revolucionaron sin duda cómo se realizó la proyección de imagen de las muestras biológicas, todavía hay retos que superar. Por ejemplo, tormenta y la palma pueden alcanzar sus mejores resoluciones espaciales después de la fijación de muestras biológicas y así presentar una representación estática de las proteínas marcada con fluorescencia, que es una limitación similar de la microscopia electrónica. Además, para conseguir alta resolución espacial para cada proteína marcada con fluorescencia en células vivas, las muestras deben ser reflejadas en tasa de fotogramas muy largo que es capaces de captar la dinámica de la proteína. Por lo tanto, es necesario superar estos obstáculos técnicos principales.
Para obtener una alta resolución espacio-temporal que está bien preparado para detectar movimiento rápido proteínas o ARN en células vivas, hemos desarrollado la microscopía de superresolución velocidad en nuestro laboratorio (figura 1)6,7, 8. varios avances técnicos importantes en microscopía de velocidad previamente nos han permitido rastrear con éxito transporte NUCLEOCITOPLASMÁTICO de pequeñas moléculas, proteínas, mRNA y virus a través de nativos nuclear poro complejos (PNJ)6, 7 , 8. brevemente, se utilizarán las siguientes características de la microscopia de la velocidad para seguir el rápido movimiento de macromoléculas a través de estructuras rotationally simétrico secundario-micrómetro en células vivas, como NPCs y cilios primarios: (1) una inclinación o un iluminación vertical PSF permite la excitación de las moléculas individuales dentro de un volumen pequeño del límite de difracción en el plano focal (figura 1); (2) el PSF inclinado puede evitar grandemente la fluorescencia fuera de enfoque y así mejorar la relación señal a ruido. (3) la densidad óptica de 100-500 kW / cm2 en la iluminación PSF permite miles de fotones a recogerse en solo fluoróforos con velocidades de detección rápida (> 500 Hz). Velocidad de la detección (4) el ayuno también reduce el error de localización espacial de una sola molécula (< 10 nm) en la determinación de las trayectorias espaciales de mover moléculas fluorescentes en células vivas, ya que la difusión molecular es uno de los principales factores que causan las imperfecciones de la localización de una sola molécula de moléculas en movimiento. Algoritmos de transformación (5) consolidada 2D a 3D nos permiten ofrecer mapas de densidad de probabilidad espacial 3D de rutas de transporte para las moléculas en el NPC o el cilio primario. Cabe mencionar que nuestro proceso de conversión entre el cartesiano y el sistema de coordinación cilíndrico se usa para generar una densidad de probabilidad espacial 3D mapa en lugar de 3D sola molécula seguimiento (figura 2). Anteriormente, datos de microscopía electrónica han revelado que el NPC9,10 y el cilio primario11 ambos tienen una estructura de simetría de rotación. En principio, difusión al azar de moléculas en movimiento a través del NPC o cilio primario debe tener distribuciones rotationally simétricas. Como se muestra en la figura 2, un número elevado de difusión al azar de las moléculas dentro del cilindro podría generar distribuciones rotationally simétricas en la vista de corte transversal que en el NPC, aún más dando como resultado un aproximadamente uniforme espacial distribución dentro de cada subregión muy pequeño entre dos anillos vecinos (figura 2E). Esta distribución uniforme conduce que la distribución espacial a lo largo de la dimensión θ en el sistema cilíndrico es constante. Entonces las coordenadas 3D (X, R, θ) se pueden simplificar para las coordenadas 2D (R, X, constante). En realidad, nuestro proceso de conversión entre el cartesiano y los sistemas cilíndricos es de 2D (X, Y) 2D (R, X, constante). El θ constante, se refiere a la densidad espacial p en la figura 2E, se calcula utilizando la ecuación A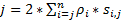 .
.
En definitiva, una sola molécula seguimiento tiene amplia aplicación en la investigación biológica, por lo tanto, es natural que se desarrollará una gran cantidad de técnicas para llenar nichos biológicos específicos12,13,14. Tal es el caso de microscopía de la velocidad. Anteriormente, cuando se combina con un algoritmo de transformación 3D, esta técnica se desarrolló para resolver rutas de transporte 3D de moléculas a través de los PNJs, una estructura biológica rotationally simétricos y sub-diffraction-tamaño6en tránsito. En este papel, cilios primarios aparecen ser organelos excelente modelo así. Cilios primarios son orgánulos cilíndricos, antena-como (~ 125 radio de nm) que se proyectan desde la superficie de las células mamíferas más15,16,17. Son responsables de recibir las señales externas y transmitir una respuesta intracelular típicamente asociada con el crecimiento y metabolismo15,16. Por lo tanto, del flujo de proteínas estructurales, reciclaje de receptores transmembranales y la transmisión de los mensajeros intracelulares son responsabilidades vitales de los cilios primarios. En la coyuntura entre los cilios primarios y el cuerpo celular es una barrera crítica de la selectividad, llamada zona de transición o TZ, a través del cual este transporte de la proteína debe ocurrir11,18,19, 20. Además de la función bloquea de la TZ, al menos dos procesos de transporte, Transporte intraflagelar y difusión pasiva, se cree que son responsables del movimiento de la proteína a través de esta región16,21, 22. desde un punto de vista de salud humana, la pérdida de cilios primarios y posterior desregulación de señalización corriente abajo es característica de muchos tipos de cáncer. Además, muchas enfermedades genéticas, como síndrome de Bardet-Biedl y enfermedad de riñón policística, están asociadas con la proteína defectuosa transporte23. El tamaño límite de sub- difracción y el complejo proceso de transporte de la proteína selectiva a través de la TZ hacen los cilios primarios el primer objetivo de esta técnica. En este trabajo métodos, demostramos el seguimiento de una proteína transmembrana ciliar, receptor de somatostatina 3 (SSTR3)24, marcado externamente con un componente de IFT, IFT2025, marcado con una molécula GFP fusionada y Alexa Fluor 647.
Protocolo
1. NIH-3T3 preparación de células para la microscopia de la velocidad de la acción
- 1,5 semanas antes el experimento, recupera un cultivo fresco de las células NIH-3T3 de un stock congelado descongelación a 37 º C y transfiriendo las células a un matraz de cultivo celular de 25 cm2 con 3 mL de medio modificado Eagle de Dulbecco (DMEM) suplementado con 110 mg/mL piruvato de sodio, 2 mM glutamina, suero bovino fetal 10% y 1% de penicilina/estreptomicina.
- Incube las células a 37 ° C en un incubador de 5% CO2 .
- Dividir celdas en el 80% de confluencia, sobre cada dos días, por lo menos tres veces antes del día experimental para asegurar la homogeneidad del ciclo celular. Trypsinize células con tripsina 0.25% por 2 min a 37 ° C, Aspire tripsina y reemplazarlo con 2 mL de medio. Pipeta el medio repetidamente para romper para arriba racimos de célula, quitar el número deseado de células y llevar el volumen total de los medios de comunicación de nuevo hasta 3 mL.
Nota: NIH-3T3 previamente fueron genéticamente modificados para expresar entre-4, una proteína que se localiza a la TZ26, fundida en la terminal C a mCherry. mCherry es un fluoróforo que puede ser excitado con 561 iluminación nm localizar cuantitativamente la barrera de la selectividad TZ y orientar los cilios primarios. - Dos días antes del experimento, placa las células en un fondo de cristal de 35 mm plato en confluencia de 60-70% con 1,5 mL del mismo medio como paso 1.1 y vuelva las células a la incubadora.
- Un día antes del experimento, químicamente transfectar las células con el plásmido deseado. Mezclar 500-1000 ng de plásmido deseado (ver Nota más abajo) en una proporción de 1: 2.5 con el reactivo de transfección en 0,25 mL de suero reducido medio sin antibióticos durante 30 minutos medios aspirado desde los 35 mm de vidrio, plato de fondo y reemplazarlo con el plásmido de 0.25 mL / mezcla de reactivo de transfección y una extra 1,25 mL de medios suero reducido sin antibióticos. Suero reducido los medios de comunicación sirven para facilitar una exitosa transfección, induciendo el crecimiento de cilios primarios, así como mantener las células vivas lo suficiente para llevar a cabo el experimento. Vuelva las células a la incubadora para el experimento al día siguiente.
Nota: Cuando se realiza el seguimiento de una sola molécula de IFT20, un plásmido que contiene que un IFT20 genéticamente fusionado en su terminal C a GFP es usada25. Cuando se realiza una sola molécula de SSTR3, un plásmido que contiene un SSTR3 genéticamente fusionado en su N terminal a un dominio de receptor péptido (AP) y terminal C a GFP es usado22. Además de la construcción de SSTR3, un plásmido que contiene el ligase de la biotina BirA debe expresarse conjuntamente y los medios de comunicación de transfección deben complementarse con biotina μm 10. BirA luego conecta biotina el dominio AP de recién sintetizadas moléculas AP-SSTR3-GFP en el nivel de la sala de emergencia. Alexa647 conjugado a tres de los cuatro sitios de unión a biotina de estreptavidina, en promedio, puede completarse entonces a los medios de comunicación antes de la proyección de imagen a la etiqueta fluorescente las moléculas AP-SSTR3-GFP en la superficie externa de la célula22,27 . GFP y AlexaFluor647 se utilizan en este método; sin embargo, otras sondas fluorescentes pueden utilizarse si tienen semejantemente alta foto-estabilidad y rendimiento cuántico. - Si utiliza el constructo SSTR3 externamente marcados, eliminar los medios de comunicación de la parte inferior de vidrio plato 1 h antes del experimento, lavan la celda 5 veces con 1 mL de tampón fosfato salino (PBS) y añadir 1 mL de suero reducidos medios suplementados con 1 μm Alexa647 conjugado estreptavidina.
- No más de 15 minutos antes del experimento, eliminar los medios de comunicación desde el plato de fondo de cristal y lave las células transfected y etiquetadas 5 veces con 1 mL de PBS.
- Coloque 1 mL de tampón de proyección de imagen (20 mM HEPES, 110 mM KOAc, 5 m m de NaOAc, MgOAc, de 2 mM 1 mM EGTA, pH 7.3) en el plato de fondo de vidrio.
Nota: En el buffer de imagen, las células son viables por no más de 3 h. Por lo tanto, sólo 2 h de experimentos se realizan en cada plato.
2. microscopia de velocidad
Nota: La configuración de la microscopia de velocidad incluye un microscopio de fluorescencia invertido equipado con un objetivo apocromática 1,4-NA 100 x inmersión en aceite, un láser de He-Ne de nm 35 que se 633 mW, 50 mW 488 nm y 561-nm láser de estado sólido, un aumento de la multiplicación de la en-viruta dispositivo de carga acoplada la cámara y un paquete de software del microscopio para adquisición de datos y el procesamiento (figura 1). Para la proyección de imagen de canal individual, GFP, mCherry y Alexa647 son excitados por 488 nm, 561 nm, o 633 nm, respectivamente. Para el rastreo de molécula única, único punto de iluminación se utiliza para rastrear moléculas individuales marcada con fluorescencia. Para la proyección de imagen de epifluorescencia, se coloca una lente cóncava en el camino de iluminación láser para expandir el haz en un campo uniforme de la iluminación. Las emisiones de fluorescencia son recogidas por el mismo objetivo, filtradas por un filtro dicroico (405/488/561/635) y un filtro de emisión (405/488/561/635) y fotografiadas con la cámara CCD arriba operando a 500 Hz para el seguimiento de la molécula sola o 2 Hz para proyección de imagen de epifluorescencia.
- Fije la placa inferior a la etapa del microscopio y localizar una célula que expresa correctamente las construcciones deseadas. Una vez que se ha encontrado un celular adecuado, alinee el punto mCherry NPHP4 en la base de los cilios primarios con la ubicación en el plano de proyección de imagen que corresponde a la iluminación del punto único del láser.
- Capturar una imagen de epifluorescencia de mCherry NPHP4 y IFT20-GFP o AP-SSTR3-GFP utilizando la función "Snap" en la pestaña de "Cámara" de la ventana de "Controles de enfoque" Si utiliza el paquete de software de microscopia digital (véase Tabla de materiales).
Nota: Estas imágenes actuará como referencia para la ubicación subsiguiente a la sola molécula. - Una vez que se obtienen las imágenes de referencia, localmente reducen la concentración de las moléculas individuales etiquetadas. Foto-blanquee el TZ con 1 mW láser de iluminación para 20 s o hasta que la intensidad de fluorescencia es cercano de la fluorescencia de fondo.
Nota: Cuando la concentración precisa puede ser controlada, 0.1-1 nM etiquetada las moléculas individuales se utilizan. - Para prepararse para el seguimiento de molécula simple, reducir el poder de iluminación láser de ~0.15 mW para las moléculas individuales con GFP o ~0.5 mW para moléculas con Alexa647.
- Tan pronto como la potencia del láser y los parámetros de la proyección de imagen son conjunto, máxima ganancia e intensificación y velocidad de fotogramas de 2 ms, imagen de molécula única, realizar el láser de iluminación adecuada y registrar no photobleached, etiquetado como moléculas individuales que son transportados a través de la región de photobleached de la TZ haciendo clic en el botón "Stream" en la pestaña de "Cámara" de la ventana de "Controles de foco".
Nota: No más de 2 minutos de video debe ser capturado para minimizar los efectos de ciliar deriva a un nivel insignificante. - Después de capturar el video de sola molécula, procesa los videos usando un algoritmo de ajuste gaussiano 2D, como la visión por el laboratorio de Gelles, que precisamente se localiza el centroide de la excitación de cada molécula individual PSF en una zona que abarca de interés (AOI).
- Seleccione todas las ubicaciones de molécula única con precisión < 10 nm y corregir el centro de cilios basado en la distribución de localidades de molécula única dispone de una función Gaussiana 2D.
Nota: Usando 2D para algoritmo de transformación 3D, las rutas de transporte 3D de rutas IFT20-GFP y AP-SSTR3 claramente aparecen en la membrana ciliar ciliar o axonemal, respectivamente.
3. 2D a 3D transformación
- Una vez que se recogen varias miles localizaciones de moléculas (señal a ruido ratio > 11) en tránsito en el cilio, seleccione el eje largo del cilio como la dimensión de X. Hacer un histograma de dimensión Y de los lugares y obtener las sumas de bin en 10 incrementos de nm.
Nota: El 2D a 3D transformación pueden evaluarse por la mano o cualquier software o lenguaje de programación. Los autores han aplicado con éxito la transformación en Matlab y Python 2.7.
Resultados
Esta sección muestra los datos obtenidos de la realización de microscopia de la velocidad en la TZ de cilios primarios para el estudio de la ruta de transporte de SSTR3 conectados por un ~ 15 nm externos vinculador Alexa647 (figura 3A). Sirve el doble propósito de verificar el algoritmo de transformación 3D. Alexa647 debe sólo etiqueta la superficie externa del cilio primario y por lo tanto, la ruta de transporte 3D debe revelar una ruta...
Discusión
Este protocolo describe el uso de la microscopia de velocidad para el cilio primario, un orgánulo celular señalización que es altamente dependiente sobre el transporte eficiente de la proteína. Microscopia de velocidad puede proporcionar alta resolución ubicaciones (< 10 nm) para las moléculas marcada con fluorescencia a medida que pasan a través de la iluminación de punto único centrada en el TZ. Previamente se ha aplicado para estudiar la proteína trata mediante el NPC6,
Divulgaciones
Los autores no declaran conflictos de interés.
Agradecimientos
Agradecemos a Dr. Kristen Verhey (Universidad de Michigan, Ann Arbor) y el Dr. Gregory Pazour (Facultad de medicina de la Universidad de Massachusetts) para proporcionar algunos plásmidos. El proyecto fue apoyado por subvenciones de los institutos nacionales de salud (NIH GM097037, GM116204 y GM122552 a W.Y.).
Materiales
| Name | Company | Catalog Number | Comments |
| 25 cm2 tissue culture dish | Corning | VV-01936-00 | |
| Penicillin/streptomycin | ThermoFisher | 15140122 | |
| Fetal bovine serum | ThermoFisher | 10438018 | |
| DMEM | ThermoFisher | 10566-016 | |
| OPTIMEM | ThermoFisher | 31985062 | |
| Trypsin | ThermoFisher | 25300054 | |
| Phosphate buffered saline | Sigma-Aldrich | P3813-1PAK | |
| Transit LT1 | Mirus | MIR 2300 | |
| 35 mm glass bottom dish | MatTek | P35GCOL-0-14-C | |
| AlexaFluor 647-conjugated streptavidin | ThermoFisher | S21374 | |
| Biotin | Sigma-Aldrich | B4501-100MG | |
| 633 nm He-Ne laser | Melles Griot | 25-LHP-928-249 | |
| 561 nm solid state laser | Coherent | OBIS 561-50 LS | |
| 488 nm solid state laser | Coherent | 1185053 | |
| Inverted fluorescence microscope | Olympus | IX81 | |
| 1.4-NA 100× oil-immersion apochromatic objective | Olympus | UPLSAPO 100× | |
| On-chip multiplication gain charge-coupled-device camera | Roper Scientific | Cascade 128+ | |
| Dichroic filter | Semrock | Di01- R405/488/561/635-25x36 | |
| Emission filter | Semrock | NF01-405/488/561/635-25X5.0 | |
| Slidebook 6.0 | Intelligent Imaging Innovations | digital microscopy software |
Referencias
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annu Rev Biochem. 78, 993-1016 (2009).
- Leung, B. O., Chou, K. C. Review of super-resolution fluorescence microscopy for biology. Appl Spectrosc. 65, 967-980 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440, 935-939 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313, 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3, 793-796 (2006).
- Ma, J., Yang, W. Three-dimensional distribution of transient interactions in the nuclear pore complex obtained from single-molecule snapshots. Proc Natl Acad Sci USA. 107, 7305-7310 (2010).
- Ma, J., Goryaynov, A., Sarma, A., Yang, W. Self-regulated viscous channel in the nuclear pore complex. Proc Natl Acad Sci USA. 109, 7326-7331 (2012).
- Ma, J., et al. High-resolution three-dimensional mapping of mRNA export through the nuclear pore. Nat Comm. 4, (2013).
- Akey, C. W., Radermacher, M. Architecture of the Xenopus nuclear pore complex revealed by three-dimensional cryo-electron microscopy. J Cell Biol. 122, 1-19 (1993).
- Akey, C. W. Interactions and structure of the nuclear pore complex revealed by cryo-electron microscopy. J Cell Biol. 109, 955-970 (1989).
- Czarnecki, P. G., Shah, J. V. The ciliary transition zone: from morphology and molecules to medicine. Trends Cell Biol. 22, 201-210 (2012).
- Elf, J., Li, G. -. W., Xie, X. S. Probing transcription factor dynamics at the single-molecule level in a living cell. Science. 316, 1191-1194 (2007).
- Anzalone, A., Annibale, P., Gratton, E. 3D orbital tracking in a modified two-photon microscope: an application to the tracking of intracellular vesicles. J Vis Exp. , (2014).
- Ritter, J. G., Veith, R., Veenendaal, A., Siebrasse, J. P., Kubitscheck, U. Light sheet microscopy for single molecule tracking in living tissue. PloS one. 5, 11639 (2010).
- Marshall, W. F., Nonaka, S. Cilia: tuning in to the cell's antenna. Curr Biol. 16, 604-614 (2006).
- Scholey, J. M., Anderson, K. V. Intraflagellar transport and cilium-based signaling. Cell. 125, 439-442 (2006).
- Yang, T. T., et al. Superresolution pattern recognition reveals the architectural map of the ciliary transition zone. Sci Rep. 5, 14096 (2015).
- Craige, B., et al. CEP290 tethers flagellar transition zone microtubules to the membrane and regulates flagellar protein content. J Cell Biol. 190, 927-940 (2010).
- Kee, H. L., et al. A size-exclusion permeability barrier and nucleoporins characterize a ciliary pore complex that regulates transport into cilia. Nat Cell Biol. 14, 431-437 (2012).
- Najafi, M., Maza, N. A., Calvert, P. D. Steric volume exclusion sets soluble protein concentrations in photoreceptor sensory cilia. Proc Natl Acad Sci USA. 109, 203-208 (2012).
- Nachury, M. V., Seeley, E. S., Jin, H. Trafficking to the ciliary membrane: how to get across the periciliary diffusion barrier. Annu Rev Cell Dev Biol. 26, 59-87 (2010).
- Ye, F., et al. Single molecule imaging reveals a major role for diffusion in the exploration of ciliary space by signaling receptors. Elife. 2, 00654 (2013).
- Ross, A. J., et al. Disruption of Bardet-Biedl syndrome ciliary proteins perturbs planar cell polarity in vertebrates. Nat Genetics. 37, 1135-1140 (2005).
- Handel, M., et al. Selective targeting of somatostatin receptor 3 to neuronal cilia. Neuroscience. 89, 909-926 (1999).
- Follit, J. A., Tuft, R. A., Fogarty, K. E., Pazour, G. J. The intraflagellar transport protein IFT20 is associated with the Golgi complex and is required for cilia assembly. Mol Biol Cell. 17, 3781-3792 (2006).
- Awata, J., et al. NPHP4 controls ciliary trafficking of membrane proteins and large soluble proteins at the transition zone. J Cell Sci. 127, 4714-4727 (2014).
- Howarth, M., Ting, A. Y. Imaging proteins in live mammalian cells with biotin ligase and monovalent streptavidin. Nat Protoc. 3, 534-545 (2008).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy. Science. 319, 810-813 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados