Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Anwendung des High-Speed-Höchstauflösung Geschwindigkeit Mikroskopie in Live primäre Zilie
In diesem Artikel
Zusammenfassung
Vor kurzem abgebildet wir die dreidimensionale (3D) räumlichen Positionen der Transportwege für verschiedene Proteine in primäre Zilien in lebenden Zellen translocating. Hier leben diese Papier-Details, die in der Versuchsanordnung, den Prozess der biologischen Proben und der Datenanalyse für die 3D Höchstauflösung Fluoreszenz imaging Ansatz neu angewendet primäre Zilien.
Zusammenfassung
Die primäre Zilie ist eine Vorwölbung Mikrotubuli-basierte auf der Oberfläche vieler eukaryotischer Zellen und enthält einer einzigartige Ergänzung der Proteine diese Funktion kritisch in Zelle Motilität und Signalisierung. Da Zilien nicht in der Lage eigene Protein zu synthetisieren sind, müssen fast 200 einzigartige ciliary Proteine zwischen dem Cytosol und primäre Zilien verschleppt werden. Es ist jedoch immer noch eine technische Herausforderung für dreidimensionale (3D) Standorte der Transportwege für diese Proteine in live primäre Zilien aufgrund der Einschränkungen der derzeit bestehenden Karte Techniken. Um die Herausforderung zu erobern, haben vor kurzem wir entwickelt und beschäftigt eine High-Speed-virtuelle 3D Höchstauflösung Mikroskopie, Einpunkt-Kante-Erregung Sub Beugung (Geschwindigkeit) Mikroskopie, um festzustellen, die 3D räumliche Lage der Transportwege für bezeichnet Beide cytosolischen und Membranproteine in primäre Zilien von lebenden Zellen. In diesem Artikel zeigen wir die detaillierte Setup SPEED-Mikroskopie, die Vorbereitung der Zellen mit dem Ausdruck Fluoreszenz-Protein-Label ciliary Proteine, die Echtzeitverfolgung der Einzelmolekül-einzelne Proteine in live Zilie und die Erreichung Karten 3D räumliche Wahrscheinlichkeitsdichte der Transportwege für ciliary Proteine.
Einleitung
Da Ernst Abbe im Jahre 1873 erklärte, hat die Auflösung der konventionellen Lichtmikroskopie angenommen wurde, beschränkt sich auf etwa 200 nm durch Lichtbeugung aus dem Ziel1,2. Derzeit Höchstauflösung lichtmikroskopische Techniken brechen diese Einschränkung und ermöglicht die Erfassung von dynamischen Bildern mit Sub-Beugung (< 200 nm) Auflösung. Die Techniken in der Regel fallen in zwei Hauptkategorien: stimulierte Emission Depletion (STED) Mikroskopie-basierte Ansätze, die Sub-Beugung Beleuchtung Volumen durch nichtlineare optische Reaktion des Fluorophore Proben3; generieren und photoaktiviert Lichtmikroskopie (PALM) und stochastische optische Rekonstruktion Mikroskopie (STORM)-basierte Höchstauflösung Techniken, die mathematische Funktionen zum Zentroiden des Fluorophore lokalisieren und dann rekonstruieren diese Zentroide nutzen Super-Resolution Bilder4,5zu bilden. Derzeit sind aufgrund der relativ unkomplizierten optische Aufbau PALM und Sturm ausgiebig beschäftigt durch die Aktivierung nur einer kleinen Teilmenge der Fluorophore in jedem Frame ein langes Video eines biologischen Präparates. Dies ermöglicht genauere Lokalisierung von 2D "glockenförmig" Einbau von fluoreszierenden Ort bezeichnet die Point-spread-Funktion (PSF), Fluoreszent-markierten Proteine in jedem Frame des Videos. Die 2D Position jedes Molekül Eindringmittel beschriftet kann dann auf einen einzigen Abbildungsebene Höchstauflösung Bilder des biologischen Vorbereitung1,2überlagert werden. Während diese Lokalisierung Einzelmolekül-Höchstauflösung Ansätze zur Mikroskopie sicherlich revolutioniert wie Bildgebung von biologischen Proben durchgeführt wurde, gibt es noch Herausforderungen zu bewältigen. Beispielsweise können Sturm und PALM erreichen ihre beste räumlicher Auflösung nach der Fixierung von biologischen Proben und so präsentieren eine statische Darstellung der Fluoreszent-markierten Proteine, die eine ähnliche Einschränkung der Elektronenmikroskopie. Darüber hinaus müssen die Erreichung hohen räumlichen Auflösung für jedes Fluoreszent-markierten Protein in lebenden Zellen, Proben bei sehr langen Frameraten abgebildet werden die Proteindynamik erfassen können. Daher ist es notwendig, diese wichtigsten technische Hürden zu überwinden.
Um eine hohe räumlich-zeitliche Auflösung zu erhalten, die gut geeignet für die Erkennung von schnelllebigen Proteine oder RNA in lebenden Zellen ist, entwickelten wir Höchstauflösung Geschwindigkeit Mikroskopie in unserem Labor (Abbildung 1)6,7, 8. mehrere große technische Fortschritte in der Geschwindigkeit Mikroskopie haben zuvor konnten wir erfolgreich Nucleocytoplasmic Transport von kleinen Molekülen verfolgen, Proteine, mRNA und Virus durch Native nuclear pore komplexe (NPCs)6, 7 , 8. kurz, die folgenden Merkmale des SPEED-Mikroskopie werden zur schnelllebigen Makromoleküle durch Sub-Mikrometer rotationssymmetrisch Strukturen in lebenden Zellen, wie NPCs und primäre Zilien verfolgen: (1) eine geneigte oder eine vertikale Beleuchtung PSF ermöglicht die Anregung von einzelnen Molekülen in einem kleinen Beugungsgrenze Volumen in der Brennebene (Abbildung 1). (2), die geneigt PSF können stark vermeiden Sie Out-of-Focus Fluoreszenz und damit das Signal-Rausch-Verhältnis verbessern. (3) die optische Dichte von 100 bis 500 kW / cm2 bei der Beleuchtung PSF ermöglicht Tausende von Photonen aus einzelnen Fluorophore mit schnellen Erkennung Geschwindigkeiten (> 500 Hz) gesammelt werden. (4) die schnelle Erkennung Geschwindigkeit reduziert auch die räumliche Lokalisation Einzelmolekül-Fehler (< 10 nm) bei der Bestimmung der räumlichen Trajektorien fluoreszierende Moleküle in lebenden Zellen zu bewegen, weil molekulare Diffusion einer der wichtigsten Faktoren ist Unvollkommenheiten der Einzelmolekül-Lokalisierung für die Bewegung der Moleküle verursacht. (5) etablierte 2D zu 3D Umwandlung Algorithmen ermöglichen Ihnen 3D räumliche Wahrscheinlichkeitsdichte Karten der Transportwege für Moleküle in der NPC oder der primäre Zilie zu bieten. Es ist bemerkenswert, dass unsere Konvertierung zwischen den kartesischen und den zylindrischen Koordinatensystem verwendet wird, um ein 3D räumliche Wahrscheinlichkeitsdichte Karte anstatt 3D Einzelmolekül-Tracking (Abbildung 2) zu generieren. Bisher haben Elektronenmikroskopie Daten zeigten, dass die NPC9,10 und die primäre Zilie11 beide eine rotationssymmetrische Struktur haben. Grundsätzlich sollte nach dem Zufallsprinzip diffundierende Moleküle durch die NPC oder primäre Zilie rotationssymmetrische Distributionen auch haben. Wie in Abbildung 2dargestellt, eine hohe Anzahl von nach dem Zufallsprinzip diffundierende Moleküle im Inneren des Zylinders erzeugt rotationssymmetrische Verteilungen der Querschnitt durch das in der NPC, weiter führt eine etwa gleichmäßige räumliche Verteilung innerhalb jeder sehr kleinen Sub-Region zwischen zwei benachbarten Ringen (Abb. 2E). Diese gleichmäßige Verteilung führt, dass die räumliche Verteilung entlang θ Dimension im zylindrischen System konstant ist. Dann können die 3D Koordinaten (X, R, θ) vereinfacht werden, um die 2D Koordinaten (R, X, konstante) werden. Eigentlich ist unsere Konvertierung zwischen den kartesischen und den zylindrischen Systemen von 2D (X, Y) in 2D (R, X, konstante). Die konstante θ bezieht sich auf die räumliche Dichte p in Abbildung 2E, wird berechnet, indem die Gleichung A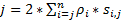 .
.
Letztlich Einzelmolekül-Tracking hat breite Anwendung in der biologischen Forschung, so ist es natürlich, dass eine Vielzahl von Techniken entwickelt, um spezifische biologische Nischen12,13,14füllen. Dies ist der Fall mit SPEED-Mikroskopie. Zuvor wurde zusammen mit einem 3D Transformationsalgorithmus diese Technik entwickelt, um 3D Transportwege von Transit-Moleküle durch die NPCs, eine sub-diffraction-Größe und rotationssymmetrische biologische Struktur6zu beheben. In diesem Papier primäre Zilien erweisen sich als ausgezeichnetes Modell Organellen sowie. Primäre Zilien sind zylindrisch, Antenne-wie Organellen (~ 125 nm Radius), die von der Oberfläche der meisten Säugetier-Zellen15,16,17zu projizieren. Sie sind verantwortlich für externe Signale empfangen und übertragen eine intrazelluläre Reaktion in der Regel verbunden mit Wachstum und Stoffwechsel15,16. Also flux von Strukturproteinen, recycling von transmembranen Rezeptoren und Übertragung von intrazellulären Botenstoffe sind wichtige Aufgaben der primären Zilien. An der Verbindungsstelle zwischen der primären Zilien und Zellkörper ist eine kritische Selektivität Schwelle, so genannte Übergangszone oder TZ, wodurch dieser Protein-Transport11,18,19erfolgen muss, 20. Neben der gating-Funktion von der TZ sind mindestens zwei Transportprozesse, intraflagellar Transport und passive Diffusion, vermutlich verantwortlich für die Bewegung des Proteins durch diese Region16,21, 22. aus Sicht der menschlichen Gesundheit, der Verlust der primäre Zilien und anschließenden Deregulierung der nachgeschalteten Signalisierung ist charakteristisch für viele Krebsarten. Darüber hinaus sind viele Erbkrankheiten wie Bardet-Bardet-Syndrom und polyzystischen Nierenerkrankung, defekte Protein Transport23zugeordnet. Die Größenbeschränkung von Sub-Beugung und den komplexen Prozess der selektiven Protein Transport durch die TZ machen die primären Zilien ein bevorzugtes Ziel für diese Technik. In diesem Methodenpapiers demonstrieren wir die Verfolgung von ciliary transmembranen Protein, Somatostatin Rezeptor 3 (SSTR3)24, extern mit Alexa Fluor 647 und Bestandteil des IFT, IFT2025, beschriftet mit einem geschmolzenen GFP-Molekül gekennzeichnet.
Protokoll
(1) NIH-3 t 3 Zelle Vorbereitung für Geschwindigkeit Mikroskopie ab Lager
- 1,5 Wochen vor dem Experiment, Wiederherstellen einer frischen Kultur NIH-3 t 3 Zellen aus einem gefrorenen bestand bei 37 ° C Auftauen und die Zellen in einem 25 cm2 Zelle Kultur Kolben mit 3 mL der Dulbeccos geändert Eagle Medium (DMEM) ergänzt mit 110 mg/mL Natrium-Pyruvat, 2 mM Glutamin fötalen Rinderserum 10 % und 1 % Penicillin/Streptomycin.
- Inkubieren Sie die Zellen bei 37 ° C in einem 5 % CO2 Inkubator.
- Teilen von Zellen bei 80 % Konfluenz, etwa alle zwei Tage mindestens dreimal vor experimentellen am Tag, um Homogenität des Zellzyklus. Trypsinize Zellen mit 0,25 % Trypsin für 2 min bei 37 ° C, Aspirieren Trypsin und ersetzen Sie es mit 2 mL des Mediums. Pipette das Medium immer wieder zu brechen Zellcluster, die gewünschte Anzahl von Zellen zu entfernen und bringen Gesamtvolumen der Medien zurück bis 3 mL.
Hinweis: NIH-3 t 3 waren zuvor gentechnisch auszudrücken NPHP-4, ein Protein, das die TZ26, verschmolzen am C-Terminus, mCherry lokalisiert. mCherry ist ein Fluorophor die mit 561 nm Beleuchtung quantitativ lokalisieren die TZ Selektivität Barriere und richten Sie die primären Zilien angeregt werden kann. - Zwei Tage vor dem Experiment, Platte die Zellen in einem 35 mm Glasboden Schale bei 60-70 % Konfluenz mit 1,5 mL des gleichen Mediums als Schritt 1.1 und Rückkehr der Zellen in den Inkubator.
- Transfizieren Sie einen Tag vor dem Experiment, chemisch die Zellen mit dem gewünschten Plasmid. 500-1000 ng des gewünschten Plasmids (siehe Hinweis unten) im Verhältnis 1: 2,5 mit Transfection Reagens in 0,25 mL reduzierte Serum-Medien ohne Antibiotika für 30 min. Aspirat Medien aus dem 35 mm Glas unten Schüssel mischen und ersetzen Sie ihn durch das Plasmid 0,25 mL / Transfection Reagens Mix plus eine zusätzliche 1,25 mL reduzierte Serum-Medien ohne Antibiotika. Reduzierte Serum Medien dient dem Zweck, eine erfolgreiche Transfektion zu erleichtern, induzierende primäre Zilien Wachstum sowie die Zellen am Leben zu halten lange genug Experiment durchführen. Zellen in den Inkubator für das Experiment am nächsten Tag zurück.
Hinweis: Bei der Einzelmolekül-Tracking der IFT20 ist ein Plasmid enthält, die eine genetisch veränderte IFT20 an der C-Terminus zu GFP verschmolzen verwendete25. Bei Einzelmolekül-tracking von SSTR3, ein Plasmid mit einem genetisch veränderten SSTR3 verschmolzen in der N-Terminus zu einer Domäne Akzeptor Peptid (AP) und C-Terminus zu GFP ist gebrauchte22. Neben dem SSTR3 Konstrukt ein Plasmid mit Biotin-Ligase BirA Co ausgedrückt werden muss und die Transfektion Medien mit 10 µM Biotin ergänzt werden. BirA legt dann Biotin auf der AP-Domäne neu synthetisierte AP SSTR3 GFP-Moleküle auf der Ebene der ER. Alexa647 auf drei der vier Biotin-Bindungsstellen an Streptavidin, konjugiert können im Durchschnitt dann an die Medien vor imaging Eindringmittel Label die AP-SSTR3-GFP-Moleküle auf der äußeren Oberfläche der Zelle22,27 ergänzt werden . GFP und AlexaFluor647 werden bei dieser Methode verwendet; jedoch können andere fluoreszierenden Sonden verwendet werden, wenn sie ähnlich hohe Lichtstabilität und Quantum ergeben haben. - Wenn mit der extern beschriftet SSTR3 Konstrukt, entfernen Sie die Medien aus den Glasboden Schale 1 h vor dem Experiment, waschen die Zelle 5 Mal mit 1 mL der Phosphat-gepufferte Kochsalzlösung (PBS) und fügen Sie 1 mL der reduzierte Serum Medien ergänzt mit 1 µM Alexa647 konjugiert Streptavidin.
- Nicht mehr als 15 min vor dem Experiment, entfernen Sie Medien aus der Glasschale unten und waschen Sie transfizierten und beschrifteten Zellen 5 Mal mit 1 mL PBS.
- Platz 1 mL bildgebenden Puffer (20 mM HEPES, 110 mM KOAc, 5 mM NaOAc, 2 mM MgOAc, 1 mM EGTA, pH 7,3) in die untere Glasschale.
Hinweis: In der Bildgebung Puffer sind Zellen tragfähig für nicht mehr als 3 h. Daher werden nur 2 h von Experimenten auf jedes Gericht durchgeführt.
2. SPEED-Mikroskopie
Hinweis: Die Geschwindigkeit Mikroskopie Setup enthält eine invertierte Fluoreszenzmikroskop ausgestattet mit einen 1,4-NA 100 × Ölimmersion apochromatische Objektive, 35 mW 633 nm He-Ne Laser, 50 mW 488 nm und 561 nm Festkörperlaser, einen Gewinn auf dem Chip Multiplikation Kamera Verbindung-Ladegerät und ein Mikroskop-Software-Paket für die Datenerfassung und Verarbeitung (Abbildung 1). Für die einzelnen Kanal Bildgebung, GLP, mCherry und Alexa647 sind begeistert von 488 nm, 561 nm oder 633 nm Laser, beziehungsweise. Für Einzelmolekül-Tracking wird Einzelpunkt-Beleuchtung verwendet, um einzelne Eindringmittel beschriftet Moleküle zu verfolgen. Für Epifluoreszenz Bildgebung befindet sich eine konkave Linse im Laser Beleuchtung Pfad, den Strahl in ein einheitliches Feld der Beleuchtung zu erweitern. Die Fluoreszenz-Emissionen werden durch das gleiche Ziel gesammelt, gefiltert durch eine dichroitische Filter (405/488/561/635) und eine Emission Filter (405/488/561/635) und abgebildet mit den oben genannten CCD-Kamera bei 500 Hz für Einzelmolekül-Tracking oder 2 Hz für Epifluoreszenz Bildgebung.
- Befestigen Sie die Glas-Bodenplatte auf die Bühne des Mikroskops und suchen Sie eine Zelle, die richtig die gewünschte Konstrukte zum Ausdruck zu bringen ist. Sobald eine geeignete Zelle gefunden worden ist, richten Sie die NPHP4-mCherry Fleck an der Basis der primären Zilien mit dem Standort auf der Abbildungsebene, die der Laser Einzelpunkt-Beleuchtung entspricht.
- Erstellen eines Epifluoreszenz-Abbildes der NPHP4-mCherry und IFT20-GFP oder AP-SSTR3-GFP mit der "Snap" Funktion im Reiter "Kamera" von den "Fokus" Fenster "" Wenn der digitalen Mikroskopie-Software-Paket zu verwenden (siehe Tabelle der Materialien).
Hinweis: Diese Bilder werden als Referenz für die anschließende Einzelmolekül-Standorte handeln. - Sobald die Referenzbilder erhalten werden, lokal reduzieren Sie die Konzentration der beschrifteten Einzelmoleküle. Foto-Bleichmittel die TZ mit 1 mW Laser Beleuchtung für 20 s oder bis der Fluoreszenzintensität des Hintergrundfluoreszenz nahekommt.
Hinweis: Wenn die genaue Konzentration kann gesteuert werden, 0,1-1 nM einzelne Moleküle gekennzeichnet sind, verwendet. - Zur Vorbereitung der Einzelmolekül-Tracking reduzieren Sie die Laserleistung Beleuchtung ~0.15 mW für einzelne Moleküle mit GFP beschriftet oder ~0.5 mW für Moleküle mit Alexa647 beschriftet.
- Sobald die Laserleistung und imaging-Parameter festgelegt sind, maximale Verstärkung und Intensivierung und 2 ms Bildrate für die Einzelmolekül-Bildgebung, den entsprechenden Beleuchtung Laser zu engagieren und erfassen nicht-Photobleached, einzelne Moleküle gekennzeichnet, wie sie transportiert werden durch die Photobleached Region der TZ durch Anklicken des Buttons "Stream" in der Registerkarte "Kamera" des Fensters "Fokus Controls".
Hinweis: Nicht mehr als 2 min Video sollte erfasst werden, um die Auswirkungen der ciliary Drift auf ein geringfügiges Maß zu minimieren. - Verarbeiten Sie nach der Aufnahme der Einzelmolekül-Video die Videos mithilfe eines 2D "glockenförmig" passenden Algorithmus, wie der Blick durch das Gelles Lab, das genau der Schwerpunkt jedes einzelne Molekül Erregung PSF in einem umfassenden Bereich von Interesse (AOI) lokalisiert.
- Wählen Sie alle Einzelmolekül-Standorte mit Präzision < 10 nm und korrigieren Sie das Zentrum der Zilien basierend auf Verteilung der Einzelmolekül-Standorte mit einer 2D Gauß-Funktion ausgestattet.
Hinweis: Mithilfe von 2D zu 3D Umwandlung Algorithmus sind die 3D Transportwege des IFT20-GFP und AP-SSTR3 Routen auf ciliary axonemal oder ciliary Membran, bzw. deutlich.
(3) 2D zu 3D Umwandlung
- Sobald mehrere tausend Lokalisierungen für Transit-Moleküle (Signal-Rausch Verhältnis > 11) in die Zilie erhoben werden, wählen Sie die Längsachse der Zilie als die X-Dimension. Ein Y-Dimension-Histogramm der Standorte zu machen und bin Summen in Schritten von 10 nm erhalten.
Hinweis: Die 2D zu 3D Umwandlung kann von Hand oder Software oder Programmiersprache ausgewertet werden. Die Autoren haben die Transformation in Matlab und Python 2.7 erfolgreich umgesetzt.
Ergebnisse
Dieser Abschnitt zeigt die Durchführung Geschwindigkeit Mikroskopie in der TZ primäre Zilien den Transportweg von SSTR3, verbunden durch ein ~ 15 nm externe Linker, Alexa647 studieren gewonnenen Daten(Abbildung 3). Es dient dem doppelten Zweck der Überprüfung des 3D-Transformation Algorithmus. Alexa647 sollten nur Label die Außenfläche der primäre Zilie und sich somit der 3D Transportweg High-Density Transportweg an dieser Stelle verra...
Diskussion
Dieses Protokoll beschreibt die Anwendung der Geschwindigkeit Mikroskopie auf die primäre Zilie, ein zellulärer Signalisierung Organell, das hoch effiziente Protein Transport abhängig ist. Geschwindigkeit-Mikroskopie kann hochauflösende (< 10 nm) für Gewebekulturen beschriftet Moleküle bieten Durchschreiten der Einzelpunkt-Beleuchtung auf die TZ zentriert. Zuvor ist angewendet worden, um das Protein des Menschenhandels durch die NPC-6,7,
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Wir danken Dr. Kristen Verhey (University of Michigan, Ann Arbor) und Dr. Gregory Pazour (University of Massachusetts Medical School) für die Bereitstellung von einigen Plasmide. Das Projekt wurde unterstützt durch Zuschüsse aus dem National Institutes of Health (NIH-GM097037, GM116204 und GM122552, W.Y.).
Materialien
| Name | Company | Catalog Number | Comments |
| 25 cm2 tissue culture dish | Corning | VV-01936-00 | |
| Penicillin/streptomycin | ThermoFisher | 15140122 | |
| Fetal bovine serum | ThermoFisher | 10438018 | |
| DMEM | ThermoFisher | 10566-016 | |
| OPTIMEM | ThermoFisher | 31985062 | |
| Trypsin | ThermoFisher | 25300054 | |
| Phosphate buffered saline | Sigma-Aldrich | P3813-1PAK | |
| Transit LT1 | Mirus | MIR 2300 | |
| 35 mm glass bottom dish | MatTek | P35GCOL-0-14-C | |
| AlexaFluor 647-conjugated streptavidin | ThermoFisher | S21374 | |
| Biotin | Sigma-Aldrich | B4501-100MG | |
| 633 nm He-Ne laser | Melles Griot | 25-LHP-928-249 | |
| 561 nm solid state laser | Coherent | OBIS 561-50 LS | |
| 488 nm solid state laser | Coherent | 1185053 | |
| Inverted fluorescence microscope | Olympus | IX81 | |
| 1.4-NA 100× oil-immersion apochromatic objective | Olympus | UPLSAPO 100× | |
| On-chip multiplication gain charge-coupled-device camera | Roper Scientific | Cascade 128+ | |
| Dichroic filter | Semrock | Di01- R405/488/561/635-25x36 | |
| Emission filter | Semrock | NF01-405/488/561/635-25X5.0 | |
| Slidebook 6.0 | Intelligent Imaging Innovations | digital microscopy software |
Referenzen
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annu Rev Biochem. 78, 993-1016 (2009).
- Leung, B. O., Chou, K. C. Review of super-resolution fluorescence microscopy for biology. Appl Spectrosc. 65, 967-980 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440, 935-939 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313, 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3, 793-796 (2006).
- Ma, J., Yang, W. Three-dimensional distribution of transient interactions in the nuclear pore complex obtained from single-molecule snapshots. Proc Natl Acad Sci USA. 107, 7305-7310 (2010).
- Ma, J., Goryaynov, A., Sarma, A., Yang, W. Self-regulated viscous channel in the nuclear pore complex. Proc Natl Acad Sci USA. 109, 7326-7331 (2012).
- Ma, J., et al. High-resolution three-dimensional mapping of mRNA export through the nuclear pore. Nat Comm. 4, (2013).
- Akey, C. W., Radermacher, M. Architecture of the Xenopus nuclear pore complex revealed by three-dimensional cryo-electron microscopy. J Cell Biol. 122, 1-19 (1993).
- Akey, C. W. Interactions and structure of the nuclear pore complex revealed by cryo-electron microscopy. J Cell Biol. 109, 955-970 (1989).
- Czarnecki, P. G., Shah, J. V. The ciliary transition zone: from morphology and molecules to medicine. Trends Cell Biol. 22, 201-210 (2012).
- Elf, J., Li, G. -. W., Xie, X. S. Probing transcription factor dynamics at the single-molecule level in a living cell. Science. 316, 1191-1194 (2007).
- Anzalone, A., Annibale, P., Gratton, E. 3D orbital tracking in a modified two-photon microscope: an application to the tracking of intracellular vesicles. J Vis Exp. , (2014).
- Ritter, J. G., Veith, R., Veenendaal, A., Siebrasse, J. P., Kubitscheck, U. Light sheet microscopy for single molecule tracking in living tissue. PloS one. 5, 11639 (2010).
- Marshall, W. F., Nonaka, S. Cilia: tuning in to the cell's antenna. Curr Biol. 16, 604-614 (2006).
- Scholey, J. M., Anderson, K. V. Intraflagellar transport and cilium-based signaling. Cell. 125, 439-442 (2006).
- Yang, T. T., et al. Superresolution pattern recognition reveals the architectural map of the ciliary transition zone. Sci Rep. 5, 14096 (2015).
- Craige, B., et al. CEP290 tethers flagellar transition zone microtubules to the membrane and regulates flagellar protein content. J Cell Biol. 190, 927-940 (2010).
- Kee, H. L., et al. A size-exclusion permeability barrier and nucleoporins characterize a ciliary pore complex that regulates transport into cilia. Nat Cell Biol. 14, 431-437 (2012).
- Najafi, M., Maza, N. A., Calvert, P. D. Steric volume exclusion sets soluble protein concentrations in photoreceptor sensory cilia. Proc Natl Acad Sci USA. 109, 203-208 (2012).
- Nachury, M. V., Seeley, E. S., Jin, H. Trafficking to the ciliary membrane: how to get across the periciliary diffusion barrier. Annu Rev Cell Dev Biol. 26, 59-87 (2010).
- Ye, F., et al. Single molecule imaging reveals a major role for diffusion in the exploration of ciliary space by signaling receptors. Elife. 2, 00654 (2013).
- Ross, A. J., et al. Disruption of Bardet-Biedl syndrome ciliary proteins perturbs planar cell polarity in vertebrates. Nat Genetics. 37, 1135-1140 (2005).
- Handel, M., et al. Selective targeting of somatostatin receptor 3 to neuronal cilia. Neuroscience. 89, 909-926 (1999).
- Follit, J. A., Tuft, R. A., Fogarty, K. E., Pazour, G. J. The intraflagellar transport protein IFT20 is associated with the Golgi complex and is required for cilia assembly. Mol Biol Cell. 17, 3781-3792 (2006).
- Awata, J., et al. NPHP4 controls ciliary trafficking of membrane proteins and large soluble proteins at the transition zone. J Cell Sci. 127, 4714-4727 (2014).
- Howarth, M., Ting, A. Y. Imaging proteins in live mammalian cells with biotin ligase and monovalent streptavidin. Nat Protoc. 3, 534-545 (2008).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy. Science. 319, 810-813 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten