需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
整合酶缺陷慢病毒载体载体的制备及其在细胞 CRISPR/Cas9-mediated 基因敲除中的表达
摘要
我们描述了整合酶缺乏慢病毒载体载体 (IDLVs) 作为向细胞传递 CRISPR/Cas9 的工具的生产策略。由于能够在细胞中调节快速和健壮的基因编辑, IDLVs 为基因的传递提供了一个更安全、同样有效的载体平台, 与整合酶能力的载体相比。
摘要
慢病毒载体载体是一种理想的选择, 提供基因编辑组件的细胞, 由于其能力稳定传感广泛的细胞和调解高水平的基因表达。然而, 它们融入宿主细胞基因组的能力增强了插入致突变的风险, 从而提高了安全性, 并限制了它们在临床环境中的使用。此外, 这些整合能力的慢病毒载体载体 (ICLVs) 提供的基因编辑组件的持续表达增加了混杂基因靶向的可能性。作为一种替代方法, 开发了新一代整合酶缺陷慢病毒载体矢量 (IDLVs), 解决了其中的许多问题。这里提出了一个新的和改进的 IDLV 平台 CRISPR 介导的基因编辑的生产协议, 并列出了这些载体的纯化和浓度所涉及的步骤, 并利用 HEK-293T 的转导和基因编辑效率细胞被证明。此协议易于伸缩, 可用于生成具有传感单元格体外和体内的高效度 IDLVs。此外, 该协议可以很容易地适应生产 ICLVs。
引言
精确的基因编辑是主要的生物医学进步的基石, 涉及发展新的策略来解决遗传疾病。在基因编辑技术的前沿是依靠使用clustered regularlyinterspaced s园艺palindromic repeats (CRISPR)/Cas9 系统的方法, 最初确定作为抗病毒遗传物质入侵的细菌免疫成分 (在引用1,2) 中进行了检查。CRISPR/Cas9 系统对其他基因编辑工具的主要优势, 如锌指核酸 (ZFNs) 和转录激活因子核酸 (TALENs) (参照3), 是质粒设计的相对简单性和CRISPR 组件的构建--这一功能推动了将基因编辑从少数专业实验室扩展到更广泛的研究社区。此外, CRISPR/Cas9 编程的简单性和其多路目标识别的能力进一步推动了它作为一种成本效益和易于使用的技术的普及。在研究人员向细胞提供这种基因编辑组件的各种方法中, 病毒载体仍然是目前最流行和最有效的系统。
慢病毒载体矢量 (lv) 已成为一种选择工具, 用于将CRISPR/Cas9系统的组件在体内用于不同的应用程序4, 5, 6, 7。一些关键功能使 lv 成为这个过程的一个流行的选择, 包括它们感染分裂和非分裂细胞的能力, 低免疫原性和极小的细胞毒性 (在参考8中被审查)。因此, LV 介导的基因治疗已被用于治疗传染性疾病, 如 HIV-1, HBV, 和 HSV-1, 以及纠正的缺陷, 如囊性纤维化和新血管性黄斑变性等人类遗传性疾病4,5,7,9,10,11. 此外, lv 已被有效地修改, 以进行多重基因编辑在不同的基因组位点使用单一的向量系统12。
然而, lv 融入宿主基因组的固有特性可能是诱变性的, 而且常常妨碍其作为转基因运载工具的效用, 特别是在临床环境中。此外, 由于稳定集成的 lv 表达他们的转基因在可持续的高水平, 这个系统是不适用于交付基因编辑组分例如 CRISPR/Cas9;过度表达的 Cas9-guide RNA (gRNA) 和类似的蛋白质, 如 ZFNs, 是与高级别的离靶效应, 其中包括不良突变13,14,15,16,17 , 并可能增强细胞毒性18。因此, 为了实现精确的基因编辑和最小的离靶效应, 有必要设计系统, 使基因编辑成分的瞬时表达。
近年来, 在单元格16、19、20、21 (在参考22中复查) 中, 已开发出各种交付平台以瞬时表达 CRISPR/Cas9。这些方法包括依赖直接将纯化的 Cas9 与适当的导 rna 一起引入细胞, 这在与质粒介导的转染16相比较中被证明更有效。研究表明, 由导 RNA/Cas9 颗粒组成的 ribonucleoprotein (RNP) 配合物在其靶上进行 DNA 解切后迅速转过来, 表明这些成分的短期表达足以达到健壮基因编辑16。可想而知, 非整合病毒载体平台, 如腺相关病毒载体 (自动增值服务) 可以提供一个可行的替代方案, 提供基因编辑机制的细胞。不幸的是, AAV 衣壳的封装能力明显低于 lv (< 5kb), 这严重阻碍了它们在单个向量中封装多组件 CRISPR 工具包的能力 (在参考8中进行了审查).值得注意的是, 添加抑制组蛋白乙酰的化合物 (例如, 丁酸钠23) 或阻碍细胞周期 (如, 咖啡因24) 已经显示增加慢病毒载体效价。尽管最近取得了一些进展, 但迄今为止发展起来的瞬态表达系统仍然受到一些缺陷的阻碍, 例如生产效率降低, 导致病毒滴度降低, 病毒的传导效率低。这些方法25。
整合酶缺乏慢病毒载体载体 (IDLVs) 是基因运载工具发展的一个重要进步, 因为它们结合了 lv 的包装能力和细胞中 AAV 样 episomal 维护的额外好处。这些功能帮助 IDLVs 在很大程度上规避了与整合向量有关的主要问题, 即持续过度表达潜在的毒性元素和整合介导的致突变性。以前已证明 IDLVs 可以成功修改以增强 episomal 基因表达式26,27。关于 IDLV 介导的 CRISPR/Cas9 交付, 低生产效价和较低表达的 episome 基因组相对于整合酶精通慢病毒载体系统限制其效用为善意工具, 以提供基因组编辑转基因结构。我们最近证明, 与 IDLV 生产相关的转基因表达和病毒效价都有明显的增强, 包括在病毒表达式卡带28内的转录因子 Sp1 的结合点。改良的 IDLVs 支持 CRISPR 介导的基因编辑体外(在 HEK-293T 细胞) 和体内(在有丝分裂后的脑神经元), 同时诱导最小的离靶突变相比, 相应的 ICLV 介导系统28。总的来说, 我们开发了一个新的, 紧凑的, 在一个 IDLV 平台上进行的 CRISPR 工具包, 并概述了使用这种运载工具, 以增强基因编辑的各种优势。
介绍了 IDLV-CRISPR/Cas9 系统的生产方案, 包括 IDLVs 的组装、纯化、浓缩、滴定等各个步骤, 以及验证这些载体的基因编辑功效的策略。该协议易于扩展, 以满足不同调查人员的需要, 旨在成功地生成 LV 矢量, 其范围为 1 x 1010传感单元 (TU)/mL。通过本协议生成的向量可以有效地感染几种不同的细胞类型, 包括难以传感器的胚胎干细胞、造血细胞 (T 细胞和巨噬细胞), 培养和在体内-注入的神经元。此外, 该议定书同样适用于类似数量的整合酶能力慢病毒载体载体的生产。
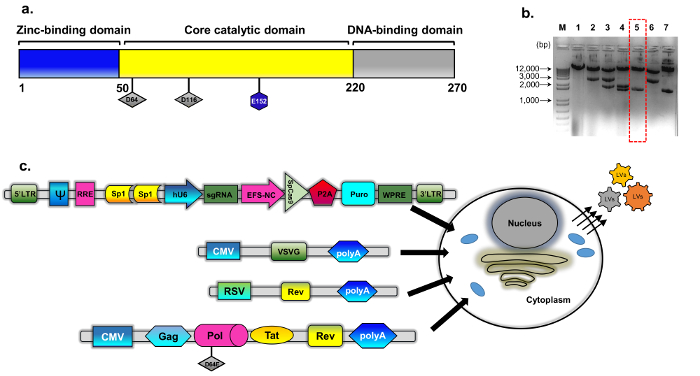
图 1: IDLV 包装.(a)野生型整合酶蛋白(b ) 的示意图该改性质粒来源于 psPAX2 (见方法, 质粒结构)。具有代表性的琼脂糖凝胶图像的克隆整合酶克隆筛选。采用标准质粒 dna 分离小试剂盒进行了 EcoRV 和 SphI 的消化, 分析了 dna 样品的制备。在INT中, D64E 替换的直接 (桑格) 排序方法进一步验证了正确消化的克隆 (数字5、虚线红色框)。整合酶包装盒被命名为 pBK43。(c)用于生成 IDLV-CRISPR/Cas9 向量的瞬变转染协议的示意图, 显示了用 VSV、包装和转基因磁带 (Sp1-CRISPR/Cas9 的多项质粒) 转移的293T 细胞。从细胞膜芽出的病毒微粒含有载体的全长 RNA (用转基因卡带表示)。第二代 IDLV 包装系统使用, 其中包括调节蛋白达和启示录转速表达式进一步补充从一个单独的卡带 (合胞病毒-转速-质粒)。Abbrev: LTR 长端重复、VSV、水泡口炎病毒 G 蛋白、pCMV 巨细胞病毒启动子;瓣肉瘤病毒促进剂;实用 (转速响应元素)。表达式盒中的其他调控元素包括 Sp1-binding 站点、转速响应元件 (实用)、土拨鼠肝炎病毒转录后水平调节元件 (WPRE)、核心伸长因子1α启动子 (EFS NC)、矢量封装元素ψ (psi), 人巨细胞病毒 (cmv) 启动子和人类 U6 启动子。请单击此处查看此图的较大版本.
研究方案
1. 培养 HEK-293T 细胞和种子细胞进行转染
注: 人类胚胎肾脏 293T (HEK-293T) 细胞生长在 DMEM, 高葡萄糖培养基辅以10% 牛小牛血清补充铁和生长促进剂, 1x 抗生素防霉溶液 (100x 溶液含有1万单位青霉素, 10 毫克链霉素和25µg 两性霉素 B 每毫升)。该培养基还辅以1x 丙酮酸钠, 1x 非必需氨基酸混合, 和2毫米 l-谷氨酰胺 (200 毫米 l-丙谷氨酰胺二肽在 0.85% NaCl)。细胞培养在100毫米组织培养板 (近似生长表面积是 55 cm²)。分培养率为 1:10, 每 2-3 天进行次培养。胰蛋白酶-EDTA 0.05% 用于分离的细胞之间的通道。为了保持实验之间的一致性, 我们建议在切换到不同批次时测试小牛血清, 并监测细胞生长、转染效率和矢量生产的任何变化。
- 使用低通道细胞启动一种新的文化 (建议不要在通道15后使用细胞, 或者如果生长减慢) 通过播种成一个10厘米的组织培养板。使用 DMEM 补充10% 血清细胞生长。生长细胞在37°c 与 5% CO2在一个标准组织培养孵化器。使用标准 hemocytometer 计算所有后续步骤的单元格数。
- 一旦细胞达到 90-95% 汇合生长, 补种成15厘米组织培养板 (下面, 步骤 1.3-1.5)。
- 补种, 从汇合板中抽出介质, 用无菌 1x PBS 轻轻冲洗。孵育细胞与2毫升的离解试剂 (例如, 胰蛋白酶-EDTA) 在37°c 为 3-5 分钟. 添加8毫升含有血清的培养基, 以灭活分离试剂, triturate 10-15 倍, 用10毫升血清吸管创建单个细胞暂停.并用重悬培养基中的细胞, 以获得大约 4 x10 6 细胞/毫升的细胞密度。
- 为了加强对基体的附着, 预涂15厘米板与0.2% 明胶, 增加8毫升的明胶每板。均匀地分布在板材的表面, 在室温下孵育10分钟, 并虹吸出液体。
- 将每块板的总容积用温热 (37 °c) HEK-293 T 介质 (参见步骤1中的注), 并通过添加2.5 毫升细胞 (总 ~ 1 x 107单元格/板) 来播种板块。孵化板材在37°c 与 5% CO2隔夜或直到 70-80% 融合被到达。
注: 可用于生产的多达六15厘米的板材。在使用超过四个板块时, 调整每版介质的音量为 ~ 20-22 毫升 (参照协议的步骤 5)。
2. 转染 HEK-293T 细胞使用磷酸钙基协议
-
转染试剂
- 准备 2x. 缓冲的解决方案 BBS (50 毫米, 280 毫米氯化钠, 1.5 毫米 Na2HPO4), 结合16.36 克氯化钠, 10.65 克 (n, n-双 (2-羟乙基)-2-氨基磺酸) 和 0.21 g 的 Na2HPO4。添加双蒸馏 H2o (dd-H2O), 最多900毫升。溶解, 滴定的 pH 值为 6.95, 1M 氢氧化钠, 并通过0.22 µM 过滤器将体积带到 1 l 过滤器。存储在-20 摄氏度。
- 准备 1M CaCl2。过滤器解决方案通过0.22 µM 过滤器。存储在4摄氏度。
- 观察一天前播种的盘子。细胞一旦到达 70-80% 融合, 就可以进行转染。
- 从盘子中吸取旧的培养基, 轻轻地添加没有血清的新鲜准备的培养基。
注: 有关所选质粒及其制备的详细信息, 请参阅补充文件 1-质粒。 - 通过将四质粒 aliquoting 成15毫升圆锥管, 制备粒料。对于单个15厘米的菜式, 请使用 CRISPR/Cas9-transfer 向量 (pBK198 或 pBK189) 的37.5 µg、µg (pBK43) 25 psPAX2-D64E、12.5 µg pMD2、G 和6.25 µg (图 1c)。
- 将312.5 µL 1M CaCl2添加到质粒组合中。为此, 最多可添加1.25 毫升无菌 dd H20。
- 慢慢地 (下降明智) 添加1.25 毫升 2x BBS 解决方案, 同时涡流的混合。室温下孵育30分钟。
- 添加转染混合物滴状到每15厘米板 (每板2.5 毫升)。轻轻地旋转盘子, 在37°c 处孵化 5% CO2为 2-3 h。此后, 每片增加2.5 毫升 (10%) 血清, 并在一夜之间继续孵化 (12-18 小时)。
注: 一些实验室使用 3% CO2孵化器来稳定介质 pH 值。但是, 在3% 和 5% CO2之间, 我们没有观察到转染效率的任何差异。此外, 投诉警察课4沉淀的大小对于转染效率至关重要;转染组合必须清楚, 然后再添加到细胞。如果混合在孵化期间变得混浊, 准备新鲜的 2x BBS (pH = 6.95)。
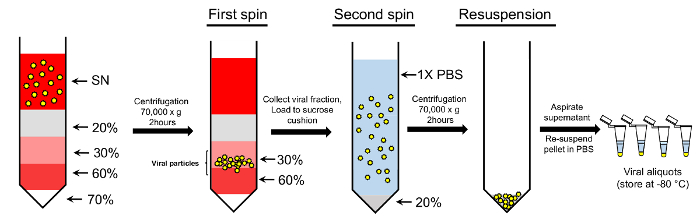
图 2: 使用双蔗糖梯度协议的病毒粒子浓度.从上清 (SN) 收集的病毒粒子被加载到梯度蔗糖梯度上。70%、60%、30% 和20% 蔗糖溶液用于创建渐变。离心后, 从 30-60% 蔗糖组分收集的微粒进一步被装载到20% 蔗糖垫和沉淀。含有纯化病毒粒子的最终颗粒在 1x PBS 中悬浮, 以便进一步使用 (请参阅文本以了解详细信息)。请单击此处查看此图的较大版本.
3. 转染后的一天
- 观察细胞, 以确保他们在这一点上接近100% 融合, 几乎没有细胞死亡。将25毫升新鲜的 DMEM + 10% 血清添加到每个盘子中, 以取代培养基。继续孵化在37°c 与 5% CO2为另外的 48 h。
注: 如果使用超过六15厘米的板 (请参阅步骤5.2、注释), 请调整步骤3.1 中介质的音量。
4. 采集病毒
- 在50毫升离心管中使用无菌10毫升组织培养液吸管和池, 从含有转染细胞的所有组织培养板中仔细收集上清液。
- 用台式离心机将悬浮液离心在 400-450 x g 处, 以10分钟的方法清除。通过0.45 µm 真空过滤装置过滤上清液。
注: 经过滤的上清液可贮存在4摄氏度, 最多4天, 然后再进行浓缩, 或 aliquoted, 储存在-80 摄氏度。IDLV-Sp1-CRISPR/Cas9 (非浓缩) 病毒制剂的预期效价应为 ~ 2 x 107涂/毫升 (参照步骤6进行滴度测定)。然而, 避免将病毒制剂置于多个冻融循环中, 因为每轮冰冻和解冻会导致功能下降 10-20%。
5. 离心的病毒微粒浓度
注: 我们采用双蔗糖提纯方法, 涉及两个步骤: 蔗糖梯度步骤和蔗糖缓冲步骤 (图 2)。
- 按以下顺序加载锥形离心管, 以创建蔗糖梯度: 0.5 毫升70% 蔗糖 (溶解 1x PBS), 0.5 毫升60% 蔗糖 (溶解在 DMEM), 1 毫升30% 蔗糖 (溶解在 DMEM) 和2毫升20% 蔗糖 (溶解在 1x PBS)。
-
小心地将含病毒的上清液添加到渐变中。由于四15厘米板上清液的总容积为100毫升, 每旋使用六离心管来处理病毒上清液的全部体积。
- 用于此步骤的每个离心管的容积容量可达30毫升 (包括蔗糖量), 在试管中均匀分布病毒上清液, 留下至少10% 个顶部以防止溢出。
- 每个实验使用四15厘米的板材时, 将培养体积调整为 20-22 毫升, 以便在六离心管中方便地容纳池内清液的最终体积。
- 将离心管填充到至少3/4 其总容积容量, 否则在离心过程中会发生管子破裂, 导致样品和/或设备损坏的可能损失。
- 平衡管与 1x PBS 和离心机样品在 7万 x g 2 小时17摄氏度 (见材料表的转子详细信息)。
注: 为了防止在加速过程中蔗糖层的破坏, 设置 ultracentrifuge 在旋转的前3分钟内缓慢地加速转子到 200 rpm。同样, 设置 ultracentrifuge, 以缓慢减速转子从 200 rpm 到 0 rpm 超过3分钟的旋转结束。 - 小心地收集 30-60% 蔗糖组分到干净的管子 (图 2)。增加冷 1x PBS 到汇集的组分并且带来容量到100毫升;混合吹打几次。
- 通过仔细地将病毒制剂放在蔗糖垫子上, 进行蔗糖缓冲步骤。为此, 增加4毫升20% 蔗糖 (在 1x PBS) 到管, 其次是 20-25 毫升的病毒溶液每管。如果管子是少于3/4 充分, 顶部与无菌 1x PBS。
- 仔细平衡和离心样品在 7万 x g 为2小时在17°c, 和以前一样。倒上清液, 让剩余的液体通过在纸巾上倒置的管子排出。
- 吸入剩余的水滴, 以除去颗粒中的所有液体。在这一步, 含有病毒的小球应该是几乎可见的小半透明斑点。
- 并用重悬颗粒通过加入70µL 的 1x PBS 到第一管, 彻底吹打悬浮, 随后将悬浮液转移到下一个管, 并与以前混合, 继续, 直到所有的球团是悬浮。
- 用另外50µL 的冷 1x PBS 冲洗管子, 并与以前混合。确保最终悬架的组合体积为120µL, 呈微乳状;通过离心在 1万 x g 三十年代在桌面离心清除它。
- 将上清液转移到新鲜的离心管, 使10µL 整除数, 并储存在-80 摄氏度。
注意: 避免在慢病毒载体样品上重复冻结解冻循环。除非需要离心, 否则应努力在组织培养罩或指定的组织培养室中使用适当的生物安全措施来执行余下的步骤 (见讨论)。
6. 病毒滴度的估计
-
p24 -酶联免疫吸附试验 (ELISA) 法
注: 根据 NIH 艾滋病疫苗计划 (HIV-1 p24抗原捕获化验试剂盒 (见材料表) 的指示, 使用高结合度96井板进行检测, 并修改29。- 第二天, 洗井三次与200µL 0.05% 吐温20在冷 pbs (pbs t 解决方案)。用100µL 单克隆抗 p24抗体, 在 1x PBS 稀释 1:1500, 在4摄氏度一夜之间孵化。
- 为消除非特定的装订, 在 PBS 中用200µL 1% BSA 阻挡板;三倍, 200 µL 0.05% 吐温20在冷 pbs (pbs t 解决方案) 在室温下至少1小时。
-
准备示例:对于浓缩的载体制剂, 稀释1µL 的样品100倍, 增加89µL 的 dd H20 和10µL 的海卫 X-100 (最后浓度 10%)。对于非浓缩制剂, 请准备十倍稀释样品 (添加80µL 的 dd H20 和10µL 的海卫 X-100 (最终浓度 10%) 到10µL 的样品)。
注: 在这个步骤中, 样品可以保存在-20 摄氏度以上, 以便以后使用。 - 通过应用2倍的连续稀释 (具有起始浓度 5 ng/毫升) 来制备 HIV-1 标准。
- 稀释浓缩样品 (从1:100 预稀释的存货) 在 RPMI 1640 补充与0.2% 吐温20和 1% BSA 建立 1:10,000, 1:50,000 和1:250,000 稀释。稀释非浓缩样品 (从1:10 预稀释的存货) 在 RPMI 1640 补充与0.2% 吐温20和 1% BSA 建立 1:500, 1:2500 和1:12,500 稀释。
- 在 triplicates 的盘子上涂抹样品, 在4摄氏度一夜之间孵化。
- 第二天, 洗井六次和孵化37摄氏度4小时与100µL 多克隆兔抗 p24抗体, 稀释1:1000 在 RPMI 1640, 10% 血清, 0.25% BSA, 2% 正常小鼠血清 (NMS)。
- 洗涤六倍以上和孵化在37°c 1 h 与山羊抗兔辣根过氧化物酶 IgG 稀释1:10,000 在 RPMI 1640 补充5% 正常山羊血清, 2% NMS, 0.25% BSA, 0.01% 吐温20。
- 洗盘子, 如上所述, TMB 过氧化物酶基质在室温下孵育15分钟。
- 通过添加100µL 1 氮盐酸来阻止反应。使用吸光度板读取器测量 450 nm 的样品吸光度。
-
荧光报告强度的测量
- 资产管制法
注: 通过流式细胞仪测量转基因细胞的平均荧光强度, 可以准确估计细胞中 GFP 信号损耗的程度。请参阅最近的论文28 , 以进行外地数据分析、演示和解释。该协议描述如下。- 在 1x PBS 中进行病毒制剂的准备工作 (从 10-1到 10-5) 进行十倍的连续稀释。
- 种子大约 5 x 105 293T 细胞在6井板的每个井在最后容量2毫升每井。添加10µL 的每个病毒稀释到细胞和孵化细胞在37°c 48 小时。
- 收获细胞为外地资产管制分析如下: 添加200µL 0.05% 胰蛋白酶-EDTA 溶液, 孵育细胞37°c 5 分钟. 添加2毫升的完整 DMEM 介质, 并收集样品到15毫升圆锥管。
- 颗粒细胞通过离心在 400 x g 在4°c, 并且并用重悬颗粒在500µL 冷 1x PBS。
- 为固定, 增加等量的4% 甲醛溶液的悬浮和孵化10分钟室温。
- 颗粒固定细胞和并用重悬在1毫升 1x PBS。使用 Ortinski et等中所述, 分析 GFP 表达式。28简要地, 使用以下公式确定病毒的功能效度:
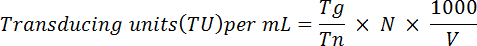
注: 这里 Tg = 数字 GFP 阳性细胞计数;总计数的细胞总数;N = 转基因的单元格总数;V = 体积 (µL) 用于转导。例如: 如果 1 x 106单元格与10µL 的病毒转基因, 2 x 104细胞被计数, 5 x 103是 GFP 阳性, 基于上述等式功能滴度将是:
- 计数 GFP 阳性细胞
- 计算用于转导的多种感染 (语言)。测试范围广泛的 mois 》杂志 (1-10), 随着 mois 》杂志的增加, 导致了更高的转导效率。
- 在转染之前, 种子 a 6 井板与大约 3-4 x 105细胞每井。一旦细胞达到 > 90% 融合 (通常在24小时内), 传感器与纯化的病毒在预先确定的 mois 》杂志。
- 孵育板在37°c 与 5% CO2在标准组织培养, 并且监视细胞在 1-7 天的定期间隔时间为 GFP 信号的变动。
- 用荧光显微镜计算 gfp 阳性细胞的数量 (计划4X 目标, 0.1 N, 40X 放大) 装有 GFP 过滤器集 (激发 wavelength-470 nm, 发射 wavelength-525 nm), 使用天真 (非转基因) 细胞来设置人口绿色荧光蛋白阴性和阳性细胞。
- 使用以下公式, 通过调整稀释系数和体积来估计最终滴度:
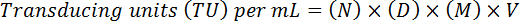
注: 这里, N = GFP 阳性细胞数, D = 稀释因子, M = 放大因子 (通常为 20X), V = 用于转导的病毒体积。例如, 对于20个 GFP 阳性细胞 (N), 在10µL 样品 (V) 的稀释 10-4 (1:10,000) 时, 在20X 倍度 (M) (D) 上计数将导致功能滴度 (20 x 104) x (20) x (10) x (100 *) = 4 x 108 TU/毫升。
(* 调整至每毫升)
- 资产管制法
结果
IDLV-CRISPR/Cas9 矢量击倒效率的验证
我们用 GFP 表达的293T 细胞作为模型, 以验证 CRISPR/Cas9-mediated 基因敲除的效率。gfp + 细胞是通过转导 HEK-293T 细胞与 pLenti GFP (vBK201a) 在0.5 的语言 (图 3b, "无病毒" 面板)。sgRNA-to-GFP/Cas9 一体矢量盒包装成 IDLV 或 ICLV 颗粒, 生产效率由 p24 ELISA 评估 (见上文;图 3a)。含有 Sp1-binding ...
讨论
IDLVs 已经开始成为体内基因编辑的首选载体, 特别是在遗传疾病的背景下, 这主要是由于与这些媒介相关联的突变风险低, 与整合交付平台22,28. 在当前手稿中, 我们试图详细说明最近在实验室28中开发的改进的 IDLV-CRISPR/Cas9 系统的生产相关的协议。
对现有平台的修改
使用此方法, 我们能够在 1 x ...
披露声明
USC-499-P 专利 (1175) 是由南卡罗来纳州大学提交的关于本手稿中所描述的工作。
致谢
我们要感谢生物学系, 杜克大学医学院和院长办公室的基础科学, 杜克大学。我们还感谢杜克病毒媒介核心成员对手稿的评论。质粒 pLenti CRISPRv2 是来自冯张 (广研院) 的礼物。LV 包装系统, 包括质粒 psPAX2, VSV, pMD2. g 和 pRSV 是来自 Trono (瑞士 EPFL) 的一种礼物。这项工作的财政支助由南卡罗来纳大学医学院提供, 补助金 RDF18080-E202 (B. K)。
材料
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| xMark Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| BD FACS | Becton Dickinson | 338960 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| 0.45-μm filter unit, 500mL | Corning | 430773 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| SW32Ti swinging-bucket rotor | Beckman Coulter | 369650 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 50mL conical centrifuge tubes | Corning | 430291 | |
| High-binding 96-well plates | Corning | 3366 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 100mm TC-Treated Culture Dish | Corning | 430167 | |
| 0.22 μM filter unit, 1L | Corning | 430513 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Tissue culture pipettes, 5 mL | Corning | 4487 | |
| Tissue culture pipettes, 10 mL | Corning | 4488 | |
| Tissue culture pipettes, 25 mL | Corning | 4489 | |
| Hemacytometer with cover slips | Cole-Parmer | UX-79001-00 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| 293FT cells | Thermo Fisher Scientific | R70007 | |
| DMEM, high glucose media | Gibco | 11965 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| Antibiotic-antimycotic solution, 100X | Sigma Aldrich | A5955-100ML | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| BES (N, N-bis (2-hydroxyethyl)-2-amino-ethanesulfonic acid) | Sigma Aldrich | B9879 - BES | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Normal mouse serum, Sterile, 500mL | Equitech-Bio | SM30-0500 | |
| Goat serum, Sterile, 10mL | Sigma | G9023 | Working concentration 1:1000 |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| psPAX2 | Addgene | 12260 | |
| pMD2.G | Addgene | 12259 | |
| pRSV-Rev | Addgene | 12253 | |
| lentiCRISPR v2 | Addgene | 52961 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsrGI | New England Biolabs | R0575S | |
| BsmBI | New England Biolabs | R0580S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
参考文献
- Horvath, P., Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 327 (5962), 167-170 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat Rev Genet. 11 (3), 181-190 (2010).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Bellec, J., et al. CFTR inactivation by lentiviral vector-mediated RNA interference and CRISPR-Cas9 genome editing in human airway epithelial cells. Curr Gene Ther. 15 (5), 447-459 (2015).
- Kennedy, E. M., et al. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease. Virology. 476, 196-205 (2015).
- Roehm, P. C., et al. Inhibition of HSV-1 Replication by Gene Editing Strategy. Sci Rep. 6, 23146 (2016).
- Wang, W., et al. CCR5 gene disruption via lentiviral vectors expressing Cas9 and single guided RNA renders cells resistant to HIV-1 infection. PLoS One. 9 (12), e115987 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Adv Genet. 87, 125-197 (2014).
- Lebbink, R. J., et al. A combinational CRISPR/Cas9 gene-editing approach can halt HIV replication and prevent viral escape. Sci Rep. 7, 41968 (2017).
- Xu, L., et al. CRISPR/Cas9-Mediated CCR5 Ablation in Human Hematopoietic Stem/Progenitor Cells Confers HIV-1 Resistance In Vivo. Mol Ther. , (2017).
- Yiu, G., Tieu, E., Nguyen, A. T., Wong, B., Smit-McBride, Z. Genomic Disruption of VEGF-A Expression in Human Retinal Pigment Epithelial Cells Using CRISPR-Cas9 Endonuclease. Invest Ophthalmol Vis Sci. 57 (13), 5490-5497 (2016).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), e147 (2014).
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol. 32 (3), 279-284 (2014).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Schaefer, K. A., et al. Unexpected mutations after CRISPR-Cas9 editing in vivo. Nat Methods. 14 (6), 547-548 (2017).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Ther. 23 (7), 627-633 (2016).
- Platt, R. J., et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159 (2), 440-455 (2014).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Res. 43 (13), 6450-6458 (2015).
- Nelson, C. E., Gersbach, C. A. Engineering Delivery Vehicles for Genome Editing. Annu Rev Chem Biomol Eng. 7, 637-662 (2016).
- Jaalouk, D. E., Crosato, M., Brodt, P., Galipeau, J. Inhibition of histone deacetylation in 293GPG packaging cell line improves the production of self-inactivating MLV-derived retroviral vectors. Virol J. 3, 27 (2006).
- Ellis, B. L., Potts, P. R., Porteus, M. H. Creating higher titer lentivirus with caffeine. Hum Gene Ther. 22 (1), 93-100 (2011).
- Hoban, M. D., et al. Delivery of Genome Editing Reagents to Hematopoietic Stem/Progenitor Cells. Curr Protoc Stem Cell Biol. 36, 1-10 (2016).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Mol Ther. 16 (12), 1968-1976 (2008).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proc Natl Acad Sci U S A. 106 (44), 18786-18791 (2009).
- Ortinski, P. I., O'Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Mol Ther Methods Clin Dev. 5, 153-164 (2017).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Mol Ther. 19 (3), 547-556 (2011).
- Berkhout, B., Verhoef, K., van Wamel, J. L., Back, N. K. Genetic instability of live, attenuated human immunodeficiency virus type 1 vaccine strains. J Virol. 73 (2), 1138-1145 (1999).
- Gomez-Gonzalo, M., et al. The hepatitis B virus X protein induces HIV-1 replication and transcription in synergy with T-cell activation signals: functional roles of NF-kappaB/NF-AT and SP1-binding sites in the HIV-1 long terminal repeat promoter. J Biol Chem. 276 (38), 35435-35443 (2001).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. J Biol Chem. 280 (22), 21545-21552 (2005).
- Ortinski, P. I., Lu, C., Takagaki, K., Fu, Z., Vicini, S. Expression of distinct alpha subunits of GABAA receptor regulates inhibitory synaptic strength. J Neurophysiol. 92 (3), 1718-1727 (2004).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. J Virol. 71 (8), 6113-6127 (1997).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. J Virol. 68 (4), 2632-2648 (1994).
- Xu, W., Russ, J. L., Eiden, M. V. Evaluation of residual promoter activity in gamma-retroviral self-inactivating (SIN) vectors. Mol Ther. 20 (1), 84-90 (2012).
- . Testing for Replication Competent Retrovirus (RCR)/Lentivirus (RCL) in Retroviral and Lentiviral Vector Based Gene Therapy Products - Revisiting Current FDA Recommendations Available from: https://sites.duke.edu/dvvc/files/2016/05/FDA-recommendation-for-RCR-testing.pdf (2017)
- . Biosafety in Microbiological and Biomedical Laboratories Available from: https://www.cdc.gov/biosafety/publications/bmbl5/bmbl.pdf (2017)
- Dull, T., et al. A third-generation lentivirus vector with a conditional packaging system. J Virol. 72 (11), 8463-8471 (1998).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。