JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
CRISPR/Cas9-중재 유전자 녹아웃 셀 분할에 대 한 Integrase 불충분 한 Lentiviral 벡터의 생산을 위한 프로토콜
요약
우리는 셀에 CRISPR/Cas9를 제공 하기 위한 차량으로 integrase 불충분 한 lentiviral 벡터 (IDLVs)의 생산 전략을 설명 합니다. 셀에서 편집 하는 신속 하 고 강력한 유전자를 중재 하는 능력, IDLVs 안전 제시와 유전자 배달에 대 한 동등 하 게 효과적인 벡터 플랫폼 integrase 유능한 벡터에 비해.
초록
Lentiviral 벡터 셀 안정적으로 세포의 광범위 한 범위를 시험 하 고 높은 수준의 유전자 발현의 중재에 대 한 자신의 능력 때문에 유전자 편집 구성 요소를 제공 하기 위한 이상적인 선택입니다. 그러나, 호스트 세포 게놈으로 통합 하는 능력 강화 insertional 변이의 위험 하 고 따라서 안전 우려를 제기 하 고 임상 조정에 있는 그들의 사용을 제한. 또한, 유전자 편집 구성의 영구 식 무차별 유전자 타겟팅의 확률이 통합 관할 lentiviral 벡터 (ICLVs) 증가 의해 전달. 대신, integrase 불충분 한 lentiviral 벡터 (IDLVs)의 새로운 세대 개발 되었습니다 많은 이러한 문제를 해결 하는. 여기 CRISPR 중재 유전자 편집 및 목록에 대 한 새로운 기능 및 향상 된 IDLV 플랫폼의 생산 프로토콜 단계 정화에 관여 하 고 설명 하는 같은 벡터의 농도 변환 및 HEK 293T를 사용 하 여 유전자 편집 효율성 셀 시연 했다. 이 프로토콜은 쉽게 확장 가능한 고 시험 세포 생체 외 고 에 비보수 있는 높은 titer IDLVs를 생성 하는 데 사용할 수 있습니다. 또한,이 프로토콜은 ICLVs의 생산을 위해 쉽게 적용할 수 있습니다.
서문
정확한 유전자 편집 유전 질병을 해결 하기 위해 새로운 전략의 개발을 포함 하는 주요 생물 의학 발전의 초석을 형성 한다. 유전자 편집 기술의 최전선 이다는 c광택 regularly-난nterspaced s읽힌 palindromic repeats (CRISPR)의 사용에 의존 하는 방법 / 처음 확인 된 Cas9 시스템 으로 바이러스 유전 물질 (참조1,2검토)의 침공에 대 한 세균의 구성 요소입니다. 아연 손가락 nucleases (ZFNs) 등 전사 활성 제 같은 이펙터 nucleases (TALENs) (참조3에서 검토 한 결과), 다른 유전자 편집 도구 CRISPR/Cas9 시스템의 주요 이점은 이다 플라스 미드 디자인의 상대적 단순 하 고 CRISPR 구성 요소 건설-기능 유전자 편집, 몇 가지 전문된 실험실에서 훨씬 더 광범위 한 연구 공동체의 확장을 강화 했다. 또한, CRISPR/Cas9 프로그래밍의 단순 하 고 다중화 대상 인식에 대 한 용량 추가 비용 효율적이 고 사용 하기 쉬운 기술로 인기를 연료 있습니다. 셀에 같은 유전자 편집 구성 요소를 제공 하는 연구자를 사용할 수 있는 다양 한 방법 중에서 바이러스 성 벡터 남아 지금까지 가장 인기 있는 하 고 효율적인 시스템.
Lentiviral 벡터 (LVs) CRISPR/Cas9 시스템에 비보에 대 한 다양 한 응용 프로그램4,5,,67의 구성 요소를 제공 하는 선택의 수단으로 떠오르고 있다. 몇 가지 주요 기능 LVs 셀 분할 및 분할 비, 낮은 immunogenicity 및 최소한의 세포 독성 (참조8에서 검토) 감염을 그들의 능력을 포함 하 여이 프로세스에 대 한 인기 있는 선택을 하십시오. 결과적으로, LV-중재 유전자 치료는 고용, HBV, 및 HSV-1, 등 전염 성 질병의 치료 뿐만 아니라 인간의 유전 질병, 낭 성 섬유 증, 신 혈관 황 반 변성 등을 기본 결함의 수정에 4 , 5 , 7 , 9 , 10 , 11. 게다가, LVs 효과적으로 수정 된 다중 유전자 단일 벡터 시스템12을 사용 하 여 고유한 게놈 loci에서 편집을 수행 하.
그러나, 호스트 게놈으로 통합 하는 LVs의 고유의 속성 기지가 될 수 있으며 종종 오답 임상 설정에서 특히 transgene 배달 차량으로 그들의 유틸리티. 또한, 안정적 통합 LVs 지속적으로 높은 수준에서 그들의 transgenes 표현, 이후이 시스템은 적합 CRISPR/Cas9; 같은 구성 요소를 편집 하는 유전자의 전달 Cas9 가이드 RNA (gRNA), 그리고 ZFNs, 같은 유사한 단백질의 overexpression 오프 대상 효과, 바람직하지 않은 돌연변이13,14,,1516 를 포함 하는 높은 수준의와 관련 된 , 17 그리고 잠재적으로 세포 독성18을 향상 시킬 수 있습니다. 따라서, 정확 하 게 달성 하기 위해 최소한의 대상에서 효과, 유전자 편집 그것은 구성 요소를 편집 하는 유전자의 변이 표현에 대 한 수 있도록 시스템을 설계 하는 것이 필수적.
최근 몇 년 동안, 전송 플랫폼의 다양 한 정도 셀16,19,,2021 (참조22검토) CRISPR/Cas9을 표현 하기 위해 개발 되었습니다. 직접 소개 적절 한 가이드 RNAs 함께 순화 Cas9 셀으로 타겟 유전자를 플라스 미드 중재 transfection16에 비해 편집에 더 효과적일 표시 했다에 의존 하는 방법이 포함 됩니다. 그 ribonucleoprotein (RNP)을 증명 하는 연구 단지의 구성 된 가이드 RNA/Cas9 입자는 이러한 부품의 단기 식 달성 하기에 충분을 제안 하는 그들의 목표에서 DNA 분열 중재 후 급속 하 게 인계 16편집 하는 강력한 유전자. 비 통합 adeno 관련 바이러스 성 벡터 (AAVs) 같은 바이러스 성 벡터 플랫폼 세포에 유전자 편집 기계를 제공 하는 대안을 제공할 수 있습니다. 불행히도, AAV capsids LVs 보다 크게 낮은 포장 기능 보유 (< 5 kb)는 심각 하 게 단일 벡터 (검토 참조8) 내에서 다중 구성 요소 CRISPR 툴킷을 포장 하는 그들의 능력을 방해 한다. 히스톤 deacetylases (예를 들면, 나트륨 낙 산 염23)을 억제 하거나 (예를 들어, 카페인24) 세포 주기를 방해 하는 화합물의 추가 lentiviral titers을 증가 표시 되었습니다 지적 가치가 있다. 몇 가지 단점, 낮은 생산 효율성 등 감소 바이러스 titers을 통해 생성 하는 바이러스의 낮은 변환 효율성 있는 최근 진도도 불구 하 고 지금까지 개발 된 과도 식 시스템 장애가 여전히는 같은25를접근 한다.
LVs의 포장 능력을 결합 하 여 셀에서 AAV 같은 episomal 유지 보수의 추가 혜택으로 Integrase 불충분 한 lentiviral 벡터 (IDLVs) 유전자 배달 차량 개발에서 큰 발전을 나타냅니다. 이러한 기능을 통합 벡터, 마주-à-마주 연속 overexpression 잠재적으로 차적인 요소 및 통합 중재 변이와 관련 된 주요 문제를 크게 우회 하는 IDLVs 있습니다. 그것은 이전 IDLVs 성공적으로 episomal 유전자 식26,27향상을 수정할 수 있습니다 시연 했다. CRISPR/Cas9 IDLV 중재 배달에 관하여 낮은 생산 titers 및 낮은 표현의 integrase 능숙 lentiviral 시스템 상대적인 episome 부담 게놈 게놈 편집 제공 하기 위한 진실 fide 도구로 그들의 유틸리티 제한 유전자 변형 구문입니다. 우리는 최근 transgene 표현과 IDLV 생산과 관련 된 바이러스 titers 크게 바이러스 성 식 카세트28내 녹음 방송 요인 s p 1에 대 한 사이트 바인딩을 포함 하 여 향상 된 시연. 수정 된 IDLVs 튼튼하게 CRISPR 중재 하는 유전자는 해당 ICLV 중재에 비해 최소한의 대상에서 돌연변이 유도 하는 동안 생체 외에서 (HEK 293T 세포)에서 그리고 vivo에서 (포스트 mitotic 뇌 신경)에 편집 지원 시스템28. 전반적으로, 우리는 소설을 개발, 소형, 모든-에-하나의 CRISPR 툴킷 IDLV 플랫폼에서 실시 하 고 향상 된 유전자 편집에 대 한 배달 차량을 사용 하 여 다양 한 이점을 설명.
여기, IDLV-CRISPR/Cas9 시스템의 생산 프로토콜은 설명, 어셈블리, 정화, 농도, 그리고이 벡터의 유전자 편집 효능을 확인 하기 위해 전략 뿐만 아니라 IDLVs, 적정에 관련 된 다양 한 단계를 포함 하 여. 이 프로토콜은 다른 수 사관 들의 요구를 충족 하도록 쉽게 확장 가능한 고 성공적으로 1 x 1010 단위 (TU) 시험의 범위에서 titers와 LV 벡터를 생성 하도록 설계 되었습니다 / mL. 이 프로토콜을 통해 생성 된 벡터 효율적으로 여러 다른 세포 유형, 어려운 transduce 배아 줄기 세포, 조 혈 모 세포 (T 세포와 대 식 세포)를 포함 하 여 그리고 교양 및 시내 vivo-감염에 이용 될 수 있다 주입된 하는 신경. 또한, 프로토콜은 동등 하 게 잘 비슷한 수량 integrase 유능한 lentiviral 벡터의 생산에 적합 합니다.
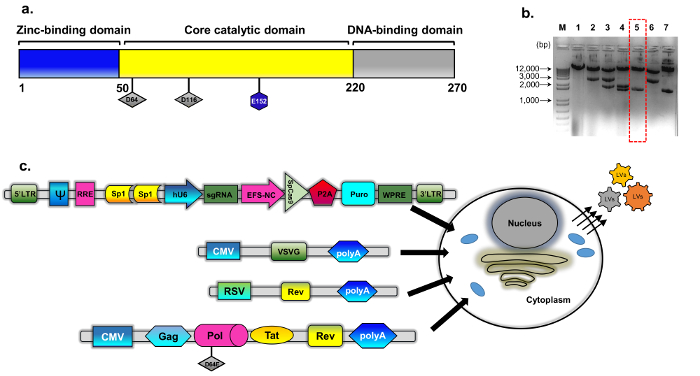
그림 1: IDLV 포장. 수정 된 플라스 미드는 psPAX2에서 파생 된 야생 타입 integrase 단백질 (b) 의 (a) 회로도 (방법, 플라스 미드 건설에 대 한 자세한 내용은 참조). 클론 돌연변이 integrase 클론에 대 한 검사의 대표 agarose 젤 이미지. DNA 샘플 표준 플라스 미드 DNA 분리 미니 키트를 사용 하 여 준비 EcoRV와 SphI 소화에 의해 분석 되었다. 제대로 소화 클론 (5 번, 파선된 빨간색 상자) 직접 (생어) 연속 D64E 대체 INT에 대 한 추가 확인 했습니다. Integrase 불충분 한 포장 카세트 pBK43 선정 됐다. (c) 과도 transfection 프로토콜의 회로도 고용 IDLV-CRISPR/Cas9 벡터 생성 하 293T 세포 VSV G, 포장, 및 transgene 카세트 (s p 1-CRISPR/Cas9-에-하나의 플라스 미드)와 페 보여주는. 바이러스 성 입자를 세포 막에서 밖으로 새싹 (transgene 카세트에서 표현) 벡터의 길이 RNA 포함. 그리고 IDLV-포장 시스템의 두 번째 세대 사용, 규제 단백질 문신 포함 목사 계 식 추가 별도 카세트 (RSV-레 브-플라스 미드)에서 보완. Abbrev: LTR 긴 터미널 반복, VSV G vesicular 구내 염 바이러스 G-단백질, pCMV 세포 발기인; 라우 스 육 종 바이러스 (RSV) 발기인; RRE-(계 응답 요소)입니다. 다른 규제 요소 식 카세트에 포함 Sp1 바인딩 사이트, 레 브 응답 요소 (RRE), 마 형 간염 바이러스 Posttranscriptional 규제 요소 (WPRE), 코어-신장 요인 1α 발기인 (EFS-NC), 벡터 포장 요소 ψ (psi), 인간의 세포 (hCMV) 발기인, 그리고 인간의 U6 발기인. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
프로토콜
1. HEK 293T 세포 배양과 Transfection에 대 한 셀을 시드
참고: 인간 미 발달 신장 293T DMEM, 10% 소 송아지 혈 청 철 및 성장 발기인 및 항생제 antimycotic 솔루션 x 1 보충으로 보충 하는 높은 포도 당 미디어에서에서 재배 되 고 (HEK 293T) 세포 (100 x 솔루션 포함 10000 단위 페니실린 10 mg 스와 25 µ g 암포 B mL 당). 미디어는 또한 1 x 나트륨 pyruvate, 1 x 비 필수 아미노산 믹스, 그리고 2 mM L-글루타민 보충 (재고 200 mM L-alanyl-L-글루타민 dipeptide 0.85 %NaCl에서). 셀 (대략 성장 표면적은 55 c m ²) 100 m m 조직 문화 접시에 교양. 1시 10분의 하위 재배 비율 하위 모든 2-3 일 배양 사용 됩니다. 트립 신-EDTA 0.05% 통행 사이 세포의 분리에 사용 됩니다. 실험 간에 일관성을 유지, 세포 성장, transfection 효율에에서 어떤 변화를 모니터링 하 고 생산 벡터 다른 많은/일괄 전환 시 테스트 종 아리 세라 추천 우리.
- 10cm 조직 문화 접시에 뿌리기에 의해 낮은 통로 셀 (그것 후에 통로 15 또는 성장 감속 경우 셀을 사용 하지 않도록 권장)를 사용 하 여 새로운 문화를 시작 합니다. DMEM 세포 성장에 대 한 10% 혈 청으로 보충을 사용 합니다. 5% CO2 표준 조직 문화 인큐베이터에서 37 ° C에서 세포 성장. 표준 hemocytometer를 사용 하 여 셀에 대 한 모든 후속 단계.
- 일단 셀 도달 90-95 %confluent 성장, 15 cm로 다시 조직 문화 접시 (아래 단계 1.3-1.5).
- 다시 시드하, aspirate 미디어는 합칠에서 접시와 부드럽게 살 균 1 x PBS와 린스. 3-5 분 추가 8 mL 혈 청 분리 시 비활성화 하 여 단일 셀을 만드는 데 10 mL 혈 청 학적인 피 펫으로 10-15 회를 triturate 포함 된 미디어에 대 한 37 ° C에서 셀 분리 시 약 (예를 들어, 트립 신-EDTA)의 2 개 mL를 품 어 현 탁 액입니다. 약 4 x 106 셀/mL의 셀 밀도를 문화 미디어에 셀 resuspend
- 기판에 부착을 강화 하려면 사전에 0.2% 젤라틴, 젤라틴 접시 당 8 mL을 추가와 15 cm 판 코트. 접시의 표면에 걸쳐 균일 하 게 확산, 10 분 동안 실내 온도에 품 어 그리고 액체에서 사이 펀.
- 따뜻한 (37 ° C) HEK 293 T 미디어 (1 단계 아래 참고 참조) 25 mL에 각 격판덮개의 전체 볼륨을 가져오고 셀 (총 ~ 1 x 107 셀/플레이트)의 2.5 mL을 추가 하 여 판 씨. 하룻밤 또는 70-80 %confluency이 때까지 접시 5% CO2 와 37 ° c를 품 어.
참고: 6 15 cm 접시 최대 생산을 위해 사용할 수 있습니다. 4 개 이상 접시 (근거에 대 한 프로토콜의 5 단계를 참조)를 사용 하는 경우 20 ~ 22 ml 접시 당 미디어의 볼륨을 조정 합니다.
2. transfecting HEK 293T 세포 칼슘 인산 염 기반 프로토콜을 사용 하 여
-
Transfection 시 약
- BES 버퍼 솔루션 게시판 (50mm BES, 280 m m NaCl, 1.5 m m 나2HPO4) x 2를 준비 하려면 16.36 g의 NaCl, 10.65 BES의 결합 (N, N-비스 (2-hydroxyethyl)-2-아미노-ethanesulfonic 산), 나2HPO40.21 g. 추가 이중 증 류 H2O (dd-H2O) 최대 900 mL. 해산, 6.95 1 M NaOH로 pH를 적정와 볼륨 0.22 μ M 필터 장치 통해 1 L. 필터. -20 ° c.에 게
- 1 M CaCl2를 준비 합니다. 솔루션 0.22 μ M 필터를 통해 필터링. 4 ° c.에 게
- 하루 전에 시드 했다 번호판을 관찰 합니다. 그들은 70-80 %confluency 도달 세포 transfection 준비 되어 있다.
- 판에서 오래 된 미디어를 발음 하 고 부드럽게 혈 청 하지 않고 갓 미디어를 추가 합니다.
참고: 선택한 플라스 미드 및 그들의 준비에 대 한 자세한 보충 파일 1-플라스 미드 를 참조. - 15 mL 원뿔 튜브에 aliquoting 4 플라스 미드에 의해 플라스 미드 믹스를 준비 합니다. 한 15 cm 접시 준비에 대 한 사용은 CRISPR/Cas9-전송 벡터 (pBK198 또는 pBK189), 25 µ g의 pBK43의 37.5 µ g (psPAX2-D64E), 12.5 µ g pMD2.G, 및 이전 (그림 1c)의 6.25 µ g.
- 플라스 미드에 312.5 µ L 1 M CaCl2 를 추가 합니다. 이 살 균 dd-H20의 최대 1.25 mL를 추가 합니다.
- 천천히 (drop-wise) vortexing 믹스 2 x BBS 솔루션의 1.25 mL을 추가. 실 온에서 30 분 동안 품 어.
- 각 15 cm 플레이트 (접시 당 2.5 mL)에 dropwise transfection 혼합물을 추가 합니다. 플레이트를 부드럽게 소용돌이 5% CO2 2-3 h 37 ° C에서 품 어. 그 후, 접시 당 혈 청 2.5 mL (10%)를 추가 하 고 잠복기 계속 하룻밤 (12 월 18 일 h).
참고: 일부 연구소 미디어 pH를 안정 시키기 위해 3% CO2 인큐베이터를 사용 합니다. 그러나, 우리 3%와 5% CO2사이 transfection 효율에 어떤 차이 관찰 하지 않습니다. 또한, 카 포4 침전의 크기가 transfection 효율;에 대 한 중요 한 transfection 혼합 셀에 그것의 추가 하기 전에 명확 하 게 있다. 혼합 되 면 흐린 인큐베이션 기간 동안, 준비 신선한 2 x BBS (pH = 6.95).
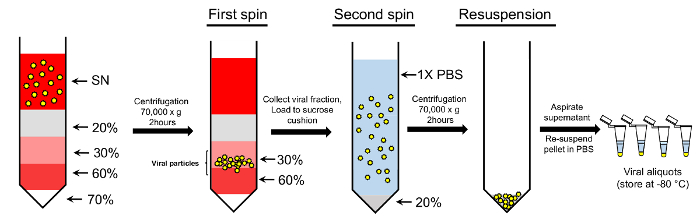
그림 2: 더블-자당 그라데이션 프로토콜을 사용 하 여 바이러스 성 입자의 농도. 상쾌한 (SN)에서 수집 된 바이러스 성 입자는 그라데이션 자당 기온 변화도에 로드 됩니다. 70%, 60%, 30%, 및 20% 자당 솔루션은 그라디언트를 만드는 데 사용 됩니다. 원심 분리, 다음 30-60% 자당 분수에서 수집 하는 입자는 추가 20% 자당 쿠션에 로드 하 고 시 켰 던. 순화 된 바이러스 입자를 포함 하는 마지막 펠 릿 추가 사용에 대 한 1 x PBS에 resuspended (대 한 자세한 내용은 텍스트 참조). 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
3입니다. 다음날 Transfection
- 그들은 100 %confluency 시점에서 아무 세포 죽음에 거의 근접 하는 보장 하기 위해 세포를 관찰 합니다. 각 접시에 신선한 DMEM + 10% 혈 청 25 mL를 추가 하 여 미디어를 교체 합니다. 5% CO2 는 추가 48 h에 대 한 37 ° C에서 배양을 계속 한다.
참고: 이상의 6 15 cm 번호판 (주의 단계 5.2 참조)를 사용 하는 경우 단계 3.1에서에서 미디어의 볼륨을 조정 합니다.
4. 바이러스를 수확
- 조심 스럽게 transfected 세포 조직 문화 피펫은 멸 균 10 mL 및 50 mL 원심 분리기 튜브에서 수영장을 사용 하 여 포함 된 모든 조직 배양 접시에서는 상쾌한을 수집 합니다.
- 탁상 원심 분리기를 사용 하 여 10 분 동안 400-450 x g에서 원심 분리에 의해 중단을 취소 합니다. 0.45 μ m 진공 필터 장치 통해 상쾌한 필터링.
참고: 필터링 된 상쾌한 수 농도, 계속 하기 전에 최대 4 일 동안 4 ° C에서 저장 또는 aliquoted-80 ° c.에 저장 IDLV-s p 1-CRISPR/Cas9 (비-농축) 바이러스 준비의 예상된 titers 2 ~ 10 배 해야7 화/mL (titer 결정을 위한 6 단계 참조). 그러나, 쓰는 중지 및 재개 기능 titers에 10-20% 손실에 결과의 각 라운드로 여러 freeze-thaw 주기를 바이러스 준비 하지 마십시오.
5입니다. Ultracentrifugation에 의해 바이러스 성 입자의 농도
참고: 두 단계를 포함 하는 정화의 더블 자당 방법 활용: 자당 그라데이션 단계와 자당 쿠션 단계 (그림 2).
- 자당 기온 변화도 만들려면 다음 순서로 원뿔 ultracentrifugation 튜브 로드: 0.5 mL 70% 자당 (1 x PBS 해산) 0.5 mL 60% 자당 (DMEM에 녹아 있는), 1 mL 30% 자당 (DMEM에 녹아 있는), 그리고 2 mL 20% 자당 (1 x PBS에 용 해).
-
그라디언트를 신중 하 게 바이러스를 포함 하는 상쾌한을 추가 합니다. 4 15 cm 접시에서 상쾌한의 총 볼륨은 100 mL, 스핀 당 6 ultracentrifugation 튜브 표면에 뜨는 바이러스의 전체 볼륨을 처리 하는 데 사용.
- 이 단계에 사용 하는 각 ultracentrifugation 튜브는 최대 30 mL (를 포함 하 여 자당의 볼륨)의 볼륨의 용량, 튜브, 적어도 10%를 떠나 중 동등 하 게 바이러스 상쾌한 배포 headspace 흘림을 방지 하기 위해.
- 풀링된 바이러스 상쾌한의 마지막 볼륨 쉽게 6 ultracentrifugation 튜브 내에서 수용 될 수 있도록 각 실험에 대 한 더 이상의 15 cm 번호판을 사용 하는 경우 문화 볼륨 접시 당 20 ~ 22 mL을 조정 합니다.
- 채우기 ultracentrifugation 적어도 3-4에 그들의 총 볼륨 용량 튜브, 튜브의 파손 원심 분리, 샘플 및/또는 장비 손상의 가능한 손실을 하는 동안 발생할 수 있습니다.
- 70000 x g 17 ° C에서 2 h에서 PBS 및 원심 분리기 샘플 x 1 튜브 균형 (회전자 세부 사항에 대 한 재료의 표 참조).
참고: 가속 중 자당 계층의 중단을 방지 하기 위해 천천히 가속 화 하기 위해로 터 200 rpm 회전 급강하의 처음 3 분 동안 ultracentrifuge를 설정 합니다. 유사 하 게, 천천히 0 rpm 회전 급강하의 끝에 3 분 이상 200 rpm에서 회전자를 감속에 ultracentrifuge를 설정 합니다. - 신중 하 게 깨끗 한 튜브 (그림 2)에 30-60% 자당 분수를 수집 합니다. 감기 1 추가 풀링된 분수 PBS x 100 ml; 볼륨을가지고 여러 번 왔다 갔다 pipetting으로 혼합.
- 신중 하 게 자당 쿠션에 바이러스 준비를 레이어 링 여 자당 쿠션 단계를 진행 합니다. 이 위해, 뒤에 튜브 당 바이러스 솔루션의 20 ~ 25 mL 튜브에 (1 x PBS)에서 20% 자당의 4 mL를 추가 합니다. 튜브는 3-4 분 미만 하는 경우 전체, 살 균 1 x PBS와 충전을.
- 신중 하 게 균형 그리고 원심에서 70000 x g 17 ° C에서 2 시간에 대 한 샘플 전에. 부는 상쾌한 어 고 나머지 액체를 종이 타 올에 튜브를 거꾸로 하 여 배출.
- 펠 릿에서 모든 액체를 제거 하려면 나머지 방울을 발음. 이 단계에서 바이러스를 포함 하는 펠 릿 겨우 작은 반투명 반점으로 표시 되어야 합니다.
- 1 x PBS의 70 µ L 첫 번째 튜브와 철저 하 게 정지를 pipetting, 이후 다음 튜브에 정지를 전송 및 추가 계속 하기 전에, 모든 펠 릿 resuspended 때까지 혼합 하 여 펠 릿을 resuspend.
- 이전과 PBS와 믹스 감기 1의 추가 50 µ L 튜브를 헹 구 십시오. 최종 서 스 펜 션의 결합 된 볼륨 ~ 120 µ L 이며, 약간 밀키; 나타납니다 확인 30에 대 한 10000 x g에서 원심 분리 하 여 취소 탁상 microcentrifuge에 s.
- 신선한 microfuge 관에는 상쾌한 전송, 10 µ L aliquots, 고-80 ° c.에서 그들을 저장합니다
참고: lentiviral 샘플에 반복된 freeze-thaw 주기를 수행 하지 마십시오. 원심 분리가 필요한 때를 제외 하 고 노력 해야 조직 문화 후드의 나머지 단계를 수행 하려고 또는 적절 한 biosafety 조치 (내용 참조)를 사용 하 여 조직 문화 객실을 지정.
6입니다. 바이러스 Titers의 추정
-
p24 -효소-연결 된 immunosorbent 분석 결과 (ELISA) 메서드
참고: 분석 결과 수행으로 에이즈-1 p24 를 위한 NIH 에이즈 백신 프로그램의 지시 당 항 원 캡처 시험 키트 ( 재료의 표참조) 수정29높은 바인딩 96 잘 접시를 사용 하 여.- 다음 날 씻어 200 µ L 0.05%로 세 번 우물 차가운 PBS (PBS-T 솔루션)에 트윈 20. 1:1500 1 x PBS에 희석에 항 체 단일 클로 널 반대로 p24 의 100 µ L 플레이트 코트와 4 ° c.에서 밤새 품 어
- 일반적인 바인딩 제거, 차단 PBS;에서 200 µ L 1 %BSA 접시 200 µ L 0.05%로 세 번 세척 실 온에서 1 시간 이상에 대 한 차가운 PBS (PBS-T 솔루션)에 트윈 20.
-
샘플을 준비: 집중된 벡터 준비, 대 한 약 1 µ L 샘플의 희석 Triton X-100 (최종 농도 10%)의 dd-H20과 10 µ L의 89 µ L을 추가 하 여. 비 집중 준비에 대 한 ten-fold 희석된 샘플 (샘플의 10 µ L 트라이 톤 X-100 (최종 농도 10%)의 dd-H20과 10 µ L의 80 µ L 추가)을 준비 합니다.
참고: 샘플을 저장할 수 있습니다이 단계에서-20 ° C에서 나중 사용을 위해 시간의 연장된 기간에 대 한. - 에이즈-1 표준 적용 (시작 농도 5 ng/mL)와 2-fold 직렬 희석 하 여 준비 합니다.
- 설정 1:10, 000, 0.2% 트윈 20, 1 %BSA 보충 RPMI 1640 년에 (1: 100 미리 희석된 주식)에서 집중된 샘플을 희석 1:50, 000, 그리고 1:250,000 희석. 비 집중 샘플 희석 (1시 10분에서 미리 희석 주식) 설정 1: 500, 1: 2500, 그리고 1:12,500 희석 하 0.2% 트윈 20, 1 %BSA 보충 RPMI 1640 년에.
- Triplicates에서 접시에 샘플을 적용 하 고 하룻밤 4 ° c.에 품 어
- 다음 날, 세척 우물 6 번 하 고 37 ° C 4 h 100 µ L polyclonal 토끼 안티-p24 항 체, RPMI 1640, 10 %FBS, 0.25 %BSA, 및 2% 정상 마우스의 혈 청 (NMS)에 희석된 1:1000에서 품 어.
- 6 배 위와 같이 세척 하 고 5% 정상 염소 혈 청, 2 %NMS, 0.25 %BSA 및 0.01% 보충 RPMI 1640에 IgG 희석 1:10,000 염소 안티 토끼 양 고추냉이 과산화 효소와 함께 1 시간 동안 37 ° C에서 품 어 트윈 20.
- 위의 접시를 세척 하 고 15 분 동안 실 온에서 TMB 과산화 효소 기질으로 품 어.
- 1 N HCL의 100 µ L을 추가 하 여 반응을 중지 합니다. 450에 샘플의 흡 광도 측정 nm에서 흡 광도 사용 하 여 플레이트 리더.
-
기자는 형광 강도 측정
- FACS 메서드
참고: 셀에 GFP 신호 고갈의 범위 수 있습니다 정확 하 게 추정 하실 cytometry 통해 transduced 셀의 의미 형광 강도 측정 하 여. FACS 데이터 분석, 프레 젠 테이 션, 및 해석에 대 한 최근 종이 28 을 참조 하십시오. 프로토콜은 다음과 같이 설명 되어 있습니다.- 1 x PBS에 바이러스 준비 (10-510-1 )에서 준비의 ten-fold 직렬 희석을 확인 합니다.
- 잘 당 2 mL의 최종 볼륨에서 6 잘 플레이트의 각 음에 약 5 x 105 293T 세포 씨앗 셀에 각 바이러스 희석의 10 µ L을 추가 하 고 셀 48 h에 대 한 37 ° C에서 품 어.
- FACS 분석에 대 한 셀을 다음과 같이 수확: 0.05% 트립 신-EDTA 용액의 200 µ L을 추가 하 고 37 ° C 5 분 추가 2 mL의 완전 한 DMEM 미디어에서 세포를 품 어 15 mL 원뿔 튜브 샘플을 수집.
- 4 ° C에 400 x g에서 원심 분리 하 여 세포를 작은 고 차가운 1 x PBS의 500 µ L에 펠 릿을 resuspend.
- 고정,이 서 스 펜이 션에 4% 포름알데히드 솔루션의 동일한 볼륨을 추가 하 고 실 온에서 10 분 동안 품 어.
- 고정된 셀 펠 렛 및 1 x PBS의 1 mL에 resuspend. Ortinski 외 에 설명 된 대로 GFP 식 FACS 악기를 사용 하 여 분석 28 잠시, 수식을 사용 하 여 다음 바이러스 기능 titer를 결정:
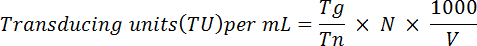
참고: 여기 Tg = GFP-긍정적인 세포 계산; Tn = 계산; 세포의 총 수 N = 불리고; 세포의 총 수 V = (µ L)에서 볼륨을 사용 하는 변환에 대 한. 예: 1 x 106 세포 바이러스의 10 µ L로 불리고 있었다, 2 x 104 의 세포 라면 계산 및 5 x 103 GFP-긍정적인, 기능 titer 것 위의 방정식에 따라 했다:
- GFP-긍정적인 세포 계산
- 감염 (MOI) 변환에 대 한 사용의 다양성을 계산 합니다. MOIs 높은 변환 효율성이 증가 광범위 한 범위 MOIs (1-10)를 테스트 합니다.
- Transfection, 전에 대략 3-4 배 잘 당 105 셀 6 잘 플레이트 씨앗. 일단 셀 도달 > 90 %confluency (24 시간) 이내 transduce pre-determined MOIs에서 순화 된 바이러스.
- GFP 신호에 변화에 대 한 1-7 일에 대 한 표준 조직 문화, 5% CO2 와 37 ° C에서 접시와 정기적으로 모니터 세포를 품 어.
- GFP-긍정적인 세포의 수를 계산 하는 형광 현미경 (계획 4 X 목표, 0.1 한다 40 배 확대) 장착 GFP 필터 세트 (여기 파장 470 nm, 방출 파장-525 nm), 순진한 (유엔 transduced) 셀을 사용 하 여 인구를 설정 GFP 부정과 긍정적인 세포.
- 희석 비율 및 다음 수식을 사용 하 여 볼륨을 조정 하 여 최종 titer를 예상:
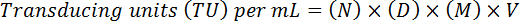
참고: 여기에서, N = GFP-긍정적인 세포, D의 수 희석 요인, M = 확대 비율 (보통 20 X), V = = 변환에 사용 되는 바이러스의 볼륨. 예를 들어 20 GFP-긍정적인 세포 (N) 10-4 (1:10, 000) 20 X 10 µ L 샘플 (V)의 희석에서 계산에 대 한 배율 (M) (D) 귀 착될 것입니다 (10) x x x (20) (20 x 104)의 기능 titer (100 *) = 4 × 108 화/mL.
(* mL 당 조정 하기)
- FACS 메서드
결과
IDLV-CRISPR/Cas9 벡터의 녹아웃 효율성의 유효성 검사
우리는 유전자 녹아웃 CRISPR/Cas9-중재의 효율성을 확인 하기 위해 모델 GFP 표현 293T 세포를 사용. GFP + 셀 HEK 293T 세포 pLenti-GFP (vBK201a) 0.5 (그림 3b, "no 바이러스" 패널)의 나에의 변환에 의해 생성 되었다. SgRNA-GFP/Cas9 모든-에-하나의 벡터 카세트 IDLV 또는 ICLV 입자에 포장 되었다 그리고 생?...
토론
IDLVs는 vivo에서 유전자-편집, mutagenesis 배달 플랫폼22 통합에 비해 이러한 벡터와 관련 된의 낮은 위험 때문에 크게 유전 질병의 맥락에서 특히 선택의 차량으로 등장 하기 시작 했습니다. , 28. 현재 원고, 우리는 우리의 실험실28에 최근 개발한 향상 된 모든-에-하나의 IDLV-CRISPR/Cas9 시스템의 생산과 관련 된 프로토콜에 자세히 설명...
공개
USC-499-P 특허 (1175)이이 원고에 설명 된 작업에 관하여 사우스 캐롤라이나 대학에 의해 제기 되었다.
감사의 말
우리는 신경 생물학의 학과 듀크 대학의과 대학 및 기초 과학, 듀크 대학에 대 한 학장의 사무실 감사 하 고 싶습니다. 우리는 또한 원고에 의견에 대 한 공작 바이러스 성 벡터 코어의 회원을 감사합니다. 플라스 미드 pLenti CRISPRv2 Feng 장 (브로드 연구소)에서 선물 했다. LV-포장 시스템 플라스 미드 psPAX2를 포함 하 여, VSV G, pMD2.G, pRSV 계 Didier Trono (EPFL, 스위스)에서 종류 선물 했다. 이 작품에 대 한 재정 지원에의 사우스 캐롤라이나의과 대학에 의해 제공 된 RDF18080 E202 (B.K) 부여 합니다.
자료
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| xMark Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| BD FACS | Becton Dickinson | 338960 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| 0.45-μm filter unit, 500mL | Corning | 430773 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| SW32Ti swinging-bucket rotor | Beckman Coulter | 369650 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 50mL conical centrifuge tubes | Corning | 430291 | |
| High-binding 96-well plates | Corning | 3366 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 100mm TC-Treated Culture Dish | Corning | 430167 | |
| 0.22 μM filter unit, 1L | Corning | 430513 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Tissue culture pipettes, 5 mL | Corning | 4487 | |
| Tissue culture pipettes, 10 mL | Corning | 4488 | |
| Tissue culture pipettes, 25 mL | Corning | 4489 | |
| Hemacytometer with cover slips | Cole-Parmer | UX-79001-00 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| 293FT cells | Thermo Fisher Scientific | R70007 | |
| DMEM, high glucose media | Gibco | 11965 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| Antibiotic-antimycotic solution, 100X | Sigma Aldrich | A5955-100ML | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| BES (N, N-bis (2-hydroxyethyl)-2-amino-ethanesulfonic acid) | Sigma Aldrich | B9879 - BES | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Normal mouse serum, Sterile, 500mL | Equitech-Bio | SM30-0500 | |
| Goat serum, Sterile, 10mL | Sigma | G9023 | Working concentration 1:1000 |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| psPAX2 | Addgene | 12260 | |
| pMD2.G | Addgene | 12259 | |
| pRSV-Rev | Addgene | 12253 | |
| lentiCRISPR v2 | Addgene | 52961 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsrGI | New England Biolabs | R0575S | |
| BsmBI | New England Biolabs | R0580S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
참고문헌
- Horvath, P., Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 327 (5962), 167-170 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat Rev Genet. 11 (3), 181-190 (2010).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Bellec, J., et al. CFTR inactivation by lentiviral vector-mediated RNA interference and CRISPR-Cas9 genome editing in human airway epithelial cells. Curr Gene Ther. 15 (5), 447-459 (2015).
- Kennedy, E. M., et al. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease. Virology. 476, 196-205 (2015).
- Roehm, P. C., et al. Inhibition of HSV-1 Replication by Gene Editing Strategy. Sci Rep. 6, 23146 (2016).
- Wang, W., et al. CCR5 gene disruption via lentiviral vectors expressing Cas9 and single guided RNA renders cells resistant to HIV-1 infection. PLoS One. 9 (12), e115987 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Adv Genet. 87, 125-197 (2014).
- Lebbink, R. J., et al. A combinational CRISPR/Cas9 gene-editing approach can halt HIV replication and prevent viral escape. Sci Rep. 7, 41968 (2017).
- Xu, L., et al. CRISPR/Cas9-Mediated CCR5 Ablation in Human Hematopoietic Stem/Progenitor Cells Confers HIV-1 Resistance In Vivo. Mol Ther. , (2017).
- Yiu, G., Tieu, E., Nguyen, A. T., Wong, B., Smit-McBride, Z. Genomic Disruption of VEGF-A Expression in Human Retinal Pigment Epithelial Cells Using CRISPR-Cas9 Endonuclease. Invest Ophthalmol Vis Sci. 57 (13), 5490-5497 (2016).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), e147 (2014).
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol. 32 (3), 279-284 (2014).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Schaefer, K. A., et al. Unexpected mutations after CRISPR-Cas9 editing in vivo. Nat Methods. 14 (6), 547-548 (2017).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Ther. 23 (7), 627-633 (2016).
- Platt, R. J., et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159 (2), 440-455 (2014).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Res. 43 (13), 6450-6458 (2015).
- Nelson, C. E., Gersbach, C. A. Engineering Delivery Vehicles for Genome Editing. Annu Rev Chem Biomol Eng. 7, 637-662 (2016).
- Jaalouk, D. E., Crosato, M., Brodt, P., Galipeau, J. Inhibition of histone deacetylation in 293GPG packaging cell line improves the production of self-inactivating MLV-derived retroviral vectors. Virol J. 3, 27 (2006).
- Ellis, B. L., Potts, P. R., Porteus, M. H. Creating higher titer lentivirus with caffeine. Hum Gene Ther. 22 (1), 93-100 (2011).
- Hoban, M. D., et al. Delivery of Genome Editing Reagents to Hematopoietic Stem/Progenitor Cells. Curr Protoc Stem Cell Biol. 36, 1-10 (2016).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Mol Ther. 16 (12), 1968-1976 (2008).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proc Natl Acad Sci U S A. 106 (44), 18786-18791 (2009).
- Ortinski, P. I., O'Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Mol Ther Methods Clin Dev. 5, 153-164 (2017).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Mol Ther. 19 (3), 547-556 (2011).
- Berkhout, B., Verhoef, K., van Wamel, J. L., Back, N. K. Genetic instability of live, attenuated human immunodeficiency virus type 1 vaccine strains. J Virol. 73 (2), 1138-1145 (1999).
- Gomez-Gonzalo, M., et al. The hepatitis B virus X protein induces HIV-1 replication and transcription in synergy with T-cell activation signals: functional roles of NF-kappaB/NF-AT and SP1-binding sites in the HIV-1 long terminal repeat promoter. J Biol Chem. 276 (38), 35435-35443 (2001).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. J Biol Chem. 280 (22), 21545-21552 (2005).
- Ortinski, P. I., Lu, C., Takagaki, K., Fu, Z., Vicini, S. Expression of distinct alpha subunits of GABAA receptor regulates inhibitory synaptic strength. J Neurophysiol. 92 (3), 1718-1727 (2004).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. J Virol. 71 (8), 6113-6127 (1997).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. J Virol. 68 (4), 2632-2648 (1994).
- Xu, W., Russ, J. L., Eiden, M. V. Evaluation of residual promoter activity in gamma-retroviral self-inactivating (SIN) vectors. Mol Ther. 20 (1), 84-90 (2012).
- . Testing for Replication Competent Retrovirus (RCR)/Lentivirus (RCL) in Retroviral and Lentiviral Vector Based Gene Therapy Products - Revisiting Current FDA Recommendations Available from: https://sites.duke.edu/dvvc/files/2016/05/FDA-recommendation-for-RCR-testing.pdf (2017)
- . Biosafety in Microbiological and Biomedical Laboratories Available from: https://www.cdc.gov/biosafety/publications/bmbl5/bmbl.pdf (2017)
- Dull, T., et al. A third-generation lentivirus vector with a conditional packaging system. J Virol. 72 (11), 8463-8471 (1998).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유