Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un protocole pour la Production de vecteurs LENTIVIRAUX intégrase déficient pour CRISPR/Cas9-mediated Gene Knockout dans les cellules en Division
Dans cet article
Résumé
Nous décrivons la stratégie de production de vecteurs LENTIVIRAUX intégrase déficient (IDLVs) comme des véhicules de livraison CRISPR/Cas9 aux cellules. Avec une capacité de médiation rapide et robuste de gène d’édition dans les cellules, IDLVs présentent une plus sûre et plateforme vector tout aussi efficaces pour la livraison de gène par rapport aux vecteurs capables d’intégrase.
Résumé
Vecteurs LENTIVIRAUX sont un choix idéal pour la prestation des modules d’édition de gène aux cellules en raison de leur capacité pour stablement transduction une vaste gamme de cellules et de médiation de hauts niveaux d’expression de gène. Cependant, leur capacité à s’intégrer dans le génome de la cellule hôte augmente le risque de mutagénicité insertional soulève des questions de sécurité et limite leur utilisation en milieu clinique. En outre, l’expression persistante de gène-montage de composants livrés par ces hausses de vecteurs LENTIVIRAUX capables d’intégration (ICLVs) la probabilité de ciblage de gènes ubiquistes. Comme alternative, une nouvelle génération de vecteurs LENTIVIRAUX intégrase déficient (IDLVs) a été développée qui aborde un grand nombre de ces préoccupations. Le protocole de production d’une plateforme jusque nouvel et améliorée pour les gènes CRISPR montage et liste les étapes de la purification et de concentration de ces vecteurs est décrite ici transduction et leurs efficacité édition de gène à l’aide de HEK-293 t cellules a été démontrée. Ce protocole est facilement évolutif et peut être utilisé pour générer des IDLVs de titre élevé qui sont capables de faire des cellules in vitro et in vivo. En outre, le présent protocole peut être facilement adapté pour la production de ICLVs.
Introduction
Gène précis édition constitue la pierre angulaire des grandes avancées biomédicales qui impliquent le développement de nouvelles stratégies pour lutter contre les maladies génétiques. À l’avant-garde des technologies de modification génétique est la méthode en s’appuyant sur l’utilisation de la clustered regularly -jenterspaced short palindromic repeats (CRISPR) / système de Cas9 qui a été initialement identifié dans le cadre de l’immunité contre l’invasion virale matériel génétique (révisé dans les références1,2) bactérienne. Un avantage majeur du système CRISPR/Cas9 autres gène-montage des outils, tels que les nucléases de doigt de zinc (ZFNs) et les nucléases d’effecteur comme activateur de transcription (TAPS) (révisés en référence3), est la relative simplicité de conception de plasmide et construction des composants de CRISPR — une caractéristique qui a alimenté l’expansion du gène-montage de quelques laboratoires spécialisés à une communauté de recherche beaucoup plus large. En outre, la simplicité de programmation CRISPR/Cas9 et sa capacité de reconnaissance de cible multiplexé ont alimenté plus sa popularité comme une technologie rentable et facile à utiliser. Parmi les diverses méthodes disponibles aux chercheurs de livrer ces composantes de l’édition de gène aux cellules, vecteurs viraux demeurent de loin le plus populaire et efficace système.
Vecteurs LENTIVIRAUX (LVs) sont apparus comme le véhicule de choix pour fournir les composants de CRISPR/Cas9 système in vivo pour diverses applications4,5,6,7. Les principales fonctionnalités font LVs un choix populaire pour ce processus, y compris leur capacité à infecter des cellules en division et non de division, faible immunogénicité tant minime toxicité cellulaire (revu en référence8). Ainsi, la thérapie génique induite par le LV a été employée dans le traitement de maladies infectieuses comme le VIH-1 et HSV-1, HBV, ainsi que dans la correction des défauts sous-jacents des maladies héréditaires humaines, telles que la fibrose kystique et la dégénérescence maculaire neo-vasculaire 4 , 5 , 7 , 9 , 10 , 11. par ailleurs, LVs ont été effectivement modifiées pour effectuer des retouches de gène multiplex locus génomiques distincts à l’aide d’un vecteur unique système12.
Cependant, la propriété inhérente de LVs à intégrer dans le génome hôte peut être mutagène et handicape souvent leur utilité comme vecteurs de transgène, notamment dans les milieux cliniques. En outre, depuis intégrée de façon stable LVs expriment leur transgènes durablement élevés, ce système est inadapté pour la livraison de gène-montage de composants tels que les CRISPR/Cas9 ; la surexpression de l’ARN Cas9-guide (gRNA) et des protéines semblables tels que ZFNs, sont associées à des niveaux élevés d’effets hors cible, incluent des mutations indésirables13,14,15,16 , 17 et peut potentiellement améliorer la cytotoxicité18. Par conséquent, pour atteindre précis édition de gène avec des effets minimes hors cible, il est impératif de concevoir des systèmes qui permettent l’expression transitoire du gène montage des composants.
Ces dernières années, une variété de plates-formes de diffusion ont été développés pour exprimer transitoirement CRISPR/Cas9 dans cellules16,19,20,21 (revu en référence22). Il s’agit de méthodes qui dépendent directement introduire Cas9 purifiée ainsi que le guide approprié ARN dans des cellules, qui s’est avéré plus efficace à l’édition de gène ciblé par rapport à médiation plasmidique transfection16. Des études ont démontré que la ribonucléoprotéine (RNP) complexes comprenant guident RNA/Cas9 particules sont rapidement remis après médiation de clivage de l’ADN à leurs cibles, ce qui suggère que l’expression à court terme de ces composants est suffisante pour atteindre gène robuste édition16. En théorie, sans intégration des plates-formes de vecteurs viraux tels que les vecteurs viraux adéno-associés (AAVs) peuvent fournir une alternative viable pour livrer les machines édition de gène aux cellules. Malheureusement, les capsides AAV possèdent significativement plus faible capacité de l’emballage que LVs (< 5 Ko), qui entrave gravement leur aptitude pour empaqueter le toolkit CRISPR multicomposants au sein d’un unique vecteur (revu en référence8). Il est à noter qu’ajout de composés qui inhibent les histones désacétylases (p. ex., le butyrate de sodium23) ou entraver le cycle cellulaire (p. ex., caféine24) ont été montré pour augmenter des titres des gènes. Malgré les progrès récents, les systèmes d’expression transitoire développés jusque sont encore entravés par plusieurs défauts, tels que l’efficacité de la production plus faible, conduisant à des titres viraux réduits et efficacité faible transduction des vicitimes du virus ces approches25.
Vecteurs LENTIVIRAUX intégrase déficient (IDLVs) représentent une avancée majeure dans le développement de véhicules de livraison-gène, car ils combinent la capacité de l’emballage de LVs avec l’avantage supplémentaire d’entretien épisomiques AAV-comme dans les cellules. Ces fonctionnalités permettent IDLVs de contourner en grande partie les principaux enjeux rattachés à l’intégration des vecteurs, surexpression continue vis-àvis des éléments potentiellement génotoxiques et mutagénicité induite par l’intégration. Il a été précédemment démontré que IDLVs peuvent être modifiées avec succès pour améliorer l’expression de gène épisomiques26,27. En ce qui concerne la livraison de médiation jusque CRISPR/Cas9, titres de production faible et plus faible expression des génomes épisomes-la charge par rapport aux systèmes des gènes intégrase-proficient limite leur utilité comme outils de bona fide pour la livraison du génome-montage constructions transgéniques. Nous avons récemment démontré que transgene expression tant des titres viraux associés à production jusque sont considérablement renforcés par l’inclusion de sites pour le facteur de transcription Sp1 dans l’expression virale cassette28de liaison. Les IDLVs mis à jour le fermement appuyé gènes CRISPR édition aussi bien in vitro (dans les cellules HEK-293 t) et in vivo (dans les neurones du cerveau post-mitotiques), tout en induisant des mutations hors cible minimales par rapport à la médiation ICLV correspondant 28de systèmes. Dans l’ensemble, nous avons développé un roman, compact, tout-en-un toolkit CRISPR exploite une plateforme jusqu’et décrit les divers avantages d’utiliser un tel véhicule de livraison pour l’édition de gène amélioré.
Ici, le protocole de production du système jusque-CRISPR/Cas9 est décrit, y compris les différentes étapes impliquées dans l’Assemblée, purification, concentration et titrage de IDLVs, ainsi que des stratégies pour valider l’efficacité de gène-édition de ces vecteurs. Ce protocole est facilement évolutif pour répondre aux besoins des différents enquêteurs et est conçu pour générer avec succès les vecteurs LV avec des titres dans l’ordre de 1 x 1010 unités (TU) de transduction / mL. Les vecteurs des vicitimes du présent protocole peuvent être utilisés pour infecter efficacement plusieurs types de cellules différentes, y compris de difficile-à-transduce les cellules souches embryonnaires, les cellules hématopoïétiques (les cellules T et les macrophages) et mis en culture et en vivo- neurones injectés. En outre, le protocole est également bien adapté pour la production de vecteurs LENTIVIRAUX intégrase-compétente en quantités similaires.
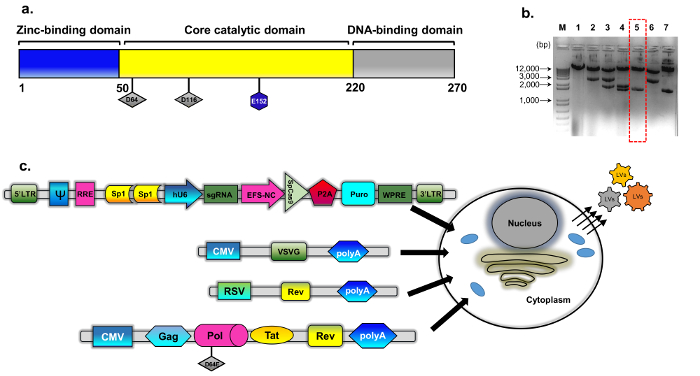
Figure 1 : emballage jusque. (a) schéma du type sauvage intégrase protéine (b) le plasmide modifié a été dérivé de psPAX2 (voir méthodologie, construction plasmidique pour plus de détails). Image de gel d’agarose représentant des clones sélectionnés pour les clones de l’intégrase muté. Des échantillons d’ADN préparés à l’aide d’un mini-kit isolement ADN plasmidique standard ont été analysés par digestion avec EcoRV et SphI. Le clone correctement digérés (numéro 5, boîte rouge en pointillés) a été davantage vérifié par séquençage direct (Sanger) pour la substitution de D64E en INT. La cassette de l’intégrase déficients en emballage s’appelait pBK43. (c) schéma du protocole de transfection transitoire utilisées pour générer des vecteurs jusque-CRISPR/Cas9, montrant des cellules 293 t transfectées avec VSV-G, emballage et cassettes transgène (Sp1-CRISPR/Cas9 tout-en-un plasmide). Les particules virales qui bourgeonnent en dehors de la membrane cellulaire contiennent l’ARN pleine longueur du vecteur (exprimé de la cassette du transgène). La deuxième génération du système d’emballage jusqu’a été utilisée, qui comprend les protéines régulatrices Tat et expression Rev Rev. est complétée d’une cassette séparée (RSV-REV-plasmide). ABBREV: virus de répéter, VSV-G, la stomatite vésiculeuse LTR-Long-terminal G-protéine, promoteur du CMVp-cytomégalovirus ; Promoteur de sarcome de Rous de virus (VRS) ; RRE-(élément de réponse de Rev). Autres éléments régulateurs sur la cassette d’expression comprennent des sites de liaison du Sp1, élément Rev Response (RRE), Woodchuck Hepatitis Virus post-transcriptionnel réglementation élément (WPRE), un promoteur de 1α facteur core-élongation (EFS-NC), l’emballage de vecteur élément ψ (lb/po2), promoteur de cytomégalovirus (hCMV) humain et humain promoteur U6. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Protocole
1. mise en culture des cellules HEK-293 t et l’ensemencement pour la Transfection de cellules
NOTE : Rein embryonnaires humaines 293 t (HEK-293 t) cellules croissent en DMEM, médias de forte concentration de glucose additionné de sérum de veau 10 % additionné de promoteurs de croissance et de fer et 1 x solution antibiotique-antimycosiques (100 x solution contient 10 000 pénicilline unités streptomycine 10 mg et 25 µg amphotéricine B par mL). Les médias sont également additionné de pyruvate de sodium 1 x, 1 x acide aminé non essentiel mix et 2 mM de L-Glutamine (dipeptide stock 200 mM L-alanyl-L-glutamine à 0,85 % NaCl). Les cellules sont cultivées en plaques de culture de tissu de 100 mm (superficie approximative de croissance est de 55 cm²). Un ratio de culture secondaire de 01:10 est utilisé avec la culture sous tous les 2-3 jours. La trypsine-EDTA 0,05 % est utilisé pour la dissociation des cellules entre les passages. Pour maintenir la cohérence entre les expériences, nous recommandons test sérum de veau lors du passage d’un lot/lot différent et surveiller tout changement dans la croissance cellulaire, l’efficacité de transfection et vector production.
- Démarrez une nouvelle culture en utilisant des cellules de passage bas (il est recommandé de ne pas utiliser de cellules après passage 15 ou si la croissance ralentit) par semis dans une plaque de culture de tissu de 10 cm. Utilisez DMEM additionné de 10 % de sérum pour la croissance cellulaire. La croissance de cellules à 37 ° C, avec 5 % de CO2 dans un incubateur de culture de tissus standard. Utilisez un hémocytomètre standard pour compter les cellules pour toutes les étapes subséquentes.
- Une fois les cellules atteignent 90-95 % croissance confluente, réensemencer en 15 cm plaques de culture de tissus (ci-dessous, étapes 1,3 - 1,5).
- Pour réamorcer, aspirer les médias de la plaque confluente et rincer doucement avec du PBS stérile 1 x. Incuber les cellules avec 2 mL de réactif dissociation (par exemple, la trypsine-EDTA) à 37 ° C pendant 3-5 min. ajoutez 8 mL de milieux contenant du sérum pour inactiver le réactif de dissociation et triturer les 10 - 15 fois avec une pipette sérologique pour créer une cellule unique de 10 mL suspension. Remettre en suspension des cellules dans les milieux de culture pour obtenir une densité cellulaire d’environ 4 x 106 cellules/mL.
- Pour améliorer l’adhérence au substrat, préalablement recouvrir les plaques de 15 cm avec 0,2 % de gélatine, ajoutant 8 mL de gélatine par plaque. Répartis uniformément sur la surface de la plaque et incuber à température ambiante pendant 10 min siphonner le liquide.
- Porter le volume total de chaque plaque à 25 mL avec tiède (37 ° C) médias HEK-293 T (voir la Note sous étape 1) et les plaques des graines en ajoutant 2,5 mL de cellules (total ~ 1 x 107 cellules/plaque). Incuber les plaques à 37 ° C, avec 5 % de CO2 pendant la nuit ou jusqu'à ce que la confluence de 70 à 80 % est atteint.
NOTE : Jusqu'à six planches de 15 cm peut être utilisé pour la production. Régler le volume des médias par plaque à ~ 20-22 mL lors de l’utilisation de plus de quatre assiettes (se reporter à l’étape 5 du protocole pour raison d’être).
2. transfectant HEK-293 t des cellules à l’aide d’un protocole à base de Phosphate de Calcium
-
Réactifs de transfection
- Pour préparer 2 x solution tamponnée BES BBS (50 mM BES, 280 mM NaCl, 1,5 mM Na2HPO4), combiner 16,36 g de NaCl, 10,65 g de BES (N, N-bis (2-hydroxyéthyl) -2-amino-ethanesulfonic acide) et 0,21 g de Na2HPO4. Ajouter bidistillée H2O (dd-H2O) jusqu'à 900 mL. Dissoudre, titrer à pH 6,95 avec 1 NaOH M et porter le volume à 1 L. filtre par filtre de 0,22 µM. Conserver à-20 ° C.
- Préparer 1M CaCl2. Filtrer la solution via un filtre de 0,22 µM. Conserver à 4 ° C.
- Observer les plaques qui ont été ensemencés un jour avant. Les cellules sont prêtes pour la transfection dès qu’ils atteignent la confluence de 70 à 80 %.
- Aspirer les anciens médias des plaques et ajouter doucement les médias fraîchement préparés sans sérum.
Remarque : Consultez le Fichier complémentaire 1-plasmides pour plus de détails sur les plasmides choisis et leur préparation. - Préparer le mélange de plasmide de plasmides toutes les quatre dans un tube conique de 15 mL. Pour une préparation de plat unique de 15 cm, utilisez 37,5 µg le vecteur CRISPR/Cas9-transfert (pBK198 ou pBK189), du 25 µg de pBK43 (psPAX2-D64E), 12,5 µg pMD2.G et 6,25 µg de pREV (Figure 1C).
- Ajouter 312,5 µL 1M CaCl2 pour le mix de plasmide. Pour cela, ajouter jusqu'à 1,25 mL stérile JJ-H20.
- Lentement (goutte-à-goutte) ajouter 1,25 mL de solution de x BBS 2 alors que l’agitation du mélange. Incuber 30 min à température ambiante.
- Ajouter le mélange de transfection goutte à chaque plaque de 15 cm (2,5 mL par plaque). Doucement tournoyer les plaques et incuber à 37 ° C, avec 5 % de CO2 pendant 2-3 h. Par la suite, ajouter 2,5 mL (10 %) de sérum par plaque et continuer en incubant pendant la nuit (12-18 h).
Remarque : Certains laboratoires utilisent 3 % CO2 incubateurs pour stabiliser le pH de médias. Cependant, nous n’observons pas une différence dans l’efficacité de transfection entre 3 % et 5 % de CO2. En outre, la taille du CaPO4 précipite est cruciale pour l’efficacité de la transfection ; le mélange de transfection doit être clair avant son ajout dans les cellules. Si le mélange devient trouble durant l’incubation, préparer les frais 2 x BBS (pH = 6,95).
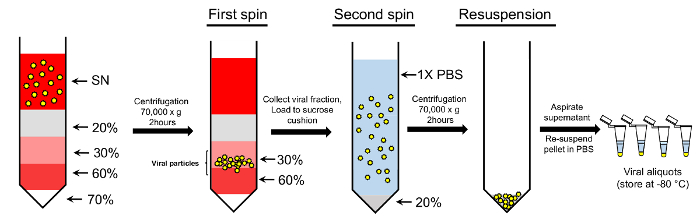
Figure 2 : Concentration des particules virales à l’aide de double-saccharose protocole dégradé. Les particules virales prélevés dans le surnageant (SN) sont chargés sur gradient gradient de saccharose. solutions de saccharose de 70 %, 60 %, 30 % et 20 % sont utilisées pour créer le dégradé. Après centrifugation, les particules prélevés dans les fractions de saccharose de 30 à 60 % sont plus chargés sur un coussin de saccharose de 20 % et précipités. Le granule final contenant des particules virales purifiées est resuspendu dans du PBS 1 x pour l’utilisation de l’autre (voir le texte pour plus de détails). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
3. jour après la Transfection
- Observer les cellules pour s’assurer qu’ils sont presque 100 % confluence à ce stade avec peu ou pas de mort cellulaire. Remplacer le support en ajoutant 25 mL de DMEM fraîche + 10 % de sérum de chaque plaque. Continuer en incubation à 37 ° C, avec 5 % de CO2 pendant 48 h supplémentaires.
Remarque : Régler le volume des médias à l’étape 3.1 si vous utilisez plus de six planches de 15 cm (voir point 5.2, Note).
4. Virus de récolte
- Recueillir soigneusement le surnageant de toutes les plaques de culture de tissu contenant les cellules transfectées à l’aide d’une pipette de vitroplants stérile de 10 mL et de la piscine dans des tubes à centrifuger 50 mL.
- Désactivez la suspension par centrifugation à 400-450 x g pendant 10 min à l’aide d’une centrifugeuse de table. Filtrer le surnageant à travers une unité de filtration sous vide de 0,45 µm.
Remarque : Le surnageant filtré peut être stockées à 4 ° C jusqu'à 4 jours avant de procéder à la concentration, ou aliquotés et stockées à-80 ° C. Les titres attendus de jusque-Sp1-CRISPR/Cas9 (non concentré) les préparations virales doivent être ~ 2 x 107 TU/mL (se reporter à l’étape 6 pour la détermination du titre). Cependant, éviter de soumettre les préparations virales à plusieurs cycles de gel-dégel, comme chaque cycle de gel et de dégel entraîne une perte de 10 à 20 % dans des titres fonctionnels.
5. la concentration de particules virales par Ultracentrifugation
Remarque : Nous utilisons une méthode de double-saccharose de purification qui comporte deux étapes : une étape de dégradé de saccharose et un pas de coussin de saccharose (Figure 2).
- Charger les tubes coniques ultracentrifugation dans l’ordre suivant pour créer un gradient de saccharose : 0,5 mL 70 % de sucrose (dissous en solution 1 PBS x), 0,5 mL 60 % de saccharose (dissous en DMEM), 1 mL 30 % de saccharose (dissous en DMEM) et 2 mL 20 % de sucrose (dissous dans du PBS 1 x).
-
Ajouter délicatement le surnageant contenant le virus au gradient. Comme le volume total de liquide surnageant de quatre plaques de 15 cm est de 100 mL, utiliser six tubes ultracentrifugation par rotation pour traiter le volume complet du virus surnageant.
- Chaque tube d’ultracentrifugation utilisé pour cette étape a une capacité de volume allant jusqu'à 30 ml (y compris le volume de saccharose), distribuer le surnageant viral également entre les tubes, en laissant au moins 10 % headspace pour éviter toute fuite.
- Réglez le volume de la culture à ~ 20-22 mL par plaque lors de l’utilisation de plus de quatre plaques de 15 cm pour chaque expérience, afin que le volume final du surnageant viral mis en commun peut facilement être logé au sein de six tubes d’ultracentrifugation.
- Ultracentrifugation remplissage tubes au moins trois quarts leur capacité de volume total, sans quoi la rupture des tubes peut survenir lors de la centrifugation, ce qui entraîne une perte possible de dommages d’échantillon ou de l’équipement.
- Équilibrer les tubes avec 1 x PBS et centrifuger les échantillons à 70 000 x g pendant 2 h à 17 ° C (voir Table des matières pour plus de détails du rotor).
Remarque : Pour éviter des perturbations de la couche de saccharose pendant l’accélération, définir l’ultracentrifugeuse pour accélérer lentement le rotor à 200 tr/min durant les 3 premières minutes de l’essaimage. De même, définir l’ultra-centrifugeuse à décélérer lentement le rotor de 200 tr/mn à 0 tr/min pendant 3 min à la fin de l’essorage. - Recueillir soigneusement fractions de saccharose de 30 à 60 % dans des tubes propres (Figure 2). Ajouter froid 1 x PBS aux fractions mis en commun et faire apparaître le volume à 100 mL ; la composition de pipetage et descendre plusieurs fois.
- Passez à l’étape de coussin de saccharose en superposant soigneusement la préparation virale sur un coussin de saccharose. Pour ce faire, ajouter 4 mL de 20 % de sucrose (dans du PBS 1 x) dans le tube, suivi par environ 20-25 mL de la solution virale par tube. Si les tubes sont moins de trois quarts plein, compléter avec du PBS stérile 1 x.
- Soigneusement équilibrer et centrifuger les échantillons à 70 000 x g pendant 2 h à 17 ° C, comme avant. Décanter le liquide surnageant et laisser le reste liquide s’écouler en inversant les tubes sur du papier absorbant.
- Aspirer les gouttes restantes afin d’évacuer tout le liquide de la pastille. À cette étape, les granules contenant le virus devraient être à peine visibles sous forme de petites taches translucides.
- Remettre en suspension les boulettes en ajoutant 70 µL de PBS 1 x dans le premier tube et soigneusement pipetage la suspension, par la suite transférer la suspension pour le tube suivant et mélange comme avant, jusqu'à ce que toutes les granules sont remises en suspension.
- Rincer les tubes avec un supplémentaire 50 µL de froid 1 x PBS et mélange comme avant. Veiller à ce que le volume combiné de la suspension définitive est ~ 120 µL et apparaît légèrement laiteux ; l’effacer par centrifugation à 10 000 x g pendant 30 s sur une micro-centrifugeuse sur table.
- Transférer le surnageant dans un tube à centrifuger fraîches, faire 10 µL d’extraits et les stocker à-80 ° C.
Remarque : Évitez d’effectuer des cycles de gel-dégel répétés sur les échantillons des gènes. Sauf si la centrifugation est nécessaire, des efforts doivent être faits pour effectuer les étapes restantes de vitroplants hottes ou désignés chambres de culture de tissus à l’aide de mesures de biosécurité appropriées (voir Discussion).
6. estimation des titres viraux
-
p24 -enzyme-liée méthode d’immunosorbent assay (ELISA)
Remarque : Le dosage est effectué à l’aide de plaques de 96 puits de haute-liaison que selon les instructions du programme NIH sida vaccin pour le VIH-1 p24 analyse de saisie d’antigène nécessaire (voir Table des matières) avec modifications29.- Le lendemain, laver les puits trois fois avec 200 µL 0,05 % Tween 20 dans du PBS froid (solution de PBS-T). Recouvrir la plaque avec 100 µL de l’anticorps monoclonal anti-p24 à une dilution de 1:1500 dans du PBS 1 x et incuber une nuit à 4 ° C.
- Afin d’éliminer les liaisons non spécifiques, bloquer la plaque avec 200 µL 1 % BSA dans du PBS ; laver trois fois avec 200 µL 0,05 % Tween 20 dans du PBS froid (solution de PBS-T) pendant au moins 1 h à température ambiante.
-
Préparer les échantillons : Pour les préparations concentrées vector, diluer 1 µL de l’échantillon 100 fois en ajoutant 89 µL de JJ-H20 et 10 µL de Triton X-100 (concentration finale de 10 %). Pour les préparations non concentré, préparer des échantillons dilués dix fois (ajouter 80 µL de JJ-H20 et 10 µL de Triton X-100 (concentration finale de 10 %) à 10 µL de l’échantillon).
Remarque : Les échantillons peuvent être conservés à-20 ° C à cette étape pour une longue période de temps pour un usage ultérieur. - Élaborer des normes de VIH-1 par l’application d’une facteur 2 dilutions (avec un départ ng/mL de concentration 5).
- Diluer les échantillons concentrés (à partir de stocks préalablement dilué au 1/100) en RPMI 1640 additionné de 0,2 % Tween 20 et 1 % de BSA d’établir à 01:10, 000, 01:50, 000 et des dilutions de 1/250 000. Diluer les échantillons non concentré (de 01:10, préalablement dilué les stocks) en RPMI 1640 additionné de 0,2 % Tween 20 et 1 % de BSA pour établir la dilution 1/500, 1:2500 et 1:12,500.
- Demander des échantillons sur la plaque en géométrie et incuber une nuit à 4 ° C.
- Le lendemain, laver les puits six fois et incuber à 37 ° C pendant 4 h avec 100 µL anticorps polyclonal de lapin anti-p24 anticorps, dilué 1/1000 dans RPMI 1640, 10 % FBS, 0,25 % de BSA et sérum de souris normales 2 % (NMS).
- Laver 6 fois comme indiqué ci-dessus et incuber à 37 ° C pendant 1 h avec la peroxydase de raifort anti-lapin chèvre IgG dilué 1/10 000 en milieu RPMI 1640 additionné de sérum de chèvre normal de 5 %, 2 % NMS, BSA de 0,25 % et 0,01 % Tween 20.
- Laver la plaque, comme indiqué ci-dessus et incuber avec substrat peroxydase TMB à température ambiante pendant 15 min.
- Arrêter la réaction en ajoutant 100 µL de 1 N HCL. Mesurer l’absorbance d’échantillon à 450 nm en utilisant l’absorbance sur plaque de lecteur.
-
Mesure de l’intensité de fluorescence journaliste
- Méthode de FACS
NOTE : L’étendue de l’appauvrissement de la couche de signal GFP dans des cellules peut être estimé avec précision en mesurant l’intensité de la fluorescence moyenne des cellules transduits par cytométrie en flux. Veuillez consulter le document récent 28 pour l’analyse de données de FACS, présentation et interprétation. Le protocole est décrit comme suit.- Faire une dilution en série dix fois de la préparation (à partir de 10-1 à 10-5) de la préparation virale dans du PBS 1 x.
- Graines d’environ 5 x 105 293 t cellules dans chaque puits d’une plaque de 6 puits dans un volume final de 2 mL par puits. Ajouter 10 µL de chaque dilution virale aux cellules et incuber les cellules à 37 ° C pendant 48 h.
- Récolte des cellules pour l’analyse des FACS comme suit : ajouter 200 µL de solution de trypsine-EDTA 0,05 % et incuber les cellules à 37 ° C pendant 5 min. Ajouter 2 mL d’un média DMEM complet et prélever des échantillons dans des tubes coniques 15 mL.
- Cellules de granule par centrifugation à 400 x g à 4 ° C et Resuspendre le culot dans 500 µL de PBS 1 x contre le rhume.
- Pour la fixation, ajouter un volume égal de solution de formaldéhyde de 4 % à cette suspension et incuber pendant 10 min à température ambiante.
- Les cellules fixes de granule et remettre en suspension dans 1 mL de PBS 1 x. Analyser l’expression de la GFP à l’aide d’un instrument de FACS, comme décrit dans Ortinski et al. 28 a brièvement, utilisez la formule suivante pour déterminer le titre fonctionnel des virus :
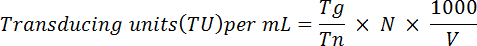
NOTE : Ici Tg = nombre de cellules positives à GFP comptés ; TN = nombre total de cellules comptées ; N = nombre total de cellules transduites ; V = volume (µL) utilisé pour la transduction. Par exemple : si 1 x 106 cellules étaient transduites avec 10 µL de virus, 2 x 104 cellules ont été comptés et 5 x 103 GFP-positive, basée sur l’équation qui précède le titre fonctionnel serait :
- Comptage des cellules positives GFP
- Calculer la multiplicité d’infection (MOI) utilisée pour la transduction. Tester une large gamme de MOIs (1-10), avec une augmentation de MOIs, ce qui entraîne une plus grande efficacité de transduction.
- Avant la transfection, amorcer une plaque 6 puits avec environ 3-4 x 105 cellules par puits. Une fois les cellules atteignent > confluence de 90 % (généralement moins de 24 h), transduce avec le virus purifié au MOIs pré-déterminé.
- Incuber 1 à 7 jours pour les modifications du signal GFP plaque à 37 ° C, avec 5 % de CO2 dans une culture de tissu standard et moniteur de cellules à intervalles réguliers.
- Compter le nombre de cellules GFP-positives avec un microscope à fluorescence (objectif PLAN 4 X, N.A 0.1, un grossissement de 40 X) équipé d’un jeu de filtre GFP (excitation d’onde-470 nm, émission nm de longueur d’onde-525), utilisant des cellules (non transduits) naïf pour définir la population des cellules de GFP-négatifs et positifs.
- Estimer le titre final en ajustant pour le facteur de dilution et le volume, à l’aide de la formule suivante :
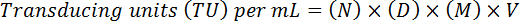
NOTE : Ici, N = nombre de cellules de GFP-positives, D = facteur de dilution, M = facteur de grossissement (habituellement 20 X), V = volume du virus utilisé pour la transduction. Par exemple, pour 20 cellules de GFP-positif (N) dénombrées à une dilution de 10-4 (01:10, 000) dans un échantillon de 10 µL (V) à 20 X grossissement (M) (D) se traduirait par un titre fonctionnel (20 x 10-4) x (20) x (10) x (100 *) = 4 x 108 TU/mL.
(* pour s’adapter à / mL)
- Méthode de FACS
Résultats
Validation de l’élimination directe-efficacité des vecteurs jusque-CRISPR/Cas9
Nous avons utilisé les cellules 293 t exprimant GFP comme modèle pour valider l’efficacité de la débouchure de gène CRISPR/Cas9-médiée. GFP + cellules ont été générés par transduction des cellules HEK-293 t avec pLenti-GFP (vBK201a) à un MOI de 0,5 (Figure 3 b, panneau « no-virus »). La cassette de sgRNA-à-GFP/Cas9 tout-en-un vecteur a é...
Discussion
IDLVs ont commencé à émerger en tant que véhicule de choix pour l’in vivo gene-édition, en particulier dans le contexte des maladies génétiques, due en grande partie au faible risque de mutagenèse associée à ces vecteurs par rapport à l’intégration de plateformes de livraison22 , 28. dans le manuscrit actuel, nous avons cherché à détailler le protocole lié à la production de l’all-in-one jusque-CRISPR/Cas9 système amélioré qui a...
Déclarations de divulgation
USC-499-P de brevet (1175) a été déposée par la University of South Carolina, en ce qui concerne les travaux décrits dans ce manuscrit.
Remerciements
Nous tenons à remercier le département de neurobiologie, Duke University School of Medicine et Décanat en sciences fondamentales, Université Duke. Nous remercions également les membres du noyau Duke vecteur Viral pour commentaires sur le manuscrit. Plasmide pLenti CRISPRv2 était le cadeau de Feng Zhang (Broad Institute). Le système de LV-emballage, y compris le psPAX2 de plasmides, VSV-G, pMD2.G et pRSV-Rev était un gentil cadeau de Didier Trono (EPFL, Suisse). Soutien financier à ce travail a été fourni par l’Université d’école de médecine en Caroline du Sud, accorder RDF18080-E202 (BA).
matériels
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| xMark Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| BD FACS | Becton Dickinson | 338960 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| 0.45-μm filter unit, 500mL | Corning | 430773 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| SW32Ti swinging-bucket rotor | Beckman Coulter | 369650 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 50mL conical centrifuge tubes | Corning | 430291 | |
| High-binding 96-well plates | Corning | 3366 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 100mm TC-Treated Culture Dish | Corning | 430167 | |
| 0.22 μM filter unit, 1L | Corning | 430513 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Tissue culture pipettes, 5 mL | Corning | 4487 | |
| Tissue culture pipettes, 10 mL | Corning | 4488 | |
| Tissue culture pipettes, 25 mL | Corning | 4489 | |
| Hemacytometer with cover slips | Cole-Parmer | UX-79001-00 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| 293FT cells | Thermo Fisher Scientific | R70007 | |
| DMEM, high glucose media | Gibco | 11965 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| Antibiotic-antimycotic solution, 100X | Sigma Aldrich | A5955-100ML | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| BES (N, N-bis (2-hydroxyethyl)-2-amino-ethanesulfonic acid) | Sigma Aldrich | B9879 - BES | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Normal mouse serum, Sterile, 500mL | Equitech-Bio | SM30-0500 | |
| Goat serum, Sterile, 10mL | Sigma | G9023 | Working concentration 1:1000 |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| psPAX2 | Addgene | 12260 | |
| pMD2.G | Addgene | 12259 | |
| pRSV-Rev | Addgene | 12253 | |
| lentiCRISPR v2 | Addgene | 52961 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsrGI | New England Biolabs | R0575S | |
| BsmBI | New England Biolabs | R0580S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
Références
- Horvath, P., Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 327 (5962), 167-170 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat Rev Genet. 11 (3), 181-190 (2010).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Bellec, J., et al. CFTR inactivation by lentiviral vector-mediated RNA interference and CRISPR-Cas9 genome editing in human airway epithelial cells. Curr Gene Ther. 15 (5), 447-459 (2015).
- Kennedy, E. M., et al. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease. Virology. 476, 196-205 (2015).
- Roehm, P. C., et al. Inhibition of HSV-1 Replication by Gene Editing Strategy. Sci Rep. 6, 23146 (2016).
- Wang, W., et al. CCR5 gene disruption via lentiviral vectors expressing Cas9 and single guided RNA renders cells resistant to HIV-1 infection. PLoS One. 9 (12), e115987 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Adv Genet. 87, 125-197 (2014).
- Lebbink, R. J., et al. A combinational CRISPR/Cas9 gene-editing approach can halt HIV replication and prevent viral escape. Sci Rep. 7, 41968 (2017).
- Xu, L., et al. CRISPR/Cas9-Mediated CCR5 Ablation in Human Hematopoietic Stem/Progenitor Cells Confers HIV-1 Resistance In Vivo. Mol Ther. , (2017).
- Yiu, G., Tieu, E., Nguyen, A. T., Wong, B., Smit-McBride, Z. Genomic Disruption of VEGF-A Expression in Human Retinal Pigment Epithelial Cells Using CRISPR-Cas9 Endonuclease. Invest Ophthalmol Vis Sci. 57 (13), 5490-5497 (2016).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), e147 (2014).
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol. 32 (3), 279-284 (2014).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Schaefer, K. A., et al. Unexpected mutations after CRISPR-Cas9 editing in vivo. Nat Methods. 14 (6), 547-548 (2017).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Ther. 23 (7), 627-633 (2016).
- Platt, R. J., et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159 (2), 440-455 (2014).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Res. 43 (13), 6450-6458 (2015).
- Nelson, C. E., Gersbach, C. A. Engineering Delivery Vehicles for Genome Editing. Annu Rev Chem Biomol Eng. 7, 637-662 (2016).
- Jaalouk, D. E., Crosato, M., Brodt, P., Galipeau, J. Inhibition of histone deacetylation in 293GPG packaging cell line improves the production of self-inactivating MLV-derived retroviral vectors. Virol J. 3, 27 (2006).
- Ellis, B. L., Potts, P. R., Porteus, M. H. Creating higher titer lentivirus with caffeine. Hum Gene Ther. 22 (1), 93-100 (2011).
- Hoban, M. D., et al. Delivery of Genome Editing Reagents to Hematopoietic Stem/Progenitor Cells. Curr Protoc Stem Cell Biol. 36, 1-10 (2016).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Mol Ther. 16 (12), 1968-1976 (2008).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proc Natl Acad Sci U S A. 106 (44), 18786-18791 (2009).
- Ortinski, P. I., O'Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Mol Ther Methods Clin Dev. 5, 153-164 (2017).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Mol Ther. 19 (3), 547-556 (2011).
- Berkhout, B., Verhoef, K., van Wamel, J. L., Back, N. K. Genetic instability of live, attenuated human immunodeficiency virus type 1 vaccine strains. J Virol. 73 (2), 1138-1145 (1999).
- Gomez-Gonzalo, M., et al. The hepatitis B virus X protein induces HIV-1 replication and transcription in synergy with T-cell activation signals: functional roles of NF-kappaB/NF-AT and SP1-binding sites in the HIV-1 long terminal repeat promoter. J Biol Chem. 276 (38), 35435-35443 (2001).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. J Biol Chem. 280 (22), 21545-21552 (2005).
- Ortinski, P. I., Lu, C., Takagaki, K., Fu, Z., Vicini, S. Expression of distinct alpha subunits of GABAA receptor regulates inhibitory synaptic strength. J Neurophysiol. 92 (3), 1718-1727 (2004).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. J Virol. 71 (8), 6113-6127 (1997).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. J Virol. 68 (4), 2632-2648 (1994).
- Xu, W., Russ, J. L., Eiden, M. V. Evaluation of residual promoter activity in gamma-retroviral self-inactivating (SIN) vectors. Mol Ther. 20 (1), 84-90 (2012).
- . Testing for Replication Competent Retrovirus (RCR)/Lentivirus (RCL) in Retroviral and Lentiviral Vector Based Gene Therapy Products - Revisiting Current FDA Recommendations Available from: https://sites.duke.edu/dvvc/files/2016/05/FDA-recommendation-for-RCR-testing.pdf (2017)
- . Biosafety in Microbiological and Biomedical Laboratories Available from: https://www.cdc.gov/biosafety/publications/bmbl5/bmbl.pdf (2017)
- Dull, T., et al. A third-generation lentivirus vector with a conditional packaging system. J Virol. 72 (11), 8463-8471 (1998).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon