È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un protocollo per la produzione di vettori lentivirali integrasi-carenti per CRISPR/Cas9-mediata Knockout del Gene nelle cellule in divisione
In questo articolo
Riepilogo
Descriviamo la strategia di produzione di vettori lentivirali integrasi-carente (IDLVs) come veicoli per la consegna di CRISPR/Cas9 alle cellule. Con una capacità di mediare rapido e robusto gene modifica nelle celle, IDLVs presentare una più sicura e piattaforma altrettanto efficace vettore per la consegna del gene rispetto ai vettori integrasi-competente.
Abstract
Vettori lentivirali sono la scelta ideale per fornire componenti di modifica del gene delle cellule dovuto la loro capienza per stabile trasducendo un ampio intervallo di celle e alti livelli di espressione genica di mediazione. Tuttavia, la loro capacità di integrarsi nel genoma della cellula ospite aumenta il rischio di mutagenesi inserzionale e quindi solleva preoccupazioni di sicurezza e limita il loro uso nelle regolazioni cliniche. Più ulteriormente, l'espressione persistente della modifica del gene componenti trasportati da questi aumenti di vettori lentivirali integrazione-competente (ICLVs) la probabilità di promiscua gene-targeting. In alternativa, è stata sviluppata una nuova generazione di vettori lentivirali integrasi-carente (IDLVs) che affronta molte di queste preoccupazioni. Qui il protocollo di produzione di una piattaforma IDLV nuova e migliorata per CRISPR-mediata del gene editing ed elenco i passaggi coinvolti nella purificazione e concentrazione di tali vettori è descritto e loro trasduzione e modifica del gene efficienza utilizzando HEK 293T cellule è stata dimostrata. Questo protocollo è facilmente scalabile e può essere utilizzato per generare alto titolo IDLVs che sono in grado di trasdurre cellule in vitro ed in vivo. Inoltre, questo protocollo può essere facilmente adattato per la produzione di ICLVs.
Introduzione
Gene preciso editing costituisce la pietra miliare dei principali progressi biomedici che coinvolgono lo sviluppo di nuove strategie per affrontare le malattie genetiche. At l'avanguardia delle tecnologie di modifica del gene è il metodo basandosi sull'uso della clustered regularly -ionterspaced short palindromic repeats (CRISPR) / sistema di Cas9 che inizialmente è stato identificato come componente dell'immunità batterica contro l'invasione di materiale genetico virale (rivisto in riferimenti1,2). Un grande vantaggio del sistema CRISPR/Cas9 rispetto ad altri strumenti di modifica del gene, quali nucleasi a dita di zinco (ZFNs) e nucleasi di attivatore-come effettore di trascrizione (TALENs) (rivista in riferimento3), è la relativa semplicità del design di plasmide e costruzione di componenti CRISPR — una caratteristica che ha alimentato l'espansione del gene-editing da pochi laboratori specializzati per una comunità di ricerca molto più ampia. Inoltre, la semplicità di programmazione CRISPR/Cas9 e la sua capacità per riconoscimento multiplex target hanno ulteriormente alimentato la sua popolarità come una tecnologia conveniente e facile da usare. Tra i vari metodi disponibili per i ricercatori di consegnare tali componenti modifica del gene delle cellule, vettori virali restano di gran lunga il sistema più popolare ed efficace.
Vettori lentivirali (LVs) sono emersi come il veicolo della scelta per fornire i componenti di CRISPR/Cas9 sistema in vivo per diverse applicazioni4,5,6,7. Diverse caratteristiche chiave rendono LVs una scelta popolare per questo processo inclusa la capacità di infettare sia separazione e divisione di celle, bassa immunogenicità e minima tossicità cellulare (rivisto in riferimento8). Di conseguenza, la terapia genica mediata da LV è stata impiegata nei trattamenti di malattie infettive come l'HIV-1, HBV e HSV-1, come pure nella correzione dei difetti alla base di malattie ereditarie umane, come la fibrosi cistica e degenerazione maculare neo-vascolare 4 , 5 , 7 , 9 , 10 , 11. Inoltre, LVs sono stati modificati in modo efficace per eseguire multisala gene editing presso distinti loci genomici utilizzando un singolo vettore sistema12.
Tuttavia, la proprietà intrinseca di LVs per integrare nel genoma ospite possa essere mutagenica e spesso ostacola la loro utilità come veicoli di consegna del transgene, specialmente nelle regolazioni cliniche. Inoltre, poiché LVs stabilmente integrato esprimono loro transgeni a livelli elevati in modo sostenibile, questo sistema è inadeguato per la consegna del gene di modifica di componenti quali CRISPR/Cas9; sovraespressione di RNA Cas9-guida (gRNA) e di proteine simili come ZFNs, sono associati con i livelli elevati di effetti fuori bersaglio, che comprendono mutazioni indesiderate13,14,15,16 , 17 e può potenzialmente migliorare la citotossicità18. Pertanto, per raggiungere precisi gene-editing con effetti minimi fuori bersaglio, è imperativo per progettare sistemi che consentono l'espressione transitoria del gene componenti editing.
Negli ultimi anni, una varietà di piattaforme di distribuzione sono stati sviluppati per esprimere transitoriamente CRISPR/Cas9 in cellule16,19,20,21 (recensione in riferimento22). Questi includono metodi che si basano sull'introduzione direttamente Cas9 purificata con il RNAs guida appropriata nelle cellule, che è stato indicato per essere più efficace al gene-editing mirato rispetto al plasmide-mediata transfezione16. Gli studi hanno dimostrato che ribonucleoproteina (RNP) complessi costituiti guidano RNA/Cas9 particelle sono girate rapidamente dopo che mediano la fenditura del DNA alle loro destinazioni, suggerendo che a breve termine espressione di questi componenti è sufficiente per raggiungere gene robusto16di editing. In teoria, non integrando piattaforme vettore virale adeno-associato di vettori virali (AAVs) in grado di fornire una valida alternativa per fornire macchinari modifica del gene alle cellule. Purtroppo, capsidi AAV possiedono capacità di imballaggio significativamente più bassa rispetto alla LVs (< 5kb), che ostacola gravemente la loro possibilità di confezionare il toolkit multi-componente CRISPR all'interno di un singolo vettore (rivisto in riferimento8). Vale la pena notare che l'aggiunta di composti che inibiscono istone deacetilasi (ad es., sodio butirrato23) o ostacolare il ciclo cellulare (per esempio, caffeina24) hanno dimostrato di aumentare i titoli lentivirali. Nonostante i recenti progressi, i sistemi di espressione transiente sviluppati finora sono ancora ostacolati da varie carenze, come l'efficienza di produzione inferiore, che portano a titoli virali ridotti e l'efficienza di trasduzione basso dei virus generato attraverso tali approcci25.
Integrasi-carenti vettori lentivirali (IDLVs) rappresentano un avanzamento importante nello sviluppo di veicoli di consegna del gene, come essi combinano la capacità di confezionamento di LVs con il beneficio aggiunto di manutenzione episomal AAV-come nelle cellule. Queste funzionalità consentono di IDLVs in gran parte eludere i problemi principali connessi con l'integrazione di vettori, vis-à-vis continua sovraespressione di potenzialmente genotossici elementi e integrazione-mediata mutagenicità. È stato precedentemente dimostrato che IDLVs possa essere modificata correttamente per migliorare il gene episomal espressione26,27. Per quanto riguarda la consegna di IDLV-mediata CRISPR/Cas9, i titoli di bassa produzione e bassa espressione di genomi episome-sopportate relativi agli impianti lentivirali integrasi-competente limita la loro utilità come strumenti di bona fide per la consegna di modifica del genoma costrutti transgenici. Recentemente abbiamo dimostrato che sia l'espressione del transgene e titoli virali associati IDLV produzione aumentano significativamente con l'inclusione di siti per il fattore di trascrizione Sp1 all'interno l'espressione virale cassetta28di legame. Il IDLVs modificate robustamente supportate CRISPR-mediata del gene editing sia in vitro (in cellule HEK 293T) e in vivo (nei neuroni post-mitotici cervello), mentre induce mutazioni di fuori bersaglio minime rispetto ai corrispondenti ICLV-mediata sistemi28. Nel complesso, abbiamo sviluppato un romanzo, compatto, all-in-one toolkit CRISPR trasportato su una piattaforma IDLV e delineati i vari vantaggi dell'utilizzo di un veicolo di consegna per l'editing avanzato gene.
Qui, il protocollo di produzione del sistema IDLV-CRISPR/Cas9 è descritto, compreso i vari passaggi nell'Assemblea, la purificazione, la concentrazione e la titolazione di IDLVs, nonché strategie per convalidare l'efficacia della modifica del gene di questi vettori. Questo protocollo è facilmente scalabile per soddisfare le esigenze di diversi ricercatori ed è progettato per generare dei vettori-LV con i titoli con successo nella gamma di 1 x 1010 transducing unità (TU) / mL. I vettori generati attraverso questo protocollo possono essere utilizzati per infettare in modo efficiente diversi tipi di cellule diverse, tra cui difficile per trasdurre staminali embrionali, cellule ematopoietiche (cellule T e macrofagi) e coltivate e vivo in- neuroni iniettati. Inoltre, il protocollo è ugualmente adatto per la produzione di vettori lentivirali integrasi-competente in quantità simili.
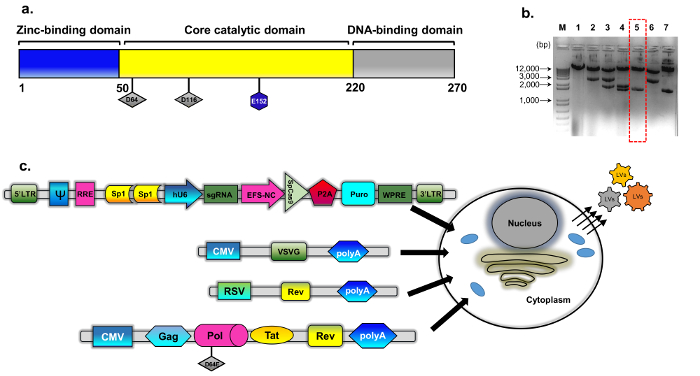
Figura 1: imballaggio di IDLV. (a) schema di tipo selvaggio dell'integrasi proteina (b) il plasmide modificato è stato derivato da psPAX2 (vedere Metodi, costruzione del plasmide per dettagli). Immagine di gel di agarosio rappresentante dei cloni schermati per cloni mutati integrasi. Campioni di DNA preparati utilizzando un mini-kit di isolamento del DNA del plasmide standard sono stati analizzati tramite digestione con EcoRV e SphI. Il clone correttamente digerito (numero 5, casella rossa tratteggiata) è stata ulteriormente verificato mediante sequenziamento diretto (Sanger) per la sostituzione di D64E in INT. La cassetta di imballaggio dell'integrasi-carente è stata denominata pBK43. (c) schema del protocollo trasfezione transiente impiegato per generare dei vettori-IDLV-CRISPR/Cas9, mostrando 293T cellule trasfettate con VSV-G, imballaggi e cassette transgene (Sp1-CRISPR/Cas9 all-in-one plasmide). Le particelle virali che bud fuori dalla membrana cellulare contengono il RNA Full-Length del vettore (espresso dal cassetto transgene). La seconda generazione del IDLV-packaging system è stata utilizzata, che comprende le proteine regolarici Tat ed espressione Rev Rev. è inoltre integrata da una cassetta separata (RSV-REV-plasmide). Abbrev: virus di stomatite vescicolare ripetere, VSV-G, LTR-Long-terminale G-proteina, promotore pCMV-citomegalovirus; Promotore di Rous sarcoma virus (RSV); RRE-(elemento di risposta Rev). Altri elementi regolatori della cassetta di espressione comprendono siti di legame per Sp1, elemento Rev Response (RRE), marmotta epatite Virus Posttranscriptional Regulatory Element (WPRE), promotore di un nucleo-allungamento fattore 1 α (EFS-NC), l'imballaggio di vettore elemento ψ (psi), promotore umano di citomegalovirus (hCMV) e promotore umano U6. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
1. coltura delle cellule HEK 293T e semina di cellule per la transfezione
Nota: Rene embrionale umano 293T cellule (HEK 293T) sono coltivate in DMEM, media alto glucosio completato con siero di vitello 10% completato con promotori di crescita e di ferro e 1 x soluzione antibiotico antimicotico (100 x soluzione contiene penicillina di 10.000 unità la streptomicina 10 mg e 25 µ g amfotericina B / mL). I mezzi di comunicazione è anche completato con 1 x sodio piruvato, mix di aminoacidi non essenziali 1x e 2 mM L-Glutammina (dipeptide magazzino 200 mM L-alanil-L-Glutammina in 0,85% NaCl). Le cellule sono coltivate in piastre di coltura del tessuto di 100 mm (area di superficie di crescita approssimativa è di 55 cm ²). Un rapporto di sub-coltivazione di 01:10 viene utilizzato con sub-coltura ogni 2-3 giorni. Tripsina-EDTA 0,05% è utilizzato per la dissociazione di cellule tra passaggi. Per mantenere la coerenza tra gli esperimenti, si consigliano di test sieri di vitello quando si passa ad un lotto diverso e monitorare le eventuali modifiche nella crescita delle cellule, l'efficienza di trasfezione e produzione di vettore.
- Avviare una nuova cultura utilizzando cellule di passaggio basso (si consiglia di non utilizzare cellule dopo passaggio 15 o se la crescita rallenta) tramite semina in una piastra di coltura del tessuto di 10 cm. Utilizzare DMEM completati con 10% di siero per la crescita cellulare. Crescere le cellule a 37 ° C con 5% CO2 in un incubatore standard di coltura del tessuto. Utilizzare un emocitometro standard per contare le celle per tutti i passaggi successivi.
- Una volta che le cellule raggiungono il 90-95% di crescita confluente, reseeding in 15cm piastre di coltura tissutale (sotto, passi 1.3 - 1.5).
- Per reinizializzare, aspirare media dalla piastra confluente e risciacquare delicatamente con PBS 1X sterile. Incubare le cellule con 2 mL di un reagente di dissociazione (ad es., tripsina-EDTA) a 37 ° C per 3-5 min aggiungere 8 mL di file multimediali contenenti siero per inattivare il reagente di dissociazione e triturare 10 - 15 volte con una pipetta sierologica di 10 mL per creare una singola cella sospensione. Risospendere le cellule in coltura per ottenere una densità di cella di circa 4 x 106 cellule/mL.
- Per migliorare l'aderenza al substrato, pre-cappotto le piastre di 15cm con gelatina di 0,2%, l'aggiunta di 8 mL di gelatina per piastra. Distribuire uniformemente su tutta la superficie della piastra, incubare a temperatura ambiente per 10 min e sifone fuori il liquido.
- Portare il volume totale di ciascuna piastra a 25 mL con tiepida (37 ° C) media T HEK-293 (Vedi nota sotto passaggio 1) e le piastre del seme con l'aggiunta di 2,5 mL di cellule (per un totale di 1 ~ x 107 cellule/piastra). Incubare le piastre a 37 ° C con 5% di CO2 durante la notte o fino a quando non viene raggiunto il confluency di 70-80%.
Nota: Fino a sei zolle di 15 cm può essere utilizzato per la produzione. Regolare il volume dei media per piastra a ~ 20-22 mL quando si utilizza più di quattro piastre (fare riferimento al punto 5 del protocollo per la spiegazione razionale).
2. trasfettando HEK-293T cellule utilizzando un protocollo basato su fosfato di calcio
-
Reagenti di transfezione
- Per preparare la soluzione tamponata BES BBS (50mm BES, 280 mM NaCl, 1,5 mM Na2HPO4): 2x, combinare 16,36 g di NaCl, 10,65 g di BES (N, N-bis (2-idrossietil) -2-ammino-ethanesulfonic acido) e 0,21 g di Na2HPO4. Aggiungere bidistillata H2O (dd-H2O) fino a 900 mL. Sciogliere, titolare a pH 6,95 con 1 M NaOH e portare il volume a 1 L. filtro tramite unità di filtro 0,22 µM. Conservare a-20 ° C.
- Preparare 1M CaCl2. Filtrare la soluzione attraverso un filtro da 0,22 µM. Conservare a 4 ° C.
- Osservare le piastre che sono state seminate un giorno prima. Cellule sono pronte per la transfezione quando raggiungono la confluenza di 70-80%.
- Aspirare i vecchi media dalle piastre e aggiungere delicatamente preparata media senza siero.
Nota: Per informazioni sui plasmidi selezionati e la loro preparazione, vedere File supplementari 1-plasmidi . - Preparare la miscela di plasmide dai plasmidi aliquotare i quattro in una provetta conica da 15 mL. Per la preparazione un piatto unico di 15 cm, utilizzare 37,5 µ g il CRISPR/Cas9-trasferimento vettoriale (pBK198 o pBK189), 25 µ g di pBK43 (psPAX2-D64E), 12,5 µ g pMD2.G e 6,25 µ g di precedente (Figura 1C).
- Aggiungere il mix di plasmide 312.5 µ l 1M CaCl2 . Per questo, è possibile aggiungere fino a 1,25 mL di sterile dd-H20.
- Lentamente aggiungere (drop-wise) 1,25 mL di 2 x BBS soluzione mentre nel Vortex il mix. Incubare per 30 min a temperatura ambiente.
- Aggiungere la miscela di transfezione goccia a goccia per ogni piastra 15 cm (2,5 mL per piastra). Agitare delicatamente le piastre e incubare a 37 ° C con 5% CO2 per 2-3 h. Successivamente, aggiungere 2,5 mL (10%) di siero per piastra e continuare l'incubazione durante la notte (12-18 h).
Nota: Alcuni laboratori utilizzano 3% CO2 incubatrici per stabilizzare il pH di media. Tuttavia, non osserviamo alcuna differenza nell'efficienza di trasfezione tra 3% e 5% CO2. Inoltre, la dimensione del CaPO4 precipita è cruciale per l'efficienza di trasfezione; il mix di transfezione deve essere chiaro prima della sua aggiunta sulle celle. Se il mix diventa torbida durante l'incubazione, preparare fresco 2 x BBS (pH = 6.95).
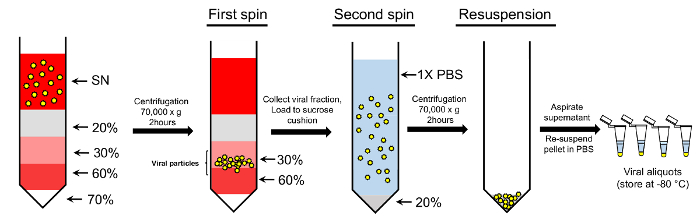
Figura 2: concentrazione delle particelle virali mediante protocollo gradiente double-saccarosio. Le particelle virali raccolte dal surnatante (SN) sono caricate su gradiente gradiente di saccarosio. 70%, 60%, 30% e 20% soluzioni di saccarosio vengono utilizzate per creare la sfumatura. Dopo centrifugazione, le particelle raccolte dalle frazioni di saccarosio di 30-60% sono ulteriormente caricate su un cuscino di saccarosio 20% e precipitate. Il pellet finale contenente particelle virali purificate è risospesi in PBS 1X per ulteriore utilizzo (Vedi testo per ulteriori dettagli). Clicca qui per visualizzare una versione più grande di questa figura.
3. giorno dopo trasfezione
- Osservare le cellule per garantire che sono quasi confluency di 100% a questo punto con poca o nessuna morte delle cellule. Sostituzione supporti aggiungendo 25 mL di DMEM fresco + 10% di siero per ogni piatto. Continuare in incubazione a 37 ° C con 5% CO2 per ulteriori 48 ore.
Nota: Regolare il volume dei media nel passaggio 3.1 se utilizza più di sei piatti di 15cm (Vedi punto 5.2, nota).
4. Virus di raccolta
- Con attenzione e raccogliere il surnatante da tutte le piastre di coltura del tessuto contenente cellule transfected utilizzando una pipetta di coltura del tessuto sterile da 10 mL e una piscina in provette centrifuga da 50 mL.
- Deselezionare la sospensione mediante centrifugazione a 400-450 x g per 10 min utilizzando una centrifuga da tavolo. Filtrare il surnatante attraverso un'unità di aspirazione filtro da 0,45 µm.
Nota: Il surnatante filtrato può essere conservato a 4 ° C fino a 4 giorni prima di procedere con la concentrazione, o aliquotati e conservati a-80 ° C. I titoli previsti dell'IDLV-Sp1-CRISPR/Cas9 (non concentrato) virale preparazioni dovrebbero essere ~ 2 x 107 TU/mL (fare riferimento al passaggio 6 per la determinazione del titolo). Tuttavia, evitare di sottoporre i preparativi virali a più cicli di gelo-disgelo, come ogni ciclo di congelamento e scongelamento provoca una perdita di 10-20% nei titoli funzionali.
5. la concentrazione di particelle virali dall'ultracentrifugazione
Nota: Utilizziamo un metodo di doppio-saccarosio di purificazione che prevede due fasi: una fase di gradiente di saccarosio e un passo di cuscino di saccarosio (Figura 2).
- Caricare i tubi conici ultracentrifugazione nell'ordine seguente per creare un gradiente di saccarosio: 0,5 mL 70% di saccarosio (disciolto 1X PBS), 0,5 mL 60% saccarosio (disciolto in DMEM), saccarosio 30% 1 mL (disciolto in DMEM) e saccarosio al 20% 2 mL (disciolto in PBS 1X).
-
Aggiungere il supernatante contenente virus attentamente alla sfumatura. Come il totale volume del surnatante da quattro piatti di 15 cm è 100 mL, è possibile utilizzare sei ultracentrifugazione tubi per giro per elaborare l'intero volume del surnatante virus.
- Ogni tubo di ultracentrifugazione utilizzata in questa fase ha una capacità di volume di fino a 30 mL (tra cui il volume di saccarosio), distribuire il surnatante virale equamente tra tubi, lasciando almeno il 10% dello spazio di testa per evitare fuoriuscite.
- Regolare il volume di cultura a ~ 20-22 mL per piastra quando si utilizza più di quattro piatti da 15 cm per ogni esperimento, così che il volume finale del pool surnatante virale può facilmente essere ospitato all'interno di sei tubi di ultracentrifugazione.
- Ultracentrifugazione di riempimento tubi per almeno tre quarti di loro capacità di volume totale, altrimenti la rottura dei tubi può verificarsi durante la centrifugazione, con conseguente possibile perdita di danni del campione e/o attrezzature.
- Bilanciare i tubi con 1x PBS e centrifugare i campioni a 70.000 x g per 2 h a 17 ° C (Vedi Tabella materiali particolari del rotore).
Nota: Per evitare la rottura dello strato saccarosio durante l'accelerazione, impostare l'ultracentrifuga per accelerare lentamente il rotore a 200 giri/min durante il primo min 3 dello spin. Allo stesso modo, impostare l'ultracentrifuga per decelerare lentamente il rotore da 200 giri/min a 0 giri/min oltre 3 minuti alla fine del giro. - Raccogliere con cura le frazioni di saccarosio di 30-60% nelle provette pulite (Figura 2). Aggiungere a freddo 1 x PBS per frazioni raccolte e portare il volume a 100 mL; mescolare pipettando su e giù più volte.
- Procedere con il passaggio del cuscino di saccarosio di stratificazione con attenzione la preparazione virale su un cuscino di saccarosio. Per questo, è necessario aggiungere 4 mL di saccarosio al 20% (in 1X PBS) al tubo, seguito da ~ 20-25 mL della soluzione virale per tubo. Se i tubi sono meno di tre quarti pieno, rabboccare con PBS 1X sterile.
- Attentamente equilibrio e centrifugare i campioni a 70.000 x g per 2 h a 17 ° C, come prima. Versare il sovranatante e lasciare il restante liquido invertendo i tubi su carta assorbente.
- Aspirare le goccioline restanti al fine di rimuovere tutto il liquido dal pellet. A questo punto, il pellet contenente virus dovrebbe essere appena visibile come piccole macchie traslucide.
- Risospendere il pellet con l'aggiunta di 70 µ l di PBS 1x al primo tubo e accuratamente la sospensione di pipettaggio, successivamente trasferire la sospensione al prossimo tubo e miscelazione come prima, continuando fino a quando tutti i pellet sono risospese.
- Sciacquare i tubi con un ulteriore 50 µ l di freddo 1X PBS e mix come prima. Assicurarsi che il volume totale della sospensione finale è ~ 120 µ l e appare leggermente lattiginoso; deselezionarla per centrifugazione a 10.000 x g per 30 s in una microcentrifuga da tavolo.
- Trasferire il surnatante in una provetta di microfuge fresco, fare 10 aliquote µ l e conservarli a-80 ° C.
Nota: Evitare di eseguire cicli ripetuti di congelamento-scongelamento sui campioni di vettori lentivirali. Tranne quando la centrifugazione è obbligatorio, gli sforzi dovrebbero essere fatto per eseguire i passaggi rimanenti in cappe di coltura del tessuto, o designati camere di coltura del tessuto utilizzando misure di biosicurezza appropriato (vedi discussione).
6. stima dei titoli virali
-
p24 -enzima-collegata dell'immunosorbente (ELISA) metodo
Nota: Il dosaggio è eseguito su utilizzando piastre da 96 pozzetti alta-associazione come secondo le istruzioni del programma NIH AIDS vaccino per l'HIV-1 p24 antigene Capture Assay Kit (Vedi Tabella materiali) con modifiche29.- Il giorno dopo, lavare i pozzetti tre volte con 200 µ l 0.05% Tween 20 in PBS freddo (soluzione di PBS-T). Rivestire la piastra con 100 µ l di anticorpo monoclonale anti-p24 ad una diluizione di 1: 1500 in PBS 1X e incubare per una notte a 4 ° C.
- Per eliminare il legame non specifico, bloccare la piastra con 200 µ l 1% BSA in PBS; lavare tre volte con 200 µ l 0.05% Tween 20 in PBS freddo (soluzione di PBS-T) per almeno 1 h a temperatura ambiente.
-
Preparare campioni: Per i preparati concentrati vettoriale, diluire 1 µ l di campione 100 volte aggiungendo 89 µ l di dd-H20 e 10 µ l di Triton X-100 (concentrazione finale di 10%). Per i preparati non concentrato, preparare dieci volte campioni diluiti (aggiungere 80 µ l di dd-H20 e 10 µ l di Triton X-100 (concentrazione finale di 10%) a 10 µ l di campione).
Nota: I campioni possono essere conservati a-20 ° C a questo passaggio per un lungo periodo di tempo per un successivo utilizzo. - Preparare l'HIV-1 standard applicando una diluizione seriale 2 volte (con un'iniziale concentrazione 5 ng/mL).
- Diluire i campioni concentrati (da scorte pre-diluito 1: 100) in RPMI 1640 completati con 0,2% Tween 20 e 1% BSA per stabilire 01:10, 000, 01:50, 000 e 1:250,000 diluizioni. Diluire i campioni non concentrati (da 01:10 pre-diluito scorte) in RPMI 1640 completati con 0,2% Tween 20 e 1% BSA per stabilire diluizioni 1: 500, 1: 2500 e 1:12,500.
- Applicare i campioni sul piatto in triplici copie e incubare per una notte a 4 ° C.
- Il giorno dopo, lavare i pozzetti sei volte e incubare a 37 ° C per 4 h con 100 µ l policlonali di coniglio anti-p24 anticorpo, diluita 1: 1000 in RPMI 1640, 10% FBS, 0,25% BSA e siero di topo normale 2% (NMS).
- Lavare sei volte come sopra e incubare a 37 ° C per 1 h con perossidasi di rafano anti-coniglio di capra IgG diluito 1: 10.000 in RPMI 1640 completato con siero di capra normale 5%, 2% NMS, 0,25% BSA e 0.01% Tween 20.
- Lavare la piastra, come sopra e incubare con TMB substrato perossidasi a temperatura ambiente per 15 min.
- Fermare la reazione aggiungendo 100 µ l di 1 N HCL. Campione di assorbanza a 450 nm utilizzando l'assorbanza piastra lettore.
-
Misura dell'intensità di fluorescenza reporter
- Metodo di FACS
Nota: La misura di svuotamento del segnale GFP in cellule può essere stimata con precisione misurando intensità media di fluorescenza delle cellule trasdotte tramite flusso cytometry. Fare riferimento alla recente carta 28 per analisi di dati di FACS, presentazione e interpretazione. Il protocollo è descritto come segue.- Fai una diluizione seriale dieci volte della preparazione (da 10-1 a 10-5) della preparazione virale in PBS 1X.
- Semi di circa 5 x 105 293T cellule in ciascun pozzetto di una piastra a 6 pozzetti in un volume finale di 2 mL per pozzetto. Aggiungere 10 µ l di ciascuna diluizione virale alle cellule e incubare le cellule a 37 ° C per 48 h.
- Raccogliere le cellule per l'analisi di FACS come segue: aggiungere 200 µ l di soluzione di tripsina-EDTA 0,05% e incubare le cellule a 37 ° C per 5 min, aggiungere 2 mL di un completo media DMEM e raccogliere campioni in provette coniche da 15 mL.
- Pellet di cellule mediante centrifugazione a 400 x g a 4 ° C e risospendere il pellet in 500 µ l di PBS 1X freddo.
- Per il fissaggio, aggiungere un volume equivalente di soluzione di formaldeide 4% a questa sospensione e incubare per 10 minuti a temperatura ambiente.
- A pellet celle fisse e risospendere in 1 mL di PBS 1X. Analizzare l'espressione di GFP utilizzando uno strumento di FACS, come descritto in Ortinski et al. 28 brevemente, utilizzare la seguente formula per determinarne titolo funzionale il virus':
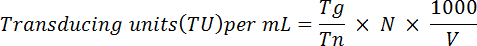
Nota: Qui Tg = numero di cellule GFP-positive ha contato; TN = numero totale di cellule contate; N = numero totale di cellule trasformata; V = volume (µ l) utilizzato per la trasduzione. Ad esempio: se 1 x 106 celle sono state trasdotte con 10 µ l di virus, 2 x 104 cellule erano contati e 5 x 103 erano GFP-positive, basate sull'equazione sopra il titolo funzionale sarebbe:
- Conteggio di cellule GFP-positive
- Calcolare la molteplicità di infezione (MOI) utilizzato per la trasduzione. Testare una vasta gamma di MOIs (1-10), con l'aumento risultante in una maggiore efficienza di trasduzione MOIs.
- Prima della trasfezione, seme una piastra a 6 pozzetti con circa 3-4 x 105 cellule per pozzetto. Una volta che le cellule raggiungono > confluency di 90% (di solito entro 24 h), trasdurre con il virus purificato al MOIs pre-determinato.
- Incubare la piastra a 37 ° C con 5% CO2 in una cultura di tessuto standard e le cellule di monitor a intervalli regolari per 1-7 giorni per i cambiamenti nel segnale GFP.
- Contare il numero di cellule GFP-positive con un microscopio a fluorescenza (piano 4 X obiettivo, 0,1 Nardi, ingrandimento 40x) dotato di un set di filtri GFP (eccitazione lunghezza d'onda-470 nm, emissione lunghezza d'onda-525 nm), utilizzando cellule (ONU-trasdotte) ingenuo per impostare la popolazione di cellule GFP-negativo e positive.
- Stimare il titolo finale regolando per il fattore di diluizione e il volume, utilizzando la seguente formula:
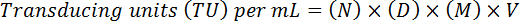
Nota:, N = numero di cellule GFP-positive, D = fattore di diluizione, M = fattore di ingrandimento (di solito 20 X), V = volume del virus utilizzati per la trasduzione. Ad esempio, per 20 cellule GFP-positive (N) ha conteggiate a una diluizione di 10-4 (01:10, 000) in un campione di 10 µ l (V) 20 X ingrandimento (M) (D) comporterebbe un titolo funzionale di (20 x 104) x (20) x (10) x (100 *) = 4 x 108 TU/mL.
(* per regolare / mL)
- Metodo di FACS
Risultati
Convalida dell'eliminazione diretta-efficienza dei vettori IDLV-CRISPR/Cas9
Abbiamo usato cellule 293T che esprimono GFP come un modello per convalidare l'efficienza di knockout di gene CRISPR/Cas9-mediata. Cellule GFP + sono state generate da trasduzione delle cellule HEK 293T con pLenti-GFP (vBK201a) presso un MOI di 0,5 (Figura 3b, pannello di "no-virus"). La cassetta di sgRNA-a-GFP/Cas9 all-in-one vector è stata confezionata in partic...
Discussione
IDLVs hanno cominciato ad emergere come il veicolo della scelta per in vivo gene-editing, soprattutto nel contesto delle malattie genetiche, in gran parte a causa del basso rischio di mutagenesi associata con questi vettori rispetto all'integrazione di piattaforme di consegna22 , 28. nel manoscritto attuale, abbiamo cercato di dettagliare il protocollo associato alla produzione del migliore sistema IDLV-CRISPR/Cas9 all-in-one che recentemente è stato sv...
Divulgazioni
USC-499-P di brevetto (1175) è stata depositata dalla University of South Carolina, in relazione ai lavori descritti in questo manoscritto.
Riconoscimenti
Vorremmo ringraziare il dipartimento di neurobiologia, Duke University School of Medicine e del decanato per scienza di base, Duke University. Ringraziamo anche i membri del nucleo vettore virale Duke per commenti sul manoscritto. Plasmide pLenti CRISPRv2 era regalo da Feng Zhang (Broad Institute). Il sistema di LV-imballaggio tra cui il psPAX2 di plasmidi, VSV-G, pMD2.G e OSPRO-Rev è stato un regalo gentile da Didier Trono (EPFL, Svizzera). Sostegno finanziario per questo lavoro è stato fornito dall'Università di South Carolina School Of Medicine, concedere RDF18080-E202 (Bambi).
Materiali
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| xMark Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| BD FACS | Becton Dickinson | 338960 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| 0.45-μm filter unit, 500mL | Corning | 430773 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| SW32Ti swinging-bucket rotor | Beckman Coulter | 369650 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 50mL conical centrifuge tubes | Corning | 430291 | |
| High-binding 96-well plates | Corning | 3366 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 100mm TC-Treated Culture Dish | Corning | 430167 | |
| 0.22 μM filter unit, 1L | Corning | 430513 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Tissue culture pipettes, 5 mL | Corning | 4487 | |
| Tissue culture pipettes, 10 mL | Corning | 4488 | |
| Tissue culture pipettes, 25 mL | Corning | 4489 | |
| Hemacytometer with cover slips | Cole-Parmer | UX-79001-00 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| 293FT cells | Thermo Fisher Scientific | R70007 | |
| DMEM, high glucose media | Gibco | 11965 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| Antibiotic-antimycotic solution, 100X | Sigma Aldrich | A5955-100ML | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| BES (N, N-bis (2-hydroxyethyl)-2-amino-ethanesulfonic acid) | Sigma Aldrich | B9879 - BES | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Normal mouse serum, Sterile, 500mL | Equitech-Bio | SM30-0500 | |
| Goat serum, Sterile, 10mL | Sigma | G9023 | Working concentration 1:1000 |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| psPAX2 | Addgene | 12260 | |
| pMD2.G | Addgene | 12259 | |
| pRSV-Rev | Addgene | 12253 | |
| lentiCRISPR v2 | Addgene | 52961 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsrGI | New England Biolabs | R0575S | |
| BsmBI | New England Biolabs | R0580S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
Riferimenti
- Horvath, P., Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 327 (5962), 167-170 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat Rev Genet. 11 (3), 181-190 (2010).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Bellec, J., et al. CFTR inactivation by lentiviral vector-mediated RNA interference and CRISPR-Cas9 genome editing in human airway epithelial cells. Curr Gene Ther. 15 (5), 447-459 (2015).
- Kennedy, E. M., et al. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease. Virology. 476, 196-205 (2015).
- Roehm, P. C., et al. Inhibition of HSV-1 Replication by Gene Editing Strategy. Sci Rep. 6, 23146 (2016).
- Wang, W., et al. CCR5 gene disruption via lentiviral vectors expressing Cas9 and single guided RNA renders cells resistant to HIV-1 infection. PLoS One. 9 (12), e115987 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Adv Genet. 87, 125-197 (2014).
- Lebbink, R. J., et al. A combinational CRISPR/Cas9 gene-editing approach can halt HIV replication and prevent viral escape. Sci Rep. 7, 41968 (2017).
- Xu, L., et al. CRISPR/Cas9-Mediated CCR5 Ablation in Human Hematopoietic Stem/Progenitor Cells Confers HIV-1 Resistance In Vivo. Mol Ther. , (2017).
- Yiu, G., Tieu, E., Nguyen, A. T., Wong, B., Smit-McBride, Z. Genomic Disruption of VEGF-A Expression in Human Retinal Pigment Epithelial Cells Using CRISPR-Cas9 Endonuclease. Invest Ophthalmol Vis Sci. 57 (13), 5490-5497 (2016).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), e147 (2014).
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol. 32 (3), 279-284 (2014).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Schaefer, K. A., et al. Unexpected mutations after CRISPR-Cas9 editing in vivo. Nat Methods. 14 (6), 547-548 (2017).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Ther. 23 (7), 627-633 (2016).
- Platt, R. J., et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159 (2), 440-455 (2014).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Res. 43 (13), 6450-6458 (2015).
- Nelson, C. E., Gersbach, C. A. Engineering Delivery Vehicles for Genome Editing. Annu Rev Chem Biomol Eng. 7, 637-662 (2016).
- Jaalouk, D. E., Crosato, M., Brodt, P., Galipeau, J. Inhibition of histone deacetylation in 293GPG packaging cell line improves the production of self-inactivating MLV-derived retroviral vectors. Virol J. 3, 27 (2006).
- Ellis, B. L., Potts, P. R., Porteus, M. H. Creating higher titer lentivirus with caffeine. Hum Gene Ther. 22 (1), 93-100 (2011).
- Hoban, M. D., et al. Delivery of Genome Editing Reagents to Hematopoietic Stem/Progenitor Cells. Curr Protoc Stem Cell Biol. 36, 1-10 (2016).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Mol Ther. 16 (12), 1968-1976 (2008).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proc Natl Acad Sci U S A. 106 (44), 18786-18791 (2009).
- Ortinski, P. I., O'Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Mol Ther Methods Clin Dev. 5, 153-164 (2017).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Mol Ther. 19 (3), 547-556 (2011).
- Berkhout, B., Verhoef, K., van Wamel, J. L., Back, N. K. Genetic instability of live, attenuated human immunodeficiency virus type 1 vaccine strains. J Virol. 73 (2), 1138-1145 (1999).
- Gomez-Gonzalo, M., et al. The hepatitis B virus X protein induces HIV-1 replication and transcription in synergy with T-cell activation signals: functional roles of NF-kappaB/NF-AT and SP1-binding sites in the HIV-1 long terminal repeat promoter. J Biol Chem. 276 (38), 35435-35443 (2001).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. J Biol Chem. 280 (22), 21545-21552 (2005).
- Ortinski, P. I., Lu, C., Takagaki, K., Fu, Z., Vicini, S. Expression of distinct alpha subunits of GABAA receptor regulates inhibitory synaptic strength. J Neurophysiol. 92 (3), 1718-1727 (2004).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. J Virol. 71 (8), 6113-6127 (1997).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. J Virol. 68 (4), 2632-2648 (1994).
- Xu, W., Russ, J. L., Eiden, M. V. Evaluation of residual promoter activity in gamma-retroviral self-inactivating (SIN) vectors. Mol Ther. 20 (1), 84-90 (2012).
- . Testing for Replication Competent Retrovirus (RCR)/Lentivirus (RCL) in Retroviral and Lentiviral Vector Based Gene Therapy Products - Revisiting Current FDA Recommendations Available from: https://sites.duke.edu/dvvc/files/2016/05/FDA-recommendation-for-RCR-testing.pdf (2017)
- . Biosafety in Microbiological and Biomedical Laboratories Available from: https://www.cdc.gov/biosafety/publications/bmbl5/bmbl.pdf (2017)
- Dull, T., et al. A third-generation lentivirus vector with a conditional packaging system. J Virol. 72 (11), 8463-8471 (1998).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon