Method Article
CRISPR 的染色质环结构重组
* 这些作者具有相同的贡献
摘要
染色质循环在基因调控中起着重要作用;然而, 没有技术上的进步, 允许选择性和可逆的修饰染色质循环。在这里, 我们描述了一个强大的体系, 染色质循环重组使用 CRISPR-dCas9 (CLOuD9), 证明有选择和可逆调节基因表达在靶点。
摘要
最近的研究已经清楚地表明, 长范围的三维染色质循环相互作用在基因表达调控中起着重要作用, 但是循环是否负责或者基因表达改变的结果仍然是未知。直到最近, 染色质循环如何影响基因活性和细胞功能的调控是相对含糊不清的, 现有方法的限制, 以控制这些结构, 防止深入探索这些相互作用。为了解决这种不确定性, 我们设计了一种利用 CRISPR-dCas9 (CLOuD9) 进行选择性和可逆染色质循环重组的方法。CLOuD9 系统的活力表现为成功地将 CLOuD9 构造定位到目标基因组位点, 以调节局部染色质构象。重要的是, 有能力逆转诱导接触和恢复内源性染色质构象也被证实。基因表达的调制通过这种方法建立了调节细胞基因表达的能力, 并强调了这一技术在创造稳定的全新的染色质循环, 显著影响基因的巨大潜力在癌症和发展的语境中表达。
引言
细胞核内染色质折叠与基因组的特定组织之间的关系近年来得到了极大的关注, 因为它已被证明与基因表达1,2密切相关。虽然基因活性与染色质结构调控的精确关系尚不清楚, 但据推测, 由于动态三维染色质组织的染色体接触之间的相互作用, 为基因调控功能3。事实上, 这种效应已经在人类球蛋白基因轨迹上得到了很好的证明, 在那里, 轨迹控制区域 (LCR) 通过在两个区域之间创建一个染色质循环来调节球蛋白基因在发育特定方式中的活动。然而, 在这两个地区, 还不清楚染色质循环是否是基因表达改变的原因或结果。
到目前为止, 研究这一现象的挑战仍未解决。例如, 其他诱导染色质循环的尝试涉及修改线性 DNA 序列或复杂的程序, 需要丰富的背景知识的特定元素, 以促进循环5,6, 7,8。此外, 虽然以前的工作表明染色质循环驱动基因表达在特定和限制的上下文7,8, 染色质循环影响转录全球的水平是不确定的。近年来, 尽管对长期循环对基因表达的影响的兴趣不断增加, 但关于建立和保留染色质接触以改变基因活性的问题仍然存在。
我们所设计的技术使用核酸酶缺乏聚类定期 interspaced 短回文重复 (CRISPR) CRISPR 相关蛋白 9 (dCas9), 以允许广泛适用的目标, 任何基因组的基因座9。该技术消除了与线性 DNA 序列的修改有关的复杂问题, 并且在没有对特定循环组件有重大先验知识的情况下可以访问。最值得注意的是, 该工具具有普遍性, 广泛适用于发展中公认的染色质循环以及各种疾病, 如癌症。通过可逆改变循环结构, 有效地调节基因表达, 证明了 CLOuD9 的威力。
研究方案
1. gRNA 设计
- 选择标准的 gRNA 设计工具来设计指南 rna10。使用以前开发的 CLOuD9 构造9 (即至少1的金黄色葡萄球菌(CSA) 和 1 s 化脓(CSP) 结构) 对每个实验进行互补对。
- 在所选的联机设计工具中, 使用 Protospacer 相邻的母题 (PAM) 序列 "NGG", 其引导长度为 20 bp, 用于 CSP 和 PAM 序列 "NNGRRT", 并引导长度为21的 CSA。
- 根据特异性评分和设计指南选择参考线, 以最大化获得工作指南的机会。
- 从寡聚糖制造商订购2寡核苷酸: 对于前向寡聚, 添加 "caccg" 到指南序列的 5 ' 末尾, 对于反向寡聚, 在订购前将 "aaac" 添加到 5 ' 端和 "c" 到 3 ' 端。
- 最初, 设计 3-5 gRNAs 为每个感兴趣的区域, 分布在 250-1000 bp 区域。评估 gRNA 目标效率如下:
- 克隆鉴定 gRNAs 为活性 Cas9 质粒 (见步骤 3), 瞬时染成293T 细胞 (见步骤 4)11。
- 生长细胞为 2-3 d 在 Dulbecco 的改良鹰培养基 (DMEM) 与10% 胎牛血清 (血清) 和1% 青霉素-链霉素 (枝链球菌), 然后收获和提取基因组 DNA12。
- 聚合酶链反应 (PCR) 放大靶区。在1.5% 琼脂糖凝胶上运行产品并纯化合适的带。
- 执行 T7 限制性化验, 如 Guschin 所述. 协议13,14。
注意: 有效的指南是那些决心在目标地点切割 DNA 的人。
2. 细胞培养
- 保持细胞处于健康和积极的分裂状态。
- 为 K562s, 文化在罗斯威尔公园纪念学院 (RPMI) 1640 个媒介与10% 个血清和1% 支笔链球菌。
- 生长 K562s 在一个25厘米2倾斜颈烧瓶, 以维护和调整细胞密度为40万细胞每毫升每天。
- 在 K562s 与 CLOuD9 构造转基因后, 添加2µg/毫升嘌呤霉素和100µg/毫升潮霉素到维护介质。
- 对于 293Ts, Dulbecco 的改良鹰培养基 (DMEM) 的培养, 有10% 只血清和1% 枝链球菌。
- 生长293Ts 在 10 cm2板材和分裂, 当汇合。
- 为 K562s, 文化在罗斯威尔公园纪念学院 (RPMI) 1640 个媒介与10% 个血清和1% 支笔链球菌。
3. 质粒制备和 gRNA 插入15
注: 质粒图可在附录中使用, 在示例实验中采用的引物可在补充表 1中获得。
- 混合5µg 的慢病毒载体 CRISPR 质粒与3µL BsmBI, 3 µL 碱性磷酸酶, 6 µL 10x 缓冲, 0.6 µL 新鲜准备100毫米。将总容积60µL 与 ddH2O, 孵育37°c 30 分钟, 以消化和 dephosphorylate 质粒。
- 凝胶纯化 ddH2O 的消化质粒和洗脱。
- 混合1µL 的每一个配对的指南在100µM 与1µL 10x T4 结扎缓冲, 6.5 µL2O, 0.5 ddH µL T4, 达到 10 PNK 总容量。孵育在37°c 为30分钟, 然后95°c 为5分钟, 然后舷梯下来到25°c 在5°c/分钟。
注: 这将退火对指南。 - 稀释退火的导轨 (从步骤 3.3) 1:200 在 ddH2O。
- 1µL 的消化质粒从步骤3.2 与0.5 µL 稀释结扎指南从步骤 3.4, 2.5 µL 2x 缓冲, 1 µL ddH2O 和0.5 µL 连接酶, 使5.5 µL 总反应。在室温下孵育10分钟, 结扎 BsmBI 消化质粒的导则。
- 将新结扎的质粒从步骤3.5 转化为 Stbl3 菌, 并利用任何质粒制备方法进行放大。
4. 慢病毒北疆生产
- 75万293T 细胞每井为一个六井板使用 hemocytometer 和种子他们在 DMEM 与10% 血清和1% 笔链球菌。每个构造都使用一个井。
- 24小时播种后, 将培养基改成新鲜无抗生素 DMEM, 10% 血清。
- 在一管, 稀释11µL 脂基转染试剂与150µL 的 OptiMEM 培养基, 每反应11。
- 在一个单独的管, 稀释2µg 的 CLOuD9 慢病毒载体载体质粒从步骤 3.6, 0.35 µg pMDLg/pRRE, 1 µg 的 pRSV-转速, 0.65 µg 的 pMD2 培养基, 每反应150。
- 每个反应, 添加稀释慢病毒载体质粒混合物稀释脂基转染试剂在1:1 的比例, 并孵化5分钟11。
- 将每个混合复合体的整个体积从步骤4.5 添加到步骤4.2 中无抗生素293T 细胞的各自井中。
- 48小时后, 通过吹打收集病毒生产介质到一个新鲜的管和旋转下来在 300 x g 5 分钟。离心后, 将上清液移至新鲜管, 以去除残余细胞碎片。
- 立即使用病毒制作培养基进行靶细胞转导, 或在-80 摄氏度处冷冻以供将来使用。
5. 细胞的慢病毒北疆转导
- 添加250µL 的每一个病毒结构的兴趣 (包括互补 CLOuD9 质粒) 到15毫升圆锥管, 其中包含8万细胞 CLOuD9 应用。
- 将每个锥形到1毫升的介质总容积与无抗生素的培养基结合在一起, 并将凝聚胺添加至1-8 µg/毫升之间的最终浓度 (凝聚胺所使用的细胞类型耐受的最大浓度)。
- 在室温下, 旋转细胞在 800 x g 处为30分钟, 然后通过吹打并用重悬, 而不去除病毒上清。将整个细胞悬浮液移到细胞培养板上。
- 24小时后转导, 自旋细胞下降 300 x g 在室温下5分钟。
- 在常规细胞培养培养基中吸入病毒上清和并用重悬细胞。
- 第二天, 添加嘌呤霉素 (1 µg/毫升293T 细胞) 和潮霉素 (25 µg/毫升293T 细胞) 到细胞培养基中选择双转基因细胞。在实验开始之前, 实验确定了给定细胞类型的嘌呤霉素和潮霉素的适当浓度。
- 在任何下游实验之前, 在选择介质中保持细胞至少3维, 并在所有实验期间保持在选择介质中。
6. 细胞二聚化和洗涤
- 添加1毫米脱落酸 (ABA) 或当量的二甲基亚砜 (亚砜), 用于控制细胞培养, dimerize CLOuD9 选择和转基因细胞。使用 ABA 在6月的时间内收到, 保持寒冷, 并保护它从光在整个使用。如果需要, 可以使用2毫米 ABA。
- 在实验期间, 每天改变媒体以提供新鲜的 ABA (或用于控制的亚砜)。
注意: 尚未观察到二聚化长度的限制。
- 在实验期间, 每天改变媒体以提供新鲜的 ABA (或用于控制的亚砜)。
- 反向二聚化通过去除含有 ABA 和洗涤细胞的介质, 用足够的磷酸盐缓冲盐水 (PBS) 覆盖板表面, 两次。此后, 在无 ABA 的培养基中培养细胞保持不二聚状态。
7. 免疫沉淀和 immunoprecipitations
注意: 使所有缓冲区新鲜, 并在使用前立即。
- 完成二聚化实验后, 收集和旋转细胞, 然后吸入上清液。
- 交联和冻结细胞
- 用新鲜材料在室温下在 PBS 中制作1% 甲醛的新库存。每200万个细胞, 在室温下轻轻地并用重悬1毫升1% 甲醛, 而旋转时则为10分钟。
注: 使用最小体积10毫升交联不到1000万个细胞。 - 将甘氨酸添加到最终浓度为0.125 米的淬火交联, 然后在室温下孵化5分钟, 同时旋转。
- 向下旋转细胞, 用冰冷的 PBS 清洗1-2x。细胞颗粒可以在液氮中凝固并储存在-80 摄氏度或立即使用。
注: 热会逆转甲醛 crosslinks。如果被冲出, 细胞可以直接储存在-80 °c, 而不冻结。
- 用新鲜材料在室温下在 PBS 中制作1% 甲醛的新库存。每200万个细胞, 在室温下轻轻地并用重悬1毫升1% 甲醛, 而旋转时则为10分钟。
- 制备抗体/珠共轭
注: 优化选择抗体的条件 (i. e, 测试不同的裂解缓冲细胞裂解和执行 IP) 可能是值得的。两个常见的缓冲器, 法纳姆裂解缓冲器和修改后的马国贤缓冲器, 可对 IP 效率和超声波效率产生显著影响。所有的缓冲成分都可以在材料表中找到。此外, 请记住, 染色质超声波可能需要几个小时才能完成。- 并用重悬珠由涡流简要地适当为选择的抗体。
- 将适量的珠子转移到1.5 毫升的试管中。作为出发点, 每1µg 抗体使用20µL 珠。还没有添加抗体。
注: 使用10µg 抗体与200µL 的珠子为1毫克的总裂解液作为一个出发点, 除非它是已知的抗体是或多或少有效比这意味着。对于低丰度蛋白质, 这个比例可能需要调整。 - 如果使用法纳姆裂解缓冲器, 请在 IP 稀释缓冲器中洗3x。如果使用马国贤裂解缓冲液, 请在马国贤裂解缓冲液中冲洗3x。
- 并用重悬珠在与步骤 7.3.3 (IP 稀释或马国贤缓冲器) 相同缓冲区的最终卷中, 即 > 1x 和 < 5x 初始卷。即对于100µL 初始珠体积, 使用100和500µL 最终泥沙体积。
- 添加抗体的洗涤珠现在在适当的浓度。对于一个良好的 HA 或标志抗体的 IP CLOuD9 构造, 使用抗体在 1:50 (µg 蛋白: µg 蛋白裂解) 和孵化之间 8 + h 和隔夜4摄氏度, 而旋转。另外, 在室温下孵育2-4 小时。
- 从珠子上取出上清, 然后丢弃。
- 洗净珠3x 与洗涤缓冲。
- 并用重悬在最小的缓冲容积中使用的抗体 (IP 稀释或马国贤缓冲) 过夜孵化。保持冷, 直到结合蛋白裂解。
- 油脂实验染色质
- 首先溶解细胞, 增加肿胀缓冲细胞在冰上10分钟, 每隔几分钟弹细胞以防止沉淀。
注: 一般情况下, 500 µL 的肿胀缓冲器被添加到2500万个细胞, 并从那里进行缩放, 但不使用少于500µL 缓冲 < 2500万细胞。 - Dounce 手动和逆时针, 每13x。
- 然后将原子核向下旋转4摄氏度, 5 分钟, 1500 x g. 完全吸出上清, 重复清除剩余的上清, 然后在 mg 中称量细胞颗粒。
- 将蛋白酶抑制剂添加到核裂解缓冲液中 (法纳姆缓冲器或马国贤缓冲液, 选择一个)。
- 添加10x 量的核裂解缓冲液颗粒状核到并用重悬 (例如, 添加1毫升的核裂解缓冲到0.100 克颗粒)。简要地漩涡和溶解为10分钟在冰。
注意: 请记住超声波管的尺寸。理想体积通常 < 1 毫升, 因此样品可能需要被划分, 如果球团是非常大的。- 为共同 IP, 油脂实验原子核简要地溶解材料和淬火 SDS 到允许免疫沉淀与2-3 容量稀释缓冲的水平, 然后进入步7.4.6。对于非常敏感的抗体, 添加更多的稀释缓冲, 以进一步减少 SDS 浓度。
注: 当裂解液清除时停止超声波;它将从不透明/半透明的白色变为完全透明。尽量保持样品的冷, 不要超过油脂实验。 - 对于芯片, 彻底油脂实验 dna 获得 150-1000 bp dna 片段, 然后进行步骤7.4.6。
- 为共同 IP, 油脂实验原子核简要地溶解材料和淬火 SDS 到允许免疫沉淀与2-3 容量稀释缓冲的水平, 然后进入步7.4.6。对于非常敏感的抗体, 添加更多的稀释缓冲, 以进一步减少 SDS 浓度。
- 在4摄氏度时, 以最大速度旋转10分钟不溶物。
- 将可溶性材料转移到新鲜的管子上。
- 对于 Co IP, 测量蛋白质的浓度, 并在步骤7.5 中进行下拉。
- 对于芯片, 删除10µL 整除的清除裂解, 并添加40µL IP 洗脱缓冲器和6µL 5 米氯化钠, 总共56ul。煮沸15分钟在95°c, 增加5-10 µL 3 M NaOAc pH 5, 并且清理在 PCR 纯化专栏。通过测量 260 nm 的吸光度并对样品进行标准化, 测量 DNA 浓度。然后在步骤7.5 中进行下拉。
注: 额外的裂解液可以在-80 摄氏度凝固并在以后使用, 但最好的结果是从新鲜的裂解液中获得。如果结冰, 不要多次冷冻/解冻裂解液。
- 首先溶解细胞, 增加肿胀缓冲细胞在冰上10分钟, 每隔几分钟弹细胞以防止沉淀。
- 拉
- 将适量的蛋白质裂解液转移到新鲜的试管中。如果使用法纳姆裂解缓冲器, 稀释2.1 卷的 IP 稀释缓冲器 (上)。
- 留出100µL 的清液输入控制, 并存储在4摄氏度, 直到第二天。
- 添加珠/抗体共轭从步骤7.3.8 到样品现在。
- 旋转样品在4°c 过夜, 并确保有足够的容积为液体运动在1.5 毫升管。
- 洗涤和洗脱
- 夜间旋转后, 将上清液保存为约束力分数。然后在 IP 稀释缓冲器中清洗珠子3-5x。
- 在室温下制备 IP 洗脱缓冲器, 无抑制剂。
- 洗脱配合物与50µL 洗脱缓冲在漩涡震动67°c 15 分钟. 转移洗脱到一个新的管, 并重复与另50µL. 将两者结合在一起, 共100µL 洗脱。
注: 如果有太多的背景从这个洗脱程序, 热量可以减少或消除在晃动/孵化。这通常会降低靶蛋白的产量, 但明显降低了背景。
- 反向 crosslinks 加热67摄氏度 > 4 小时。
注意: 这对于联合 IPs 来说是不必要的, 但会有帮助。- 为共同知识产权, 以确保在利益目标之间的完全二聚化, 运行 eluates 在 SDS 页凝胶和探针抗体对 HA 标签或标志标签, 如所示, 所有在 1: 1000。然后继续步骤8。
- 对于芯片 qPCR, 为了确保每个 CRISPR-dCas9 部件的正确定位和目标化, 在10µL 中, 在柱上清洁洗脱产品并洗脱. 对纯化 DNA 进行实时定量聚合酶链反应 (qPCR)。在提交的数据中使用的引物可在补充表 1中获得。然后继续步骤8。
8. RNA 提取和定量 PCR
- 探讨基因表达的 CLOuD9-induced 变化, 首先从控制细胞颗粒和二聚细胞中分离纯化总 RNA。
- 用反向转录17进行纯化 RNA 的互补 DNA (cDNA), 并进行 qPCR 分析。辅助表 1提供了底漆。
9. 染色体构象捕获试验
- 观察 CLOuD9 诱导的基因组基因座接触频率的变化, 进行染色体构象捕获 (3C) 测定8。
- 用定量实时 PCR 技术对三种生物复制中的每一个副本进行3C 结扎产品的量化。将样本规范化为来自蛋白轨迹的3C 信号。辅助表 1提供了底漆。
结果
CLOuD9 诱导可逆β-球蛋白启动子-LCR 循环.适当使用 CLOuD9 系统, 通过添加或去除 ABA 到细胞培养介质 (图 1a), 诱导互补 CSA 和 CSP CLOuD9 结构的可逆接触。CSA 和 CSP 构造 (图 1b) 使用标准 CRISPR gRNAs 本地化到适当的基因组区域。考虑到人类球蛋白轨迹的大量文献, 以及在发展过程中发生的频繁染色体折叠和重排, 该区域被选择来证明 CLOuD9 系统的实用性。此外, K562 细胞线被选中, 因为它已被证明始终如一地表达高水平的胎儿γ球蛋白基因, 而不是β-球蛋白基因, 通常表达在健康的成年红谱系细胞。利用 K562 细胞, 可以通过尝试恢复细胞系中β-球蛋白基因的表达来检测 CLOuD9 修饰基因表达的能力。
在诱导二聚化之前, 采用染色质免疫沉淀定量 PCR (芯片 qPCR), 以确保每个 CLOuD9 成分的准确定位和靶向 (补充图 1)。此外, 免疫沉淀 (co IP) 与和没有 ABA 验证 CSA 和 CSP 二聚化在配体的存在, 以及可逆性在没有配体 (图 1c和补充图 2)。24 h 添加 ABA 后, β-球蛋白和 LCR 之间的接触量在二聚化部分的细胞中以染色体构象捕获 (3C) 来测量, 而不是仅包含两个 CSA 或 CSP 结构的控制, 从而验证了靶点染色质变化的特异性 (图 1c和补充图 34)。创建 lcr 球蛋白交互并没有完全消除内生 lcr 球蛋白联系人, 而是添加到原始联系人中, 如以前报告的8。除了目标 LCR 和β-球蛋白启动子区域内的确切区域 (补充图 56) 之外, 观察到的β-球蛋白/LCR 接触增加了多达 72 h 的二聚化。最后, 在去除 ABA 后, 对系统的可逆性进行了3C 的确认, 显示了内源构象的完全更新 (图 1d和补充图 3-6)。
我们认为 K562 细胞中的成功可能是染色质区域球蛋白轨迹位置的结果 (图 1d), 因此利用第二个细胞线来探索这个想法。CLOuD9 系统被应用到 HEK 293T 细胞在异色和不表达球蛋白基因的区域 (图 1e)。结果与 K562s 中观察到的相似;在24小时后用 ABA (图 1e和补充图 3) 测量了更多的β-球蛋白 LCR 关联, 为 CLOuD9's 在不同细胞环境中功能的强健能力提供了证据, 尽管最初的染色质状态或构象。
额外的基因座被测试, 以确保广泛适用的 CLOuD9, 包括 Oct4 启动器和远端 5 ' 增强剂内293T 细胞。以前, 在这个细胞线上没有可检测到的 Oct4 表达, 而且没有任何内源接触的描述。与远端 5 ' 增强剂接触引起的胚胎干细胞中 Oct4 表达的证据激发了这一实验, 并在β-球蛋白轨迹18上观察到了同样的结果。Oct4 远端增强剂和启动子之间的接触是在 CLOuD9 被激活的细胞中发现的, 而不是控制单元 (图 1f)。此外, 还发现, Oct4 启动子和远端 5 ' 增强剂相互作用也促使 3 ' 增强剂接触 Oct4 启动子。这一事件与 3 ' 增强剂在内源性基因活化10中与 Oct4 promoter/5 远端增强物相互作用的证据一致。
CLOuD9 诱导基因位点的上下文特异改变.在确认 CLOuD9 系统确实诱发基因位点的染色体接触后, 我们试图研究循环对基因表达的影响。据文献记载, 球蛋白和 Oct4 基因的转录取决于 LCR 和球蛋白基因位点之间的联系, 以及远端 5 ' 增强剂和 Oct4 启动子之间的接触, 分别为1、11。因此, 我们假设, 使用 CLOuD9 系统驱动染色质环形成在这些地区, 将导致引人注目的基因表达。
在这两个基因座中, qPCR 表明 ABA 诱导的染色质循环推动了293T 细胞 Oct4 表达的增加, 在 K562 细胞中的β球蛋白表达, 虽然不是 293Ts (图 2a)。尽管 aba 在细胞培养中的添加量在24小时内增加了β-球蛋白表达的显著性, 但在 aba 的冲洗后, 其表达持续增加了72个小时, 并可逆转 (图 2a)。除了控件的所有 K562 单元格都遵循这种趋势, 无论二聚化组件位于 LCR 和β球蛋白启动子区域 (图 2a和补充图 78)。为支持这些发现, H3K4me3 和 RNA qPCR 在 K562s 和293Ts 的β-球蛋白轨迹上的芯片--II. 与观察到的转录变化 (图 2c-f) 相对应。
CLOuD9 建立稳定的染色质循环.虽然短期回路感应与 CLOuD9 清楚地跟随了期望, 是否长期诱导循环有区别作用仍然被观察。为了调查这一情况, 细胞在 ABA 的存在下培养了10天。虽然 K562s 和293Ts 在β-球蛋白轨迹和 LCR 相对于控制之间的接触频率增加 (图 3a, b和补充图 910), 但β-球蛋白表达式的改变仍然只观察在 K562 单元格中 (图 3c)。然而, 有趣的是, 据指出, K562s 的长期二聚化, 转录强烈上调, 已不再可逆 (图 3a和补充图 9-11)。然而, 在 293Ts, 如果在长期二聚化后没有观察到转录的改变, 染色质接触的诱导改变仍然是可逆的 (图 3b和补充图 9)。
总的来说, 在 ABA 摘除10天后, 基因表达仅有小的下降, 二聚化之前的基因表达水平仍显著高于 (图 3c)。因此, K562 细胞, 但不是293T 细胞, 显示 H3K4me3 和 RNA 在β-球蛋白位点的持续变化的芯片 qPCR, 相比对照, 即使在10天的 ABA 去除 (图 3d-f)。因此, 我们的结果表明, 染色质循环的稳定性意味着更可持续的基因表达。
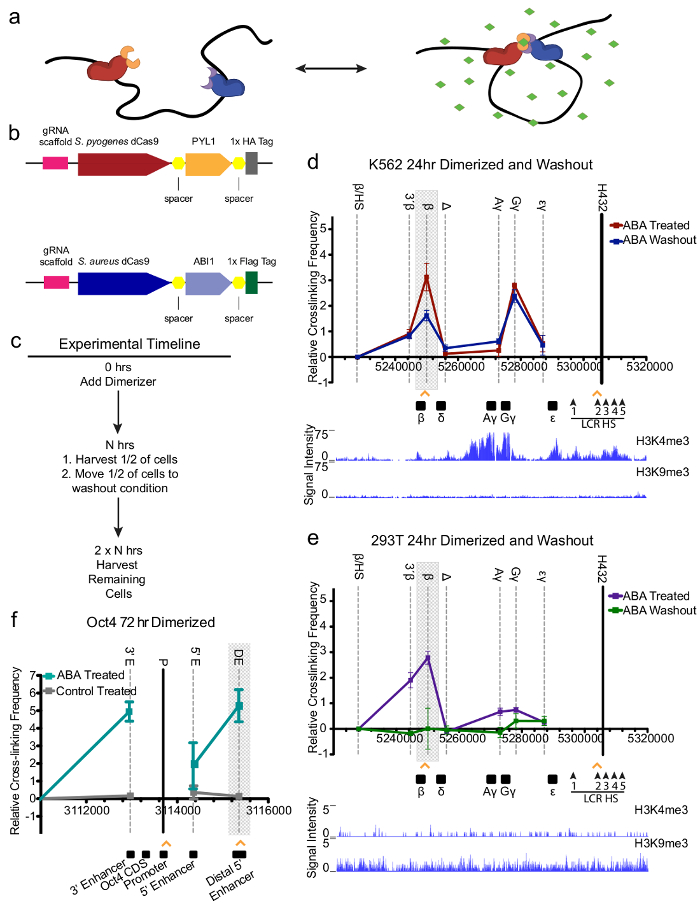
图 1: CLOuD9 诱导可逆β-球蛋白启动子-LCR 循环。(a)脱落酸的增加 (ABA, 绿色) 带来两个互补的 CLOuD9 构造 (CLOuD9 化脓 (CSP), CLOuD9 的金黄色葡萄球菌 (CSA), 红色和蓝色分别) 进入接近, 重塑染色质结构。去除 ABA 恢复内源染色质构象。(b) CLOuD9 构造结合 CRISPR-dCas9 技术从金黄色葡萄球菌和化脓与可逆 dimerizeable PYL1 和 ABI1 领域。(c) CLOuD9 二聚化实验的时间线。(d) 3C 测定 K562 细胞中β-球蛋白的交联频率, 在24小时后, ABA (红色) 和随后的冲洗 (蓝色) 显示诱导β-球蛋白/LCR 接触的可逆性 (以灰色突出显示)。橙色箭头表示特定的 CLOuD9 构造目标区域。EcoRI 片段包含了1-4 的 LCR (黑条) 的敏感点, 被用作锚区域。它的交联频率与其他被表明的 EcoRI 片断 (名字在图的顶部) 被评估了。人类β-球蛋白基因和 LCR 敏感点被描述在图的底部与染色体位置坐标。来自 H3K4me3 和 H3K9me3 的芯片序列数据表明, 该区域在 K562s euchromatic. (e)在 HEK 293T 细胞中, 染色质结构的类似可逆变化见, 尽管 H3K4me3 和 H3K9me3 芯片序列数据表明球蛋白区域在该单元格类型中异色。(f) 3C 测定 Oct4 72 小时后293T 细胞中的全轨迹交联频率, 显示诱导的 Oct4/distal 增强剂接触 (灰色突出显示)。橙色箭头表示特定的 CLOuD9 构造目标区域。将含有 Oct4 启动子 (黑条) 的 MboI 片段用作锚区。它的交联频率与其他被表明的 MboI 片断 (名字在图的顶部) 被评估了。人 Oct4 区域在图的底部被描述以染色体位置坐标。所有的3C 结果都是从至少三个独立实验中获得的。3C 值被规范化为蛋白。对于β球蛋白, 锚片段与包含β/HS 片段的片段之间的相互作用频率设置为零。对于 Oct4, Oct4 相互作用区域外的锚点与负控制片段之间的相互作用频率设置为零。错误条指示 s.d. n=3。此数字已从摩根、孙燕姿等图 1中修改. 9.请点击这里查看这个数字的更大版本.
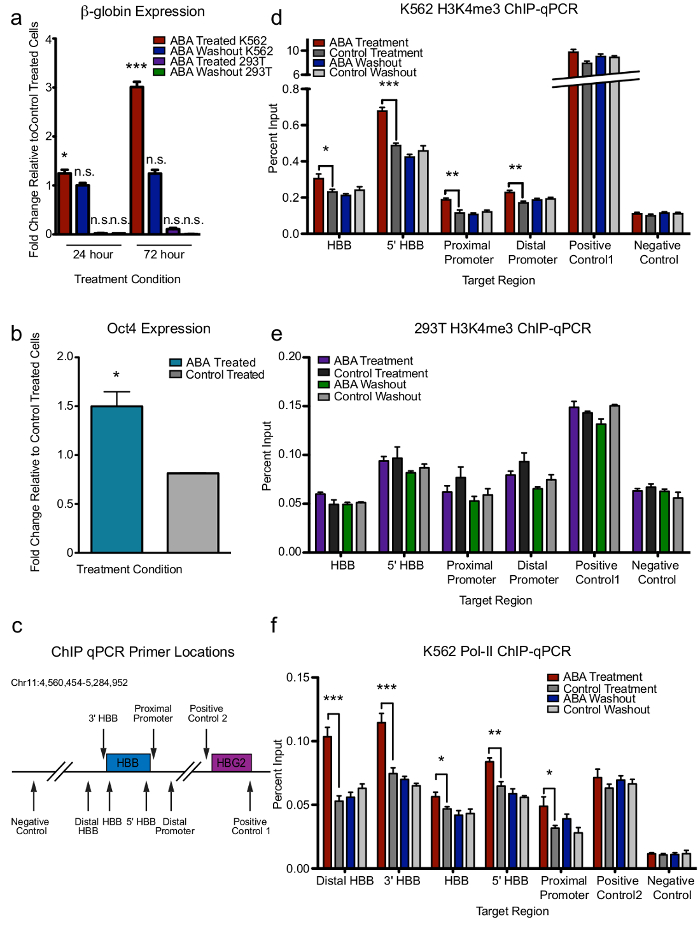
图 2: CLOuD9 诱导基因表达和染色质状态的上下文特异改变。(a) CLOuD9-induced 染色质循环在β-球蛋白的位置导致β球蛋白在 K562s 的可逆诱导, 但不在 293Ts. 给予控制治疗细胞的意义。二尾学生的t测试 * P < 0.05, t = 3.418, df = 5;P < 0.0001, t = 10.42 df = 5;生理盐水不重要。错误条表示 s.d. n = 3。(b)在同一部位 CLOuD9-induced 循环后293Ts 观察到 Oct4 表达的诱导。给出了相对于控制治疗细胞的意义。二尾学生的t测试 * P < 0.05, t = 4.562, df = 2。误差条表示 s.d. (c)芯片 qPCR 底漆位置沿β球蛋白基因体的示意图。(d、e)芯片-qPCR 在 K562s 的β-球蛋白轨迹上显示 H3K4me3 的可逆变化, 但在 CLOuD9-induced 循环后293Ts。双尾学生的t检验 < 0.05, ** p < 0.001, *** p < 0.0001。误差条表示 s.d. (f) CLOuD9 介导的β-球蛋白转录在 K562s 中的变化与 RNA-球蛋白基因体的整个整体的增加有关。双尾学生的t检验 < 0.05, ** p < 0.001, *** p < 0.0001。错误条指示 s.d。这一数字已从摩根、孙燕姿等图 2中修改. 9.请点击这里查看这个数字的更大版本.
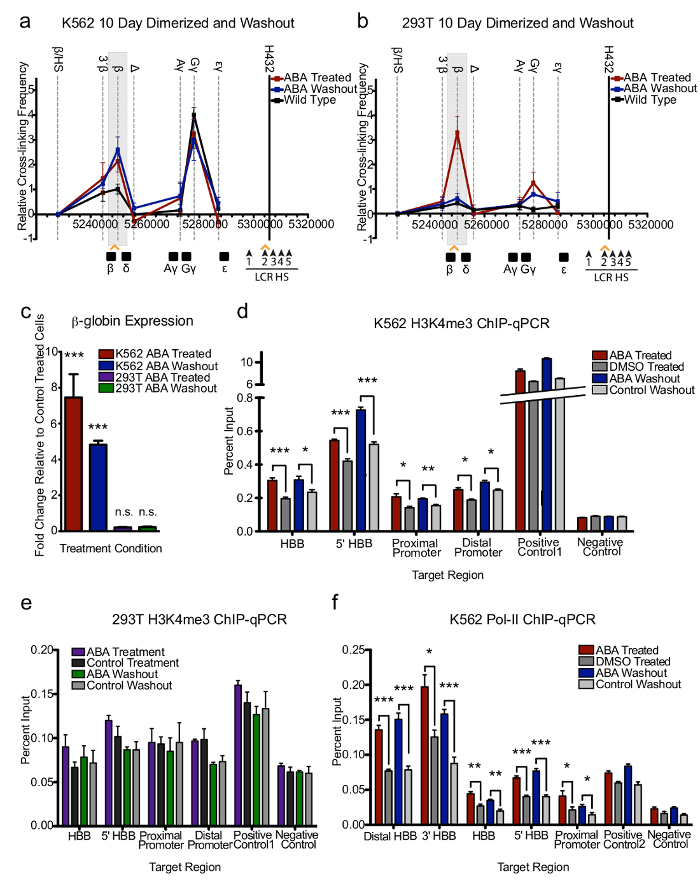
图 3: CLOuD9 建立稳定的染色质循环, 在长期二聚化下维持强健的基因表达。(a, b) 3C 测定表明, 在 K562s 但不是 293Ts, CLOuD9-induced 染色质循环在10天的 aba 治疗后变得不可逆转, 即使 aba 被移除多达10天。所有的3C 结果都是从至少三个独立实验中获得的。3C 值归一化为蛋白, 锚点与包含β/HS 片段的片段之间的相互作用频率设置为零。错误条表示 s.d. n = 3。(c) K562s 回路稳定, 结果持续表达β-球蛋白, 甚至继10天的 ABA 冲洗。在293Ts 中观察不到β-球蛋白表达的变化. 相对于控制治疗细胞的意义。二尾学生的t测试 ** P < 0.0001, t = 5.963, df = 5;生理盐水非显著性(d)芯片 qPCR 显示在 CLOuD9-induced 循环10天后, 对 K562s 环的β-球蛋白轨迹的 H3K4me3 标记的增加是持续的. 两尾学生的t检验 < 0.05, ** p< 0.001, ** P < 0.0001。(e) 293Ts 的芯片 qPCR 观察到长期二聚化后 H3K4me3 信号没有明显的变化. (f)在 K562s 长期循环诱导后, β-球蛋白轨迹的占用增加了。经过10天的配体冲洗。双尾学生的t检验 < 0.05, ** p < 0.001, *** p < 0.0001。所有误差线都指示 s.d。这一数字已从摩根、孙燕姿等图 3中修改. 9.请点击这里查看这个数字的更大版本.
补充图 1: CLOuD9 构造本地化到其预期目标区域.染色质免疫沉淀和定量 PCR 的 CLOuD9 构造表明正确的定位, 其预期的基因座。此数字已从摩根、孙燕姿等的补充图 1中修改. 9.请点击这里下载这个数字.
补充图 2: CLOuD9 构造可逆的关联, 以回应 ABA 的治疗.immunoprecipitations 表明 dCas9 蛋白在72小时后 ABA 治疗后的关联, 在随后的 72 h 的配体冲洗后逆转。此数字已从摩根、孙燕姿等的补充图 2中修改. 9.请点击这里下载这个数字.
补充图 3: 控制治疗导致染色质接触没有变化.用控制剂亚砜治疗24小时, 可使 K562 细胞或 HEK 293Ts 中的内源染色质构象无任何变化. 3C 值归一化为蛋白, 锚片段与片段之间的相互作用频率包括β/HS 片段被设置为零。误差线表示 SD n = 3。此数字已从摩根、孙燕姿等的补充图 3中修改. 9.请点击这里下载这个数字.
补充图 4: 控制 CLOuD9 转基因细胞显示染色质循环没有改变.将两个 CLOuD9 结构定向到 LCR 或β-球蛋白启动子, 在 ABA 治疗相对于控制治疗的情况下, 诱导3C 的染色质结构无显著变化。3C 值归一化为蛋白, 锚点与包含β/HS 片段的片段之间的相互作用频率设置为零。误差线表示 SD n = 3。此数字已从摩根、孙燕姿等的补充图 4中修改. 9.请点击这里下载这个数字.
补充图 5: CLOuD9 染色质循环在72小时二聚化后保持可逆.3C. K562s 的检测表明, 在 ABA 治疗72小时后, CLOuD9 诱导的β-球蛋白/LCR 接触的可逆性。3C 值归一化为蛋白, 锚点与包含β/HS 片段的片段之间的相互作用频率设置为零。误差线表示 SD n = 3。此数字已从摩根、孙燕姿等的补充图 5中修改. 9.请点击这里下载这个数字.
补充图 6: CLOuD9 诱导的β-球蛋白/LCR 循环不受球蛋白目标站点的影响.将 CSA 和 CSP 结构定向到 LCR 或β-球蛋白启动子的交替区域, 在 ABA 治疗72小时后, 在回路诱导中产生类似的可逆变化。3C 值归一化为蛋白, 锚点与包含β/HS 片段的片段之间的相互作用频率设置为零。误差线表示 SD n = 3。此数字已从摩根、孙燕姿等的补充图 6中修改. 9.请点击这里下载这个数字.
补充图 7: 无论球蛋白目标部位如何, CLOuD9 诱导的基因表达改变都是持续的.将 CSA 和 CSP 结构定向到β-球蛋白启动子和 LCR 的交替区域, 对72小时二聚化后基因表达的诱导没有影响。然而, 虽然观察到长期 (10 天) 二聚化对基因表达强度的影响, 但在随后的配体冲洗10天后, 相对于控制治疗细胞的β-球蛋白的含量也有很大的增加。给出了相对于控制治疗细胞的意义。**p < 0.001, t = 10.25, df = 5;p < 0.0001, 左到右 t = 8.697, df = 6, t = 40.31, df = 7;生理盐水不重要。所有误差线都指示 SD。此数字已从摩根、孙燕姿等的补充图 7中修改. 9.请点击这里下载这个数字.
补充图 8: 控制 CLOuD9 转基因细胞显示β-球蛋白表达式没有改变.将两个 CLOuD9 结构定向到 LCR 或β-球蛋白启动子, 在 ABA 治疗相对于控制治疗后诱导β-球蛋白表达无显著变化。给出了相对于控制治疗细胞的意义。生理盐水不重要。此数字已从摩根、孙燕姿等的补充图 8中修改. 9.请点击这里下载这个数字.
补充图 9: 长期控制治疗不会导致染色质接触的变化.用10天的控制剂亚砜治疗, 在 K562 细胞或 HEK 293Ts 中, 不导致内源性染色质构象改变 3C. 3C 值归一化为蛋白, 锚片段与片段之间的相互作用频率包括β/HS 片段被设置为零。误差线表示 SD n = 3。此数字已从摩根、孙燕姿等的补充图 9中修改. 9.请点击这里下载这个数字.
补充图 10: 长期 CLOuD9 诱导的β球蛋白/LCR 循环不受球蛋白目标站点的影响.将 CSA 和 CSP 结构定向到 LCR 或β-球蛋白启动子的交替区域, 可在10天的 ABA 处理后和10天后配体冲刷后, 以3C 的方式显示类似的持续回路诱导。3C 值归一化为蛋白, 锚点与包含β/HS 片段的片段之间的相互作用频率设置为零。误差线表示 SD n = 3。此数字已从摩根、孙燕姿等的补充图 10中修改. 9.请点击这里下载这个数字.
补充图 11: CLOuD9 构造不可逆转的关联, 以响应长期 ABA 治疗.immunoprecipitations 在 ABA 治疗10天后和10天的配体清除后, 证明了 CSA 和 CSP dCas9 蛋白的不可逆关联。此数字已从摩根、孙燕姿等的补充图 11中修改. 9.请点击这里下载这个数字.
补充表 1: gRNAs、qRT PCR、3C 和芯片 qPCR 的引物序列清单.本表已从摩根、孙燕姿等补充表 1中修改. 9.请点击这里下载此表格
讨论
CLOuD9 染色质循环的最关键步骤是: 1) 设计或使用正确的 gRNAs, 2) 每天变化的媒体 CLOuD9-transduced 细胞, 包括 aba 或亚砜, 3) 保持 ABA 的新鲜度, 和 4) 执行准确和仔细评估染色质构象。
CLOuD9 的极限主要存在于为选择的目标区域设计指南的能力。导 rna 执行的重要任务, 本地化的 dCas9 的组成部分, 以目标 DNA 区域是二聚和效果的指南是基于他们的具体目标地点。如果没有适当的 gRNA 组件, CLOuD9 系统将无法形成可逆的诱导回路。因此, 通过为每个感兴趣的区域设计多个参考线, 并将指南传播到 250-1000 bp 区域, 至少可以确保一个成功的指南。指南的位置也是准确的结果的组成部分。重要的是要避免在转录因子结合部位或其他关键区域的指南, 以防止背景影响, 如向上或向下调节转录。此外, CLOuD9 构造的精确位置可以轻微影响目标基因的转录。这强调了测试每个目标区域的多对指南的重要性, 以确定最健壮的对为实验目的。此外, 在每一对目标区域中, CSA 结构都应以 gRNAs 为目标, 而 CSP 构造应以 gRNAs 为靶向特异性化脓.
为了确保准确的结果和正确的二聚化, 在 CLOuD9 结构的转导后保持细胞环境的新鲜度也很重要。每天的媒体变化和新鲜的 dimerizer (或控制) 的添加, 确保互补的结构将保持在接近和保持改变的染色质构象。此外, 保证 ABA 是新鲜的, 并根据制造商的协议适当存储 (在6月内打开, 保持寒冷, 免受光照) 是取得真实结果的必要条件。
值得注意的是, CLOuD9 的 ABA dimerizer 与 ABI 和 PYL 二聚化蛋白一起使用, 而不是更普遍使用的 FRB 和 FKBP 系统。由于对癌细胞的毒性, FRB/FKBP 系统 rapalog 的必要性将限制 CLOuD9 的适用性。替代 ABI/PYL 系统规避了这一限制, 有效地使 CLOuD9 能够更广泛地利用。
总的来说, 我们开发了 CLOuD9, 一种独特和健壮的技术, 可以强行但可逆地建立远程目标基因组的联系人之间的接触。通过诱导染色质循环, 我们还表明, CLOuD9 可以用来修改基因表达在适当的细胞环境。这种技术的适应性允许对任何两个基因座之间的相互作用进行无限制的研究, 而不需要事先了解循环区域或循环机制。此外, CLOuD9's 独特的可逆性证明可以进一步检查疾病和发展的循环机制。虽然已经清楚地证明了染色质循环的靶向效应, 但仍有数据提供对非目标循环的影响以及随后对目标循环的影响的了解。
我们的数据仅说明了这一工具的一些应用, 但暗示了染色质排列是基因表达的主要基本思想。本技术可用于研究和揭示基因调控中染色质结构的细微差别, 从而提高了染色质折叠在基因转录中作用的整体理解。更好地理解转录动力学的细微之处, 可以引导癌症、遗传性疾病和先天性疾病的研究和治疗, 在这种情况下, 不同的染色质组装无疑会改变基因表达20, 21,22,23。随后利用 CLOuD9 技术的工作将进一步阐明染色质领域的排列和动态, 以及它们如何推动折叠, 以维持稳定的基因在发育和疾病中的表达。
披露声明
作者没有什么可透露的。
致谢
我们感谢 h. 昌, 破产, s. Tavazoie, 弗林, 巴蒂斯塔, e. 卡洛, 和整个王实验室的技术支持和关键阅读的手稿。S.L.M. 通过 NSFGRF (DGE-114747)、NDSEGF (FA9550-11-C-0028) 和国家癌症研究所 (1F99CA222541-01) 在这项工作中得到了支持。K.C.W. 是由巴斯威威基金的医学科学家的职业奖支持, 是唐纳德 E 和迪莉娅 b。
材料
| Name | Company | Catalog Number | Comments |
| RPMI 1640 media | Life Technologies | 11875-119 | For K562 cell culture |
| DMEM media | Life Technologies | 11995-065 | 1X, for 293T cell culture |
| lentiCRISPR v2 | Addgene plasmid | #52961 | For CLOuD9 plasmid development |
| pRSV-Rev | Addgene plasmid | #12253 | For lentivirus production |
| pMD2.G | Addgene plasmid | #12259 | For lentivirus production |
| pMDLg/pRRE | Addgene plasmid | #12251 | For lentivirus production |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668-019 | For lentivirus production |
| anti-HA antibody | Cell Signaling | 3724 | For immunoprecipitation |
| anti-Flag antibody | Sigma | F1804 | For immunoprecipitation |
| DNeasy Blood and Tissue Kit | Qiagen | 69504 | For DNA extraction |
| TRIzol | Life Technologies | 15596-018 | For RNA extraction |
| RNeasy Kit | Qiagen | 74106 | For RNA extraction |
| Superscript VILO | Life Technologies | 11754-050 | For cDNA |
| SYBR Green I MasterMix | Roche | 4707516001 | For qPCR analysis |
| Light Cycler 480II | Roche | For qPCR analysis | |
| anti-H3K4me3 antibody | AbCam | ab8580 | For ChIP-qPCR |
| anti-RNA Pol-II antibody | Active Motif | 61083 | For ChIP-qPCR |
| EDTA free protease inhibitor | Roche | 11873580001 | For protein extraction |
| 4-12% Tris Glycine gel | Biorad | Any size, For western blot | |
| anti-Rabbit HRP antibody | Santa Cruz | sc-2030 | For western blot |
| anti-mouse HRP antibody | Cell Signaling | 7076S | For western blot |
| K562 and H3K293 ChIP-Seq data | Encode | ENCSR000AKU | For ChIP-seq analysis |
| K562 and H3K293 ChIP-Seq data | Encode | ENCSR000APE | For ChIP-seq analysis |
| K562 and H3K293 ChIP-Seq data | Encode | ENCSR000FCJ | For ChIP-seq analysis |
| K562 and H3K293 ChIP-Seq data | GEO | GSM1479215 | For ChIP-seq analysis |
| Dynabeads Protein A for Immunoprecipitation | Thermo Fisher Scientific | 10001D | For immunoprecipitation |
| Dynabeads Protein G for Immunoprecipitation | Thermo Fisher Scientific | 10004D | For immunoprecipitation |
| RNA Clean & Concentrator-5 | Zymo Research | R1015 | For RNA purification |
| Pierce 16% Formaldehyde Methanol-free | Thermo Fisher Scientific | 28908 | For crosslinking |
| PX458 Plasmid | Addgene | 48138 | Suggested active Cas9 plasmid for gRNA cloning, but any active Cas9 plasmid will do |
| QIAquick PCR Purification Kit | Qiagen | 28104 | For PCR purification |
| FastDigest BsmBI | Thermo Fisher Scientific | FD0454 | For cloning guide RNAs |
| FastAP | Thermo Fisher Scientific | EF0651 | For cloning guide RNAs |
| 10X FastDigest Buffer | Thermo Fisher Scientific | B64 | For cloning guide RNAs |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | For cloning guide RNAs |
| 10X T4 Ligation Buffer | NEB | B0202S | For cloning guide RNAs |
| T4 PNK | NEB | M0201S | For cloning guide RNAs |
| 2X Quick Ligase Buffer | NEB | B2200S | For cloning guide RNAs |
| Quick Ligase | NEB | M2200S | For cloning guide RNAs |
| Buffers | |||
| Farnham lysis buffer | 1% Tris-Cl pH 8.0, 1% SDS, 1% protease inhibitor water solution (non-EDTA), and 1 mM EDTA in water | ||
| Modified RIPA buffer | 1% NP40/Igepal, 0.5% sodium deoxycholate, 0.1% SDS, 1 mM EDTA, and 1% protease inhibitor water solution (non-EDTA) in PBS pH 7.8 or 7.4 | ||
| IP dilution buffer | 0.01% SDS, 1.1% Triton-X 100, 1.2 mM EDTA, 16.7 mM Tris-HCl pH 8.0, 167 mM NaCl, 0.1x protease inhibitor | ||
| Wash buffer | 100 mM Tris pH 9, 100 mM LiCl, 1% NP-40, and 1% sodium deoxycholate | ||
| Swelling buffer | 0.1 M Tris pH 7.5, 10 mM potassium acetate, 15 mM magnesium acetate, 1% NP-40 | ||
| Dilution buffer | 0.01% SDS, 1.1% Triton X-100, 1.2 mM EDTA, 16.7 mM Tris pH 8 and 167 mM NaCl | ||
| IP elution buffer | 1% SDS, 10% NaHCO3 |
参考文献
- Krivega, I., Dean, A. Chromatin looping as a target for altering erythroid gene expression. Ann. N.Y. Acad. Sci. 1368, 31-39 (2016).
- Matharu, N., Ahituv, N. Minor loops in major folds: Enhancer-promoter looping, chromatin restructuring, and their association with transcriptional regulation and disease. PLoS Genet. 11, e1005640 (2015).
- Dekker, J., Martí-Renom, M. A., Mirny, L. A. Exploring the three-dimensional organization of genomes: interpreting chromatin interaction data. Nat. Rev Genet. 14, 390-403 (2013).
- Kim, A., Dean, A. Chromatin loop formation in the b-globin locus and its role in globin gene transcription. Mol Cells. 34, 1-5 (2012).
- Petrascheck, M., et al. DNA looping induced by a transcriptional enhancer in vivo. Nucleic Acids Res. 33, 3743-3750 (2005).
- Ameres, S. L., et al. Inducible DNA-loop formation blocks transcriptional activation by an SV40 enhancer. EMBO J. 24, 358-367 (2005).
- Deng, W., et al. Controlling Long-Range Genomic Interactions at a Native Locus by Targeted Tethering of a Looping Factor. Cell. 149, 1233-1244 (2012).
- Deng, W., et al. Reactivation of developmentally silenced globin genes by forced chromatin looping. Cell. 158, 849-860 (2014).
- Morgan, S. L., et al. Manipulation of Nuclear Architecture through CRISPR-Mediated Chromosomal Looping. Nature Communications. 8, (2017).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biol. 17 (1), 148 (2016).
- . . Lipofectamine 2000 Reagent. , (2013).
- . . DNeasy Blood & Tissue Kit Handbook. , (2006).
- . . Determining Genome Targeting Efficiency using T7 Endonuclase I (M0302). , (2016).
- Guschin, D. Y., et al. A rapid and general assay for monitoring endogenous gene modification. Methods Mol Biol. 649, 247-256 (2010).
- Shalem, O., Sanjana, N. E., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. , 83-87 (2014).
- . . RNeasy Mini Handbook. , (2012).
- . . SuperScript VILO cDNA Synthesis Product Information Sheet. , (2015).
- Theunissen, T. W., et al. Systematic identification of culture conditions for induction and maintenance of naive human pluripotency. Cell Stem Cell. 15, 471-487 (2014).
- Mokry, M., et al. Integrated genome-wide analysis of transcription factor occupancy, RNA polymerase II binding and steady state RNA levels identify differentially regulated functional gene classes. Nucleic Acids Res. 40, 148-158 (2012).
- Drier, Y., et al. An oncogenic MYB feedback loop drives alternate cell fates in adenoid cystic carcinoma. Nat Genet. 48, 265-272 (2016).
- Ryan, R. J. H., et al. Detection of enhancer-associated rearrangements reveals mechanisms of oncogene dysregulation in B-cell lymphoma. Cancer Discov. 5, 1058-1071 (2015).
- Montavon, T., et al. A regulatory archipelago controls Hox genes transcription in digits. Cell. 147, 1132-1145 (2011).
- Lupianez, D. G., et al. Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions. Cell. 161, 1012-1025 (2015).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。