Method Article
CRISPR-vermittelten Reorganisation der Chromatinstruktur mit Schleife
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Chromatin looping spielt eine bedeutende Rolle in der Genregulation; Allerdings gab es keine technologische Fortschritte, die eine selektive und reversible Änderung des Chromatins Schleifen ermöglichen. Hier beschreiben wir ein leistungsfähiges System für Chromatin Schleife Reorganisation Loci gezielt mit CRISPR-dCas9 (CLOuD9) gezeigt, um selektiv und reversibel Genexpression zu modulieren.
Zusammenfassung
Jüngste Studien haben deutlich gezeigt, dass Langstrecken, dreidimensionale Chromatin looping Interaktionen spielen eine wichtige Rolle bei der Regulation der Genexpression, aber ob die Schleife für verantwortlich ist oder ein Ergebnis von Veränderungen in der Genexpression nach wie vor ist unbekannt. Bis vor kurzem wie Chromatin Schleifen die Regulierung der Genaktivität und zelluläre Funktion beeinflusst wurde relativ unklar und Einschränkungen in vorhandenen Methoden, diese Strukturen zu manipulieren verhindert eingehende Erforschung dieser Interaktionen. Um diese Unsicherheit zu beseitigen, wir entwickelten eine Methode für die selektive und reversible Chromatin Schleife Re-Organisation mit CRISPR-dCas9 (CLOuD9). Die Dynamik des Systems CLOuD9 wurde durch erfolgreiche Lokalisierung von CLOuD9 Konstrukte genomic Loci um lokale Chromatin Konformation modulieren Ziel nachgewiesen. Wichtig ist, ist die Fähigkeit zum Umkehren des induzierten Kontakts und Wiederherstellen der endogenen Chromatin-Konformation auch bestätigt worden. Modulation der Genexpression mit dieser Methode stellt die Fähigkeit zur zellulären Genexpression regulieren und unterstreicht das große Potenzial für Anwendungen dieser Technologie bei der Schaffung stabiler de Novo Chromatin-Schleifen, die deutlich gen beeinflussen Ausdruck im Kontext von Krebs und Entwicklung.
Einleitung
Die Beziehung zwischen Chromatin Falten in den Zellkern und die spezifische Organisation des Genoms hat großes Interesse in den letzten Jahren sammelte, wie sich gezeigt hat, eng verbunden mit der Gen-Expression-1,-2sein. Während die genaue Beziehung zwischen Genaktivität und Modulation der Chromatinstruktur unklar bleibt, hat wurde vermutet, dass die Wechselwirkungen zwischen chromosomalen Kontakte durch dynamische dreidimensionale Chromatin Organisation dienen ein gen regulatorische Funktion3. In der Tat, solche Wirkung auch auf die menschliche Globin-gen-Locus, nachgewiesen wo der Locus Kontrollregion (LCR) die Aktivität der Globin-Gene Entwicklungsgeschichtlich gezielt reguliert durch die Schaffung einer Chromatin-Schleife zwischen den beiden Regionen4. Jedoch in dieser und anderen Regionen ist es unklar, ob eine Ursache oder Folge von Veränderungen in der Genexpression Chromatin looping ist.
Bis jetzt blieb die Herausforderungen bei der Untersuchung dieses Phänomens. Andere Versuche zur Induktion Chromatin Schleifen beteiligt z. B. Veränderung der linearen DNA-Sequenz oder komplizierte Verfahren erfordern eine Fülle von Hintergrundwissen über bestimmte Elemente, die Schleife5,6zu erleichtern, 7,8. Während bisherige Arbeit hat vorgeschlagen, dass das Chromatin Fahrt gen-Expression in eine spezifische und eingeschränkten Rahmen7,Schleifen ist8, das Niveau an welchem Chromatin looping Transkription Global wirkt sich darüber hinaus ungewiss. Obwohl Interesse an die Auswirkungen der Langstrecken Schleifen auf die Genexpression in den letzten Jahren kontinuierlich gewachsen ist, bestehen unbeantwortete Fragen über den Aufbau und die Beibehaltung von Chromatin Kontakte Genaktivität ändern.
Die Technologie, die wir entwickelt haben beschäftigt die Nuklease mangelhaft gruppierten regelmäßig dazwischen kurze palindromische Wiederholungen (CRISPR) - CRISPR - associated Protein 9 (dCas9), um alle genomic Loci9breit anwendbar gezielt zu ermöglichen. Diese Technologie eliminiert die komplexen Fragen im Zusammenhang mit Änderungen der linearen DNA-Sequenz und ist ohne wesentliche Vorkenntnisse bestimmter Schleife Komponenten zu erreichen. Vor allem ist das Tool universal und breit anwendbar auf Chromatin Loops in Entwicklung sowie in einer Vielzahl von Krankheiten, wie Krebs erkannt. Die Macht der CLOuD9 belegen reversibel verändern die Struktur der Schleifen Genexpression effektiv zu modulieren.
Protokoll
1. gRNA Design
- Wählen Sie ein standard gRNA-Design-Tool, den Guide RNAs10zu entwerfen. Komplementäre Paare der zuvor entwickelten CLOuD9 Konstrukte9 verwenden (d. h. mindestens 1 S. Aureus (CSA) und 1 S. Pyogenes (CSP) konstruieren) für jedes Experiment.
- Innerhalb der ausgewählten Online-Design-Tool, verwenden die Protospacer angrenzenden Motiv (PAM) Sequenz "NGG" mit einer Anleitung Länge von 20 bp für CSP und die PAM-Sequenz "NNGRRT" mit einer Anleitung Länge von 21 für CSA.
- Wählen Sie Führer anhand der Spezifität der Gäste und design Guides auf beiden Strängen, Chancen auf eine funktionierende Anleitung zu maximieren.
- Bestellen Sie 2 Oligonukleotide pro Guide von einem Oligo-Hersteller: für die vorwärts Oligo, fügen Sie "Caccg", 5'-Ende der Anleitung Folge, und für die umgekehrte Oligo, hinzufügen "Aaac", die 5'-Ende und "c" am 3'-Ende vor der Bestellung.
- Entwerfen Sie zunächst 3-5 gRNAs für die einzelnen Regionen von Interesse, über eine 250-1000 bp Region verteilt. Die gRNA Ausrichtung auf Effizienz wie folgt zu bewerten:
- Klon identifiziert gRNAs in eine aktive Cas9 Plasmid (siehe Schritt 3) und vorübergehend transfizieren in 293T Zellen (siehe Schritt 4)11.
- Wachsen Sie Zellen für 2-3-d in Dulbeccos geändert Eagle Medium (DMEM) mit 10 % fetalen bovine Serum (FBS) und 1 % Penicillin-Streptomycin (Stift Strep), dann ernten Sie und extrahieren Sie genomische DNA12zu.
- Verstärken Sie Zielregionen durch Polymerase-Kettenreaktion (PCR). Führen Sie Produkte auf einem 1,5 % Agarosegel und reinigen Sie entsprechenden Bands zu.
- Einen T7 Endonuklease Assay durchführen, wie beschrieben in der Guschin Et Al. Protokoll Nr.13,14.
Hinweis: Effiziente Führer sind die DNA an den Zielstandort schneiden will.
2. die Zellkultur
- Zellen in ein gesundes und aktiv Teilen Zustand zu erhalten.
- Für K562s, Kultur in Roswell Park Memorial Institute (RPMI) 1640 Medien mit 10 % FBS und 1 % Stift strep.
- Wachsen Sie K562s in einem 25 cm2 abgeschrägten Hals Kolben für Wartung und passen Sie die Zelldichte auf 400.000 Zellen pro mL pro Tag.
- Nachdem K562s mit ausgestrahlt wurde haben CLOuD9 konstruiert, Wartung Medien 2 µg/mL Puromycin und 100 µg/mL Hygromycin hinzufügen.
- Für 293Ts, Kultur in Dulbeccos geändert Eagle Medium (DMEM) mit 10 % FBS und 1 % Stift strep.
- 293Ts in 10 cm2 Platten wachsen und beim konfluierende aufgeteilt.
- Für K562s, Kultur in Roswell Park Memorial Institute (RPMI) 1640 Medien mit 10 % FBS und 1 % Stift strep.
(3) Plasmid Vorbereitung und gRNA Insertion15
Hinweis: Plasmid-Karten sind erhältlich im Anhang und Primer verwendet in der Beispiel-Experimente gibt es in ergänzenden Tabelle1.
- Mix 5 µg des Lentivirale CRISPR-Plasmids mit 3 µL BsmBI, 3 µL alkalische Phosphatase, 6 µL 10-fach Puffer und 0,6 µL frisch zubereitete 100 mm DTT. Das Gesamtvolumen 60 µL mit DdH2O bringen und Inkubation bei 37 ° C für 30 min zu verdauen und dephosphorylate das Plasmid.
- Gel reinigen das verdaute Plasmid und eluieren in DdH2O.
- Mix 1 µL jedes der gekoppelten Handbücher bei 100 µM mit 1 µL 10 x T4 Ligation Puffer, 6,5 µL DdH2O und 0,5 µL T4 PNK, 10 µL Gesamtvolumen zu erreichen. Inkubation bei 37 ° C für 30 min, dann 95 ° C für 5 min, dann auf 25 ° C bei 5 ° C/min Rampe.
Hinweis: Dies wird das Paar Guides Tempern. - Verdünnen Sie die geglühten Führer (aus Schritt 3.3) 1: 200 in DdH2O.
- Mix 1 µL des verdauten Plasmids aus Schritt 3.2 mit 0,5 µL verdünnter aufgespaltenen Guides aus Schritt 3.4, 2,5 µL 2 x Puffer, 1 µl DdH2O und 0,5 µL Ligase, 5,5 µL Gesamt Reaktion zu machen. Inkubieren Sie dies bei Raumtemperatur für 10 min in die Führungen auf dem Plasmid BsmBI verdaut zu verbinden.
- Neu aufgespaltenen Plasmid aus Schritt 3.5 in Stbl3 Bakterien verwandeln und mit einer beliebigen Plasmid Vorbereitung Methode zu verstärken.
4. Lentivirus Produktion
- Graf 750.000 293T Zellen pro Bohrloch für ein sechs-Platte mit einem Hemocytometer und Säen sie in DMEM mit 10 % FBS und 1 % Stift strep. Verwenden Sie einen Brunnen für jedes Konstrukt.
- 24 h nach der Aussaat, ändern Sie Medien in frischen antibiotikafreien DMEM mit 10 % FBS.
- Verdünnen Sie in einem Rohr 11 µL Lipid-basierte Transfection Reagens mit 150 µL des OptiMEM Mediums pro Reaktion11.
- In einem separaten Rohr 2 µg des Plasmids CLOuD9 Lentivirale Vektor aus Schritt 3.6, 0,35 µg pMDLg/pRRE, 1 µg pRSV-Rev und 0,65 µg des pMD2.G mit 150 µL des OptiMEM Mediums pro Reaktion11zu verdünnen.
- Für jede Reaktion verdünnter Lipid-basierte Transfection Reagens im Verhältnis 1:1 verdünnten Lentivirale Plasmid-Mischungen hinzu, und inkubieren Sie 5 min11.
- Fügen Sie das gesamte Volumen der jeder gemischten Komplex aus Schritt 4.5 in die jeweiligen Vertiefungen der antibiotikafreien 293T Zellen aus Schritt 4.2.
- 48 h sammeln durch pipettieren in ein frisches Tubus und Spin down bei 300 X g für 5 min später, virale Produktion Medien. Nach Zentrifugation bewegen des Überstands zu einem frischen Schlauch, verbleibende Zelle Ablagerungen zu entfernen.
- Virale Produktion Media für die Transduktion von Zielzellen sofort verwenden oder Einfrieren bei-80 ° C für eine spätere Verwendung.
5. Lentivirus Transduktion von Zellen
- Eine 15 mL konische Röhrchen mit 80.000 Zellen für die Anwendung von CLOuD9 fügen Sie 250 µL jedes virale Konstrukt von Interesse (bestehend aus komplementären CLOuD9 Plasmide hinzu).
- Bringen Sie das Gesamtvolumen der Medien in jedem kegelförmig bis 1 mL mit Antibiotika-freie Medien, und fügen Sie Polybrene, eine Endkonzentration von zwischen 1 bis 8 µg/mL (die maximale Konzentration des Polybrene toleriert durch die Zelle Art genutzt werden müssen experimentell ermittelt werden).
- Spin-Zellen bei 800 X g für 30 min bei Raumtemperatur, dann durch pipettieren ohne Entfernen des viralen Überstands aufzuwirbeln. Verschieben Sie die gesamte Zellsuspension auf eine Zellplatte Kultur.
- 24 h nach Transduktion, Spin-Zellen nach unten bei 300 X g für 5 min bei Raumtemperatur.
- Aspirieren Sie virale überstand und aufzuwirbeln Sie Zellen in regelmäßigen Zellkulturmedien.
- Am nächsten Tag fügen Sie Puromycin (1 µg/mL für 293T Zellen) und Hygromycin (25 µg/mL für 293T Zellen hinzu) Zellkulturmedium für doppelt ausgestrahlt Zellen auswählen. Experimentell bestimmen Sie die entsprechenden Konzentrationen von Puromycin und Hygromycin für einen bestimmten Zelltyp vor Beginn der Experimente.
- Zellen im Auswahl-Media für mindestens 3 d vor alle nachgelagerten Experimente und halten im Auswahl-Media für die Dauer aller Experimente.
6. Zelle Dimerisierung und Auswaschen
- Fügen Sie 1 mM Abscisic Säure (ABA) oder gleichwertigem Umfang von Dimethyl Sulfoxid (DMSO) für Steuerelemente, Zellkultur, die CLOuD9 Dimere ausgewählt und Zellen ausgestrahlt. Verwenden Sie ABA innerhalb von 6 Monaten ab Datum des Eingangs, halten Sie kalt und schützen Sie es vor Licht während der Benutzung. 2 mM ABA kann genutzt werden, wenn nötig.
- Medien täglich frische ABA (oder DMSO für Steuerelemente) für die Dauer des Experiments zu ändern.
Hinweis: Keine Begrenzung auf der Länge der Dimerisierung ist noch nicht beobachtet worden.
- Medien täglich frische ABA (oder DMSO für Steuerelemente) für die Dauer des Experiments zu ändern.
- Umgekehrter Dimerisierung durch die Beseitigung von Medien mit ABA und waschen Zellen mit ausreichend Phosphat gepufferte Kochsalzlösung (PBS) auf die Oberfläche der Platte, zweimal decken. Danach Zellkulturen in ABA-freie Medien zu einen un-dimerized Zustand zu halten.
(7) Immunopräzipitation und Co-Immunperoxidase
Hinweis: Stellen Sie alle Puffer frisch und unmittelbar vor der Verwendung.
- Nach Abschluss der Dimerisierung Experimente, sammeln und spin-down Zellen, dann Aspirieren überstand.
- Crosslink und Einfrieren Zellen
- Machen Sie einen frischen Vorrat an 1 % Formaldehyd in PBS bei Raumtemperatur mit frischen Materialien. Aufschwemmen Sie für jede 2 Millionen Zellen sanft mit 1 mL 1 % Formaldehyd bei Raumtemperatur für genau 10 min beim Drehen.
Hinweis: Verwenden Sie ein Mindestvolumen von 10 mL für Vernetzung weniger als 10 Millionen Zellen. - Vernetzung mit dem Zusatz von Glycin, eine Endkonzentration von 0,125 M, gefolgt von Inkubation für 5 min bei Raumtemperatur während der Drehung zu stillen.
- Spin-down Zellen und 1-2 X mit eiskaltem PBS waschen. Zelle Pellets können dann Snap in flüssigem Stickstoff eingefroren und bei-80 ° C gelagert oder sofort verwendet werden.
Hinweis: Wärme wird Formaldehyd Querverbindungen umzukehren. Wenn gehetzt, können Zellen direkt bei-80 ° C gespeichert werden, ohne die Snap einfrieren.
- Machen Sie einen frischen Vorrat an 1 % Formaldehyd in PBS bei Raumtemperatur mit frischen Materialien. Aufschwemmen Sie für jede 2 Millionen Zellen sanft mit 1 mL 1 % Formaldehyd bei Raumtemperatur für genau 10 min beim Drehen.
- Antikörper/Wulst-Konjugat vorbereiten
Hinweis: Optimierung Bedingungen für ausgewählte Antikörper (z. B. Testen verschiedene Lyse Puffer für Zelle Lysis und Durchführung IP) können sich lohnen. Zwei gemeinsame Puffer, Farnham Lyse Puffer und modifiziert RIPA Puffer, können erhebliche Auswirkungen auf die IP und Beschallung Effizienz haben. Alle Puffer Kompositionen finden Sie in der Tabelle der Materialien. Darüber hinaus denken Sie daran, dass Chromatin Beschallung kann mehrere Stunden in Anspruch nehmen.- Aufschwemmen Sie Perlen durch Vortexen kurz nach Bedarf für den gewählten Antikörper.
- Übertragen Sie einen angemessenen Betrag von Perlen für das Experiment auf eine 1,5 mL-Tube. Als Ausgangspunkt verwenden Sie 20 µL Perlen für jeden 1 µg des Antikörpers. Fügen Sie noch keine Antikörper.
Hinweis: Verwenden Sie 10 µg Antikörper mit 200 µL Perlen für 1 mg insgesamt lysate als Ausgangspunkt, es sei denn, es bekannt ist, dass der Antikörper mehr oder weniger effizient ist, als würde dies bedeuten. Für niedrige Fülle Proteine kann dieses Verhältnis angepasst werden müssen. - Wenn Farnham Lyse Puffer verwenden, waschen Sie 3 X in IP-Verdünnungspuffer. Wenn RIPA Lyse Puffer verwenden, waschen Sie 3 X im RIPA Lyse Puffer.
- Aufschwemmen Perlen in einem Endvolumen des gleichen Puffers ab Schritt 7.3.3 (IP-Verdünnung oder RIPA-Puffer), die > 1 X und < 5 x des Ausgangsvolumens. d. h. Für 100 µL erste Perle Volumen zwischen 100 und 500 µL endgültige Wiederfreisetzung Volume verwenden.
- Gewaschene Perlen jetzt an eine geeignete Konzentration Antikörper hinzufügen. Für eine gute HA oder Flag Antikörper für IP-CLOuD9 Konstrukte verwenden Antikörper bei 01:50 (µg Protein: µg Protein lysate) und inkubieren Sie zwischen 8 + h und über Nacht bei 4 ° C, während der Drehung. Alternativ 2-4 h bei Raumtemperatur inkubieren.
- Den überstand von Perlen entfernen und entsorgen.
- Wash Perlen 3 X mit der Waschpuffer.
- Aufschwemmen Sie in einem minimalen Volumen des Puffers für die Übernachtung Inkubation mit dem Antikörper (IP-Verdünnung oder RIPA Puffer) genutzt. Halten Sie Kälte bis mit Protein lysate kombiniert.
- Beschallen Chromatin
- Lösen Sie zuerst Zellen mit dem Zusatz von Schwellungen Puffer zu Zelle Pellets für 10 min auf Eis, schnippen Zellen alle paar Minuten absetzen zu verhindern.
Hinweis: In der Regel 500 µL Puffer Schwellung ist hinzugefügt, um 25 Millionen Zellen und skaliert von dort, aber tun nicht Nutzung von weniger als 500 µL Puffer für < 25 Millionen Zellen. - Dounce manuell im Uhrzeigersinn und gegen den Uhrzeigersinn, 13 x jeweils.
- Spin-Kerne, bei 4 ° C für 5 min bei 1.500 x g. überstand vollständig abzusaugen und wiederholen, um verbleibende Überstand entfernen dann wiegen die Zelle Pellet in mg.
- Kerne Lyse Puffer Protease-Inhibitor hinzufügen (Farnham Puffer oder RIPA Puffer oben, wählen Sie eine).
- Gebeizte Kerne zu Aufschwemmen 10 X Anzahl der Kerne Lyse Puffer hinzufügen (z. B. hinzufügen 1 mL der Kerne Lyse Puffer ein 0,100 g Granulat). Kurz Wirbel und lösen Sie für 10 min auf Eis.
Hinweis: Denken Sie daran die Größe des Rohres Beschallung. Ideale Lautstärke ist oft < 1 mL, so Proben müssen aufgeteilt werden, wenn Pellets sehr groß sind.- Beschallen Sie für Co-IP Kerne kurz zu solubilisieren Material und stillen SDS auf ein Niveau, das Immunopräzipitation mit 2-3 Bände der Verdünnungspuffer erlaubt, dann fahren Sie mit Schritt 7.4.6. Fügen Sie für sehr empfindliche Antikörper mehr Verdünnungspuffer zur weiteren Reduzierung die Konzentration von SDS hinzu.
Hinweis: Stop Beschallung bei lysate löscht; Es ändert sich von einer opak/transluzent weiß, völlig transparent. Proben so kühl wie möglich halten und nicht über beschallen. - Für ChIP gründlich Beschallen Sie DNA 150-1000 bp DNA-Fragmente zu, dann fahren Sie mit Schritt 7.4.6.
- Beschallen Sie für Co-IP Kerne kurz zu solubilisieren Material und stillen SDS auf ein Niveau, das Immunopräzipitation mit 2-3 Bände der Verdünnungspuffer erlaubt, dann fahren Sie mit Schritt 7.4.6. Fügen Sie für sehr empfindliche Antikörper mehr Verdünnungspuffer zur weiteren Reduzierung die Konzentration von SDS hinzu.
- Ausgliederung von unlöslichen Material mit maximaler Geschwindigkeit für 10 min bei 4 ° C.
- Übertragen Sie lösliche Material auf einem frischen Rohr.
- Messen Sie für Co-IP die Konzentration des Proteins und fahren Sie mit dem Pulldown in Schritt 7,5.
- Für ChIP, Entfernen einer 10 µL Aliquot der lysate gelöscht, und fügen Sie 40 µL IP-Elution Buffer und 6 µL 5 M NaCl, für eine Gesamtmenge von 56ul. Kochen Sie für 15 min bei 95 ° C, 5-10 µL 3 M NaOAc pH 5 hinzufügen und Aufräumen auf einer PCR-Reinigung-Spalte. Messen die Konzentration von DNA durch die Messung der Absorption bei 260 nm und über Proben zu standardisieren. Fahren Sie dann mit Pulldown in Schritt 7,5.
Hinweis: Extra lysate Snap bei-80 ° C eingefroren und verwendet werden kann zu einem späteren Zeitpunkt, aber die besten Ergebnisse aus den frischen lysate. Wenn das Einfrieren, nicht Einfrieren/Auftauen lysate mehr als einmal.
- Lösen Sie zuerst Zellen mit dem Zusatz von Schwellungen Puffer zu Zelle Pellets für 10 min auf Eis, schnippen Zellen alle paar Minuten absetzen zu verhindern.
- Pulldown
- Übertragen Sie eine entsprechende Menge an Protein lysate auf ein frisches Rohr. Verdünnen Sie Farnham Lyse Puffer verwenden, 2,1 Mengen an IP-Verdünnungspuffer (siehe oben).
- 100 µL des Überstandes für Eingabesteuerelement beiseite, und bis zum nächsten Tag bei 4 ° C lagern.
- Perle/Antikörper Konjugat aus Schritt 7.3.8 Proben jetzt hinzufügen.
- Drehen Sie Proben bei 4 ° C über Nacht und sicherzustellen Sie, dass genügend Volumen für flüssige Bewegung in 1,5 mL-Tuben.
- Waschen und eluieren
- Nach der Übernachtung Drehung speichern Sie überstand als unverbindliche Bruchteil. Dann Perlen Waschen 3-5 X in Verdünnungspuffer IP.
- IP-Elution Puffer bei Raumtemperatur ohne Inhibitoren vorzubereiten.
- Eluieren komplexe mit 50 µL Elution Buffer auf einen Wirbel bei 67 ° C für 15 min. Transfer Elution zu einem neuen Schlauch erschüttert und wiederholen Sie dies mit einem anderen 50 µL. kombinieren sie beide für eine insgesamt 100 µL Elution.
Hinweis: Wenn es zu viel Hintergrund von diesem Elution Verfahren gibt, Hitze verringert oder beseitigt während schütteln/Inkubation. Dies verringert den Ertrag des Zielproteins in der Regel aber verringert deutlich den Hintergrund.
- Reverse Querverbindungen durch Erhitzen bei 67 ° C > 4 h.
Hinweis: Dies ist nicht unbedingt notwendig für Co IPs aber kann hilfreich sein.- Führen Sie für Co-IP volle Dimerisierung zwischen den Zielen des Interesses, um sicherzustellen Eluate auf einem SDS-Page Gel und Sonde mit Antikörpern gegen den HA-Tag oder Tag der Flagge, wie angegeben, auf 1: 1.000. Fahren Sie mit Schritt 8.
- Für ChIP-qPCR um korrekte Lokalisierung und Ausrichtung der einzelnen CRISPR-dCas9 Komponenten zu gewährleisten, reinigen Sie Elution Produkt auf einer Säule und eluieren in 10 µL. Perform in Echtzeit quantitative Polymerase-Kettenreaktion (qPCR) gereinigte DNA. Primer verwendet in den dargestellten Daten stehen in ergänzenden Tabelle1. Fahren Sie mit Schritt 8.
(8) RNA-Extraktion und Quantitative PCR
- Um die CLOuD9-induzierten Veränderungen in der Genexpression zu untersuchen, zunächst zu isolieren und Gesamt-RNS von Kontrolle Zelle Pellets und dimerized Zelle Pellets zu reinigen.
- Komplementäre DNA (cDNA) aus der gereinigten RNA durch reverse Transkription17 machen und qPCR Analysen durchzuführen. Primer sind in ergänzenden Tabelle1zur Verfügung.
9. Chromosom Konformation Capture-Assay
- Änderungen in der Häufigkeit der genomic Loci Kontakte induziert durch CLOuD9 beobachten, Chromosom Konformation erfassen (3C) führen zu Proben8.
- 3C Ligatur Produkte in zwei Sätzen von Duplikaten für jede der drei biologische Wiederholungen durch quantitative Real-Time PCR zu quantifizieren. Normalisieren Sie Proben auf die 3C-Signale von den Tubulin-Locus. Primer sind in ergänzenden Tabelle1zur Verfügung.
Ergebnisse
CLOuD9 induziert reversible β-Globin-Promoter-LCR looping. Angemessene Verwendung von CLOuD9 System induziert reversible Kontakt des ergänzenden CSA und CSP CLOuD9 konstruiert durch Hinzufügen oder Entfernen von ABA, Zellkulturmedien (Abbildung 1a). CSA und CSP-Konstrukte (Abbildung 1 b) sind entsprechende genomische Regionen mit standard CRISPR gRNAs lokalisiert. In Anbetracht der riesigen Dokumentation der menschliche Globin Locus sowie häufige chromosomale Falten und Neuordnung, die es während der Entwicklung auftritt, wurde diese Region gewählt, um das Dienstprogramm des CLOuD9 Systems zu demonstrieren. Darüber hinaus wurde die Zellinie K562 ausgewählt, weil sich gezeigt hat, konsequent hohe Niveaus des fetalen γ-Globin-Gens, im Gegensatz zu den β-Globin-gen zum Ausdruck bringen, die in der Regel in den gesunden Erwachsenen erythroiden Linie Zellen ausgedrückt wird. Mithilfe der K562-Zellen kann die Möglichkeit von CLOuD9, Genexpression verändern untersucht werden, indem er versucht, die Ausdruck des β-Globin-Gens bei dieser Zelllinie wiederherzustellen.
Vor der Induktion von Dimerisierung, Chromatin Immunopräzipitation-quantitative PCR (ChIP-qPCR) eingesetzt wurde, um die genaue Lokalisierung und Ausrichtung der einzelnen CLOuD9 Komponenten zu gewährleisten (ergänzende Abbildung1). Darüber hinaus überprüft co-Immunopräzipitation (Co-IP) mit und ohne ABA CSA und CSP Dimerisierung im Beisein von Liganden sowie Reversibilität in der Abwesenheit des Liganden (Abbildung 1 c und ergänzende Abbildung2). 24 h nach dem Hinzufügen von ABA, besseren Kontakt zwischen β-Globin und LCR erschien, gemessen an Chromosom Konformation erfassen (3 C) in den Zellen mit beiden Dimerisierung teilen, aber nicht die Steuerelemente, die mit nur zwei CSA oder CSP Konstrukte, damit die Validierung der Spezifität der Chromatin-Änderung für die Ziel-Websites (Abbildung 1 c und ergänzende Angaben 3,4). Die LCR-β-Globin-Interaktion erstellen nicht vollständig eliminieren die endogenen LCR-Globin-Kontakt, aber stattdessen an den ursprünglichen Kontakt als bisher gemeldeten8hinzugefügt. Anstieg der β-Globin/LCR Kontakte wurden bis zu 72 h Dimerisierung, unabhängig von der genauen Region innerhalb der gezielten LCR und β-Globin Promoter-Region (Ergänzende Figuren 5,6) beobachtet. Zu guter Letzt wurde die Reversibilität des Systems mit 3C bestätigt, nach dem Entfernen der ABA, die eine vollständige Erneuerung der endogenen Konformation (Abbildung 1 d und ergänzende Abbildungen 3-6) zeigte.
Wir hielten der Erfolg gezeigt in den K562-Zellen möglicherweise ein Ergebnis des Standortes Globin Locus in einer Region von Euchromatin (Abbildung 1 d), also eine zweite Zelllinie verwendet wurde, um diese Idee zu erkunden. CLOuD9 System war HEK 293T Zellen in Regionen zugewiesen, die heterochromatischen und Globin-Gene (Abbildung 1e) nicht ausdrücklich. Das Ergebnis war ähnlich wie in K562s beobachtet wurde; mehr β-Globin LCR Verbände waren gemessen an 3C nach 24 Stunden mit ABA (Abb. 1e und ergänzende Abbildung 3), den Nachweis für CLOuD9 robuste Funktionsfähigkeit in verschiedenen zellulären Umgebungen, trotz den Originalzustand Chromatin oder Konformation.
Weitere Loci wurden getestet, um die breite Anwendbarkeit von CLOuD9, einschließlich der Oct4-Veranstalter und eine distale 5' Enhancer in den 293T Zellen zu gewährleisten. Bisher gab es keine nachweisbaren Oct4 Ausdruck in dieser Zelllinie und darüber hinaus keine endogenen Kontakte beschrieben. Nachweis der Oct4 Ausdruck in embryonale Stammzellen, die aus Kontakt mit dem distalen 5' Enhancer motiviert dieses Experiment, und das gleiche Ergebnis wurde an der β-Globin Locus18beobachtet. Kontakt zwischen dem Oct4 distalen Enhancer und Förderer wurde in die Zellen CLOuD9 aktiviert, aber nicht der Kontrollzellen (Abb. 1f) identifiziert. Darüber hinaus wurde festgestellt, dass der Oct4 Projektträger und der distalen 5' Enhancer Interaktion auch, eine 3'-Enhancer aufgefordert, der Oct4-Veranstalter zu kontaktieren. Diese Veranstaltung steht im Einklang mit Beweise dafür, dass die 3'-Enhancer mit dem Oct4 Promotor/5 interagiert "während der endogenen gen Aktivierung10komplexe distale Enhancer.
CLOuD9 induziert Kontext spezifische Veränderungen im Gen-Loci. Nachdem Sie bestätigt haben, dass das CLOuD9-System in der Tat chromosomalen Kontakte auf Gen-Loci induzieren, haben wir versucht, die Schleifen-Effekt auf die Genexpression zu untersuchen. Es wurde dokumentiert, dass Transkription für die Globin und Oct4 Gene sind die Kontakte zwischen der LCR und Globin gen-Loci und der distalen 5' Enhancer und Oct4 Förderer, bzw.1,11abhängig. So, wir die Hypothese aufgestellt, dass mit der CLOuD9 überzeugende Genexpression System Laufwerk Chromatin Schleifenbildung in jeder dieser Regionen führen würde.
In beiden Loci RT-qPCR zeigte, dass ABA induziert Chromatin Schleifen fuhr Anstieg Oct4 Ausdruck in den 293T Zellen und in β-Globin Ausdruck in K562-Zellen, wenn auch nicht in 293Ts (Abbildung 2a). Obwohl die Zugabe von ABA zu Zell-Kultur für weniger als 24 h erhöhte β-Globin Ausdruck deutlich Ausdruck weiter an stetig bis zu 72 Stunden und auf ABA Auswaschen (Abbildung 2a) reversibel war. Alle K562 Zellen mit Ausnahme der Steuerelemente folgten diesem Trend, egal wo die Dimerisierung Komponenten in den LCR und β-Globin-Promoter-Regionen (Abbildung 2a und ergänzende Zahlen 7,8) befanden. Zur Unterstützung dieser Erkenntnisse entsprach ChIP-qPCR H3K4me3 und RNA Pol-II in der β-Globin Locus in K562s und 293Ts mit beobachteten Veränderungen in der Transkription (Abbildung 2 c-f).
CLOuD9 schafft stabile Chromatin Schleifen. Obwohl kurzfristig Schleife Induktion mit CLOuD9 offenbar Erwartungen, gefolgt ob langfristige Induktion von looping differenzielle Auswirkungen gehabt blieb um zu beachten. Um dies zu untersuchen, wurden die Zellen in Anwesenheit von ABA für 10 Tage kultiviert. Während K562s und 293Ts höhere Kontaktfrequenz zwischen der β-Globin Locus und die LCR im Vergleich zu Kontrollen (Abbildung 3a, b und Ergänzende Figuren 9,10) ausgestellt, wurden Änderungen im β-Globin Ausdruck nur noch beobachtet. in K562-Zellen (Abb. 3 c). Interessant ist, aber es wurde beobachtet, dass langfristige Dimerisierung in K562s, wo Transkription stark hochreguliert war, war nicht mehr reversibel (Abbildung 3a und ergänzende Zahlen 9-11). Jedoch induzierte in 293Ts, wo keine Veränderung in der Transkription nach langfristigen Dimerisierung beobachtet wurde, Veränderungen im Chromatin Kontakte blieb reversibel (Abb. 3 b und ergänzende Abbildung 9).
Insgesamt wurde nur ein kleiner Rückgang der Genexpression nach 10 Tagen der ABA Entfernung beobachtet, die deutlich höher als Gen Ausdruck vor Dimerisierung (Abb. 3 c) blieb. In diesem Sinne K562-Zellen, aber nicht 293T Zellen zeigte nachhaltige Veränderungen in H3K4me3 und RNA Pol-II in der β-Globin Locus von ChIP-qPCR, im Vergleich zu Kontrollen, auch nach 10 Tagen der ABA Entfernung (Abbildung 3d-f). So, unsere Ergebnisse zeigen, dass die Stabilität der Chromatin-Schleife mehr nachhaltige Genexpression impliziert.
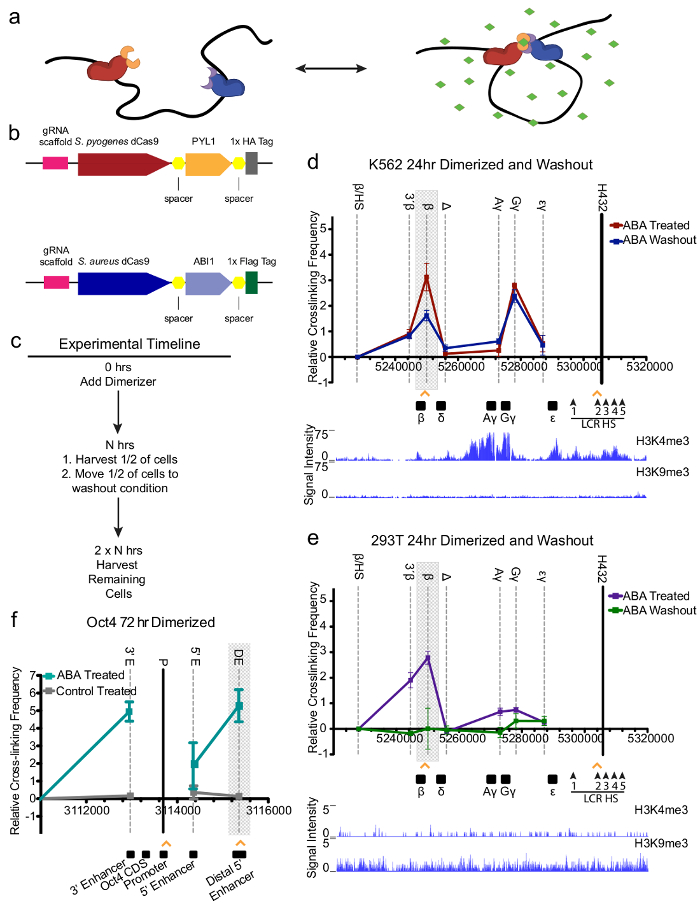
Abbildung 1: CLOuD9 induziert reversible β-Globin-Promoter-LCR Schleifen. (a) Ergänzung der Abscisic Säure (ABA, grüne) bringt zwei komplementäre CLOuD9 Konstrukte (CLOuD9 S. Pyogenes (CSP), CLOuD9 S. Aureus (CSA), rot und blau, beziehungsweise) in die Nähe, Umbau Chromatinstruktur. Entfernung von ABA stellt die endogenen Chromatin-Konformation. (b) CLOuD9 Konstrukte kombinieren CRISPR-dCas9-Technologie von S. Aureus und S. Pyogenes mit reversibel dimerizeable PYL1 und ABI1-Domänen. (c) Chronik von CLOuD9 Dimerisierung Experimente. (d) 3 C Test Messung β-Globin Locus-weite Vernetzung Frequenzen in K562-Zellen nach 24 Stunden nach der Behandlung mit ABA (rot) und anschließende Auswaschen (blau) zeigt die Reversibilität der induzierten β-Globin/LCR-Kontakte (grau hinterlegt). Orange Pfeilspitzen zeigen spezifische CLOuD9 Konstrukt Zielregionen. Das EcoRI-Fragment mit Überempfindlichkeit Standorten 1-4 der LCR (schwarze Balken) diente als Anker-Region. Seine Frequenz der Vernetzung mit anderen angegebenen EcoRI-Fragmenten (Namen oben auf der Grafik) wurden beurteilt. Die menschlichen β-Globin-Gene und LCR Überempfindlichkeit Websites sind auf der Unterseite des Diagramms mit chromosomalen Positionskoordinaten dargestellt. Daten vom ChIP-Seq von H3K4me3 und H3K9me3 zeigen, dass diese Region euchromatisch in K562s (e) ähnlich ist reversible Veränderungen in Chromatinstruktur in HEK 293T Zellen, trotz Beweise aus H3K4me3 und H3K9me3 ChIP-Seq-Daten zu sehen sind, die die Globin die Region ist in einer solchen Zelle heterochromatischen. (f) 3 C Test Messen Oct4 Locus-weite Vernetzung Frequenzen im 293T Zellen nach 72 h Behandlung mit ABA (rot) zeigt Oct4/distalen Enhancer-Kontakte (grau hinterlegt) induziert. Orange Pfeilspitzen zeigen spezifische CLOuD9 Konstrukt Zielregionen. Das MboI Fragment mit der Oct4 Promoter (schwarze Balken) diente als Anker-Region. Seine Frequenz der Vernetzung mit anderen angegebenen MboI Fragmenten (Namen oben auf der Grafik) wurden beurteilt. Die menschlichen Oct4-Regionen sind auf der Unterseite des Diagramms mit chromosomalen Positionskoordinaten dargestellt. Alle 3C Ergebnisse wurden von mindestens drei unabhängigen Experimenten erhalten. 3C-Werte wurden an Tubulin normalisiert. Für β-Globin waren Interaktion Frequenzen zwischen den Anker-Fragment und das Fragment umfasst das β/HS-Fragment auf NULL gesetzt. Für Oct4 waren Interaktion Frequenzen zwischen den Anker-Fragment und eine Negativkontrolle Fragment außerhalb des Oct4 interagierenden auf NULL gesetzt. Fehlerbalken zeigen s.d. n = 3. Diese Zahl wurde von Abbildung 1 in Morgan, Stefanie L., Et Al. modifiziert 9. Bitte klicken Sie hier für eine größere Version dieser Figur.
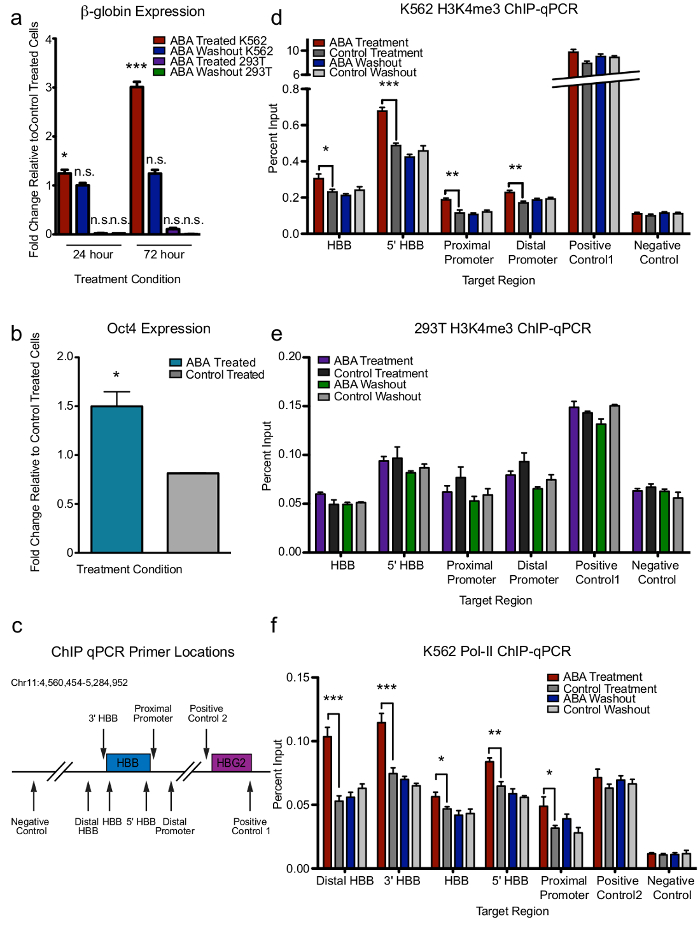
Abbildung 2: CLOuD9 induziert Kontext spezifische Veränderungen im Gen-Ausdruck und Chromatin-Zustand. (a) CLOuD9-induzierte Chromatin Schleifen an der β-Globin Locus resultiert die reversible Induktion der β-Globin Ausdruck in K562s aber nicht in 293Ts Bedeutung gegeben im Vergleich zu Kontrollzellen behandelt. Zweiseitigen Student t-Tests * P < 0,05, t = 3.418, df = 5; P < 0,0001, t = 10,42 df = 5; n.s. nicht signifikant. Fehlerbalken zeigen s.d. n = 3. (b) Induktion von Oct4 Ausdruck im Anschluss an 293Ts, CLOuD9-induzierte beobachtet wurde Schleifen an den gleichen Ort. Bedeutung erhält im Vergleich zu Kontrollzellen behandelt. Zweiseitigen Student t-Tests * P < 0,05, t = 4.562, df = 2. Fehlerbalken zeigen s.d. (c) schematische Darstellung der ChIP-qPCR Grundierung Orten entlang der β-Globin gen Körper. (d, e) ChIP-qPCR zeigt reversible Veränderungen in H3K4me3 an der β-Globin Locus in K562s aber nicht in 293Ts nach CLOuD9-induzierte Schleifen. Zweiseitigen Student t-Tests * P < 0,05, ** P < 0,001 *** P < 0,0001. Fehlerbalken zeigen s.d. (f) CLOuD9 vermittelte Änderungen β-Globin-Transkription in K562s mit einem Anstieg der RNA Pol-II-Belegung über die Gesamtheit des β-Globin gen Körpers entsprechen. Zweiseitigen Student t-Tests * P < 0,05, ** P < 0,001 *** P < 0,0001. Fehlerbalken zeigen s.d. Diese Zahl wurde von Abbildung 2 in Morgan, Stefanie L., Et al.modifiziert. 9. Bitte klicken Sie hier für eine größere Version dieser Figur.
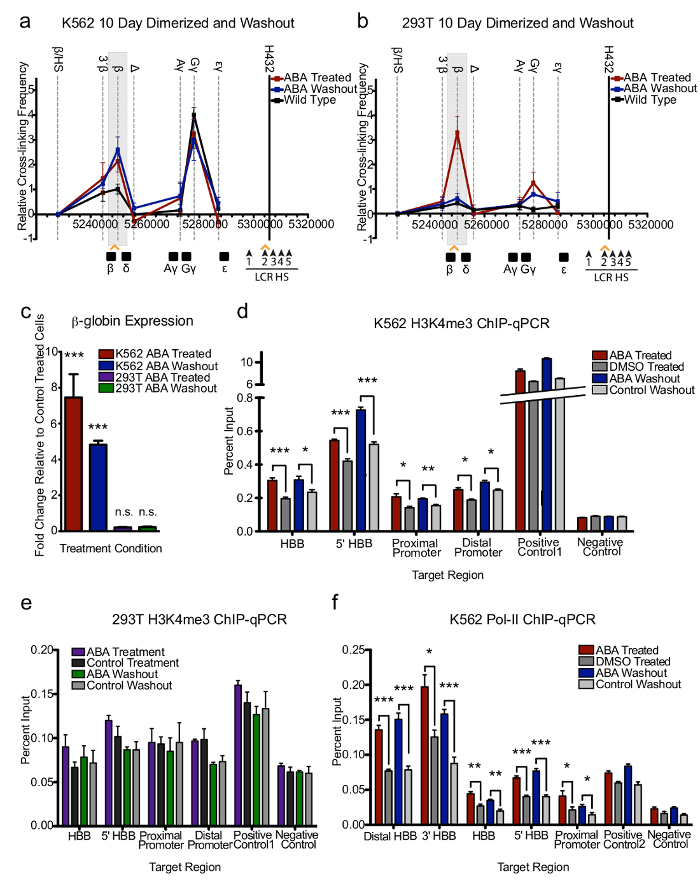
Abbildung 3: CLOuD9 schafft stabile Chromatin-Schleifen, die robuste Genexpression nach langfristigen Dimerisierung aufrecht zu erhalten. (a, b) 3 C-Test zeigt, dass in K562s aber nicht 293Ts, CLOuD9-induzierte Chromatin Schleife wird irreversibel nach 10 Tagen der ABA Behandlung, auch wenn ABA für bis zu 10 zusätzliche Tage entfernt ist. Alle 3C wurden Ergebnisse von mindestens drei unabhängigen Experimenten erhalten. 3C-Werte wurden an Tubulin normalisiert, und Interaktion Frequenzen zwischen den Anker-Fragment und das Fragment umfasst das β/HS-Fragment auf NULL gesetzt wurden. Fehlerbalken zeigen s.d. n = 3. (c) Schleife Stabilisierung im K562s führt zu hartnäckig Ausdruck der β-Globin, auch nach 10 Tagen der ABA auswaschen. Keine Änderungen im β-Globin Ausdruck werden in 293Ts. Bedeutung relativ behandelt Kontrollzellen beobachtet. Zweiseitigen Student t-Tests *** P < 0,0001, t = 5.963, df = 5; n.s. nicht signifikant (d) ChIP-qPCR zeigen Erhöhungen der H3K4me3 Marken über die β-Globin Locus in Reaktion auf CLOuD9-induzierte Schleifen werden gestützt, nach 10 Tagen der Liganden Auswaschung in K562s zweiseitigen Student t-Tests * P < 0,05, ** P < 0,001 *** P < 0,0001. (e) keine signifikanten Veränderungen in H3K4me3 signalisiert folgende langfristige Dimerisierung verzeichneten 293Ts (f) erhöhte RNA Pol-II-Belegung der β-Globin Locus nach langfristigen Schleife Induktion von ChIP-qPCR blieb in K562s Anschluss an 10 Tagen von Liganden auswaschen. Zweiseitigen Student t-Tests * P < 0,05, ** P < 0,001 *** P < 0,0001. Alle Balken Fehler s.d. Diese Zahl wurde von Abbildung 3 in Morgan, Stefanie L., Et al.modifiziert. 9. Bitte klicken Sie hier für eine größere Version dieser Figur.
Ergänzende Abbildung1: CLOuD9 Konstrukte zu lokalisieren, um ihre beabsichtigten Zielregionen. Chromatin Immunopräzipitation und quantitative PCR von CLOuD9 Konstrukte zeigen korrekte Lokalisierung ihrer beabsichtigten genomic Loci. Diese Zahl wurde von ergänzenden Abbildung1 in Morgan, Stefanie L., Et al.modifiziert. 9. Klicken Sie bitte hier, um diese Zahl zu downloaden.
Ergänzende Abbildung2: CLOuD9 Konstrukte reversibel zu verbinden, in Reaktion auf die Behandlung von ABA. Co-Immunperoxidase demonstriert die Vereinigung der dCas9 Proteine, die folgenden 72 h ABA Behandlung ist umgekehrt folgenden weiteren 72 h von Liganden auswaschen. Diese Zahl wurde von ergänzenden Abbildung2 in Morgan, Stefanie L., Et al.modifiziert. 9. Klicken Sie bitte hier, um diese Zahl zu downloaden.
Ergänzende Abbildung 3: Kontrolle der Behandlung induziert keine Veränderungen im Chromatin Kontakte. Behandlung mit DMSO, Schädlingsbekämpfungsmittel, 24 Stunden lang keine Änderungen in der endogenen Chromatin-Konformation durch 3C in den beiden K562-Zellen auslöst oder HEK 293Ts. 3C Werte wurden auf Tubulin und Interaktion Frequenzen zwischen den Anker-Fragment und das Fragment normalisiert umfasst das β/HS-Fragment auf NULL gesetzt wurden. Fehlerbalken zeigen SD n = 3. Diese Zahl wurde von ergänzenden Abbildung 3 in Morgan, Stefanie L., Et al.modifiziert. 9. Klicken Sie bitte hier, um diese Zahl zu downloaden.
Ergänzende Abbildung 4: Kontrolle CLOuD9 ausgestrahlt Zellen zeigen keine Veränderungen im Chromatin looping. Leitung zwei CLOuD9 baut entweder die LCR oder der β-Globin-Veranstalter keine wesentlichen Änderungen in der Chromatinstruktur durch 3C nach ABA Behandlung relativ Kontrollbehandlung auslöst. 3C-Werte wurden an Tubulin normalisiert, und Interaktion Frequenzen zwischen den Anker-Fragment und das Fragment umfasst das β/HS-Fragment auf NULL gesetzt wurden. Fehlerbalken zeigen SD n = 3. Diese Zahl wurde von ergänzenden Abbildung 4 in Morgan, Stefanie L., Et al.modifiziert. 9. Klicken Sie bitte hier, um diese Zahl zu downloaden.
Ergänzende Abbildung 5: CLOuD9 Chromatin Schleife bleibt nach 72 Stunden Dimerisierung reversibel. 3C-Probe in K562s zeigt, Reversibilität der CLOuD9 β-Globin/LCR Kontakte nach 72 Stunden nach der Behandlung ABA induziert. 3C-Werte wurden an Tubulin normalisiert, und Interaktion Frequenzen zwischen den Anker-Fragment und das Fragment umfasst das β/HS-Fragment auf NULL gesetzt wurden. Fehlerbalken zeigen SD n = 3. Diese Zahl wurde von ergänzenden Abbildung 5 in Morgan, Stefanie L., Et al.modifiziert. 9. Klicken Sie bitte hier, um diese Zahl zu downloaden.
Ergänzende Abbildung 6: CLOuD9 induzierte β-Globin/LCR Schleife wird nicht durch Globin Zielsite beeinträchtigt. Regie, CSA und CSP auf Alternative Regionen der LCR oder der β-Globin Projektträger führt zu ähnlichen reversible Veränderungen in Schleife Induktion von 3C nach 72 Stunden nach der Behandlung ABA konstruiert. 3C-Werte wurden an Tubulin normalisiert, und Interaktion Frequenzen zwischen den Anker-Fragment und das Fragment umfasst das β/HS-Fragment auf NULL gesetzt wurden. Fehlerbalken zeigen SD n = 3. Diese Zahl wurde von ergänzenden Abbildung 6 in Morgan, Stefanie L., Et al.modifiziert. 9. Klicken Sie bitte hier, um diese Zahl zu downloaden.
Ergänzende Abbildung 7: CLOuD9 induzierte Veränderungen in der Genexpression sind unabhängig von der Ziel-Site Globin getragen. Regie, CSA und CSP Alternative Regionen des β-Globin-Promotors konstruiert und LCR hat keinen Einfluss auf die Induktion der Genexpression nach 72 h Dimerisierung. Während einige auf die Stärke der Genexpression folgende langfristige (10 Tage) Dimerisierung beobachtet wurde beeinflussen, waren hohe β-Globin relativ Kontrollzellen behandelt jedoch nachhaltige folgende nachfolgenden Liganden Auswaschung für 10 weitere Tage. Bedeutung erhält im Vergleich zu Kontrollzellen behandelt. p < 0,001, t = 10,25, df = 5; p < 0,0001, von links nach rechts t = 8.697, df = 6, t = 40.31, df = 7; n.s. nicht signifikant. Alle Balken Fehler SD Diese Zahl wurde von ergänzenden Abbildung 7 in Morgan, Stefanie L., Et al.modifiziert. 9. Klicken Sie bitte hier, um diese Zahl zu downloaden.
Ergänzende Abbildung 8: Kontrolle CLOuD9 ausgestrahlt Zellen zeigen keine Veränderungen im β-Globin Ausdruck. Leitung zwei CLOuD9 baut entweder die LCR oder der β-Globin-Promoter induziert keine wesentlichen Änderungen im β-Globin Ausdruck nach ABA Behandlung relativ zur Kontrolle der Behandlung. Bedeutung erhält im Vergleich zu Kontrollzellen behandelt. n.s. nicht signifikant. Diese Zahl wurde von ergänzenden Abbildung 8 in Morgan, Stefanie L., Et al.modifiziert. 9. Klicken Sie bitte hier, um diese Zahl zu downloaden.
Ergänzende Abbildung 9: langfristige Kontrolle der Behandlung induziert keine Veränderungen im Chromatin Kontakte. Behandlung mit DMSO, Schädlingsbekämpfungsmittel, 10 Tage lang keine Veränderung in der endogenen Chromatin Konformation durch 3C in den beiden K562-Zellen auslöst oder HEK 293Ts. 3C Werte wurden auf Tubulin und Interaktion Frequenzen zwischen den Anker-Fragment und das Fragment normalisiert umfasst das β/HS-Fragment auf NULL gesetzt wurden. Fehlerbalken zeigen SD n = 3. Diese Zahl wurde von ergänzenden Abbildung 9 in Morgan, Stefanie L., Et al.modifiziert. 9. Klicken Sie bitte hier, um diese Zahl zu downloaden.
Ergänzende Abbildung 10: Long-Term CLOuD9 induzierte β-Globin/LCR Schleife wird nicht durch Globin Zielsite beeinträchtigt. Regie, CSA und CSP für alternative Regionen der LCR baut oder der β-Globin-Promotor resultiert ebenso nachhaltig Schleife Induktion beweist 3 C nach 10 Tagen der ABA Behandlung und 10 Tage der nachfolgenden Liganden auswaschen. 3C-Werte wurden an Tubulin normalisiert, und Interaktion Frequenzen zwischen den Anker-Fragment und das Fragment umfasst das β/HS-Fragment auf NULL gesetzt wurden. Fehlerbalken zeigen SD n = 3. Diese Zahl wurde von ergänzenden Abbildung 10 in Morgan, Stefanie L., Et al.modifiziert. 9. Klicken Sie bitte hier, um diese Zahl zu downloaden.
Ergänzende Abbildung 11: CLOuD9 Konstrukte irreversibel zu assoziieren, als Reaktion auf ABA Langzeitbehandlung. Co-Immunperoxidase demonstrieren irreversible Verband der CSA und CSP dCas9 Proteine nach 10 Tagen der ABA Behandlung und 10 nachfolgende Tage des Liganden auswaschen. Diese Zahl wurde von ergänzenden Abbildung 11 in Morgan, Stefanie L., Et al.modifiziert. 9. Klicken Sie bitte hier, um diese Zahl zu downloaden.
Ergänzende Tabelle1: Liste der Primer-Sequenzen für gRNAs, qRT-PCR, 3C und ChIP qPCR. Diese Tabelle wurde von ergänzenden Tabelle1 in Morgan, Stefanie L., Et al.modifiziert. 9. Bitte klicken Sie hier, um diese Tabelle herunterladen
Diskussion
The Most wichtige Schritte im CLOuD9 Chromatin Schleifen sind: (1) entwerfen oder mit Hilfe der richtigen gRNAs, 2) wechselnde Medien täglich auf CLOuD9 ausgestrahlt Zellen, einschließlich ABA oder DMSO, 3) Erhaltung der Frische der ABA, und (4) genaue und sorgfältige Beurteilung der Durchführung Chromatin-Konformation.
Die Grenzen der CLOuD9 befinden sich hauptsächlich in der Fähigkeit, Leitfäden für die Zielregion der Wahl zu gestalten. Guide-RNAs führen die wichtige Aufgabe der Lokalisierung der dCas9 Komponenten DNA Zielregionen zu dimerized werden und die Wirksamkeit der Guides basieren auf ihre spezifischen Ziel-Site. Ohne die richtige gRNA Komponenten induziert die CLOuD9 System nicht reversibel bilden können Schleifen. So wird durch mehrere Hilfslinien für die einzelnen Regionen von Interesse Gestaltung und Verbreitung der Führer über einen Bereich von 250-1000 bp, mindestens eine erfolgreiche Führer sichergestellt werden. Leitfaden-Lage ist auch integraler Bestandteil der genaue Ergebnisse. Es ist wichtig zu vermeiden, Führungen befindet sich in Transkription Faktor Bindungsstellen oder anderen kritischen Regionen um Hintergrundeffekte wie oben oder down-Regulation der Transkription zu verhindern. Darüber hinaus kann die genaue Lage des Konstrukts CLOuD9 leicht Transkription des Zielgens auswirken. Dies unterstreicht die Bedeutung des Tests mehrere Paare von Leitfäden für jedes Zielgebiet, das robusteste paar um zu Versuchszwecken zu identifizieren. Darüber hinaus in jedem Paar Zielregionen, das CSA-Konstrukt sollte mit gRNAs für S. Aureusgezielt und das CSP-Konstrukt sollte mit gRNAs für S. Pyogenes für targeting Spezifität ausgerichtet sein.
Um genaue Ergebnisse und korrekte Dimerisierung zu gewährleisten, ist es auch wichtig, die Frische der zellulären Umgebungen nach der Transduktion der CLOuD9 Konstrukte zu erhalten. Tägliche Medienwechsel und die Zugabe von frischen Dimerizer (oder Control) sorgt dafür, dass die komplementäre Konstrukte in der Nähe bleiben und die veränderten Chromatin-Konformation bewahren. Darüber hinaus ist es wichtig, authentische Resultate, garantieren die ABA ist frisch und entsprechend nach den Hersteller-Protokoll (eröffnet innerhalb von 6 Monaten, kalt, lichtgeschützt aufbewahrt) gespeichert wurden.
Insbesondere wurde ABA Dimerizer für CLOuD9 mit ABI und PYL Dimerisierung Proteine verwendet, anstatt mehr allgemein FRB und FKBP-System genutzt. Die Notwendigkeit einer Rapalog für die FRB/FKBP-System würde die Anwendbarkeit der CLOuD9, wegen der Giftigkeit zu Krebszellen beschränkt haben. Das Alternativsystem ABI/PYL umgangen diese Einschränkung, effektiv ermöglicht CLOuD9 breiter nutzbar sein.
Gemeinsam entwickelten wir CLOuD9, eine einzigartige und robuste Technologie, die gewaltsam können aber reversibel schaffen Kontakte zwischen langfristigen Ziel genomic Loci. Durch Induktion Chromatin Schleifen, zeigen wir auch, dass CLOuD9 genutzt werden können, um gen-Expression in den entsprechenden zellulären Kontext ändern. Die Anpassungsfähigkeit der Technologie ermöglicht die uneingeschränkte Untersuchung der Wechselwirkungen zwischen jeden zwei genomic Loci, ohne Vorkenntnisse in der Schleife Regionen oder looping Mechanismen. CLOuD9 einzigartige nachgewiesene Reversibilität ermöglicht darüber hinaus weitere Untersuchung der Schleifen Mechanismen in Krankheit und Entwicklung. Während die zielgenaue Auswirkungen von Chromatin looping eindeutig nachgewiesen wurden, ist noch Daten bietet Einblick in die Auswirkungen der Ziel-looping und die weiteren Auswirkungen auf die Schlaufen am Ziel zu sein.
Unsere Daten nur wenige Anwendungen dieses Tool zeigt aber impliziert die große zugrunde liegende Idee, dass Chromatin Anordnung der Genexpression bezeichnend ist. Unsere Technologie kann verwendet werden, um zu studieren und die Nuancen der Chromatinstruktur in Genregulation und verbessert das allgemeine Verständnis der Rolle des Chromatins Falten in der Transkription von Genen zu offenbaren. Ein besseres Verständnis für die Feinheiten der transkriptionellen Dynamik kann eine Vorreiterrolle in der Forschung und Behandlung von Krebs, Erbkrankheiten und angeborene Erkrankungen, in denen verschiedene, die Chromatin Assembly zweifellos Gen Ausdruck20verändert, 21,22,23. Folgearbeiten CLOuD9 Technologie beleuchten weitere Details über die Anordnung und die Dynamik von Chromatin Domains und wie sie fahren Falten zu stabilen Genexpression in Entwicklung und Krankheit aufrecht zu erhalten.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir danken H. Chang, T. Oro, S. Tavazoie, R. Flynn, s. Batista, E. Calo und die gesamte Wang-Labor für technische Unterstützung und kritische Lektüre des Manuskripts. S.L.M. wurde in dieser Arbeit durch die NSFGRF (DGE-114747), NDSEGF (FA9550-11-C-0028) und das National Cancer Institute (1F99CA222541-01) unterstützt. K.C.W. wird durch den Career Award für Mediziner aus dem Burroughs Wellcome Fonds unterstützt und ist ein Donald E. und Delia B. Baxter Foundation Faculty Scholar.
Materialien
| Name | Company | Catalog Number | Comments |
| RPMI 1640 media | Life Technologies | 11875-119 | For K562 cell culture |
| DMEM media | Life Technologies | 11995-065 | 1X, for 293T cell culture |
| lentiCRISPR v2 | Addgene plasmid | #52961 | For CLOuD9 plasmid development |
| pRSV-Rev | Addgene plasmid | #12253 | For lentivirus production |
| pMD2.G | Addgene plasmid | #12259 | For lentivirus production |
| pMDLg/pRRE | Addgene plasmid | #12251 | For lentivirus production |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668-019 | For lentivirus production |
| anti-HA antibody | Cell Signaling | 3724 | For immunoprecipitation |
| anti-Flag antibody | Sigma | F1804 | For immunoprecipitation |
| DNeasy Blood and Tissue Kit | Qiagen | 69504 | For DNA extraction |
| TRIzol | Life Technologies | 15596-018 | For RNA extraction |
| RNeasy Kit | Qiagen | 74106 | For RNA extraction |
| Superscript VILO | Life Technologies | 11754-050 | For cDNA |
| SYBR Green I MasterMix | Roche | 4707516001 | For qPCR analysis |
| Light Cycler 480II | Roche | For qPCR analysis | |
| anti-H3K4me3 antibody | AbCam | ab8580 | For ChIP-qPCR |
| anti-RNA Pol-II antibody | Active Motif | 61083 | For ChIP-qPCR |
| EDTA free protease inhibitor | Roche | 11873580001 | For protein extraction |
| 4-12% Tris Glycine gel | Biorad | Any size, For western blot | |
| anti-Rabbit HRP antibody | Santa Cruz | sc-2030 | For western blot |
| anti-mouse HRP antibody | Cell Signaling | 7076S | For western blot |
| K562 and H3K293 ChIP-Seq data | Encode | ENCSR000AKU | For ChIP-seq analysis |
| K562 and H3K293 ChIP-Seq data | Encode | ENCSR000APE | For ChIP-seq analysis |
| K562 and H3K293 ChIP-Seq data | Encode | ENCSR000FCJ | For ChIP-seq analysis |
| K562 and H3K293 ChIP-Seq data | GEO | GSM1479215 | For ChIP-seq analysis |
| Dynabeads Protein A for Immunoprecipitation | Thermo Fisher Scientific | 10001D | For immunoprecipitation |
| Dynabeads Protein G for Immunoprecipitation | Thermo Fisher Scientific | 10004D | For immunoprecipitation |
| RNA Clean & Concentrator-5 | Zymo Research | R1015 | For RNA purification |
| Pierce 16% Formaldehyde Methanol-free | Thermo Fisher Scientific | 28908 | For crosslinking |
| PX458 Plasmid | Addgene | 48138 | Suggested active Cas9 plasmid for gRNA cloning, but any active Cas9 plasmid will do |
| QIAquick PCR Purification Kit | Qiagen | 28104 | For PCR purification |
| FastDigest BsmBI | Thermo Fisher Scientific | FD0454 | For cloning guide RNAs |
| FastAP | Thermo Fisher Scientific | EF0651 | For cloning guide RNAs |
| 10X FastDigest Buffer | Thermo Fisher Scientific | B64 | For cloning guide RNAs |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | For cloning guide RNAs |
| 10X T4 Ligation Buffer | NEB | B0202S | For cloning guide RNAs |
| T4 PNK | NEB | M0201S | For cloning guide RNAs |
| 2X Quick Ligase Buffer | NEB | B2200S | For cloning guide RNAs |
| Quick Ligase | NEB | M2200S | For cloning guide RNAs |
| Buffers | |||
| Farnham lysis buffer | 1% Tris-Cl pH 8.0, 1% SDS, 1% protease inhibitor water solution (non-EDTA), and 1 mM EDTA in water | ||
| Modified RIPA buffer | 1% NP40/Igepal, 0.5% sodium deoxycholate, 0.1% SDS, 1 mM EDTA, and 1% protease inhibitor water solution (non-EDTA) in PBS pH 7.8 or 7.4 | ||
| IP dilution buffer | 0.01% SDS, 1.1% Triton-X 100, 1.2 mM EDTA, 16.7 mM Tris-HCl pH 8.0, 167 mM NaCl, 0.1x protease inhibitor | ||
| Wash buffer | 100 mM Tris pH 9, 100 mM LiCl, 1% NP-40, and 1% sodium deoxycholate | ||
| Swelling buffer | 0.1 M Tris pH 7.5, 10 mM potassium acetate, 15 mM magnesium acetate, 1% NP-40 | ||
| Dilution buffer | 0.01% SDS, 1.1% Triton X-100, 1.2 mM EDTA, 16.7 mM Tris pH 8 and 167 mM NaCl | ||
| IP elution buffer | 1% SDS, 10% NaHCO3 |
Referenzen
- Krivega, I., Dean, A. Chromatin looping as a target for altering erythroid gene expression. Ann. N.Y. Acad. Sci. 1368, 31-39 (2016).
- Matharu, N., Ahituv, N. Minor loops in major folds: Enhancer-promoter looping, chromatin restructuring, and their association with transcriptional regulation and disease. PLoS Genet. 11, e1005640 (2015).
- Dekker, J., Martí-Renom, M. A., Mirny, L. A. Exploring the three-dimensional organization of genomes: interpreting chromatin interaction data. Nat. Rev Genet. 14, 390-403 (2013).
- Kim, A., Dean, A. Chromatin loop formation in the b-globin locus and its role in globin gene transcription. Mol Cells. 34, 1-5 (2012).
- Petrascheck, M., et al. DNA looping induced by a transcriptional enhancer in vivo. Nucleic Acids Res. 33, 3743-3750 (2005).
- Ameres, S. L., et al. Inducible DNA-loop formation blocks transcriptional activation by an SV40 enhancer. EMBO J. 24, 358-367 (2005).
- Deng, W., et al. Controlling Long-Range Genomic Interactions at a Native Locus by Targeted Tethering of a Looping Factor. Cell. 149, 1233-1244 (2012).
- Deng, W., et al. Reactivation of developmentally silenced globin genes by forced chromatin looping. Cell. 158, 849-860 (2014).
- Morgan, S. L., et al. Manipulation of Nuclear Architecture through CRISPR-Mediated Chromosomal Looping. Nature Communications. 8, (2017).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biol. 17 (1), 148 (2016).
- . . Lipofectamine 2000 Reagent. , (2013).
- . . DNeasy Blood & Tissue Kit Handbook. , (2006).
- . . Determining Genome Targeting Efficiency using T7 Endonuclase I (M0302). , (2016).
- Guschin, D. Y., et al. A rapid and general assay for monitoring endogenous gene modification. Methods Mol Biol. 649, 247-256 (2010).
- Shalem, O., Sanjana, N. E., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. , 83-87 (2014).
- . . RNeasy Mini Handbook. , (2012).
- . . SuperScript VILO cDNA Synthesis Product Information Sheet. , (2015).
- Theunissen, T. W., et al. Systematic identification of culture conditions for induction and maintenance of naive human pluripotency. Cell Stem Cell. 15, 471-487 (2014).
- Mokry, M., et al. Integrated genome-wide analysis of transcription factor occupancy, RNA polymerase II binding and steady state RNA levels identify differentially regulated functional gene classes. Nucleic Acids Res. 40, 148-158 (2012).
- Drier, Y., et al. An oncogenic MYB feedback loop drives alternate cell fates in adenoid cystic carcinoma. Nat Genet. 48, 265-272 (2016).
- Ryan, R. J. H., et al. Detection of enhancer-associated rearrangements reveals mechanisms of oncogene dysregulation in B-cell lymphoma. Cancer Discov. 5, 1058-1071 (2015).
- Montavon, T., et al. A regulatory archipelago controls Hox genes transcription in digits. Cell. 147, 1132-1145 (2011).
- Lupianez, D. G., et al. Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions. Cell. 161, 1012-1025 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten