Method Article
CRISPR induite par la réorganisation de la Structure de la chromatine boucle
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Le bouclage de la chromatine joue un rôle important dans la régulation des gènes ; Toutefois, on n’a aucun progrès technologiques qui permettent une modification sélective et réversible de la chromatine boucles. Nous décrivons ici un système puissant pour re-Organisation de la chromatine boucle utilisant CRISPR-dCas9 (CLOuD9), démontré de manière sélective et réversible modulent l’expression des gènes à ciblées locus.
Résumé
Des études récentes ont démontré que la chromatine à longue portée, en trois dimensions, une boucle de jeu d’interactions entre un rôle important dans la régulation de l’expression des gènes, mais si une boucle est responsable ou un résultat d’altération dans l’expression des gènes est toujours inconnu. Jusqu'à tout récemment, comment la chromatine bouclage affecte la régulation de l’activité des gènes et de la fonction cellulaire a été relativement ambigu et limites des méthodes existantes pour manipuler ces structures empêchent une exploration approfondie de ces interactions. Pour dissiper cette incertitude, nous avons conçu une méthode pour la réorganisation de la boucle sélectif et réversible de la chromatine en utilisant CRISPR-dCas9 (CLOuD9). Le dynamisme du système CLOuD9 a été démontré par localisation réussie de CLOuD9 constructions pour cibler les locus génomiques pour moduler la conformation de la chromatine local. Ce qui est important, la possibilité d’inverser le contact induit et restaurer la conformation de la chromatine endogène a également été confirmée. Modulation de l’expression génique avec cette méthode établit la capacité à réguler l’expression génique cellulaire et met en évidence le grand potentiel pour les applications de cette technologie dans la création de boucles de chromatine qui affectent notablement gène stable de novo expression dans le contexte du cancer et de développement.
Introduction
La relation entre la chromatine pliage dans le noyau et l’organisation spécifique du génome a suscité d’intérêt important ces dernières années, puisqu’il a été démontré pour être étroitement liées à l’expression de gène1,2. Alors que la relation exacte entre l’activité des gènes et la modulation de la structure de la chromatine reste imprécie, il a émis l’hypothèse que les interactions entre les contacts chromosomiques par suite de l’organisation de la dynamique de la chromatine en trois dimensions servent un fonction de régulation de gène3. En effet, un tel effet a été bien démontré dans le locus du gène globine humaine, où la région de contrôle du locus (LCR) réglemente l’activité des gènes de globine développemental concrètement en créant une boucle de la chromatine entre deux régions4. Toutefois, à la fois ceci et d’autres régions, on ignore si le bouclage de la chromatine est une cause ou la conséquence des altérations dans l’expression des gènes.
Jusqu'à présent, les défis dans l’étude de ce phénomène restaient en suspens. Par exemple, les autres tentatives d’induire la chromatine boucles impliqués modification de la séquence d’ADN linéaire ou procédures compliquées nécessitant une abondance de connaissances de base sur des éléments spécifiques qui facilitent en boucle5,6, 7,8. En outre, alors que les travaux précédents a suggéré que la chromatine en boucle l’expression des gènes en voiture dans un contexte spécifique et limité7,8, le niveau à laquelle la chromatine bouclage affecte transcription au niveau mondial est incertain. Bien que l’intérêt pour l’impact du bouclage à long terme sur l’expression génique a continuellement augmenté ces dernières années, des questions sans réponse sur l’établissement et le maintien des contacts de la chromatine pour modifier l’activité des gènes persistent.
La technologie que nous avons conçu emploie la nucléase déficient en cluster régulièrement dois‑je courtes répétitions palindromique (CRISPR) - CRISPR - associées protéine 9 (dCas9), afin de permettre le ciblage largement applicable d’un locus génomiques9. Cette technologie élimine les problèmes complexes liés à des modifications de la séquence d’ADN linéaire et est accessible sans connaissance préalable significative des composantes particulières de bouclage. Plus particulièrement, l’outil est universelle et largement applicable aux boucles de chromatine reconnues en développement ainsi qu’une variété de maladies comme le cancer. La puissance de CLOuD9 attestent réversiblement modifier la structure de boucles pour effectivement moduler l’expression des gènes.
Protocole
1. gRNA Design
- Sélectionnez un outil de conception standard gRNA pour concevoir le guide RNAs10. Utilisez des paires complémentaires des constructions CLOuD9 développé précédemment9 (p. ex., construisent d’au moins 1 S. aureus (CSA) et 1 S. pyogenes (CSP)) pour chaque expérience.
- Dans l’outil de conception en ligne sélectionnés, utilisez la séquence de Motif Adjacent Protospacer (PAM) « NGG » d’une longueur de guide de 20 bp pour CSP et la PAM de séquence « NNGRRT » avec une longueur de guide de 21 pour l’ASC.
- Choisissez les guides basées sur la spécificité marquer et guide sur les deux brins de maximiser ses chances d’obtenir un guide de travail de conception.
- Commander 2 oligonucléotides par guide par un fabricant d’oligo : pour les oligo avant, ajouter « caccg » à l’extrémité 5' de la séquence de guide et pour les oligo inverse, ajouter « aaac » à l’extrémité 5' et le « c » à l’extrémité 3' avant de commander.
- D’abord, la conception gRNAs 3-5 pour chacune des régions d’intérêt, répartis sur une région de Pb 250-1000. Évaluer la gRNA ciblant l’efficacité comme suit :
- GRNAs clone identifié dans un plasmide de Cas9 actif (voir étape 3) et transitoirement transfecter dans les cellules 293 t (voir étape 4)11.
- La croissance de cellules pendant 2-3 jours dans de Dulbecco modifié Eagle (DMEM) avec 10 % sérum fœtal (SVF) et 1 % la pénicilline-streptomycine (strep stylo), puis récolter et extraire l’ADN génomique12.
- Amplifier les régions ciblées par polymerase chain reaction (PCR). Lancer des produits sur un gel d’agarose de 1,5 % et purifier les bandes appropriées.
- Effectuer un test d’endonucléase T7 comme décrit dans la Guschin, et al. Protocole n°13,14.
NOTE : Guides efficaces sont ceux qui sont déterminés à couper l’ADN sur le site cible.
2. Culture cellulaire
- Maintenir les cellules dans un milieu sain et divisent activement l’État.
- Pour K562s, la culture dans les médias de Roswell Park Memorial Institute (RPMI) 1640 avec 10 % de SVF et 1 % plume de strep.
- Pousser K562s dans une fiole de cou incliné 25 cm2 pour l’entretien et ajuster la densité cellulaire de 400 000 cellules / mL chaque jour.
- Après que K562s ont été transduites avec constructions de CLOuD9, ajouter 2 puromycine µg/mL et 100 µg/mL hygromycine entretien media.
- Pour 293Ts, la culture dans de Dulbecco modifié Eagle (DMEM) avec 10 % de SVF et 1 % plume de strep.
- Croissance 293Ts en plaques de 10 cm2 et séparer quand confluentes.
- Pour K562s, la culture dans les médias de Roswell Park Memorial Institute (RPMI) 1640 avec 10 % de SVF et 1 % plume de strep.
3. plasmide préparation et gRNA Insertion15
Remarque : Les cartes de plasmide sont disponibles dans l’annexe et amorces utilisées dans les expériences de l’exemple sont disponibles en supplémentaire tableau 1.
- Mix 5 µg du plasmide CRISPR lentiviral avec 3 µL de BsmBI, 3 µL de la Phosphatase alcaline, 6 µL de mémoire tampon 10 x et 0,6 µL de fraîchement préparée 100 mM TNT. Porter le volume total de 60 µL avec FD2O et incuber à 37 ° C pendant 30 min à digérer et déphosphorylent le plasmide.
- Gel de purifier le plasmide digéré et éluer à FD2O.
- Mélanger 1 µL de chacune des guides appariés à 100 µM avec 1 µL de 10 x 0,5 µL T4 ligature tampon et 6,5 µL de ddH2O T4 PNK, pour atteindre le volume total de 10 µL. Incuber à 37 ° C pendant 30 min, puis à 95 ° C pendant 5 min, puis rampe jusqu'à 25 ° C à 5 ° C/min.
Remarque : Cela va recuire la paire de guides. - Diluer au 1 : 200 guides recuit (à l’étape 3.3) FD2O.
- Mélanger 1 µL du plasmide digéré à l’étape 3.2 avec 0,5 µL des guides ligaturés dilués de l’étape 3.4, 2,5 µL de 2 x tampon 1 µl de ddH2O et 0,5 µL de Ligase, de faire une réaction totale 5,5 µL. Ceci incuber à température ambiante pendant 10 min à ligaturer les guides pour le plasmide BsmBI digéré.
- Transformer le plasmide nouvellement ligaturé à l’étape 3.5 Stbl3 bactéries et amplifier à l’aide de n’importe quelle méthode de préparation de plasmide.
4. lentivirus Production
- Comte 750 000 293 t cellules / puits pour un six-puits plaque utilisant un hémocytomètre et les graines en DMEM avec 10 % de SVF et 1 % plume de strep. Utiliser un puits pour chaque construction.
- 24h après l’ensemencement, changer les médias au frais sans antibiotique DMEM avec 10 % FBS.
- Dans un tube, diluer 11 µL de réactif de transfection à base de lipides avec 150 µL du milieu OptiMEM, par réaction,11.
- Dans une éprouvette, diluer 2 µg du plasmide vecteur lentiviral CLOuD9 à l’étape 3.6, 0,35 µg de 0,65 µg de pMD2.G avec 150 µL du milieu OptiMEM, par réaction11pMDLg/pRRE et 1 µg de pré-rinçage-Rev.
- Pour chaque réaction, les mélanges de plasmide des gènes dilués s’ajoute le réactif de transfection de lipides à base dilué au rapport 1:1 et incuber pendant 5 min11.
- Ajouter la totalité du volume de chaque complexe mixte de l’étape 4.5 dans leurs puits respectifs des cellules 293 t sans antibiotiques de l’étape 4.2.
- 48 h plus tard, recueillir des médias production virale en pipettant également, dans un nouveau tube et spin down à 300 x g pendant 5 min. Après centrifugation, déplacez le surnageant pour un nouveau tube pour enlever les débris de cellules résiduelles.
- Utiliser les médias de production virale immédiatement pour la transduction des cellules cibles ou congeler à-80 ° C pour une utilisation future.
5. lentivirus Transduction des cellules
- Ajouter 250 µL de chaque construction virale d’intérêt (comprenant des plasmides de CLOuD9 complémentaires) à un tube conique de 15 mL, contenant 80 000 cellules d’application CLOuD9.
- Porter le volume total des médias chacune conique à 1 mL avec les médias sans antibiotiques et ajouter polybrene à une concentration finale d’entre 1 à 8 µg/mL (la concentration maximale de polybrene tolérée par le type de cellule utilisé devra être déterminée expérimentalement).
- Faites tourner les cellules à 800 g pendant 30 min à température ambiante, puis remettre en suspension en pipettant également, sans retirer le surnageant viral. Déplacez la suspension de toute cellule sur une plaque de culture cellulaire.
- transduction après 24h, cellules de spin down à 300 x g pendant 5 min à température ambiante.
- Aspirer le surnageant viral et remettre en suspension des cellules dans les milieux de culture cellulaire ordinaire.
- Le lendemain, ajouter la puromycine (1 µg/mL pour les cellules 293 t) et hygromycine (25 µg/mL pour les cellules 293 t) au milieu de culture cellulaire pour sélectionner des cellules doublement transduits. Expérimentalement, déterminer les concentrations appropriées de puromycine et hygromycine pour un type donné de cellules avant de commencer les expériences.
- Maintenir les cellules en médias de sélection pendant au moins 3 jours avant d’expériences en aval et maintenir dans les médias de sélection pour la durée de toutes les expériences.
6. la dimérisation et lavent par des cellules
- Ajouter 1 mM acide abscissique (ABA) ou un volume équivalent de diméthylsulfoxyde (DMSO) pour les contrôles à la culture cellulaire se dimériser le CLOuD9 sélectionné et transduites cellules. Utiliser l’ABA dans les six mois de la date de réception, garder au froid et protéger de la lumière tout au long de l’utilisation. 2 mM ABA peut être utilisé si nécessaire.
- Changement médias tous les jours pour fournir les frais ABA (ou le DMSO pour les contrôles) pendant la durée des expériences.
Remarque : Aucune limite sur la longueur de dimérisation n’a encore été observé.
- Changement médias tous les jours pour fournir les frais ABA (ou le DMSO pour les contrôles) pendant la durée des expériences.
- Inverser une dimérisation par la suppression de médias contenant ABA et laver les cellules avec assez tamponnée au phosphate salin (PBS), pour couvrir la surface de la plaque, deux fois. Par la suite, les cellules en culture dans des milieux sans ABA pour maintenir un état de non-dimérisé.
7. immunoprécipitation et Co-Immunoprécipitation
Remarque : Assurez-vous de tous les tampons fraîche et immédiatement avant utilisation.
- Après la réalisation d’expériences de dimérisation, recueillir et tournez en bas des cellules, puis aspirer le surnageant.
- Crosslink et geler les cellules
- Faire un stock frais de 1 % de formaldéhyde dans du PBS à la température ambiante à l’aide de matériel frais. Pour chaque cellules 2 millions, resuspendre doucement avec 1 mL de 1 % de formaldéhyde à température ambiante pendant exactement 10 min, tout en tournant.
Remarque : Utilisez un volume minimal de 10 mL pour la réticulation inférieur à 10 millions de cellules. - Étancher la réticulation avec l’ajout de la glycine à une concentration finale de 0,125 M, suivie d’une incubation de 5 min à température ambiante, pendant la rotation.
- Faites tourner les cellules et laver 1-2 fois avec du PBS glacée. Granulés de la cellule peuvent alors être snap congelés dans l’azote liquide et stockées à-80 ° C, soit utilisée immédiatement.
Remarque : Chaleur inversera crosslinks de formaldéhyde. Si précipité, les cellules peuvent être stockés directement à-80 ° C sans geler le composant logiciel enfichable.
- Faire un stock frais de 1 % de formaldéhyde dans du PBS à la température ambiante à l’aide de matériel frais. Pour chaque cellules 2 millions, resuspendre doucement avec 1 mL de 1 % de formaldéhyde à température ambiante pendant exactement 10 min, tout en tournant.
- Préparer le conjugué d’anticorps/perle
Remarque : Les conditions optimisation d’anticorps choisi (c.-à-d., test de mémoires tampons de lysis différents pour la lyse cellulaire et exécutant IP) peuvent être utiles. Deux tampons communes, tampon de lyse de Farnham et tampon RIPA modifiée, peuvent avoir un effet significatif sur l’efficacité de la propriété intellectuelle et l’efficience de la sonication. Toutes les compositions de tampon se trouvent dans la Table des matières. En outre, gardez à l’esprit que la chromatine sonication peut prendre plusieurs heures pour terminer.- Remettre en suspension les perles au vortex brièvement en fonction de l’anticorps choisi.
- Transférer une quantité appropriée de perles pour l’expérience dans un tube de 1,5 mL. Comme point de départ, utiliser 20 µL de perles pour chaque 1 µg d’anticorps. N’ajoutez pas encore l’anticorps.
Remarque : Utiliser 10 µg anticorps avec 200 µL de perles pour 1 mg du total lysat comme point de départ, sauf s’il est connu que l’anticorps est plus ou moins efficace que cela impliquerait. Pour les protéines de faible abondance, ce ratio peut devoir être ajustée. - Si vous utilisez le tampon de lyse de Farnham, laver 3 x dans le tampon de dilution IP. Si vous utilisez le tampon de lyse RIPA, laver 3 x dans le tampon de lyse RIPA.
- Remettre en suspension les perles dans un volume final de la même mémoire tampon de l’étape 7.3.3 (dilution d’IP ou tampon RIPA) qui est > 1 x et < 5 fois son volume initial. c'est-à-dire Pour 100 µL perle initiale de volume, utiliser entre 100 et 500 µL resuspension finale volume.
- Ajoutez l’anticorps à perles lavés maintenant à une concentration appropriée. Pour un bon anticorps HA ou drapeau pour des constructions de CLOuD9 IP, utiliser des anticorps à 01:50 (µg protéine : protéine µg lysat) et incuber entre 8 + h et durant la nuit à 4 ° C, pendant la rotation. Par ailleurs, incuber pendant 2-4 h à température ambiante.
- Retirez le surnageant de perles et jetez-le.
- Perles de lavage 3 x avec le tampon de lavage.
- Remettre en suspension dans un volume minimal de la mémoire tampon utilisée pour une nuit d’incubation avec l’anticorps (dilution d’IP ou tampon RIPA). Garder au froid jusqu'à ce que combinés avec des lysats de protéines.
- Laisser agir la chromatine
- Tout d’abord de lyser les cellules avec l’ajout de gonflement tampon aux pastilles de cellule sur la glace pendant 10 min, pichenette cellules chaque quelques min pour éviter de s’installer.
Remarque : Généralement, 500 µL de tampon de gonflement est ajouté aux cellules 25 millions et mise à l’échelle de là, mais le faire pas utilisation de moins de 500 µL de tampon pour < 25 millions de cellules. - Dounce manuellement dans le sens horaire et dans le sens antihoraire, 13 x chacun.
- Puis des noyaux de spin down à 4 ° C pendant 5 min à 1 500 x g. aspirer le surnageant complètement et répétez l’opération pour enlever le surnageant restant, puis pesez le culot cellulaire en mg.
- Ajouter inhibiteur de la protéase de tampon de lyse des noyaux (buffer tampon Farnham ou RIPA, ci-dessus, choisir une).
- Ajouter 10 x montant du tampon de lyse des noyaux à granulés noyaux pour remettre en suspension (par exemple, ajouter 1 mL de tampon de lyse des noyaux d’une boulette de 0,100 g). Brièvement de vortex et lyse pendant 10 min sur la glace.
Remarque : N’oubliez pas la taille du tube sonication. Volume idéal est souvent < 1 mL, échantillons devrez peut-être être divisé si les pellets sont très grandes.- Pour Co-IP, soniquer noyaux brièvement pour solubiliser matériel et étancher SDS à un niveau qui permette l’immunoprécipitation avec 2-3 volumes de tampon de dilution, puis passez à l’étape 7.4.6. Pour les anticorps très sensibles, ajoutez plus tampon de dilution pour réduire la concentration de la SDD.
Remarque : Arrêter la sonication quand lysat efface ; Il passera de l’opaque/translucide blanc complètement transparent. Conserver les échantillons aussi froid que possible et non plus de laisser agir. - Pour la puce, soigneusement soniquer ADN pour obtenir des fragments d’ADN de 150-1000 bp, puis passez à l’étape 7.4.6.
- Pour Co-IP, soniquer noyaux brièvement pour solubiliser matériel et étancher SDS à un niveau qui permette l’immunoprécipitation avec 2-3 volumes de tampon de dilution, puis passez à l’étape 7.4.6. Pour les anticorps très sensibles, ajoutez plus tampon de dilution pour réduire la concentration de la SDD.
- Essorer les matières insolubles à vitesse maximale pendant 10 min à 4 ° C.
- Transférer les matières solubles dans un tube frais.
- Pour Co-IP, mesurer la concentration de protéine et procéder à la liste déroulante étape 7,5.
- Pour la puce, enlever une quantité de 10 µL de dégagé lysat et ajouter 40 µL de tampon d’élution IP et 6 µL 5 M NaCl à celle pour un total de 56ul. Faire bouillir pendant 15 minutes à 95 ° C, ajouter 5 à 10 µL de 3M NaOAc pH 5 et nettoyer sur une colonne de purification de PCR. Mesurer la concentration de l’ADN en mesurant l’absorbance à 260 nm et uniformiser sur échantillons. Passez à menu déroulant à l’étape 7,5.
NOTE : Extra lysat peut être snap congelé à-80 ° C et utilisé à une date ultérieure, mais obtenir les meilleurs résultats sont obtenus à partir de lysat fraîches. Si le point de congélation, ne pas de gel/dégel lysat plus d’une fois.
- Tout d’abord de lyser les cellules avec l’ajout de gonflement tampon aux pastilles de cellule sur la glace pendant 10 min, pichenette cellules chaque quelques min pour éviter de s’installer.
- Menu déroulant
- Transférer une quantité appropriée de lysat protéique dans un tube frais. Si vous utilisez le tampon de lyse de Farnham, diluer en 2,1 volumes de tampon de dilution IP (voir ci-dessus).
- Mettre de côté 100 µL de liquide surnageant contrôle d’entrée et conserver à 4 ° C jusqu’au lendemain.
- Ajouter perle/anticorps conjugué de l’étape 7.3.8 aux échantillons.
- Faire pivoter les échantillons à 4 ° C durant la nuit et ce qu'il y ait assez de volume pour le mouvement des liquides dans des tubes de 1,5 mL.
- Laver et éluer
- Après rotation pendant la nuit, enregistrez le surnageant sous la fraction non contraignante. Puis lavage perles x 3-5 dans le tampon de dilution IP.
- Préparer la mémoire tampon d’élution IP à température ambiante, sans inhibiteurs.
- Éluer complexes avec 50 µL de tampon d’élution secouée sur un vortex à 67 ° C pendant 15 min. transfert d’élution dans un nouveau tube et répéter avec une autre 50 µL. combiner les deux pour une élution totale de 100 µL.
Remarque : S’il y a trop d’arrière-plan de cette procédure d’élution, chaleur peut être réduit ou éliminé pendant l’agitation/incubation. Généralement, cela réduit le rendement de la protéine cible mais diminue sensiblement l’arrière-plan.
- Inverser les croisements par chauffage à 67 ° C pendant > 4 h.
NOTE : Ceci n’est pas strictement nécessaire pour IPs Co mais peut être utile.- Pour Co-IP, afin d’assurer une dimérisation complète entre les cibles des terroristes, exécuter éluats sur un gel SDS-page et la sonde avec des anticorps contre la balise HA ou drapeau, comme indiqué, tout à 1:1 000. Passez à l’étape 8.
- Pour puce-qPCR, afin d’assurer la localisation correcte et le ciblage de chaque composant CRISPR-dCas9, nettoyer le produit élution sur colonne et éluer à 10 µL. Perform PCR en temps réel quantitative (qPCR) d’ADN purifié. Les amorces utilisées dans les données présentées sont disponibles en supplémentaire tableau 1. Passez à l’étape 8.
8. RNA Extraction et PCR Quantitative
- Afin d’étudier les changements dans l’expression des gènes CLOuD9, tout d’abord, d’isoler et de purifier l’ARN total des pastilles de cellule de contrôle et cellule dimérisé boulettes.
- Faire l’ADN complémentaire (ADNc) à partir de l’ARN purifié par transcription inverse17 et effectuer des analyses de qPCR. Amorces sont disponibles dans le tableau1 supplémentaire.
9. analyse de saisie de Conformation du chromosome
- Pour observer les changements dans la fréquence des contacts de locus génomiques induite par CLOuD9, effectuer du chromosome conformation capture (3C) des analyses de8.
- Quantifier les produits ligature 3C dans deux séries de doublons pour chacun des trois réplicats biologiques par quantitative PCR en temps réel. Normaliser les échantillons aux signaux 3c du locus tubuline. Amorces sont disponibles dans le tableau1 supplémentaire.
Résultats
CLOuD9 induit réversible β-globine promoteur-LCR bouclage. Utilisation appropriée du système CLOuD9 induit contact réversible de l’ASC complémentaire et CSP CLOuD9 construit grâce à l’ajout ou la suppression de l’ABA de milieux de culture cellulaire (Figure 1 a). Constructions de l’ASC et CSP (Figure 1 b) sont localisées à des régions génomiques appropriées à l’aide de standard CRISPR gRNAs. Compte tenu de la vaste documentation du locus globine humaine ainsi que le pliage chromosomiques fréquentes et le réarrangement qui se produit là au cours du développement, cette région a été choisie pour démontrer l’utilité du système CLOuD9. En outre, la lignée cellulaire K562 a été choisie car il a été montré pour constamment expriment des niveaux élevés du gène γ-globine, plutôt que le gène de la β-globine qui est généralement exprimé dans les cellules de lignée érythroïde adulte en bonne santé. En utilisant les cellules K562, la capacité de CLOuD9 à modifier l’expression génétique peut être examinée en tentant de restaurer l’expression du gène β-globine dans cette lignée cellulaire.
Avant induction de dimérisation, chromatine immunoprécipitation-quantitative PCR (ChIP-qPCR) a été utilisé pour assurer la localisation précise et le ciblage de chaque composant de CLOuD9 (supplémentaire Figure 1). En outre, co-immunoprécipitation (Co-IP) avec et sans ABA vérifié dimérisation CSA et CSP en présence du ligand comme réversibilité en l’absence du ligand (Figure 1C et 2 supplémentaires). 24h après l’ajout d’ABA, contact plus étroit entre la LCR et de β-globine est apparu tel que mesuré par la capture de conformation de chromosome (3C) dans les cellules avec les deux parties de dimérisation, mais pas les commandes contenant uniquement deux constructions CSA ou CSP, validant ainsi la spécificité de la modification de la chromatine pour les sites visés (Figure 1C et complémentaires Figures 3, 4). Création de l’interaction de la LCR-β-globine n’éliminait pas complètement le contact LCR-globine endogène, mais au lieu de cela, ajouté au contact, comme indiqué précédemment8. Augmentations dans les contacts de la β-globine/LCR ont été observées pendant 72 h de dimérisation, quelle que soit la région exacte au sein de la région promotrice ciblée LCR et la β-globine (chiffres supplémentaires 5,6). Enfin, la réversibilité du système a été confirmée avec 3C après avoir enlevé l’ABA, qui ont montré un renouvellement complet de la conformation endogène (Figure 1D et Figures supplémentaires 3-6).
On considère que le succès apparaît dans les cellules K562 pourraient être le résultat de l’emplacement de locus de globine dans une région d’euchromatine (Figure 1D), donc une deuxième ligne de la cellule a été utilisée pour étudier cette idée. Le système de CLOuD9 a été appliqué aux cellules 293 t HEK dans régions hétérochromatiques et n’expriment pas les gènes de la globine (Figure 1e). Le résultat était similaire à ce qui a été observé dans la K562s ; des associations de β-globine LCR ont été mesurées par 3C après 24h avec ABA (Figure 1e et supplémentaire Figure 3), fournissant des preuves de capacité robuste de CLOuD9 de fonctionner dans des environnements cellulaires différents, malgré l’état de la chromatine original ou conformation.
Autres loci ont été testés pour s’assurer de l’applicabilité large de CLOuD9, y compris le promoteur Oct4 et un exhausteur distale 5' dans les cellules 293 t. Auparavant, il a été sans expression Oct4 détectable dans cette lignée cellulaire et en outre, aucun contact endogène décrit. Preuve d’expression Oct4 dans les cellules souches embryonnaires résultant d’un contact avec le renforceur distale 5' motivé cette expérience, et le même résultat a été observé à la β-globine locus18. Contact entre le Oct4 promoteur et exhausteur de distale a été identifié dans les cellules de CLOuD9 activé, mais pas les cellules de contrôle (Figure 1f). En outre, il a été observé que le promoteur Oct4 et distale 5' interaction enhancer a également suscité un exhausteur de 3' à contacter le promoteur Oct4. Cet événement est conforme à la preuve que l’amplificateur de 3' interagit avec le promoteur Oct4/5 ' complexe au cours de l' activation de gènes endogènes10distale enhancer.
CLOuD9 induit des altérations spécifiques de contexte au locus. Après avoir vérifié que le système de CLOuD9 n’induit en effet chromosomiques contacts au locus, nous avons cherché à examiner effets des boucles sur l’expression génique. Il a été établi que la transcription de la globine et gènes Oct4 sont subordonnées sur les contacts entre les LCR et la globine locus et entre le rehausseur de distale 5' et le promoteur Oct4, respectivement1,11. Ainsi, nous avons émis l’hypothèse qu’en utilisant le CLOuD9 système de formation de boucle de la chromatine en voiture dans chacune de ces régions se traduirait par l’irrésistible l’expression des gènes.
Dans les deux loci, RT-qPCR a démontré que la chromatine ABA induit une boucle a conduit des augmentations Oct4 expression dans les cellules 293 t et dans l’expression de la β-globine dans les cellules K562, mais pas dans 293Ts (Figure 2 a). Bien que l’ajout de l’ABA dans la cellule culture pour aussi peu que 24 expression h β-globine accrue considérablement, expression a continué à augmenter régulièrement jusqu'à 72 heures et était réversible après lavage ABA (Figure 2 a). Toutes les cellules K562 sauf pour les contrôles de suivi cette tendance, peu importe où les composants de dimérisation étaient situés dans les régions promotrices LCR et de β-globine (Figure 2 a et complémentaires Figures 7, 8). À l’appui de ces conclusions, ChIP-qPCR de H3K4me3 et RNA Pol-II au locus β-globine chez K562s et 293Ts a correspondu avec des altérations observées dans la transcription (Figure 2c-f).
CLOuD9 établit des boucles de chromatine stable. Bien qu’à court terme induction boucle avec CLOuD9 suivi clairement les attentes, si induction à long terme du bouclage avait des effets différentiels sont restés à observer. Pour étudier cela, les cellules ont été cultivées en présence de l’ABA pendant 10 jours. Tandis que les K562s et les 293Ts étaient plus grande fréquence de contact entre le locus de β-globine et la LCR par rapport aux témoins (Figure 3 a, b et Figures complémentaires 9,10), étaient encore seulement observé des altérations des expression de la β-globine dans les cellules K562 (Figure 3C). Fait intéressant, cependant, on a constaté qu’à long terme une dimérisation dans K562s, où transcription a fortement augmenté, n’était aucun plus longue réversible (Figure 3 a et complémentaire Figures 9-11). Toutefois, en 293Ts, où aucune altération dans la transcription a été observée après dimérisation à long terme, provoquent des modifications dans les contacts de la chromatine restés réversibles (Figure 3 b et supplémentaires Figure 9).
Dans l’ensemble, seulement une faible diminution de l’expression des gènes a été observée après 10 jours de retrait de ABA, ce qui est resté significativement plus élevés que les niveaux d’expression de gène avant dimérisation (Figure 3C). Conformément à cela, cellules K562, mais pas les cellules 293 t, ont montré des altérations soutenues dans H3K4me3 et RNA Pol-II au locus β-globine par ChIP-qPCR, par rapport aux témoins, même après 10 jours de l’enlèvement de l’ABA (Figure 3d-f). Par conséquent, nos résultats indiquent que la stabilité de la boucle de la chromatine implique plus l’expression des gènes durable.
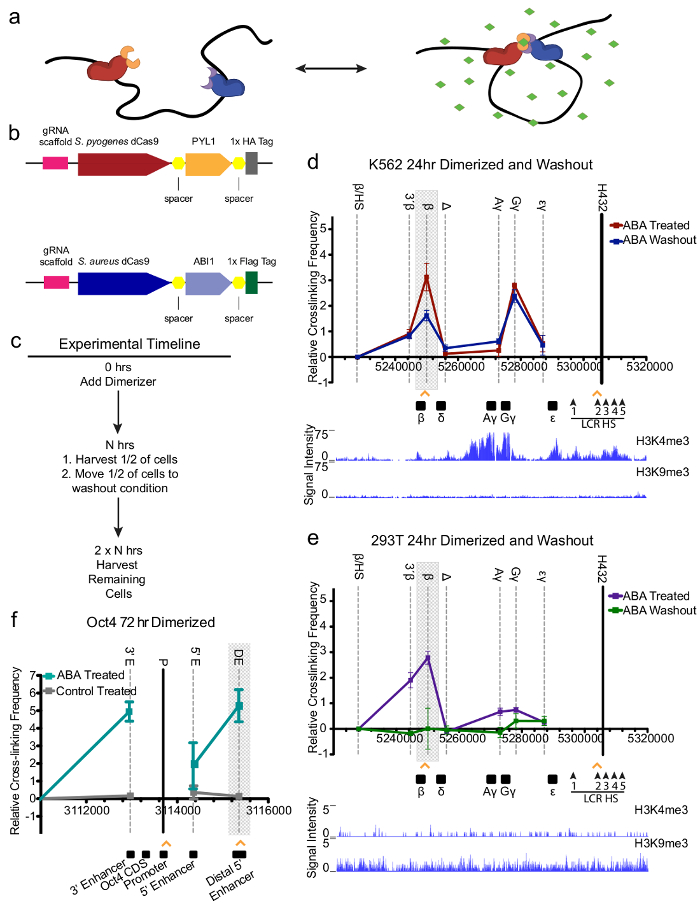
Figure 1: CLOuD9 induit réversible β-globine promoteur-LCR en boucle. (a) ajout d’acide abscissique (ABA, vert) a conduit deux concepts complémentaires de CLOuD9 (CLOuD9 s. pyogenes (CSP), CLOuD9 S. aureus (CSA), rouge et bleu, respectivement) en proximité, transformant la structure de la chromatine. Suppression de l’ABA restaure la conformation de la chromatine endogène. (b) CLOuD9 constructions allient technologie CRISPR-dCas9 de S. aureus et S. pyogenes réversiblement dimerizeable domaines PYL1 et ABI1. (c) chronologie de CLOuD9 dimérisation des expériences. (d) 3 C de dosage mesure β-globine réticulation de locus à l’échelle des fréquences dans les cellules K562 après 24 h de traitement à l’ABA (rouge) et lavage ultérieur (bleu) montrant la réversibilité des induits par contacts de β-globine/LCR (affichés en gris). Pointes de flèche orange indiquent des régions spécifiques CLOuD9 construct cible. Le fragment EcoRI contenant une hypersensibilité des sites 1 à 4 de la LCR (barre noire) a été utilisé comme la région de l’ancre. Sa fréquence de réticulation avec autres fragments de EcoRI indiqués (noms sur le dessus de la graphique) ont été évaluées. Les gènes de la β-globine humaine et les sites d’hypersensibilité LCR sont représentés sur la partie inférieure du graphique avec coordonnées de position chromosomique. Données de ChIP-seq de H3K4me3 et H3K9me3 montrent que cette région est euchromatique K562s. (e) comme changements réversibles dans la structure de la chromatine sont vus dans les cellules HEK 293 t, malgré des données H3K4me3 et H3K9me3 ChIP-seq qui la globine la région est hétérochromatique dans ce type de cellule. (f) 3C analyse mesure Oct4 réticulation de locus à l’échelle des fréquences en 293 t cellules après que 72 h de traitement à l’ABA (rouge) montrant induit Oct4/distale contacts enhancer (affichés en gris). Pointes de flèche orange indiquent des régions spécifiques CLOuD9 construct cible. Le fragment MboI contenant le promoteur Oct4 (barre noire) a été utilisé comme la région de l’ancre. Sa fréquence de réticulation avec autres fragments de MboI indiqués (noms sur le dessus de la graphique) ont été évaluées. Les régions Oct4 humaines sont représentées sur la partie inférieure du graphique avec coordonnées de position chromosomique. Tous les résultats de 3C proviennent d’au moins trois expériences indépendantes. 3C valeurs ont été normalisées de tubuline. Pour la β-globine, fréquences d’interaction entre le fragment de l’ancre et le fragment qui englobe le fragment β/HS ont été fixées à zéro. Pour Oct4, fréquences de l’interaction entre le fragment d’ancrage et un fragment de contrôle négatif à l’extérieur de la région d’interaction Oct4 ont été fixées à zéro. Barres d’erreur indiquent s.d. n = 3. Ce chiffre a été modifié de la Figure 1 en Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
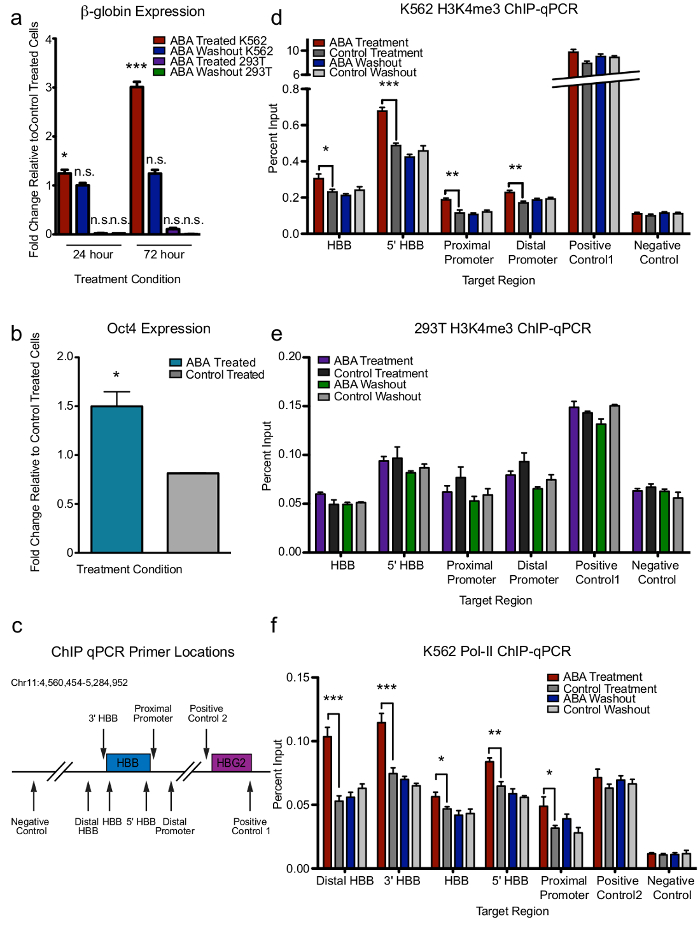
Figure 2: CLOuD9 induit des altérations spécifiques de contexte dans l’état de la chromatine et de l’expression génique. b chromatine CLOuD9 induite par le bouclage du locus de β-globine résulte en l’induction réversible d’expression de la β-globine en K562s mais pas en 293Ts. signification donnée par rapport aux hématies traitées. De l’étudiant à deux t-tests * P < 0,05, t = 3.418, df = 5 ; P < 0,0001, t = 10,42 df = 5 ; n.s. non significative. Barres d’erreur indiquent s.d. n = 3. (b) Induction de Oct4 expression a été observée en 293Ts la suite CLOuD9 induite par une boucle au même locus. Signification est donnée par rapport aux hématies traitées. De l’étudiant à deux t-tests * P < 0,05, t = 4.562, df = 2. Barres d’erreur indiquent s.d. (c) représentation schématique du ChIP-qPCR emplacements de primer le long du corps du gène β-globine. (d, e) Puce-qPCR montre réversibles altérations H3K4me3 du locus de β-globine en K562s mais pas en 293Ts suite CLOuD9 induite par le bouclage. De l’étudiant à deux t-tests * P < 0,05, ** P < 0,001, *** P < 0,0001. Barres d’erreur indiquent s.d. (f) CLOuD9 médiée par des altérations dans la transcription de la β-globine dans K562s correspondent aux augmentations en occupation RNA Pol-II à travers l’ensemble de l’organisme du gène β-globine. De l’étudiant à deux t-tests * P < 0,05, ** P < 0,001, *** P < 0,0001. Barres d’erreur indiquent s.d. Ce chiffre a été modifié par la Figure 2 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
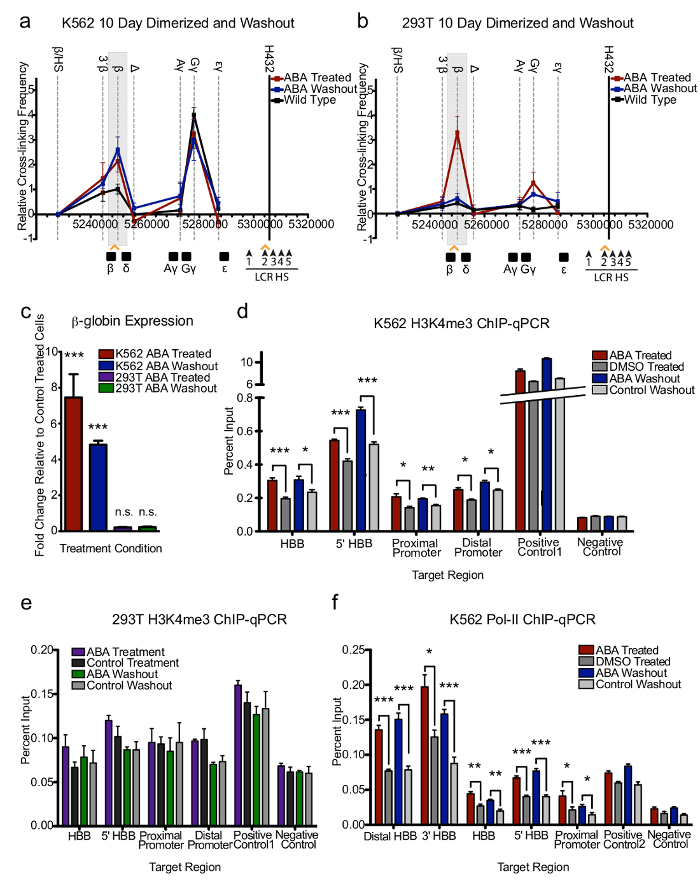
Figure 3: CLOuD9 établit des boucles de chromatine stable qui soutiennent l’expression des gènes robuste après dimérisation à long terme. (a, b) 3C test montre que dans K562s mais pas 293Ts, chromatine CLOuD9 induite par une boucle devienne irréversible après 10 jours de traitement à l’ABA, même si l’ABA est retiré pendant jusqu'à 10 jours supplémentaires. Tous les 3C obtenus depuis au moins trois expériences indépendantes. 3C valeurs ont été normalisées à la tubuline et fréquences de l’interaction entre le fragment de l’ancre et le fragment qui englobe le fragment β/HS ont été mis à zéro. Barres d’erreur indiquent s.d. n = 3. (c) stabilisation de boucle en K562s entraîne une expression persistante de β-globine, même après 10 jours de sevrage de l’ABA. Aucun changement dans l’expression de la β-globine n’est observées chez 293Ts. signification donnée par rapport aux hématies traitées. De l’étudiant à deux t-essais *** P < 0,0001, t = 5.963, df = 5 ; augmentation non significative (d) ChIP-qPCR montrant n.s. H3K4me3 marques sur le locus de β-globine en réponse à CLOuD9 induite par le bouclage se nourrissent après 10 jours de sevrage de ligand dans K562s. bilatéral de student t-tests * P < 0,05, ** P < 0,001, *** P < 0,0001. (e) aucune altération significative en H3K4me3 des signaux à long terme qui suit dimérisation ont été observés par ChIP-qPCR en 293Ts. (f) occupation augmenté RNA Pol-II du locus β-globine après induction de boucle à long terme a été maintenue chez K562s après 10 jours de sevrage de ligand. De l’étudiant à deux t-tests * P < 0,05, ** P < 0,001, *** P < 0,0001. Toutes les barres d’erreur indiquent s.d. Ce chiffre a été modifié par la Figure 3 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Supplémentaire Figure 1 : localiser les constructions de CLOuD9 dans leurs régions cibles. Immunoprécipitation de la chromatine et quantitative PCR de CLOuD9 constructions montrent une localisation correcte à leurs locus génomiques prévues. Ce chiffre a été modifié par supplémentaire Figure 1 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour télécharger ce chiffre.
Supplémentaire Figure 2 : constructions de CLOuD9 associent réversiblement en réponse à un traitement ABA. Co-Immunoprécipitation démontrant l’association des protéines dCas9 suivants à 72 h de traitement à l’ABA est inversée suivantes qui suit 72 h de lavage de ligand. Ce chiffre a été modifié par supplémentaires Figure 2 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour télécharger ce chiffre.
Supplémentaire Figure 3 : traitement de contrôle n’induit aucune modification dans les contacts de la chromatine. Traitement avec le DMSO, agent de contrôle, pendant 24 heures n’induit aucune modification dans la conformation de la chromatine endogène par 3C dans deux cellules K562 ou HEK 293Ts. 3C valeurs ont été normalisées à la tubuline et fréquences de l’interaction entre le fragment de l’ancre et le fragment englobant le fragment β/HS ont été mis à zéro. Barres d’erreur indiquent SD. n = 3. Ce chiffre a été modifié par supplémentaire Figure 3 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour télécharger ce chiffre.
Supplémentaire Figure 4 : contrôle CLOuD9 transduites cellules ne montrent aucune altération dans le bouclage de la chromatine. Mise en scène deux CLOuD9 construit soit la LCR ou le promoteur de la β-globine n’induit aucun changement significatif dans la structure de la chromatine en 3C après un traitement ABA par rapport au traitement de la commande. 3C valeurs ont été normalisées à la tubuline et fréquences de l’interaction entre le fragment de l’ancre et le fragment qui englobe le fragment β/HS ont été mis à zéro. Barres d’erreur indiquent SD. n = 3. Ce chiffre a été modifié par supplémentaires Figure 4 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour télécharger ce chiffre.
Supplémentaire Figure 5 : boucle de chromatine CLOuD9 reste réversible après 72 heures de dimérisation. 3C test chez K562s montre la réversibilité de CLOuD9 induite par la β-globine/LCR contacts après 72 heures de traitement à l’ABA. 3C valeurs ont été normalisées à la tubuline et fréquences de l’interaction entre le fragment de l’ancre et le fragment qui englobe le fragment β/HS ont été mis à zéro. Barres d’erreur indiquent SD. n = 3. Ce chiffre a été modifié par supplémentaire Figure 5 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour télécharger ce chiffre.
Supplémentaires Figure 6 : CLOuD9 bouclage induite de la β-globine/LCR n'est pas touché par les site de cible de globine. Mise en scène CSA et CSP construit d’autres régions de la LCR ou la β-globine promoteur entraîne des changements réversibles semblables dans l’induction de la boucle par 3C après 72 heures de traitement à l’ABA. 3C valeurs ont été normalisées à la tubuline et fréquences de l’interaction entre le fragment de l’ancre et le fragment qui englobe le fragment β/HS ont été mis à zéro. Barres d’erreur indiquent SD. n = 3. Ce chiffre a été modifié par supplémentaires Figure 6 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour télécharger ce chiffre.
Supplémentaires Figure 7 : CLOuD9 induit des altérations dans l’expression des gènes sont soutenues indépendamment de site cible globine. Mise en scène CSA et CSP construit d’autres régions du promoteur β-globine et LCR n’a aucune incidence sur l’induction de l’expression génique après 72 h de dimérisation. Cependant, bien que certains d’incidence sur la force de l’expression des gènes suivants dimérisation de longue durée (10 jours) a été observée, des niveaux élevés de β-globine par rapport aux hématies traitées sont soutenue lavage de ligand ultérieurs suivants pendant 10 jours supplémentaires. Signification est donnée par rapport aux hématies traitées. p < 0,001, t = 10,25, df = 5 ; p < 0,0001, de gauche à droite t = 8.697, df = 6, t = 40,31, df = 7 ; n.s. non significative. Toutes les barres d’erreur indiquent SD Ce chiffre a été modifié par supplémentaires Figure 7 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour télécharger ce chiffre.
Supplémentaire Figure 8 : contrôle CLOuD9 transduites cellules ne présentent aucune altération dans l’expression de la β-globine. Mise en scène deux CLOuD9 construit soit la LCR ou le promoteur de la β-globine n’induit aucun changement significatif dans l’expression de β-globine après un traitement ABA par rapport au traitement de la commande. Signification est donnée par rapport aux hématies traitées. n.s. non significative. Ce chiffre a été modifié par supplémentaire Figure 8 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour télécharger ce chiffre.
Supplémentaires Figure 9 : traitement de contrôle à long terme n’induit aucune modification dans les contacts de la chromatine. Traitement avec le DMSO, agent de contrôle, pendant 10 jours n’induit aucun changement de conformation de la chromatine endogène par 3C dans deux cellules K562 ou HEK 293Ts. 3C valeurs ont été normalisées à la tubuline et fréquences de l’interaction entre le fragment de l’ancre et le fragment englobant le fragment β/HS ont été mis à zéro. Barres d’erreur indiquent SD. n = 3. Ce chiffre a été modifié par supplémentaires Figure 9 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour télécharger ce chiffre.
Supplémentaire Figure 10 : long-term CLOuD9 bouclage induite de la β-globine/LCR n'est pas touché par les site de cible de globine. Diriger le CSA et CSP construit d’autres régions de la LCR ou les résultats de promoteur de β-globine dans prolongée de même boucle induction comme en témoignent les 3C après 10 jours de traitement à l’ABA et 10 jours de lavage ligand ultérieures. 3C valeurs ont été normalisées à la tubuline et fréquences de l’interaction entre le fragment de l’ancre et le fragment qui englobe le fragment β/HS ont été mis à zéro. Barres d’erreur indiquent SD. n = 3. Ce chiffre a été modifié par supplémentaire Figure 10 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour télécharger ce chiffre.
Supplémentaire Figure 11 : constructions de CLOuD9 irréversiblement associent en réponse à un traitement à long terme ABA. Co-Immunoprécipitation démontrant la liaison irréversible des protéines dCas9 CSA et CSP après 10 jours de traitement à l’ABA et de 10 jours consécutifs de lavage de ligand. Ce chiffre a été modifié par supplémentaire Figure 11 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour télécharger ce chiffre.
Supplémentaire tableau 1 : liste des amorces séquences pour gRNAs, qRT-PCR, 3C et puce qPCR. Cette table a été modifiée de supplémentaire tableau 1 dans Morgan, Stefanie L., et al. 9. s’il vous plaît cliquez ici pour télécharger ce tableau
Discussion
The Most sont des étapes cruciales dans le bouclage de la chromatine de CLOuD9 : conception 1) ou en utilisant le gRNAs correct, médias 2) changeant tous les jours sur les cellules transduites de CLOuD9, y compris ABA ou le DMSO, 3) maintenir la fraîcheur de l’ABA et 4) exécution d’évaluations exactes et prudents de conformation de la chromatine.
Les limites de CLOuD9 résident principalement dans la capacité de concevoir des guides de la région cible de choix. ARN Guide effectuer la tâche importante de localiser les composants dCas9 de régions d’ADN cibles pour être mélanges et l’efficacité des guides reposent sur leur site de cible spécifique. Sans les composants appropriés gRNA, le CLOuD9 système ne sera pas en mesure de former de façon réversible induit des boucles. Ainsi, en concevant plusieurs guides pour chaque région d’intérêt et en répartissant les guides sur une région de 250-1000 bp, au moins un succès guide sera assuré. Guide emplacement est également partie intégrante des résultats précis. Il est important d’éviter les guides localisées dans les sites de fixation de facteurs de transcription ou d’autres régions critiques afin d’éviter des effets de fond tels que vers le haut ou vers le bas de la régulation de la transcription. En outre, la localisation précise de la construction de CLOuD9 peut influer légèrement de transcription du gène cible. Cela souligne l’importance de tester plusieurs paires de guides pour chaque région cible, pour identifier la paire plus robuste à des fins expérimentales. De plus, dans chaque paire de régions cibles, la construction CSA devrait être ciblée avec gRNAs pour Staphylococcus aureuset la construction CSP devrait être ciblée avec gRNAs pour S. pyogenes afin de cibler la spécificité.
Pour garantir des résultats précis et correcte dimérisation, il est également important de maintenir la fraîcheur des milieux cellulaires suite à la transduction des constructions CLOuD9. Changement de médias quotidiens et l’ajout de frais dimerizer (ou contrôle) s’assure que les constructions complémentaires restera à proximité et préserver la conformation de la chromatine altérée. En outre, garantissant l’ABA est frais et a été stockée correctement selon le protocole du fabricant (ouvert dans les 6 mois, conservés à froid, protégées de la lumière) est essentiel à l’obtention de résultats authentiques.
Notamment, la dimerizer ABA pour CLOuD9 a été utilisé avec les protéines de dimérisation ABI et PYL, plutôt que le plus couramment utilisé FRB et FKBP système. La nécessité d’une rapalog pour le système de la FRB/FKBP aurait limité l’applicabilité de CLOuD9, en raison de la toxicité pour les cellules cancéreuses. Le système alternatif d’ABI/PYL de contourner cette limitation, permettant effectivement CLOuD9 à être plus largement utilisable.
Collectivement, nous avons développé CLOuD9, une technologie unique et robuste qui peut par la force, mais réversible créer des contacts entre locus génomiques cible à longue distance. Grâce à induire la chromatine boucles, nous démontrons que CLOuD9 peut être utilisée pour modifier l’expression génétique dans le contexte cellulaire approprié. La capacité d’adaptation de la technologie permet l’étude sans restriction des interactions entre n’importe quel deux locus génomiques, sans nécessiter de connaissances préalables de la boucle des régions ou le parcours des mécanismes. En outre, réversibilité démontrée unique de CLOuD9 permet davantage l’examen des mécanismes en boucle dans la maladie et le développement. Alors que les effets sur la cible de la chromatine en boucle ont été clairement démontrés, il est encore d’être données qui offrent un aperçu sur les effets de bouclage hors cible et l’impact ultérieur sur les boucles sur la cible.
Nos données illustre seulement quelques applications de cet outil, mais implique l’idée majeure que l’arrangement de chromatine est révélateur de l’expression des gènes. Notre technologie permet d’étudier et de révéler les nuances de la structure de la chromatine dans la régulation des gènes, ce qui améliore la compréhension globale du rôle de la chromatine pliage dans la transcription des gènes. Une meilleure compréhension des subtilités de transcriptional dynamique peut montrer la voie dans la recherche et le traitement du cancer, les maladies héréditaires et congénitales, dans laquelle la chromatine distincte l’Assemblée modifie sans doute gene expression20, 21,22,23. Les travaux ultérieurs utilisant la technologie de CLOuD9 illuminera plus détails sur l’organisation et la dynamique des domaines de la chromatine et comment ils conduisent pliage pour soutenir l’expression des gènes stable dans le développement et la maladie.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions H. Chang, T. Oro, S. Tavazoie, R. Flynn, P. Batista, E. Calo et tout laboratoire Wang pour le support technique et de la lecture critique du manuscrit. S.L.M. a été soutenue dans ce travail par le biais de la NSFGRF (DGE-114747), NDSEGF (FA9550-11-C-0028) et le National Cancer Institute (1F99CA222541-01). K.C.W. est soutenu par une bourse de carrière pour les scientifiques médicaux du fonds Burroughs Wellcome et est un Donald E. et Delia B. Baxter Fondation Faculty Scholar.
matériels
| Name | Company | Catalog Number | Comments |
| RPMI 1640 media | Life Technologies | 11875-119 | For K562 cell culture |
| DMEM media | Life Technologies | 11995-065 | 1X, for 293T cell culture |
| lentiCRISPR v2 | Addgene plasmid | #52961 | For CLOuD9 plasmid development |
| pRSV-Rev | Addgene plasmid | #12253 | For lentivirus production |
| pMD2.G | Addgene plasmid | #12259 | For lentivirus production |
| pMDLg/pRRE | Addgene plasmid | #12251 | For lentivirus production |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668-019 | For lentivirus production |
| anti-HA antibody | Cell Signaling | 3724 | For immunoprecipitation |
| anti-Flag antibody | Sigma | F1804 | For immunoprecipitation |
| DNeasy Blood and Tissue Kit | Qiagen | 69504 | For DNA extraction |
| TRIzol | Life Technologies | 15596-018 | For RNA extraction |
| RNeasy Kit | Qiagen | 74106 | For RNA extraction |
| Superscript VILO | Life Technologies | 11754-050 | For cDNA |
| SYBR Green I MasterMix | Roche | 4707516001 | For qPCR analysis |
| Light Cycler 480II | Roche | For qPCR analysis | |
| anti-H3K4me3 antibody | AbCam | ab8580 | For ChIP-qPCR |
| anti-RNA Pol-II antibody | Active Motif | 61083 | For ChIP-qPCR |
| EDTA free protease inhibitor | Roche | 11873580001 | For protein extraction |
| 4-12% Tris Glycine gel | Biorad | Any size, For western blot | |
| anti-Rabbit HRP antibody | Santa Cruz | sc-2030 | For western blot |
| anti-mouse HRP antibody | Cell Signaling | 7076S | For western blot |
| K562 and H3K293 ChIP-Seq data | Encode | ENCSR000AKU | For ChIP-seq analysis |
| K562 and H3K293 ChIP-Seq data | Encode | ENCSR000APE | For ChIP-seq analysis |
| K562 and H3K293 ChIP-Seq data | Encode | ENCSR000FCJ | For ChIP-seq analysis |
| K562 and H3K293 ChIP-Seq data | GEO | GSM1479215 | For ChIP-seq analysis |
| Dynabeads Protein A for Immunoprecipitation | Thermo Fisher Scientific | 10001D | For immunoprecipitation |
| Dynabeads Protein G for Immunoprecipitation | Thermo Fisher Scientific | 10004D | For immunoprecipitation |
| RNA Clean & Concentrator-5 | Zymo Research | R1015 | For RNA purification |
| Pierce 16% Formaldehyde Methanol-free | Thermo Fisher Scientific | 28908 | For crosslinking |
| PX458 Plasmid | Addgene | 48138 | Suggested active Cas9 plasmid for gRNA cloning, but any active Cas9 plasmid will do |
| QIAquick PCR Purification Kit | Qiagen | 28104 | For PCR purification |
| FastDigest BsmBI | Thermo Fisher Scientific | FD0454 | For cloning guide RNAs |
| FastAP | Thermo Fisher Scientific | EF0651 | For cloning guide RNAs |
| 10X FastDigest Buffer | Thermo Fisher Scientific | B64 | For cloning guide RNAs |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | For cloning guide RNAs |
| 10X T4 Ligation Buffer | NEB | B0202S | For cloning guide RNAs |
| T4 PNK | NEB | M0201S | For cloning guide RNAs |
| 2X Quick Ligase Buffer | NEB | B2200S | For cloning guide RNAs |
| Quick Ligase | NEB | M2200S | For cloning guide RNAs |
| Buffers | |||
| Farnham lysis buffer | 1% Tris-Cl pH 8.0, 1% SDS, 1% protease inhibitor water solution (non-EDTA), and 1 mM EDTA in water | ||
| Modified RIPA buffer | 1% NP40/Igepal, 0.5% sodium deoxycholate, 0.1% SDS, 1 mM EDTA, and 1% protease inhibitor water solution (non-EDTA) in PBS pH 7.8 or 7.4 | ||
| IP dilution buffer | 0.01% SDS, 1.1% Triton-X 100, 1.2 mM EDTA, 16.7 mM Tris-HCl pH 8.0, 167 mM NaCl, 0.1x protease inhibitor | ||
| Wash buffer | 100 mM Tris pH 9, 100 mM LiCl, 1% NP-40, and 1% sodium deoxycholate | ||
| Swelling buffer | 0.1 M Tris pH 7.5, 10 mM potassium acetate, 15 mM magnesium acetate, 1% NP-40 | ||
| Dilution buffer | 0.01% SDS, 1.1% Triton X-100, 1.2 mM EDTA, 16.7 mM Tris pH 8 and 167 mM NaCl | ||
| IP elution buffer | 1% SDS, 10% NaHCO3 |
Références
- Krivega, I., Dean, A. Chromatin looping as a target for altering erythroid gene expression. Ann. N.Y. Acad. Sci. 1368, 31-39 (2016).
- Matharu, N., Ahituv, N. Minor loops in major folds: Enhancer-promoter looping, chromatin restructuring, and their association with transcriptional regulation and disease. PLoS Genet. 11, e1005640 (2015).
- Dekker, J., Martí-Renom, M. A., Mirny, L. A. Exploring the three-dimensional organization of genomes: interpreting chromatin interaction data. Nat. Rev Genet. 14, 390-403 (2013).
- Kim, A., Dean, A. Chromatin loop formation in the b-globin locus and its role in globin gene transcription. Mol Cells. 34, 1-5 (2012).
- Petrascheck, M., et al. DNA looping induced by a transcriptional enhancer in vivo. Nucleic Acids Res. 33, 3743-3750 (2005).
- Ameres, S. L., et al. Inducible DNA-loop formation blocks transcriptional activation by an SV40 enhancer. EMBO J. 24, 358-367 (2005).
- Deng, W., et al. Controlling Long-Range Genomic Interactions at a Native Locus by Targeted Tethering of a Looping Factor. Cell. 149, 1233-1244 (2012).
- Deng, W., et al. Reactivation of developmentally silenced globin genes by forced chromatin looping. Cell. 158, 849-860 (2014).
- Morgan, S. L., et al. Manipulation of Nuclear Architecture through CRISPR-Mediated Chromosomal Looping. Nature Communications. 8, (2017).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biol. 17 (1), 148 (2016).
- . . Lipofectamine 2000 Reagent. , (2013).
- . . DNeasy Blood & Tissue Kit Handbook. , (2006).
- . . Determining Genome Targeting Efficiency using T7 Endonuclase I (M0302). , (2016).
- Guschin, D. Y., et al. A rapid and general assay for monitoring endogenous gene modification. Methods Mol Biol. 649, 247-256 (2010).
- Shalem, O., Sanjana, N. E., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. , 83-87 (2014).
- . . RNeasy Mini Handbook. , (2012).
- . . SuperScript VILO cDNA Synthesis Product Information Sheet. , (2015).
- Theunissen, T. W., et al. Systematic identification of culture conditions for induction and maintenance of naive human pluripotency. Cell Stem Cell. 15, 471-487 (2014).
- Mokry, M., et al. Integrated genome-wide analysis of transcription factor occupancy, RNA polymerase II binding and steady state RNA levels identify differentially regulated functional gene classes. Nucleic Acids Res. 40, 148-158 (2012).
- Drier, Y., et al. An oncogenic MYB feedback loop drives alternate cell fates in adenoid cystic carcinoma. Nat Genet. 48, 265-272 (2016).
- Ryan, R. J. H., et al. Detection of enhancer-associated rearrangements reveals mechanisms of oncogene dysregulation in B-cell lymphoma. Cancer Discov. 5, 1058-1071 (2015).
- Montavon, T., et al. A regulatory archipelago controls Hox genes transcription in digits. Cell. 147, 1132-1145 (2011).
- Lupianez, D. G., et al. Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions. Cell. 161, 1012-1025 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon