Method Article
クロマチンのループ構造の再編を CRISPR 媒介
* これらの著者は同等に貢献しました
要約
遺伝子発現制御に重要な役割を果たしているクロマチンのループただし、クロマチンのループの選択的かつ可逆的な変更を可能にする技術の進歩はいません。ここでクロマチンのループの再編成のための強力なシステムを記述対象遺伝子座に CRISPR dCas9 (CLOuD9) を使用して、選択的かつ可逆的に遺伝子発現を調節することが実証します。
要約
最近の調査示した、長距離、三次元クロマチンのループの相互作用の再生がループが担当かどうかまたは遺伝子発現の変化の結果、遺伝子発現の調節に重要な役割はまだ明らかに不明です。最近まで、比較的あいまいなされている遺伝子活性と細胞機能の調節のしくみクロマチンのループおよびこれらの構造体を操作する既存のメソッドの制限を防ぐこれらの相互作用の詳細な調査。この不確実性を解決するには、我々 は選択的かつ可逆的なクロマチン ループ再構成法を設計 CRISPR dCas9 (CLOuD9) を使用します。CLOuD9 システムのダイナミズムは、CLOuD9 の構成要素がローカル クロマチン構造を調節する遺伝子座をターゲットのローカリゼーションの成功によって実証されています。重要なは、逆に誘導の接触および内因性クロマチン構造を復元する機能が確認されています。この方法で遺伝子発現の調節細胞の遺伝子発現を調節する能力を確立し、安定したde novo遺伝子に著しく影響するクロマチンのループの作成にこの技術の応用のための大きな可能性を強調がんと開発のコンテキストにおける式。
概要
クロマチン核ゲノムの特定の組織の折りたたみとの関係は、遺伝子式1,2と密接に関連することが示されていると近年、大きな関心を集めています。動的三次元クロマチン組織の結果として染色体の連絡先との相互作用を提供する一方、遺伝子活動とクロマチン構造の変調との正確な関係は不明のまま、えられています、遺伝子調節機能3。確かに、そのような効果は人間グロビン遺伝子座、軌跡制御領域 (LCR) が 2 つの領域4間クロマチンのループを作成することによって発達の特定の方法でグロビン遺伝子の活性を制御するどこでも実証されています。ただし、両方のこれとその他の地域は、それは不明原因や遺伝子発現の変化の結果がクロマチンのループかどうか。
今までは、この現象を勉強の課題未解決の。たとえば、クロマチンのループを誘発する他の試みに関与線形 DNA シーケンスまたは豊富なループ5,6、容易にするための特定の要素に関する背景知識を必要とする複雑な手続きを変更します。 7,8。さらに、前作は、クロマチンのループ ドライブ特定と制限されたコンテキスト7,遺伝子の発現が示唆された8がクロマチンのループに影響を与える転写グローバル レベルはあります。遺伝子発現に対する長期的ループの影響で関心は、近年継続的に成長している、けれども確立と遺伝子活動を変更するクロマチン連絡先を維持についての疑問が保持されます。
我々 は、設計技術をすべてのゲノムの軌跡9対象に広く適用できるようにヌクレアーゼ欠損クラスター化された空間定期的に短い回文繰り返し (CRISPR) - CRISPR - 関連付けられている蛋白質 9 (dCas9) を採用しています。この技術は線形 DNA シーケンスの変更に関連する複雑な問題を排除する、特定のコンポーネントがループの重要な事前の知識なしでアクセスできます。特に、ツールはユニバーサルとクロマチンのループが開発にさまざまながんなどの病気の認識に広く適用。CLOuD9 の力を発揮して、元に戻せる効果的に遺伝子発現を調節するループの構造を変えることによって。
プロトコル
1. gRNA デザイン
- ガイド Rna10をデザインする標準的な gRNA デザイン ツールを選択します。開発済み CLOuD9 構造9の相補的なペアを使用して (すなわち、少なくとも 1黄色ブドウ球菌(CSA) および 1 s. 化膿(CSP) を構築) 各実験のため。
- 選択したオンライン デザイン ツール内でガイド長さ 20 の Protospacer 隣接するモチーフ (PAM) シーケンス"NGG"を使用して、CSP と PAM の bp は、CSA の 21 のガイドの長さで"NNGRRT"をシーケンスします。
- 選択ガイドの特異性に基づくスコアし、設計の作業ガイドを得ることのチャンスを最大化する両方のストランドのガイド。
- オリゴ メーカーからガイドあたり 2 オリゴヌクレオチドを注文: 前方の oligo の"caccg"5' 端にガイド シーケンスと逆のオリゴを追加、5' 端と 3' 端に"c"を注文する前に「aaac」を追加。
- 当初は、250-1000 bp 地域に広がって、興味の地域ごとの 3-5 gRNAs をデザインします。効率を次のようにターゲット gRNA を評価します。
- アクティブの Cas9 プラスミドにクローン識別 gRNAs (手順 3 参照) 293 t 細胞 (手順 4. を参照) に transfect 一過性と11。
- 2-3 d でダルベッコ変更イーグル培地 (DMEM) 10% 牛胎児血清 (FBS) と 1% ペニシリン-ストレプトマイシン (ペン連鎖球菌) のための細胞を成長し、収穫し、ゲノム DNA12を抽出します。
- ポリメラーゼの連鎖反応 (PCR) によって対象となる領域を増幅します。1.5% の agarose のゲルの製品を実行し、適切なバンドを浄化します。
- Guschin で説明したように T7 エンドヌクレアーゼ分析を実行ら。プロトコル13,14。
注: 効率的なガイドでは、ターゲット ・ サイトで DNA を切るために定めします。
2. 細胞培養
- 健康的で活発に分裂状態の細胞を維持します。
- K562s、10 %fbs と 1% ペン連鎖球菌でロズウェル パーク記念研究所 (RPMI) 1640 メディア文化。
- メンテナンスのため 25 cm2斜め首フラスコに K562s を成長し、毎日 1 mL あたり 400,000 セルにセル密度を調整します。
- K562s で導入したされている CLOuD9 を構築、保守メディアに 2 μ G/ml ピューロマイシンと 100 μ g/mL hygromycin を追加します。
- 293Ts、文化でダルベッコ変更イーグル培地 (DMEM) 10 %fbs と 1% ペン連鎖球菌。
- 10 cm2板の 293Ts を成長し、合流するときに分割します。
- K562s、10 %fbs と 1% ペン連鎖球菌でロズウェル パーク記念研究所 (RPMI) 1640 メディア文化。
3. プラスミッドの準備と gRNA 挿入15
注: プラスミド マップ付録であり、例実験で利用されているプライマー、補足表 1で利用可能です。
- BsmBI、アルカリホスファターゼ、バッファー、x 10 の 6 μ L と作りたて 100 mM DTT の 0.6 μ L の 3 μ L の 3 μ L でレンチの CRISPR プラスミドのミックス 5 μ。DdH2O 60 μ L に総量を持参し、消化し、プラスミドを dephosphorylate に 30 分間の 37 ° C で孵化させなさい。
- ゲルの消化のプラスミッドの浄化し、には ddH2o.
- T4 結紮バッファー、ddH2O、6.5 μ L、0.5 μ L x 10 の 1 μ L で 100 μ M で一対のガイドのそれぞれのミックス 1 μ L T4 PNK、10 μ L の総ボリュームに到達します。37 ° c、5 分間、95 ° C、30 分間インキュベートし、ランプを 5 ° C/分で 25 ° C まで。
注: これはガイドのペアをアニールします。 - 希釈する ddH2O. 焼なましガイド (ステップ 3.3) から 1: 200
- ステップ 3.4、バッファー x、ddH2O、1 μ l のリガーゼ、0.5 μ L 5.5 μ L の全反応を作る 2 の 2.5 μ L から希薄結紮ガイドの 0.5 μ L で 3.2 から消化のプラスミッドのミックス 1 μ。これを消化 BsmBI プラスミドにガイドを縛ること 10 分間室温でインキュベートします。
- Stbl3 細菌にステップ 3.5 から新しく縛られたプラスミッドを変換し、増幅するプラスミッドの準備方法を使用します。
4. レンチ ウイルス生産
- 6 ウェルのウェルあたりのカウント 750,000 293 t 細胞診断をプレートし、10 %fbs と 1% ペン連鎖球菌で DMEM でそれらを播きます。各構成要素の 1 つの井戸を使用します。
- 10% の新鮮な抗生物質無料 DMEM にメディアを変更, 播種後 24 h FBS。
- 1 つの管で 150 μ L 反応11あたりの OptiMEM 中の脂質ベースのトランスフェクション試薬の 11 μ L を希釈します。
- 別のチューブでステップ 3.6、0.65 μ g pMD2.G の OptiMEM 中、反応11あたりの 150 μ L で、1 μ g pRSV-Rev の pMDLg/pRRE 0.35 μ g から CLOuD9 レンチウイルスベクターのプラスミッドの 2 μ g を希釈します。
- 各反作用のため 1:1 の比率で希釈した脂質ベースのトランスフェクション試薬を希釈レンチ ウイルス プラスミドの混合物を追加し、115 分間インキュベートします。
- 4.2 のステップから抗生物質無料 293 t 細胞のそれぞれの井戸へのステップ 4.5 から各混合複合体の全体のボリュームを追加します。
- 48 時間後、ダウン 5 分 300 x g で新鮮なチューブとスピンにピペッティングによるウイルス製造メディアを収集します。遠心分離の後残留細胞の残骸を削除する新鮮なチューブに上清を移動します。
- すぐに標的細胞の伝達ウイルス製造メディアを利用または将来使用するため-80 ° C で凍結します。
5. レンチ ウイルス細胞の伝達
- CLOuD9 申請 80,000 セルを含む 15 mL の円錐管に関心 (補完 CLOuD9 プラスミドで構成) の各ウイルス構築の 250 μ L を追加します。
- メディアの総容量は、各抗生物質フリーのメディアと 1 mL に円錐形 1-8 μ g/mL (polybrene 使用電池の種類によって許容の最大濃度は、実験的に決定する必要があります) の間の最終的な集中に polybrene を追加。
- 室温で 30 分間 800 x g でセルをスピンし、ウイルス上清を削除せずピペッティングによって再懸濁します。全体の細胞懸濁液を細胞培養プレートに移動します。
- 24 h 後伝達、室温で 5 分間 300 x g でダウン スピン細胞。
- ウイルス上清を吸引し、通常の細胞培養媒体の細胞を再懸濁します。
- 次の日に二重導入された細胞を選択する培ピューロマイシン (293 t 細胞を 1 μ g/mL) と hygromycin (293 t 細胞を 25 μ g/mL) を追加します。実験ピューロマイシン hygromycin 実験を開始する前に指定されたセルの種類の適切な濃度で判断します。
- 下流実験前に少なくとも 3 d 選択メディアの細胞を維持し、すべての実験の期間の選択メディアの維持します。
6. 細胞の活性化とを洗う
- 1 mM アブシジン酸 (ABA) を追加またはジメチルスルホキシド (DMSO) コントロール、CLOuD9 を dimerize に細胞培養のための同等のボリュームを選択し、セルを導入します。ABA を使用して、領収書の日付の 6 ヶ月以内、冷たい、維持、使用中の光から保護します。2 mM ABA は、必要な場合に利用できます。
- 毎日新鮮な ABA (またはコントロールの DMSO) 実験の持続期間のためにメディアを変更します。
注: 二量体化の長さの制限はまだ観測されていません。
- 毎日新鮮な ABA (またはコントロールの DMSO) 実験の持続期間のためにメディアを変更します。
- 逆にメディア ABA を含む十分なリン酸緩衝生理食塩水洗浄セル (PBS) 2 回、板の表面をカバーするための取り外しによって二量体化。その後、国連パイエルス状態を維持するために ABA 自由な媒体の細胞を培養します。
7. 免疫沈降と Co-immunoprecipitations
注: は、新鮮なすぐに使用する前にすべてのバッファーを作る。
- 二量体化の実験が完了した後、収集し、細胞、スピンダウン、上清を吸引します。
- クロスリンクと細胞を凍結
- 新鮮な素材を利用して常温で PBS で 1% のホルムアルデヒドの新鮮な在庫を作る。200 万セル全てに対して優しく再懸濁します丁度 10 分室温で 1% のホルムアルデヒドの 1 ml を回転させながら。
注: は、未満 1000 万セル架橋の 10 mL の最小量を使用します。 - 0.125 M、回転させながら、室温で 5 分間インキュベーション後の最終的な集中にグリシンを加えると架橋を癒します。
- 細胞をスピンし、氷冷 PBS で 1-2 x を洗ってください。細胞ペレット スナップ液体窒素で凍結し、-80 ° C で保存またはすぐに使用できます。
注: 熱ホルムアルデヒド クロスリンクが逆になります。急いで、スナップ凍結せず、セルは-80 ° C で直接格納できます。
- 新鮮な素材を利用して常温で PBS で 1% のホルムアルデヒドの新鮮な在庫を作る。200 万セル全てに対して優しく再懸濁します丁度 10 分室温で 1% のホルムアルデヒドの 1 ml を回転させながら。
- 抗体/ビーズ共役を準備します。
注: 選択した抗体 (すなわち、別の換散バッファーのセル換散のためのテストと IP を実行する) ための最適化条件は価値がある可能性があります。2 つの共通バッファー、ファーナムの換散バッファー変更 RIPA バッファーは IP 効率および超音波処理効率に大きな影響を持つことができます。すべてのバッファー組成は、表の材料で見つけることができます。また、覚えておいて、クロマチン超音波処理は完了まで数時間かかる場合があります。- ビーズを選ばれた抗体を必要に応じて簡単にボルテックスによって再懸濁します。
- 実験用ビーズの適切な量を 1.5 mL チューブに転送します。開始点として抗体のすべて 1 μ g のビーズの 20 μ L を使用します。抗体はまだ追加しないでください。
注: は、抗体がこれが意味するだろうよりももっとまたはより少なく効率的な知られている限り、ビーズの 200 μ L で開始点として溶解液 1 mg の 10 μ g 抗体が使用します。低豊富な蛋白質のためこの比率は調整する必要があります。 - ファーナムの換散バッファーを使用して、IP 希釈バッファーで 3 x を洗います。RIPA 溶解バッファーを使用している場合は、RIPA 溶解バッファーで 3 x を洗います。
- (IP 希釈または RIPA バッファー) 7.3.3 のステップから同じバッファーの最終巻でのビードを再停止しなさい > 1 x と < ボリュームの初期 x 5。すなわち100 μ L 初期ビーズ ボリューム 100 と 500 μ L の最終的な再懸濁のボリュームを使用します。
- 適切な濃度で洗浄ビーズに抗体を追加します。良い HA またはフラグ抗体を IP CLOuD9 構成、1:50 で抗体を使用 (μ g タンパク質: μ g 蛋白ライセート) 8 + h と 4 ° C で回転させながら一晩の間孵化させなさいと。また、室温で 2-4 時間インキュベートします。
- ビーズから上澄みを削除し、破棄します。
- 洗浄は、洗浄バッファーで 3 x をビーズします。
- 一晩インキュベート (IP 希釈または RIPA バッファー) 抗体の利用バッファーの最小ボリュームに再懸濁します。ライセートの蛋白質と結合するまで寒さを維持します。
- クロマチンを超音波照射します。
- 最初 10 分間氷の上細胞ペレットにバッファーを腫れの細胞を溶解、沈降を防ぐためにすべてのいくつかの分を細胞にフリックします。
注: 一般に、500 μ L のバッファーを膨潤を 2500 万セルに追加され、そこから、拡大/縮小、か 500 μ L では未満使用しないバッファーの < 2500 万セル。 - Dounce は時計回りと反時計回りの両方手動で各 13。
- 1,500 x g に 5 分の 4 ° C でダウン スピン核、上清を完全に吸引し残りの上清を除去するを繰り返し、mg 細胞ペレットの重量を量る。
- プロテアーゼ阻害剤を核の換散バッファーに追加 (ファーナム バッファーまたは RIPA バッファー上、いずれかを選択)。
- 再懸濁しますに小球形にされた核に核換散バッファーの 10 の x 量を追加 (0.100 g ペレットを 1 mL の核溶解バッファーを追加するなど)。簡単に渦と氷で 10 分間溶解します。
注意: は超音波処理管のサイズに注意します。理想的なボリュームは多くの場合、< 1 mL のサンプルは、ペレットが非常に大きい場合に分割する必要がありますので。- Co - ip は、材料を可溶化し希釈バッファーのボリュームを 2-3 で免疫沈降が許可されるレベルに SDS を消す簡単に核を超音波照射し、7.4.6 のステップへ進みます。非常に敏感な抗体は、SDS の濃度をさらに削減するより多くの希釈バッファーを追加します。
注: 超音波処理を停止は、ライセートをクリア。それは完全に透明、不透明・半透明白から変更されます。サンプル可能のように冷たいしはない以上超音波。 - チップは、徹底的に 150-1000 bp の DNA のフラグメントを取得する DNA を超音波、7.4.6 のステップへ進みます。
- Co - ip は、材料を可溶化し希釈バッファーのボリュームを 2-3 で免疫沈降が許可されるレベルに SDS を消す簡単に核を超音波照射し、7.4.6 のステップへ進みます。非常に敏感な抗体は、SDS の濃度をさらに削減するより多くの希釈バッファーを追加します。
- 4 ° C で 10 分間の最大速度で不溶性物質をスピンします。
- 新鮮な管に溶ける材料を転送します。
- Co - ip は、タンパク質の濃度を測定し、7.5 の手順でプルダウンに進みます。
- チップ、削除の 10 μ 因数ライセートをクリアし、40 μ L の IP 溶出バッファーと 6 μ L 5 M 追加 56ul の合計のために塩化ナトリウム。95 ° C で 15 分間沸騰 3 M NaOAc ph 5、5-10 μ L を追加し、PCR 精製カラムのクリーンアップします。260 で吸光度を測定することによって DNA の濃度の測定 nm サンプル全体で標準化と。7.5 の手順でプルダウンに進みます。
注: 余分な新鮮なライセートから最高の結果が得られますが、スナップ-80 ° C で凍結し、使用できます後でライセート。場合は、凍結はないフリーズ/フリーズ解除ライセートより複数回。
- 最初 10 分間氷の上細胞ペレットにバッファーを腫れの細胞を溶解、沈降を防ぐためにすべてのいくつかの分を細胞にフリックします。
- プルダウン
- 新鮮なチューブに蛋白ライセートの適切な量を転送します。ファーナムの換散バッファーを使用している場合は、IP 希釈バッファー (上記) の 2.1 ボリュームで希釈します。
- 脇に入力コントロールの上澄みを 100 μ l 添加し翌日まで 4 ° C で保存します。
- 7.3.8 のステップからのビーズ/抗体の共役をサンプルを今すぐ追加します。
- 一晩で 4 ° C のサンプルを回転させるし、1.5 mL チューブに液体の動きに十分なボリュームがあることを確認します。
- 洗うし、溶出
- 一晩回転後法的拘束力の一部として上清を保存します。その後、洗浄ビーズ IP 希釈バッファーで 3-5 x です。
- 室温、阻害剤なしで IP 溶出バッファーを準備します。
- 新しい管へ 15 分転送溶出 67 ° C で渦に動揺 50 μ L 溶出バッファーで溶出が錯体と繰り返し別 50 μ L。 それらの両方合計 100 μ L の溶出。
注: この溶出プロシージャからあまりにも多くのバック グラウンドがある場合熱することができますを削減または排除揺れ/孵化中。これは一般的に標的タンパク質の収量が減少が、バック グラウンドを大幅に削減は。
- 67 ° C で加熱することにより逆にクロスリンク > 4 h。
注: これは共同 IPs 厳密に必要はありませんが、役立つことができます。- Co-IP の興味のターゲット間におけるダイマー形成を強固にする、実行波 SDS ページのゲルとフラグの札または HA タグに対する抗体をプローブ 1: 1,000 で示されているように。手順 8 に進みます。
- チップ qPCR、正しいローカリゼーションおよび各 CRISPR dCas9 コンポーネントのターゲットを確実にきれいに列に溶出製品と 10 μ L。 実行リアルタイムの量的なポリメラーゼの連鎖反応 (qPCR) 精製 DNA に。補足表 1のプライマー、表示されているデータの活用があります。手順 8 に進みます。
8. RNA の抽出および量的な PCR
- CLOuD9 による遺伝子発現の変化を調べるためには、まず、分離、制御細胞ペレットとパイエルス細胞ペレットの両方からの総 RNA の浄化します。
- 逆のトランスクリプション17によって浄化された RNA から相補的 DNA (cDNA) を作るし、qPCR 解析を実行します。プライマーは、補足表 1にあります。
9 染色体構造の捕獲の試金
- CLOuD9 によるゲノム遺伝子座接触の頻度の変化を観察してを染色体構造キャプチャ (3 C) を実行するには、8の試金します。
- 定量的リアルタイム PCR による 3 つの生物学的複製のそれぞれの重複の 2 つのセットで 3 C 結紮製品を定量化します。チューブリンの軌跡から 3 C 信号にサンプルを正規化します。プライマーは、補足表 1にあります。
結果
CLOuD9 誘導リバーシブル β-グロビン プロモーター-LCR ループします。CLOuD9 システムの適切な使用は補完の CSA の可逆接触を誘発し、CSP CLOuD9 を追加または細胞培養媒体 (図 1 a) に ABA の除去を構築します。CSA および CSP 構造 (図 1 b) は、標準の CRISPR gRNAs を使用して適切なゲノム領域にローカライズされます。考えると人間のグロビン遺伝子座と同様、頻繁に染色体を折るとが開発中に発生する転位の膨大なドキュメント、CLOuD9 システムの有用性を実証するこの地域が選ばれました。また、k562 細胞は、一貫して健康的な成人の赤血球系細胞では、通常表現 β グロビン遺伝子ではなく、胎児 γ グロビン遺伝子の高レベルを表現する示されているために選ばれました。K562 細胞を使用して、この細胞における β-グロビン遺伝子の発現を復元しようとして CLOuD9 が遺伝子発現を変更できる機能を調べることができます。
ダイマー化、クロマチン免疫沈降定量的 PCR (チップ qPCR) を用いている定位と CLOuD9 の各コンポーネントのターゲットを確保するための誘導の前に (補足図 1)。さらに、co 法 (Co IP) ABA とは、配位子として配位子 (補足図 2と図 1 c ) の不在で可逆性の存在下での CSA と CSP のダイマー化を検証しました。ABA を追加した後 24 h β-グロビンと、LCR の大きい接触登場両方の二量体化の部分を細胞内染色体構造キャプチャ (3 C) によって測定されるが、2 つだけ CSA または CSP 構造を検証を含むコントロールではなく、(図 1 cと補助図 3, 4) は、ターゲット サイトのクロマチンの変化の特異性。LCR-β-グロビンの相互作用を作成する内因性の LCR グロビン接触を完全に排除していないが、代わりに、以前に報告された8として、元の連絡先を追加しました。72 h までターゲットを絞った LCR および β-グロビン プロモーター領域 (補足図 5, 6) 内で正確な地域に関係なく、二量体の β-グロビン/LCR 連絡先の増加が認められました。最後に、ABA は、内因性の構造 (図 1 dと補助図 3-6) の完全リニューアルを示したを削除した後 3 c システムの可逆性を確認しました。
我々 はこのアイデアを探索する 2 番目のセル行が利用されたので、K562 細胞に示す成功ユークロマチン (図 1 d) の地域でグロビン遺伝子座位置の結果であるかもしれませんと見なされます。CLOuD9 システムは、多色、グロビン遺伝子 (図 1e) を発現していない地域での HEK 293 t 細胞に適用されました。K562s; で観察されたものに類似していた結果多くの β-グロビン LCR 団体は ABA (図 1eおよび補足の図 3) が 24 h 後 3 C で測定した CLOuD9 の元のクロマチンの状態にもかかわらず、さまざまな携帯電話環境で機能する堅牢な能力の証拠を提供または構造。
CLOuD9、Oct4 プロモーター、293 t 細胞内の遠位 5' エンハンサーなどの広範な適用を確保するため追加の軌跡を調べた。以前は、なかった細胞検出 Oct4 式とさらに、内因性連絡先記載。胚性幹細胞エンハンサー遠位 5' との接触から生じる、Oct4 の発現の証拠動機この実験と同じ結果が観測された β-グロビン遺伝子座18。連絡先、Oct4 の遠位エンハンサーやプロモーター CLOuD9 有効なセルがない制御の細胞 (図 1 階) で確認されました。また、Oct4 プロモーターやエンハンサー遠位 5' 相互作用また Oct4 プロモーターに連絡する 3' エンハンサーを求めが観察されました。このイベントは、3' エンハンサーはやり取り Oct4 プロモーター/5 と整合的 ' 遠位エンハンサー内因性遺伝子活性化10の間に複雑な。
CLOuD9 誘導遺伝子座位コンテキスト特定変更します。CLOuD9 システムに遺伝子座の染色体の連絡先が誘発する実際にことを確認し、我々 はループの遺伝子発現に及ぼす影響を調べるしようと思う。それは、グロビンと Oct4 遺伝子の転写は、LCR とグロビン遺伝子座および遠位 5' エンハンサーとそれぞれ1,11、Oct4 プロモーター間の接触によって決まりますが記載されています。したがって、我々 はこれらの領域のそれぞれのドライブ クロマチン ループ形成システムと説得力のある遺伝子発現、CLOuD9 を使用する仮説。
両方の軌跡 RT qPCR 実証、アブシジン酸誘導によるクロマチンのループ Oct4 式 293 t 細胞、K562 細胞における β-グロビン式運転増加 293Ts ではない (図 2 a)。しかし ABA として 24 h 増加 β-グロビン式の文化を大幅に、セルに式を追加 72 時間まで着実に増加を続けているし、ABA ウォッシュ アウト (図 2 a) に可逆的であった。K562 細胞のコントロール以外のすべては二量体化コンポーネントが LCR および β-グロビンのプロモーター領域 (図 2 aおよび補足図 7, 8) に位置していたに関係なく、このような傾向が続きます。これらの所見を支持する H3K4me3 と K562s と 293Ts β グロビン遺伝子座で RNA Pol II のチップ qPCR 転写 (図 2 cf) で観測された変化と対応しました。
CLOuD9 が安定したクロマチンのループを確立します。CLOuD9 と短期的なループ誘導明確に続いて期待が観察される残ったループの長期的な誘導が差動効果を持っていたかどうか。これを調べるためには、細胞は 10 日間 ABA の存在下で培養しました。Β-グロビン表現の変化が観察されただけまだ K562s と 293Ts の両方は、β-グロビン遺伝子座とコントロール (図 3 a、 bおよび補足図 9, 10) を基準にして LCR の接触頻度の増加を展示、K562 細胞 (図 3 c)。興味深いことに、しかし、それを認めたが、長期で転写亢進していた強く、K562s の二量体化はなかった長い可逆 (3 a を図と補助図 9-11)。ただし、293Ts、次の長期的な二量体化転写で改造が認められたない、クロマチン連絡先を可逆的に残った (図 3 bと補足図 9) の変化を誘導しました。
全体的に、遺伝子発現の小さな減少のみが二量体化 (図 3 c) 前に遺伝子発現レベルが有意に残った ABA 除去 10 日後観測されました。これに合わせて K562 細胞がない 293 t 細胞、H3K4me3 とチップ-qPCR による β-グロビン遺伝子座で RNA Pol II 持続的な変化を示した ABA 除去 (図 3 df) 10 日後もコントロールと比較してください。したがって、我々 の結果は、クロマチンのループの安定性がより持続可能な遺伝子表現を意味することを示します。
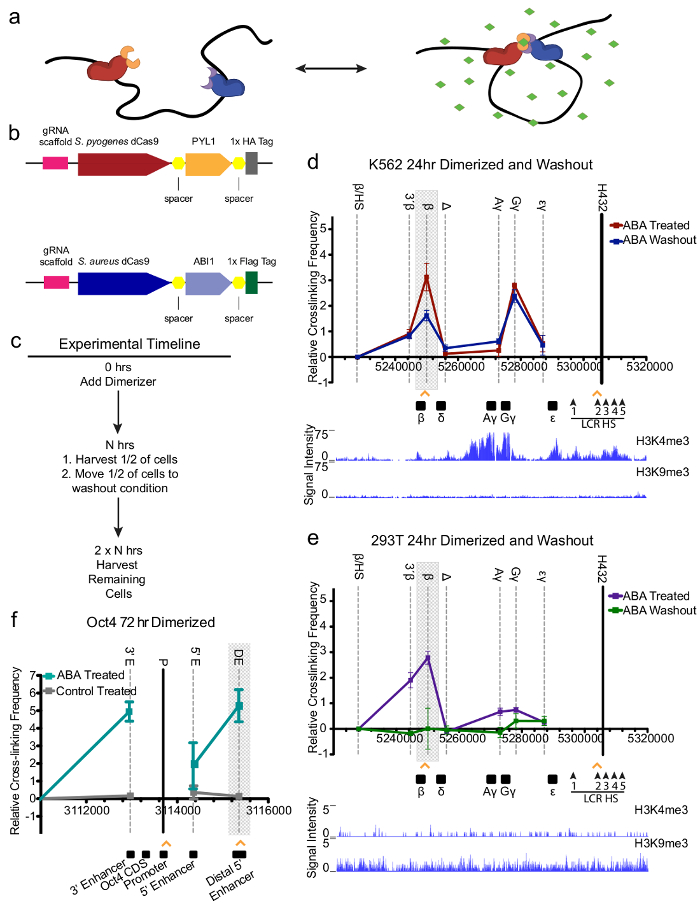
図 1: CLOuD9 リバーシブル β-グロビン プロモーター-LCR ループを誘導します。(a)アブシジン酸 (ABA、緑) の付加をもたらす 2 つの相補的な CLOuD9 構造 (CLOuD9 S 化膿 (CSP)、CLOuD9 S. 球菌 (CSA)、赤と青のそれぞれ) に近接、クロマチン構造を改造します。ABA の除去は、内因性クロマチン構造を復元します。(b) CLOuD9 構造技術を組み合わせて CRISPR dCas9黄色ブドウ球菌とs. 化膿から可逆 dimerizeable した PYL1 と ABI1 ドメイン。(c) CLOuD9 タイムライン二量体化実験。(d) 3 C 分析 K562 細胞における β-グロビン ローカス ワイド架橋の測定周波数 ABA (赤) と治療の 24 時間後とその後ウォッシュ アウト (青) の可逆性を示す誘導 β-グロビン/LCR 連絡先 (灰色で強調表示されます)。オレンジ色の矢印は、特定 CLOuD9 構築ターゲット領域を示します。LCR (黒いバー) の過敏症のサイト 1-4 を含む EcoRI フラグメントはアンカー領域として使用されました。他の示された EcoRI フラグメント (グラフの上部にある名前) 架橋頻度を評価しました。ヒト β グロビン遺伝子と LCR の過敏症のサイトは、染色体の位置座標とグラフの下部に描かれています。H3K4me3 と H3K9me3 チップ seq からデータを示すこの地域は K562s (e)と同様にユークロマチン H3K4me3 と H3K9me3 チップ seq データからの証拠にもかかわらず、HEK 293 t 細胞のクロマチン構造の可逆的な変化が見られるが、グロビン地域は、この型のセルの多色です。293 t Oct4 ローカス ワイド架橋周波数を測定(f) 3 C 分析細胞後 72 時間を示す ABA (赤) と治療の Oct4/遠位エンハンサー連絡先 (灰色で強調表示されます)。オレンジ色の矢印は、特定 CLOuD9 構築ターゲット領域を示します。Oct4 プロモーター (黒いバー) を含む mboi を検索フラグメントはアンカー領域として使用されました。他の示された mboi を検索フラグメント (グラフの上部にある名前) 架橋頻度を評価しました。人間 Oct4 地域は、染色体の位置座標とグラフの下部に描かれています。3 C の結果のすべては、少なくとも 3 つの独立した実験から得られました。3 C の値は、チューブリンに正規化されました。Β-グロビン、アンカーのフラグメントと β/HS フラグメントを含むフラグメント間の相互作用の周波数がゼロに設定されました。Oct4、アンカー フラグメントと Oct4 相互作用領域の外側のネガティブ コントロール フラグメント間の相互作用の周波数がゼロに設定されました。誤差範囲を示す標準偏差 n = 3。この図は、図 1モルガン、ステファニー L.、らから変更されています。9.この図の拡大版を表示するのにはここをクリックしてください。
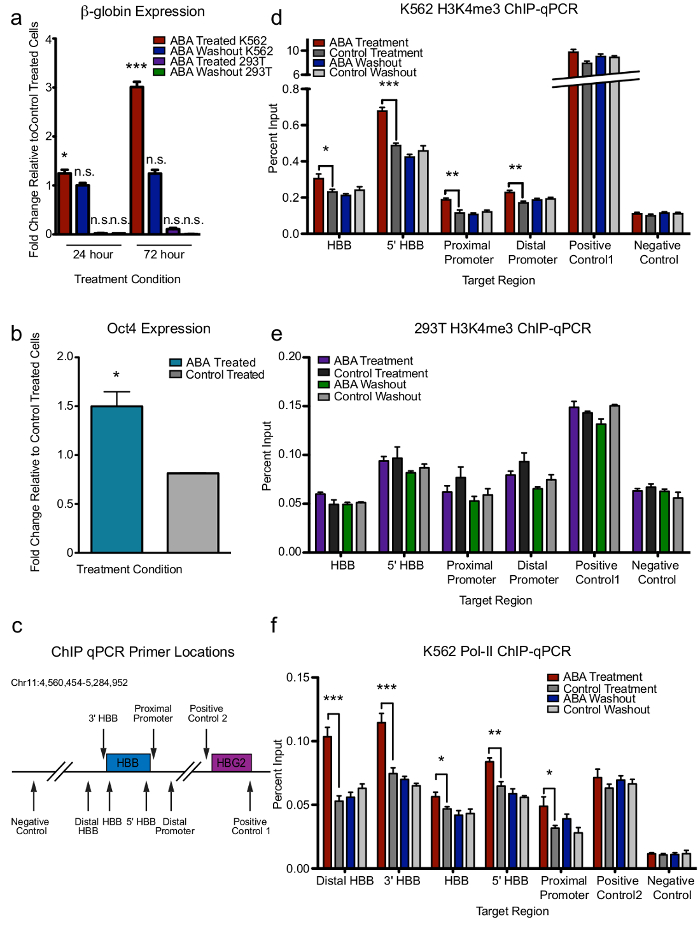
図 2: CLOuD9 コンテキスト特定遺伝子の発現とクロマチンの状態変化を誘導します。(a) CLOuD9 誘因クロマチン β グロビン遺伝子座でループは β-グロビン式 293Ts 駆除処理されたセルを基準にして与えられた意義ではなく K562s での可逆的な誘導の結果します。2-スチューデントの両側t-テスト * P < 0.05、 t = 3.418、df = 5;P < 0.0001、 t = 10.42 df = 5;n. s. 有意ではないです。誤差範囲を示す標準偏差n = 3。(b)誘導の Oct4 CLOuD9 誘起 293Ts 次のように観察された式は、同じ軌跡でループです。意義は、駆除処理されたセルを基準にして与えられます。2-スチューデントの両側t-テスト * P < 0.05、 t = 4.562、df = 2。エラーバーは標準偏差(c) β-グロビン遺伝子体に沿ってチップ qPCR プライマー場所の概略図を示します。(d, e)チップ qPCR 可逆変化 293Ts ではなく K562s で β グロビン遺伝子座で H3K4me3 CLOuD9 誘起ループを次に示します。2-スチューデントの両側t-テスト * P < 0.05 * * P < 0.001、* * * P < 0.0001。誤差範囲を示す標準偏差(f) CLOuD9 K562s における β-グロビン転写を介した変化は β グロビン遺伝子の体の全体にわたって RNA Pol II の占有率の増加に対応します。2-スチューデントの両側t-テスト * P < 0.05 * * P < 0.001、* * * P < 0.0001。誤差範囲を示す標準偏差.この図は、図 2モーガン、ステファニー L.、らから変更されています。9.この図の拡大版を表示するのにはここをクリックしてください。
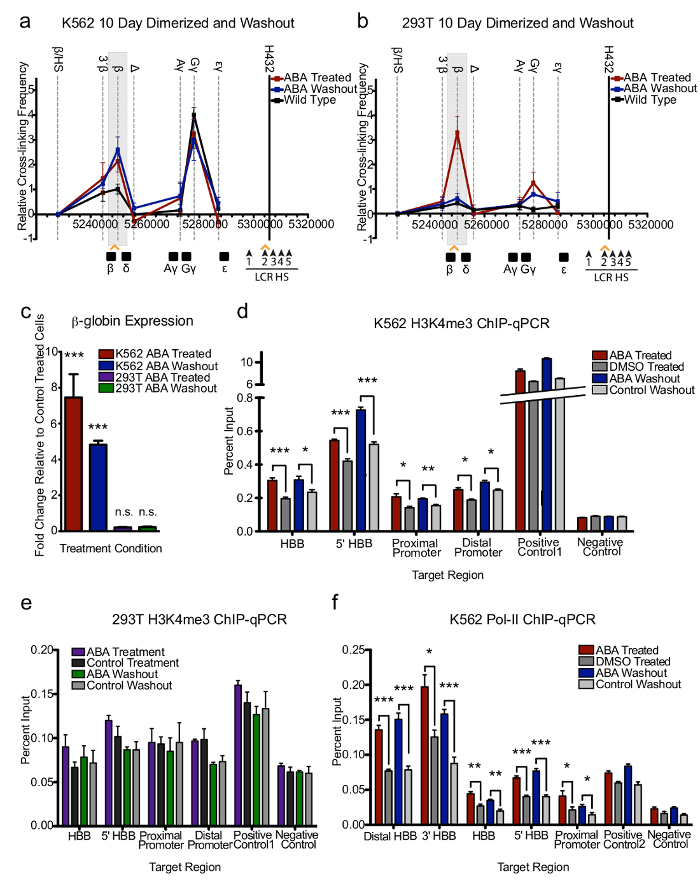
図 3: CLOuD9 が長期的な二量体化に続く堅牢な遺伝子発現を支える安定したクロマチンのループを確立します。(a、b) 3 C 分析は、K562s でもない 293Ts、CLOuD9 誘因クロマチンのループになる不可逆的な ABA 治療 10 日後さらに最大 10 日間の ABA が削除されるときにもを示します。すべての 3 C 結果が少なくとも 3 つの独立した実験から得られました。3 C の値は、チューブリンに正規化され、アンカーのフラグメントと β/HS フラグメントを含むフラグメント間の相互作用の周波数がゼロに設定されました。誤差範囲を示す標準偏差 n = 3。(c) K562s でループの安定化と、β-グロビンも ABA ウォッシュ アウトの 10 日間、次の固定式。293Ts 駆除処理されたセルを基準にして与えられた意義では、β-グロビン発現の変化は観察されなかった.2-スチューデントの両側t-テスト * * * P < 0.0001、t = 5.963、df = 5;CLOuD9 誘起ループに対して β-グロビン遺伝子座上 H3K4me3 マーク n. s. (d)非重要なチップ qPCR 表示増加が持続 K562s. 2 スチューデントのtのリガンド ウォッシュ アウトの 10 日間、次のテスト * P < 0.05 * * P< 0.001、* * * P < 0.0001。(e) H3K4me3 の大幅な変化信号を次の長期的な二量体化は、293Ts (f)長期的なループ誘導 β グロビン遺伝子座の増加 RNA Pol II 占有チップ qPCR で観察した K562s で維持されていた配位子のウォッシュ アウトの 10 日間、次の。2-スチューデントの両側t-テスト * P < 0.05 * * P < 0.001、* * * P < 0.0001。すべての誤差範囲を示す標準偏差.この図は図 3モーガン、ステファニー L.、らから変更されています。9.この図の拡大版を表示するのにはここをクリックしてください。
補足図 1: CLOuD9 構造をその対象地域にローカライズします。クロマチン免疫沈降法と定量的 PCR の CLOuD9 構造は、彼らの意図されていたゲノム遺伝子座への正しいローカライズをデモンストレーションします。この図は、モーガン、ステファニー L.、ら補足図 1から変更されています。9.この図をダウンロードするここをクリックしてください。
補足図 2: CLOuD9 構造は可逆的 ABA の処置への応答に関連付けます。Co immunoprecipitations ABA 処理の次の 72 h は dCas9 蛋白質の協会を示す逆次以降リガンド ウォッシュ アウトの 72 h。この図は、モーガン、ステファニー L.、ら補足図 2から変更されています。9.この図をダウンロードするここをクリックしてください。
補足図 3: クロマチン連絡先の変化を誘発するコントロール治療がありません。24 時間コントロール エージェント、DMSO を治療、K562 細胞の 3 C による内因性クロマチン構造の変化を誘導するないまたは HEK 293Ts 3 C 値は、チューブリンとアンカーのフラグメント、フラグメント間の相互作用の周波数を正規化されました。ゼロに設定された β/HS フラグメントを網羅します。誤差範囲を示す SD. n = 3。この図は、モーガン、ステファニー L.、ら補足図 3から変更されています。9.この図をダウンロードするここをクリックしてください。
補足図 4: コントロール CLOuD9 導入細胞クロマチンのループで変化を示さないです。2 つを演出 CLOuD9 のコンス トラクターのいずれかを LCR または β-グロビン プロモーター 3 C 以下のコントロール治療を基準にして ABA 処理によってクロマチン構造に大きな変化を誘発するないです。3 C の値は、チューブリンに正規化され、アンカーのフラグメントと β/HS フラグメントを含むフラグメント間の相互作用の周波数がゼロに設定されました。誤差範囲を示す SD. n = 3。この図は、補足図 4モーガン、ステファニー L.、らから変更されています。9.この図をダウンロードするここをクリックしてください。
補足図 5: 二量体化の 72 時間後元に戻せる状態のまま CLOuD9 クロマチンのループします。K562s の 3 C 分析を示します CLOuD9 の可逆性は、ABA 処理の 72 時間後に β-グロビン/LCR 連絡先を誘発しました。3 C の値は、チューブリンに正規化され、アンカーのフラグメントと β/HS フラグメントを含むフラグメント間の相互作用の周波数がゼロに設定されました。誤差範囲を示す SD. n = 3。この図は、モーガン、ステファニー L.、ら補足図 5から変更されています。9.この図をダウンロードするここをクリックしてください。
補足図 6: CLOuD9 グロビン ターゲット サイトで誘導 β-グロビン/LCR ループは影響されません。3 C 以下の ABA 処理の 72 時間によって、LCR またはループ誘導のような可逆的な変化で β-グロビン プロモーター結果の代替領域に構築 CSA と CSP を演出します。3 C の値は、チューブリンに正規化され、アンカーのフラグメントと β/HS フラグメントを含むフラグメント間の相互作用の周波数がゼロに設定されました。誤差範囲を示す SD. n = 3。この図は、モーガン、ステファニー L.、らの補足の図 6から変更されています。9.この図をダウンロードするここをクリックしてください。
補足図 7: 遺伝子発現の誘導変化がグロビン ターゲット ・ サイトに関係なく持続 CLOuD9 。Β-グロビン プロモーターの代替領域に構築 CSA と CSP を演出し、LCR は、次の 72 時間二量体化の遺伝子発現の誘導に影響を与えません。ただし、いくつか次長期 (10 日) 二量体化が観察された遺伝子発現強度への影響、高レベルの β-グロビン治療コントロールのセルと相対的あった持続次以降リガンド [ウォッシュ アウト] 10 追加日間。意義は、駆除処理されたセルを基準にして与えられます。p < 0.001、t = 10.25、df = 5;p < 0.0001、左右 t = 8.697, df = 6、t = 40.31、df = 7;n. s. 有意ではないです。すべての誤差範囲を示すの SDこの図は、補助図の 7モルガン、ステファニー L.、らから変更されています。9.この図をダウンロードするここをクリックしてください。
補足図 8: コントロール CLOuD9 導入細胞 β-グロビン式で変化を示さないです。2 つを演出 CLOuD9 のコンス トラクターのいずれかを LCR または β-グロビン プロモーターにコントロール治療を基準にして ABA の処置に続く β-グロビン発現の重要な変化が誘導するないです。意義は、駆除処理されたセルを基準にして与えられます。n. s. 有意ではないです。この図は、モーガン、ステファニー L.、ら補足図 8から変更されています。9.この図をダウンロードするここをクリックしてください。
補足図 9: クロマチン連絡先の変化を誘発する長期管理治療がありません。10 日間コントロール エージェント、DMSO を治療、K562 細胞の 3 C による内因性クロマチン構造の変化を誘導するないまたは HEK 293Ts 3 C 値は、チューブリンとアンカーのフラグメント、フラグメント間の相互作用の周波数を正規化されました。ゼロに設定された β/HS フラグメントを網羅します。誤差範囲を示す SD. n = 3。この図は、モーガン、ステファニー L.、ら補足図 9から変更されています。9.この図をダウンロードするここをクリックしてください。
補足図 10: 長期 CLOuD9 グロビン ターゲット サイトで誘導 β-グロビン/LCR ループは影響されません。3 C 10 日 ABA 処理と後配位子のウォッシュ アウトの 10 日間、次に示すように β-グロビン プロモーター結果同様にループ誘導を持続や、LCR の代替領域にコンストラクト CSA と CSP を演出します。3 C の値は、チューブリンに正規化され、アンカーのフラグメントと β/HS フラグメントを含むフラグメント間の相互作用の周波数がゼロに設定されました。誤差範囲を示す SD. n = 3。この図は、モーガン、ステファニー L.、ら補足図 10から変更されています。9.この図をダウンロードするここをクリックしてください。
補足図 11: CLOuD9 構造が不可逆的長期的な ABA の処置への応答に関連付けます。Co-immunoprecipitations ABA 治療の 10 日と配位子のウォッシュ アウトの 10 以降日 CSA と CSP の dCas9 蛋白質の不可逆的な協会を示します。この図は、モーガン、ステファニー L.、らの補足の図 11から変更されています。9.この図をダウンロードするここをクリックしてください。
補足表 1: プライマーのリストは gRNAs、qRT PCR、3 C、およびチップの qPCR のシーケンスします。このテーブルは、補足表 1モルガン、ステファニー L.、らから変更されています。9しますこの表をダウンロードするここをクリックして下さい。
ディスカッション
The most CLOuD9 クロマチンのループで重要な手順は、: 1) 設計または正しい gRNAs を使用して、CLOuD9 導入細胞、ABA、DMSO などで毎日 2) 変化するメディア 3) ABA の鮮度を維持すること、4) の正確かつ慎重な評価を行うクロマチン構造。
CLOuD9 の制限は、選択の対象地域にガイドを設計する能力に主に存在します。ガイド Rna は二量体化するためにターゲット DNA の領域に dCas9 コンポーネントのローカライズの重要なタスクを実行し、ガイドの有効性が自分の特定のターゲット サイトに基づいています。適切な gRNA のコンポーネントなしでは、システムは元に戻せる状態を形成することはできません CLOuD9 はループを誘発しました。したがって、興味の地域ごとの複数のガイドを設計してガイドを広がって 250-1000 bp の領域で、少なくとも 1 つ成功ガイドが確保されます。ガイド位置も正確な結果に不可欠です。転写調節の上下など、背景の効果を防ぐために転写因子結合部位やその他の重要な地域に位置するガイドを避けるために重要です。また、CLOuD9 構成要素の正確な位置は若干ターゲット遺伝子の転写に影響を与えます。これは実験目的のための最も堅牢なペアを識別する、ターゲット地域のガイドの複数のペアをテストの重要性を強調します。さらに、対象地域の各ペアでは、黄色ブドウ球菌gRNAs と CSA コンストラクトをターゲットし、CSP 構造をターゲットとする gRNAs とs. 化膿の特異性の対象です。
正確な結果と正しいダイマー形成を強固にするには、また CLOuD9 構成要素の伝達に続く細胞環境の鮮度を維持することが重要としてます。毎日メディア変更し、新鮮な dimerizer (またはコントロール) の添加により、相補的な構造が近接で維持され、変更されたクロマチン構造を保持します。さらに、ABA は新鮮と (開いた風邪、光から保護維持、6 ヶ月以内) 製造元のプロトコルに従って適切に保存されている保証は、本格的な結果を得ることに不可欠です。
特によりよく活用 FRB と FKBP システムではなく、CLOuD9 の ABA dimerizer は ABI と PYL の二量化蛋白質で使用されました。FRB/FKBP システム、rapalog の必要性には、限られたがん細胞への毒性のため、CLOuD9 の適用がでしょう。ABI/PYL の代替システムより広く活用する CLOuD9 を効果的に有効にする、このような制限を回避しました。
総称して、CLOuD9、強制的にすることができますが、元に戻せる長距離ターゲット ゲノムにおける遺伝子座間の連絡先を作成するユニークな堅牢な技術を開発しました。クロマチンのループを誘導を介して我々 はまた CLOuD9 を適切な細胞における遺伝子発現を変更する利用できることを示します。技術の適応性無制限ループ領域やメカニズムをループの事前の知識を必要とせずの任意の 2 つの遺伝子座位間の相互作用に関することができます。さらに、CLOuD9 のユニークな実証可逆性により病気と開発におけるループ メカニズムの検討、さらに。クロマチンのループの対象の効果を明確に実証されている、あるまだターゲットのループに及ぼすターゲットをループと後続への影響の洞察を提供しているデータであること。
我々 のデータはのみこのツールのいくつかのアプリケーションを示していますが、クロマチン配置は遺伝子発現を示す主要な基になるアイデアを意味します。当社の技術は、研究し、遺伝子、遺伝子の転写に折りたたみクロマチンの役割の全体的な理解を向上におけるクロマチン構造の微妙な違いを明らかにする使用できます。研究と治療のがん、遺伝性疾患、先天性疾患、どの明確なクロマチン アセンブリを間違いなく遺伝子式20、変更の方法につながることができます転写ダイナミクスの機微の理解を深める 21,22,23。CLOuD9 技術を活用した後続作業がクロマチン ドメインとどのように彼らは折り畳み式の開発と病気の両方で安定した遺伝子発現を維持するためにドライブのダイナミクスの配置の詳細については、さらに点灯します。
開示事項
著者が明らかに何もありません。
謝辞
私たちは、h. チャン、T. オロ、s. Tavazoie、r. フリン、p. バティスタ、E. カロ、テクニカル サポートや原稿の重要な読書のための全体の王研究室に感謝します。S.L.M. は、国立癌研究所 (1F99CA222541-01)、NDSEGF (FA9550-11-C-0028)、NSFGRF (DGE-114747) この作品でサポートされています。K.C.W. はバローズ ・ ウェルカム財団から医療科学者のキャリア賞に支えられて、ドナルド E. とデリア B. バクスター財団国際学部奨学生。
資料
| Name | Company | Catalog Number | Comments |
| RPMI 1640 media | Life Technologies | 11875-119 | For K562 cell culture |
| DMEM media | Life Technologies | 11995-065 | 1X, for 293T cell culture |
| lentiCRISPR v2 | Addgene plasmid | #52961 | For CLOuD9 plasmid development |
| pRSV-Rev | Addgene plasmid | #12253 | For lentivirus production |
| pMD2.G | Addgene plasmid | #12259 | For lentivirus production |
| pMDLg/pRRE | Addgene plasmid | #12251 | For lentivirus production |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668-019 | For lentivirus production |
| anti-HA antibody | Cell Signaling | 3724 | For immunoprecipitation |
| anti-Flag antibody | Sigma | F1804 | For immunoprecipitation |
| DNeasy Blood and Tissue Kit | Qiagen | 69504 | For DNA extraction |
| TRIzol | Life Technologies | 15596-018 | For RNA extraction |
| RNeasy Kit | Qiagen | 74106 | For RNA extraction |
| Superscript VILO | Life Technologies | 11754-050 | For cDNA |
| SYBR Green I MasterMix | Roche | 4707516001 | For qPCR analysis |
| Light Cycler 480II | Roche | For qPCR analysis | |
| anti-H3K4me3 antibody | AbCam | ab8580 | For ChIP-qPCR |
| anti-RNA Pol-II antibody | Active Motif | 61083 | For ChIP-qPCR |
| EDTA free protease inhibitor | Roche | 11873580001 | For protein extraction |
| 4-12% Tris Glycine gel | Biorad | Any size, For western blot | |
| anti-Rabbit HRP antibody | Santa Cruz | sc-2030 | For western blot |
| anti-mouse HRP antibody | Cell Signaling | 7076S | For western blot |
| K562 and H3K293 ChIP-Seq data | Encode | ENCSR000AKU | For ChIP-seq analysis |
| K562 and H3K293 ChIP-Seq data | Encode | ENCSR000APE | For ChIP-seq analysis |
| K562 and H3K293 ChIP-Seq data | Encode | ENCSR000FCJ | For ChIP-seq analysis |
| K562 and H3K293 ChIP-Seq data | GEO | GSM1479215 | For ChIP-seq analysis |
| Dynabeads Protein A for Immunoprecipitation | Thermo Fisher Scientific | 10001D | For immunoprecipitation |
| Dynabeads Protein G for Immunoprecipitation | Thermo Fisher Scientific | 10004D | For immunoprecipitation |
| RNA Clean & Concentrator-5 | Zymo Research | R1015 | For RNA purification |
| Pierce 16% Formaldehyde Methanol-free | Thermo Fisher Scientific | 28908 | For crosslinking |
| PX458 Plasmid | Addgene | 48138 | Suggested active Cas9 plasmid for gRNA cloning, but any active Cas9 plasmid will do |
| QIAquick PCR Purification Kit | Qiagen | 28104 | For PCR purification |
| FastDigest BsmBI | Thermo Fisher Scientific | FD0454 | For cloning guide RNAs |
| FastAP | Thermo Fisher Scientific | EF0651 | For cloning guide RNAs |
| 10X FastDigest Buffer | Thermo Fisher Scientific | B64 | For cloning guide RNAs |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | For cloning guide RNAs |
| 10X T4 Ligation Buffer | NEB | B0202S | For cloning guide RNAs |
| T4 PNK | NEB | M0201S | For cloning guide RNAs |
| 2X Quick Ligase Buffer | NEB | B2200S | For cloning guide RNAs |
| Quick Ligase | NEB | M2200S | For cloning guide RNAs |
| Buffers | |||
| Farnham lysis buffer | 1% Tris-Cl pH 8.0, 1% SDS, 1% protease inhibitor water solution (non-EDTA), and 1 mM EDTA in water | ||
| Modified RIPA buffer | 1% NP40/Igepal, 0.5% sodium deoxycholate, 0.1% SDS, 1 mM EDTA, and 1% protease inhibitor water solution (non-EDTA) in PBS pH 7.8 or 7.4 | ||
| IP dilution buffer | 0.01% SDS, 1.1% Triton-X 100, 1.2 mM EDTA, 16.7 mM Tris-HCl pH 8.0, 167 mM NaCl, 0.1x protease inhibitor | ||
| Wash buffer | 100 mM Tris pH 9, 100 mM LiCl, 1% NP-40, and 1% sodium deoxycholate | ||
| Swelling buffer | 0.1 M Tris pH 7.5, 10 mM potassium acetate, 15 mM magnesium acetate, 1% NP-40 | ||
| Dilution buffer | 0.01% SDS, 1.1% Triton X-100, 1.2 mM EDTA, 16.7 mM Tris pH 8 and 167 mM NaCl | ||
| IP elution buffer | 1% SDS, 10% NaHCO3 |
参考文献
- Krivega, I., Dean, A. Chromatin looping as a target for altering erythroid gene expression. Ann. N.Y. Acad. Sci. 1368, 31-39 (2016).
- Matharu, N., Ahituv, N. Minor loops in major folds: Enhancer-promoter looping, chromatin restructuring, and their association with transcriptional regulation and disease. PLoS Genet. 11, e1005640 (2015).
- Dekker, J., Martí-Renom, M. A., Mirny, L. A. Exploring the three-dimensional organization of genomes: interpreting chromatin interaction data. Nat. Rev Genet. 14, 390-403 (2013).
- Kim, A., Dean, A. Chromatin loop formation in the b-globin locus and its role in globin gene transcription. Mol Cells. 34, 1-5 (2012).
- Petrascheck, M., et al. DNA looping induced by a transcriptional enhancer in vivo. Nucleic Acids Res. 33, 3743-3750 (2005).
- Ameres, S. L., et al. Inducible DNA-loop formation blocks transcriptional activation by an SV40 enhancer. EMBO J. 24, 358-367 (2005).
- Deng, W., et al. Controlling Long-Range Genomic Interactions at a Native Locus by Targeted Tethering of a Looping Factor. Cell. 149, 1233-1244 (2012).
- Deng, W., et al. Reactivation of developmentally silenced globin genes by forced chromatin looping. Cell. 158, 849-860 (2014).
- Morgan, S. L., et al. Manipulation of Nuclear Architecture through CRISPR-Mediated Chromosomal Looping. Nature Communications. 8, (2017).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biol. 17 (1), 148 (2016).
- . . Lipofectamine 2000 Reagent. , (2013).
- . . DNeasy Blood & Tissue Kit Handbook. , (2006).
- . . Determining Genome Targeting Efficiency using T7 Endonuclase I (M0302). , (2016).
- Guschin, D. Y., et al. A rapid and general assay for monitoring endogenous gene modification. Methods Mol Biol. 649, 247-256 (2010).
- Shalem, O., Sanjana, N. E., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. , 83-87 (2014).
- . . RNeasy Mini Handbook. , (2012).
- . . SuperScript VILO cDNA Synthesis Product Information Sheet. , (2015).
- Theunissen, T. W., et al. Systematic identification of culture conditions for induction and maintenance of naive human pluripotency. Cell Stem Cell. 15, 471-487 (2014).
- Mokry, M., et al. Integrated genome-wide analysis of transcription factor occupancy, RNA polymerase II binding and steady state RNA levels identify differentially regulated functional gene classes. Nucleic Acids Res. 40, 148-158 (2012).
- Drier, Y., et al. An oncogenic MYB feedback loop drives alternate cell fates in adenoid cystic carcinoma. Nat Genet. 48, 265-272 (2016).
- Ryan, R. J. H., et al. Detection of enhancer-associated rearrangements reveals mechanisms of oncogene dysregulation in B-cell lymphoma. Cancer Discov. 5, 1058-1071 (2015).
- Montavon, T., et al. A regulatory archipelago controls Hox genes transcription in digits. Cell. 147, 1132-1145 (2011).
- Lupianez, D. G., et al. Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions. Cell. 161, 1012-1025 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved