Method Article
你在哪里切的问题: 眼地标性小鼠视网膜空间定位的解剖与分析指南
摘要
该协议提供了一个全面的解剖和分析指南, 使用深眼地标, s-视蛋白免疫组化, Retistruct 和自定义代码, 以准确和可靠地定向孤立的小鼠视网膜在解剖空间。
摘要
准确、可靠地识别孤立的小鼠视网膜的空间方向对于许多视觉神经科学的研究都是很重要的, 包括视网膜细胞的密度和大小梯度的分析, 方向选择方向的调整。神经节细胞和一些视网膜疾病的地形退化模式的检查。然而, 文献中还有许多不同的眼部解剖方法, 用于鉴别和标记小鼠视网膜方向。虽然这种研究中使用的定位方法常常被忽视, 但如果不报告视网膜定向是如何确定的, 则在试图比较研究数据时, 可能会导致文学上的差异和混淆。表面的眼部地标, 如角膜烧伤, 通常使用, 但最近被证明是不可靠的比更深层次的标志, 如直肌, 脉络膜裂, 或 s 视蛋白梯度。在这里, 我们提供了一个全面的指导, 使用深眼地标, 以准确地解剖和记录的空间方向的孤立的小鼠视网膜。我们还比较了两种 s-视蛋白抗体的有效性, 并包括了视蛋白免疫组化的协议。因为视网膜的方向根据 s-视蛋白梯度要求视网膜重建与 Retistruct 软件和自转以自定义代码, 我们已经提出了使用这两个程序所需的重要步骤。总的来说, 本议定书的目标是提供一套可靠和可重复的方法, 用于精确的视网膜定向, 这是适应大多数实验性协议的。这项工作的一个首要目标是标准化视网膜定向方法, 以供将来研究之用。
引言
视网膜神经科学的一个重要的, 有时被忽视的方面是正确的定位和分析孤立的全视网膜, 无论是视网膜的方向在电生理记录室或组织学幻灯片。这对于涉及老鼠视网膜的研究尤其重要, 这是目前最广泛使用的哺乳动物视觉系统调查模型。最近的发现表明, 小鼠视网膜不是空间上的均匀的, 而是有功能上不同的视网膜细胞类型的密度和大小梯度, 如黑视素神经节细胞, 瞬态的非α神经节细胞, 锥 opsins1,2 ,3,4,5。因此, 用于确定视网膜方向的方法可能会影响细胞类型或视蛋白分布2、3、6、方向选择性方向调谐的实验结果。神经节细胞7,8,9, 和地形模式视网膜变性10,11,12,13,14.事实上, 没有报告视网膜定向是如何报告的, 可能会导致文学的差异和混淆时, 试图比较数据之间的研究。因此, 研究人员要报告视网膜定位的方法是非常重要的, 这样可以准确地解释这些研究的结果。
视网膜定向通常是指在眼球摘除1、3、12、15、16、17 前, 在背、腹、鼻或颞部角膜上评分. ,18,19或通过切割或染色深解剖眼睛的地标, 如眼外肌6,7, 脉络膜裂20,21, 或s-视蛋白梯度2,3。直肌可用于鉴别背, 腹, 鼻和颞部视网膜通过做一个深的缓解切口, 平分的附件的上级直肌, 下直肌, 内侧直肌, 或侧直肌, 分别。然而, 对于大多数实验, 使用一个直肌是足够的定向视网膜22。脉络膜裂是眼睛发育的残余物, 可以被看作是眼睛背面的一条微弱的水平线。这条线的每个末端在鼻子或地球23的世俗极性终止。最后, 视蛋白表达不对称分布于小鼠腹侧视网膜, 视蛋白抗体可用于在免疫组化实验中揭示腹壁视网膜1。
最近的工作由 Stabio,等等。22表明, 浅表眼地标如角膜烧伤是一种较不可靠的方法定位视网膜在解剖空间, 很可能是由于人的错误和变异, 使角膜烧伤时, 使用的时间和内侧canthi 作为参考点。相反, 深的地标, 如直肌, 脉络裂, 和 s-视蛋白梯度, 已被证明是更可靠和准确的地标定向视网膜22。然而, 鉴定这些解剖地标需要独特的解剖步骤, 在文献中没有详细描述。因此, 本议定书的目的是提供一个全面的教程, 如何使用上直肌, 脉络膜裂, 和 s-视蛋白梯度, 以准确地识别的空间方向的小鼠视网膜。此外, 我们还包括了两种 s-视蛋白抗体的有效性的比较, 以及视蛋白免疫组化的协议。
依靠精确的视网膜方向进行研究的另一个挑战是在录音室、盘子或滑梯上压扁 wholemount 视网膜所需的大的减压切口。这可以为分析三维结构, 当它被成像为平面二维结构时, 引入挑战。一个名为 Retistruct24的程序可以用来返回一个平坦的 wholemount 视网膜到它的三维结构, 然后从它收集的数据进行分析。因此, 本协议的一节专门强调了使用 Retistruct 软件重建视蛋白 immunostained 小鼠视网膜所需的步骤。我们还包括了使用我们的自定义 MATLAB 脚本的一部分协议, 这是开发, 以准确地旋转和东方的老鼠视网膜染色的视蛋白。
研究方案
这里描述的所有方法都已获得阿克伦大学机构动物护理和使用委员会 (IACUC) 的批准。
1. 使用上直肌的标志性识别视网膜方向
注: 高级直肌是背视网膜的标志性 (表 1)。如果实验不需要背视网膜的标记, 跳过步骤1并继续步骤2。
- 遵循您批准的机构动物护理和使用委员会的协议, 老鼠安乐死。
- 为了确定地球的一般方位, 在安乐死后立即在角膜-巩膜边界附近的鼻腔和颞 canthi 之间直接做一个烧伤标记 (图 1a)。将烧灼笔加热十秒钟, 然后将笔的笔尖接触到背角膜不到一秒钟即可使烧伤标记。
注意: 把烧灼笔放在角膜上太久会导致地球穿刺。
注意: 虽然有些烧灼笔确实发出光, 但在材料表中列出的烧灼笔在加热时不会发出任何光, 这使得它成为暗适应实验的安全选择。 - 对于摘除, 使用弯曲钳轻轻地把眼睛从它的插座, 并抓住地球从下面。不要切断视神经以除去地球;相反, 慢慢地从它的插座中抬起地球仪, 同时将它轻轻地从左向右移动, 直到地球从插座上松开。
注: 这项议案将允许直肌在地球最终完全从插座中被移除时, 保持附着在全球。视神经也会经常附着在地球上。 - 在含有解剖培养基的培养皿中, 将附着的直肌转移到全球。一定要跟踪哪个眼睛是左眼, 哪个是右眼。
注: 刀应使用适当的解剖培养基, 使其与实验性的协议一致。 - 在解剖范围内, 视觉定位角膜背烧伤并识别与之相关联的直肌 (图 1A)。
- 使用解剖剪刀或20克 (0.9 毫米 x 25 毫米) 针 (见材料表), 刺穿角膜在烧伤标记。对对开的肌肉进行深部的减压, 使其向视神经的方向切割。一个孤立的和重建的视网膜与这个切口显示在图 1B和1C。
- 开始使用两套镊子 (见材料表) 来隔离视网膜, 在步骤1.6 中轻轻地撕开穿刺孔, 直到部分视网膜暴露出来。
注意: 这是很重要的, 这是轻轻地做, 因为撕裂太用力会导致解除削减进一步撕裂。 - 用镊子将视网膜从巩膜中分离出来, 直到巩膜完全切除。用镊子除去虹膜、晶状体、玻璃体和任何残留的结构, 直到视网膜完全分离。
注意: 该协议可能在此处暂停。如果组织将被固定的 s-视蛋白免疫组化, 继续步骤3.5。
2. 利用脉络膜裂隙标志识别视网膜方向
注: 脉络膜裂隙存在于眼睛背面的巩膜上, 从颞极到鼻极 (图 2B和2C;表 1)。
- 遵循批准的机构动物护理和使用委员会议定书的老鼠安乐死。
- 为了确定地球的一般方位, 在安乐死后立即在角膜-巩膜边界附近的鼻腔和颞 canthi 之间直接做一个烧伤标记 (图 2a)。将烧灼笔加热十秒钟, 然后将笔的笔尖接触到背角膜不到一秒钟即可使烧伤标记。
注意: 把烧灼笔放在角膜上太久会导致地球穿刺。 - Enucleate 的眼睛和转移地球的培养皿中含有解剖培养基。一定要跟踪哪个眼睛是左眼, 哪个是右眼。
注: 刀应使用适当的解剖培养基, 使其与实验性的协议一致。 - 视觉定位和识别眼睛背面的脉络膜裂 (图 2B, 2C)。
注: 脉络膜裂也可见在眼罩下红外线20。 - 将地球定位在培养皿中, 使背部的烧伤位于上级, 如眼睛仍然在老鼠中。
注意: 背部烧伤的存在允许识别鼻腔和颞侧的地球, 只要它是一个右或左眼已被记录: 如果是右眼, 鼻腔脉络裂将在烧伤和时势的权利脉络裂将在烧伤的左侧。如果是左眼, 颞脉络膜裂将在烧伤的右侧, 鼻腔脉络裂将在烧伤的左侧。
使用解剖剪刀或20克 (0.9 x 25 毫米) 针 (见材料表), 使一个穿刺在地球上的背部烧伤位于。 - 对角膜背烧伤处的视神经进行浅部减压切口。这个切口将垂直于脉络膜裂, 允许在隔离后识别背视网膜 (图 2D)。
- 对视神经进行以下两个深层的减轻切口: 一是通过将解剖剪刀的叶片衬在眼睛背面的颞脉络膜裂线上, 一条由鼻脉络膜将解剖剪刀的刀片衬里起来。眼后的裂隙线。这些切口显示在图 2D和2E的一个孤立和重建的视网膜上。
注: 交替时, 可在颞脉络膜裂处进行深切口, 在鼻腔脉络裂处可进行浅切口, 使角膜背部烧伤不必要。这使得视网膜的准确定位, 减少了减轻削减。 - 开始使用两套镊子 (见材料表) 来隔离视网膜, 在步骤2.7 和2.8 中轻轻地撕开穿刺孔, 直到部分视网膜外露。
注意: 这是很重要的, 这是轻轻地做, 因为撕裂太用力会导致解除削减 (s) 进一步撕裂。 - 用镊子将视网膜从巩膜中分离出来, 直到巩膜完全切除。用镊子除去虹膜、晶状体、玻璃体和任何残留的结构, 直到视网膜完全分离。
注意: 该协议可能在此处暂停。如果组织将被固定的 s-视蛋白免疫组化, 继续步骤3.5。
3. 在小鼠视网膜上标记 S-视蛋白梯度
注意: s-视蛋白光敏表达不对称地分布于腹面视网膜1, 使其成为视网膜腹半部分的极好标记。这种方法只适用于固定和 immunostained 组织 (表 1)。以下步骤可应用于使用上述任何方法解剖的视网膜。
- 遵循批准的机构动物护理和使用委员会议定书的老鼠安乐死。
- 安乐死后, 立即 enucleate 眼睛, 并将地球放置在培养皿中, 解剖培养基。在视网膜解剖后, 一定要跟踪眼睛的左眼和右眼, 以确定视网膜方向。
注: 刀应使用适当的解剖培养基, 使其与实验性的协议一致。 -
开始使用两套镊子 (材料表) 来隔离视网膜, 在角膜的一部分暴露之前, 轻轻地撕开一个洞。
注意: 这是很重要的, 这是轻轻地做, 因为撕裂太用力会导致视网膜撕裂。 - 用镊子将视网膜从巩膜中分离出来, 直到巩膜完全切除。用镊子除去虹膜、晶状体、玻璃体和任何残留的结构, 直到视网膜完全分离。
注意: 该协议可能在此处暂停。如果使用视网膜进行活体实验, 请在执行以下步骤之前进行实验。 - 使用解剖剪刀, 使四减轻削减在视网膜上, 使它将躺在平坦。通过将视网膜的每一个角轻轻地压在镊子上, 将视网膜神经节细胞端上硝化膜 (材料表) 上。
注意: 当使用 s-视蛋白梯度为视网膜方向时, 减轻切口的位置可以是任意的。 - 使用镊子, 将安装的视网膜转移到第一个井中的24井板 (材料表) 填充1毫升4% 多聚甲醛 (材料表) 固定。将24井板放在室温 (材料表) 的轨道振动筛上, 将视网膜固定在40分钟左右。
注: 所有以下洗涤和孵化步骤应完成与24井板在一个轨道振动筛。 - 在室温下将视网膜洗净15分钟, 然后将其转移到填充1毫升0.1 米 PBS 的第二井。重复此步骤两次, 依次将视网膜转移到0.1 米 PBS 填充的第三和第四井。
- 将已安装的视网膜转移到含有1毫升阻断溶液的第五井 (1.7% X-100 和5.2% 只驴正常血清0.1 米 PBS; 见材料表), 并在4°c 过夜。
- 添加兔抗 s-视蛋白主要抗体 (见材料表) 到阻塞溶液浓度为 1:500, 孵育三天, 在4摄氏度。
- 将多余的原发抗体从视网膜中冲洗六次, 按顺序将其放入六个井中, 1 毫升0.1 米 PBS, 每室温下10分钟。
- 安置视网膜在一个井与新鲜的阻拦的解答 (1.7% 海卫 X-100 和5.2% 驴子正常血清在 0.1 M PBS) 和增加驴子抗兔 Alexa-594 二级抗体 (参见材料表)。用次级抗体在4摄氏度的夜间孵育视网膜。
- 将多余的二次抗体从视网膜中冲洗六次, 按顺序将其放置在六口水井中, 1 毫升新鲜0.1 米 PBS, 每室温下都有10分钟。
- 使用镊子, 将安装的视网膜转移到含有0.1 米 PBS 的培养皿中。在视网膜和细胞膜之间轻轻插入镊子的尖端, 直到视网膜不再附着, 从硝化膜中释放视网膜。
- 用镊子轻轻地将视网膜贴在玻璃显微镜上, 直到视网膜粘在玻璃上, 然后从培养皿中取出幻灯片。
- 用 Aquamount 遮住滑梯上的视网膜, 用 #1 盖住它. 5 盖玻片。将幻灯片放在幻灯片托盘中 (见材料表), 并允许它在室温下坐一个小时。
- 在不使用时, 将幻灯片返回到冰箱并存储在幻灯片托盘中 (见材料表) 4 摄氏度。在幻灯片 coverslipped 24 小时后, 用指甲油封住幻灯片的两侧以防止干燥。
注意: 协议可以在这里暂停。
4. 用视蛋白重建的视网膜 Immunostained 识别视网膜方向
- 用共焦显微镜或 epifluorescent 显微镜与照相机附件 (见材料表) 可视化 s-视蛋白梯度, 使整个视网膜在一个图像中可见 (图1B、2D、3A和3D). 这可以通过在低放大率的切片中对视网膜进行成像, 然后将图像拼接在一起。
- 命名视网膜, 使其可识别。例如, 命名第一个要重建的视网膜 "Retina1"。
- 下载并安装 ImageJ 在 https://imagej.nih.gov/ij/download.html。
- 为需要重建的每个视网膜创建单个文件夹, 但保留文件夹为空。例如, 创建标题为 "Retina1" 的文件夹。重建此视网膜所需的所有文件将在后续步骤中放置在此文件夹中。
注意: 这些文件夹应该包含的文件只有 Retistruct 要分析的文件。除下面详细说明的任何文件之外, Retistruct 软件都无法打开视网膜。 - 选择文件→打开 ImageJ 的视网膜图像, 然后选择 "Retina1"。
- 在不对图像进行任何更改的情况下, 将其保存为 "图像. png", 然后选择 "文件→保存为→ png" 以 "视网膜 1" 文件夹命名。
注: 该文件必须命名为 "图像. png", 以便 Retistruct 软件识别为重建视网膜的文件。 - 使用分段线工具勾勒出视网膜的边缘。通过点击在视网膜边界上的两个相邻斑点, 分割线工具将基本上 "连接点" 之间的两个相邻的斑点, 创建一个轮廓。重复, 直到整个视网膜被概述。将视网膜轮廓保存为标题为 "Retina1" 的文件夹, 选择 "分析→工具→ roi 管理器→添加 [t] →更多→保存"。
注意: 视网膜的边缘可以被识别在视蛋白染色过渡到背景的地方。 - 按照步骤4.7 中的指示, 使用分段线工具来勾勒出光学盘的边框。将光盘轮廓保存为 "Retina1" 文件夹, 选择 "分析→工具→ roi 管理器→添加 [t] →更多→保存"。
注意: 视盘被确定为视网膜中央的小孔, 根据解剖质量的不同而有所不同。
注意: Retistruct 重建所需的所有文件 ("图像. png"、"大纲. roi" 和 "od roi") 现在应该保存在文件夹 "Retina1" 中。 - 要下载、安装和打开 Retistruct 程序, 请按照 Sterratt 的补充材料部分 Retistruct 用户指南中概述的说明进行操作. 24
- 出现 Retistruct 窗口后, 单击窗口左上方的 "打开" 图标, 然后选择目录文件夹 "Retina1"。
- 将弹出一个图像窗口, 指示不存在缩放条。单击 "关闭", 将在框中显示视网膜图像。通过单击窗口右上的 "属性" 按钮, 将轮廓颜色更改为可见的颜色 (图 5a), 可视化视网膜轮廓。
- 重要: 指定视网膜是从右眼还是左眼在面板左侧 (图 5a)。
- 单击左侧的 "添加撕裂" 按钮, 并通过单击三个顶点的撕裂 (图 5a) 指定在视网膜中撕裂或切口的位置。这将创建连接剪切的三个顶点的线条。对视网膜的所有切口重复。
- 通过点击视网膜轮廓的任意点来指定背侧视网膜。一个大写字母 "D" 将出现在轮廓的那个点上 (图 5B)。
注意: 视网膜的背面是较暗的一半, 与 s-视蛋白梯度相反。然而, Retistruct 背视网膜的标记不是识别视网膜背半的可靠方法, 因此在这个步骤中, "背部" 的标记可以是任意的。 - 通过点击屏幕左上方的 "重建视网膜" 按钮重建视网膜 (图 5B)。重建后的视网膜的极图将出现, 切口与轮廓的颜色相同 (图 5C)。
- 单击屏幕右侧的 "保存" 按钮, 使重建的视网膜和与之关联的所有数据都保存在 "Retina1" 文件夹目录中 (图 5D)。
- 通过单击右侧面板中的 "PDF" 按钮保存重建后的视网膜 (图 5D)。将出现一个框, 要求尺寸规格。以下步骤可接受默认大小。此操作将将重建后的视网膜保存为 "Retina1" 文件夹目录中的 "image.polar.pdf"。
- 在画图程序 (或其他图像操作程序) 中打开 "image.polar.pdf", 并使用 "画图桶" 工具 (或类似) 将重建后的视网膜的背景改变为黑色。将重建后的视网膜保存为. tif 文件, 如 "Retina1" 文件夹目录中的 "Retina1_reconstructed. tif"。
注意: 协议可以在这里暂停。 - 下载用于旋转称为 "Retina_Rotator" 的视网膜的 MATLAB 代码 (参见补充材料)。将代码文件放在其自己的文件夹中, 文件夹中没有其他文件。
- 打开 MATLAB, 2007b 版或更高版本。双击代码文件以在 MATLAB 中打开它。在命令窗口中, 键入 "Retina_Rotator", 然后按 enter 键。将出现一个搜索窗口。
注意: 代码特定于. tif 文件。如果要旋转的文件格式不正确, 则代码不会正确旋转视网膜。请参见步骤4.17 和4.18。以正确的格式保存重建的视网膜。 - 打开要旋转的文件。例如, 选择 "Retina1_reconstructed. tif"。然后, 该代码将分析重建后的视网膜, 并在原始文件所在的文件夹中自动将旋转的视网膜保存为 "Retina1_reconstructed_rotated. tif"。
- 在代码完成对视网膜的分析之后, 还会出现一个窗口, 显示在旋转前后的视网膜图像进行比较 (图 3B和3C;图 3E和3F)。
注意: 这个代码旋转重建的视网膜, 使腹 (最亮) 的一半是在底部和背部 (暗) 的一半是在顶部, 从而准确地定位视网膜根据 s-视蛋白梯度1。如果视网膜是从右眼或左眼被记录在案, 鼻腔和颞极的位置也可以从这个方向的方法推断 (图 3)。
结果
单一的减压切口, 平分的直肌, 准确和可靠地识别背侧视网膜 (图 1)。脉络膜裂准确、可靠地识别鼻、颞部视网膜, 并沿颞、鼻脉络膜裂深泄切口 (图 2)。在这个例子中, 在背视网膜上也做了一个减压切口, 以确定视网膜的背侧/腹轴 (图 2D, 垂直箭头)。这些过程的步骤显示为将来 dissectors 复制的目的。视蛋白免疫组化 (图 3A 和3D), 重建与 Retistruct 软件 (3B, 3E) 和精确旋转的自定义 MATLAB 代码 (3C, 3F) 允许确定视网膜的腹侧和背半部分, 以及鼻腔和颞极, 如果它是已知的视网膜是从右或左眼 (图 3)。我们还比较了两种常用的 s-视蛋白抗体, 以有效地标记 s-视蛋白锥 (图 4A D): 山羊抗 s-视蛋白原抗体和兔抗 s-视蛋白原抗体有效标签视蛋白锥 (图 4E) 在同一鼠标。
在视蛋白 immunostained 重建的视网膜上确定了减压切口, 并将其位置与视蛋白梯度确定的定位进行了比较。使用我们的自定义 MATLAB 代码 (参见补充材料), 视网膜被准确地旋转, 以便最高浓度的 s-视蛋白染色位于腹部, 从而放置真正的背部在 90° (为上级直肌), 真正的鼻在 0° (鼻脉络裂) 和真颞在 180° (颞脉络膜裂)。根据视蛋白梯度对视网膜进行旋转后, 用 ImageJ 角工具确定各切口角的值。计算每个减压切口类型的平均角度, 然后在极图上绘制每个减压切口类型的平均值 (图 6)。平均来说, 直肌切开后的 96.3 4.3° (n = 11) 的背极被确定 (图 6)。鼻脉络膜裂为 6.7 5.8°, 颞脉络膜裂为 172.0 4.4° (n = 9) 的颞极;图 6)。
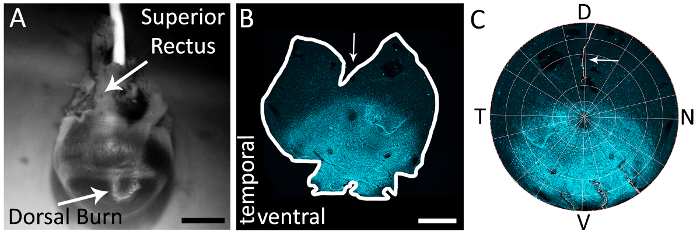
图 1: 使用上位直肌, 准确地识别右眼的背视网膜.(A) 在角膜巩膜边界附近用烧灼笔尖笔 (白色箭头) 制成的背角膜烧伤的例子。在这个视图 (白色箭头) 也可以看到优越的直肌。(B) 以平分上直肌为例, 通过在背视网膜上进行减压切口的全安装视网膜。箭头描绘了由平分上直肌的背部视网膜所做的深层减压切口。视网膜染原抗体山羊抗 s-视蛋白 (见材料表) 和二级抗体驴抗山羊 Alexa 594 (见材料表; 励磁: 590 nm; 发射: 620 nm) (青色)。视网膜被成像与一个 epifluorescent 显微镜与得克萨斯红色过滤器 (595 毫微米)。(C) 在 Retistruct 中重建的视网膜, 并以自定义的 MATLAB 代码旋转 (参见补充材料) 与上直肌减压切口可见 (白色箭头)。背, V: 腹侧, T: 颞, 鼻。刻度条 = 1 毫米.请点击这里查看这个数字的大版本.

图 2: 利用脉络膜裂缝准确识别右眼视网膜的鼻腔和颞极.(A) 在角膜巩膜边界附近用烧灼笔尖笔进行的背角膜烧伤的例子。(B) 在巩膜上的眼睛背面可见脉络裂 (白色箭头)。背角膜烧伤也可见, 在这个观点, 位于90°从颞脉络膜裂。(C) 在巩膜上眼睛背面可见的脉络裂, 从视神经到角膜巩膜边界。(D) 带有山羊抗 s-视蛋白的视网膜 (见材料表) 和二级抗体驴抗山羊 Alexa 594 (见材料表; 励磁: 590 nm; 发射: 620 nm) (青色), 脉络膜裂伤 (水平箭头) 和背部减压切口 (垂直箭头)。视网膜被成像与一个 epifluorescent 显微镜与得克萨斯红色过滤器 (595 毫微米)。(E) 在 Retistruct 中重建的视网膜, 并用自定义的 MATLAB 代码 (见补充材料) 进行旋转, 背部减压切口和鼻腔和颞脉络膜裂隙切割可见 (白色箭头)。背, V: 腹侧, T: 颞, 鼻。刻度条 = 1 毫米.请点击这里查看这个数字的大版本.
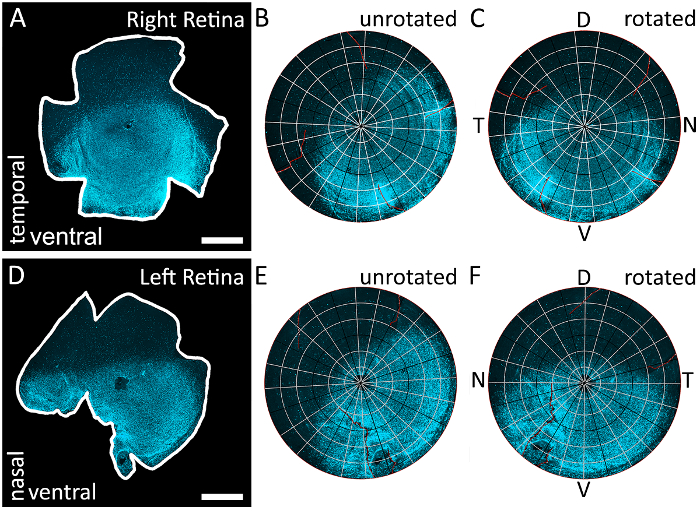
图 3: 使用 s-视蛋白梯度来识别视网膜的所有四个极.(a) 从右眼中 immunostained 的视网膜的一个例子, 它已被标记为视蛋白, 并用 epifluorescent 显微镜与德州红色过滤器 (595 nm) 进行成像。这种视网膜的切口是任意的, 因为地形方向是由 s-视蛋白梯度决定的。(B) 用 Retistruct 重建视网膜的结果。请注意, 视蛋白渐变没有正确对齐, 因为视网膜尚未通过自定义的 MATLAB 代码运行 (请参阅补充材料)。(C) 将视网膜与自定义代码旋转的结果。视网膜已经旋转, 使最高浓度的 s-视蛋白染色位于底部, 并确定为腹侧视网膜。因为视网膜是从右眼, 颞极位于90°逆时针从背极和鼻杆位于90°顺时针从背极。(D) 从左眼中 immunostained 的视网膜的一个例子, 它已被标记为视蛋白, 并以德州红色过滤器 (595 nm) 进行成像。这种视网膜的切口是任意的, 因为地形方向是由 s-视蛋白梯度决定的。(E) 用 Retistruct 数字重建视网膜 D 的结果。请注意, 视蛋白渐变没有正确对齐, 因为该视网膜尚未由自定义代码旋转。(F) 在 D 中用自定义代码旋转视网膜的结果。视网膜已经旋转, 使最高浓度的 s-视蛋白染色位于底部, 并确定为腹侧视网膜。因为视网膜是从左眼, 鼻杆位于90°逆时针从背极和颞极位于90°顺时针从背极。背, V: 腹侧, T: 颞, 鼻。刻度条 = 1 毫米.请点击这里查看这个数字的大版本.

图 4: 标记视蛋白锥的两种主要 s-视蛋白抗体的比较.(a) 视网膜上沾有山羊抗 s-视蛋白原抗体 (见材料表)。 (B) 同一老鼠的另一视网膜上沾有兔抗 s-视蛋白的原抗体 (见材料表)。(C) 一个具有代表性的区域 (0.1 x 0.1 毫米2), 其视网膜上沾有山羊抗 s-视蛋白的原发性抗体。在 epifluorescent 显微镜下拍摄的40X 放大图像。(D) 一个具有代表性的区域 (0.1 x 0.1 毫米2), 其视网膜上沾有兔抗 s-视蛋白 (见材料表), 是一种主要的抗体替代品。图像是在 epifluorescent 显微镜下拍摄的, 比例为40X。(E) 两种抗体都标有相同数量的 s 锥外段, 因为在任何经测试的视网膜上, 山羊抗 s-视蛋白和兔抗 s-视蛋白染色的阳性 s 锥数没有显著差异怪癖 (n = 2;方差分析与后Bonferroni 试验;p > 0.05)。刻度条 = 1 毫米 (A B);25µm (C D)。请单击此处查看此图的较大版本.
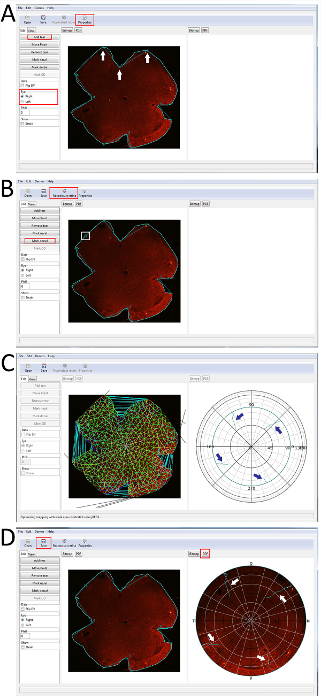
图 5: 使用 Retistruct 软件重建视网膜 immunostained 与视蛋白的视觉指南.(a) 在 Retistruct 中打开的视网膜, 轮廓可见, 并添加了 "撕裂"。"泪花" 的点用叠加的白色箭头表示。所有切口在这视网膜是任意的, 因为没有特别地标被用于标记视网膜方向在解剖期间。重要按钮用红色勾勒出来。(B) 视网膜上有 "泪", 视网膜边缘有 "D" 的背视网膜。请注意, "重建视网膜" 按钮现在可见。重要按钮用红色勾勒出来。(C) 重建视网膜的过程。重建后的视网膜的极图将出现在右侧, 显示出青色的减轻切口 (叠加在澄清切口位置的蓝色箭头)。(D) 通过 Retistruct 运行视网膜的最终结果。原 wholemount 视网膜留在左侧, 重建的视网膜出现在右侧。解除削减是可见的青色 (白色箭头叠加, 以澄清切割位置)。请单击此处查看此图的较大版本.
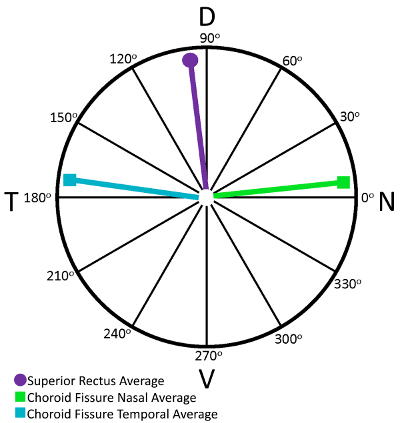
图 6: 直肌和脉络膜裂可用于准确定位小鼠视网膜.从上腹直肌减压切口或脉络膜裂隙中获得的从 Retistruct 重建的视网膜上的角度的极图。在视蛋白 immunostained 重建的视网膜上发现了减轻切口, 并将其位置与 s-视蛋白梯度的位置进行了比较。使用自定义的 MATLAB 代码准确地旋转视网膜, 使视蛋白染色的最高浓度位于腹部, 真正的背 (90°为优越的直肌), 真正的鼻 (为鼻脉络膜裂的回旋) 和真实的时间 (180°为颞脉络膜裂) 是为每个视网膜确定的。在 ImageJ 中确定了每个减压切口角的值, 并计算了每个减压切口类型的平均角度。在 96.3 4.3° (n = 11) 中, 直肌切开后发现背极。鼻脉络膜裂 6.7 5.8°, 颞脉络膜裂为 172.0 4.5° (n = 9) 的颞极。请单击此处查看此图的较大版本.
| 深地标 | 角膜烧伤部位 | 视网膜极确定 | 实验应用 |
| 高级直肌 | 背 | 背 | 居住或固定 |
| 鼻脉络膜裂 | 背 | 鼻 | 居住或固定 |
| 颞脉络膜裂 | 背 | 时间 | 居住或固定 |
| s-视蛋白梯度 | 没有 | 背, 腹, 鼻, 颞 | 固定 |
表 1: 深部地标, 他们识别的视网膜的极, 以及它们是否可以用于活体或固定的组织应用。
讨论
目前还没有全面的标准化的协议来确定和标记孤立的小鼠视网膜在解剖空间的方向。这里详细的协议试图填补这个空白, 标准化和详细说明如何使用深解剖地标作为参考点, 以可靠地识别视网膜方向。结果表明, 本协议中的深部解剖标志提供了一种更准确、更可靠的方法来定位小鼠视网膜, 而不是像角膜烧伤22这样的表面标志物。因此, 依赖角膜烧伤视网膜定向的研究可能比依赖于诸如直肌和脉络膜裂隙等地标的研究有更大的方向性错误。这种差异突出了这一标准化议定书对解释结果的必要性和意义, 并对依赖准确的视网膜定向的研究进行比较。总体而言, 标准化的协议将为视觉研究人员提供一个共同的方法, 从而消除了在数据获取中存在混淆变量, 这种变化可能发生在使用非标准化方法来识别视网膜取向。
这里提出的方法很容易重复, 适用于许多类型的实验协议。事实上, 该协议的最大优点之一就是它的适应性。由于脉络膜裂、s-视蛋白表达、直肌等标志物均已被发现可靠地识别视网膜方位22最适合的标志性实验参数可选择优化数据采集 (表1). 此外, 可以结合解剖方法, 进一步阐明视网膜的方向。例如, 脉络膜裂隙切口可以与 s-视蛋白免疫组化相结合, 以适应视网膜的所有四个极: 鼻和颞半球可以通过脉络膜裂隙的切割来识别, 而 s-视蛋白免疫组织化学可以识别腹侧和背半球。然而, 该协议的适应性可能受到生理实验的时间敏感性的制约。由于确定一个地标的时间, 做一个角膜烧伤, 并执行减轻削减可能导致重大组织死亡的活体实验, 其中一些解剖方法可能是低于最佳。幸运的是, 一旦一个刀已经熟悉了脉络膜裂或上直肌解剖方法, 确定的深地标和使减轻削减迅速成为解剖例程的一部分, 并没有显著增加解剖长度。虽然我们承认, 这里详细的步骤可以增加时间对非常时间敏感的实验, 我们建议使用 s-视蛋白梯度的后视网膜定向后, 组织的生存能力不再是一个问题 (图3).视蛋白视网膜染色是一种有效的方法来定位视网膜, 因为它可以识别所有四个极点: s-视蛋白染色将视网膜分为背部和腹侧极, 并根据视网膜是否能识别鼻腔和颞极。是从右或左眼 (图 3)。因此, 我们认为该协议提供了可靠和可重复的一套方法, 以准确的视网膜方向, 可以满足任何实验参数。
与任何改良的视网膜解剖一样, 解剖方法的有效性受到刀的准确性和被隔离的组织质量的限制。如果在解剖过程中丢失了任何组织, 或者视网膜过于错位, 无法精确重建, Retistruct 和 MATLAB 程序将无法可靠地重建或定位视网膜。因此, 在使用该方法进行数据收集实验之前, 必须进行解剖法的实践。虽然这里解释的解剖类型并不困难, 但必须进行练习, 以确保识别视网膜方向的重复性与特定的地标。此外, 刀在开始收集数据之前, 必须在视觉上识别出解剖学地标, 以确定正确的地标正在使用。检查某一特定刀的准确性的一种方法是使脉络裂切口或优越的直肌切开, 然后将切口的位置与 s-视蛋白梯度进行比较, 因为它是一个固定的标记, 因此不依赖于 dissectio 的准确性。潜在的 dissectors 还可以将其重建的视网膜与重建后的视网膜的例子进行比较, 并进行精确的标志性切割, 如图 1 和图 2所示。本质上, 一个潜在的刀应该执行本协议中概述的特定解剖类型的步骤, 无论是直肌或脉络膜裂法, 并将结果与 s-视蛋白梯度进行比较, 以确定特别刀。因为如果刀不确定地标的位置, 它可能导致不准确的视网膜方向, 默认情况下, 会影响数据的收集和解释。
披露声明
作者没有什么可透露的。
致谢
我们要感谢布列塔尼天和杰西卡 Onyak 为他们的技术援助和刘医生好心让我们使用他的 epifluorescent 显微镜。支持的鸣谢: NIH R15EY026255-01 和卡尔 Kirchgessner 基金会。
材料
| Name | Company | Catalog Number | Comments |
| 0.1 M Phosphate Buffered Saline | Sigma-Aldrich | P5244 | |
| Axioplan2 Epifluorescent Microscope | Zeiss | N/A | |
| Clear Nailpolish | N/A | N/A | |
| Corning LSE Low Speed Orbital Shaker | Sigma-Aldrich | CLS6780FP | |
| Costar TC-Treated 24-well Plates | Sigma-Aldrich | CLS3524 | |
| Dissection Microscope | Olympus | SZ51 | |
| Donkey anti-Goat Alexa 594 | Life Technologies | A11058 | |
| Donkey anti-Rabbit Alexa 594 | Life Technologies | A21207 | |
| Donkey Normal Serum | Millipore | 566460 | Use at 5.2% (52 μL with 86 μL of 20% Triton X-100 and 863 μL of 0.1M PBS for 1 mL of blocking solution) |
| Fisherbrand Superfrost Plus Microscope Slides | Fisher Scientific | 12-550-15 | |
| Goat anti-s-opsin | Santa Cruz Biotechnologies | sc-14363 | Not commerically available as of 2017 |
| Graefe Curved Forceps | Fine Science Tools | 11052-10 | |
| ImageJ or FIJI | National Institute of Health | N/A | Freely available software |
| Low Temperature Cautery Ophthalmic Fine Tip Cauterizer | Bovie Medical Corporation | AA00 | |
| MATLAB | MathWorks | N/A | At least version 2007b or later |
| Micro Cover Glasses | VWR International | 48393-241 | |
| Micro Slide Trays | VWR International | 82020-913 | |
| Moira Ultra Fine Forceps | Fine Science Tools | 11370-40 | |
| Nitrocellulose membrane | Millipore | HAWP04700 | |
| Paraformaldehyde | Electron Microscopy Sciences | 15714-S | Use at 4% (25 μL and 875 μL of 0.1 M PBS for 1 mL of fixative) |
| PrecisionGlide Needle 20G (0.90 mm x 25 mm) | BD PrecisionGlide | 305175 | |
| Pyrex Glass Petri Dish | Sigma-Aldrich | CLS3160152 | |
| R | The R Project for Statistical Computing | N/A | Freely available software; version 3.4.3 or later |
| Rabbit anti-s-opsin | Millipore | ABN1660 | |
| Retiga R3 Microscope Camera | Qimaging | 01-RET-R3-R-CLR-14-C | |
| Retistruct | N/A | N/A | Freely available software compatiable with Windows 7 or Windows 10 |
| Shandon Aqua-Mount Slide Mounting Media | Fisher Scientific | 14-390-5 | |
| Triton X-100 | Sigma-Aldrich | T8787 | Use 1.7% (86 μL of 20% Triton-X with 52 μL of Donkey Normal Serum and 863 μL of 0.1 M PBS for 1 mL of blocking solution) |
| Vannas Spring Dissection Scissors | Fine Science Tools | 15000-03 | |
| 5MP USB Microscope Digital Camera | AmScope | MU500 | To be used with the Olympus Dissection Microscope |
参考文献
- Applebury, M. L., et al. The murine cone photoreceptor: A single cone type expresses both S and M opsins with retinal spatial patterning. Neuron. 27 (3), 513-523 (2000).
- Hughes, S., Watson, T. S., Foster, R. G., Peirson, S. N., Hankins, M. W. Nonuniform distribution and spectral tuning of photosensitive retinal ganglion cells of the mouse retina. Curr Biol. 23 (17), 1696-1701 (2013).
- Sondereker, K. B., Onyak, J. R., Islam, S. W., Ross, C. L., Renna, J. M. Melanopsin ganglion cell outer retinal dendrites: Morphologically distinct and asymmetrically distributed in the mouse retina. J Comp Neurol. 525 (17), 3653-3665 (2017).
- Bleckert, A., Schwartz, G. W., Turner, M. H., Rieke, F., Wong, R. O. L. Visual space is represented by nonmatching topographies of distinct mouse retinal ganglion cell types. Current Biology. 24 (3), 310-315 (2014).
- Warwick, R. A., Kaushansky, N., Sarid, N., Golan, A., Rivlin-Etzion, M. Inhomogeneous Encoding of the Visual Field in the Mouse Retina. Curr Biol. 28 (5), 655-665 (2018).
- Valiente-Soriano, F. J., et al. Distribution of melanopsin positive neurons in pigmented and albino mice: evidence for melanopsin interneurons in the mouse retina. Front Neuroanat. 8, 131(2014).
- Sabbah, S., et al. A retinal code for motion along the gravitational and body axes. Nature. 546 (7659), 492-497 (2017).
- Vaney, D. I., Sivyer, B., Taylor, W. R. Direction selectivity in the retina: Symmetry and asymmetry in structure and function. Nat Rev Neurosci. 13 (3), 194-208 (2012).
- Huberman, A. D., et al. Genetic identification of an On-Off direction-selective retinal ganglion cell subtype reveals a layer-specific subcortical map of posterior motion. Neuron. 62 (3), 327-334 (2009).
- Ueki, Y., Ramirez, G., Salcedo, E., Stabio, M. E., Lefcort, F. Loss of Ikbkap causes slow, progressive retinal degeneration in a mouse model of familial dysautonomia. eNeuro. 3 (5), (2016).
- Maiorano, N. A., Hindges, R. Restricted perinatal retinal degeneration induces retina reshaping and correlated structural rearrangement of the retinotopic map. Nat Commun. 4, 1938(2013).
- Hadj-Said, W., et al. Quantitative and topographical analysis of the losses of cone photoreceptors and retinal ganglion cells under taurine depletion. Invest Ophthalmol Vis Sci. 57 (11), 4692-4703 (2016).
- Tao, Y., et al. The temporal topography of the N-Methyl- N-nitrosourea induced photoreceptor degeneration in mouse retina. Sci Rep. 5, 18612(2015).
- Risner, M. L., Pasini, S., Cooper, M. L., Lambert, W. S., Calkins, D. J. Axogenic mechanism enhances retinal ganglion cell excitability during early progression in glaucoma. Proc Natl Acad Sci U S A. , (2018).
- Estevez, M. E., et al. Form and function of the M4 cell, an intrinsically photosensitive retinal ganglion cell type contributing to geniculocortical vision. J Neurosci. 32 (39), 13608-13620 (2012).
- Kolesnikov, A. V., Kefalov, V. J. Transretinal ERG recordings from mouse retina: Rod and cone photoresponses. J Vis Exp. (61), (2012).
- Lin, B., Wang, S. W., Masland, R. H. Retinal ganglion cell type, size, and spacing can be specified independent of homotypic dendritic contacts. Neuron. 43 (4), 475-485 (2004).
- Ortin-Martinez, A., et al. Number and distribution of mouse retinal cone photoreceptors: differences between an albino (Swiss) and a pigmented (C57/BL6) strain. PLoS One. 9 (7), 102392(2014).
- Zhang, H., et al. The degeneration and apoptosis patterns of cone photoreceptors in rd11 Mice. J Ophthalmol. 2017, 9721362(2017).
- Wei, W., Elstrott, J., Feller, M. B. Two-photon targeted recording of GFP-expressing neurons for light responses and live-cell imaging in the mouse retina. Nat Protoc. 5 (7), 1347-1352 (2010).
- Wang, J., et al. Anatomy and spatial organization of Muller glia in mouse retina. J Comp Neurol. 525 (8), 1759-1777 (2017).
- Stabio, M. E., et al. A novel map of the mouse eye for orienting retinal topography in anatomical space. J Comp Neurol. 526 (11), (2018).
- Lamb, T. D., Collin, S. P., Pugh, E. N. Evolution of the vertebrate eye: Opsins, photoreceptors, retina and eye cup. Nat Rev Neurosci. 8 (12), 960-976 (2007).
- Sterratt, D. C., Lyngholm, D., Willshaw, D. J., Thompson, I. D. Standard anatomical and visual space for the mouse retina: Computational reconstruction and transformation of flattened retinae with the Retistruct package. PLoS Comput Biol. 9 (2), 1002921(2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。