Method Article
多年生草试验中土壤、根际和根系微生物群落的分离与分析
摘要
详细描述了植物根系从田间开挖以及样品加工到 endosphere、根际和土壤的情况, 包括 DNA 提取和数据分析方法。本文旨在使其他实验室能够利用这些技术来研究土壤、endosphere 和根际微生物。
摘要
植物和土壤微生物学研究对了解微生物在农业生产力中的作用越来越重要。本手稿的目的是提供详细说明如何快速取样土壤, 根际和 endosphere 的复制田间试验和分析可能发生的变化, 在微生物群落由于样本类型, 治疗和植物基因型。用于演示这些方法的实验包括包含两个纯、暖季草 (柳枝柳枝稷和须芒草 gerardii)和低多样性草混合物 (gerardii、Sorghastrum) 的复制场地块。垂穗和Bouteloua curtipendula)。简单地, 植物被挖掘出来, 各种根被切开并放置在磷酸盐缓冲物中, 然后摇动以收集根际。根被带到实验室的冰和表面消毒漂白和乙醇 (乙醇)。用离心法过滤和浓缩根际。从根球周围挖出的泥土被放进塑料袋里, 带到实验室里, 那里有少量的泥土用于 DNA 提取。DNA 从根、土壤和根际提取, 然后用引物放大 16S rRNA 基因的 V4 区。Amplicons 的顺序, 然后分析与开放的访问生物信息学工具。这些方法使研究人员能够测试微生物群落的多样性和组成因样本类型、治疗和植物基因型而异。利用这些方法和统计模型, 表明根系、根际和土壤微生物群落存在显著差异。这里提供了一套完整的步骤来收集野外样本, 分离, 提取, 量化, 放大, 序列 DNA, 并分析微生物群落多样性和组成的复制田间试验。
引言
微生物研究对于理解和操纵生态系统过程, 如养分循环、有机质周转和土壤病原体的发育或抑制有重要意义1、2。这一研究领域也为了解土壤微生物对自然植物群落和农田生产力的影响提供了巨大的潜力。尽管有许多研究集中在自然生态系统中的土壤微生物群中, 但在农田3中, 对植物根际和 endosphere 微生物的关注较少。在内布拉斯加州, 农业主导着整个州大部分地区的景观, 对这些土壤进行研究, 农业上重要的农作物成为研究的重要课题。本方法的目的是为研究人员提供一组标准的协议来描述农田中的微生物, 以确定植物根系如何改变根际和 endosphere 的微生物群落, 并最终了解这些微生物在土壤健康和植物生产力中发挥的作用。
此处提出的方法与其他4、5中使用的方法略有不同, 因为本文旨在了解哪些微生物完全位于根内, 以及它们与直接在根外的微生物有何不同。根 际。本研究中使用的扩增子测序确定了 DNA 样本中发现的微生物分类, 并允许调查人员根据样本类型或治疗来确定群落的变化。此协议与 Lundberg 所使用的非常相似的协议之间的关键区别之一 。6是, 这个协议使用表面杀菌与漂白剂和乙醇去除根际根的超声波。其他还使用了有效的表面杀菌7,8,9,10。这些方法比其他方法更有利, 但略有不同。这些方法特别适合于大型野外试验, 因为有足够的人每天可以处理超过150场地块, 在划分成 endosphere、根际和土壤时增加约450个样本。本手稿详细描述了用于实地取样的方法, 处理实验室中的材料, 提取和序列 DNA, 并简要概述了分析结果测序数据的步骤。
研究方案
1. 现场介绍
- 在收集期间描述实验现场站点。使用 GPS 确定域 (纬度、经度和高度) 的位置。
- 描述取样深度、取样时间和土壤质地。
- 环境因素对微生物群落的形成起着重要的作用。记录气候信息, 如年平均气温、年降水量、往年作物轮作、耕作做法、施肥方法和现场历史。自动气象站或其他设备是有用的记录每天的降雨量和气温在一个生长季节。
2. 土壤、根际和根场样品的收集和处理
- 植物的挖掘。
- 将洗涤盘和水桶贴上标签, 其中包含有关要取样的植物材料的信息。包括绘图号、植物基因型和植物种类等信息。将标签桶带到绘图区, 并将洗涤盘放在现场的固定工作站后面。
- 用铲子刺穿土壤, 深度为30厘米, 以切割在土壤中保持植物的任何侧根。近似体积为18厘米3。随机选择和收集两个植物每个情节从不同的区域内的情节。
- 利用铲子挖掘植物根系, 将根球放在标记的桶中。把被挖掘的根球带上标记的水桶回到现场的工作站。切断和丢弃地上植物的生物量。
- 从根部清除土壤和收集大块土。
- 摇动根部, 手动移除土壤或使用铁锹或手持的分蘖从根部去除土壤。摇动根部足以去除非常沙土中的土壤。在加工站附近戴上手套并放置根部。
- 摇根后, 土壤的大部分将在洗涤锅里。在洗涤盘中混合土壤, 用手持的分蘖分解任何泥土。将不含碎片的泥土样本放置在标签的 17.7 x 19.5 厘米拉链储存袋中, 放置在阴凉处或冰上。
- 根和根际的汇集。
- 使用修剪剪刀消毒在70% 乙醇, 消费的各种根, 大约4到6根每株植物和每个根约 9-12 厘米长度。将切除根 (切割所需的) 放置在含有35毫升蒸压的标签50毫升管中, 磷酸盐缓冲器 (6.33 克/升 NaH2PO4, 8.5 克/升 Na2HPO4无水, pH = 6.5, 200 µl/升表面活性剂)。
注: 表面活性剂 (见材料表) 在热处理磷酸盐缓冲液后添加。根球的体积和根长度的变化取决于植物的年龄和植物种类。 - 摇管2分钟, 从根部表面释放根际。用镊子在70% 乙醇消毒, 去除根从管, 在纸巾上涂抹, 并放置在一个新的, 标记为50毫升管。把包含根际的管和含有根的管子放在冰上。
- 使用修剪剪刀消毒在70% 乙醇, 消费的各种根, 大约4到6根每株植物和每个根约 9-12 厘米长度。将切除根 (切割所需的) 放置在含有35毫升蒸压的标签50毫升管中, 磷酸盐缓冲器 (6.33 克/升 NaH2PO4, 8.5 克/升 Na2HPO4无水, pH = 6.5, 200 µl/升表面活性剂)。
3. 实验室现场样品的处理
- 田间采集后根部的表面灭菌。
注: 从现场返回后, 应尽快进行表面杀菌。如果不可能在同一天表面消毒根, 贮存根在4摄氏度, 直到处理。- 添加大约35毫升50% 漂白剂 + 0.01% 吐温20到50毫升根管收集在该领域。摇动50毫升管30到60秒。倒入50% 漂白剂, 加入35毫升70% 乙醇。再摇30到60秒。
- 倒入70% 乙醇, 加入35毫升无菌、超纯水。摇1分钟。重复水洗两次。
注: 为了确保我们的表面灭菌处理是足够的, 我们镀样品的水从上次冲洗和观察没有增长的细菌 (p. 王, 未发表的数据, 2014)。其他调查人员使用类似方法9、10测试了根杀菌效率。 - 在干净的纸巾上涂抹根干。对每个样品使用干净的纸巾。
注: 纸巾在使用前可以消毒。我们不消毒纸巾。然而, 我们使用1纸巾每样品和保持毛巾包装, 直到使用。 - 使用无菌钳和修剪剪刀, 把根切成大约5毫米, 并将切割根部放在一个干净的, 标记为15毫升圆锥管。将样品贮存在摄氏-80 摄氏度, 直至进一步加工。
- 加工根际样品。
- 摇动50毫升管, 包含根际样品从田间并用重悬整个样品。使用无菌, 100 µm 网状细胞过滤器 (见材料表), 过滤悬浮样品到一个新的50毫升管。
- 在室温下离心管在 3000 x g 处5分钟。立即倒掉并丢弃上清。
- 将根际颗粒放在50毫升的冰管中。加入1.5 毫升无菌磷酸盐缓冲液 (不含表面活性剂) 到根际颗粒和涡旋悬浮。
- 将悬浮液吸管到一个干净的, 标记的, 2 毫升的离心管。在室温下, 旋转管在 15871 x g 处为2分钟。立即倒入上清, 并将管子排在干净的纸巾上。
- 将小球储存在-20 摄氏度, 直到进一步加工。
注: 步骤 3.2.2-3.2.4, 以减少样品管大小的存储。与50毫升管相比, 存储较小的2毫升管的空间效率更高。
- 处理土壤样品进行 DNA 提取和土壤分析。
- 使用无菌金属铲, 填充一个干净, 标记2毫升管约3克的土壤 DNA 提取。避免任何小的根碎片和碎片。将土壤样品贮存在摄氏-20 摄氏度。在每个样品之间冲洗70% 乙醇金属刮刀。
- 在一个干净的洗涤锅里, 把泥土袋空成堆积的筛子 (见材料表), 在较小的筛子上方筛上较大的筛子, 并通过两个筛子手工筛选土壤。用刷子仔细清洗样品之间的筛子。
- 在 17.7 x 19.5 厘米的拉链袋中预留100到125克的被筛土壤, 用于未来土壤理化和纹理分析。将土壤袋放置在4摄氏度, 用于短期贮存。
- 土壤水分的土壤样品处理。
- 将一个标有褐色的纸袋包装在秤上。测量40到 45 g 被筛的土壤入褐色纸袋子。将棕色纸袋和土壤的重量记录在数据表上, 并将袋子放在烘干烤箱中, 设置为 55-60 摄氏度。
- 72小时后, 从烘干烤箱中取出袋子。让土壤袋冷却至少30分钟, 然后权衡。
- 要记录重量, 请将刻度减至零, 并将棕色纸袋放在刻度上。使用公式计算每个样品的土壤含水量百分比:
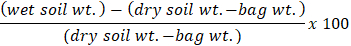
注: 土壤水分法是从凯洛格生物站长期生态研究 (LTER)。知识库 LTER 为其网站上的研究人员提供了广泛的既定协议 (https://Lter.kbs.msu.edu/)。
4. 制备用于 DNA 提取的加工根样。
- 研磨冷冻根材料, 使样品均匀。
- 将液氮倒入塑料烧杯中, 用干净的铲子, 在干净的砂浆和杵中, 使样品在整个研磨过程中冻结。
- 将冷冻组织放入砂浆中, 用杵研磨成细粉。不断地在研磨过程中添加液氮以保持样品冷冻。
- 用刮刀将地面组织放置在一个干净的, 标记为2毫升的管。存储在-80 摄氏度。
5. 96 井格式提取土壤和根际样品中的 DNA
- 将土壤样品装入96井板中。
- 用70% 乙醇和1% 漂白剂擦拭工作区域。在这些步骤中佩戴实验室手套。从-20 摄氏度的储藏中取出土壤样品, 并允许在冰桶中解冻。
- 从带有 DNA 提取试剂盒的96井萃取板上取下密封垫盖。将密封垫盖在2纸巾之间, 在不使用时保持清洁。为避免污染, 使用8井胶粘剂的 PCR 条覆盖96井萃取板12柱。
- 将一个不育的重量漏斗 (大小 SM) 在一个刻度上, 并将200到250毫克的土壤称量出来。
注: 这些无菌漏斗在一侧扁平, 因此漏斗在鳞片上平整。漏斗被泥土填满, 直接放进井里。这种技术避免了样品的丢失, 减少了溢出, 防止了交叉污染。 - 要揭开萃取板的第一井, 小心地提起胶条, 将填充的称重漏斗的颈部放在适当的井中, 轻轻地将土壤样品引导到适当的井中。更换胶条盖好。
- 重复这个过程的每一个良好的板块, 使用一个新的, 不育漏斗为每个样品, 直到该板块填充。保留一个井空作为提取空白控件。
注: 每个萃取板上都留有一个井, 作为负 (空白) 控制。对试剂盒试剂11中可能存在的污染物的控制。 - 在萃取板上更换密封垫盖, 并将该板材贮存在-20 摄氏度, 直到准备好进行 DNA 提取。
- 将根际样品装入96井板中。
- 从-20 摄氏度的储藏中去除根际样品, 并允许在冰桶中解冻。
- 按照以上步骤5.1.2 准备 DNA 提取板。
- 将干净的纸擦在秤上, 然后将无菌金属铲抹在秤上。用刮刀小心地从样品管中挖出一些根际颗粒。把刮刀退回到鳞片上, 并在根际样品的200和250毫克之间称量。
- 小心地提起胶条, 揭开萃取板的第一井, 将填充的铲入井中, 用无菌牙签将根际物质刮入适当的井中。
- 在水中冲洗金属铲, 然后在样品间乙醇70%。对板块的每一个井重复这个过程, 直到盘子填满, 留下一个井空作为提取空白控制。
- 96井格式提取根际和土壤样品中的 DNA。
- 利用为土壤优选的试剂盒提取土壤和根际 DNA (见材料表), 遵循制造商的协议。
注意: 我们使用这个特定的试剂盒来隔离土壤和根际 DNA, 因为专有试剂能够去除土壤中的腐植酸和其他功能强大的 PCR 抑制剂。
- 利用为土壤优选的试剂盒提取土壤和根际 DNA (见材料表), 遵循制造商的协议。
- DNA 量化。
- 根据制造商的协议, 量化92样品和4浓度标准 (见材料表; 标准包括在内)。
- 根据制造商的协议, 将剩余的4个样品从盘子中取出, 以容纳四标准的水井 (见材料表)。
6. 从根样品中提取96井格式的 DNA。
- 将根样品装入96井板中。
- 用70% 乙醇和1% 漂白剂擦拭工作区域。在这些步骤中戴上手套。
- 把样品放在桶里, 放在干冰里, 保持地面根部的样品冻结。
- 用液氮填充塑料烧杯, 在塑料烧杯中放置抗静电 microspatulas 和无菌称重漏斗 (尺寸 XSM) 降温。
- 从带套件的96井萃取珠板上取下密封垫盖, 并将其放在2纸巾之间, 在不使用时保持清洁。
注意: 此套件的更新版本, 在我们进行这些 DNA 提取的时间之后发布, 要求用户提供提取珠板。在材料表中, 我们列出了订购这些物料所需的供应商目录信息。 - 为避免污染, 使用8井胶粘剂的 PCR 条覆盖96井萃取板12柱。
- 将萃取珠板放在干冰上, 将样品保存在井中冷冻。
- 小心地提起胶条, 揭开萃取板的第一井, 将填充的称重漏斗的颈部放入适当的井中, 再加入3勺地根组织。更换胶条盖好。
注: 在称重之前, 土壤和根际在冰上解冻, 而植物材料则被重冷冻。冷冻植物组织, 尤其是少量的, 很难在不解冻的情况下进行秤重。在基层组织进行了重量测试, 以确定有多少铲勺是足够的。请注意, 该套件的制造商不需要确切数量的组织, 但建议约50毫克. 不同的植物样本类型将有所不同, 用户将需要确定适当的数量。 - 对板块的每一个井重复这个过程, 直到盘子填满。
- 储存板在-20 °c, 直到准备 DNA 提取。
- 根组织的 DNA 提取。
- 根据制造商的协议, 用为植物优化的套件 (见材料表) 提取 DNA。
注意: 我们使用这个套件专门为植物组织, 以达到最大产量从根样品。与土壤和根际不同的是, 腐植酸和其他污染物对根系组织的问题较小。
- 根据制造商的协议, 用为植物优化的套件 (见材料表) 提取 DNA。
- 将 DNA 量化为步骤5.4。
7. 放大和测序分离的 DNA。
- 用证明阅读聚合酶 (见材料表) 放大16S 基因的 V4 区域, 如 Gohl等。12条码样品与不同的索引底漆和池排序前。序列使用 Gohl et al12描述的方法使用两步 PCR 过程与 V4 引物 (补充文件 1)。
注意: 这些步骤的一些方法在其他12、13、14中详细描述, 因此不会在此描述。对于根样本, 添加肽拮抗剂, 以减少质 DNA 的数量从植物组织, 这已经被充分描述15。在测序中使用了两个控制, 一个负控制, 其中包括萃取板空白控制 (参见步骤5.1.5 后的注意事项), 以及已知细菌 DNA 种群的模拟社区 (见材料表), 作为一个积极的控制。
注意: 在大多数情况下, MiSeq 试剂盒 v3 用于 2 X 300 基配对端模式。对于本手稿中的样品, Illumina HiSeq 2500 用于快速模式与250配对端 (2 x 250) 模式。所有样品都在同一条车道上测序。 - 通过管道进行微生物群落分析 (USEARCH v9.2.64、QIIME v1.9.1 和 RStudio v3.4.3 16) 处理测序数据。
- 使用 USEARCH17准备排序数据。
注: USEARCH 可在线提供完整的说明 (https://www.drive5.com/usearch/)。 - 使用索引读取或条形码解排序数据, 以将 Illumina 读取分配给示例。
- 合并配对端读取以获得一致的序列。使用命令: usearch-fastq_mergepairs * R1 * fastq 重新标记备选 @ fastq_maxdiffs 10-fastq_minmergelen 230-fastq_maxmergelen 320-fastq_pctid 80-fastqout 合并。
注意: 参数是通过引用 USEARCH 指令手册来设置的。 - 从测序数据中除去引物, 以避免在底漆序列中进行替代, 这可能是由 PCR 反应引起的。使用命令: usearch-fastx_truncate 合并. stripleft 19-stripright 20-fastqout 剥离。
- 过滤测序数据以去除低质量的读数并保持高质量的操作分类单元 (OTU) 序列。使用命令: usearch-fastq_filter 剥离. fastq_maxee 1.0-fastaout 过滤。
- 使用 USEARCH17准备排序数据。
- 在 USEARCH 中生成 OTUs。
- 执行 dereplication 以标识唯一的 OTU 序列集。使用命令: usearch fastx_uniques 筛选 fastaout 独有 sizeout 重新标记备选。
- 群集 OTUs 与 97-100% 序列相似性, 以指定唯一的 OTUs。使用命令: usearch-cluster_otus 独有 minsize 2-otus otus 重新标记备选。
注意: 此步骤还包括从群集 OTUs 中移除单克隆, 并将合体从排序数据中移除。 - 在 USEARCH 中创建一个 OTU 表。使用命令: usearch-usearch_global 被剥去. otus. 发线 + id 0.97-otutabout otutable.txt。
注意: 此命令生成一个表, 其中每个示例的所有 OTUs 的读取次数 (计数) 数。OTU 表用于下游步骤, 包括差异丰度分析和微生物多样性分析。OTU 表的示例显示在补充图2中。 - 在 QIIME v1.9.1 18进行稀疏分析。
注: 稀疏曲线是用 OTU 表计算的, 以确定测序的深度是否正确地取样了微生物群落。稀疏曲线的示例显示在补充图 3中。 - 进行阿尔法多样性分析18。在 QIIME v1.9.1 中使用 alpha_rarefaction 来计算每个样本中微生物群落的多样性。
注意: 此分析计算多样性指数, 如香农19、辛普森20和 Chao121。 - 进行 beta 多样性分析18,22。使用 Python 脚本: beta_diversity_through_plots QIIME v1.9.1 Bray–Curtis 相异矩阵。
注: 本分析比较了样品间的微生物群落组成。 - 对各组进行统计分析。使用 PERMONOVA 计算的距离矩阵, 使用阿尼斯和方差分析功能在纯素包装23 v2.4.5 在 RStudio16。使用纯素包装中的 capscale 函数对主坐标 (CAP) 分析进行规范化分析。在 RStudio 中使用 ggplot2 包24 v2.2.1 可视化数据。
结果
这份手稿中的代表性成果来自于2012年在内布拉斯加州-林肯农业研究分部农场附近建立的一个实地网站, 在米德, NE. 在实验之前, 该网站已被管理为玉米-大豆轮作.该研究地点位于三种不同类型的土壤上, 但分析的数据是, 如果测量土壤性质的所有变化是由于所实施的处理。
现场网站包含两个, 纯, 柳枝稷 (P. 柳枝稷cv 自由) 和大白羊草 (A. gerardii) 以及一个低多样性的草混合物含有大白羊草, indiangrass (垂穗), 和' 山丘 ' sideoats 格兰玛 (B. curtipendula)。三暖季草地块是在随机完整的块设计, 复制了三次。嵌套在三不同的草地块是二氮气 (N) 受精处理, 是 56 (N1) 和 112 (N2) 公斤 N ha-1的应用的尿素。在生长季节结束时, 微生物取样时, 土壤中含有8.0 到 1.1 (平均的) ppm 硝酸盐在被受精的地块112公斤 n 公顷-1和 6.8 @ 0.7 (平均 + SD) ppm 硝酸盐在被施肥的地块与56公斤 n 公顷-1。这些地块每年受精一次。暖季草地块被指定为主要地块 (8000 米2) 和 N 处理是分裂地块 (4000 米2)。大白羊草被播种为50:50 混合的 ' 金矿 ' 和 ' 金矿 ' 和 Indiangrass 被播种了作为50:50 混合的 ' 侦察员 ' 和 ' 勇士 '。这些地块是在2012年种植的, 第一个 N 应用发生在2013年春季。
土壤和根系取样是在2014年9月15日进行的。下面所述的工作是在一个被设置为三复制的分块随机设计的领域进行的 (图 1)。endosphere 所有样品的平均测序深度如下: 4871 5711 (平均 * SD), 根际: 40726 @ 14684, 土壤: 38184 @ 9043。使用所描述的方法, 这些实验中最大的变异来源之一是在样本类型之间发现的微生物群落的差异 (图 2)。在这个代表性的数据集, 根际和土壤似乎比 endosphere 更相似的组合 (图 2A)。然而, 根际和土壤微生物群落组成也有很大的差异 (p = 0.001) (图 2B)。以样本类型分析的实验结果表明, 总变异率为26%。
阿尔法多样性分析表明, 与土壤和根际相比, endosphere 中微生物群落的样本多样性较低 (图 3)。白羊草和柳枝稷的 endosphere 样品之间, 任何隔间的草种之间的多样性唯一显著差异 (图 3)。相对丰度分析 (图 4) 突出了Proteobacteria在所有样本类型中跟随放线菌的优势。土壤和根际也以Acidobacteria和Chloroflexi为主, 而 endosphere 的Bacteriodetes相对丰度较大。
在本实验中, 植物生长了两种不同的氮肥, 因此我们分析了数据, 以确定是否有治疗效果。治疗效果占总变异的 12%, 但没有显著差异, 虽然在协调两种治疗看起来不同 (图 5)。这突出了统计分析对这些数据集的重要性, 而不是视觉检验或定性判断。
利用约束的协调方法, 对植物组织和土壤微生物群中植物影响的差异进行了可视化研究。统计差异是通过 PERMANOVA 分析确定的特定变量, 如物种, 导致明显不同的微生物群落组成之间的样本。在对所有样本类型进行分析的同时, 由于植物种类的不同, 在微生物群落组成上发现了显著差异 (图 6)。在本实验中, 植物种类的变异量为6.7%。最后, 对每个样本类型分别进行分析, 以确定哪种样本类型可能会推动植物物种的显著效应。只有在 endosphere 有一个高度显著的差异 (p = 0.001) 之间的微生物群落组成的不同植物种类 (图 7)。在其他样本类型中, 个别分析时, 物种效应并不显著。在 endosphere 中, 由于物种的变异率为 27%, 而在根际 (18%) 和土壤 (15%) 中的百分比则较低。这进一步强调了单独分析每个组织类型的重要性。
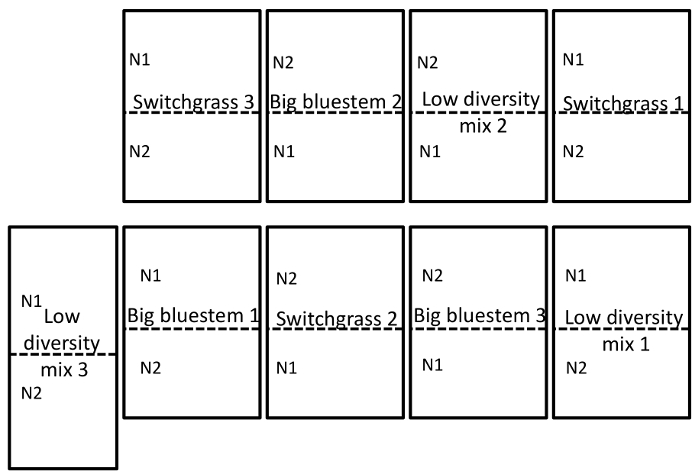
图 1: 实验场设计的示例.试验场设计, 说明了在内布拉斯加州-林肯东部内布拉斯加大学研究和推广中心附近的一三个实地网站的一个随机完整块设计, NE。有关完整站点说明, 请参阅 "结果" 部分。N1 是低 (56 公斤 n 公顷-1尿素) 和 N2 (112 公斤 n 公顷-1尿素) 是更高的氮气率被应用。请单击此处查看此图的较大版本.
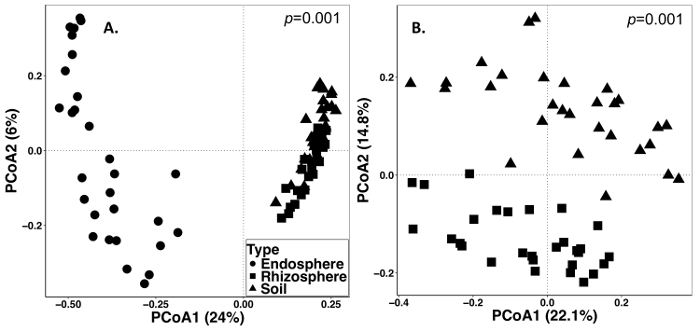
图 2: 在 2014年, 从多年生牧草取样的不同样品类型中, 包括 endosphere、根际和土壤中的微生物组成进行了β分集分析比较.用 QIIME1.9.1 中的 Python 脚本进行了分析, 生成了布雷-柯蒂斯相异矩阵。在 RStudio 中, PCoA 了基于布雷-柯蒂斯相异矩阵的主坐标分析方法。PCoA1 和 PCoA2 表示 PCoA 分析解释的第一和第二最大方差。PERMANOVA 统计分析, 以确定样本类型之间的意义, p值显示在右上角。图中的每个符号代表了每个样本的整个微生物群落。(A) Endosphere、根际和土壤样本类型一起分析。所有87样品都是稀薄到486序列每样品。(B)共同分析了根际和土壤样本。所有59个样品都是稀薄的8231序列。请单击此处查看此图的较大版本.
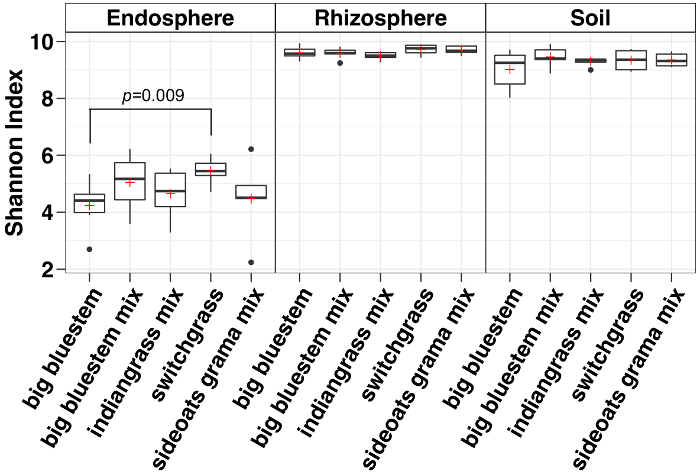
图 3: 用香农指数对 endosphere、根际和土壤中的每种物种进行阿尔法多样性分析.分析是使用 QIIME1.9.1 中的 Python 脚本进行的。对 endosphere、根际和土壤样本类型分别进行了稀疏处理, 每个样品的486、17154和8231个序列。框表示第二十五和第七十五百分点 (第一和第三分位数)。方框内的水平线表示中值, 红色加号表示平均值。胡须显示的数据范围, 排除异常 (显示为黑点), 下跌超过1.5 倍的四分位范围 (n = 6 为每个样本, 除了 sideoats 格兰玛混合, 其中n = 5)。endosphere 五种的香农指数均低于根际和土壤。用非参数魏氏秩和试验确定物种间的意义, 并在盒顶部显示出物种间的显著差异。请单击此处查看此图的较大版本.
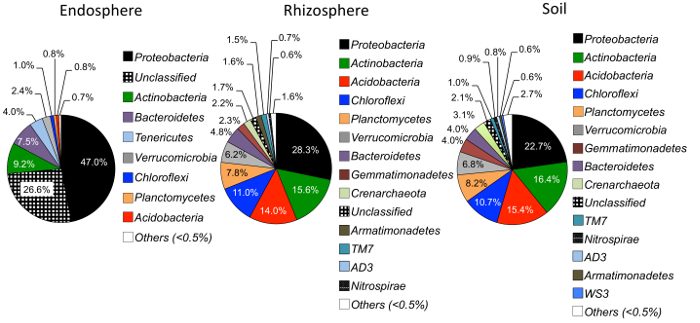
图 4: endosphere、根际和土壤中的相对丰度.对样品进行了分析, 比较了不同样品类型的微生物门的丰度 (每种样品的n = 29)。分析是使用 QIIME1.9.1 中的 Python 脚本从 OTU 表中进行的。饼图中的不同颜色表示门。该百分比表示每个样本类型中每个语系的相对丰度。使用核糖体数据库项目分类器 (RDP)25对该语系信息进行了注释。请单击此处查看此图的较大版本.
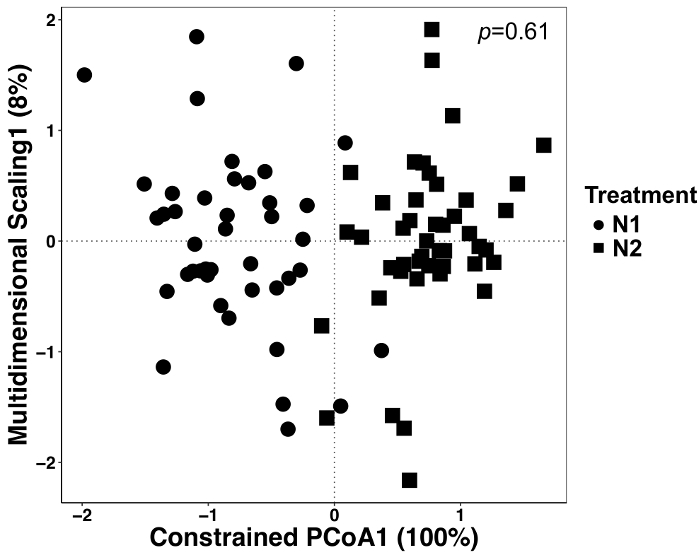
图 5: 在所有样本类型之间使用处理作为约束因子的分析.对主坐标 (CAP) 分析进行了规范化分析, 以确定治疗中微生物群落组成是否存在差异。为每 n 治疗, n = 42 为 N1 (56 公斤 n 公顷-1) 和N = 45 为 N2 (112 公斤 n ha-1)。在 QIIME1.9.1 中使用 python 脚本生成了布雷-柯蒂斯相异矩阵。以 RStudio 的因子为基础, 采用基于布雷-柯蒂斯不同矩阵的 CAP 分析方法对其进行了约束。PERMANOVA 分析, 以确定治疗差异是否显著, p值显示在右上角。请单击此处查看此图的较大版本.
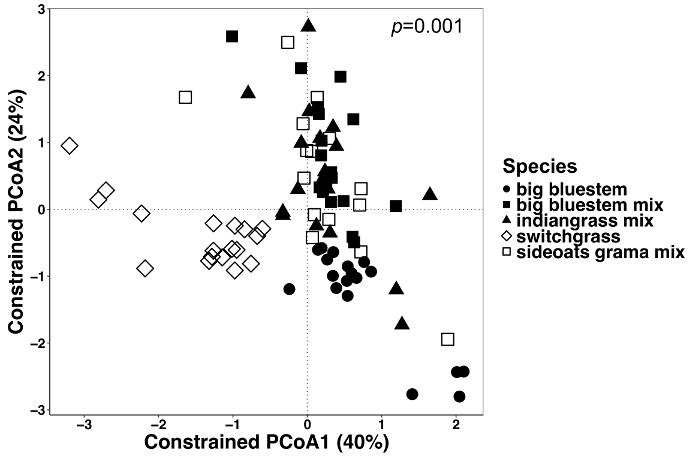
图 6: 在所有样本类型之间使用植物种类作为约束因子的分析.进行了分析, 以确定是否有不同的微生物群落组成的植物物种之间的所有样本类型。对所有样本类型 (endosphere、根际和土壤) 进行了主坐标排序和上限分析, 采用了布雷-柯蒂斯不同矩阵。在 QIIME1.9.1 中使用 Python 脚本生成了布雷-柯蒂斯相异矩阵。在 RStudio 的基础上, 通过对植物物种的约束, 建立了基于布雷-柯蒂斯相异矩阵的 CAP 分析方法。PERMANOVA 统计分析, 以确定植物物种之间的意义, P 值显示在右上角。图中的每个符号代表了该样本的整个微生物群落。n = 18 为每个种类在所有样品类型除了n = 15 为 sideoats 格兰玛混合物。请单击此处查看此图的较大版本.
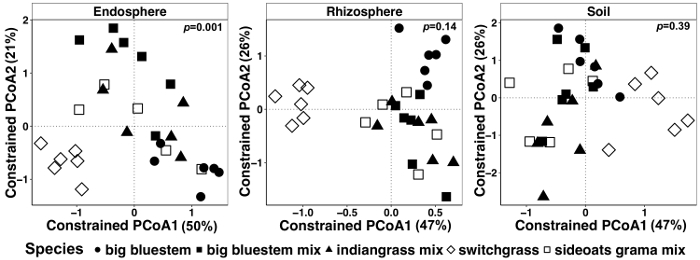
图 7: 使用物种作为每个样本类型的约束因子的 CAP 分析示例.用布雷-柯蒂斯不同矩阵对每个样本类型 (endosphere、根际和土壤) 进行主坐标排序和上限分析。每样样品在 endosphere、根际和土壤中分别为486、17154和8231。物种被用作制约协调的因素。PERMANOVA 统计分析, 以确定各样本类型植物种类之间的意义, p值显示在右上角。图中的每个符号代表每个样本的整个微生物群落。样品大小是n = 29 为每个样品类型, n = 6 为每个样品类型的植物种类除了 sideoats 格兰玛混合物 (n = 5)。请单击此处查看此图的较大版本.
讨论
本手稿中描述的方法应能使科学家容易进入土壤和植物 metagenomics 的领域。多年来, 我们已经改进了我们的方法, 因为进行了这篇手稿中描述的实验。一个改变是, 我们现在预先标签的管, 然后走出去的领域样本。我们的实验室使用条形码系统和标签打印机。标签打印机不仅节省了标签管的时间, 而且使所有的东西更容易跟踪和正确识别样本, 而不反复无常的人类手写。另一个关键点是, 我们试图处理的材料后, 尽快从外地回来。我们的目的是冻结用于 DNA 分析的土壤, 对根部进行消毒和冷冻, 并在从田间返回后12小时内过滤和冻结根际。dna 提取程序是漫长的许多步骤, 特别是对土壤和根际, 所以我们购买了机器人 (翠鸟 Flex, ThermoFisher), 最大限度地减少手的 dna 提取协议, 减少人类的错误, 可能会引入,并改善了不同批次的土壤、根或根际处理方式的一致性。在使用植物材料时, 确定要研究的根类型或采取各种根类型来获得 "代表性样本" 是很重要的。在进行 dna 提取时, 保持根和叶子处于冰冻状态是很重要的, 因为在填充96井 dna 萃取板时, 样品之间没有交叉污染。另一个需要考虑的重要因素是在设计野外试验时使用的复制次数, 并在可能的情况下使用完整的随机设计26。由于高场变异性, 可能需要大量的复制来检测小的差异。最后, 根据我们的经验, 在挖掘树根时确保土壤不太湿是必不可少的。如果土壤是饱和的水, 它不仅是混乱的工作, 但它也很难定义的根际和清除土壤从根。
在开发这些方法的早期进行的一项修改是, 而不是用手工摇动管子来释放我们升级到 vortexers 的根际, 以使该领域的工作更容易, 并在更标准化的方面每管被搅动的时间和方式。扩增子测序方法的一个局限性是, 结果的分类解析通常是有限的, 许多 OTUs 是未知的, 或者只在家庭或属的水平上知道。这一研究领域正在迅速发展, 因此必须认识到新的和发展的方法, 特别是数据分析, 这可能会提高结果的分辨率。
这些协议只用于研究细菌和古菌, 而不是真菌。使用不同的引物进行扩增将允许研究真菌群落使用相同的 DNA 样本27,28。这些方法不需要购买大量的设备, 因为这些方法可以简化。我们在这里描述的方法主要是确定 "谁在那里", 但该领域正在迅速演变为问有关功能的重要问题, 这可能是通过使用猎枪排序方法, 隔离和测试功能微生物, 或测序整个微生物基因组。
代表的结果突出了微生物群落的差异, 可以用所描述的方法来识别。在数据分析22中使用 beta-多样性方法, 在样本类型之间显示了成分差异。在 endosphere、根际和土壤中含有独特的微生物群落3的大多数其他研究中都清楚地看到了这些差异。计算了香农多样性指数, 确定了 endosphere、根际和土壤中各植物种类中微生物种类的丰度和均匀性。如本研究所示, 在许多其他方面, 阿尔法多样性在土壤中最高, 在根际略有下降, 然后在 endosphere3,5,29显著减少。这些结果表明, 本文所描述的方法适用于识别 endosphere、根际和土壤中的成分变化。
Proteobacteria的优势是 endosphere 和土壤30、31、32研究中的一个常见发现。Endosphere 的微生物种类普遍较低, Proteobacteria的相对丰度较高。这再次突显出, 这里的结果代表了文献中的其他发现。这项研究的治疗效果没有显著差异, 这两个主要原因可能是, 治疗所施加的差异不够大, 无法产生足够的变异来检测, 而且这种取样是在生长季节, 当田地可能有充足的时间把氮气绘制到类似的水平, 这是在本赛季结束时测量的。在另一项研究中, 使用相同的施肥率在较长的时间内, 只有相对较小的变化, 微生物的组成被测量33。其他研究表明, 由于氮肥34,35, 真菌和细菌群落的变化。
已知植物种类在确定其微生物3、32、36时发挥作用, 甚至在不同植物基因型之间的微生物群落变异之间存在微小差异。单一物种37。在本研究中, 植物物种间的微生物群落组成存在显著差异。在所有的样本类型中, 柳枝稷具有最明显的微生物组成, 但在 endosphere 中, 物种间的差异仅有统计学意义。如果有更多的复制可供分析, 根际群落组成可能变得很重要。
这里描述的联合野外、实验室和分析协议为研究不同因素对土壤、根际和 endosphere36的微生物群落组成的影响提供了有力的方法。在研究微生物方面, 特别是在农业领域, 还有大量工作要做。关于土壤微生物量如何改变产量的重要问题尚未得到充分阐明。甚至关于作物轮作如何影响土壤微生物的最基本问题, 时间如何改变微生物群, 非生物胁迫如何改变微生物群, 土壤类型如何与这些因素相互作用来改变微生物, 以及是否有美国某些农作物或地区的普遍微生物都是开放的问题。这些方法也将有助于流行病学研究, 以确定致病和有益细菌的存在和持续。这些方法的另一个未来地平线将是开始整合这里描述的 DNA 方法与植物和微生物 RNA 和代谢物数据。对更多的变量进行进一步的改进和测试对于进一步优化这些协议将是非常重要的。
披露声明
作者没有什么可透露的。
致谢
这篇手稿的发展得到了国家科学基金会 EPSCoR Rhizobiome 创新奖 OIA-1557417 中心的支持。数据收集得到了内布拉斯加大学-林肯分校、农业研究和发展基金以及美国农业部提供的孵化补助金的资助。我们也承认美国农业部提供的支持, 农业和食品研究倡议2011-68005-30411 号竞争性赠款来自美国农业部粮食和农业研究所, 以建立和管理这些领域。
材料
| Name | Company | Catalog Number | Comments |
| Dneasy PowerSoil HTP 96 Kit | Qiagen/MoBio | 12955-4 | Extraction kit for soil and rhizosphere |
| Dneasy PowerPlant HTP 96 Kit | Qiagen/MoBio | 13496-4 | Extraction kit for roots |
| D-Handle Digging shovel, 101 cm L | Fiskars | 9669 | |
| Rapid Tiller, 40 cm L | Truper | 34316 | |
| Ziploc Bags, 17.7 cm x 19.5 cm | Ziploc | NA | |
| Cooler | Any | NA | |
| Wash pan | Any | NA | |
| Plastic bucket | Any | NA | |
| Gloves (work and lab) | Any | NA | |
| 20 cm diameter Soil sieve #8, 2360 μm mesh size | Dual Manufacturing Co., Chicago IL | US8-8FS | |
| 20 cm diameter Soil Sieve #4, 4750 μm mesh size | Dual Manufacturing Co., Chicago IL | US8-4FS | |
| portable generator | Honda brand works well | NA | |
| Sterile cell strainers 100 μm mesh size | Fisher Scientific | 22-363-549 | |
| NaH2PO4·H2O | VWR | 0823 | |
| Na2HPO4 | VWR | 0404 | |
| Silwet L-77 | Lehman Seeds | VIS-30 | Surfactant |
| Autoclaves | Any | NA | |
| Drying Oven | Any | NA | |
| Scale | Any | Any | |
| Bleach | CLOROX - household strength | NA | |
| Tween 20 | Any | NA | |
| Liquid Nitrogen | Any | NA | |
| Dry Ice pellets | Any | NA | |
| Ethanol | Any | NA | |
| 11 cm precision fine point tweezers | Fisher | 17456209 | |
| 18 cm Straight point specimen forceps | VWR | 82027-436 | |
| 13.5 cm Pruning Scissors | Fiskars | 9921 | |
| 2 mL tube | Any | NA | |
| 15 mL PP conical tube | MIDSCI | C15B | |
| 50 mL PP conical tube | MIDSCI | C50B | |
| Ultrapure water | Millipore-sigma | Milli-Q Integral, Q-POD | |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32854 | For DNA quantification of removed samples |
| QuantiFluor dsDNA System | Promega | E2670 | For DNA quantification |
| 96-Well Black with Clear Flat Bottom Plates | Corning | 3631 | |
| pPNA PCR Blocker | PNA Bio | PP01-50 | |
| mPNA PCR Blocker | PNA Bio | MP01-50 | |
| Genomic DNA from Microbial Mock Community B (Even Low Concentration) v5.1L, for 16s rRNA Gene sequencing | BEI Resources | HM782D | |
| Adhesive 8 well-strips for plates | VWR | 89134-434 | |
| Stainless steel beads, 3.2 mm dia | Next Advance | SSB32 | |
| 1 ml Assay block (DNA extraction plate for the Qiagen/MoBio Dneasy PowerPlant HTP Kit) | CoStar | 3959 | |
| antistatic PP weighing funnel, size small for soil/rhizosphere | TWD Tradewinds, INC | ASWF1SPK | |
| antistatic PP weighing funnel, size x-small for root/leaf | TWD Tradewinds, INC | ASWFXSCS | |
| Genie 2 Digital Vortex | Scientific Industries | SI-0236 | |
| Vortex adapter for 50 mL tubes | Scientific Industries | SI-H506 | |
| Mortar (100 mL) and pestle | Any | NA | |
| Metal micro-spatula | VWR | 80071-672 | |
| Disposable antistatic microspatulas | VWR | 231-0106 | |
| Brown Paper bag 2# (10.95 cm x 6.19 cm x 20 cm) | Duro | 18402 | |
| 5424 Centrifuge for 2 mL tube | Eppendorf | 22620461 | |
| Centrifuge for 96-well plate | Sigma4-16S | 81510 | |
| Centrifuge rotor for 50 mL tubes | Sigma4-16S | 12269 - Biosafe | |
| KAPA HiFi DNA polymerase | Kapa Biosystems |
参考文献
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: the microbial ecology of the rhizosphere. Nature Reviews Microbiology. 11, 789-799 (2013).
- Fierer, N. Embracing the unknown: disentangling the complexities of the soil microbiome. Nature Review Microbiology. 15 (10), 579-590 (2017).
- Wang, P., et al. Shifts in microbial communities in soil, rhizosphere and roots of two major crop systems under elevated CO2 and O3. Scientific Reports. 7 (1), 15019 (2017).
- White, L. J., Jothibasu, K., Reese, R. N., Brözel, V. S., Subramanian, S. Spatio Temporal influence of isoflavonoids on bacterial diversity in the soybean Rhizosphere. Molecular Plant-Microbe Interactions. 28 (1), 22-29 (2015).
- Edwards, J., et al. Structure, variation, and assembly of the root-associated microbiomes of rice. Proceedings of the National Academy of Science of the United States of America. 112 (8), E911-E920 (2015).
- Lundberg, D. S., et al. Defining the core Arabidopsis thaliana root microbiome. Nature. 488, 86-90 (2012).
- Bougoure, D. S., Cairney, J. W. Assemblages of ericoid mycorrhizal and other root-associated fungi from Epacris pulchella (Ericaceae) as determined by culturing and direct DNA extraction from roots. Environmental Microbiology. 7 (6), 819-827 (2005).
- Doty, S. L., et al. Diazotrophic endophytes of native black cottonwood and willow. Symbiosis. 47 (1), 23-33 (2009).
- Gottel, N. R., et al. Distinct microbial communities within the endosphere and rhizosphere of populus deltoides roots across contrasting soil types. Applied and Environmental Microbiology. 77 (17), 5934-5944 (2011).
- Xin, G., Glawe, D., Doty, S. L. Characterization of three endophytic, indole-3-acetic acid-producing yeasts occurring in Populus trees. Mycological Research. 113, 973-980 (2009).
- Salter, S. J., et al. Reagent and laboratory contamination can critically impact sequence-based microbiome analyses. BMC Biolology. 12, 87 (2014).
- Gohl, D. M., et al. Systematic improvement of amplicon marker gene methods for increased accuracy in microbiome studies. Nature Biotechnology. 34 (9), 942-949 (2016).
- Kozich, J. J., Westcott, S. L., Baxter, N. T., Highlander, S. K., Schloss, P. D. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform. Applied Environmental Microbiology. 79 (17), 5112-5120 (2013).
- Caporaso, J. G., et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms. ISME Journal. 6 (8), 1621-1624 (2012).
- Lundberg, D. S., Yourstone, S., Mieczkowski, P., Jones, C. D., Dangl, J. L. Practical innovations for high-throughput amplicon sequencing. Nature Methods. 10 (10), 999-1002 (2013).
- Edgar, R. C. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature Methods. 10 (10), 996-998 (2013).
- Caporaso, J. G., et al. QIIME allows analysis of high-throughput community sequencing data. Nature Methods. 7 (5), 335-336 (2010).
- Shannon, C. A mathematical theory of communication. Bell System Technical Journal. 27, 379-423 (1948).
- Simpson, E. H. Measurement of diversity. Nature. 163, 688 (1949).
- Chao, A. Non-parametric estimation of the number of classes in a population. Scandanavian Journal of Statistics. 11, 265-270 (1984).
- Legendre, P. Studying beta diversity: Ecological variation partitioning by multiple regression and canonical analysis. Journal of Plant Ecology. 1 (1), 3-8 (2008).
- Oksanen, J., et al. The vegan package. Community Ecology Package. 10, 631-637 (2007).
- Wickham, H. ggplot2: Elegant graphics for data analysis. Ggplot2: Elegant Graphics for Data Analysis. , 1-212 (2009).
- Cole, J. R., et al. The Ribosomal Database Project (RDP-II): sequences and tools for high-throughput rRNA analysis. Nucleic Acids Research. 33, D294-D296 (2006).
- Wagner, M. R., et al. Host genotype and age shape the leaf and root microbiomes of a wild perennial plant. Nature Communications. 7, 12151 (2016).
- O'Brien, H. E., Parrent, J. L., Jackson, J. A., Moncalvo, J. M., Vilgalys, R. Fungal community analysis by large-scale sequencing of environmental samples. Applied and Environmental Microbiology. 71 (9), 5544-5550 (2005).
- Taylor, D. L., et al. A first comprehensive census of fungi in soil reveals both hyperdiversity and fine-scale niche partitioning. Ecological Monographs. 84 (1), 3-20 (2014).
- Lebeis, S. L., et al. Salicylic acid modulates colonization of the root microbiome by specific bacterial taxa. Science. 349 (6250), 860-864 (2015).
- Ofek-Lalzar, M., et al. Niche and host-associated functional signatures of the root surface microbiome. Nature Commununications. 5, 4950 (2014).
- Niu, B., Paulson, J. N., Zheng, X., Kolter, R. Simplified and representative bacterial community of maize roots. Proceedings of the National Academy of Science of the United States of America. 114 (12), E2450-E2459 (2017).
- Fitzpatrick, C. R., et al. Assembly and ecological function of the root microbiome across angiosperm plant species. Proceedings of the National Academy of Science of the United States of America. 115 (6), E1157-E1165 (2018).
- Ramirez, K. S., Lauber, C. L., Knight, R., Bradford, M. A., Fierer, N. Consistent effects of nitrogen fertilization on soil bacterial communities in contrasting systems. Ecology. 91 (12), 3463-3470 (2010).
- Paungfoo-Lonhienne, C., et al. Turning the table: Plants consume microbes as a source of nutrients. PLoS One. 5 (7), e11915 (2010).
- Yeoh, Y. K., et al. The core root microbiome of sugarcanes cultivated under varying nitrogen fertilizer application. Environmental Microbiology. 18 (5), 1338-1351 (2016).
- Bulgarelli, D., Schlaeppi, K., Spaepen, S., Ver Loren van Themaat, E., Schulze-Lefert, P. Structure and functions of the bacterial microbiota of plants. Annual Review of Plant Biology. 64, 807-838 (2013).
- Peiffer, J. A., et al. Diversity and heritability of the maize rhizosphere microbiome under field conditions. Proceedings of the National Academy of Science of the United States of America. 110 (16), 6548-6553 (2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。