Method Article
Isolamento e análise de comunidades microbianas na rizosfera, solo e raízes em experiências de grama perene
Neste Artigo
Resumo
Escavação de raízes de plantas do campo, bem como processamento de amostras em endosphere, rizosfera e solo são descritas em detalhes, incluindo os métodos de análise de dados e extração de DNA. Este papel é projetado para permitir que outros laboratórios de usar essas técnicas para o estudo do solo, endosphere e microbiomes da rizosfera.
Resumo
Planta e solo microbiome estudos estão se tornando cada vez mais importantes para compreender que os microorganismos papéis desempenhar na produtividade agrícola. O objetivo deste manuscrito é fornecer detalhes sobre como rapidamente rizosfera, solo e endosphere de ensaios de campo replicados da amostra e analisar as mudanças que podem ocorrer em comunidades microbianas devido ao tipo de amostra, tratamento e genótipo da planta. O experimento usou para demonstrar esses métodos consiste em parcelas de campo replicada contendo dois, puro, morno-estação gramíneas (Panicum virgatum e Andropogon gerardii) e uma mistura de grama baixa diversidade (a. gerardii, Sorghastrum nutans, e Bouteloua curtipendula). Resumidamente, as plantas são escavadas, uma variedade de raízes são cortados e colocados em tampão fosfato e em seguida agitado para coletar a rizosfera. As raízes são trazidas para o laboratório no gelo e superfície sterilized com descorante e etanol (EtOH). A rizosfera é filtrada e concentrada por centrifugação. Solo escavado de em torno da esfera de raiz é colocado em sacos de plástico e trouxe para o laboratório onde uma pequena quantidade de solo é levada para extrações de DNA. DNA é extraído da rizosfera, solo e raízes e então amplificado com os primers para a região de V4 do gene 16S rRNA. Amplicons são sequenciados e, em seguida, analisados com ferramentas de Bioinformática de acesso aberto. Estes métodos permitem aos pesquisadores testar como a diversidade da comunidade microbiana e composição varia devido ao tipo de amostra, tratamento e genótipo da planta. Usando estes métodos juntamente com modelos estatísticos, os resultados representativos demonstram que existem diferenças significativas nas comunidades microbianas de raízes, rizosfera e solo. Métodos aqui apresentados fornecem um conjunto completo de etapas sobre como coletar amostras de campo, isolar, extrair, quantificar, amplificar e sequenciar o DNA e analisam a diversidade da comunidade microbiana e composição em ensaios de campo replicadas.
Introdução
Microbiome pesquisa tem implicações importantes para a compreensão e manipulação de processos do ecossistema como nutriente ciclismo, volume de matéria orgânica e o desenvolvimento ou a inibição de patógenos de solo1,2. Esta área de investigação também possui grande potencial para a compreensão dos impactos de micróbios do solo sobre a produtividade de comunidades vegetais naturais e agroecossistemas. Embora existam muitos estudos que focaram a microbiome solo em ecossistemas naturais, menos têm incidido sobre os micróbios de rizosfera e endosphere de plantas em agroecossistemas3. Em Nebraska, agricultura domina a paisagem em grande parte do estado, que faz com que os estudos destes solos onde as culturas agronomicamente importantes são cultivadas um tema vital para a pesquisa. O objetivo deste trabalho de métodos é fornecer a pesquisadores com um conjunto padrão de protocolos para descrever os micróbios presentes nos agroecossistemas, para determinar como as raízes das plantas modificam as comunidades microbianas na rizosfera e endosphere e eventualmente compreende as funções que estes micróbios jogar em produtividade de saúde e a planta do solo.
O método aqui apresentado difere ligeiramente métodos usados por outros4,5 em que este papel destina-se a aprendizagem que micróbios são exclusivamente no interior da raiz e como eles diferem de micróbios imediatamente fora a raiz na rizosfera. A sequenciação de amplicons utilizada neste estudo identifica os microbianos táxons encontrados na amostra de DNA e permite que os investigadores determinar como as comunidades mudam dependendo do tipo de amostra ou tratamento. Uma das principais diferenças entre o presente protocolo e um protocolo muito semelhante usado por Lundberg et al . 6 é que em vez de sonication, este protocolo utiliza esterilização de superfície com água sanitária e álcool para remover a rizosfera das raízes. Outros também usaram superfície esterilização efetivamente7,8,9,10. Esses métodos não são mais vantajosos do que outros métodos, mas ligeiramente diferente. Esses métodos são particularmente adequados para experimentos de campo grande, porque é possível processar mais de 150 parcelas de campo por dia, que adiciona até aproximadamente 450 amostras quando particionado em endosphere, rizosfera e solo com bastante gente. Este manuscrito descreve detalhadamente os métodos usados no campo da amostra, processar o material no laboratório, extrair e sequenciar o DNA e fornece uma breve visão geral das etapas para analisar os dados resultantes de sequenciamento.
Protocolo
1. o campo Descrição do Site
- Descreva sites campo experimental durante os períodos de recolha. Determinar a localização do campo (latitude, longitude e altitude) usando um GPS.
- Descreva a textura do solo, tempo de amostragem e profundidade de amostragem.
- Fatores ambientais desempenham um papel importante na formação de comunidades microbianas. Gravar informações climáticas, tais como a temperatura média anual, precipitação anual, rotação de culturas dos anos anteriores, as práticas de plantio direto, método de fertilização e a história do campo local. Automatizado de estações meteorológicas ou outros dispositivos são úteis para registar a temperatura e precipitação diária durante uma estação de crescimento.
2. coleta e processamento de amostras de campo de raiz, rizosfera e solo
- Escavação de plantas.
- Rotule um pan de lavagem e um balde com uma nota auto-adesiva contendo informações sobre o material de planta a amostrar. Incluir informações como o número de lote, genótipo da planta e espécies de plantas. Levar o balde etiquetado para a trama e deixar a panela de lavagem para trás na estação de trabalho estabelecida no campo.
- Perfure o solo com uma pá a uma profundidade de 30 cm para cortar qualquer uma das raízes laterais segurando a planta no solo. O volume aproximado é de 18 cm3. Aleatoriamente escolhe e coletar duas plantas por parcela de diferentes áreas dentro da parcela.
- Escavar as raizes da planta, aproveitando a pá e colocar a esfera de raiz no balde rotulado. Traz o balde rotulado com a esfera de raiz escavado volta para a estação de trabalho no campo. Corte e elimine a biomassa da planta acima do solo.
- Remoção de solo de raízes e coleta de solo em massa.
- Agite as raízes para manualmente remover o solo ou use uma pá ou um rebento à mão para remover o solo das raízes. Agitando as raízes é suficiente para remover o solo em solos muito arenosos. Use luvas e coloque raízes perto da estação de processamento.
- Depois de cumprimentar as raízes, a maior parte do solo será na lavagem de panela. Misture o solo na lavagem panela e separar qualquer torrões de solo com um rebento à mão. Coloca uma amostra de solo que é livre de detritos em um saco de armazenamento do zipper 17,7 x 19,5 cm rotulado e lugar em um lugar fresco ou no gelo.
- Coleção de raízes e rizosfera.
- Usando uma tesoura de poda, esterilizada em 70% EtOH, impostos especiais de consumo uma variedade de raízes, aproximadamente 4 a 6 raízes por planta e cada raiz cerca de 9 a 12 cm de comprimento. Coloque as raízes extirpadas (corte conforme necessário para caber) em um tubo de 50ml rotulados contendo 35 mL de tampão de fosfato autoclavados, (6,33 g/L NaH2PO4, 8,5 g/L, Na2HPO4 anidro, pH = 6,5, 200 surfactante µ l/L).
Nota: O surfactante (ver Tabela de materiais) foi adicionado após autoclavagem o tampão de fosfato. O volume da esfera de raiz e comprimentos de raiz variam dependendo da idade da planta e espécies de plantas. - Agite os tubos por 2 min liberar a rizosfera da superfície das raízes. Com Pinças esterilizadas em 70% EtOH, remover as raízes do tubo, borrão brevemente sobre toalhas de papel e coloque em um novo, rotulado de tubo de 50 mL. Coloque ambos o tubo contendo a rizosfera e aquele que contém as raízes no gelo.
- Usando uma tesoura de poda, esterilizada em 70% EtOH, impostos especiais de consumo uma variedade de raízes, aproximadamente 4 a 6 raízes por planta e cada raiz cerca de 9 a 12 cm de comprimento. Coloque as raízes extirpadas (corte conforme necessário para caber) em um tubo de 50ml rotulados contendo 35 mL de tampão de fosfato autoclavados, (6,33 g/L NaH2PO4, 8,5 g/L, Na2HPO4 anidro, pH = 6,5, 200 surfactante µ l/L).
3. processamento de campo amostras no laboratório
- Esterilização de superfície de raízes após a coleta de campo.
Nota: Realize esterilização superfície logo que possível após o retorno do campo. Se não for possível a superfície esterilizar as raízes no mesmo dia, loja raízes a 4 ° C até processada.- Adicione aproximadamente 35 mL de água sanitária 50% + 0,01% Tween 20 para tubos de raiz 50 mL coletadas no campo. Agite os tubos de 50 mL por 30 a 60 segundos. Despeje água sanitária 50% e o Adicionar 35 mL de 70% EtOH. Agitar durante mais de 30 a 60 segundos.
- Despeje 70% EtOH e adicionar 35 mL de água estéril, ultrapura. Agitar durante 1 minuto. Repeti a lavagem de água mais duas vezes.
Nota: Para garantir o que nosso tratamento de superfície de esterilização é suficiente, nós chapeado amostras da água do último enxaguamento e não observado nenhum crescimento de bactérias (P. Wang, dados não publicados, 2014). Outros investigadores têm testado para eficiência de esterilização de raiz usando semelhantes métodos9,10. - Borre seco de raízes em toalhas de papel limpo. Use uma toalha de papel limpa para cada amostra.
Nota: Toalhas de papel podem ser esterilizadas antes da utilização. Nós não esterilizar nossas toalhas de papel. No entanto, usamos 1 toalha de papel por amostra e manter toalhas até usado. - Usar Pinças esterilizadas e tesoura de poda, corte as raízes em aproximadamente peças de 5 mm e lugar corta raízes em um tubo cónico de limpo, etiquetado 15 mL. Armazenar as amostras a-80 ° C até mais processada.
- Amostras de rizosfera de processamento.
- Agite os tubos de 50 mL contendo amostras de rizosfera de campo para Ressuspender a amostra inteira. Usando um filtro estéril, 100 µm-malha de célula (ver Tabela de materiais), filtro a ressuspensão amostra para um novo tubo de 50 mL.
- Centrifuga os tubos a 3000 x g durante 5 min à temperatura ambiente. Imediatamente despeje e descartar o sobrenadante.
- Lugar de pellets de rizosfera em tubos de 50 mL no gelo. Adicione 1,5 mL de tampão de fosfato estéril (sem surfactante) a rizosfera pelotas e vórtice para suspender.
- Pipetar o líquido suspenso em um tubo de microcentrífuga limpos, etiquetado, 2 mL. Gire os tubos a 15.871 x g por 2 min à temperatura ambiente. Imediatamente, decantar o sobrenadante e escorrer os tubos em toalhas de papel limpo.
- Armazene as pelotas a-20 º C até ulteriormente.
Nota: Etapas 3.2.2 - 3.2.4 são feitas para reduzir o tamanho do tubo de amostra para o armazenamento. É o espaço mais eficiente para armazenar os tubos de 2 mL menores em comparação com os tubos de 50 mL.
- Processamento de amostras para análise de solo e extração de DNA.
- Usando uma espátula de metal estéril, encha um tubo de 2ml limpo, etiquetado com cerca de 3 g de solo para extração de DNA. Evite qualquer raiz pequenos pedaços e detritos. Guarde esta amostra de solo a-20 ° C. Espátula de metal enxaguar em 70% EtOH entre cada amostra.
- Em uma panela de lavagem limpa, esvazie o saco do solo em peneiras empilhadas (ver Tabela de materiais), peneira maior em cima da peneira menor e manualmente, peneirar o solo através de duas peneiras. Use um pincel para limpar cuidadosamente as peneiras entre amostras.
- Reserve 100 a 125 g de solo peneirado num saco com zíper para futuro solo físico-químico e análise de textura 17,7 x 19,5 cm. Coloque os sacos do solo a 4 ° C para o armazenamento a curto prazo.
- Processamento de amostras de solo para a umidade do solo.
- Um saco de papel marrom etiquetados em uma escala de Tara. Medem de 40 a 45 g de solo peneirado dentro do saco de papel marrom. Registrar o peso do saco de papel marrom e solo em uma folha de dados e conjunto de sacos de lugar em um forno de secagem para 55-60 ° C.
- Após 72 horas, retire os sacos do forno de secagem. Permitir que os sacos de solo esfriar pelo menos 30 min e pesar.
- Para registrar o peso, tare a balança para zero e colocar os sacos de papel marrom na escala. Calcule a percentagem de umidade do solo para cada amostra, usando a fórmula:
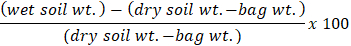
Nota: O método de umidade do solo é de Kellogg biológica estação de longo termo pesquisa ecológica (KBS LTER). KBS LTER fornece uma vasta gama de protocolos estabelecidos para os pesquisadores em seu site (https://Lter.kbs.msu.edu/).
4. preparação de raiz transformado amostras para extrações de DNA.
- Moa o material congelado raiz para que a amostra é homogénea.
- Despeje o nitrogênio líquido em um copo de plástico com espátulas limpas e em um pilão amostras congeladas durante todo o processo de moagem de manter limpo.
- Coloque o tecido congelado no almofariz e triture com o pilão em um pó fino. Continuamente adicione nitrogênio líquido durante a moagem para manter as amostras congeladas.
- Use uma espátula para colocar o tecido do chão em um tubo limpo, etiquetado 2 mL. Loja a-80 ° C.
5. extração de DNA de solo e da rizosfera amostras em formato de 96 poços
- Carregar amostras de solo em placas de 96 poços.
- Limpe a área de trabalho com 70% de EtOH e 1% de alvejante. Use luvas de laboratório durante essas etapas. Remover as amostras de solo de armazenamento-20 ° C e deixe para descongelar em um balde de gelo.
- Remova a tampa vedadora de esteira de uma placa de 96 poços de extração que é fornecida com o kit de extração de DNA. Coloque a tampa vedadora esteira entre 2 toalhetes de papel para manter limpo enquanto não estiver em uso. Para evitar a contaminação, use adesivo 8 poços PCR tiras para cobrir as 12 colunas da placa de 96 poços de extração.
- Tare um pesar estéril-funil (tamanho SM) em uma escala e pesar 200 a 250 mg de solo.
Nota: Estes funis estéril são achatados de um lado, para que o funil plana estabelece uma escala. O funil é enchido com o solo e colocado diretamente em um poço. Esta técnica evita a perda de amostras, minimiza derramamento e evita a contaminação cruzada. - Para descobrir o primeiro poço da placa de extração, levante com cuidado a fita adesiva, lugar no pescoço do pesar-funil cheio para o apropriado bem e delicadamente guiar a amostra de solo o bem apropriado. Substitua a fita adesiva para cobrir o poço.
- Repita este processo para cada poço da placa, usando um funil de novo e estéril para cada amostra, até que o prato está cheio. Deixa um vazio bem como um controle de extração em branco.
Nota: Um bem é deixado em branco em cada placa de extração para servir como um controle negativo (em branco). Este controles de contaminantes que possam estar presentes no kit de reagentes11. - Substitua a tampa vedadora esteira na placa de extração e armazenar a placa a-20 ° C até que esteja pronto para extração de DNA.
- Carregar amostras de rizosfera em placas de 96 poços.
- Remover as amostras de rizosfera de armazenamento-20 ° C e deixe para descongelar em um balde de gelo.
- Siga os passos acima para preparar o prato de extração de DNA 5.1.2.
- Coloque uma compressa de papel limpo em escala e, em seguida, tare uma espátula metálica estéril na escala. Use a espátula para colher alguns da pelota de rizosfera de um tubo de amostra cuidadosamente. Retornar a espátula para a escala e pesam entre 200 e 250 mg da amostra rizosfera.
- Levante com cuidado a fita adesiva para descobrir o primeiro poço da placa de extração, a espátula cheia do ângulo dentro do poço e raspar o material da rizosfera no apropriado bem com um palito de dente estéril.
- Lave a espátula de metal na água, seguida por 70% EtOH entre amostras. Repita este processo para cada poço da placa até que a placa está cheia, deixando um vazio bem como um controle de extração em branco.
- Extração de DNA de amostras de rizosfera e solo em formato de 96 poços.
- Extrato de solo e rizosfera DNA usando um kit otimizado para o solo (ver Tabela de materiais), seguindo o protocolo do fabricante.
Nota: Nós usamos este kit específico para isolar o solo e da rizosfera DNA devido à capacidade dos reagentes proprietárias para remover o ácido húmico e outros poderosos inibidores PCR encontrados no solo.
- Extrato de solo e rizosfera DNA usando um kit otimizado para o solo (ver Tabela de materiais), seguindo o protocolo do fabricante.
- Quantificação de DNA.
- Quantificar 92 amostras e 4 concentrações de padrões usando um kit (ver Tabela de materiais; padrões estão incluídos), de acordo com o protocolo do fabricante.
- Quantificar as restantes 4 amostras que foram removidas da placa para acomodar poços para os quatro padrões usando um kit (veja a Tabela de materiais), de acordo com o protocolo do fabricante.
6. extração de DNA de raiz amostras em formato de 96 poços.
- Carregar amostras de raiz em placas de 96 poços.
- Limpe a área de trabalho com 70% de EtOH e 1% de alvejante. Use luvas durante essas etapas.
- Manter amostras de raiz do solo congeladas em todos os momentos, colocando as amostras em um balde com gelo seco.
- Encha um copo de plástico com nitrogênio líquido e coloque microspatulas antiestático e pesar-funis estéril (tamanho XSM) no copo de plástico para refrigerar para baixo.
- Remova a tampa vedadora esteira da placa de grânulo de 96 poços de extração que é fornecida com o kit e coloque-a entre 2 toalhetes de papel para manter limpo enquanto não estiver em uso.
Nota: A versão mais recente deste kit, lançado logo após o tempo que realizamos estas extrações de DNA, exige que o usuário fornecer as placas de grânulo de extração. Na Tabela de materiais, listamos as informações de catálogo de fornecedores necessárias para pedir esses itens. - Para evitar a contaminação, use adesivo 8 poços PCR tiras para cobrir as 12 colunas da placa de 96 poços de extração.
- Coloque a placa do grânulo de extração em gelo seco para manter as amostras em poços congelados.
- Levante cuidadosamente a fita adesiva para descobrir o primeiro poço da placa de extração, coloque no pescoço do pesar-funil cheio no bem apropriado e adicionar 3 colheres de espátula de tecido de raiz do chão. Substitua a fita adesiva para cobrir o poço.
Nota: O solo e da rizosfera são descongelados no gelo antes de pesar, Considerando que o material vegetal é pesado para fora congelado. Tecidos de vegetais congelados, especialmente em pequenas quantidades é difícil pesar em uma escala sem descongelar. Pesa testes foram feitos em tecido de raiz do solo para determinar quantas colheres de espátula eram suficientes. Observe que o fabricante do kit não requer uma quantidade exata de tecido, mas recomenda-se ao redor de 50 mg. tipos de amostra de diferentes plantas irão variar e o usuário precisará determinar a quantidade adequada. - Repita este processo para cada poço da placa até que o prato está cheio.
- Armazenar a placa a-20 ° C até que esteja pronto para extração de DNA.
- Extração de DNA de tecido de raiz.
- Extrair DNA usando um kit otimizado para plantas (ver Tabela de materiais) seguindo o protocolo do fabricante.
Nota: Nós usamos este kit que é projetado especificamente para o tecido de planta alcançar o máximo rendimento de amostras de raiz. Ao contrário do solo e da rizosfera, o ácido húmico e outros contaminantes estão a menos de um problema para o tecido de raiz.
- Extrair DNA usando um kit otimizado para plantas (ver Tabela de materiais) seguindo o protocolo do fabricante.
- Quantificar o DNA como na etapa 5.4.
7. amplificação e sequenciamento do DNA isolado.
- Amplificar a região V4 do gene 16S com uma prova de leitura do polymerase (ver Tabela de materiais), conforme descrito em Göhl et al 12 amostras de código de barras com diferentes iniciadores de indexação e piscina antes de sequenciamento. Sequência usando os métodos descritos por Göhl et al12 usando um processo PCR de dois passos com primers V4 (1 de arquivo suplementar).
Nota: Alguns dos métodos para essas etapas são descritas em detalham em outro lugar12,13,14 e, portanto, não vão ser descritos aqui. Para as amostras de raiz, bloqueadores PNA foram adicionados para reduzir a quantidade de plastid DNA amplificado a partir do tecido de planta, que já havia sido totalmente descrito15. Dois controles foram usados no sequenciamento, um controlo negativo, que incluía as extração placa em branco controles (ver nota após etapa 5.1.5), e uma comunidade de simulação de uma população conhecida de DNA bacteriano (ver Tabela de materiais), que serviu como um positivo controle.
Nota: Na maioria dos casos, o v3 MiSeq Kits de reagentes são utilizados no modo de base final emparelhados de 2 X 300. Para as amostras neste manuscrito, utilizou-se a Illumina HiSeq 2500 em modo rápido com 250 modo de cobertura-final (2 x 250). Toda a amostra foram sequenciados na mesma pista. - Processe os dados de sequenciamento através de um gasoduto para análises de comunidade microbiana (USEARCH v9.2.64, QIIME v1.9.1 e RStudio v3.4.3 16).
- Prepare os dados de sequenciamento usando USEARCH17.
Nota: USEARCH está disponível on-line com instruções completas (https://www.drive5.com/usearch/). - Demultiplex de dados de sequenciamento usando índice leituras ou códigos de barras para atribuir Illumina lê para amostras.
- Mescle o lê emparelhado-final para obter as sequências de consenso. Use o comando: usearch-fastq_mergepairs * R1*.fastq-remarcagem @ - fastq_maxdiffs 10 - fastq_minmergelen 230 - fastq_maxmergelen 320 - fastq_pctid 80 - fastqout merged.fq.
Nota: Os parâmetros são definidos através de referenciar o manual de instruções USEARCH. - Remova os primers a partir dos dados de sequenciamento para evitar substituições nas sequências de cartilha, que podem ser causadas pela reação de PCR. Use o comando: usearch-fastx_truncate merge.fq - stripleft 19 - stripright 20 - fastqout stripped.fq.
- Filtre dados de sequenciamento para remover as leituras de baixa qualidade e manter sequências de unidade taxonômica operacional (OTU) de alta qualidade. Use o comando: usearch-fastq_filter stripped.fq-fastq_maxee 1.0 - fastaout filtered.fa.
- Prepare os dados de sequenciamento usando USEARCH17.
- Gere OTUs em USEARCH.
- Execute a desreplicação para identificar o conjunto de sequências OTU exclusivos. Use o comando: usearch-fastx_uniques filtered.fa - fastaout uniques.fa - sizeout-reclassificar Uniq.
- OTUs de cluster com similaridade da sequência 97 – 100% para designar os OTUs exclusivos. Use o comando: usearch-cluster_otus uniques.fa - minsize 2 - otus otus.fa-reclassificar Otu.
Nota: Este passo incorpora também a remoção de singletons dos OTUs clusterizados e remoção de quimeras de dados de sequenciamento. - Crie uma tabela OTU em USEARCH. Use o comando: usearch-usearch_global stripped.fq - db otus.fa-plus - identificação 0.97 - otutabout otutable.txt da costa.
Nota: Este comando gera uma tabela com o número de leituras (contagem) de OTUs todas para cada amostra. A tabela OTU é usada por etapas a jusante, incluindo análises de abundância diferencial e análises de diversidade microbiana. Exemplo de tabela OTU é mostrado na figura suplementar 2. - Realizar uma análise de rarefação em QIIME v1.9.1 18.
Nota: A curva de rarefação é calculada usando a tabela OTU para determinar se a profundidade do sequenciamento amostras adequadamente a comunidade microbiana. Exemplo de curvas de rarefação são mostrados na Figura suplementar 3. - Realizar uma análise de diversidade alfa18. Use alpha_rarefaction.py em QIIME v1.9.1 para calcular a diversidade da comunidade microbiana dentro de cada amostra.
Nota: Esta análise calcula os índices de diversidade como Shannon19, Simpson20e Chao121. - Realizar um beta diversidade análise18,22. Use o script Python: beta_diversity_through_plots.py na matriz de dissimilaridade de Bray-Curtis de v1.9.1 QIIME.
Nota: Esta análise compara a composição da comunidade microbiana entre amostras. - Realizar análises estatísticas entre grupos. Use matrizes de distância calculadas para PERMONOVA usando a função adonis e anova no vegan pacote23 v2.4.5 em RStudio16. Realizar análise canônica da análise de coordenadas principal (CAP), usando a função capscale no pacote vegan. Visualize dados usando ggplot2 pacote24 v 2.2.1 em RStudio.
Resultados
Os resultados representativos apresentados neste manuscrito vêm de um site de campo estabelecido em 2012, na Universidade de Nebraska-Lincoln agricultura pesquisa divisão fazenda perto de Mead, NE. antes do experimento, o site tinha sido gerenciados como uma rotação milho-soja . O local de estudo foi localizado em três diferentes tipos de solos, mas os dados foram analisados, como se todas as alterações nas propriedades do solo medida deveram-se a tratamentos impostos.
O site de campo continha dois, puro, carrinhos de "switchgrass" (p. virgatum cv Liberty) e bluestem grande (a. gerardii), bem como uma mistura de grama baixa diversidade contendo bluestem grande, indiangrass (S. nutans) e 'Butte' sideoats grama ( B. curtipendula). As três parcelas de grama morno-estação foram em um design de bloco completo estudo randomizado que era replicado três vezes. Aninhado em três parcelas diferentes de grama foram dois tratamentos de fertilização de nitrogênio (N), que foram 56 (N1) e 112 (N2) kg N ha-1 de ureia aplicada. No momento da amostragem microbiome no final da estação de crescimento, o nitrato de ppm (média ± DP) de 8,0 ± 1,1 solo contido nas tramas fertilizados com 112 kg N ha-1 e 6,8 ± 0,7 (média + SD) nitrato ppm nas tramas fertilizados com 56 kg N ha-1. As parcelas tinham sido fertilizadas uma vez por ano. A grama de morno-estação parcelas foram designadas como as parcelas principais (8000 m2) e N tratamentos foram as parcelas de separação (4000 m2). Bluestem grande foi semeada como uma mistura de 50: 50 de 'Bonanza' e 'Mina de ouro' e Indiangrass foi semeada como uma mistura de 50: 50 de 'Scout' e 'Guerreiro'. As parcelas foram plantadas em 2012, e a primeira aplicação de N ocorreu na primavera de 2013.
O solo e a amostragem de raiz foi realizado em 15 de setembro de 2014. O trabalho descrito abaixo foi realizado em um campo que foi criado como um divisão de pontos casualizado com três repetições (Figura 1). A profundidade média de sequenciamento de todas as amostras para endosphere foram os seguintes: 4871 ± 5711 (média ± DP), rizosfera: 40726 ± 14684, solo: 38184 ± 9043. Uma das maiores fontes de variação nesses experimentos, usando os métodos descritos, é a diferença em comunidades microbianas encontradas entre tipos de amostra (Figura 2). Neste conjunto de dados representativos, a rizosfera e solo parecem ser mais similar em composição ao outro do que o endosphere (Figura 2A). No entanto, havia também altamente significativa (p = 0,001) diferenças na composição da comunidade microbiana entre rizosfera e solo (Figura 2B). A variação total contabilizada nesses experimentos analisados por tipo de amostra foi de 26%.
Diversidade alfa análise mostrou que as comunidades microbianas no endosphere foram menores na diversidade da amostra em relação ao solo e da rizosfera (Figura 3). As diferenças significativas apenas na diversidade entre as espécies de grama em qualquer compartimento estavam entre as amostras de endosphere do bluestem grande e switchgrass, com switchgrass tendo significativamente maior diversidade de espécies microbianas (Figura 3). A análise de abundância relativa (Figura 4) destaca o domínio de Proteobacteria seguido de Actinobacteria em todos os tipos de amostra. Solo e da rizosfera também são dominadas por Acidobacteria e Chloroflexi Considerando que o endosphere tinha uma maior abundância relativa de Bacteriodetes.
Neste experimento, as plantas foram cultivadas com duas diferentes quantidades de fertilizante N e, portanto, foram analisados os dados para determinar se houve efeitos do tratamento. Efeitos do tratamento foram responsáveis por 12% da variação total, mas não foram significativamente diferentes, embora a ordenação dos dois tratamentos olhem diferente (Figura 5). Isto ressalta a importância de análises estatísticas para estes conjuntos de dados ao invés de inspeção visual ou julgamentos qualitativos.
Planta-influenciado diferenças na microbiome de tecidos vegetais e solo foram visualizadas usando um método restrito de ordenação. Diferenças estatísticas foram determinadas utilizando uma análise de TÁXONS para testar se as variáveis específicas, tais como espécie, resultam na composição da comunidade microbiana significativamente diferente entre as amostras. Quando todos os tipos de amostra foram analisados em conjunto, uma diferença altamente significativa foi encontrada na composição da comunidade microbiana devido a espécies de plantas (Figura 6). Neste experimento, a quantidade de variação explicada por espécies vegetais foi de 6,7%. Finalmente, cada tipo de amostra foi analisado individualmente para determinar qual dos tipos de amostra pode estar dirigindo o efeito de espécies de plantas significativas. Só no endosphere estava lá uma diferença altamente significativa (p = 0,001) entre as composições de comunidade microbiana de diferentes espécies de plantas (Figura 7). Nos outros tipos de amostra, o efeito das espécies não foi significativo quando analisados individualmente. Na endosphere, a variação percentual devido a espécie era 27%, Considerando que foi menor na rizosfera (18%) e no solo (15%). Isto ainda mais destaca a importância de analisar cada tipo de tecido individualmente.
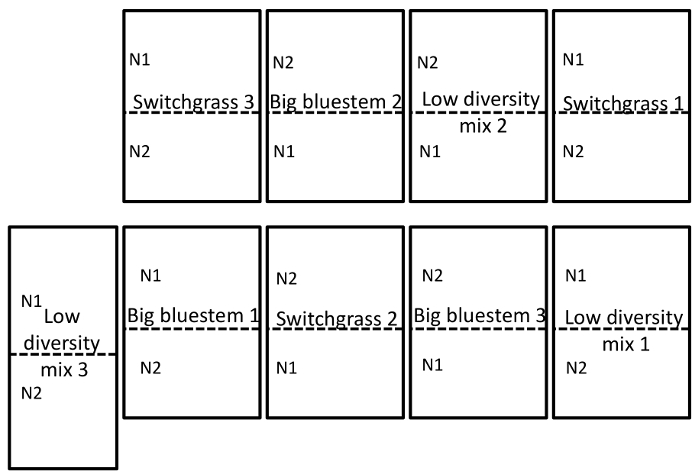
Figura 1: exemplo do campo experimental design. Projeto de campo experimental, ilustrando um design de bloco completo estudo randomizado em triplicado do campo site localizado na Universidade de Nebraska-Lincoln Oriental Nebraska centro de pesquisa e extensão perto Mead, NE. Para Descrição completa do site, consulte a seção de resultados. N1 é a baixa (56 kg N ha-1 ureia) e N2 (112 kg N ha-1 ureia) é a taxa mais elevada de nitrogênio que foi aplicada. Clique aqui para ver uma versão maior desta figura.
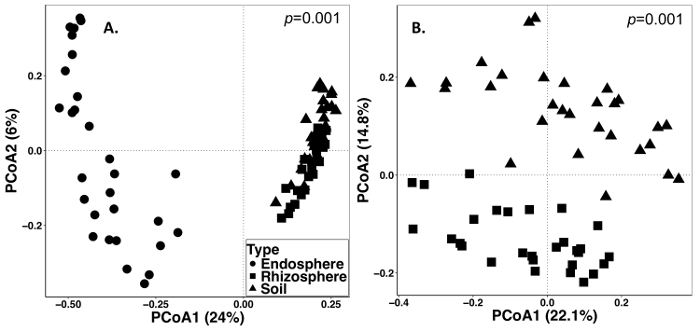
Figura 2: análise de diversidade Beta comparando a composição microbiana em tipos diferentes de amostra incluindo endosphere, rizosfera e solo da amostragem de grama perene em 2014. A análise foi realizada usando um script Python em QIIME1.9.1 para produzir a matriz de dissimilaridade Bray-Curtis. Análise de coordenadas principais (PCoA) com base na matriz de dissimilaridade Bray-Curtis foi visualizado em RStudio. PCoA1 e PCoA2 indicam a primeira e segunda maior variância explicada pela análise PCoA. TÁXONS análise estatística foi realizada para determinar o significado entre tipos de amostra, e o valor de p é mostrado no canto superior direito. Cada símbolo nas figuras representam toda a comunidade microbiana para cada amostra. (A) tipos de amostra Endosphere, rizosfera e solo foram analisados em conjunto. Todos os 87 amostras foram escasseadas de 486 sequências por amostra. (B) amostras de rizosfera e solo foram analisadas em conjunto. Todas as 59 amostras foram escasseadas com 8231 sequências. Clique aqui para ver uma versão maior desta figura.
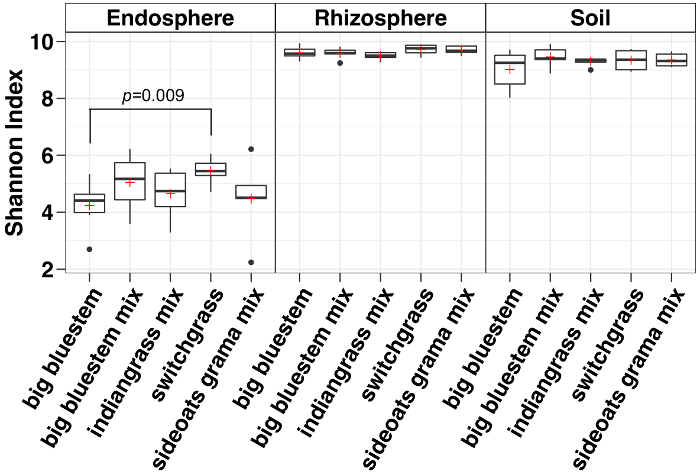
Figura 3: análise de diversidade alfa usando o índice de Shannon para cada espécie na rizosfera, solo e endosphere. A análise foi realizada utilizando um script Python no QIIME1.9.1. Rarefação foi feita para o endosphere, rizosfera e tipos de amostra de solo respectivamente com 486, 17154 e 8231 sequências por amostra. Caixas indicam os percentis 25 e 75 (primeiros e terceiros quartis). A linha horizontal dentro da caixa denota a mediana e o vermelho, além disso, mostra a média. Bigodes mostram o intervalo de dados excluindo valores atípicos (que são mostrados como pontos pretos) que caiu mais de 1,5 vezes o intervalo interquartil (n = 6 para cada amostra, exceto para sideoats grama combinação onde n = 5). O índice de Shannon de todas as cinco espécies no endosphere eram inferiores a rizosfera e o solo. Não-paramétrico Wilcoxon teste de classificação soma foi utilizada para determinar a significância entre as espécies e somente as diferenças significativas entre as espécies foram mostradas em cima das caixas. Clique aqui para ver uma versão maior desta figura.
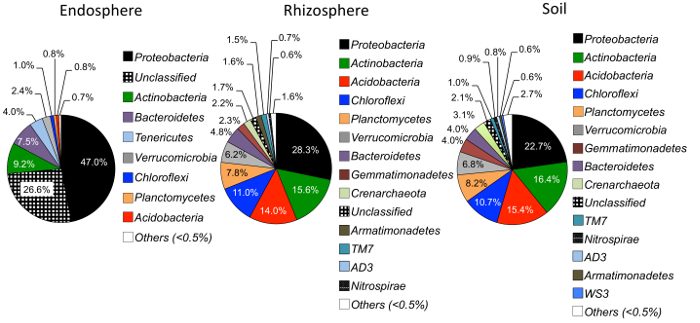
Figura 4: abundância relativa ao nível de filo no endosphere, rizosfera, solo e. As amostras foram analisadas para comparar a abundância dos filos microbianas entre tipos diferentes de amostras (n = 29 para cada tipo de amostra). A análise foi realizada utilizando um script Python no QIIME1.9.1 da tabela OTU. As cores diferentes dentro do gráfico de pizza denotam os filos. A porcentagem indica a abundância relativa de cada filo em cada tipo de amostra. As informações de filo foi anotadas usando o projeto de banco de dados Ribosomal classificador (RDP)25. Clique aqui para ver uma versão maior desta figura.
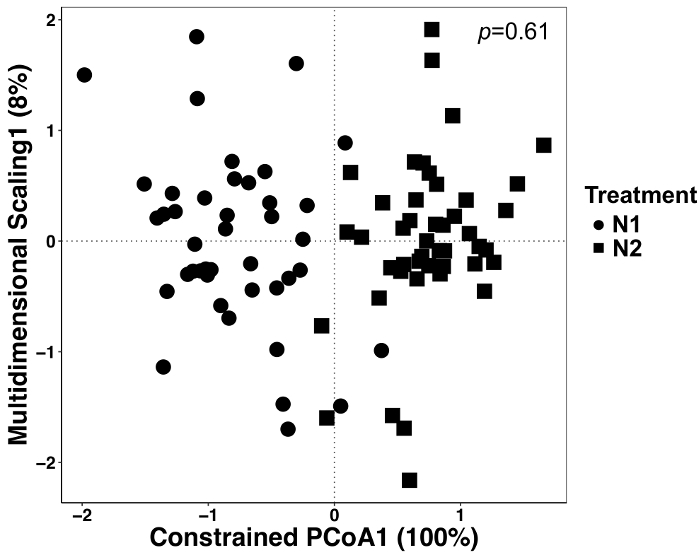
Figura 5: análise usando o tratamento como fator de restrição entre todos os tipos de amostra. Realizou-se análise canônica da análise de coordenadas principal (CAP) para determinar se houve diferenças na composição da comunidade microbiana entre os tratamentos. Para cada tratamento de N, n = 42 para N1 (56 kg N ha-1) e n = 45 para N2 (112 kg N ha-1). A matriz de dissimilaridade Bray-Curtis foi gerada usando um script python no QIIME1.9.1. Análise de CAP com base na matriz de dissimilaridade Bray-Curtis foi feito por restringir o tratamento como o fator em RStudio. Análise de TÁXONS foi realizada para determinar se as diferenças de tratamento foram significativas, e o valor de p é mostrado no canto superior direito. Clique aqui para ver uma versão maior desta figura.
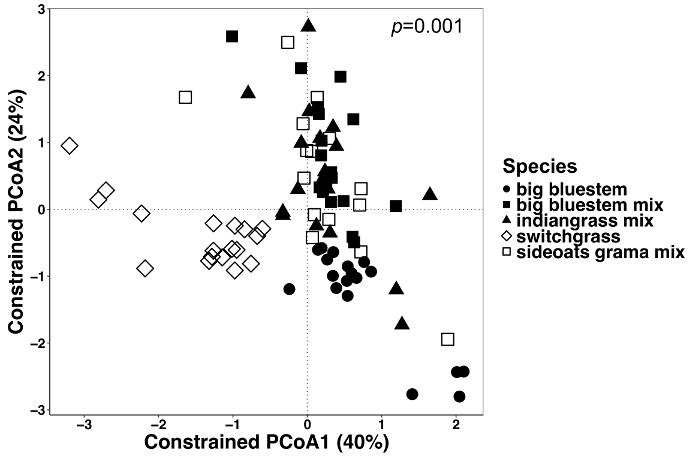
Figura 6: análise utilizando espécies vegetais como fator de restrição entre todos os tipos de amostra. Análise foi realizada para determinar se houve diferenças na composição microbiana Comunidade entre espécies de plantas em todos os tipos de amostra. Ordenação de coordenadas principal e CAP análise de todos os tipos de amostra (endosphere, rizosfera e solo) foram feitos usando uma matriz de dissimilaridade Bray-Curtis. A matriz de dissimilaridade Bray-Curtis foi gerada usando o script Python no QIIME1.9.1. Análise de CAP com base na matriz de dissimilaridade Bray-Curtis foi feito por restringir as espécies de plantas como o fator em RStudio. TÁXONS análise estatística foi realizada para determinar o significado entre espécies de plantas, e o valor de P é mostrado no canto superior direito. Cada símbolo nas figuras representa toda a comunidade microbiana para aquela amostra. n = 18 para cada espécie de todos os tipos de amostra, exceto n = 15 para a mistura de grama de sideoats. Clique aqui para ver uma versão maior desta figura.
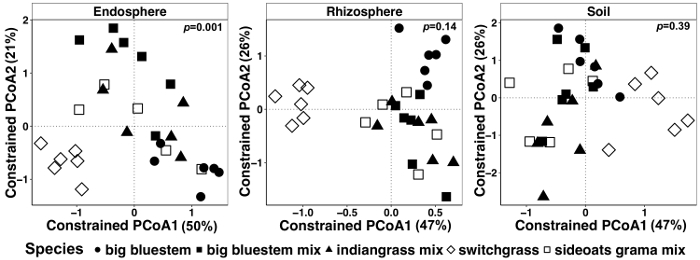
Figura 7: exemplo de análise de CAP usando espécies como fator de restrição para cada tipo de amostra individualmente. Ordenação de coordenadas principal e análise de CAP de cada tipo de amostra (endosphere, rizosfera e solo) usando a matriz de dissimilaridade Bray-Curtis. Cada tipo de amostra foi escasseado 486, 17154 e 8231 leituras por amostra, respectivamente, em endosphere, rizosfera e solo. Espécie foi usado como o fator para restringir a ordenação. TÁXONS análise estatística foi realizada para determinar o significado entre espécies de plantas em cada tipo de amostra, e o valor de p é mostrado no canto superior direito. Cada símbolo na figura representa toda a comunidade microbiana para cada amostra. Tamanho da amostra é n = 29 para cada tipo de amostra, n = 6 para cada espécie de planta em cada tipo de amostra, exceto para a mistura de grama de sideoats (n = 5). Clique aqui para ver uma versão maior desta figura.
Discussão
Os métodos descritos neste manuscrito deverão permitir aos cientistas para facilmente entrar no campo de metagenômica de solo e planta. Ao longo dos anos, nós refinaram nossos métodos desde a realização do experimento descrito neste manuscrito. Uma mudança é que agora pre-rotulamos os tubos antes de sair para o campo de amostra. Nosso laboratório utiliza um sistema de código de barras e uma impressora de etiqueta. A impressora de etiqueta não só economiza tempo quando rotulagem tubos, mas também torna tudo mais fácil para controlar e identificar corretamente as amostras sem os caprichos da escrita de mão humana. Outro ponto crítico é que tentar processar o material depois de trazer de volta do campo logo que possível. Nosso objetivo é congelar o solo utilizado para análise de DNA, esterilizar e congelar as raízes e filtrar e congelar a rizosfera dentro de 12 a 36 horas depois de voltar do campo. Os procedimentos de extração de DNA são longos com muitos degraus, particularmente para o solo e da rizosfera, então nós compramos um robô (Kingfisher Flex, ThermoFisher) que minimiza as mãos na hora dos protocolos de extração de DNA, reduz o erro humano que pode ser introduzido, e melhora a consistência na maneira diferentes lotes de solo, raízes ou rizosfera são processados. Ao trabalhar com material vegetal é importante decidir sobre o tipo de raiz para ser estudado ou tomar uma variedade de tipos de raiz de uma "amostra representativa". Manutenção de raízes e folhas no estado congelado quando conduzir as extrações de DNA é importante, porque está assegurando que não haja nenhuma contaminação cruzada entre amostras ao encher-se placas de extração de DNA de 96 poços. Outro fator importante a considerar é o número de repetições deve ser usado quando projetar experimentos de campo e usando um completo randomizados projeto sempre que possível26. Devido à variabilidade de alto campo pode ser necessário ter um grande número de repetições para detectar pequenas diferenças. Finalmente, nossa experiência é essencial para certificar-se que os solos não são demasiado molhados quando escavando as raízes. Se os solos estão saturados de água não só é confuso para trabalhar com, mas também é muito difícil definir a rizosfera e remover o solo das raízes.
Uma modificação que foi feita logo no início durante o desenvolvimento desses métodos foi em vez de tremer os tubos à mão para liberar a rizosfera que nós atualizado para vortexers alimentado por um gerador a gás para facilitar o trabalho no campo e mais padronizada em termos de o tempo e a maneira que cada tubo foi agitado. Uma limitação da abordagem de sequenciamento de amplicon é que a resolução taxonômica dos resultados é muitas vezes limitada e OTUs muitos são conhecidos desconhecidos ou apenas no nível de família ou género. Neste campo de pesquisa está evoluindo rapidamente, por isso é importante estar ciente de abordagens novas e em desenvolvimento, particularmente para análise de dados, que podem melhorar a resolução dos resultados.
Esses protocolos são somente para estudar as bactérias e archaea, não de fungos. O uso de diferentes primers para amplificação permitirá o estudo das comunidades fúngicas, usando o mesmo DNA amostras27,28. Esses métodos não exigem a compra de grandes quantidades de equipamento porque os métodos podem ser simplificados. Os métodos que descrevemos aqui são principalmente para determinar "quem é", mas o campo está a evoluir rapidamente para perguntas importantes sobre a função, que pode ser resolvida usando métodos de sequenciamento de espingarda, isolamento e testar a funcionalidade do micróbios, ou toda microbiana genomas de sequenciamento.
Os resultados representativos destacam as diferenças em comunidades microbianas que podem ser identificadas usando os métodos descritos. Usando uma abordagem de beta-diversidade para a análise de dados22, diferenças de composição foram mostradas entre tipos de amostra. Essas diferença foram claramente observados na maioria dos outros estudos onde endosphere, rizosfera e solo contêm comunidades microbianas original3. O índice de diversidade de Shannon foi calculado para determinar a abundância e a uniformidade das espécies microbianas presentes dentro de cada espécie de planta no endosphere, rizosfera e solo. Como mostrado neste estudo e em muitos outros, diversidade alfa é mais alta no solo, diminuindo ligeiramente na rizosfera e em seguida diminuir significativamente no endosphere3,5,29. Estes resultados indicam que os métodos descritos aqui são adequados para identificar mudanças composicionais no endosphere, rizosfera e solo.
O domínio das proteobactérias é um achado comum em estudos de31,30,endosphere e solo32. Endosphere geralmente tem uma baixa diversidade de espécies microbianas com uma maior abundância relativa as proteobactérias. Este novo destaca que os resultados aqui são representativas de outros achados na literatura. Os efeitos de tratamento neste estudo não foram significativamente diferentes e dois motivos principais para isso podem ser que as diferenças impostas pelos tratamentos não eram grandes o suficiente para gerar variação suficiente para detectar e que esta amostragem foi feita no final da época, quando os campos podem ter tido tempo suficiente para sacar o nitrogênio para níveis semelhantes, que é o que foi medido no final da temporada de crescimento. Em outro estudo utilizando taxas semelhantes de fertilização por um longo período de tempo, apenas relativamente pequenas mudanças na composição da microbiome foram medidos33. Outros estudos mostraram alterações nas comunidades fúngicas e bacterianas devido ao nitrogênio fertilizante34,35.
Espécies de plantas são conhecidos por desempenhar papéis na determinação de suas microbiomes3,32,36 e até mesmo pequenas diferenças na variação de comunidade microbiana tem sido demonstradas entre genótipos de plantas diferentes dentro de um única espécie37. Neste estudo, verificou-se uma diferença significativa na composição da comunidade microbiana entre espécies de plantas. Em todos os tipos de amostra pareceu que switchgrass tinha a composição microbiana mais distinta, mas as diferenças entre as espécies apenas foram estatisticamente significativas no endosphere. Composição da comunidade de rizosfera pode ter se tornado significativa se mais repetições estavam disponíveis para análise.
O campo combinado, laboratório e protocolos analíticos, descritos aqui fornecem um método poderoso para estudar a influência de fatores diferentes como a composição de comunidades microbianas em solos, rizosfera e a endosphere de raízes36. Há uma grande quantidade de trabalho a ser feito na área de estudo microbiomes, particularmente em campos agrícolas. Questões importantes sobre como os rendimentos são alterados por microbiome o solo ainda tem que ser totalmente elucidado. Mesmo as perguntas mais básicas sobre como as rotações de culturas influenciam a microbiome solo, como o sincronismo altera a microbiome, estresse abiótico como altera a microbiome, como tipo de solo interage com estes fatores para alterar a microbiome e se existem micróbios universais em certas culturas ou regiões dos EUA são todas questões abertas. Esses métodos também será útil para estudos epidemiológicos identificar a presença e persistência de bactérias patogénicas e benéficas. Outro horizonte de futuro para esses métodos será para começar a integrar os métodos de DNA descritos aqui com planta e micróbio RNA e dados do metabólito. Melhoria adicional e teste de variáveis mais será importantes para otimização mais desses protocolos.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
O desenvolvimento deste manuscrito é suportado pelo National Science Foundation EPSCoR Center para a raiz e o prêmio de inovação Rhizobiome OIA-1557417. A coleta de dados foi apoiada por fundos da Universidade de Nebraska-Lincoln, pesquisa agrícola e desenvolvimento e por uma concessão de hachura do USDA. Também reconhecemos o apoio do USDA-ARS e apoio foi prestado pela agricultura e comida pesquisa iniciativa competitiva Grant no. 2011-68005-30411 do USDA, Instituto Nacional de alimentos e agricultura para estabelecer e gerenciar esses campos.
Materiais
| Name | Company | Catalog Number | Comments |
| Dneasy PowerSoil HTP 96 Kit | Qiagen/MoBio | 12955-4 | Extraction kit for soil and rhizosphere |
| Dneasy PowerPlant HTP 96 Kit | Qiagen/MoBio | 13496-4 | Extraction kit for roots |
| D-Handle Digging shovel, 101 cm L | Fiskars | 9669 | |
| Rapid Tiller, 40 cm L | Truper | 34316 | |
| Ziploc Bags, 17.7 cm x 19.5 cm | Ziploc | NA | |
| Cooler | Any | NA | |
| Wash pan | Any | NA | |
| Plastic bucket | Any | NA | |
| Gloves (work and lab) | Any | NA | |
| 20 cm diameter Soil sieve #8, 2360 μm mesh size | Dual Manufacturing Co., Chicago IL | US8-8FS | |
| 20 cm diameter Soil Sieve #4, 4750 μm mesh size | Dual Manufacturing Co., Chicago IL | US8-4FS | |
| portable generator | Honda brand works well | NA | |
| Sterile cell strainers 100 μm mesh size | Fisher Scientific | 22-363-549 | |
| NaH2PO4·H2O | VWR | 0823 | |
| Na2HPO4 | VWR | 0404 | |
| Silwet L-77 | Lehman Seeds | VIS-30 | Surfactant |
| Autoclaves | Any | NA | |
| Drying Oven | Any | NA | |
| Scale | Any | Any | |
| Bleach | CLOROX - household strength | NA | |
| Tween 20 | Any | NA | |
| Liquid Nitrogen | Any | NA | |
| Dry Ice pellets | Any | NA | |
| Ethanol | Any | NA | |
| 11 cm precision fine point tweezers | Fisher | 17456209 | |
| 18 cm Straight point specimen forceps | VWR | 82027-436 | |
| 13.5 cm Pruning Scissors | Fiskars | 9921 | |
| 2 mL tube | Any | NA | |
| 15 mL PP conical tube | MIDSCI | C15B | |
| 50 mL PP conical tube | MIDSCI | C50B | |
| Ultrapure water | Millipore-sigma | Milli-Q Integral, Q-POD | |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32854 | For DNA quantification of removed samples |
| QuantiFluor dsDNA System | Promega | E2670 | For DNA quantification |
| 96-Well Black with Clear Flat Bottom Plates | Corning | 3631 | |
| pPNA PCR Blocker | PNA Bio | PP01-50 | |
| mPNA PCR Blocker | PNA Bio | MP01-50 | |
| Genomic DNA from Microbial Mock Community B (Even Low Concentration) v5.1L, for 16s rRNA Gene sequencing | BEI Resources | HM782D | |
| Adhesive 8 well-strips for plates | VWR | 89134-434 | |
| Stainless steel beads, 3.2 mm dia | Next Advance | SSB32 | |
| 1 ml Assay block (DNA extraction plate for the Qiagen/MoBio Dneasy PowerPlant HTP Kit) | CoStar | 3959 | |
| antistatic PP weighing funnel, size small for soil/rhizosphere | TWD Tradewinds, INC | ASWF1SPK | |
| antistatic PP weighing funnel, size x-small for root/leaf | TWD Tradewinds, INC | ASWFXSCS | |
| Genie 2 Digital Vortex | Scientific Industries | SI-0236 | |
| Vortex adapter for 50 mL tubes | Scientific Industries | SI-H506 | |
| Mortar (100 mL) and pestle | Any | NA | |
| Metal micro-spatula | VWR | 80071-672 | |
| Disposable antistatic microspatulas | VWR | 231-0106 | |
| Brown Paper bag 2# (10.95 cm x 6.19 cm x 20 cm) | Duro | 18402 | |
| 5424 Centrifuge for 2 mL tube | Eppendorf | 22620461 | |
| Centrifuge for 96-well plate | Sigma4-16S | 81510 | |
| Centrifuge rotor for 50 mL tubes | Sigma4-16S | 12269 - Biosafe | |
| KAPA HiFi DNA polymerase | Kapa Biosystems |
Referências
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: the microbial ecology of the rhizosphere. Nature Reviews Microbiology. 11, 789-799 (2013).
- Fierer, N. Embracing the unknown: disentangling the complexities of the soil microbiome. Nature Review Microbiology. 15 (10), 579-590 (2017).
- Wang, P., et al. Shifts in microbial communities in soil, rhizosphere and roots of two major crop systems under elevated CO2 and O3. Scientific Reports. 7 (1), 15019 (2017).
- White, L. J., Jothibasu, K., Reese, R. N., Brözel, V. S., Subramanian, S. Spatio Temporal influence of isoflavonoids on bacterial diversity in the soybean Rhizosphere. Molecular Plant-Microbe Interactions. 28 (1), 22-29 (2015).
- Edwards, J., et al. Structure, variation, and assembly of the root-associated microbiomes of rice. Proceedings of the National Academy of Science of the United States of America. 112 (8), E911-E920 (2015).
- Lundberg, D. S., et al. Defining the core Arabidopsis thaliana root microbiome. Nature. 488, 86-90 (2012).
- Bougoure, D. S., Cairney, J. W. Assemblages of ericoid mycorrhizal and other root-associated fungi from Epacris pulchella (Ericaceae) as determined by culturing and direct DNA extraction from roots. Environmental Microbiology. 7 (6), 819-827 (2005).
- Doty, S. L., et al. Diazotrophic endophytes of native black cottonwood and willow. Symbiosis. 47 (1), 23-33 (2009).
- Gottel, N. R., et al. Distinct microbial communities within the endosphere and rhizosphere of populus deltoides roots across contrasting soil types. Applied and Environmental Microbiology. 77 (17), 5934-5944 (2011).
- Xin, G., Glawe, D., Doty, S. L. Characterization of three endophytic, indole-3-acetic acid-producing yeasts occurring in Populus trees. Mycological Research. 113, 973-980 (2009).
- Salter, S. J., et al. Reagent and laboratory contamination can critically impact sequence-based microbiome analyses. BMC Biolology. 12, 87 (2014).
- Gohl, D. M., et al. Systematic improvement of amplicon marker gene methods for increased accuracy in microbiome studies. Nature Biotechnology. 34 (9), 942-949 (2016).
- Kozich, J. J., Westcott, S. L., Baxter, N. T., Highlander, S. K., Schloss, P. D. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform. Applied Environmental Microbiology. 79 (17), 5112-5120 (2013).
- Caporaso, J. G., et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms. ISME Journal. 6 (8), 1621-1624 (2012).
- Lundberg, D. S., Yourstone, S., Mieczkowski, P., Jones, C. D., Dangl, J. L. Practical innovations for high-throughput amplicon sequencing. Nature Methods. 10 (10), 999-1002 (2013).
- Edgar, R. C. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature Methods. 10 (10), 996-998 (2013).
- Caporaso, J. G., et al. QIIME allows analysis of high-throughput community sequencing data. Nature Methods. 7 (5), 335-336 (2010).
- Shannon, C. A mathematical theory of communication. Bell System Technical Journal. 27, 379-423 (1948).
- Simpson, E. H. Measurement of diversity. Nature. 163, 688 (1949).
- Chao, A. Non-parametric estimation of the number of classes in a population. Scandanavian Journal of Statistics. 11, 265-270 (1984).
- Legendre, P. Studying beta diversity: Ecological variation partitioning by multiple regression and canonical analysis. Journal of Plant Ecology. 1 (1), 3-8 (2008).
- Oksanen, J., et al. The vegan package. Community Ecology Package. 10, 631-637 (2007).
- Wickham, H. ggplot2: Elegant graphics for data analysis. Ggplot2: Elegant Graphics for Data Analysis. , 1-212 (2009).
- Cole, J. R., et al. The Ribosomal Database Project (RDP-II): sequences and tools for high-throughput rRNA analysis. Nucleic Acids Research. 33, D294-D296 (2006).
- Wagner, M. R., et al. Host genotype and age shape the leaf and root microbiomes of a wild perennial plant. Nature Communications. 7, 12151 (2016).
- O'Brien, H. E., Parrent, J. L., Jackson, J. A., Moncalvo, J. M., Vilgalys, R. Fungal community analysis by large-scale sequencing of environmental samples. Applied and Environmental Microbiology. 71 (9), 5544-5550 (2005).
- Taylor, D. L., et al. A first comprehensive census of fungi in soil reveals both hyperdiversity and fine-scale niche partitioning. Ecological Monographs. 84 (1), 3-20 (2014).
- Lebeis, S. L., et al. Salicylic acid modulates colonization of the root microbiome by specific bacterial taxa. Science. 349 (6250), 860-864 (2015).
- Ofek-Lalzar, M., et al. Niche and host-associated functional signatures of the root surface microbiome. Nature Commununications. 5, 4950 (2014).
- Niu, B., Paulson, J. N., Zheng, X., Kolter, R. Simplified and representative bacterial community of maize roots. Proceedings of the National Academy of Science of the United States of America. 114 (12), E2450-E2459 (2017).
- Fitzpatrick, C. R., et al. Assembly and ecological function of the root microbiome across angiosperm plant species. Proceedings of the National Academy of Science of the United States of America. 115 (6), E1157-E1165 (2018).
- Ramirez, K. S., Lauber, C. L., Knight, R., Bradford, M. A., Fierer, N. Consistent effects of nitrogen fertilization on soil bacterial communities in contrasting systems. Ecology. 91 (12), 3463-3470 (2010).
- Paungfoo-Lonhienne, C., et al. Turning the table: Plants consume microbes as a source of nutrients. PLoS One. 5 (7), e11915 (2010).
- Yeoh, Y. K., et al. The core root microbiome of sugarcanes cultivated under varying nitrogen fertilizer application. Environmental Microbiology. 18 (5), 1338-1351 (2016).
- Bulgarelli, D., Schlaeppi, K., Spaepen, S., Ver Loren van Themaat, E., Schulze-Lefert, P. Structure and functions of the bacterial microbiota of plants. Annual Review of Plant Biology. 64, 807-838 (2013).
- Peiffer, J. A., et al. Diversity and heritability of the maize rhizosphere microbiome under field conditions. Proceedings of the National Academy of Science of the United States of America. 110 (16), 6548-6553 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados