Method Article
절연 및 토양, Rhizosphere, 및 다년생 잔디 실험에 뿌리에서 미생물 커뮤니티의 분석
요약
필드에서 식물 뿌리의 발굴 뿐만 아니라 endosphere, rhizosphere, 및 토양 샘플의 처리는 DNA 추출 및 데이터 분석 방법 등 자세하게에서 설명 되어 있습니다. 이 문서는 토양, endosphere, rhizosphere microbiomes의 연구에 대 한 이러한 기법을 사용 하 여 다른 실험실을 사용 하도록 설계 되었습니다.
초록
식물 및 토양 미생물 연구 이해 역할 미생물 농업 생산성에서 재생에 대 한 점점 더 중요 한 되 고 있다. 이 논문의 목적은 빠르게 토양, rhizosphere, 및 endosphere 복제 된 현장 실험의 샘플 유형, 치료, 및 식물 유전자 미생물 지역 사회에서 발생할 수 있는 변경 내용을 분석 하는 방법에 정보를 제공 하도록. 이러한 메서드를 보여 주기 위해 사용 하는 실험 웜 시즌 잔디 2, 순수를 포함 하는 복제 필드 플롯의 구성 (Panicum virgatum 및 Andropogon gerardii) 및 낮은 다양성 잔디 혼합물 (A. gerardii, Sorghastrum nutans, 및 Bouteloua curtipendula). 간단히, 식물 발굴, 다양 한 뿌리는 잘라 고 인산 염 버퍼에 배치 다음 동요는 rhizosphere를 수집 하. 뿌리는 얼음 및 표면 소독 표 백제와 에탄올 (EtOH) 실험실에 주어진 다. rhizosphere 필터링 하 고 원심 분리에 의해 집중. 루트 볼 주위에서 발굴된 토양 비닐 봉지에 배치 하 고 토양의 작은 금액을 DNA 추출에 대 한 촬영은 연구실로 가져. DNA는 뿌리, 토양, 및 rhizosphere에서 추출 하 고 16S rRNA 유전자의 v 4 영역에 대 한 뇌관으로 증폭. Amplicons 시퀀싱, 다음 오픈 액세스 생물 정보학 도구와 분석. 이러한 방법을 연구원 미생물 지역 사회 성과 구성 샘플 유형, 치료, 인해 다릅니다 어떻게 테스트 하 고 유전자를 식물. 통계 모델과 함께 이러한 방법을 사용 하 여, 대표적인 결과 입증 뿌리, rhizosphere, 및 토양 미생물 지역 사회에 중요 한 차이가 있다. 여기에 제시 하는 방법 단계 필드 샘플을 수집, 분리, 추출, 계량, 증폭, 및 DNA 시퀀싱 하는 방법에 대 한의 완전 한 세트를 제공 하 고 미생물 지역 사회 성과 복제 필드 재판에서 조성 분석.
서문
미생물 연구 이해 하 고 조작 영양소 사이클링, 유기 물 회전율, 및 개발 또는 토양 병원 균1,2의 저해 등 생태계의 프로세스에 대 한 중요 한 의미를 갖는다. 연구의이 분야는 또한 천연 식물 지역 사회와 agroecosystems의 생산성에 토양 미생물의 영향을 이해 하기 위한 큰 잠재력을 보유 하고있다. 자연 생태계에서 토양 미생물에 초점을 맞춘 많은 연구는, 적은 agroecosystems3공장 rhizosphere 및 endosphere 미생물에 집중 했다. 네브라스카, 농업 농업으로 중요 한 작물 연구에 대 한 중요 한 주제를 재배는 이러한 토양의 연구를 만드는 국가의 큰 부분에 걸쳐 풍경을 지배. 이 방법 종이의 목표는 식물의 뿌리에는 rhizosphere와 endosphere, 그리고 결국 미생물 지역 사회를 수정 하는 방법을 확인 하려면 agroecosystems에 미생물을 설명 하는 프로토콜의 표준 세트와 연구자를 제공 하기 이러한 미생물 토양 건강 및 공장 생산성에서 재생 하는 기능을 이해 합니다.
여기에 제시 된 방법4,5 에서이 종이 어떤 미생물 학습 겨냥은 독점적으로 내부 루트 및 루트 밖에 즉시 미생물에서 서로 어떻게 다른 사람에 의해 사용 되는 방법에서 약간 차이가 있는 rhizosphere입니다. 이 연구에 사용 하는 amplicon 시퀀싱 DNA 샘플에서 발견 미생물 taxa 고 커뮤니티 샘플 형식 또는 치료에 따라 변경 하는 방법을 결정 하는 조사를 허용 합니다. 이 프로토콜 Lundberg 외 에서 사용 하는 매우 비슷한 프로토콜의 주요 차이점 중 하나 6 그 대신 쥡니다,이 프로토콜 사용 하 여 표면 살 균 표 백제와 에탄올 뿌리에서는 rhizosphere를 제거 하는. 다른 사람 또한 효과적으로 표면 살 균 사용7,8,,910. 이러한 메서드는 하지만 약간 다른 다른 방법 보다 더 유리한 있습니다. 이러한 메서드는 충분 한 사람들이 함께 그것은 endosphere, rhizosphere, 및 토양으로 분할 될 때 약 450 샘플까지 추가 하루 150 필드 플롯을 처리할 수 있기 때문에 특히 큰 필드 실험에 적합 합니다. 이 원고 분야에서 샘플, 실험실에서 자료 처리 하 고 추출 하 고, DNA 시퀀싱 하는 데 사용 하는 방법을 자세히 설명 하 고 결과 시퀀싱 데이터를 분석 하는 단계에 대 한 간략 한 개요를 제공 합니다.
프로토콜
1. 필드 사이트 설명
- 컬렉션 기간 동안 실험 분야 사이트를 설명 합니다. 필드 (위도, 경도 및 고도)의 위치를 GPS를 사용 하 여.
- 샘플링 깊이, 샘플링 시간, 및 토양 질감을 설명 합니다.
- 환경 요인 미생물 지역 사회 형성에 중요 한 역할을 한다. 연례 평균 온도, 연례 강 수, 이전 년 윤작, 경작 관행, 정의 메서드 및 필드 사이트의 역사 기후 정보를 기록 합니다. 자동화 된 날씨 방송국 또는 다른 장치는 성장 시기를 통해 일일 강우량과 온도 기록 유용 합니다.
2. 수집 및 토양, Rhizosphere, 및 루트 필드 샘플의 처리
- 식물의 발굴입니다.
- 라벨 세척 팬 및 통 샘플링 수를 식물 재료에 대 한 정보가 들어 있는 스티커 메모와 함께. 플롯 번호와 같은 정보를 포함, 유전자, 식물 및 식물의 종. 음모에 레이블이 양동이 하 고 필드에 설정 된 워크스테이션에서 세척 팬을 남겨두고.
- 토양에서 식물을 들고 옆 뿌리의 잘라 30 cm의 깊이에 삽으로 흙을 피어스. 대략적인 볼륨 18 c m3이다. 임의로 선택한 플롯 내의 다른 지역에서 음모 당 2 개의 식물을 수집 합니다.
- 삽을 활용 하 여 식물의 뿌리를 발굴 하 고 레이블이 양동이에 루트 공을 두고. 분야에서 워크스테이션에 다시 발굴된 루트 공을 레이블이 양동이 가져와. 잘라 하 고 지상 식물 바이오 매스를 삭제.
- 뿌리와 대량 토양에서 토양의 제거.
- 흔들어 뿌리를 수동으로 토양을 제거 하거나 삽 또는 소형 경 운 기를 사용 하 여 뿌리에서 토양을 제거 합니다. 뿌리를 흔들어 아주 모래 토양에서 토양을 제거 해도 됩니다. 장갑을 착용 하 고 뿌리 처리 역 근처 장소.
- 뿌리를 흔들어, 후 씻어 냄비에 토양의 대량 있을 것입니다. 토양 세척 팬에서 혼합 하 고 어떤 토양 clods 소형 타 병으로 헤어. 얼음에 레이블, 17.7 x 19.5 cm 지퍼 스토리지 가방 및 장소 또는 장소에 파편이 면제 되는 토양의 샘플을 놓습니다.
- 뿌리와 rhizosphere의 컬렉션입니다.
- 70에 멸 균 전 정이 위를 사용 하 여 %EtOH, 소비 세 뿌리, 약 4 ~ 6 뿌리 식물 당 및 각 루트 길이 약 9-12 cm의 다양 한. 압력가 마로 소독, 인산 염 버퍼의 35 mL를 포함 하는 레이블이 50 mL 튜브에 삭제 뿌리 (절단에 맞게 필요에 따라) 장소 (6.33 g/L NaH2포4, 8.5 g/L Na2HPO4 무수, pH = 6.5, 200 µ l/L 계면 활성 제).
참고:는 계면 활성 제 ( 재료의 표참조) 추가 되었다 압력가 마로 소독 한 후 인산 염 버퍼. 루트 볼 볼륨 루트 길이 식물 연령과 식물 종에 따라 다릅니다. - 뿌리의 표면에서 rhizosphere 출시를 2 분 동안 튜브를 흔들어. 70%에서 소독 집게와 EtOH, 튜브, 오 점 짧게에서 뿌리에 종이 타 올, 그리고 새에 제거 표시 50 mL 튜브. 둘 다 얼음에 뿌리를 포함 하는 rhizosphere를 포함 하는 튜브를 놓습니다.
- 70에 멸 균 전 정이 위를 사용 하 여 %EtOH, 소비 세 뿌리, 약 4 ~ 6 뿌리 식물 당 및 각 루트 길이 약 9-12 cm의 다양 한. 압력가 마로 소독, 인산 염 버퍼의 35 mL를 포함 하는 레이블이 50 mL 튜브에 삭제 뿌리 (절단에 맞게 필요에 따라) 장소 (6.33 g/L NaH2포4, 8.5 g/L Na2HPO4 무수, pH = 6.5, 200 µ l/L 계면 활성 제).
3. 처리 분야의 실험실에서 샘플
- 필드 컬렉션 후 뿌리의 표면 소독
참고: 필드에서 반환 된 후 최대한 빨리 표면 살 균을 수행 합니다. 표면 수는 같은 날에는 뿌리를 소독, 저장 처리 될 때까지 4 ° C에서 뿌리.- 약 35 mL 50% 표 백제 + 0.01% 트윈 20 50 mL 루트 튜브를 수집 필드에 추가 합니다. 30 ~ 60 초간 50 mL 튜브를 흔들어. 50% 표 백제를 부 어 고 추가 70%의 35 mL EtOH. 또 다른 30-60 초 동안 흔들어입니다.
- 부 어 70 %EtOH 살 균, 초순 물 35 mL를 추가. 1 분 동안 흔들어. 물 세척 두 번 더 반복 합니다.
참고: 우리의 표면 살 균 처리는 충분 한 보장 하기 위해, 우리가 마지막 린스에서 물 샘플을 도금 고 (P. 왕 되지 않은 데이터, 2014) 박테리아의 성장이 관찰. 다른 조사자는 비슷한 방법9,10를 사용 하 여 루트 살 균 효율에 대 한 테스트. - 깨끗 한 종이 타월에 뿌리 건조 되 리. 각 샘플에 대 한 깨끗 한 종이 타월을 사용 합니다.
참고: 종이 타 올은 사용 하기 전에 멸 균 될 수 있다. 우리는 우리의 종이 수건 소독 하지 않습니다. 그러나, 우리는 샘플 당 1 종이 타월을 사용 하 고 수건 포장 사용 될 때까지 계속. - 소독 집게와 약 5mm 조각 장소로 뿌리를 잘라 가지 치기가 위를 사용 하 여 깨끗 하 고 레이블 15 mL 원뿔 튜브에 뿌리를 잘라. 추가 처리 될 때까지-80 ° C에서 샘플을 저장 합니다.
- Rhizosphere 샘플을 처리.
- 전체 샘플을 resuspend 필드에서 rhizosphere 샘플을 포함 하는 50 mL 튜브를 흔들. 살 균, 100 µ m 메쉬 셀 스 트레이너를 사용 하 여 (참조 테이블의 자료), 필터 새 50 mL 튜브에 resuspended 샘플.
- 실 온에서 5 분 동안 3000 x g에서 튜브 원심 즉시 해제와 삭제는 상쾌한.
- 얼음에 50 mL 튜브에 rhizosphere 펠 릿을 배치 합니다. Rhizosphere 펠 릿을 일시 중단 하는 소용돌이 (계면 활성 제) 없이 멸 균 인산 염 버퍼의 1.5 mL를 추가 합니다.
- 피펫으로 깨끗 하 고 레이블, 2 mL microfuge 관으로 일시 중단 된 액체. 실 온에서 2 분 동안 15,871 x g에서 튜브를 회전 합니다. 즉시 부는 상쾌한에 어 및 드레인 깨끗 한 종이 타월에 튜브.
- 추가 처리 될 때까지-20 ° C에서 펠 릿을 저장 합니다.
참고: 단계 3.2.2-3.2.4 수행 됩니다 저장을 위한 샘플 튜브 크기를 줄이기. 그것은 더 많은 공간 저장 작은 2 mL 튜브 50 mL 튜브에 비해 효율적입니다.
- DNA 추출 및 토양 분석을 위한 토양 샘플을 처리.
- 살 균 금속 주걱을 사용 하 여, 약 3 세대 DNA 추출에 대 한 토양의 깨끗 하 고 레이블 2 mL 튜브를 채우십시오. 어떤 작은 루트 조각과 파편 하지 마십시오. -20 ° c.에이 토양 샘플을 저장 70%에 린스 금속 주걱 EtOH 각 샘플 사이의.
- 깨끗 한 세척 팬에서 빈의 가방 토양에 누적된 체 ( 재료의 표참조), 작은 체 위에 큰 체 하 고 수동으로 두 체를 통해 토양 체질. 브러시를 사용 하 여 샘플 사이 체를 신중 하 게 청소.
- 미래 토양 물리 화학 및 텍스처 분석 17.7 x 19.5 cm 지퍼 백에 따로 체질된 토양의 100 ~ 125 g 설정 합니다. 단기 저장을 위해 4 ° C에서 토양의 가방을 놓습니다.
- 토양 수 분에 대 한 토양 샘플을 처리.
- 규모에 레이블이 갈색 종이 봉지 포장 갈색 종이 봉지에 체질 한 토양의 40 ~ 45 g을 측정 합니다. 갈색 종이 봉지 및 데이터 시트에는 토양의 무게를 기록 하 고 장소 가방 건조 오븐에서 55-60 ° c.로 설정
- 72 시간 후 건조 오븐에서 가방을 제거 합니다. 적어도 30 분을 기다린 다음 무게를 토양 가방을 수 있습니다.
- 무게를 녹음 하려면 0 규모를 포장 하 고 규모에 갈색 종이 봉지를 놓습니다. 수식을 사용 하 여 각 샘플에 대 한 토양 수 분의 비율을 계산:
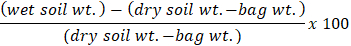
참고: 토양 수 분 방법은 Kellogg 생물학 역 긴 장기 생태 연구 (KBS 필터)에서 이다. KBS 필터 연구원은 그들의 웹사이트 (https://Lter.kbs.msu.edu/)에 대 한 설립된 프로토콜의 넓은 범위를 제공합니다.
4. 준비 된 루트의 DNA 추출에 대 한 샘플링합니다.
- 샘플은 균질 성 언된 루트 자료를 갈기.
- 깨끗 한 박격포와 유 봉 샘플 과정에 걸쳐 냉동 유지 하에 깨끗 한 주걱과 플라스틱 비 커에 액체 질소를 부 어.
- 냉동된 조직 박격포에 배치 하 고 벌금 파우더로는 유 봉과 갈기. 지속적으로 액체 질소 냉동 샘플을 계속 연마를 통해 추가 합니다.
- 주걱을 사용 하 여 깨끗 하 고 레이블 2 mL 튜브에 지상 조직 배치. -80 ° c.에 게
5. 토양 및 96-잘 형식에서 Rhizosphere 샘플에서 DNA 추출
- 96 잘 접시로 토양 샘플을 로드.
- 70 %EtOH 1% 표 백제와 함께 작업 영역 아래로 닦으십시오. 이 단계 동안 실험실 장갑을 착용 한다. -20 ° C 저장에서 토양 샘플을 제거 하 고 얼음 양동이에 해빙 하는 것을 허용.
- DNA 추출 키트와 함께 제공 되는 96-잘 추출 접시에서 봉인 매트 커버를 제거 합니다. 동안 사용 중인 깨끗 하 게 유지 2 종이 잎사귀 사이 봉인 매트 커버를 놓습니다. 오염 방지, 접착 8 잘 PCR를 사용 하 여 추출 96 잘 접시의 12 열 커버를 제거 합니다.
- Tare 메 마른 체중-퍼 널 (크기 SM) 규모와 밖으로 토양의 200 ~ 250 밀리 그램 무게.
참고: 이러한 살 균 퍼 널은 결합 1 개의 측에 그래서 깔때기 규모에 평평 낳는다. 깔때기는 흙으로 가득 하 고 우물에 직접 배치. 이 기술은 샘플의 손실 방지, 흘림, 최소화 그리고 교차 오염을 방지. - 추출 판의 첫 번째 잘 폭로, 신중 하 게 접착 스트립을 들어올려, 장소에 적절 한 채워진된 무게 깔때기의 목 음, 그리고 부드럽게 토양 샘플으로 가이드 적절 한 우물. 잘 커버 접착제 스트립을 교체 합니다.
- 접시 가득 때까지 각 샘플에 대 한 새로운, 살 균 퍼 널을 사용 하 여 판의 모든 잘이 과정을 반복 합니다. 하나는 잘 추출 빈 컨트롤로 비워 놓을.
참고: 하나 잘은 비어 부정적인 (빈) 제어 역할을 모든 추출 접시에 있습니다. 이 키트 시 약11에 있을 수 있는 오염 물질에 대 한 제어 합니다. - 추출 접시에 봉인 매트 커버를 장착 하 고 DNA 추출에 대 한 준비까지-20 ° C에서 접시를 저장.
- 96 잘 접시로 rhizosphere 샘플을 로드.
- -20 ° C 저장에서 rhizosphere 샘플을 제거 하 고 얼음 양동이에 해빙 하는 것을 허용.
- 5.1.2 DNA 추출 접시를 준비 하려면 위의 단계를 따릅니다.
- 규모, 깨끗 한 종이 지우기 장소 다음 눈금에 살 균 금속 주걱을 포장. 주걱을 사용 하 여 신중 하 게 푸 샘플 튜브에서 rhizosphere 펠 릿의 일부 다. 규모에는 주걱을 사이 200과 250 mg rhizosphere 샘플의 무게.
- 조심 스럽게 들어올립니다 밝히기 추출 판의 첫 번째 잘, 잘,으로 채워진된 주걱 각도 및 살 균 이쑤시개와 적절 한 우물에 rhizosphere 자료를 긁어 접착제 스트립.
- 린스 물, 70% 뒤에 금속 주걱 EtOH 샘플 사이. 접시 가득 하나 잘 추출 빈 컨트롤로 빈 떠날 때까지 접시의 모든 잘이 과정을 반복 합니다.
- 96-잘 포맷의 rhizosphere 및 토양 샘플에서 DNA의 추출.
- 추출 토양 및 rhizosphere DNA를 사용 하 여 키트에 대 한 최적화 된 토양 ( 재료의 표참조), 다음 제조 업체의 프로토콜.
참고: 우리는 humic 산 및 토양에서 발견 다른 강력한 PCR 억제제를 제거 하는 독자적인 시 약의 능력 때문에 토양 및 rhizosphere DNA 분리이 특정 키트를 사용 합니다.
- 추출 토양 및 rhizosphere DNA를 사용 하 여 키트에 대 한 최적화 된 토양 ( 재료의 표참조), 다음 제조 업체의 프로토콜.
- DNA의 정량화입니다.
- 92 샘플 및 키트를 사용 하 여 표준의 4 농도 계량 ( 재료의 테이블을참조 기준은 포함), 제조 업체의 프로토콜에 따라.
- 나머지 4 샘플 키트를 사용 하 여 4 개의 표준에 대 한 웰 스에 맞게 접시에서 제거 된 계량 ( 재료의 표참조), 제조 업체의 프로토콜에 따라.
6. 루트에서 DNA 추출 96 잘 형식으로 샘플링합니다.
- 96 잘 접시로 루트 샘플을 로드.
- 70 %EtOH 1% 표 백제와 함께 작업 영역 아래로 닦으십시오. 이러한 단계 중 장갑을 착용 합니다.
- 지상 루트 샘플 드라이 아이스와 함께 양동이에 샘플을 배치 하 여 항상 동결을 유지.
- 액체 질소로 플라스틱 비 커를 진정 플라스틱 비 커에 정전기 방지 microspatulas 및 메 마른 체중-퍼 널 (XSM 크기) 장소.
- 키트와 함께 제공 되는 96-잘 추출 구슬 판을에서 봉인 매트 커버를 제거 하 고 사용에 없는 동안 깨끗 하 게 유지 2 종이 잎사귀 사이 장소.
참고: 이러한 DNA 추출, 수행 시간 이후에 출시 된이 키트의 새 버전 추출 구슬 플레이트를 제공 하는 사용자를 필요 합니다. 테이블의 자료에서 우리는 이러한 항목을 주문 하는 데 필요한 공급 업체 카탈로그 정보를 나열 했습니다. - 오염 방지, 접착 8 잘 PCR를 사용 하 여 추출 96 잘 접시의 12 열 커버를 제거 합니다.
- 얼 스에서 샘플을 계속 드라이 아이스에 추출 구슬 접시를 놓습니다.
- 조심 스럽게 들어 접착제 스트립 추출 판의 첫 번째 잘 폭로, 채워진된 무게 깔때기의 목 적절 한 우물에 놓고 지상 루트 조직의 3 주걱 스푼을 추가 하십시오. 잘 커버 접착제 스트립을 교체 합니다.
참고: 토양 및 rhizosphere는 해 동 얼음 무게, 이전에 식물 재료 냉동 밖으로 무게는 반면. 냉동된 공장 조직, 특히 작은 금액, 해빙 없이 규모에 무게를 어렵습니다. 무게 테스트 얼마나 많은 주걱 숟가락 충분 했다 결정 하기 위해 지상 루트 조직에서 수행 되었다. 키트의 제조 업체 조직의 정확한 금액을 필요 하지 않습니다 하지만 좋습니다 주위 50 mg. 다른 식물 샘플 종류 달라 집니다 그리고 사용자는 적절 한 금액을 결정 해야 합니다. - 접시 가득 때까지 접시의 모든 우물을 위한이 프로세스를 반복 합니다.
- DNA 추출에 대 한 준비까지-20 ° C에서 접시를 저장.
- 루트 조직의 DNA 추출입니다.
- 식물 ( 재료의 표참조)에 대 한 최적화 된 키트를 사용 하 여 DNA를 추출 제조 업체의 프로토콜을 따르고.
참고: 우리는 식물 조직에서 루트 샘플 최대 수익률을 달성 하기 위해 특별히 설계 된이 키트를 사용 합니다. 토양 및 rhizosphere와 달리 humic 산 및 다른 오염 물질은 루트 조직에 대 한 문제가 있습니다.
- 식물 ( 재료의 표참조)에 대 한 최적화 된 키트를 사용 하 여 DNA를 추출 제조 업체의 프로토콜을 따르고.
- 5.4 단계에서 DNA를 계량.
7. 확대 그리고 고립 된 DNA 시퀀싱입니다.
- 중 합 효소를 읽고 증명 V4 지역 16 유전자의 증폭 ( 재료의 표참조) Gohl 외 에 설명 된 대로 12 바코드 샘플 다른 인덱싱 뇌관 및 시퀀싱 하기 전에 수영장. Gohl 외12 V4 뇌관 (보충 파일 1)와 2 단계 PCR 프로세스를 사용 하 여 설명 하는 방법을 사용 하 여 시퀀스입니다.
참고: 일부 메서드의 다음이 단계에서 설명 하는 다른12,,1314 세부 고 따라서 여기서 설명 하지 않습니다. 루트 샘플에 대 한 PNA 차단제는 완전히 설명된15이전 되었습니다 식물 조직에서 증폭 plastid DNA의 양을 줄이기 위해 추가 되었습니다. 두 개의 컨트롤이 시퀀싱, 추출 접시 빈 컨트롤 (단계 5.1.5 후 참고 참조)를 포함 하는 부정적인 제어에 사용 되었다 세균성 DNA의 알려진된 인구의 모의 사회는 긍정적인 역임 ( 재료의 표참조) 제어 합니다.
참고: 대부분의 경우에 MiSeq 시 약 키트 v 3 2 X 300 기본 쌍된 엔드 모드에 사용 됩니다. 이 원고 샘플 Illumina HiSeq 2500 250 쌍-엔드 (2 x 250) 모드와 빠른 모드에서 사용 되었다. 모든 샘플은 동일한 차선에서 시퀀싱 했다. - 미생물 커뮤니티 분석 (USEARCH v9.2.64, QIIME v1.9.1 RStudio v3.4.3 16) 파이프라인을 통해 시퀀싱 데이터를 처리 합니다.
- USEARCH17를 사용 하 여 시퀀싱 데이터를 준비 합니다.
참고: USEARCH는 사용할 수 있는 온라인 전체 지침 (https://www.drive5.com/usearch/). - 인덱스 읽기를 사용 하 여 시퀀싱 데이터를 demultiplex 또는 할당 Illumina 바코드 샘플을 읽습니다.
- 쌍 간 읽기 합의 시퀀스를 병합 합니다. 명령을 사용 하 여: usearch-fastq_mergepairs * R1*.fastq-레이블 재지정 @-fastq_maxdiffs 10-fastq_minmergelen 230-fastq_maxmergelen 320-fastq_pctid 80-fastqout merged.fq.
참고: 매개 변수는 USEARCH 사용 설명서를 참조를 통해 설정 됩니다. - 시퀀싱 데이터를 PCR 반응에 의해 발생할 수 있습니다 뇌관 시퀀스에서 대체에서 뇌관을 제거 합니다. 명령을 사용 하 여: usearch-fastx_truncate merge.fq-stripleft 19-20 stripright-fastqout stripped.fq.
- 시퀀싱 데이터 고품질 읽기를 제거 하 고 계속 높은 품질 조작 상 분류학 단위 (OTU) 시퀀스를 필터링 합니다. 명령을 사용 하 여: usearch-fastq_filter stripped.fq-fastq_maxee 1.0-fastaout filtered.fa.
- USEARCH17를 사용 하 여 시퀀싱 데이터를 준비 합니다.
- USEARCH에서 OTUs를 생성 합니다.
- 고유한 OTU 시퀀스의 집합을 식별 하려면 dereplication을 수행 합니다. 명령을 사용 하 여: usearch-fastx_uniques filtered.fa-fastaout uniques.fa-sizeout-클럭이 고유.
- 97-100% 순서 유사성과 클러스터 OTUs 독특한 OTUs 지정 하 명령을 사용 하 여: usearch-cluster_otus uniques.fa-minsize 2-otus otus.fa-Otu 클럭이.
참고:이 단계는 또한 클러스터 OTUs에서 싱글톤의 제거 및 시퀀싱 데이터에서 키메라의 제거 포함합니다. - USEARCH에는 OTU 테이블을 만듭니다. 명령을 사용 하 여: usearch-usearch_global stripped.fq-db otus.fa-플러스-id 0.97-otutabout otutable.txt 물가.
참고:이 명령은 각 샘플에 대 한 모든 OTUs의 읽기 (수) 수는 테이블을 생성합니다. OTU 테이블 다운스트림 단계 차동 풍부 분석 및 미생물 다양성 분석을 포함 하 여 사용 됩니다. OTU 테이블의 예는 보충 그림 2에에서 표시 됩니다. - QIIME v1.9.1 18희박 분석을 수행 합니다.
주: 진공 곡선 시퀀싱의 깊이 미생물 지역 사회를 제대로 샘플링 여부를 확인 하려면 OTU 테이블을 사용 하 여 계산 됩니다. 진공 곡선의 예는 보충 그림 3에 표시 됩니다. - 18알파 다양성 분석 수행 합니다. QIIME v1.9.1에서 alpha_rarefaction.py를 사용 하 여 각 샘플 내의 미생물 커뮤니티의 다양성을 계산.
참고:이 분석 논19, 심슨20, Chao121등 다양성 지 수를 계산합니다. - Beta 다양성 분석18,22를 실시 합니다. Python 스크립트를 사용 하 여: QIIME v1.9.1 브 레이-커티스 호환성 매트릭스에 beta_diversity_through_plots.py.
참고:이 분석 샘플 사이의 미생물 커뮤니티 구성을 비교합니다. - 그룹 간의 통계적 분석을 실시 합니다. 채식주의 패키지23 v2.4.5 RStudio16에서 아도니스와 anova 함수를 사용 하 여 PERMONOVA에 대 한 계산 거리 행렬을 사용 합니다. 주 좌표 (모자) 분석 채식주의 패키지에서 capscale 함수를 사용 하 여 정식 분석을 수행 합니다. RStudio의 ggplot2 패키지24 v2.2.1를 사용 하 여 데이터 시각화.
결과
이 원고에 제시 하는 대표적인 결과 필드 사이트 미드 근처 대학 네브라스카 링컨 농업 연구 부문 농장에서 2012 년에 설립, 실험, 사이트 니 전에 옥수수-콩 순환으로 관리 했다 . 세 가지 유형의 토양에 위치한 연구 사이트 하지만 측정된 토양 속성의 모든 변경 사항을 부과 하는 치료로 인해 마치 데이터 분석.
분야 사이트 2, 순수 포함, 큰 bluestem, indiangrass (S. nutans), 그리고 '버트' sideoats grama (낮은 다양성 잔디 혼합물 뿐만 아니라 switchgrass (P. virgatum 이력서 자유)와 큰 bluestem (A. gerardii)의 의미 B. curtipendula)입니다. 3 따뜻한 시즌 잔디 플롯 세 번 복제 된 완전 한 무작위 블록 디자인에 있었다. 3 다른 잔디 플롯으로 중첩 되었고 56 (N1) 112kg (N2) N 하는 2 개의 질소 (N) 수정 처리 적용된 요소-1 했다. 성장 시기의 끝에 미생물 샘플링 시 음모에서 포함 된 토양 8.0 ± 1.1 (평균 ± SD) ppm 질산염 함께 수정 112kg N 하-1 과 6.8 ± 0.7 (평균 + SD) 음모에 ppm 질산염 함께 수정 56 kg N 하-1. 음모는 일년에 한 번을 수정 된 했다. 음모는 주요 플롯 (8000 m2) 및 N 치료로 지정 되었다 웜 시즌 잔디 분할 플롯 (4000 m2) 했다. 큰 bluestem '보 난 자'와 '금광'의 50: 50 혼합으로 시드 되었습니다 그리고 Indiangrass '스카우트'와 '전사'의 50: 50 혼합으로 시드 되었습니다. 음모는 2012 년에 설치 했다 그리고 첫 번째 N 응용 프로그램 2013의 봄.
토양 및 루트 샘플링은 2014 년 9 월 15 일에 실시 되었다. 3 복제 (그림 1)과 분할 플롯 무작위 디자인으로 설정 된 필드에 아래 설명 된 작업 실시 됐다. Endosphere에 대 한 샘플은 다음과 같이 모든 평균 시퀀싱 깊이: 4871 ± 5711 (평균 ± SD), rhizosphere: 40726 ± 14684, 토양: 38184 ± 9043. 설명, 방법을 사용 하 여 이러한 실험에 변화의 가장 큰 소스 중 하나 미생물 지역 사회 발견 샘플 형식 (그림 2) 사이에서 차이가 있습니다. 이 대표적인 데이터 세트에서 rhizosphere 및 토양 서로 구성 (그림 2A) endosphere 보다 더 비슷한 것으로 나타납니다. 그러나, 거기 또한 매우 중요 한 했다 (p = 0.001) rhizosphere와 (그림 2B) 토양 미생물 지역 사회 구성에 차이. 샘플 형식에 의해 분석 이러한 실험에서 차지 하 고 총 변화 26% 이었다.
알파 다양성 분석 미생물 지역 사회는 endosphere에서 낮은 토양와 rhizosphere (그림 3) 샘플 다양성에는 나타났다. 어떤 구획에 잔디 종 간의 다양성에만 큰 차이가 큰 bluestem의 endosphere 샘플 및 switchgrass, switchgrass 데 상당히 높은 미생물 종 다양성 (그림 3)와 함께 사이 했다. 관계 되는 풍부 분석 (그림 4) Proteobacteria Actinobacteria 뒤에 모든 샘플 종류에서의 지배를 강조 표시 합니다. 토양 및 rhizosphere는 또한에 의해 지배 Acidobacteria 와 Chloroflexi 는 endosphere Bacteriodetes의 더 큰 상대 풍부 했다 반면.
이 실험에서 두 개의 서로 다른 양의 N 비료 식물 성장 했다 고 우리 치료 효과 있었습니다 여부를 확인 하기 위해 데이터를 분석 하는 따라서. 치료 효과 총 변이의 12%를 차지 하지만 안 수에서 두 치료는 다른 (그림 5) 보지 크게 다른 아니었다. 이 이러한 데이터 집합 보다는 육안 검사 또는 질적 판단에 대 한 통계 분석의 중요성을 강조 한다.
식물 조직 및 토양의 미생물에서 식물 영향 차이 안 수의 제한 된 메서드를 사용 하 여 시각화 했다. 통계적 인 차이 사용 하 여 PERMANOVA 분석은 크게 다른 미생물 커뮤니티 구성 샘플 사이에서 종, 등의 특정 변수 결과 여부 테스트를 결정 했다. 모든 샘플 종류 함께 분석 했다 때 매우 중요 한 차이 때문에 식물 종 (그림 6) 미생물 지역 사회 구성에서 발견 되었다. 이 실험에서 변형 식물 종에 의해의 양은 6.7% 이었다. 마지막으로, 각 샘플 형식 샘플 종류의 운전 하 고 있을 중요 한 식물 종 효과 결정 하기 위해 개별적으로 분석 했다. endosphere에서 서만 거기 있 었 어 매우 중요 한 차이 (p = 0.001) (그림 7) 다른 식물 종의 미생물 커뮤니티 구성 사이. 다른 샘플 형식에서는 종 효과 개별적으로 분석 하면 중요 한 되지 않았습니다. Endosphere, 종 인 백분율 변화 했다 rhizosphere (18%) 및 토양 (15%)에서 낮은 반면 27%, 이었다. 이 더 각 조직 유형을 개별적으로 분석의 중요성을 강조 한다.
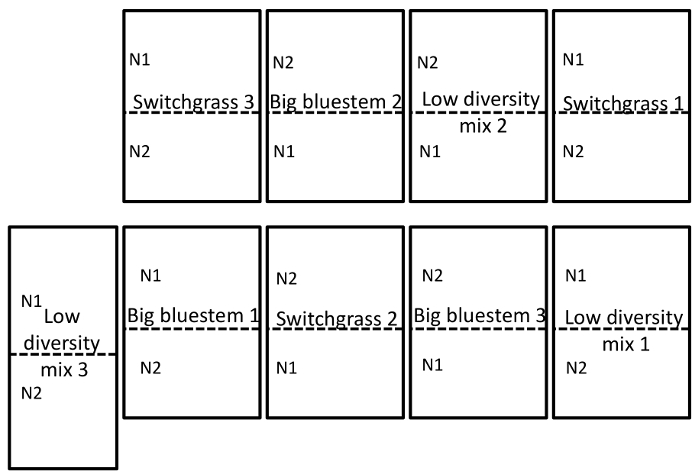
그림 1: 실험 분야 디자인의 예. 대학 네브라스카 링컨 동부 네브라스카 연구와 미드, 네브라스카 근처 센터에 위치한 필드 사이트의 3 중에서 완전 한 무작위 블록 디자인을 설명 하는 실험 분야 디자인 전체 사이트 설명에 대 한 결과 섹션을 참조. N 1은 낮은 (56 kg N 하-1 요소) 및 N2 (112 k N g 하-1 요소) 적용 된 높은 질소 비율 이다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
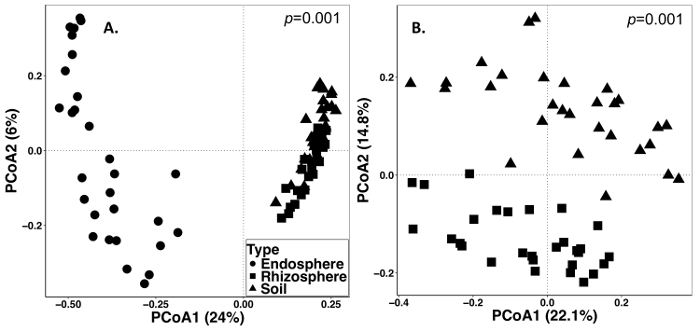
그림 2: endosphere, rhizosphere, 및 2014에 다년생 잔디 샘플링에서 토양을 포함 하 여 다른 샘플 형식에서 미생물 구성 비교 베타 다양성 분석. 분석 브 레이 커티스 호환성 매트릭스를 생성 하 QIIME1.9.1에 파이썬 스크립트를 사용 하 여 실행 되었다. 주 좌표 분석 (PCoA) 브 레이 커티스 호환성 매트릭스에 따라 RStudio에서 구상 되었다. PCoA1과 PCoA2 PCoA 분석에 의해 설명 하는 첫 번째와 두 번째로 큰 분산을 나타냅니다. PERMANOVA 통계 분석 샘플 형식 간의 중요성을 결정 하기 위해 수행 하 고 p 값 오른쪽 상단 모서리에 표시 됩니다. 그림에 각 기호는 각 샘플에 대 한 전체 미생물 지역 사회를 나타냅니다. (A) Endosphere, rhizosphere 및 토양 샘플 형식 함께 분석 했다. 모든 87 샘플 샘플 당 486 시퀀스에 올라가기 했다. (B) Rhizosphere 및 토양 샘플 함께 분석 했다. 모든 59 샘플 8231 시퀀스 올라가기 했다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
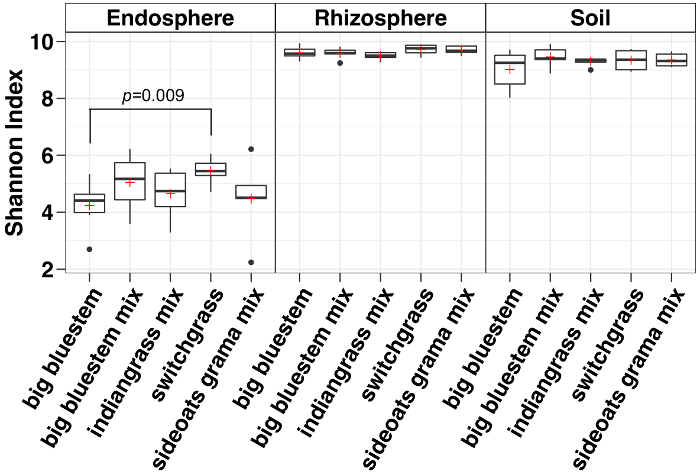
그림 3: endosphere, rhizosphere 및 토양에 각 종족에 대 한 논 지를 사용 하 여 알파 다양성 분석. 분석은 QIIME1.9.1에 파이썬 스크립트를 사용 하 여 실행 되었다. 진공은 endosphere, rhizosphere, 및 토양 샘플 형식 샘플 당 486, 17154 및 8231 시퀀스와 각각에 대 한 수행 되었다. 상자 표시 25 그리고 75 백분위 (첫 번째 및 세 번째 quartiles). 상자 안에 가로줄 중간값과 빨간색을 나타냅니다 플러스 평균을 보여 줍니다. 수염 outliers (이 검은 점 들으로 표시 됩니다) 그 보다 1.5 배 interquartile 범위를 제외 하는 데이터의 범위를 표시 (n = 6 sideoats grama 섞는다 어디를 제외 하 고 각 샘플에 대 한 n = 5). 섀 넌 인덱스는 endosphere에 있는 모든 5 종의 rhizosphere와 토양 보다 낮은 했다. 비 파라메트릭 Wilcoxon 순위 합계 테스트 종 사이 의미를 결정 하는 데 사용 했다 하 고 큰 차이가 종 상자 위에 표시 했다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
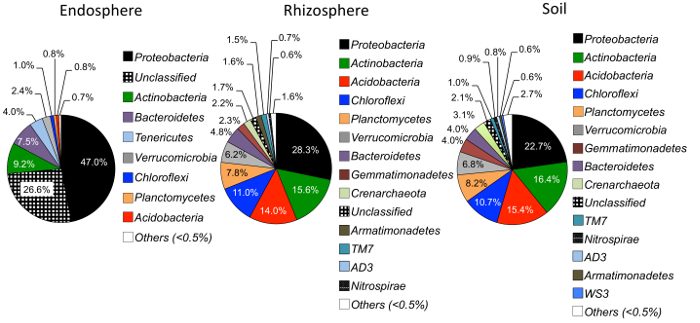
그림 4: endosphere, rhizosphere, 및 토양에 있는 문 수준에 관계 되는 풍부. 샘플 다른 샘플 종류 중에서 미생물 문의의 풍부를 비교 분석 했다 (n = 각 샘플 형식에 대 한 29). 분석은 OTU 테이블에서 QIIME1.9.1에 파이썬 스크립트를 사용 하 여 실행 되었다. 원형 차트 내부 다양 한 색상은 문의 나타냅니다. 백분율은 각 문 각 샘플 형식에의 관계 되는 풍부를 나타냅니다. 문 정보 했다 Ribosomal 데이터베이스 프로젝트 분류자 (RDP)25를 사용 하 여 주석 처리 됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
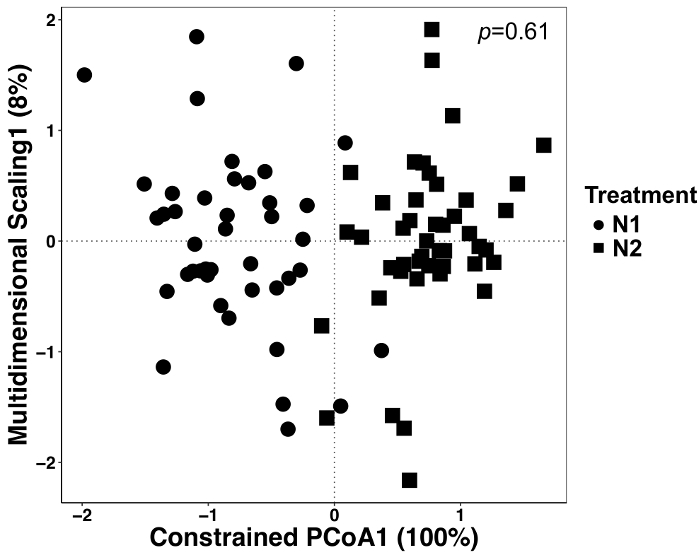
그림 5: 모든 샘플 형식 간의 제약 요인으로 치료를 사용 하 여 분석. 주 좌표 (모자) 분석의 정식 분석 치료 사이 미생물 지역 사회 구성에 차이가 있었는지 확인 하기 위해 수행 되었다. 치료를 위해 각 N, n = 42 n 1에 대 한 (56 k N g 하-1) 및 n n 2에 대 한 = 45 (112kg N 하-1). 브 레이 커티스 호환성 매트릭스는 QIIME1.9.1에 파이썬 스크립트를 사용 하 여 생성 되었습니다. 브 레이 커티스 호환성 매트릭스에 따라 모자 분석 RStudio에서 요인으로 치료를 제한 하 여 이루어졌다. PERMANOVA 분석 치료 차이 했다 중요 한, p 값은 오른쪽 상단에 표시 됩니다 있는지 확인 위해 수행 되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
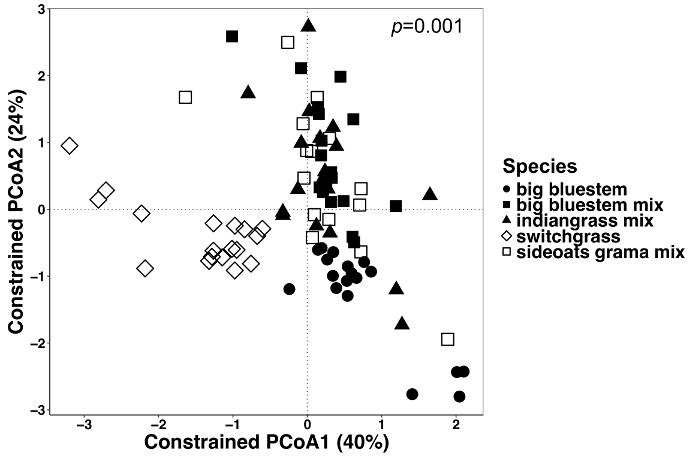
그림 6: 모든 샘플 형식 간의 제약 요인으로 식물을 사용 하 여 분석. 분석은 모든 샘플 종류에서 식물 종 사이 미생물 지역 사회 구성에 차이가 있었는지 확인 하기 위해 실시 됐다. 주 좌표 조정 및 모든 샘플 형식 (endosphere, rhizosphere, 및 토양)의 모자 분석 수행 되었다 브 레이 커티스 호환성 매트릭스를 사용 하 여. 브 레이 커티스 호환성 매트릭스 QIIME1.9.1에 파이썬 스크립트를 사용 하 여 생성 되었습니다. 브 레이 커티스 호환성 매트릭스에 따라 모자 분석 RStudio에서 요인으로 제약 하는 식물 종에 의해 이루어졌다. PERMANOVA 통계 분석 식물 종 사이 의미를 결정 하기 위해 수행 하 고 P 값 오른쪽 상단 모서리에 표시 됩니다. 각 기호는 그림에 그 샘플에 대 한 전체 미생물 지역 사회를 나타냅니다. n = n 을 제외한 모든 샘플 종류에 각 종족에 대 한 18 = 15 sideoats grama 믹스에 대 한. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
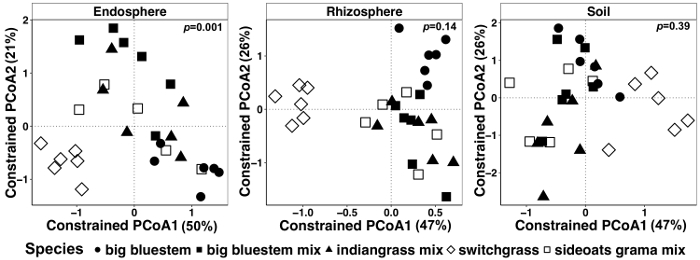
그림 7: 종을 사용 하 여 제약 요인으로 각 샘플 유형에 개별적으로 모자 분석의 예. 주 좌표 조정 및 각 샘플 형식 (endosphere, rhizosphere, 및 토양)의 모자 분석 브 레이 커티스 호환성 매트릭스를 사용 하 여. 각 샘플 종류 486, 17154, 및 endosphere, rhizosphere 및 토양에서 각각 샘플 당 8231 읽기에 올라가기는. 종은 안 수를 제한 하는 요인으로 사용 되었다. PERMANOVA 통계 분석 각 샘플 유형에서 식물 종 사이 의미를 결정 하기 위해 수행 하 고 p 값 오른쪽 상단 모서리에 표시 됩니다. 그림에서 각 기호는 각 샘플에 대 한 전체 미생물 지역 사회를 나타냅니다. 샘플 크기는 n = 29 각 샘플 유형, n = sideoats grama 믹스를 제외한 각 샘플 형식에 각 식물 종에 대 한 6 (n = 5). 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
토론
이 원고에 설명 된 방법을 쉽게 토양과 식물 metagenomics의 필드를 입력 하는 과학자를 사용 해야 합니다. 년 동안, 우리는이 원고에서 설명 하는 실험을 실시 이후 우리의 방법을 세련는. 한 변화는 우리가 지금 미리 라벨 튜브 샘플 필드에 외출 하기 전에. 우리의 실험실 바코드 시스템 및 라벨 프린터를 사용합니다. 라벨 프린터는 뿐만 아니라 시간 때 라벨 튜브, 뿐만 아니라 모든 것을 쉽게 추적 하 고 올바르게 식별 인간의 손 쓰기의 vagaries 없이 샘플을 저장 합니다. 또 다른 중요 한 요점은 우리는 다시 가능한 한 빨리 필드에서 그것를 데 려 후 자료를 처리 하려고입니다. 우리는 DNA 분석에 사용 되는 토양 동결, 소독, 뿌리를 고정 필터링 그리고는 rhizosphere 필드에서 반환 된 후 12 ~ 36 시간 이내 동결 하고자 합니다. DNA 추출 절차는 토양을 rhizosphere, 특히 많은 단계로 긴 그래서 우리 DNA 추출 프로토콜에 대 한 시간에 손을 최소화, 도입 될 수 있습니다, 인간의 오류를 감소 하는 로봇 (Kingfisher 플렉스, ThermoFisher) 구입 다른 방법에 일관성을 향상 하 고 토양, 뿌리, 또는 rhizosphere의 일괄 처리 됩니다. 공장 설비 재료를 작업할 때 그것은 공부 또는 "견본"를 루트 종류의 다양 한 루트 형식을 결정 하는 것이 중요. 유지 뿌리 그리고 때 DNA 추출 수행 하는 것이 중요, 96-잘 DNA 추출 접시를 채울 때 샘플 사이의 교차 오염 없이 보장은 냉동된 상태에서 떠난다. 고려해 야 할 또 다른 중요 한 요소는 필드 실험을 디자인 하 고 완전 한를 사용 하 여 무작위로 디자인 가능한 때 사용할 복제의 번호26. 높은 분야 다양성 때문에 그것은 작은 차이 감지 하는 복제의 다 수 필요할 수 있습니다. 마지막으로, 우리의 경험에서 뿌리를 발굴 하는 때 토양은 너무 젖은 하지 있는지 확인이 필수적입니다. 토양 물으로 포화 되는 경우 그것은 작업할 더러운 하지만 그것은 또한 매우 어려운는 rhizosphere를 정의 하 고 뿌리에서 토양을 제거 하.
초기에 이러한 방법을 개발 하는 동안 만들어진 하나의 수정 했다 대신 우리 분야에 더 많은 작업을 쉽게 하기 위해 가스 발전기에 의해 구동 vortexers 업그레이드 rhizosphere 출시를 손으로 튜브를 떨고 표준화의 관점에서 시간과 방식으로 각 튜브 동요 했다. Amplicon 시퀀싱 방법의 한 가지 한계는 결과 분류학 해상도 종종 제한 됩니다 많은 OTUs는 알 수 없는 또는 유일한 알려진 가족 또는 속 수준입니다. 연구의이 분야는 새롭고 개발 방법, 데이터 분석 결과의 해상도 향상 시킬 수 있는 특히 인식 하는 것이 중요 하다 그래서 빠르게 진화 하고있다.
이러한 프로토콜은 박테리아와 archaea, 버섯 하지 공부에 합니다. 확대를 위한 다른 프라이 머를 사용 하 여 동일한 DNA 샘플27,28를 사용 하 여 곰 팡이 사회의 연구에 대 한 허용할 것 이다. 이러한 메서드는 방법 단순화 될 수 있기 때문에 대량의 장비 구매를 필요 하지 않습니다. 우리가 설명 하는 방법 "누구 가", 결정 하는 데 주로 운항 하지만 필드의 기능을 테스트 및 샷건 시퀀싱 방법, 격리를 사용 하 여 해결 될 수 있는 함수에 대 한 중요 한 질문에 신속 하 게 진화 하 고 미생물, 또는 시퀀싱 전체 미생물 게놈입니다.
대표 결과 설명 된 방법을 사용 하 여 식별 될 수 있는 미생물 지역 사회에서 차이 강조 표시 합니다. 데이터 분석22에 베타-다양성 방식을 사용 하 여, 작곡 차이 샘플 종류 사이 표시 했다. 이러한 차이 명확 하 게 endosphere, rhizosphere, 및 토양 고유의 미생물 커뮤니티3을 포함 대부분의 다른 연구에서 관찰 되었습니다. Shannon 다양성 지 풍부 하 고 있는 각 식물 종에서 endosphere, rhizosphere, 및 토양 미생물 종의 균일성을 결정 하기 위해 계산 되었다. 이 연구 및 많은 다른 사람에서 같이, 알파 다양성은 토양는 rhizosphere에서 약간 감소 하 고 endosphere3,,529에서 크게 감소 후에 가장 많습니다. 이러한 결과 여기에 설명 된 메서드는 endosphere, rhizosphere, 및 토양에서 구성 변경 내용을 식별 하는 데 적합 한 나타냅니다.
Proteobacteria 의 지배력은 endosphere 및 토양30,,3132에 대 한 연구에서 일반적인 찾아내는 이다. Endosphere는 일반적으로 Proteobacteria의 더 높은 관계 되는 풍부를 가진 낮은 미생물 종의 다양성이 있다. 이 다시 여기 결과 문학에 있는 다른 발견의 대표는 강조 한다. 이 연구에서 치료 효과가 크게 다른 되었고 그에 대 한 두 가지 주요 이유는 치료에 의해 부과 된 차이 감지에 충분 한 변화를 생성 하기에 충분 되지 않은 고이 샘플링 했다의 끝에 있을 수 있습니다는 성장 계절 때 필드 비슷한 수준으로는 무슨 절 기의 끝에 측정 했다 질소 내려 그릴 충분 한 시간을 했다 수 있습니다. 또 다른 연구에서 시간의 긴 기간 동안 비슷한 수정 속도 사용 하 여, 단지 상대적으로 작은 변화는 미생물의 구성에 측정된33했다. 다른 연구 질소 비료34,35인해 곰 팡이 및 세균 지역 사회에서 변화를 보여왔다.
식물 종의 그들의 microbiomes3,,3236 결정에 역할을 알려지고 미생물 지역 사회 변화에도 작은 차이에서 다른 식물 genotypes 사이 입증 되었습니다가 단일 종37 본이 연구에서는 미생물 지역 사회 구성에 상당한 차이 식물 종 사이 발견 되었다. 모든 샘플 종류에 그것은 switchgrass 가장 뚜렷한 미생물 구성 했지만 종 간의 차이 endosphere에 통계적만 했다 나타났다. Rhizosphere 커뮤니티 구성 수 있습니다 더 많은 복제 분석에 사용할 수 있는 경우 중요 한 되고있다.
결합 된 필드, 실험실, 및 여기에서 설명 하는 분석 프로토콜, rhizosphere, 토양과 뿌리36의 endosphere에 미생물 커뮤니티의 구성 어떻게 다른 요인 영향 공부에 대 한 강력한 방법을 제공 합니다. 많은 농업 분야에서 특히 microbiomes의 지역에서 해야 할 일이 있다. 수익률 토양 미생물에 의해 변경 되는 방법에 대 한 중요 한 질문은 아직 완전히 해명 하. 자르기 회전 영향 토양 미생물, 타이밍은 미생물을 변경 하는 방법, 어떻게 비 생물 적인 긴장을 어떻게에 관한 가장 기본적인 질문에도 미생물, 변경 여부 및 토양 유형에 미생물을 변경할 이러한 요소와 상호 작용 하는 방법 특정 작물 및 미국 지역에서 보편적인 미생물 모든 관련 질문은. 이러한 방법은 존재 및 병원 성 및 유익한 박테리아의 지 속성을 식별 하기 위해 역학 연구도 유용할 것 이다. 이러한 방법에 대 한 또 다른 미래 지평선 DNA 방법 여기에 설명 된 식물 및 미생물 RNA 대사 산물 데이터 통합을 시작 하는 것입니다. 추가 개선 및 더 많은 변수 테스트 추가 이러한 프로토콜의 최적화를 위해 중요할 것 이다.
공개
저자는 공개 없다.
감사의 말
이 원고를의 개발은 국립 과학 재단 EPSCoR 센터 루트 및 Rhizobiome 혁신 상을 OIA-1557417에 지원 됩니다. 데이터 수집 네브래스카 링컨의 대학, 농업 연구 및 개발에서 자금 및 농 무부에서 해치 교부 금에 의해 지원 되었다. 우리는 또한 미국 농 무부-ARS에서 지원 인정 하 고 지원 설정 및 이러한 필드를 관리 하는 미국 농 무부 식품 및 농업에서 농업과 음식 연구 사업 경쟁 보조금 번호 2011-68005-30411에 의해 제공 되었다.
자료
| Name | Company | Catalog Number | Comments |
| Dneasy PowerSoil HTP 96 Kit | Qiagen/MoBio | 12955-4 | Extraction kit for soil and rhizosphere |
| Dneasy PowerPlant HTP 96 Kit | Qiagen/MoBio | 13496-4 | Extraction kit for roots |
| D-Handle Digging shovel, 101 cm L | Fiskars | 9669 | |
| Rapid Tiller, 40 cm L | Truper | 34316 | |
| Ziploc Bags, 17.7 cm x 19.5 cm | Ziploc | NA | |
| Cooler | Any | NA | |
| Wash pan | Any | NA | |
| Plastic bucket | Any | NA | |
| Gloves (work and lab) | Any | NA | |
| 20 cm diameter Soil sieve #8, 2360 μm mesh size | Dual Manufacturing Co., Chicago IL | US8-8FS | |
| 20 cm diameter Soil Sieve #4, 4750 μm mesh size | Dual Manufacturing Co., Chicago IL | US8-4FS | |
| portable generator | Honda brand works well | NA | |
| Sterile cell strainers 100 μm mesh size | Fisher Scientific | 22-363-549 | |
| NaH2PO4·H2O | VWR | 0823 | |
| Na2HPO4 | VWR | 0404 | |
| Silwet L-77 | Lehman Seeds | VIS-30 | Surfactant |
| Autoclaves | Any | NA | |
| Drying Oven | Any | NA | |
| Scale | Any | Any | |
| Bleach | CLOROX - household strength | NA | |
| Tween 20 | Any | NA | |
| Liquid Nitrogen | Any | NA | |
| Dry Ice pellets | Any | NA | |
| Ethanol | Any | NA | |
| 11 cm precision fine point tweezers | Fisher | 17456209 | |
| 18 cm Straight point specimen forceps | VWR | 82027-436 | |
| 13.5 cm Pruning Scissors | Fiskars | 9921 | |
| 2 mL tube | Any | NA | |
| 15 mL PP conical tube | MIDSCI | C15B | |
| 50 mL PP conical tube | MIDSCI | C50B | |
| Ultrapure water | Millipore-sigma | Milli-Q Integral, Q-POD | |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32854 | For DNA quantification of removed samples |
| QuantiFluor dsDNA System | Promega | E2670 | For DNA quantification |
| 96-Well Black with Clear Flat Bottom Plates | Corning | 3631 | |
| pPNA PCR Blocker | PNA Bio | PP01-50 | |
| mPNA PCR Blocker | PNA Bio | MP01-50 | |
| Genomic DNA from Microbial Mock Community B (Even Low Concentration) v5.1L, for 16s rRNA Gene sequencing | BEI Resources | HM782D | |
| Adhesive 8 well-strips for plates | VWR | 89134-434 | |
| Stainless steel beads, 3.2 mm dia | Next Advance | SSB32 | |
| 1 ml Assay block (DNA extraction plate for the Qiagen/MoBio Dneasy PowerPlant HTP Kit) | CoStar | 3959 | |
| antistatic PP weighing funnel, size small for soil/rhizosphere | TWD Tradewinds, INC | ASWF1SPK | |
| antistatic PP weighing funnel, size x-small for root/leaf | TWD Tradewinds, INC | ASWFXSCS | |
| Genie 2 Digital Vortex | Scientific Industries | SI-0236 | |
| Vortex adapter for 50 mL tubes | Scientific Industries | SI-H506 | |
| Mortar (100 mL) and pestle | Any | NA | |
| Metal micro-spatula | VWR | 80071-672 | |
| Disposable antistatic microspatulas | VWR | 231-0106 | |
| Brown Paper bag 2# (10.95 cm x 6.19 cm x 20 cm) | Duro | 18402 | |
| 5424 Centrifuge for 2 mL tube | Eppendorf | 22620461 | |
| Centrifuge for 96-well plate | Sigma4-16S | 81510 | |
| Centrifuge rotor for 50 mL tubes | Sigma4-16S | 12269 - Biosafe | |
| KAPA HiFi DNA polymerase | Kapa Biosystems |
참고문헌
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: the microbial ecology of the rhizosphere. Nature Reviews Microbiology. 11, 789-799 (2013).
- Fierer, N. Embracing the unknown: disentangling the complexities of the soil microbiome. Nature Review Microbiology. 15 (10), 579-590 (2017).
- Wang, P., et al. Shifts in microbial communities in soil, rhizosphere and roots of two major crop systems under elevated CO2 and O3. Scientific Reports. 7 (1), 15019 (2017).
- White, L. J., Jothibasu, K., Reese, R. N., Brözel, V. S., Subramanian, S. Spatio Temporal influence of isoflavonoids on bacterial diversity in the soybean Rhizosphere. Molecular Plant-Microbe Interactions. 28 (1), 22-29 (2015).
- Edwards, J., et al. Structure, variation, and assembly of the root-associated microbiomes of rice. Proceedings of the National Academy of Science of the United States of America. 112 (8), E911-E920 (2015).
- Lundberg, D. S., et al. Defining the core Arabidopsis thaliana root microbiome. Nature. 488, 86-90 (2012).
- Bougoure, D. S., Cairney, J. W. Assemblages of ericoid mycorrhizal and other root-associated fungi from Epacris pulchella (Ericaceae) as determined by culturing and direct DNA extraction from roots. Environmental Microbiology. 7 (6), 819-827 (2005).
- Doty, S. L., et al. Diazotrophic endophytes of native black cottonwood and willow. Symbiosis. 47 (1), 23-33 (2009).
- Gottel, N. R., et al. Distinct microbial communities within the endosphere and rhizosphere of populus deltoides roots across contrasting soil types. Applied and Environmental Microbiology. 77 (17), 5934-5944 (2011).
- Xin, G., Glawe, D., Doty, S. L. Characterization of three endophytic, indole-3-acetic acid-producing yeasts occurring in Populus trees. Mycological Research. 113, 973-980 (2009).
- Salter, S. J., et al. Reagent and laboratory contamination can critically impact sequence-based microbiome analyses. BMC Biolology. 12, 87 (2014).
- Gohl, D. M., et al. Systematic improvement of amplicon marker gene methods for increased accuracy in microbiome studies. Nature Biotechnology. 34 (9), 942-949 (2016).
- Kozich, J. J., Westcott, S. L., Baxter, N. T., Highlander, S. K., Schloss, P. D. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform. Applied Environmental Microbiology. 79 (17), 5112-5120 (2013).
- Caporaso, J. G., et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms. ISME Journal. 6 (8), 1621-1624 (2012).
- Lundberg, D. S., Yourstone, S., Mieczkowski, P., Jones, C. D., Dangl, J. L. Practical innovations for high-throughput amplicon sequencing. Nature Methods. 10 (10), 999-1002 (2013).
- Edgar, R. C. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature Methods. 10 (10), 996-998 (2013).
- Caporaso, J. G., et al. QIIME allows analysis of high-throughput community sequencing data. Nature Methods. 7 (5), 335-336 (2010).
- Shannon, C. A mathematical theory of communication. Bell System Technical Journal. 27, 379-423 (1948).
- Simpson, E. H. Measurement of diversity. Nature. 163, 688 (1949).
- Chao, A. Non-parametric estimation of the number of classes in a population. Scandanavian Journal of Statistics. 11, 265-270 (1984).
- Legendre, P. Studying beta diversity: Ecological variation partitioning by multiple regression and canonical analysis. Journal of Plant Ecology. 1 (1), 3-8 (2008).
- Oksanen, J., et al. The vegan package. Community Ecology Package. 10, 631-637 (2007).
- Wickham, H. ggplot2: Elegant graphics for data analysis. Ggplot2: Elegant Graphics for Data Analysis. , 1-212 (2009).
- Cole, J. R., et al. The Ribosomal Database Project (RDP-II): sequences and tools for high-throughput rRNA analysis. Nucleic Acids Research. 33, D294-D296 (2006).
- Wagner, M. R., et al. Host genotype and age shape the leaf and root microbiomes of a wild perennial plant. Nature Communications. 7, 12151 (2016).
- O'Brien, H. E., Parrent, J. L., Jackson, J. A., Moncalvo, J. M., Vilgalys, R. Fungal community analysis by large-scale sequencing of environmental samples. Applied and Environmental Microbiology. 71 (9), 5544-5550 (2005).
- Taylor, D. L., et al. A first comprehensive census of fungi in soil reveals both hyperdiversity and fine-scale niche partitioning. Ecological Monographs. 84 (1), 3-20 (2014).
- Lebeis, S. L., et al. Salicylic acid modulates colonization of the root microbiome by specific bacterial taxa. Science. 349 (6250), 860-864 (2015).
- Ofek-Lalzar, M., et al. Niche and host-associated functional signatures of the root surface microbiome. Nature Commununications. 5, 4950 (2014).
- Niu, B., Paulson, J. N., Zheng, X., Kolter, R. Simplified and representative bacterial community of maize roots. Proceedings of the National Academy of Science of the United States of America. 114 (12), E2450-E2459 (2017).
- Fitzpatrick, C. R., et al. Assembly and ecological function of the root microbiome across angiosperm plant species. Proceedings of the National Academy of Science of the United States of America. 115 (6), E1157-E1165 (2018).
- Ramirez, K. S., Lauber, C. L., Knight, R., Bradford, M. A., Fierer, N. Consistent effects of nitrogen fertilization on soil bacterial communities in contrasting systems. Ecology. 91 (12), 3463-3470 (2010).
- Paungfoo-Lonhienne, C., et al. Turning the table: Plants consume microbes as a source of nutrients. PLoS One. 5 (7), e11915 (2010).
- Yeoh, Y. K., et al. The core root microbiome of sugarcanes cultivated under varying nitrogen fertilizer application. Environmental Microbiology. 18 (5), 1338-1351 (2016).
- Bulgarelli, D., Schlaeppi, K., Spaepen, S., Ver Loren van Themaat, E., Schulze-Lefert, P. Structure and functions of the bacterial microbiota of plants. Annual Review of Plant Biology. 64, 807-838 (2013).
- Peiffer, J. A., et al. Diversity and heritability of the maize rhizosphere microbiome under field conditions. Proceedings of the National Academy of Science of the United States of America. 110 (16), 6548-6553 (2013).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유