Method Article
Изоляция и анализ микробных сообществ в почве, ризосфере и корни в экспериментах многолетние травы
В этой статье
Резюме
Раскопки корни растений от поля, а также обработки образцов в ризосфере, endosphere и почвы описаны в деталях, включая ДНК извлечения и анализа методов данных. Этот документ предназначен для включения других лабораторий использовать эти методы для исследования почвы, endosphere и microbiomes ризосфере.
Аннотация
Микрофлора исследования растений и почвы становятся все более важными для понимания роли микроорганизмов счетчик производительности в сельском хозяйстве. Эта рукопись предназначен для предоставления сведений о том, как быстро образец почвы, ризосфере и endosphere реплицированных полевых испытаний и анализировать изменения, которые могут произойти в микробных сообществ из-за образец типа, лечение и генотип растений. Эксперимент, используемый для демонстрации этих методов состоит из участков реплицированных поля, содержащие два, чистый, теплый сезон травы (просо заострённая и Бородач Жерара) и низким разнообразие травосмеси (A. Жерара, Sorghastrum поникший, и Bouteloua curtipendula). Вкратце раскопки растений, различные корни вырезать и помещаются в фосфатного буфера и затем потряс собирать ризосфере. Корни были привлечены в лабораторию на льду и поверхности, стерилизованное с отбеливателем и этанола (EtOH). Ризосфере фильтруется и сосредоточены центрифугированием. Выемки грунта вокруг корневого кома из помещаются в пластиковые мешки и принес в лабораторию, где небольшое количество почвы берется для экстракции ДНК. ДНК извлекается из корней, почвы и ризосфере и затем усиливается с праймерами для региона V4 гена 16S рРНК. Ампликонов последовательности, а затем проанализированы с открытым доступом Биоинформатика инструменты. Эти методы позволяют исследователям для тестирования, как многообразие микробных и состав изменяется из-за образец типа, лечение и завод генотипа. Используя эти методы, а также статистические модели, представитель результаты показывают, что существуют значительные различия в микробных сообществ корни, ризосфере и почвы. Представленные здесь методы предоставляют полный набор шагов для как собирать образцы поле, изолировать, извлекать, количественно, усиливают и последовательности ДНК и анализировать многообразие микробных и композиции в реплицированной полевых испытаний.
Введение
Микрофлора исследования имеет важные последствия для понимания и манипулирования экосистемных процессах таких питательных Велоспорт, оборот органического вещества и разработки или ингибирование почву патогенов1,2. Эта область исследований также имеет большой потенциал для понимания воздействия микробов почвы на продуктивность естественных растительных сообществ и агроэкосистем. Хотя существует множество исследований, которые были сосредоточены на почве микрофлора в естественных экосистемах, меньше сосредоточились на завод ризосфере и endosphere микробы в агроэкосистемах3. В штате Небраска сельское хозяйство доминирует пейзаж через большие части государства, делая исследования этих почв, где аграрно важные культуры выращиваются жизненно важной темой для исследования. Цель этой методы документа — предоставить стандартный набор протоколов для описания микробов, присутствующих в агроэкосистемах, чтобы определить, как корни растений изменить микробных сообществ в ризосфере и endosphere и в конечном итоге исследователи понимаете функции, которую эти микробы играть в производительности растений и здоровья почвы.
Метод здесь представлены незначительно отличается от методов, используемых другими5 4,в, что этот документ направлен на изучение которых микробы являются исключительно внутри корень и как они отличаются от микробов сразу за пределами корень в ризосфере. Ампликон последовательности, используемое в настоящем исследовании идентифицирует микробной таксонов в образец ДНК и позволяет детективов, чтобы определить, как общины меняется в зависимости от типа образца или лечения. Одним из ключевых различий между этим протоколом и очень подобный протокол, используемый в Лундберг и др. 6 , что вместо sonication, этот протокол использует поверхности стерилизации с отбеливателем и этанола для удаления ризосфере от корней. Другие также использовали поверхности стерилизации эффективно7,8,9,10. Эти методы не являются более выгодным, чем другие методы, но немного отличается. Эти методы являются особенно хорошо подходит для крупных полевых экспериментов, потому что с достаточно людей можно обрабатывать более 150 поле сюжетов в день, который добавляет до приблизительно 450 образцов когда разделена на endosphere, ризосфере и почвы. Эта рукопись подробно описывает методы, используемые для образца в поле, обработки материала в лаборатории, извлекать и последовательности ДНК и предоставляет краткий обзор шагов для анализа полученных данных последовательности.
протокол
1. поле Описание сайта
- Опишите опытное поле сайтов в коллекции периоды. Определить местоположение поля (Широта, Долгота и высота над уровнем моря) с использованием GPS.
- Опишите время отбора проб, почвы текстуры и глубины выборки.
- Экологические факторы играют важную роль в формировании микробных сообществ. Рекордное климатической информации как годовая средняя температура, годовое количество осадков, предыдущие годы севооборот, практики обработки, метод оплодотворения и истории на сайте поля. Автоматических метеорологических станций или другие устройства полезны для записи суточное количество осадков и температур за вегетационный период.
2. сбор и обработка почвы, ризосфере и корневой области образцов
- Раскопки растений.
- Ярлык мыть кастрюлю и ведро с записки, содержащие информацию о растительного материала для выборки. Включают такие сведения, как номер участка, генотип растений и видов растений. Нести помечены ведро на участок и оставить сковороду мыть на установленной станции в поле.
- Проколоть почву с лопатой на глубину до 30 см, чтобы вырезать любой из боковых корней, держа растение в почве. Приблизительный объем составляет 18 см3. Случайным образом выбрать и собирать два растения на участок из различных областей в пределах участка.
- Выкопать корни растений, используя лопату и корневого кома в ведро с метками. Принесите помечены ведро с экскаватором корневого кома обратно к рабочей станции в поле. Cut off и отбросить биомассы надземной растений.
- Удаление почвы коллекции массовых почвы и корни.
- Встряхните корни, чтобы вручную удалить почвы или используйте лопату или ручной культиватор для удаления почвы от корней. Покачивая корни достаточно удалить почвы очень песчаных почвах. Надевайте перчатки и место корни станции обработки.
- После встряхивания корни, основная часть почвы будет мыть кастрюлю. Смешать почвы в сковороду мыть и разрушить любой комья почвы с ручной культиватор. Поместите образец почвы, мусора в маркировку, 17,7 x 19,5 см молнию мешок хранения и место в прохладном месте или на льду.
- Сбор корней и ризосфере.
- С помощью обрезка ножницы, стерилизовать в 70% EtOH, акцизный различные корни, примерно 4-6 корни на растении и каждого корня около 9-12 см в длину. Место подакцизным корни (резка при необходимости подогнать) в обозначенные 50 мл трубки, содержащий 35 мл газобетона, фосфатного буфера (6,33 г/Л NaH2PO4, 8,5 г/Л Na2HPO4 безводный, рН = 6,5, 200 мкл/Л ПАВ).
Примечание: ПАВ (см. Таблицу материалы) был добавлен после автоклавирования фосфатного буфера. Объем корневого кома и длины корня варьируются в зависимости от возраста растений и видов растений. - Встряхните трубы для 2 мин выпустить ризосфере от поверхности корней. Пинцетом, стерилизовать в 70% EtOH, удалить корни из трубки, блот кратко на бумажные полотенца и место в новой, помечены Тюбик 50 мл. Место как трубка, содержащая ризосфере и один, содержащий корни на льду.
- С помощью обрезка ножницы, стерилизовать в 70% EtOH, акцизный различные корни, примерно 4-6 корни на растении и каждого корня около 9-12 см в длину. Место подакцизным корни (резка при необходимости подогнать) в обозначенные 50 мл трубки, содержащий 35 мл газобетона, фосфатного буфера (6,33 г/Л NaH2PO4, 8,5 г/Л Na2HPO4 безводный, рН = 6,5, 200 мкл/Л ПАВ).
3. обработка поля образцов в лаборатории
- Поверхности стерилизации корней после коллекцию полей.
Примечание: Выполнение поверхности стерилизации как можно скорее после возвращения из поля. Если это не возможно поверхности стерилизовать корни в тот же день, хранить корни на 4 ° C до обработки.- Добавьте примерно 35 мл 50% хлорки + 0.01% анимации от 20 до 50 мл трубки корень собранные на местах. Shake 50 мл трубки для 30-60 секунд. Слить 50% отбеливатель и добавить 35 мл 70% EtOH. Трясти еще 30-60 секунд.
- Слить 70% EtOH и добавить 35 мл стерильного, ультрачистая вода. Трясти за 1 минуту. Повторите еще два раза мыть водой.
Примечание: Для обеспечения нашего поверхности стерилизации лечения достаточно, мы покрытием образцы воды из последнего полоскания и отмечено никакого роста бактерий (P. Wang, неопубликованные данные, 2014). Другие исследователи протестировали для корня эффективность стерилизации с помощью аналогичных методов9,10. - Помарки сухой корни на чистой бумажные полотенца. Используйте чистое бумажное полотенце для каждого образца.
Примечание: Бумажные полотенца можно стерилизовать перед использованием. Мы не стерилизуйте наши бумажные полотенца. Однако мы используем 1 бумажное полотенце на сэмпл и держать полотенца, завернутый пока не используется. - С помощью обрезка ножницы, нарезать корни примерно 5 мм штук и место и стерильный пинцет отрезать корни в чистой, обозначенные 15 мл Конические трубки. Хранить образцы на-80 ° C до дальнейшей обработки.
- Обработка образцов ризосфере.
- Встряхните 50 мл пробирки, содержащие ризосфере образцы от поля до Ресуспензируйте весь пример. С помощью стрейнер стерильные, 100-мкм сетка клетки (см. Таблицу материалы), фильтр ресуспензированы образца в новый Тюбик 50 мл.
- Центрифуга для трубы на 3000 x g 5 мин при комнатной температуре. Немедленно слить и удалить супернатант.
- Место ризосфере окатышей в 50 мл трубки на льду. Добавьте 1,5 мл стерильного фосфатного буфера (без ПАВ) в ризосфере окатышей и вихревой приостановить.
- Накапайте приостановлено жидкость в чистую, помечены, 2-мл пробирку отцентрифугировать. Спин трубы на 15,871 x g на 2 мин при комнатной температуре. Немедленно слить супернатант и канализация трубы на чистой бумажные полотенца.
- Храните гранул при-20 ° C до дальнейшей обработки.
Примечание: Чтобы уменьшить размер образца трубки для хранения сделали шаги 3.2.2 - 3.2.4. Это больше пространства эффективно хранить меньше 2 мл пробирок, по сравнению с 50 мл трубки.
- Обработка образцов почвы для извлечения и почвы анализа ДНК.
- С помощью стерильных металлической лопаткой, заполните чистой, обозначенные 2-мл пробирку с приблизительно 3 g почвы для экстракции ДНК. Избегайте любой небольшой корневой части и мусора. Храните этот образец грунта при-20 ° C. Промойте металлический шпатель в 70% EtOH между выборками.
- В чистой мыть кастрюлю, пустой мешок из почвы в стеке сита (см. Таблицу материалы), больше сито поверх меньше сито и вручную сита почвы через оба сита. Используйте кисть, чтобы тщательно очистить сита между выборками.
- Отложите 100-125 г, котор фильтруют почвы в 17,7 x 19,5 см молнии мешок для будущих почвы физико-химических и анализа текстуры. Место мешки почвы на 4 ° C для краткосрочного хранения.
- Обработка образцов почвы для влажности почвы.
- Масса тары помечены коричневый бумажный мешок в масштабе. Мера 40-45 г, котор фильтруют почвы в коричневый бумажный мешок. Запишите вес коричневый бумажный мешок и почвы на листе данных и место мешки в сушильной печи набор до 55-60 ° C.
- После 72 часов удалите мешки из печи. Разрешить мешки почвы остыть в течение минимум 30 минут и затем взвесить.
- Для записи вес, тарные масштаба до нуля и место коричневые мешки на шкале. Рассчитайте процент влажности почвы для каждого образца, используя формулу:
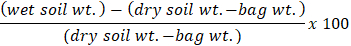
Примечание: Метод влажности почвы — от Келлогг биологических станции длинный срок экологических исследований (KBS фильтр). KBS фильтр обеспечивает широкий спектр установленных протоколов для исследователей на их веб-сайте (https://Lter.kbs.msu.edu/).
4. Подготовка обработанных корня образцов для экстракции ДНК.
- Молоть замороженные корень материал так, что образец является однородной.
- Залейте жидкий азот в пластиковый стакан с чистой шпатели и чистой ступку и пестик, чтобы сохранить образцы, заморожены на протяжении всего процесса шлифовальные.
- Замороженные ткани в ступке и растереть с пестиком в мелкий порошок. Постоянно добавьте жидкий азот всей шлифовальные хранить Замороженные пробы.
- Место местах ткани в чистой, обозначенные 2-мл пробирку лопаточкой. Хранить при температуре-80 ° C.
5. Добыча ДНК из почвы и ризосфере образцов в 96-луночных формате
- Загрузка образцов почвы в 96-луночных пластины.
- Протрите области работы с EtOH и 1% отбеливатель 70%. Надевайте перчатки лаборатории во время этих шагов. Удаление образцов почвы от-20 ° C для хранения и позволяют таять в ведёрке со льдом.
- Удалите крышку уплотнения мат из 96-луночных извлечение пластины, который поставляется с комплектом экстракции ДНК. Поместите крышку уплотнения мат между 2 бумажные салфетки в чистоте не во время использования. Чтобы избежать заражения, использовать клей 8-Ну ПЦР полосы для покрытия 12 столбцов 96-луночных извлечение пластины.
- Тарные стерильные весят воронка (размер см) в масштабе и весят, 200-250 мг почвы.
Примечание: Эти стерильных воронки выравниваются на одной стороне, поэтому воронку лежит плашмя на шкале. Воронка заполняется с почвой и помещен непосредственно в колодец. Этот метод позволяет избежать потери образцов, минимизирует утечки и предотвращает перекрестное загрязнение. - Чтобы раскрыть первую скважину извлечение пластины, осторожно поднимите клейкой лентой, место шеи заполненные весят воронка в соответствующий хорошо и мягко руководство образца грунта в соответствующие колодец. Замените клейкой лентой для покрытия хорошо.
- Повторите этот процесс для каждой скважины пластину, используя новый, стерильные воронку для каждого образца, до тех пор, пока пластины заполняется. Оставьте один хорошо пустым, как добыча пустой элемент управления.
Примечание: Один хорошо оставлено пустым на каждый пластины извлечения в качестве отрицательного контроля (пустой). Это контролирует для загрязнителей, которые могут присутствовать в комплект реагентов11. - Заменить крышку уплотнения мат на пластину добычи и хранить пластины при-20 ° C до готовности для экстракции ДНК.
- Загрузка образцов ризосфере в 96-луночных пластины.
- Удалите образцы ризосфере от-20 ° C для хранения и позволяют таять в ведёрке со льдом.
- Выполните шаг 5.1.2 выше подготовить пластину экстракции ДНК.
- Место протрите чистой бумаги в масштабе, а затем тары стерильные металлической лопаткой по шкале. Используйте шпатель для тщательно вырезать некоторые из ризосфере гранулы из образца трубки. Возвращение лопатку к шкале и весят между 200 и 250 мг ризосфере образца.
- Аккуратно поднимите клейкой лентой, чтобы раскрыть первую скважину пластины извлечения, угол заполненные шпателем в колодец и соскрести ризосфере материала в соответствующих колодец с стерильных зубочисткой.
- Промойте металлический шпатель в воде, после чего 70% EtOH между выборками. Повторите этот процесс для каждой скважины пластины до тех пор, пока пластины заполняется, оставив один хорошо пустой, как добыча пустой элемент управления.
- Извлечения ДНК из образцов ризосфере и почвы в 96-луночных формате.
- Экстракт почвы и ризосфере ДНК с помощью комплекта оптимизирован для почвы (см. Таблицу материалы), производителя протоколом.
Примечание: Мы используем этот конкретный набор для изоляции грунта и ризосфере ДНК из-за способности несвободных реагентов для удаления гуминовых кислот и других мощных ингибиторы ПЦР в почве.
- Экстракт почвы и ризосфере ДНК с помощью комплекта оптимизирован для почвы (см. Таблицу материалы), производителя протоколом.
- Количественное определение ДНК.
- Количественно 92 образцов и 4 концентрации стандартов с использованием комплекта (см. Таблицу материалов; стандартов включены), согласно протоколу от производителя.
- Количественно оставшиеся 4 образцы, которые были удалены из пластины для размещения скважин для четырех стандартов с использованием комплекта (см. Таблицу материалы), по словам производителя протокол.
6. экстракции ДНК из корня образцов в 96-луночных формате.
- Загрузка образцов корень в 96-луночных пластины.
- Протрите области работы с EtOH и 1% отбеливатель 70%. Надевайте перчатки во время этих шагов.
- Держите землю корень образцы заморожены на все времена, поместив образцов в ведро с сухим льдом.
- Заполнить пластиковый стакан с жидким азотом и антистатические microspatulas и стерильные весят воронки (размер XSM) в пластиковый стакан остыть.
- Удалите крышку уплотнения мат от пластины шарик 96-луночных экстракции, который поставляется с комплектом и поместите его между 2 бумажные салфетки в чистоте не во время использования.
Примечание: Новая версия этого комплекта, выпущенный после времени, мы провели эти экстракции ДНК, требует от пользователя на поставку пластин извлечение шарик. В Таблица материаловмы перечислили сведений о каталоге поставщика, необходимо заказать эти элементы. - Чтобы избежать заражения, использовать клей 8-Ну ПЦР полосы для покрытия 12 столбцов 96-луночных извлечение пластины.
- Место пластину из бисера добычи на сухой лед сохранить образцы в скважинах заморожены.
- Осторожно поднимите клейкой лентой раскрыть первую скважину извлечение пластины, поместите шеи заполненные весят воронка в соответствующие хорошо и добавить 3 Мастихин Совки земли корень ткани. Замените клейкой лентой для покрытия хорошо.
Примечание: Почвы и ризосфере являются талой на льду до взвешивания, тогда как растительный материал отвесил замороженные. Замороженные растительной ткани, особенно небольших сумм, трудно весить масштабе без оттаивания. Весят испытания были проведены на землю корень ткани для определения, сколько порций шпатель были достаточно. Обратите внимание, что производитель комплекта не требуется точное количество ткани, но рекомендует около 50 мг. видов различных растений образца будет меняться, и пользователю будет необходимо определить соответствующую сумму. - Повторите этот процесс для каждой скважины пластины до тех пор, пока пластины заполняется.
- Хранить пластины при-20 ° C до готовности для экстракции ДНК.
- Экстракции ДНК тканей корня.
- Извлечь ДНК с помощью комплекта, оптимизированный для растений (см. Таблицу материалы) после производителя протокол.
Примечание: Мы используем этот комплект, который предназначен специально для тканей растений для достижения максимальной урожайности от корневого образцов. В отличие от почвы и ризосфере гуминовые кислоты и другие загрязнители являются меньше проблем для тканей корня.
- Извлечь ДНК с помощью комплекта, оптимизированный для растений (см. Таблицу материалы) после производителя протокол.
- Количественного определения ДНК как шаг 5.4.
7. усиление и секвенирование изолированных ДНК.
- Усилить регионе V4 гена 16S с доказательство чтения полимеразы (см. Таблицу материалы), как описано в Голь и др. 12 штрих образцы с различными индексации грунты и бассейн перед последовательности. Последовательность, используя методы, описанные в Голь et al12 с помощью ПЦР в два этапа с праймерами V4 (дополнительный файл 1).
Примечание: Некоторые из методов для эти шаги описаны в других подробно12,13,14 и поэтому будут не описаны здесь. Для корневого образцов ПНА блокаторы были добавлены для уменьшения количества ДНК пластид, усиливается от растительной ткани, который ранее был полностью описано15. В последовательности, отрицательный контроль, которая включала извлечение пластины пустых элементов управления (см. Примечание после шага 5.1.5), использовались два элемента управления и макет сообщества известный населения бактериальной ДНК (см. Таблицу материалы), который служил как положительные управления.
Примечание: В большинстве случаев, v3 MiSeq наборов реагентов используются в режиме база парных конец 2 X 300. Для образцов в этой рукописи Illumina HiSeq 2500 был использован в быстрый режим с 250 сопряженные конец (2 x 250) режиме. Все образцы были упорядочены в той же полосе. - Процесс виртуализации данных через конвейер для анализа микробных (USEARCH v9.2.64, QIIME v1.9.1 и RStudio v3.4.3 16).
- Подготовка данных виртуализации с помощью USEARCH17.
Примечание: USEARCH доступен онлайн с подробными инструкциями (https://www.drive5.com/usearch/). - Демультиплексирования последовательности данных, с использованием индекса читает или штрих-кодов для назначения Illumina читает образцов.
- Слияние в паре конец читает получить консенсус последовательности. Используйте команду: usearch-fastq_mergepairs * R1*.fastq-переразметки @ - fastq_maxdiffs 10 - fastq_minmergelen 230 - fastq_maxmergelen 320 - fastq_pctid 80 - fastqout merged.fq.
Примечание: Параметры задаются через ссылки на USEARCH Инструкция по эксплуатации. - Удалите из последовательности данных, чтобы избежать замены в грунт последовательностей, которые могут быть вызваны реакции PCR праймеров. Используйте команду: usearch-fastx_truncate merge.fq - stripleft 19 - stripright 20 - fastqout stripped.fq.
- Фильтрация данных последовательности чтобы удалить низкого качества чтения и сохранить высокое качество оперативной таксономическая единица (ОТУ) последовательности. Используйте команду: usearch-fastq_filter stripped.fq-filtered.fa - fastaout fastq_maxee 1.0.
- Подготовка данных виртуализации с помощью USEARCH17.
- Генерировать OTUs в USEARCH.
- Выполните dereplication определить набор уникальных OTU последовательностей. Используйте команду: usearch-fastx_uniques filtered.fa - fastaout uniques.fa - sizeout-переименовали Uniq.
- OTUs кластера с 97 – 100% сходством последовательности назначить уникальный OTUs. Используйте команду: usearch-cluster_otus uniques.fa - minsize 2 - otus otus.fa-переименовали ОТУ.
Примечание: Этот шаг также включает в себя удаление одноэкземплярных классов из кластерных OTUs и удалением химер из последовательности данных. - Создайте таблицу OTU в USEARCH. Используйте команду: usearch-usearch_global stripped.fq - db otus.fa-нить плюс - id 0,97 - otutabout otutable.txt.
Примечание: Эта команда создает таблицу с количество считываний (СИГ) всех OTUs для каждого образца. OTU таблица используется для вниз по течению шаги, включая дифференциальные изобилие анализа и анализа микробного разнообразия. Дополнительные рисунке 2 приведен пример OTU таблицы. - Провести анализ разрежения в QIIME v1.9.1 18.
Примечание: Разрежения кривая рассчитывается с помощью OTU таблицы для определения ли глубина последовательности правильно образцы микробных. Пример разрежения кривых показаны Дополнительные рисунок3. - Провести анализ альфа-разнообразие18. Использование alpha_rarefaction.py в QIIME v1.9.1 для расчета разнообразие микробного сообщества в рамках каждого образца.
Примечание: Этот анализ рассчитывает индексы разнообразия Shannon19, Симпсон20и21Chao1. - Провести бета разнообразия анализ18,22. Использовать сценарий Python: beta_diversity_through_plots.py в QIIME v1.9.1 Брей – Кертис несходство матрицы.
Примечание: Этот анализ сравнивает состав микробных сообществ между выборками. - Проводить статистический анализ между группами. Использование матрицы расстояния, рассчитанные для PERMONOVA, используя функцию Адониса и дисперсионного анализа в Веганские пакет23 v2.4.5 в RStudio16. Канонический анализ анализа основных координаты (CAP), используя функцию capscale в пакете строгого вегетарианца. Визуализация данных с помощью ggplot2 пакет24 v2.2.1 в RStudio.
Результаты
Представитель результаты, представленные в этой рукописи из области сайта, создан в университете штата Небраска-Линкольн сельского хозяйства исследований отдела ферме возле МИД в 2012, NE. перед эксперимента, сайт осуществлялось как кукуруза соя вращение . Исследование сайт был расположен на трех различных типов почв, но данные были проанализированы как будто все изменения в свойствах измерений почвы из-за введенных лечения.
На сайте поле содержатся два, чистый, стоит switchgrass (P. заострённая cv свободы) и большой bluestem (A. Жерара), а также низким разнообразие травы смесь, содержащую большой bluestem, indiangrass (S. поникший) и «Butte» sideoats Грама ( B. curtipendula). Три теплый сезон травы участки находились в рандомизированных полный блок дизайн, который был воспроизведен в три раза. Вложенные в трех различных участков были два лечения удобрения азота (N), которые были 56 (N1) и 112 (N2) кг N га-1 прикладной мочевины. В то время микрофлора выборки в конце сезона выращивания, нитрата ppm (среднее ± SD) 8.0 ± 1.1 почвы содержащихся в участки оплодотворенной с 112 кг N га-1 и 6,8 ± 0,7 (mean + SD) нитрат ppm в участках удобренный с 56 кг N га-1. Участки удобряла раз в год. Теплый сезон травы, которую участки были выделены в качестве основных участков (8000 m2) и N лечения были участки Сплит (4000 m2). Большой bluestem был посеян как смесь 50: 50 «Bonanza» и «Золотой жилой» и Indiangrass был посеян как смесь 50: 50 «Разведчик» и «Воин». Участки были посажены в 2012 году, и первое приложение N произошло весной 2013 года.
Почвы и корня выборки была проведена 15 сентября 2014 года. Работа, описанная ниже было проведено на поле, который был создан как Сплит участок рандомизированных дизайна с тремя реплицирует (рис. 1). Среднее последовательности глубины все образцы для endosphere были следующие: 4871 ± 5711 (среднее ± SD), ризосфере: 40726 ± 14684, почвы: 38184 ± 9043. Одним из крупнейших источников вариации в этих экспериментах, используя методы, описанные, является разница в микробных сообществ между типами образца (рис. 2). В этот представительный набор данных ризосфере и почвы, как представляется, будет больше похожи в составе друг к другу, чем endosphere (рисунок 2A). Однако, было также весьма значительные (p = 0,001) различия в состав микробных сообществ ризосфере и почвы (рис. 2B). Общая вариации, приходилось в этих экспериментах, анализируемой образец типа был 26%.
Альфа-разнообразие анализ показал, что микробных сообществ в endosphere были ниже в образце разнообразия по сравнению с почвы и ризосферной (рис. 3). Только существенные различия в разнообразии между трава зверобоя в любом отделении были между образцы endosphere большой bluestem и switchgrass, с switchgrass, имея значительно выше разнообразия видов микроорганизмов (рис. 3). Анализ относительного изобилия (рис. 4) подчеркивает преобладание бактерии по алфавиту следуют актиномицеты во всех типах образца. Почвы и ризосфере также преобладают Acidobacteria и Chloroflexi в то время как endosphere был больше относительное обилие Bacteriodetes.
В этом эксперименте растения выращивались с двумя различное количество N-удобрения, и поэтому мы проанализировали данные, чтобы определить, имеются ли какие-либо эффекты лечения. Эффекты лечения приходилось 12% общей вариации, но не существенно отличались хотя в координации две процедуры выглядят разные (рис. 5). Это подчеркивает важность статистических анализов для этих наборов данных вместо визуального осмотра или качественных решений.
Завод влияние различий в микрофлора тканей растений и почвы были визуализируется с помощью ограниченного метода рукоположения. Статистические различия были определены с помощью ПЕРМАНОВА анализа для проверки ли конкретные переменные, такие как видов, результат в состав существенно различных микробных сообществ между выборками. Когда все типы образцов были проанализированы вместе, очень существенное различие было обнаружено в состав микробных сообществ благодаря видов растений (рис. 6). В этом эксперименте количество вариантов, приходится на виды растений был 6,7%. Наконец каждый образец типа был проанализирован индивидуально определить, какой из типов образца могут быть движущей эффект видов значительное завод. Только в endosphere был там очень существенное различие (p = 0,001) между микробных композиций различных видов растений (рис. 7). В других типах образца видов эффект не был значительным, когда анализируются индивидуально. В endosphere процент вариации из-за вида был 27%, в то время как он был ниже в ризосфере (18%) и почвы (15%). Это далее подчеркивает важность анализа каждого типа ткани индивидуально.
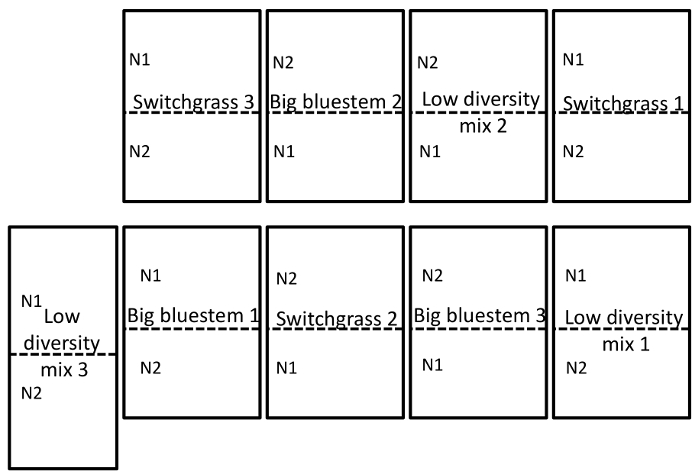
Рисунок 1: пример опытное поле дизайн. Опытное поле дизайн, иллюстрирующие рандомизированных полный блок дизайн в трех экземплярах области сайта, расположенный в университете штата Небраска-Линкольн, Восточной Небраска исследований и расширение центра вблизи мидуса, NE. Полное описание в разделе результаты. N1 является низкой (56 кг N га мочевина-1 ) и N2 (112 кг N га мочевина-1 ) является уровень азота, который был применен. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
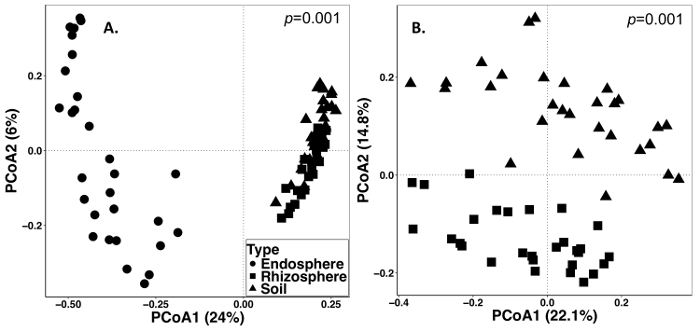
Рисунок 2: анализ разнообразия бета, сравнивая микробный состав различных образцов типов, включая endosphere, ризосфере и почвы из выборки многолетние травы в 2014 году. Анализ был проведен с использованием сценария Python в QIIME1.9.1 для производства Bray-Кертис несходство матрицы. Основные координаты анализ (PCoA), основанный на матрице несходство Брей-Кертис был визуализирован в RStudio. PCoA1 и PCoA2 указывают на первый и второй по величине дисперсии, объясняется PCoA анализа. ПЕРМАНОВА статистический анализ проводился для определения значимости между типами образца, и p значение отображается в верхнем правом углу. Каждый символ в цифры представляют собой сообщество микроорганизмов для каждого образца. (A) Endosphere, ризосфере и образец типов почвы были проанализированы вместе. Все 87 образцы были разрежена 486 последовательностей на сэмпл. (B) образцы ризосфере и почвы были проанализированы вместе. Все 59 образцы были разрежена 8231 последовательностями. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
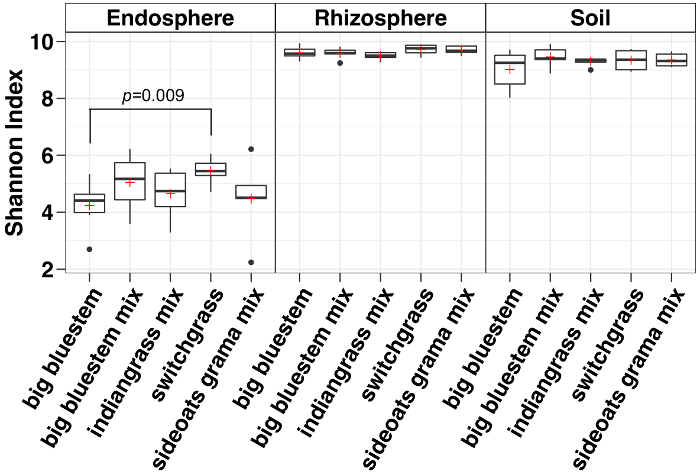
Рисунок 3: альфа разнообразия анализ с использованием индекса Шеннона для каждого вида в ризосфере и почвы и endosphere. Анализ был проведен с использованием сценария Python в QIIME1.9.1. Разрежения было сделано для endosphere, ризосфере и типов почвы образца соответственно с 486, 17154 и 8231 последовательности на сэмпл. Прямоугольники указывают 25 и 75-й процентили (первый и третий квартили). Горизонтальная линия в поле обозначает средний и красный плюс показывает среднее. Усы показывают диапазон данных, исключая выбросы (которые отображаются в виде черных точек), которые упали более чем в 1,5 раза межквартильный диапазон (n = 6 для каждого образца, за исключением для sideoats Грама смесь где n = 5). Шеннон индекс всех пяти видов в endosphere были ниже, чем ризосфере и почвы. Non параметрический Вилкоксон ранга сумма тест был использован для определения значимости между видами и только существенные различия между видами были показаны на верху коробки. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
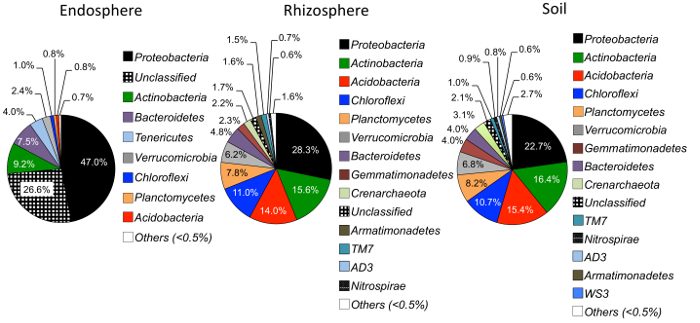
Рисунок 4: относительное изобилие Фила в на уровне почвы endosphere и ризосфере. Были проанализированы образцы для сравнения обилие микробной типы среди различных образцов типов (n = 29 для каждого образца типа). Анализ был проведен с использованием сценария Python в QIIME1.9.1 из OTU таблицы. Различные цвета внутри круговой диаграммы обозначают типы. Процент указывает относительное обилие каждый тип в тип каждого образца. Тип информации была аннотированный с использованием классификатора (RDP) рибосомной проекта базы данных25. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
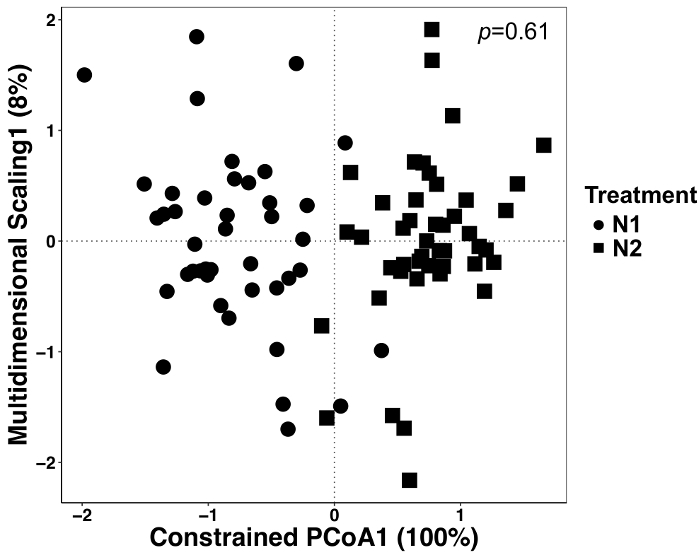
Рисунок 5: анализ с помощью лечения как сдерживающий фактор между всеми типами образца. Определить, существуют ли различия в состав микробных сообществ между процедурами был проведен канонический анализ анализа основных координаты (CAP). Для каждого лечения N, n = 42 для N1 (56 кг N га-1) и n = 45 для N2 (112 кг N га-1). Матрица несходство Брей-Кертис был создан с помощью скрипта python в QIIME1.9.1. CAP анализ, основанный на матрице несходство Брей-Кертис было сделано путем ограничения лечения как фактор в RStudio. Чтобы определить ли лечение различия значительны, и на правом верхнем углу отображается значение p был проведен ПЕРМАНОВА анализ. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
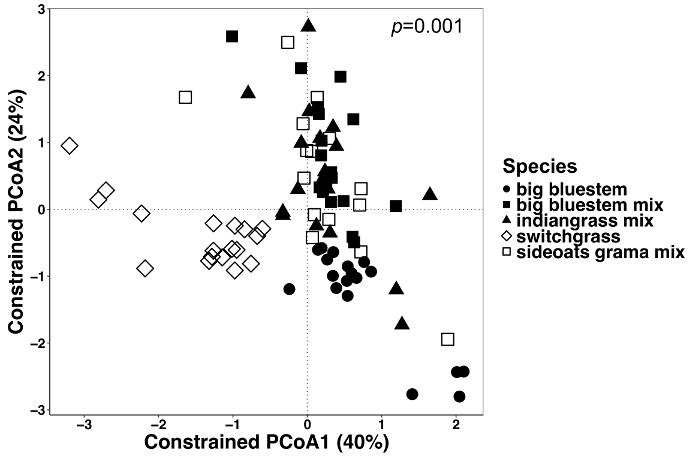
Рисунок 6: анализ с использованием видов растений как сдерживающий фактор между всеми типами образца. Чтобы определить, существуют ли различия в состав микробных между видов растений во всех типах образец был проведен анализ. Основные координаты рукоположения и Кап анализ всех типов образцов (endosphere, ризосфере и почвы) были сделаны с помощью матрицы несходство Брей-Кертис. Матрица несходство Брей-Кертис был создан с помощью скрипта Python в QIIME1.9.1. CAP анализ, основанный на матрице несходство Брей-Кертис было сделано путем ограничения видов растений как фактор в RStudio. ПЕРМАНОВА статистический анализ проводился для определения значимости между видов растений, и P значение отображается в верхнем правом углу. Каждый символ в цифрах представляет сообщество микроорганизмов для этого образца. n = 18 для каждого вида во всех типах образца за исключением n = 15 для sideoats Грама микс. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
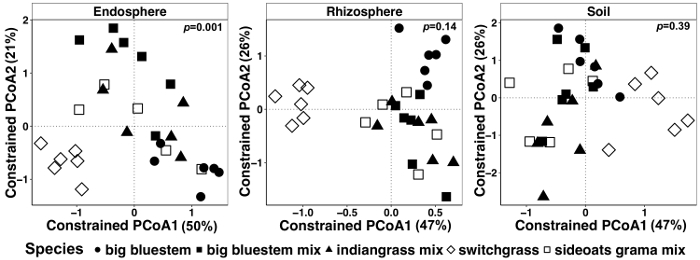
Рисунок 7: пример CAP анализа с использованием видов как сдерживающий фактор для каждого образца типа индивидуально. Основные координат координацию и Кап анализ каждого образца типа (endosphere, ризосфере и почвы) с помощью матрицы несходство Брей-Кертис. Каждый образец типа был разрежена 486, 17154 и 8231 чтений на сэмпл соответственно в endosphere, ризосфере и почвы. Вид был использован как фактор затрудняет координацию. ПЕРМАНОВА статистический анализ проводился для определения значимости между видов растений в каждом образце тип, и значение p отображается в верхнем правом углу. Каждый символ на рисунке представляет сообщество микроорганизмов для каждого образца. Это размер выборки n = 29 для каждого типа образца, n = 6 для каждого вида растений в каждый образец типа за исключением Грама смесь sideoats (n = 5). Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Обсуждение
Методы, описанные в этой рукописи должны позволить ученых легко ввести поле метагеномики почвы и растений. С годами мы уточнили наши методы после проведения эксперимента, описанные в этой рукописи. Одно из изменений является, что мы сейчас предварительно метки трубы перед выходом в поле образец. Наша лаборатория использует систему штрихкодирования и принтер этикеток. Принтер этикеток не только экономит время, когда маркировки труб, но и делает все проще отслеживать и правильно определить образцы без капризов человека почерк. Другой критической точки является, что мы пытаемся обработки материала, после чего он вернулся из поля как можно скорее. Мы стремимся заморозить почва, используемая для анализа ДНК, стерилизовать и заморозить корни и фильтровать и заморозить ризосфере 12 до 36 часов после возвращения из поля. Процедуры экстракции ДНК длительных с много шагов, особенно для почвы и ризосфере, поэтому мы купили робот (Kingfisher Flex, ThermoFisher), который минимизирует руки на время протоколы извлечения дна, уменьшает человеческой ошибки, которые могут быть введены, и улучшает последовательность в пути разные пакеты почвы, корни или ризосфере обрабатываются. При работе с материалом растений важно решить в корневом типе изучаться или принимать различные типы корневых получить «репрезентативной выборки». Корни и листья в замороженном состоянии при проведении экстракции ДНК имеет важное значение, как обеспечить есть нет перекрестного загрязнения между образцами при заполнении пластины извлечения ДНК 96-луночных. Еще один важный фактор, чтобы рассмотреть это количество реплицирует использоваться при проектировании полевых экспериментов и используя полный рандомизированных дизайна там, где это возможно26. Из-за изменчивости высокой поля может быть необходимо иметь большое количество реплицирует обнаружить небольшие различия. Наконец из нашего опыта важно убедиться, что почвы не слишком мокрая, при раскопках корни. Если почва насыщена водой это не только запутанна, чтобы работать с, но это также очень трудно определить ризосфере и устранить почву от корней.
Одно изменение, которое было сделано на раннем этапе в ходе разработки этих методов было вместо пожимая трубки вручную, чтобы освободить ризосфере, которую мы повышен до vortexers, питание от газогенератора для облегчения работы в поле и более стандартизированы по образом, что каждая трубка был взволнован и время. Ограничение ампликон последовательности подхода состоит в том, что таксономический резолюции результаты зачастую ограничен и многие OTUs неизвестных или единственный известный на уровне семьи или рода. Это поле исследований быстро развивается, поэтому очень важно быть в курсе новых и развивающихся подходов, особенно для анализа данных, которые могут повысить разрешение результатов.
Эти протоколы являются только для изучения бактерий и архей, не грибов. Использование различных Праймеры для усиления позволит исследование грибковых общин с использованием же ДНК образцов27,28. Эти методы не требуют приобретения большого количества оборудования, потому что методы могут быть упрощены. Методы, мы опишем здесь в основном для определения «кто там», но поле быстро превращается в задавать важные вопросы о функции, которые могут решаться с помощью методов секвенирования ружье, изоляции и тестирование функциональных возможностей микробы, или последовательности всей микробных геномов.
Представитель результаты подчеркивают различия в микробных сообществ, которые могут быть определены с помощью методов, описанных. С помощью бета разнообразие подход к анализу данных22, были показаны композиционные отличия между типами образца. Эти различия были четко прослеживается в большинстве других исследований, где endosphere, ризосфере и почвы содержат уникальные микробных сообществ3. Шеннон разнообразию индекс рассчитывался определить изобилие и равномерность микробных видов в рамках каждого вида растений в endosphere, ризосфере и почвы. Как показано в этом исследовании и во многих других странах, альфа-разнообразие является самым высоким в почве, слегка уменьшаясь в ризосфере и затем значительно уменьшается в endosphere3,5,29. Эти результаты показывают, что описанные здесь методы подходят для определения изменения в endosphere, ризосфере и почвы.
Преобладание бактерии по алфавиту является общий вывод в исследованиях по31,30,endosphere и почвы32. Endosphere обычно имеет меньше разнообразие видов микроорганизмов с выше относительное обилие бактерии по алфавиту. Это вновь подчеркивает, что результаты здесь являются представитель других выводов в литературе. Эффекты лечения в этом исследовании не были значительно отличается и две основные причины для этого, возможно, что различия введенных лечения не достаточно большой, чтобы генерировать достаточно вариантов для обнаружения и что этой выборки было сделано в конце вегетационный период, когда поля могут имели достаточно времени, чтобы привлечь вниз азота аналогичного уровня, который является то, что было измерено в конце сезона. В другом исследовании с использованием аналогичных оплодотворение ставок в течение более длительного периода времени только сравнительно небольшие изменения в составе микрофлора были измеренных33. Другие исследования показали изменения в грибковых и бактериальных сообществ из-за азотных удобрений34,35.
Видов растений, как известно, играют роль в определении их microbiomes3,,3236 и даже небольшие различия в микробных вариации были продемонстрированы между генотипами различных растений в пределах один вид37. В этом исследовании было обнаружено значительной разницы в состав микробных сообществ между видов растений. Во всех типах образца оказалось что switchgrass наиболее выразительной микробный состав, но различия между видами были только статистически значимой в endosphere. Ризосфере сообщества композиция может стать значительным, если больше реплицирует были доступны для анализа.
Комбинированные области, лаборатории и аналитических протоколов, описанные здесь обеспечивают мощный метод для изучения как различные факторы влияния состава микробных сообществ в почвах и ризосфере, endosphere корни36. Существует много работы предстоит сделать в области изучения microbiomes, особенно в сельскохозяйственных полей. Важные вопросы о как урожайность изменяются, микрофлора почвы еще предстоит полностью выяснены. Даже самые основные вопросы, касающиеся как севооборотов влияние микрофлора почвы, как сроки изменяет микрофлора, как абиотического стресса изменяет микрофлора, как тип почвы взаимодействует с этими факторами изменить микрофлора и есть ли Универсальные микробов в отдельных культур или регионах США являются все открытые вопросы. Эти методы также будет полезным для эпидемиологических исследований для определения присутствия и сохранение патогенных и полезных бактерий. Еще один будущий горизонт для этих методов будет приступить к интеграции ДНК методы, описанные здесь с завода и Микроб РНК и метаболит данных. Дополнительные улучшения и тестирование более переменных будет иметь важное значение для дальнейшей оптимизации этих протоколов.
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Развитие этой рукописи поддерживается Национальный научный фонд EPSCoR центр для корня и Rhizobiome инновации премии ия-1557417. Сбор данных было поддержано средств от университета Небраска-Линкольн, сельскохозяйственных исследований и разработок и Люк грант от USDA. Мы также признаем поддержку от USDA-ARS и была оказана поддержка сельского хозяйства и продовольствия исследований инициатива конкурентных Грант № 2011-68005-30411 от USDA национального института продовольствия и сельского хозяйства, создавать и управлять этими полями.
Материалы
| Name | Company | Catalog Number | Comments |
| Dneasy PowerSoil HTP 96 Kit | Qiagen/MoBio | 12955-4 | Extraction kit for soil and rhizosphere |
| Dneasy PowerPlant HTP 96 Kit | Qiagen/MoBio | 13496-4 | Extraction kit for roots |
| D-Handle Digging shovel, 101 cm L | Fiskars | 9669 | |
| Rapid Tiller, 40 cm L | Truper | 34316 | |
| Ziploc Bags, 17.7 cm x 19.5 cm | Ziploc | NA | |
| Cooler | Any | NA | |
| Wash pan | Any | NA | |
| Plastic bucket | Any | NA | |
| Gloves (work and lab) | Any | NA | |
| 20 cm diameter Soil sieve #8, 2360 μm mesh size | Dual Manufacturing Co., Chicago IL | US8-8FS | |
| 20 cm diameter Soil Sieve #4, 4750 μm mesh size | Dual Manufacturing Co., Chicago IL | US8-4FS | |
| portable generator | Honda brand works well | NA | |
| Sterile cell strainers 100 μm mesh size | Fisher Scientific | 22-363-549 | |
| NaH2PO4·H2O | VWR | 0823 | |
| Na2HPO4 | VWR | 0404 | |
| Silwet L-77 | Lehman Seeds | VIS-30 | Surfactant |
| Autoclaves | Any | NA | |
| Drying Oven | Any | NA | |
| Scale | Any | Any | |
| Bleach | CLOROX - household strength | NA | |
| Tween 20 | Any | NA | |
| Liquid Nitrogen | Any | NA | |
| Dry Ice pellets | Any | NA | |
| Ethanol | Any | NA | |
| 11 cm precision fine point tweezers | Fisher | 17456209 | |
| 18 cm Straight point specimen forceps | VWR | 82027-436 | |
| 13.5 cm Pruning Scissors | Fiskars | 9921 | |
| 2 mL tube | Any | NA | |
| 15 mL PP conical tube | MIDSCI | C15B | |
| 50 mL PP conical tube | MIDSCI | C50B | |
| Ultrapure water | Millipore-sigma | Milli-Q Integral, Q-POD | |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32854 | For DNA quantification of removed samples |
| QuantiFluor dsDNA System | Promega | E2670 | For DNA quantification |
| 96-Well Black with Clear Flat Bottom Plates | Corning | 3631 | |
| pPNA PCR Blocker | PNA Bio | PP01-50 | |
| mPNA PCR Blocker | PNA Bio | MP01-50 | |
| Genomic DNA from Microbial Mock Community B (Even Low Concentration) v5.1L, for 16s rRNA Gene sequencing | BEI Resources | HM782D | |
| Adhesive 8 well-strips for plates | VWR | 89134-434 | |
| Stainless steel beads, 3.2 mm dia | Next Advance | SSB32 | |
| 1 ml Assay block (DNA extraction plate for the Qiagen/MoBio Dneasy PowerPlant HTP Kit) | CoStar | 3959 | |
| antistatic PP weighing funnel, size small for soil/rhizosphere | TWD Tradewinds, INC | ASWF1SPK | |
| antistatic PP weighing funnel, size x-small for root/leaf | TWD Tradewinds, INC | ASWFXSCS | |
| Genie 2 Digital Vortex | Scientific Industries | SI-0236 | |
| Vortex adapter for 50 mL tubes | Scientific Industries | SI-H506 | |
| Mortar (100 mL) and pestle | Any | NA | |
| Metal micro-spatula | VWR | 80071-672 | |
| Disposable antistatic microspatulas | VWR | 231-0106 | |
| Brown Paper bag 2# (10.95 cm x 6.19 cm x 20 cm) | Duro | 18402 | |
| 5424 Centrifuge for 2 mL tube | Eppendorf | 22620461 | |
| Centrifuge for 96-well plate | Sigma4-16S | 81510 | |
| Centrifuge rotor for 50 mL tubes | Sigma4-16S | 12269 - Biosafe | |
| KAPA HiFi DNA polymerase | Kapa Biosystems |
Ссылки
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: the microbial ecology of the rhizosphere. Nature Reviews Microbiology. 11, 789-799 (2013).
- Fierer, N. Embracing the unknown: disentangling the complexities of the soil microbiome. Nature Review Microbiology. 15 (10), 579-590 (2017).
- Wang, P., et al. Shifts in microbial communities in soil, rhizosphere and roots of two major crop systems under elevated CO2 and O3. Scientific Reports. 7 (1), 15019 (2017).
- White, L. J., Jothibasu, K., Reese, R. N., Brözel, V. S., Subramanian, S. Spatio Temporal influence of isoflavonoids on bacterial diversity in the soybean Rhizosphere. Molecular Plant-Microbe Interactions. 28 (1), 22-29 (2015).
- Edwards, J., et al. Structure, variation, and assembly of the root-associated microbiomes of rice. Proceedings of the National Academy of Science of the United States of America. 112 (8), E911-E920 (2015).
- Lundberg, D. S., et al. Defining the core Arabidopsis thaliana root microbiome. Nature. 488, 86-90 (2012).
- Bougoure, D. S., Cairney, J. W. Assemblages of ericoid mycorrhizal and other root-associated fungi from Epacris pulchella (Ericaceae) as determined by culturing and direct DNA extraction from roots. Environmental Microbiology. 7 (6), 819-827 (2005).
- Doty, S. L., et al. Diazotrophic endophytes of native black cottonwood and willow. Symbiosis. 47 (1), 23-33 (2009).
- Gottel, N. R., et al. Distinct microbial communities within the endosphere and rhizosphere of populus deltoides roots across contrasting soil types. Applied and Environmental Microbiology. 77 (17), 5934-5944 (2011).
- Xin, G., Glawe, D., Doty, S. L. Characterization of three endophytic, indole-3-acetic acid-producing yeasts occurring in Populus trees. Mycological Research. 113, 973-980 (2009).
- Salter, S. J., et al. Reagent and laboratory contamination can critically impact sequence-based microbiome analyses. BMC Biolology. 12, 87 (2014).
- Gohl, D. M., et al. Systematic improvement of amplicon marker gene methods for increased accuracy in microbiome studies. Nature Biotechnology. 34 (9), 942-949 (2016).
- Kozich, J. J., Westcott, S. L., Baxter, N. T., Highlander, S. K., Schloss, P. D. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform. Applied Environmental Microbiology. 79 (17), 5112-5120 (2013).
- Caporaso, J. G., et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms. ISME Journal. 6 (8), 1621-1624 (2012).
- Lundberg, D. S., Yourstone, S., Mieczkowski, P., Jones, C. D., Dangl, J. L. Practical innovations for high-throughput amplicon sequencing. Nature Methods. 10 (10), 999-1002 (2013).
- Edgar, R. C. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature Methods. 10 (10), 996-998 (2013).
- Caporaso, J. G., et al. QIIME allows analysis of high-throughput community sequencing data. Nature Methods. 7 (5), 335-336 (2010).
- Shannon, C. A mathematical theory of communication. Bell System Technical Journal. 27, 379-423 (1948).
- Simpson, E. H. Measurement of diversity. Nature. 163, 688 (1949).
- Chao, A. Non-parametric estimation of the number of classes in a population. Scandanavian Journal of Statistics. 11, 265-270 (1984).
- Legendre, P. Studying beta diversity: Ecological variation partitioning by multiple regression and canonical analysis. Journal of Plant Ecology. 1 (1), 3-8 (2008).
- Oksanen, J., et al. The vegan package. Community Ecology Package. 10, 631-637 (2007).
- Wickham, H. ggplot2: Elegant graphics for data analysis. Ggplot2: Elegant Graphics for Data Analysis. , 1-212 (2009).
- Cole, J. R., et al. The Ribosomal Database Project (RDP-II): sequences and tools for high-throughput rRNA analysis. Nucleic Acids Research. 33, D294-D296 (2006).
- Wagner, M. R., et al. Host genotype and age shape the leaf and root microbiomes of a wild perennial plant. Nature Communications. 7, 12151 (2016).
- O'Brien, H. E., Parrent, J. L., Jackson, J. A., Moncalvo, J. M., Vilgalys, R. Fungal community analysis by large-scale sequencing of environmental samples. Applied and Environmental Microbiology. 71 (9), 5544-5550 (2005).
- Taylor, D. L., et al. A first comprehensive census of fungi in soil reveals both hyperdiversity and fine-scale niche partitioning. Ecological Monographs. 84 (1), 3-20 (2014).
- Lebeis, S. L., et al. Salicylic acid modulates colonization of the root microbiome by specific bacterial taxa. Science. 349 (6250), 860-864 (2015).
- Ofek-Lalzar, M., et al. Niche and host-associated functional signatures of the root surface microbiome. Nature Commununications. 5, 4950 (2014).
- Niu, B., Paulson, J. N., Zheng, X., Kolter, R. Simplified and representative bacterial community of maize roots. Proceedings of the National Academy of Science of the United States of America. 114 (12), E2450-E2459 (2017).
- Fitzpatrick, C. R., et al. Assembly and ecological function of the root microbiome across angiosperm plant species. Proceedings of the National Academy of Science of the United States of America. 115 (6), E1157-E1165 (2018).
- Ramirez, K. S., Lauber, C. L., Knight, R., Bradford, M. A., Fierer, N. Consistent effects of nitrogen fertilization on soil bacterial communities in contrasting systems. Ecology. 91 (12), 3463-3470 (2010).
- Paungfoo-Lonhienne, C., et al. Turning the table: Plants consume microbes as a source of nutrients. PLoS One. 5 (7), e11915 (2010).
- Yeoh, Y. K., et al. The core root microbiome of sugarcanes cultivated under varying nitrogen fertilizer application. Environmental Microbiology. 18 (5), 1338-1351 (2016).
- Bulgarelli, D., Schlaeppi, K., Spaepen, S., Ver Loren van Themaat, E., Schulze-Lefert, P. Structure and functions of the bacterial microbiota of plants. Annual Review of Plant Biology. 64, 807-838 (2013).
- Peiffer, J. A., et al. Diversity and heritability of the maize rhizosphere microbiome under field conditions. Proceedings of the National Academy of Science of the United States of America. 110 (16), 6548-6553 (2013).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены