Method Article
基于寡核苷酸的串联 RNA 分离法恢复真核 mRNA-蛋白复合物
摘要
本文介绍了一种用于恢复内在形成的 mRNA 蛋白复合物的串联 RNA 分离过程 (跳闸)。具体来说, rna 蛋白复合物在体内交联, polyadenylated rna 与寡核苷酸 (dT) 珠提取物分离, 特殊基因用修饰 RNA 反义寡核苷酸捕获。通过应用免疫印迹分析, 检测到基因的蛋白质。
摘要
RNA 结合蛋白 (限制性商业惯例) 在转录后控制基因表达中起着关键作用。因此, 基因蛋白复合物的生化特性对于理解由相互作用的蛋白质或非编码 rna 推断的 mrna 调节是必不可少的。在这里, 我们描述了串联 RNA 分离程序 (旅行), 使纯化内在形成的 mRNA 蛋白复合物从细胞提取物。两步协议包括 polyadenylated 基因与反义寡核苷酸 (dT) 珠的分离和随后捕获的 mRNA 的兴趣与 3 '-生物素化 2 '-O 甲基化反义 RNA 寡核苷酸, 然后可以隔离链亲和素珠。从酵母、线虫和人体细胞中提取体内交联 mRNA-ribonucleoprotein (mRNP) 配合物, 进一步进行 RNA 和蛋白质分析。因此, 旅行是一个多才多艺的方法, 可以适应所有类型的 polyadenylated rna 跨有机体, 以研究动态重新安排 mRNPs 强加的细胞内或环境的线索。
引言
转录后基因调控是通过 RNA 结合蛋白 (限制性商业)、非编码 rna (ncRNAs) 和基因的相互作用驱动的, 它直接影响细胞内每个转录的处理、定位、翻译和衰变1,2. 因此, 确定与特定基因相互作用的限制性商业惯例和 ncRNA, 建立所谓的 ribonucleoprotein 复合物/微粒 (RNPs), 因此是了解基因和基因表达控制的命运的关键。对 RNP 配合物的生化特性进行补充的方法3,4: "以蛋白质为中心" 的方法是以纯化特定的限制性商业惯例为基础的, "以 RNA 为中心" 的方法涉及分离亚群或个体 rna 和随后分析相互作用的蛋白质或 rna。最近, 以 RNA 为中心的方法, 以识别与 polyadenylated (聚 (A)) rna5相互作用的限制性蛋白汇, 已变得越来越流行, 通过纳入紫外线 (UV) 光交联步骤, 以稳定蛋白质 RNA基因隔离之前的相互作用, 这极大地扩大了在人类细胞6,7,8,9,10,11的限制性商业惯例目录,秀丽线虫12、酿酒酵母12、13、14等生物体15、16、17、18、 19,20。然而, 蛋白质和/或 ncRNAs 在特定成绩单上的映射仍然是一个巨大的挑战。为此, 目前使用了两种主要方法: 一方面, rna 核酸适体标签被融合到感兴趣的 rna, 使亲和纯化。因此, RNA 适配子绑定与氨基糖苷类抗生素的高度亲和性, 包括妥布霉素和链霉素21,22,23,24, 或蛋白质, 如涂层蛋白从 R17/MS2 噬菌体或细菌链亲和素 S125,26,27,28,29。虽然这种方法被证明是相对健壮和多才多艺的, 它需要克隆设计的 RNA 正在调查中, 因此不能用来捕捉自然基因。另一方面, 反义寡核苷酸 (ASOs) 早期用于恢复和鉴定高表达的原生 RNPs30,31和病毒 rna32。最近, ASOs 被应用于捕获长的非编码 rna33,34,35和体外形成 mRNPs36。以 rna 为中心的方法的一个主要限制因素是兴趣 RNA 的拷贝数字, 使低表达的基因的恢复更加疑难。这种限制可以通过推广反应来克服;但是, 这可能会导致增加背景。
在这里, 我们描述了我们最近开发的基于麻生太郎的串联 RNA 分离程序 (旅行), 以分离高选择性37 (图 1) 的本地 mRNA 蛋白复合物.该协议涉及两个连续的基于麻生太郎的净化步骤, 即用商业上可用的寡聚 (dT) 耦合珠 (基因) 的隔离, 然后用专门设计的短生物素化 2 '-甲氧基捕获特定的基因。RNA ASOs。这两个步骤的过程有助于消除污染的蛋白质, 并增加了调整和优化的机会。在这种情况下, 从酵母, 线虫和人类细胞的选择基因, 以确认在体内形成的 mRNA 蛋白复合物的旅行。
研究方案
1. 设计反义寡核苷酸 (ASOs)
- 使用可用的在线工具38分析 mRNA 的二级结构或其片段。因此, 在空框中输入核苷酸 (nt) 序列。在 "基本选项" 框中, 选择 "最小自由能" (MFE) 和分区函数, 并避免孤立的基对。在 "输出选项" 框中, 选择 "交互式 RNA 次级结构图", 最后单击 "继续" 按钮。将弹出一个新窗口, 显示感兴趣的 mRNA 的二级结构。
- 在感兴趣的 mRNA 中选择至少三个不同的 21-24 个长序列, 优先于缺乏可检测的二级结构的区域 (e., 非结构化循环) 和 3 ' 未翻译区域 (UTR)。
注: 已观察到, 在 3 ' 未翻译的区域 (UTR) 中, ASOs 退火对序列的执行情况最好。一种可能性是, 核糖体绑定到编码序列 (CDS) 有时可能会阻碍 ASOs 的退火。ASOs 退火到 5 ' UTRs 未被测试。- 选择胍/胞嘧啶比接近50% 的区域, 缺乏核苷酸串联重复, 以避免可能形成发夹或自退火。
- 手工设计 2 '-甲氧基修饰 RNA 寡核苷酸在 3 ', 完全互补的选择区域 (步骤 1.2) 在所需的目标 mRNA。
注: 2 '-甲氧基修饰使 RNA 对细胞 RNases 的抗性, 并增加双相熔化温度, 这允许严格清洗。生物素基团是捕获 ASOs 与链亲和素所必需的。- 调整 RNA 混合体的熔化温度为 ~ 60-65 °c, 并确保它具有较高的语言序列复杂度 (> 60%), 确定与适当的在线工具39。
- 使用基本的本地对齐搜索工具40 , 以搜索与转录中的其他基因的潜在麻生太郎交叉杂交。选择核苷酸爆炸, 在空框中插入序列并选择感兴趣的有机体。将其余参数保留为默认值, 然后单击 "爆炸"。
注意: 即使是部分连续对8-10 位基因可以导致交叉杂交和恢复这一 mRNA。
2. 细胞培养、紫外线照射和细胞裂解
注: 以下为酵母菌、线虫、人胚肾细胞(HEK293) 的萌发过程。然而, 它可以适应其他有机体以及, 虽然它尚未明确测试。
- 美国酿酒酵母
- 培养酵母细胞 (e., 菌株 BY4743 衍生物;马/α his3Δ1/his3Δ1 leu2Δ0/leu2Δ0 LYS2/lys2Δ0 met15Δ0/MET15 ura3Δ0/ura3Δ0 PFK2:TAP/PFK2) 在500毫升 YPD 培养基 (1% 酵母萃取物, 2% 蛋白胨, 2% d-葡萄糖) 在30°c 与恒定的震动在 220 rpm。
- 通过使用0.45 µm 尼龙过滤器或在室温 (RT) 的 3000 x g 离心, 在中测井阶段收集细胞 (光学密度在 600 nm (OD)600 ~ 0.6)。分别丢弃流动或上清。
- 用25毫升磷酸盐缓冲盐水 (PBS) 用0.45 µm 尼龙过滤器清洗细胞三次, 或通过离心在 3000 x g 处收集, 以在 RT 上提取2分钟, 然后丢弃上清。尽快收集细胞, 因为施加的压力可能会影响 RNA 蛋白的相互作用。
- 并用重悬25毫升的 PBS 中的细胞, 然后将悬浮液倒入15厘米培养皿中。将盘子放在冰上, 取下盖子, 用400兆焦耳-2的 254 nm 紫外光在紫外交联剂中暴露细胞三次, 在每一个接触周期之间用温和的混合进行2分钟的休息。
- 将细胞转移到50毫升管, 通过离心在 3000 x g 处收集细胞, 在4摄氏度时进行3分钟。扔掉上清, 保持小球。
注: 细胞可以在液氮中凝固, 储存在-80 摄氏度。 - 并用重悬4毫升的冷冻裂解缓冲液中的细胞 a (磅 a; 100 毫米三盐酸, pH 值 7.5, 500 毫米 LiCl, 10 毫米 EDTA, 1% 海卫 X-100, 5 毫米, 20 u 毫升-1 DNase I, 100 u 毫升-1 RNasin, 完全无 EDTA 蛋白酶抑制剂鸡尾酒)。
- 将细胞转化为两个2毫升的管子。添加最大值。⅔体积的冰玻璃珠和扰乱组织 lyser 的细胞在30赫兹10分钟, 在4摄氏度。
- 用热针在管子底部打一个洞, 在三十年代将裂解液转移到新鲜的1.5 毫升管上, 离心 600 x 克。
- 清除裂解液由三顺序 centrifugations 在4°c 在 3000 x g 为3分钟, 然后 5000 x g 和 1万 x g 为5分钟每个。
注: 提取物可在液氮中凝固, 储存在-80 摄氏度。
- C. 线虫
- 养殖布里斯托尔 N2 蠕虫在20°c 在线虫生长培养基 (NGM) 板材 (0.3% 氯化钠, 1.7% 琼脂, 0.25% 蛋白胨, 1 毫米 CaCl2, 5 µg/毫升胆固醇, 1 毫米 MgSO4, 25 毫米 KPO4缓冲, pH 6.0) 种子与 OP50 大肠杆菌菌株41.
- 添加5毫升的 M9 缓冲41 (0.3% 的 HPO2PO4, 0.6% Na24, 0.5% 氯化钠, 1 毫米 MgSO4) 到每个板块, 摇板并用重悬蠕虫和转移蠕虫到15毫升管。放置2µL 悬浮在幻灯片和计数蠕虫在悬浮与显微镜。然后应用因子计算每片蠕虫的数量。
- 在室温下离心 (400 x 克, 2 分钟, RT) 收集12万只蠕虫, 并丢弃上清液。
- 把虫子洗三次。因此, 添加10毫升的 M9 缓冲器, 混合反转和收集蠕虫通过离心在 400 x g 2 分钟的 RT。
- 将15毫升的 M9 缓冲液添加到蠕虫和旋转轮上, 在 RT 上放置15分钟。
- 将蠕虫转移到 NGM 板 (每板4000蠕虫), 并在紫外交联剂中暴露在 300 mJ cm-2的紫外光 (254 nm) 上。
- 将5毫升的 M9 缓冲器直接添加到板上, 并摇动板以并用重悬缓冲区中的蠕虫。用吸管将悬浮液转移到15毫升管上, 通过离心在 400 x g 上以2分钟的 RT 收集蠕虫。
- 并用重悬2毫升裂解缓冲 b 中的蠕虫 (LB b; 100 毫米三盐酸, pH 值 8.0, 150 毫米氯化钠, 1 毫米 EDTA, 0.75% IGEPAL, 1 毫米, 20 u 毫升-1 DNase I, 100 u 毫升-1 RNasin, 完全无 EDTA 蛋白酶抑制剂鸡尾酒)。
- 在装满液氮的砂浆中研磨蠕虫。在50毫升管中收集粉末, 在 RT 中解冻。
注: 液氮必须按照安全规程处理 (如油烟机和安全手套) - 清除裂解液, 包括脂肪层离心在 1.4万 x 克10分钟, 然后通过一个0.45 微米的过滤器与注射器通过澄清裂解。
- 人类培养细胞
注: 以下描述是基于 HEK293 细胞的瞬态转染 pGL3-CDKN1B-3'UTR 报告质粒, 表达 3′UTR CDKN1B/27 下游的萤火虫荧光素酶基因42。- 培养 HEK293 细胞在 Dulbecco 的改良鹰的培养基 (DMEM) 含有25毫米葡萄糖和1毫米丙酮酸钠, 补充 100 U 毫升-1青霉素, 100 µg 毫升-1链霉素和10% 胎牛血清 (血清), 并孵化在37°c 在加湿室 (孵化器) 含 5% CO2。
- 种子 ~ 3×106 HEK293 细胞在标准的 10 cm 组织培养盘在转染之前的前一天。用 hemocytometer 计数单元格。
- 混合2µg 的报告基因 (e., pGL3-p27-3'UTR) 与20µL 的转染试剂和染细胞70% 融合 (~ 7×106细胞)。
- 在收割前将细胞放置在37摄氏度的孵化器中, 以进一步48小时。
- 用血清吸管取出培养基, 用10毫升的 PBS 在37摄氏度前预热, 快速冲洗两次细胞。用血清学吸管移除 PBS。
- 将6毫升的 PBS 添加到盘中, 放置在冰上, 然后将细胞暴露在100兆焦耳厘米-2的紫外交联剂中, 以紫外线 (254 nm) 照射。
注意: 在紫外线照射的时候, 盘子必须放在冰上。 - 在 PBS 中刮掉细胞 (总共 107细胞), 并转移到15毫升管。
- 向下旋转 250 x g 的细胞10分钟, 在4摄氏度, 然后用吸管取出上清液。
注: 细胞颗粒可在液氮中凝固, 储存在-80 摄氏度, 直至使用。 - 并用重悬2毫升的预冷裂解缓冲液中的细胞 (LB A) 通过吹打上下5-6 次, 同时保持管在冰上。
- 用吸管将裂解液转移到放置在冰上的5毫升管上。
- 主题裂解到三回合超声波包括二十年代爆发在10微米振幅与三十年代冷却期间在冰。
注意: 超声波是建议, 因为它完成细胞的裂解和碎片的 DNA。 - 将裂解液转移到2毫升管和离心机在 1.5万 x g 10 分钟在4摄氏度。收集上清 (= 提取物) 和转移到一个新的管。去除分析样品 (5-10%), 进一步蛋白质和 RNA 分析。
注意: 这一步对于删除任何剩余的单元碎片是至关重要的, 可以重复。
3. 第一步: 聚 (A) RNA 分离
- 平衡1毫克的寡聚 (dT)25耦合磁性珠在500µL 各自溶解缓冲。
- 将5毫克, 10 毫克或4毫克 (蛋白质) 的酵母, C. 线虫或 HEK293 提取物, 分别与1毫克的寡核苷酸 (dT)25耦合磁性珠。在搅拌机中, 将样品搅拌10分钟, 25 摄氏度。
注: 以牛血清白蛋白 (BSA) 为参考标准, 可测定提取物的蛋白质浓度。剧烈晃动的样品防止了珠子沉淀。为了评价 mRNA 分离的特异性, 通过添加过量 (20 µg) 的 polyadenylic 酸 (pA), 可以并行进行控制实验。 - 把管子放在十年代的磁性支架上, 取出上清液。将上清液保持在冰上以供后续几轮恢复 (步骤 3.7)。
- 加入500µL 洗涤缓冲器 A (10 毫米三盐酸, pH 7.5, 600 毫米 LiCl, 1 毫米 EDTA, 0.1% 海卫 X-100) 到珠和漩涡为 5 s。
注意: LiCl 在洗涤缓冲器中的浓度会有所不同。600毫米 LiCl 建议, 但较低浓度也测试 (500 毫米为C. 线虫, 300 毫米为 HEK293)。 - 用磁铁收集珠子, 然后用500µL 洗涤缓冲 B (世界银行; 10 毫米三盐酸, pH 7.5, 600 毫米 LiCl, 1 毫米 EDTA) 洗涤珠子。
- 洗脱 RNA 在30µL 10 毫米三 HCl, pH 7.5 在80°c 为2分钟以连续的震动 (1000 rpm) 在搅拌机。立即将管放在磁性支架上, 并在十年代后收集洗脱液. 保存珠子以获得更多的净化。
注: 尽可能快地收集洗脱液, 以防止在较低温度下可能重新绑定基因到珠子上。 - 将前一步的珠子添加到步骤3.3 所收集的上清液中, 重复基因的捕获、洗涤和洗脱两次 (步骤 3.3-3.6)。eluates 从重复的回合然后被结合并且可以被存放在-80 °c。
4. 第二步: 用 3 '-生物素化 2 '-甲氧基修饰的反义 RNA 寡核苷酸捕获特定基因
注意: 为了测试 ASOs 的适用性, 可以使用从细胞分离的总 rna 进行以下过程。
- 平衡30µL 的链亲和素耦合磁珠1毫升的装订和洗涤缓冲器 (B & W 缓冲器; 10 毫米三盐酸, ph 值 7.5, 150 毫米氯化钠, 0.5 毫米 EDTA, ph 8.0), 含0.1 毫克 mL-1大肠杆菌转移 RNA (tRNA) 在一个转子上为 1 h 在Rt。
- 用750µL & W 缓冲器清洗珠子三次。
- 并用重悬在30µL & W 缓冲的珠子, 并保持在冰上, 直到使用。
- 稀释 ~ 35 µg 的总蛋白从以前的聚 (A) 隔离 (见 3) 在100µL 的 B & W 缓冲器在1.5 毫升管。
注: 对 ASOs 总 rna 的特异性进行测试, 将纯化总 rna 的 600, 添加到100µL 的 B & W 缓冲器中。 - 添加 200 pmol 的各自麻生太郎和孵化在70°c 5 分钟。
注: 高温可消除 RNA 中的二级结构, 促进了麻生太郎的退火。 - 从设备上卸下整个散热块, 并将其放在 RT 上10分钟, 慢慢降温。
- 添加30µL 平衡链亲和素耦合磁性珠从步骤4.1 到样品。
- 在25摄氏度孵育混合物30分钟, 在搅拌机中以950转每分钟恒定晃动。
注: 建议每隔10分钟轻拍管以防止珠子沉淀。 - 将管子放在磁支架上, 取出上清, 用750µL 预热的 B & W 缓冲器在55摄氏度上清洗珠三次。
注: 最佳洗涤温度可能略有差异/不同 ASOs 之间的差异。建议用非交联的总 RNA 在细胞提取物进行实验前, 对该条件进行调整。 - 洗脱 RNA 在20µL 10 毫米三 HCl, pH 7.5 在90°c 为10分钟以恒定的震动在 950 rpm 在搅拌机.将管子放在磁性支架上, 立即收集洗脱液。
注: 蛋白质分析, 添加20µL 的 1× Laemmli 缓冲珠和孵化在95摄氏度5分钟. 通过标准实验室协议应用免疫印迹分析, 对蛋白质进行监测。
结果
我们开发了一个基于麻生太郎的 RNA 分离策略, 称为行程, 以捕获特定的基因与他们的绑定蛋白质从三种不同的有机体37。从根本上说, RNA 蛋白复合物在体内通过紫外线照射的细胞在 254 nm, 和聚 (A) rna 恢复与商业可用的寡核苷酸 (dT) 耦合磁性珠, 然后的 mRNA 的兴趣被隔离3 '-生物素化 2-0 '-甲氧基修饰的反义 RNA 寡核苷酸 (图 1)。因此, 我们设计了21-24 种改进 ASOs, 完全互补的区域在选定的基因从酵母, C. 线虫和人类, 并测试他们是否适合恢复的 mRNA 的兴趣 (一份引物和 ASOs 列表中给出表1). 首先用非交联的总 RNA, 从各自的生物体中分离出单个 ASOs 的效率和特异性。在这些实验中, rna ASOs 被耦合到链亲和素共轭顺磁珠和孵化与非交联总 RNA 制备的相应的有机体。在从珠子中释放捕获的基因后, 通过反向转录 (RT)-聚合酶链反应 (PCR)37对 mRNA 靶点的存在以及不相关的控制基因进行监测。我们注意到, 两个变量, 盐浓度和洗涤缓冲温度, 发挥了关键作用, 从酵母PFK2 mRNA 捕获的效率, 测试了三不同的 ASOs (图 2A)。将盐 (NaCl) 浓度降低到25毫米, 将阴性对照 mrna (PFK1) 的回收率降低到所有 ASOs, 但同时也减少了所需的目标 mRNA PFK2 (10-15% 的输入) 的恢复。反之, 盐浓度增加到生理水平 (150 毫米 NaCl) 增加了PFK2基因的回收率高达 75%, 与麻生太郎 PFK2-2, 超过控制PFK1 mRNA 至少5倍 (图 2A)。进一步说明, 不同 ASOs 在生理盐浓度上表现出显著的 mRNA 靶向俘获效应, 强调了 ASOs 的经验验证的必要性.cep-1 和酵母RPS20基因, 使用各自的 ASOs (表 1), 说明了 mRNA 恢复对洗涤缓冲器温度的依赖性.我们观察到, 最佳洗涤温度介于50摄氏度和55摄氏度之间, 从低背景与不相关的基因和基因目标的有效恢复 (图 2B) 可见。在这一点上, 我们希望强调 ASOs 与其他基因交叉杂交的可能性。例如, DNM1 是完全互补的序列内的DNM1编码序列, 但它也部分退火与ACT1 mRNA。DNM1 ASOs 恢复两基因, 无论洗涤温度, 显示强烈的交叉杂交倾向 (图 2C)。最后, 我们使用以上描述的测试和优化选择三 ASOs, 适合于从基因中分离出的 RNA、线虫和人类细胞中的总核糖核酸来恢复各自的靶向性 (图2D).
在体外初步选择合适的 ASOs 后, 我们进行了从紫外交联生物体/细胞提取的细胞萃取物的旅行 (图 1)。具体地说, 我们测试了从三种不同的有机体中恢复三种不同的基因: 酵母PFK2 mRNA , cep-1线虫,和一个记者 mRNA (pGL3-CDKN1B-3'UTR) 轴承 3 ' UTR人CDKN1B/p27 mRNA 序列融合到一个荧光素酶 (卢克) 记者的 HEK293 细胞瞬时表达。为了控制 RNA 分离的特异性, 我们监测了几个不相关的基因的恢复, 我们进行了竞争实验, 增加了过量的 pA 细胞提取物, 这与细胞基因的结合, 以寡核苷酸 (dT)25珠在第一步净化步骤。如前所见与非交联总 RNA 样品 (图 2), rt-pcr 证实了各自基因目标 mRNA 的丰富在旅行期间, 而一些非相关的控制基因没有被丰富 (图 3A)。此外, 没有 ASOs 的珠子上都没有检测到基因, 这表明了避免不特异绑定的适当的阻止程序。在这条线上, 不相关的/被炒的 ASOs 也可以作为控制, 虽然有潜在的交叉杂交与某些基因和推断捕获的束缚蛋白, 然后要考虑到。由于最终洗脱液中的蛋白质几乎无法在银色染色的聚丙烯酰胺凝胶上可视化 (数据未显示), 所以通过应用免疫印迹分析进一步评估了先前已知的 mRNA 相互作用蛋白的存在。这包括 Pfk2p 从S. 酵母, 有选择性地结合在核糖体独立方式的PFK2 mRNA12;线虫GLD-1, 一种规范的限制性标准, 结合 3 ' UTR 序列的cep-1 mRNA 为平移规则43;和宾虚, 一个规范 mrna 稳定性和翻译的p27/CDKN1B mrna44。正如预期的那样, 所有这些蛋白都是在基因的旅行 eluates 中发现的, 这是由西方印迹分析 (图 3B)。
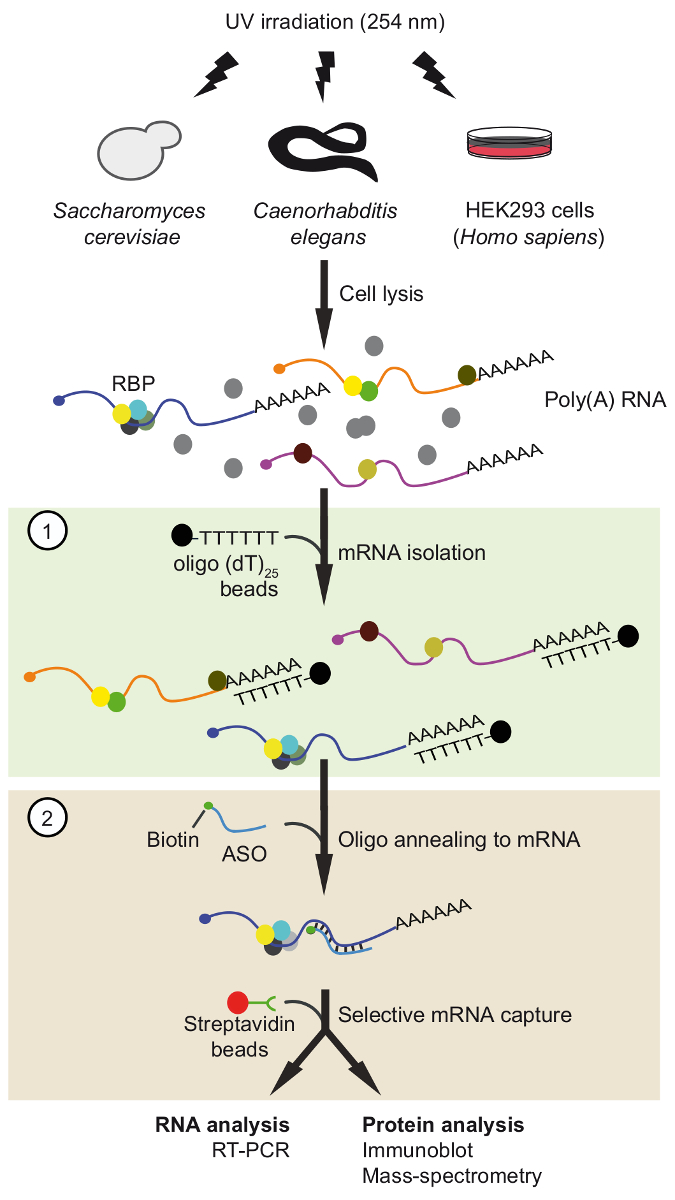
图1。行程示意图表示.蛋白质通过紫外线照射在体内与 RNA 交联。在第一步 (光绿盒), 聚 (A) RNA 蛋白复合物被回收与寡核苷酸 (dT)25珠应用严格的洗涤条件, 以消除未绑定的蛋白质。在第二步 (粉红色盒), 目标 mRNP 是拉出与生物素化反义 RNA 寡核苷酸和链亲和素珠。然后用 rt-pcr 和应用免疫印迹/质谱 (MS) 对纯化 mRNPs 进行分析, 以确定 rna 和蛋白质与感兴趣的 mRNA 相互作用。该数字是从以前的出版物37修改, 并获得许可。请单击此处查看此图的较大版本.
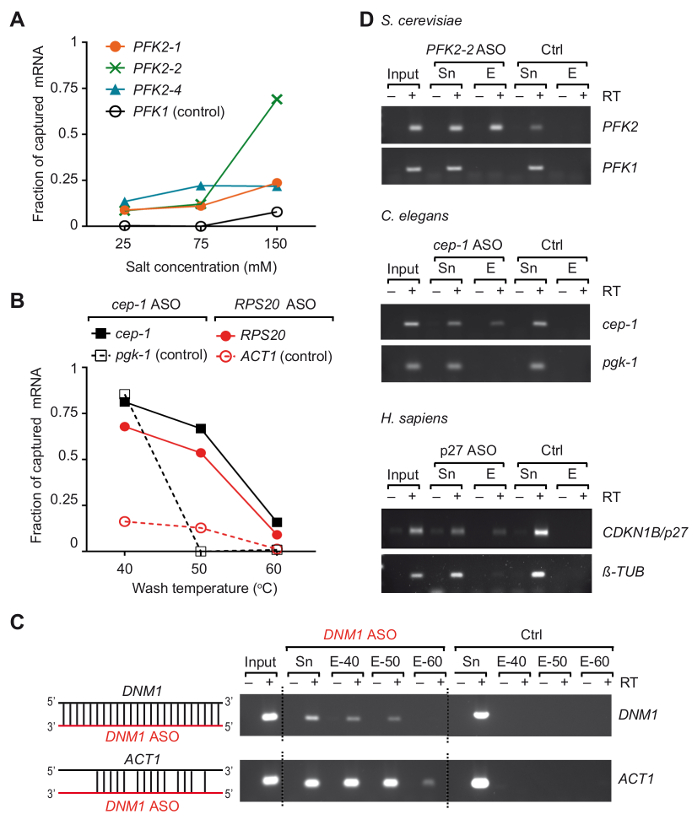
图2。用改进的反义 RNA 捕获探针从非交联细胞/生物体的总 RNA 中分离选定的基因.(A) 盐浓度对 ASOs 酵母PFK2 mRNA 捕获效率的影响。酵母细胞中的总 RNA 与所示的 ASOs 结合, 用含有指定盐 (NaCl) 浓度的缓冲液洗涤55摄氏度。绿色, 蓝色和橙色线代表PFK2 mRNA 恢复与不同的 ASOs 由 RT PCR37: PFK2-1和PFK2-2 退火在 3 ' UTR, PFK2-4在 CDS。PFK1是阴性对照 mRNA。PCR 的执行与32的放大周期和量化如前所述37。(B) eleganscep-1和酵母RPS20基因在不同洗涤温度下的分数, 分别以黑色和红色线表示。eleganspgk-1和酵母ACT1是非靶 (对照) 基因。30和 32 PCR 周期分别用于检测酵母和基因线虫。(C) DNM1 (红色) 与DNM1 mrna 中的序列杂交的示意图, 以及与ACT1 mrna 的潜在交叉杂交, 左侧显示。一种琼脂糖凝胶, 显示的产品从 RT PCR 反应 (30 周期) 检测酵母DNM1和ACT1基因洗脱从珠子被显示在右侧。输入, 总 RNA;锡, 上清后与 ASOs 孵化;E、eluates 从被洗涤的珠在指示的温度之前洗脱 (40 °c, 50 °c 和60°c)。对照实验 (Ctrl) 并行执行, 不添加麻生太郎。(D)琼脂糖凝胶, 显示 rt-pcr 产品检测基因 (右) 从酵母的总 RNA, C. 线虫, 和人类 HEK293 细胞 (智人) 捕获. 输入, 总 RNA;锡, 上清;E, eluates 从珠子上出来。PCR 是由30个放大周期的酵母基因, 32 周期为C. 线虫基因, 28 和30周期为人类蛋白和p27基因, 分别。该数字是从以前的出版物37修改, 并获得许可。请单击此处查看此图的较大版本.
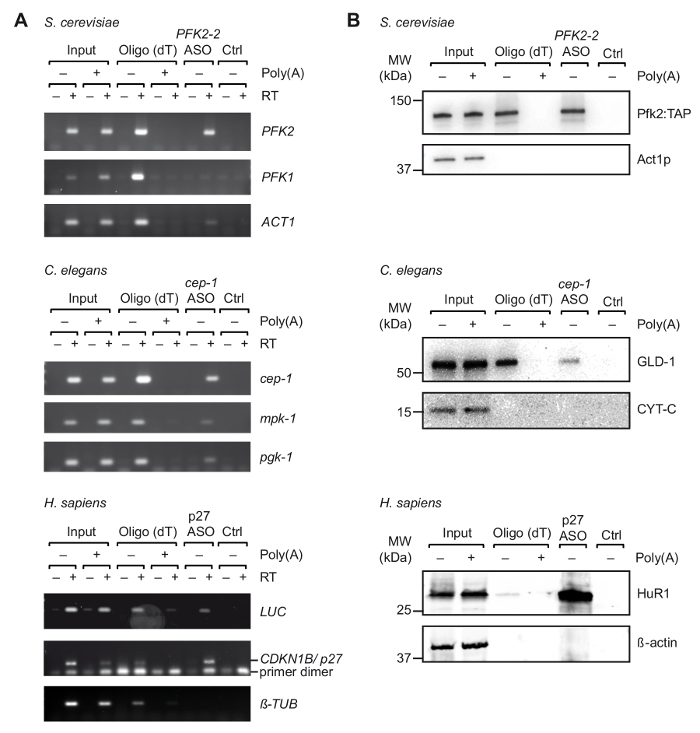
图3。从紫外交联细胞中提取的特定 mRNA-蛋白复合物进行旅行.(A) 琼脂糖凝胶用于检测基因 (右) 与 rt-pcr (dT) 捕获, 并表明 ASOs 从S. 酵母, C. 线虫和人类 (智人) 细胞提取物。输入, 总 RNA 从交联的细胞或有机体;Ctrl, 控制没有麻生太郎。聚 (a), 与聚 (a) 的竞争。采用 rt-pcr 的方法进行了37 , 35 个放大周期为卢克, 32 个周期为p27, 29 个周期为蛋白。(B) 应用免疫印迹分析具有指示抗体的 mRNA 结合蛋白 (右)。加载分数如下: 0.1%, 2.5% 和1% 的酵母, 线虫和人类投入;10%、10% 和5% 用于酵母、线虫和人寡糖 (dT) 车道;和 66% ASOs 车道。分子量 (兆瓦) 在 kilodaltons (kDa) 中标明。图以权限37重新发布。请单击此处查看此图的较大版本.
| 底漆名称 | 序列 | 目标 | 大小 | |
| Pfk2_Fwd | GTGTTAAGGGTTCACATGTCG | PFK2 的酵母 | 133 bp | |
| Pfk2_Rev | CTTCCAACCAAATGGTCAGC | PFK2 的酵母 | 133 bp | |
| Pfk1_Fwd | GGTGATTCTCCAGGTATGAATG | PFK1 的酵母 | 97 bp | |
| Pfk1_Rev | CTTCGTAACCTTCGTAAACAGC | PFK1 的酵母 | 97 bp | |
| Act1_Fwd | GTCTGGATTGGTGGTTCTATC | ACT1 的酵母 | 85 bp | |
| Act1_Rev | GGACCACTTTCGTCGTATTC | ACT1 的酵母 | 85 bp | |
| Dnm1_Fwd | CTGTGTTCGATGCATCAGAC | DNM1 的酵母 | 156 bp | |
| Dnm1_Rev | CGCACTCCAATTCTTCTCTC | DNM1 的酵母 | 156 bp | |
| Rps20_Fwd | CGCTGAACAACACAACTTGG | RPS20 的酵母 | 228 bp | |
| Rps20_Rev | GGAAGCAACAACAACTTCGAC | RPS20 的酵母 | 228 bp | |
| Cep1_Fwd | CGATGAAGAGAAGTCGCTGT | cep-1 线虫 | 110 bp | |
| Cep1_Rev | ATCTGGGAACTTTTGCTTCG | cep-1 线虫 | 110 bp | |
| Pgk1_Fwd | GCGATATTTATGTCAATGATGCTTTC | pgk-1 线虫 | 74 bp | |
| Pgk1_Rev | TGAGTGCTCGACTCCAACCA | pgk-1 线虫 | 74 bp | |
| Mpk1_Fwd | TGCTCAGTAATCGGCCATTG | mpk-1 线虫 | 74 bp | |
| Mpk1_Rev | TCCAACAACTGCCAAAATCAAA | mpk-1 线虫 | 74 bp | |
| p27_Fwd | TTTAAAAATACATATCGCTGACTTCATGG | p27 智人 | 212 bp | |
| p27_Rev | CAAAGTTTATGTGCTACATAAAAGGTAAAAA | p27 智人 | 212 bp | |
| Luc_Fwd | AATGGCTCATATCGCTCCTGGAT | 荧光素酶pγralis | 117 bp | |
| Luc_Rev | TGGACGATGGCCTTGATCTTGTCT | 荧光素酶pγralis | 117 bp | |
| β-TUBULIN_Fwd | CTGAACCACCTTGTCTCAGC | β-蛋白智人 | 136 bp | |
| β-TUBULIN_Rev | AGCCAGGCATAAAGAAATGG | β-蛋白智人 | 136 bp | |
| PFK2-1 麻生太郎 | AAUAGAAAGUGUAAUAAAAGGUCAU | 3 ' UTR PFK2 的酵母 | - | |
| PFK2-2 麻生太郎 | GUUUCAUGGGGUAGUACUUGU | 3 ' UTR PFK2 的酵母 | - | |
| PFK2-4 麻生太郎 | CUUGAAGAGGAGCGUUCAUA | PFK2 酵母 | - | |
| DNM1麻生 | UCGGUCAGUGGAGGUUCAGCGUUU | DNM1 酵母 | - | |
| RPS20麻生 | GUCGGUAAUAGCCUUCUCAUUCUUG | RPS20 酵母 | - | |
| cep-1麻生 | GUGAGAAAUGCGGUGCUUUGAAA | 3 ' UTR cep-1 线虫 | - | |
| p27麻生 | UCAUACCCCGCUCCACGUCAGUU | 3 ' UTR p27 智人 | - | |
表1。寡核苷酸序列.在这项工作中使用的 PCR 引物和 ASOs 的列表, 底漆序列, 基因靶和预期片段大小扩增后。
讨论
旅行允许用生化手段在体内对特定基因的蛋白质进行分析。虽然我们使用该方法来验证特定限制性商业惯例与不同生物体/细胞的 mRNA 目标之间的相互作用, 但也适用于研究其他类型的聚 (A) rna, 如细胞质长 ncRNAs。此外, 系统分析的束缚蛋白和/或 rna 与 MS 或 RNA 排序可以通过一个向上扩展的程序。在这方面, 我们的初步数据表明, 1 L 的酵母培养和至少1亿的人类 HEK293 细胞可以提供足够的起始材料, 以获得可靠的 MS 数据的良好表达的基因 (未发表的结果)。
所描述的行程协议包括辐照的细胞与紫外光在 254 nm, 以交联 RNA 蛋白相互作用。这使得在净化过程中实施严格的洗涤条件。然而, 虽然没有明确的测试, 我们希望注意到交联是可选的, 积极的 RNPs 也可以恢复与生理缓冲。然而, 在这种情况下, 应该考虑到在裂解物目标 RNA 上的蛋白质和 rna 的潜在重新排列。反之, 如果采用 UV 或其他交联程序 (如甲醛), rna 的完整性应该被评估为 rna 退化可能导致减少 mRNPs 的恢复。此外, UV 交联是相当低效 (~ 5%), 甚至更少的蛋白质相互作用的双链 RNA, 这可能会引入偏见, 以检测某些类别的限制性商业惯例12。最后, 紫外线照射还可以诱发蛋白质蛋白和蛋白质-DNA crosslinks, 应考虑数据解释45,46。因此, 替代的交联程序, 例如甲醛, 也可能是特别感兴趣的捕获更大的蛋白质组件在基因。
行程的一个关键特征是两步的基于麻生太郎的纯化程序来恢复特定 rna, 从而增加选择性, 降低共净化污染物的几率。例如, 一个关注的问题是将生物素化 ASOs 直接添加到细胞提取物中, 从而导致生物素结合蛋白的共纯化。这是规避在旅行中实施第一轮纯化聚 (a) rna 使用共价键耦合寡核苷酸 (dT)25磁性珠, 这允许严格的纯化条件, 为 RNA 蛋白 interactome 捕获。此外, 聚 (A) RNA 的选择消除了高丰富的 ncRNAs, 如核糖体 rna 和 tRNAs, 从而降低了样本的复杂性和潜在的交叉杂交和污染的来源。第二步, 用生物素化和修改后的 ASOs 对 mRNA 靶区进行退火, 以恢复特定的基因。另外, 修改后的 ASOs 也可以通过胺连接器直接与磁珠共价键, 减少了捕获生物素结合蛋白的倾向。然而, 根据我们的经验, 聚 (A) RNA 分数与 ASOs 在添加链亲和素珠之前的孵化率比直接添加麻生太郎偶联珠更有效地恢复 mRNPs。可能, 与固定化的寡核苷酸相比, 自由寡核苷酸动力学更有利于获得结构 RNA 中的序列。因此, 在样品孵化前, ASOs 与珠子的共价耦合可能对 RNA 靶的恢复有害, 必须进行实证检验。
基于麻生太郎的方法的一个缺点是某些目标基因的恢复效率低下。因此, 我们建议对几个 ASOs 的评价, 对不同地区的成绩单进行退火。具体来说, 我们建议的设计 2-3 ASOs, 退火的序列在 3 '-UTR 或 cd 的兴趣 mRNA。每一个麻生太郎可能表现出不同的效率, 以恢复各自的 mRNA 目标, 并应进行经验测试 (图 2A, B, 数据没有显示)。最后, 我们发现, ASOs 退火到 3 ' UTRs 的表现更好地与那些退火到 CDS。这可能是由于缺乏翻译核糖体, 而核糖体可能阻碍有效杂交在 CDS 之内, UTRs 的结合点的可及性。我们还经历了多种 ASOs 的结合可能会导致大量非目标基因的污染增加, 降低了下拉效率。在任何情况下, 选择寡核苷酸的选择性可以通过竞争实验 (例如, 增加竞争寡核苷酸) 来控制。
另外一个关注的问题是潜在的交叉杂交与不相关的 rna, 因为我们观察到, 即使部分杂交与其他基因可能导致恢复该 mRNA (图 2C)。为了评估设计的 ASOs 的适用性, 我们建议使用总 rna 进行初始体外检测, 以优化盐浓度和缓冲温度。在我们的手中, 洗涤的链亲和素耦合磁珠在缓冲含有150毫米氯化钠在55°c 表现最好, 但我们建议独立测试的条件, 每一个设计的麻生太郎。值得注意的是, 我们发现从体外实验中选择的所有 ASOs (图 2D) 适合于从交联细胞提取物中捕获各自的 mRNA 靶 (图 3), 进一步证实了在体外检测。除了为 mRNA 捕获设计合适的 ASOs 外, 其他因素也会影响选择性。因此, 与 BSA 和/或 tRNA 根据珠型规格的全面封锁程序, 可以防止不规则绑定珠。为了进一步减少对蛋白质的不特异吸收, 我们还建议使用螺管, 特别是在使用少量样品时。
一般来说, 旅行可以补充以前建立的方法捕捉 rna 与 ASOs。例如, 使用一组长 (90 ) 生物素化 DNA 寡核苷酸, 覆盖整个转录, 将非编码 RNA Xist 与来自200-800 兆紫外交联细胞的核萃取物分离出来。34,35。然而, 选择的平铺方法可能会因与不相关的 rna 交叉杂交的巨大潜力而变得有问题, 而且它不能用于选择特定的转录,例如, 交替拼接的形式。最近, 专门设计的锁定核酸 (低噪声放大剂) 或 DNA 麻生太郎 mixmers 是共价键连接到一个磁性树脂, 以恢复体外转录或高度表达核糖体 rna 从无细胞提取物;然而,在体内形成的 mRNA-蛋白复合物36的恢复中, 未进行试验。鉴于限制性商业惯例和 ncRNA 对转录后基因控制的认识增加, 我们认为, 旅行是一种有效的方法来调查细胞内 RNP 配合物的组成和动力学, 并对胞内和环境线索及其影响在健康和疾病。
披露声明
作者没有什么可透露的。
致谢
我们感谢乔纳森霍尔博士和齐默尔曼 (瑞士联邦) 为PFK2和cep-1 ASOs、Maikel 武泰的设计 p27/CDKN1B ASOs 博士和 Rafal Ciosk (Friedrich 弗雷德里希·米歇尔生物医学研究所,巴塞尔) 为 anti-GLD-1 抗体。这项工作得到了生物技术和生物科学研究理事会 (BB/K009303/1) 和皇家学会沃尔夫森研究优异奖 (WM170036) 的支持 A.P.G。
材料
| Name | Company | Catalog Number | Comments |
| MaxQ 5000 Large Incubated and Refrigerated Orbital Shaker | Thermo Fisher Scientific | SHKE5000-8CE | Floor shaker to grow yeast cells |
| BBD 6220 | Thermo Fisher Scientific | CO2 incubator to grow human cells | |
| Sonicator Soniprep150 | MSE | MSS150.CX4.5 | Sonicator/cell disruptor. Used to shear DNA in human cell lysates |
| Stratalinker 1800 | Stratagene | Stratalinker to expose cells to UV light at 254 nm | |
| Tissue Lyser | Qiagen | RETSCH MM200 | Device to mechanically disrupt yeast cells |
| Refrigerated Centrifuge | Eppendorf | 5810R | Centrifuge to spin down cells, lysates |
| Shaking incubator, Thriller | Peqlab | Thermoshaker for 1.5 mL tubes | |
| Glass beads 0.5mm | Stratech Scientific Limited | 11079105 | Lysis of yeast cells |
| Nylon Filter, 0.41 μm | Millipore | NY4104700 | Collection of yeast cells |
| Millex-HA Filter, 0.45 µm | EMD Millipore | SLHA02510 | Filter to clear nematode lysate |
| Eppendorf LoBind microcentrifuge tubes Protein 1.5 mL | Sigma-Aldrich | 22431081 | Minimise protein loss |
| 2 mL microfuge tube | Ambion | AM12425 | Yeast extract preparation and other applications |
| 5 mL tube | Thermo Fisher Scientific | 129A | Collection of HEK293 cell lysate |
| 15 mL tube | Sarstedt | 62.547.254 | Collection C. elegans |
| 50 mL plastic tube | Sarstedt | 62.554.502 | Collection yeast cells |
| DMEM, high glucose, pyruvate | Thermo Fisher Scientific | 41966 | Media for culturing HEK293 cells |
| Penicillin-Streptomycin | Sigma-Aldrich | P4333 | Used to supplement cell culture media to control bacterial contamination |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F7524 | Used to supplement cell culture media |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| RQ1 RNase-Free DNase | Promega | M6101 | DNAse I to degrade DNA from cell lysate |
| RNasin Plus RNase Inhibitor | Promega | N2611 | RNase inhibitor to avoid RNA degradation |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | Protease inhibitor cocktail to avoid protein degradation |
| Dynabeads Oligo (dT)25 | Thermo Fisher Scientific | 61011 | Magnetic beads required for the first step of mRNA-protein complex purification |
| Dynabeads M-280 Streptavidin | Thermo Fisher Scientific | 11205D | Magnetic beads required for the second step of mRNA-protein complex purification |
| E. coli tRNA | Sigma-Aldrich | 10109550001 | transfer RNA from E. coli used for blocking streptavidin beads and avoid unsepecific interactions |
| Quick Start Bradford 1x Dye Reagent | BioRad | 5000205 | To determine protein concentration within lysates, using BSA as reference |
| Bovine Serum Albumin | Sigma | A7906-100G | Used to perform the standard curve that will be used as reference for protein concentration determination |
| mouse anti-Act1 | MP Biomedicals | 869100 | Antibody for detection of yeast actin in Western blot (1:2,500) |
| mouse anti-Act1 | Sigma | A1978 | Antibody for detection of human actin in Western blot (1:2,000) |
| mouse anti-HuR | Santa Cruz | sc-5261 | Antibody for detection of HuR in Western blot (1:500) |
| mouse anti–CYC-1 | Invitrogen | 456100 | Antibody for detection of Cyc-1 in Western blot (1:1,000) |
| peroxidase anti-peroxidase soluble complex | Sigma | P1291 | Detection of Pfk2:TAP in Western blot (1:5,000) |
| HRP-conjugated sheep anti-mouse IgG | Amersham | NXA931 | HRP-coupled secondary antibody |
参考文献
- Mitchell, S. F., Parker, R. Principles and properties of eukaryotic mRNPs. Molecular Cell. 54 (4), 547-558 (2014).
- Iadevaia, V., Gerber, A. P. Combinatorial Control of mRNA Fates by RNA-Binding Proteins and Non-Coding RNAs. Biomolecules. 5 (4), 2207-2222 (2015).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biology. 15 (1), 203 (2014).
- Matia-González, A. M., Gerber, A. P., Sesma, A., von der Haar, T. Ch. 14. Fungal RNA Biology. , 347-370 (2014).
- Tsvetanova, N. G., Klass, D. M., Salzman, J., Brown, P. O. Proteome-wide search reveals unexpected RNA-binding proteins in Saccharomyces cerevisiae. PLoS One. 5 (9), (2010).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Baltz, A. G., et al. The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts. Molecular Cell. 46 (5), 674-690 (2012).
- Castello, A., et al. System-wide identification of RNA-binding proteins by interactome capture. Nature Protocols. 8 (3), 491-500 (2013).
- Liao, Y., et al. The Cardiomyocyte RNA-Binding Proteome: Links to Intermediary Metabolism and Heart Disease. Cell Reports. 16 (5), 1456-1469 (2016).
- Liepelt, A., et al. Identification of RNA-binding Proteins in Macrophages by Interactome Capture. Molecular Cell Proteomics. 15 (8), 2699-2714 (2016).
- Conrad, T., et al. Serial interactome capture of the human cell nucleus. Nature Communications. 7, 11212 (2016).
- Matia-Gonzalez, A. M., Laing, E. E., Gerber, A. P. Conserved mRNA-binding proteomes in eukaryotic organisms. Nature Structural and Molecular Biology. 22 (12), 1027-1033 (2015).
- Mitchell, S. F., Jain, S., She, M., Parker, R. Global analysis of yeast mRNPs. Nature Structural and Molecular Biology. 20 (1), 127-133 (2013).
- Beckmann, B. M., et al. The RNA-binding proteomes from yeast to man harbour conserved enigmRBPs. Nature Communications. 6, 10127 (2015).
- Wessels, H. H., et al. The mRNA-bound proteome of the early fly embryo. Genome Research. 26 (7), 1000-1009 (2016).
- Sysoev, V. O., et al. Global changes of the RNA-bound proteome during the maternal-to-zygotic transition in Drosophila. Nature Communications. 7, 12128 (2016).
- Despic, V., et al. Dynamic RNA-protein interactions underlie the zebrafish maternal-to-zygotic transition. Genome Research. 27 (7), 1184-1194 (2017).
- Zhang, Z., et al. UV crosslinked mRNA-binding proteins captured from leaf mesophyll protoplasts. Plant Methods. 12, 42 (2016).
- Reichel, M., et al. In Planta Determination of the mRNA-Binding Proteome of Arabidopsis Etiolated Seedlings. Plant Cell. 28 (10), 2435-2452 (2016).
- Koster, T., Marondedze, C., Meyer, K., Staiger, D. RNA-Binding Proteins Revisited - The Emerging Arabidopsis mRNA Interactome. Trends Plant Sciences. 22 (6), 512-526 (2017).
- Hamasaki, K., Killian, J., Cho, J., Rando, R. R. Minimal RNA constructs that specifically bind aminoglycoside antibiotics with high affinities. Biochemistry. 37 (2), 656-663 (1998).
- Bachler, M., Schroeder, R., von Ahsen, U. StreptoTag: a novel method for the isolation of RNA-binding proteins. RNA. 5 (11), 1509-1516 (1999).
- Vazquez-Pianzola, P., Urlaub, H., Rivera-Pomar, R. Proteomic analysis of reaper 5' untranslated region-interacting factors isolated by tobramycin affinity-selection reveals a role for La antigen in reaper mRNA translation. Proteomics. 5 (6), 1645-1655 (2005).
- Hartmuth, K., Vornlocher, H. P., Luhrmann, R. Tobramycin affinity tag purification of spliceosomes. Methods Molecular Biology. 257, 47-64 (2004).
- Beach, D. L., Keene, J. D. Ribotrap : targeted purification of RNA-specific RNPs from cell lysates through immunoaffinity precipitation to identify regulatory proteins and RNAs. Methods Molecular Biology. 419, 69-91 (2008).
- Slobodin, B., Gerst, J. E. A novel mRNA affinity purification technique for the identification of interacting proteins and transcripts in ribonucleoprotein complexes. RNA. 16 (11), 2277-2290 (2010).
- Slobodin, B., Gerst, J. E. RaPID: an aptamer-based mRNA affinity purification technique for the identification of RNA and protein factors present in ribonucleoprotein complexes. Methods Molecular Biology. 714, 387-406 (2011).
- Yoon, J. H., Gorospe, M. Identification of mRNA-Interacting Factors by MS2-TRAP (MS2-Tagged RNA Affinity Purification). Methods Molecular Biology. 1421, 15-22 (2016).
- Leppek, K., Stoecklin, G. An optimized streptavidin-binding RNA aptamer for purification of ribonucleoprotein complexes identifies novel ARE-binding proteins. Nucleic Acids Research. 42 (2), 13 (2014).
- Blencowe, B. J., Sproat, B. S., Ryder, U., Barabino, S., Lamond, A. I. Antisense probing of the human U4/U6 snRNP with biotinylated 2'-OMe RNA oligonucleotides. Cell. 59 (3), 531-539 (1989).
- Lingner, J., Cech, T. R. Purification of telomerase from Euplotes aediculatus: requirement of a primer 3' overhang. Proceedings of the National Academy of Sciences U S A. 93 (20), 10712-10717 (1996).
- Upadhyay, A., Dixit, U., Manvar, D., Chaturvedi, N., Pandey, V. N. Affinity capture and identification of host cell factors associated with hepatitis C virus (+) strand subgenomic RNA. Molecular Cell Proteomics. 12 (6), 1539-1552 (2013).
- Imig, J., et al. miR-CLIP capture of a miRNA targetome uncovers a lincRNA H19-miR-106a interaction. Nature Chemical Biology. 11 (2), 107-114 (2015).
- Chu, C., et al. Systematic discovery of Xist RNA binding proteins. Cell. 161 (2), 404-416 (2015).
- McHugh, C. A., et al. The Xist lncRNA interacts directly with SHARP to silence transcription through HDAC3. Nature. 521 (7551), 232-236 (2015).
- Rogell, B., et al. Specific RNP capture with antisense LNA/DNA mixmers. RNA. 23 (8), 1290-1302 (2017).
- Matia-Gonzalez, A. M., Iadevaia, V., Gerber, A. P. A versatile tandem RNA isolation procedure to capture in vivo formed mRNA-protein complexes. Methods. , 93-100 (2017).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic Acids Research. 36 (Web Server issue), W70-W74 (2008).
- Kalendar, R., Khassenov, B., Ramankulov, Y., Samuilova, O., Ivanov, K. I. FastPCR: An in silico tool for fast primer and probe design and advanced sequence analysis. Genomics. 109 (3-4), 312-319 (2017).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. Journal of Molecular Biology. 215 (3), 403-410 (1990).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Kedde, M., et al. A Pumilio-induced RNA structure switch in p27-3' UTR controls miR-221 and miR-222 accessibility. Nature Cell Biology. 12 (10), 1014-1020 (2010).
- Schumacher, B., et al. Translational repression of C. elegans p53 by GLD-1 regulates DNA damage-induced apoptosis. Cell. 120 (3), 357-368 (2005).
- Ziegeler, G., et al. Embryonic lethal abnormal vision-like HuR-dependent mRNA stability regulates post-transcriptional expression of cyclin-dependent kinase inhibitor p27Kip1. Journal of Biological Chemistry. 285 (20), 15408-15419 (2010).
- Itri, F., et al. Femtosecond UV-laser pulses to unveil protein-protein interactions in living cells. Cellular and Molecular Life Sciences. 73 (3), 637-648 (2016).
- Zhang, L., Zhang, K., Prandl, R., Schoffl, F. Detecting DNA-binding of proteins in vivo by UV-crosslinking and immunoprecipitation. Biochemical and Biophysical Research Communications. 322 (3), 705-711 (2004).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。